Method Article
细胞外基质的Glycoproteomics:使用质谱联用完整糖肽分析的方法
摘要
本文介绍一种方法来准备心血管组织样本进行质谱分析,允许(1)分析的ECM蛋白质组成,(2)识别的糖基化位,和(3)的组分特征的聚糖形式。这种方法可应用于进行微小的改进,在其他组织中的细胞外基质的研究。
摘要
纤维化是许多心血管疾病的标志,并且与细胞外基质(ECM)的加剧分泌和沉积有关。使用蛋白质组学,我们先前已经确定了150多个ECM和心血管组织ECM相关蛋白。值得注意的是,许多ECM的蛋白质糖基化。这种翻译后修饰影响蛋白质折叠,可溶性,结合和降解。我们已经开发了用于ECM蛋白质顺序提取和浓缩方法,其与随后的液相色谱串联质谱法完整糖肽(LC-MS / MS)分析兼容。该策略是基于用NaCl,SDS顺序温育用于组织去细胞化,并为ECM蛋白的增溶胍盐酸盐。在LC-MS / MS的最新进展包括片段化方法,如高能量碰撞解离(HCD)(ETD)的组合和电子转移解离,其允许细胞外基质蛋白的糖肽的直接成分分析。在本论文中,我们描述制备从组织样品的ECM的方法。该方法不仅允许蛋白谱,但也通过MS分析评估和糖基化的表征。
引言
肝纤维化是多种疾病的一个标志。成纤维细胞增殖和向高度合成的表型,其与细胞外基质(ECM)1的加剧分泌和沉积相关区分。过度沉积ECM可以继续,最初损伤已大大减少甚至后,导致功能受损。使用蛋白质组学,我们先前已经确定超过150个ECM和在心脏组织2,3 ECM相关蛋白。他们不仅是结构蛋白,而且蛋白质matricellular和蛋白酶有助于重塑连续和心脏的动态适应。值得注意的是,许多ECM的蛋白质糖基化4。这种翻译后修饰(PTM)包括加入的糖残基的某些氨基酸位置,并影响蛋白质折叠,可溶性,结合和降解5 。
有迹象表明,发生在哺乳动物中两个主要的糖基化类型。 (1)N-糖基化发生在共有序列的Asn-的Xaa-苏氨酸/丝氨酸,其中Xaa是除脯氨酸之外的任何氨基酸内天冬酰胺残(ASN)的甲酰胺基的氮。 (2)在O-糖基化,糖残基连接到丝氨酸和苏氨酸残基(丝氨酸,苏氨酸),或在较小的程度上,羟脯氨酸和羟赖氨酸。而O-糖基化可在多种蛋白质组的发生,N-糖基化被限制为分泌蛋白或膜蛋白5的胞外结构域。这使得N-糖基化有吸引力的目标研究ECM时。
蛋白质组学设置了在疾病蛋白质的变化进行分析的新标准。迄今为止,大多数蛋白质组学研究已集中在细胞内蛋白质6。这主要是由于以下原因。首先,丰富的胞内蛋白妨碍identi稀缺的ECM成分fication。这是在心脏组织,其中线粒体和肌丝蛋白占很大比例的蛋白质含量7的特别重要的。其次,积分ECM蛋白是众多交叉链接和难以溶解。最后,翻译后修饰丰富( 即糖基化)的存在改变了分子质量,电荷,和肽的电泳性能,既影响的分离和通过液相色谱串联质谱法(LC-MS / MS)鉴定。近年来,我们已经制定和完善了ECM的蛋白质顺序提取和富集方法是与随后的质谱(MS)分析兼容。该战略是基于连续孵化。
第一步是用NaCl,促进ECM相关和松散结合的ECM蛋白的提取,以及新合成的细胞外基质蛋白的离子缓冲液中进行。据我小号洗涤剂自由的,非变性的,非破坏性的细胞膜的,并适合用于进一步的生化测定8。然后,脱细胞与十二烷基硫酸钠(SDS)来实现的。在该步骤中,一个低浓度的SDS确保膜去稳定化和细胞内蛋白质的同时防止更可溶的非整体的ECM组分的破坏释放。最后,ECM蛋白与胍盐酸盐缓冲液(盐酸胍)萃取。盐酸胍是有效地从组织如腱9,软骨10,容器11,12,13和心脏2,3提取重交联的蛋白和蛋白聚糖。我们应用此生化分馏,结合LC-MS / MS,探讨在心血管疾病2重塑ECM >,3,11,12,13,14。在MS的最新进展包括新颖片段化方法,如高能量碰撞解离(HCD)和电子转移解离(ETD)的组合,其允许完整糖肽3,15的直接分析。
这里介绍一个方法来为MS分析功能,使蛋白质成分,糖基化位点的鉴定,和聚糖形式特征的分析准备ECM。相比于ECM糖基化16前面的分析,这种方法允许在糖基化分布在特定网站的方式使用MS成分变化的直接评估。我们已经采用这种方法来心血管组织。但是,它可以人所以适用,但小的修改,以便在其他组织标本ECM的研究,可以提供前所未有的见解ECM生物学。
研究方案
这项研究是经旺兹沃思地方研究伦理委员会(参考号码:06 / Q0803 / 37),并从研究和发展办公室获得机构的认可。所有患者签署知情同意书。
1.细胞外基质蛋白的提取
注:体外循环期间从心耳获得用于这些实验的人的心房组织,只是心脏的停搏后。所有样品在圣乔治医院,伦敦,英国收集。所有的组织样品必须在-80℃冷冻。不要使用固定剂,如多聚甲醛保存的样品,即交联蛋白。
- 准备所有提取缓冲液预先实验中,按表1中,为了最小化所述提取步骤之间的时间。在温度受控的环境中执行,在室温(RT)温育所有( 即,〜20℃),以确保提取之间的一致性。
- 称取20-50 mg组织的。如果几个样品进行提取,切割和一个衡量他们一个避免组织的完全解冻。使用手术刀,切块的组织成3-4更小的块( 即,2-3毫米),并一起将它们放入1.5毫升管中。
- 添加冰冷的磷酸盐缓冲盐水的500μL(PBS; 见表1和材料的数据表 ),并执行五次洗涤,以尽量减少血液污染。
- 萃取步骤1:与孵育NaCl的缓冲液
- 用PBS洗涤后,将样品放入1.5mL的管中带螺纹盖。 添加氯化钠缓冲 ( 见表1)以10倍(体积:重量)的组织重量。涡流在RT管用于以最小速度( 即,600rpm)下1个小时。
注:低涡流速度是至关重要的,以避免在该步骤期间的组织的机械破坏。使用泡沫适配器在提取过程中,将所有管。 - 转移提取物至新管和离心机中以16000×g离心10分钟,在4℃下。在-20℃下,直到使用存储提取物。简言之洗净用新鲜NaCl缓冲液的剩余的组织颗粒。使用相同类型的缓冲液( 即,NaCl缓冲液的100μL)进行洗涤,以防止与不同溶解度( 即,蛋白质用NaCl不可提取)其它蛋白质的提取。
- 洗涤后,确保完全除去缓冲液以最小化蛋白质含量与随后的提取步骤的重叠。丢弃用于洗涤的NaCl的缓冲液。
注:缓冲体积和组织重量之间的比率为可再现的提取重要。 10:1的比(V:重量)的NaCl和SDS提取和5:1的盐酸胍步骤提供足够量的蛋白质,而不饱和的缓冲区。蛋白质浓度是大约分机后1-2微克/μLraction。
- 用PBS洗涤后,将样品放入1.5mL的管中带螺纹盖。 添加氯化钠缓冲 ( 见表1)以10倍(体积:重量)的组织重量。涡流在RT管用于以最小速度( 即,600rpm)下1个小时。
- 提取步骤2:用脱细胞SDS缓冲液
- 在十次(ν:w)的添加SDS缓冲液( 表1)的组织重量;使用低浓度的SDS( 即,0.1%)是至关重要的,以避免脱细胞过程中的ECM蛋白的损失。涡流在RT管用于以最小速度( 即,600rpm)下16个小时。
注:低涡的速度减少ECM的机械破坏。 - 转移提取物到新管。离心在4℃下16000×g离心10分钟;保存在-20℃直至使用。简言之洗剩余组织粒料使用dd H 2 O操作除去SDS。保证清洗后完全去除液体。
- 在十次(ν:w)的添加SDS缓冲液( 表1)的组织重量;使用低浓度的SDS( 即,0.1%)是至关重要的,以避免脱细胞过程中的ECM蛋白的损失。涡流在RT管用于以最小速度( 即,600rpm)下16个小时。
- 提取步骤3:孵育盐酸胍缓冲
- (:W v)的组织重量在五次添加盐酸胍缓冲液( 表1)。涡流在RT管以最大速度(72个小时即 3200 RPM);剧烈涡旋促进ECM的机械破坏。
- 转移提取物到新管。离心机在16000×g下在4℃下10分钟,并储存在-20℃直至使用。
2.蛋白定量降水
注意:由于洗涤剂的存在下,根据在280nm处的吸光度测量的SDS缓冲液是具有直接蛋白质定量不相容。为了确保重现量化,用比色测定所有蛋白提取物17。
- 量化。
- 制备标准使用牛血清白蛋白(BSA)的校准曲线在适当的提取缓冲液( 即,氯化钠,SDS,或盐酸胍)17连续稀释。在此期间,解冻样品的提取物。
- 稀释的样品中提取缓冲液,以得到内浓度吸光度的线性范围内; 1:10稀释(V:V)得到十分满意的结果。稀释盐酸胍样品用等量DDH 2 O的定量的,作为比色测定法是不与> -4 M盐酸胍浓度相容。
- 使用二喹啉甲酸(BCA)基比色测定法17(参见材料的表 ),按照在96孔板中测定制造商的说明书;建议至少进行两次重复测量。
- 孵育30分钟后,取吸光度读数在570nm的波长,以便计算使用BSA标准校准曲线17中的蛋白质浓度。
- 蛋白质沉淀
- 解冻盐酸胍提取在室温。等分试样的10微克蛋白质的每个样品放入新的试管中。对于直接的糖肽的分析,等分试样50微克。添加乙醇和INCU 10倍体积的软化过夜,在-20℃。
注:盐酸胍不进一步酶促反应和大多数电泳应用程序兼容。盐酸胍的去除之前去糖基化和胰酶消化是必需的。盐酸胍在乙醇和,的溶解度相反,蛋白质的低溶解度允许有效地除去盐酸胍的同时产生大约蛋白18的98%的回收率。 - 离心将样品在16000×g的30分钟,在4℃和吸出上清液。注意不要打扰沉淀沉淀。干燥使用真空浓缩器(参见材料的表 )在室温15分钟的粒料。
注:该协议可以在这里停止,并且储存在-20℃直至使用将干燥的粒料。 - 可选地,运行凝胶电泳质量控制(QC,参见补充方法 )。
- 解冻盐酸胍提取在室温。等分试样的10微克蛋白质的每个样品放入新的试管中。对于直接的糖肽的分析,等分试样50微克。添加乙醇和INCU 10倍体积的软化过夜,在-20℃。
3.顺序去糖基化的N-糖基化位点占据的评估
- 在样品干燥(参见步骤2.2.2),制备含有脱支去糖基化酶,按照表1中的去糖基化缓冲液中。见材料表产品的详细信息。
- 添加含酶每个样品去糖基化缓冲的10μL。通过执行快速涡流和样品的降速确保适当的沉淀再悬浮。
注意:在使用脱支酶的糖单体的去除是用于随后的和完全除去O-连接的复合糖的基本和便于通过PNG酶-F N联糖的后切割。 - 孵育2小时,在25℃,以允许用于除去硫酸乙酰肝素的肝素酶通过的II.Increase温度至37℃,并孵育在温和搅拌36小时。
注意:由于低的反应体积和延长的温育时间,使用培养箱摇床和紧密地挤紧多个1.5mL的桶的50mL锥形管中,在大约45°培养箱内靠在内部ES。 - 36小时后,离心样品1分钟,在16000×g下 和蒸发 通过使用在室温真空浓缩约45分钟H 2 O,使其脱离样品。
- 用的H 2 18 O含有50 U / ml的PNG酶-F,10μL重悬干燥的样品进行切割以脱酰胺化反应的所有的天冬酰胺连接的聚糖。
注意:得到的天冬氨酸携带2.98 Da的过量质量指示N-糖基化的MS分析过程中的存在。 - 在37℃下在振荡培养箱持续搅拌孵育36个小时。
4.在溶液内胰蛋白酶消化
注意:此步骤应该两个非去糖基化( 即,用于直接糖肽分析)和去糖基化的样品进行( 即,用于聚糖OC的评估cupancy)。
- 使用总蛋白的10微克用于N-糖基化位点占据的评估(如在前面的步骤表示)。测量用于直接糖肽分析的样品中,使用蛋白质的50微克作为起始量。
注:蛋白质为10μg描述了下列步骤。根据需要扩大第evolumes( 即,对于50微克5次)。 - 变性使用9M尿素和3M脲每个样品等分试样中的蛋白质,具有分别6M尿素和2M硫脲,终浓度( 例如,对于10μL样品,20μL脲/硫脲)。
- 通过加入100mM的二硫苏糖醇(10毫摩尔DTT,3.33微升,最终浓度)降低蛋白质。孵育37℃以240rpm搅拌1个小时。
- 通过加入0.5M的碘乙酰胺(:50mM的3.7μL,最终浓度)进行烷基化之前冷却样品至室温。孵育在黑暗中1个小时。
- 使用预冷(-20℃)丙酮(样品的6倍体积),以在-20℃下过夜孵育样品。通过离心在14,000×g沉淀在4℃下25分钟。
- 吸出上清液。注意不要打扰沉淀沉淀。干燥使用在室温下30分钟的真空浓缩该蛋白颗粒。
- 重悬在0.1M三乙基碳酸氢铵(TEAB)缓冲液,pH 8.2的20微升,含有胰蛋白酶(0.01微克/微升),并在37℃和240rpm下消化过夜。
- 通过酸化,用10%三氟乙酸将样品(TFA,2微升的1%TFA的终浓度)终止消化。
5.肽清理使用C18色谱柱
注:消化后从肽混合物干扰污染物的去除降低离子抑制和改善信号噪声比和序列覆盖率。这一步应该两个非去糖基化和去糖基化SA进行mples。
- 激活在C18喷丝板的树脂(参见材料的表 ),使用每孔和离心机200μL甲醇在1,000×g下1分钟。
- 通过在1,000×g下将每80%乙腈(ACN)和0.1%TFA的阱200μL在H 2 O.离心1分钟洗净。
- 通过在1,000×g下将每1个%ACN和0.1%TFA的阱200μL在H 2 O.离心1分钟平衡。重复此步骤两次以上。
- 从第4步到含有在1,500×g下将树脂和离心1分钟孔加载样品(整个体积)。刷新流过第二次和重复离心。
- 通过在1,500×g下将每1个%ACN和0.1%TFA的阱200μL在H 2 O.离心1分钟洗净。重复此步骤两次以上。
- 洗脱用每50%ACN,0.1%TFA的阱170μL的样品中的H 2 O.离心1500×g离心1分钟。重复上一步并结合收集的洗提。
- 使用真空浓缩,在室温2小时干燥洗脱液。如果它没有被立即使用,保持干燥的样品在-80℃下直到使用。
注意:用于蛋白质鉴定去糖基样品只准备在此步骤之后,以用于LC-MS / MS。步骤6和不需要用于这些样品7。 - 解冻,重悬在2%ACN和0.05%TFA去糖基化样品中的H 2 O操作的0.5微克/微升的最终蛋白质浓度。步骤6对于非去糖基化的样品的糖肽直接分析进行。
- 任选地,过滤肽与串联质量标签(TMT)标记前;看到肽fitration的补充方法 。
6.标签与TMT(用于直接糖肽分析只)
- 解冻,重悬将干燥的粒料在50mM TEAB的50μL,以获得1微克/微升的浓度。
- ResusPED TMT零的0.8毫克小瓶(TMT 0,参见材料的表 )试剂在ACN的41μL。按照制造商的recommendantions再悬浮。
- 在50种微克的肽的比率标记肽样品至0.4mg TMT 0( 即,肽的50μL至20.5μLTMT 0)。孵育在室温1个小时。
- 通过以比6加入5%羟胺淬灭标记反应:100( 即,4.23 5%羟胺μL)。孵育在室温下15分钟。
- 干燥TMT 0 -标记的肽样品用于使用真空浓缩器在室温下1个小时。重悬在DDH 2 O的10μL
注意:由于聚糖残基,糖肽显示比非糖基化肽更高的分子量。 TMT 0增加糖的充电状态。这降低了它们的相对质荷(M / Z)的比率并有助于ETD碎片。
7。糖肽富集
- 使用在试剂盒中(参见材料的表 )提供反应缓冲液。
- 加的来自步骤6.5的结合缓冲液,每10μL样品50μL。涡glycocapture树脂溶液,直到它变均匀。使用两性离子亲水相互作用液相色谱(ZIC-HILIC)系捕获15。
- 等分试样50μL的树脂悬浮于新的1.5mL管。自旋为1分钟,在2500×g下,去除上清。添加样品的60μL( 即,用结合缓冲液将样品)添加到含有树脂颗粒的管中。混合使用移液管,并在1,200rpm下在RT温育在搅动20分钟。
- 离心2分钟,在2000×g离心,将上清液转移到新管中。保持管。加入洗涤缓冲液150μL到树脂管。混合使用移液管,并在1,200rpm下在RT温育在搅动10分钟。
- 旋在2500×g下2分钟。转让上清液到同一管(来自步骤7.4)中。重复洗涤步骤两次。
- 加入75微升洗脱缓冲液,并用吸管混合。搅拌在室温1200 rpm离心5分钟,然后在2500×g下离心管2分钟。转移上清液至新的1.5毫升管中。重复洗涤步骤,然后将洗脱液上清转移到相同的管中。
- 离心含有的洗脱液( 即,糖肽)的管2分钟在2500×g下。将上清转移到新的试管中,以确保在前面的步骤中除去任何残留的树脂。
- 干燥使用真空浓缩,在室温约2小时洗出物整个150μL。重悬干燥向下糖肽在2%ACN的15μL和0.05%TFA在DDH 2 O.
- 继续使用HCD碎片分析ECM蛋白质组合物进行LC-MS / MS和LC-MS / MS使用HCD和ETD碎裂为糖肽的表征。见第8。
8.质谱分析
- 使用HCD碎片分析ECM蛋白质组合物进行LC-MS / MS;见补充方法的细节。
- 使用HCD和ETD碎裂为糖肽的表征(详见补充方法 )进行LC-MS / MS;富集的样品应当相比于非富集的输入材料15。
注释:LC-MS / MS方法间接糖分析,直接糖分析和数据库检索的详细描述,在补充方法提供。鼓励有兴趣使用质谱特征ECM的蛋白质和多糖组成的研究人员参考以前的出版物3,11,15。
结果
协议的示意性工作流在图1中提供。
ECM提取协议
提取的效率可以通过运行等分试样上形成的Bis-Tris丙烯酰胺凝胶各提取物和使用银染色进行可视化来监测。 图2A示出了的NaCl,SDS的互补性和盐酸胍顺序提取后提取。此QC允许与样品质量如过量的蛋白质降解的潜在问题的识别。萃取后,ECM的糖蛋白是在盐酸胍提取物( 图2B)丰富。
去糖基化
为了评估去糖基化的效率,非去糖基化控制应PA运行rallel。去糖基化的时间必须是合适的,以实现完整和均匀的去除糖残基,如例示于图3A。 图3B示出了通过添加酶用于靶向较小N-和O-连接的寡糖GAG去除和去糖基化酶有效地去糖基化样品的代表例。
Glycoproteomics
为NXT / S序列肽段的占用的评估协议提高了MS( 图3C)后的糖蛋白的ECM蛋白的序列覆盖,并且允许糖蛋白的存在的最初筛选。这有助于减少对糖的搜索时间,因为数据库可以进行定制,以包含先前确定的糖蛋白。 HCD-ETD碎片被用于附接至ECM克低聚糖的鉴定和组分特征 lycoproteins。 图4A显示用于与18 O标记的去糖基化后(间接糖肽分析)的肽获得的代表性光谱。 图4B是从ECM中提取完整的糖肽(糖肽直接分析)的分析后得到的频谱的一个代表性的例子。
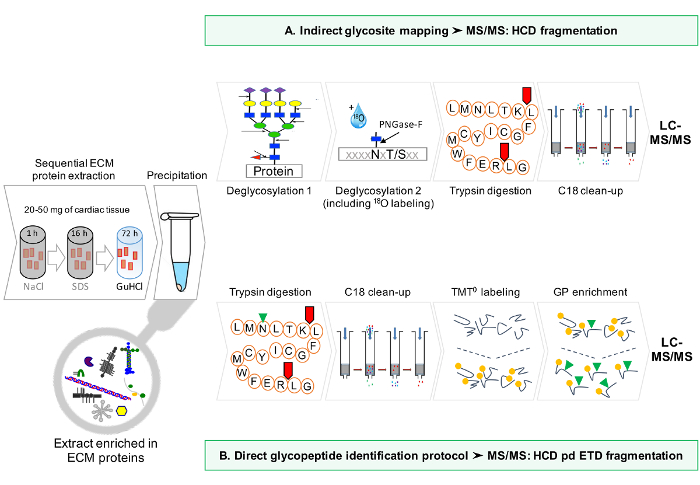
图1:方法概述。 (A)为ECM蛋白顺序富集后,LC-MS / MS分析是在去糖基化的提取物进行的。 (B)可替代地,非去糖基化ECM提取物进一步富集糖肽。 请点击此处查看该图的放大版本。
2" SRC = "/文件/ ftp_upload / 55674 / 55674fig2.jpg"/>
图2:细胞外基质蛋白的提取。 (A)从所述连续提取法("英国快步")的3种不同的提取物在它们的蛋白质含量是互补的。虽然SDS提取物中的细胞内蛋白质富集,盐酸胍提取物含有大部分ECM蛋白。成功的分离是由不同的银染色模式显示。 (B)ECM蛋白如小的富含亮氨酸的蛋白聚糖核心蛋白聚糖,糖链蛋白聚糖和mimecan在盐酸胍提取物主要是检测到的,与在SDS和NaCl提取物小存在。 请点击此处查看该图的放大版本。
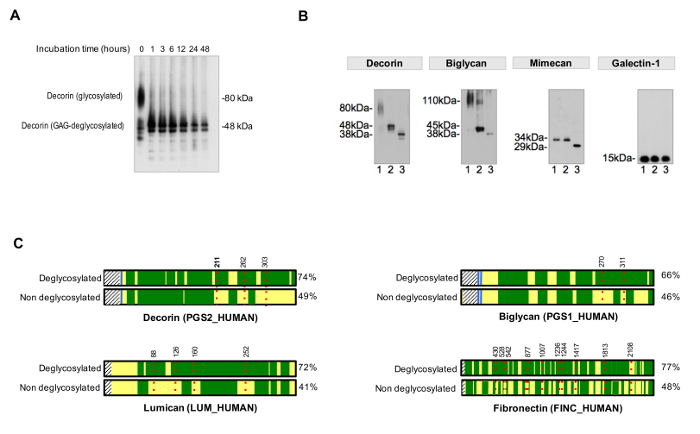
图3. GLYC分析osylation。 (A)适当的温育时间所需要的完整的去糖基化。该示例示出的温育时间从该糖蛋白核心蛋白聚糖去除糖胺聚糖链的过程中的效果。 (B)ECM的糖蛋白与大的和重复的糖胺聚糖链和短多样N-和O-连接的寡糖的装饰。在各免疫印迹的泳道1代表未处理心脏提取物。泳道2包含与该摘要糖胺聚糖酶处理的提取物。在泳道3的样品含有,此外,酶用于去除N-和O-连接的oligossacharides。半乳凝素1未糖基化的,因此存在在蛋白质大小没有变化。从证券M, 等调整。相比,非去糖基化的样品4(C)在LC-MS / MS分析,用PNGase-F在H 2 18 O的存在下处理的样品获得更好的序列覆盖率(%,在右侧)。 ð方舟绿地由LC-MS / MS代表序列覆盖。红,虚线表示glycosites,具有指示它们的氨基酸位置编号。在位置核心蛋白聚糖的糖基化的天冬酰胺的检测211(N,以粗体突出显示)中详细示出作为一个例子在图4中,请点击查看该图的放大版本。
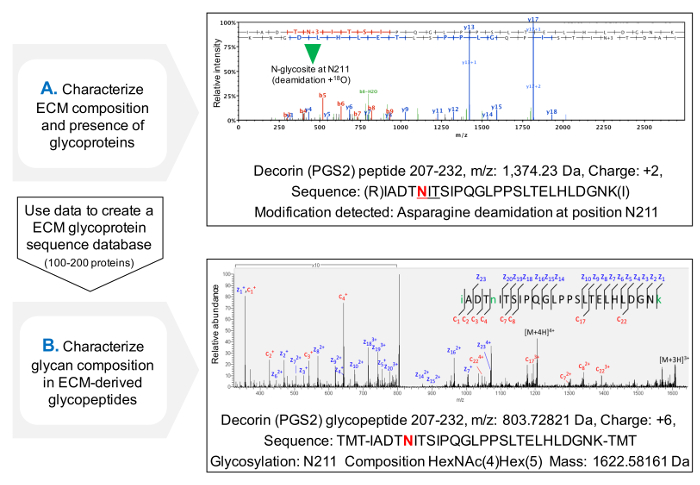
图4.糖肽分析MS。 (A)使用于ECM富集提取物鸟枪蛋白质组学方法,糖肽可以通过NXT / S序列肽段内脱酰胺化的天冬酰胺的存在来鉴定,并用18 O标记的示例示出了用于核心蛋白聚糖的含有肽的HCD MS / MS谱先前的糖基化天冬酰胺211。获得的数据可用于创建ECM糖蛋白的一个定制的数据库。 (B)HCD-ETD碎片被用于分析糖肽富集ECM提取物。的ETD MS / MS谱允许聚糖组合物的表征。 请点击此处查看该图的放大版本。
| A.股票方案 | |
| DTT(二硫苏糖醇,C 4 H 10 O 2 S 2) | 的100mM DTT在DDH 2 O 1 |
| EDTA(乙二胺四乙酸,C 10 H 16 N 2 O 8) | 的250mM EDTA在DDH 2 O,pH 8.0中。 |
| 盐酸胍(盐酸胍,CH 6 CLN 3) | -8 M盐酸胍中的DDH 2 O. |
| IAA(碘乙酰胺,C 2 H 4 INO) | 的500mM IAA在DDH 2 O 1,2 |
| 乙酸钠(乙酸钠,C 2 H 3 NaO等2) | 的1M乙酸钠在DDH 2 O,pH值5.8。 |
| 的NaCl(氯化钠,NaCl)中 | 1M NaCl的在DDH 2 O. |
| 磷酸钠二钠(磷酸二钠的Na 2 H 2 PO 4) | 的1M磷酸钠二元在DD H 2 O,pH值6.8。 |
| SDS(十二烷基硫酸钠,NAC 12 H 25 SO 4) | 1%SDS(35毫摩尔)在DDH 2 O 3 |
| TFA(三氟乙酸,C 2 HF 3 O 2) | 10%TFA(1.2 M)在DDH 2 O. |
| TEAB(三乙基碳酸氢铵,C 7 H 17 NO 3) | 1M TEAB在ddH 2 O中,pH 8.5的 |
| 硫脲(硫脲,CH 4 N 2 S) | 3 2M硫脲在DDH 2 O. |
| 的Tris-HCl(三-盐酸盐(NH(11)C 4 O 3 [HCl]的) | 的100mM的Tris-HCl中的DDH 2 O,pH 7.5中。 |
| 脲(尿素,CH 4 N 2 O) | 9M尿素在DDH 2 O. |
| B.反应缓冲液 | |
| C18清理平衡缓冲液 | 1%ACN,0.1%TFA在DDH 2 O |
| C18清理柱洗涤缓冲液 | 80%ACN,0.1%TFA的H 2ö |
| C18清理洗脱缓冲液 | 50%乙腈,0.1%TFA在DDH 2 O |
| 去糖基化缓冲液(4X) | 的600mM NaCl和200mM的磷酸钠在DDH 2 O,pH 6.8的。 |
| 盐酸胍缓冲4 | 4M的盐酸胍,50mM的醋酸钠和25mM EDTA在DDH 2 O,pH值5.8。加入1:100(V:V)蛋白酶抑制剂鸡尾酒的使用之前。 |
| NaCl缓冲液4 | 0.5M NaCl的,的10mM的Tris-HCl和25mM EDTA在DDH 2 O,pH 7.5中。加入1:100(V:V)蛋白酶抑制剂鸡尾酒的使用之前。 |
| PBS(1X) | 1.7mM的KH 2 PO 4,5mM的的Na 2 HPO 4,150mM的氯化钠,pH值7.4。添加25毫摩尔EDTA和1:100(V:V)蛋白酶抑制剂鸡尾酒的使用之前。 |
| 样品缓冲液(4X) | 100毫摩尔Tris,2%SDS,在DDH 2 O,pH 6.8的40%甘油,0.02%溴酚蓝。用前加10%β-巯基乙醇。 |
| SDS缓冲液4 | 0.1%SDS和25mM EDTA在DDH 2 O添加1:100(V:V)蛋白酶抑制剂鸡尾酒的BEF矿用。 |
| C.酶 | |
| 软骨素酶ABC 5 | 0.5 U / ml的去糖基化在缓冲液(1×) |
| 角质素5 | 0.1 U / mL,在去糖基化缓冲液(1×) |
| 肝素酶II 5 | 0.1 U / mL,在去糖基化缓冲液(1×) |
| α2-3,6,8,9 -神经氨酸酶(唾液酸酶)5 | 0.025 U / mL,在去糖基化缓冲液(1×) |
| β1,4半乳糖苷酶5 | 0.015 U / mL,在去糖基化缓冲液(1×) |
| β-N-乙酰氨基葡5 | 0.25 U / mL,在去糖基化缓冲液(1×) |
| 内切α-N-乙酰氨基葡萄(O糖苷酶)5 | 0.013 U / mL,在去糖基化缓冲液(1×) |
| PNG酶F(N-糖苷酶-F)6 | 五0 U / mL的H 2 18 O |
| 胰蛋白酶 | 0.01微克/μL在TEAB缓冲液 |
| 表格注释。 | |
| 1保持在-20℃下冷冻储液。 | |
| 2 IAA应保持避光。 | |
| 3 SDS容易结晶在<20℃。为了方便的1%SDS(原液)溶解,加温下热自来水的缓冲区。 | |
| 4个提取缓冲液可被存储在RT。添加蛋白酶抑制剂的广谱鸡尾酒使用前所示。 | |
| 5这些酶应在第一去糖基化步骤中加入。 | |
| 6 PNG酶-F应该在去糖基化的第二步骤中仅加入。 | |
表1:股票方案,反应缓冲液和酶。此表列出了前MS分析心脏ECM蛋白的提取和随后的处理(包括酶消化)所需的各储备溶液和反应缓冲液的组成。
讨论
这蛋白质组学协议已经被优化了我们实验室在过去几年。在这里,我们使用的心脏组织,但只有轻微的调整可能需要其应用到其他组织。例如,提取方案需要采取的组织的细胞结构考虑。相比血管组织的心脏组织是高度细胞。当使用血管组织中,SDS浓度可以更低( 即 0.08%)和脱细胞时间较短( 即,4小时)11,12,13。使用去糖基化酶的是ECM组合物的LC-MS / MS分析是至关重要的。然而,需要孵育时间进行调整,针对不同的组织类型。例如,肝素酶II需要延长的温育时间在25℃下使用的样品,如皮肤,它们含有丰富的基底膜蛋白( 例如集聚蛋白,基底膜蛋白聚糖)(数据未示出)时。可以在条件培养基中培养15进行从细胞中直接糖肽分析。可以不需要对于该简化subproteome的分析富集步骤。以盐酸胍提取物类似,氯化钠提取物也适合用于稍作修改glycoproteomics分析。对于ECM的蛋白质富集其他提取协议可以适用于:表征ECM糖19,20。
糖基化是最复杂的PTM 5。糖肽的间接标识是通过检测脱酰胺化的Asn的与掺入18 O以NXT / S序列子来实现的。脱酰胺的天冬酰胺在其他位置可能代表误报。同样地,N-糖基化必须的蛋白质本体的背景下考虑:含NXT / S的序列子细胞内蛋白质不会被糖基化,但可能会引起误报。由于当前变奏ħ算法不允许翻译后修饰中的预先确定的序列仅( 即在天冬酰胺NXT / S)筛选,需要数据的手动过滤。在这些位置处存在/不存在的糖基化的识别可以疾病和对照样品之间进行比较。没有酶相当于PNG酶F下O型去糖基化( 即引入上的苏氨酸或丝氨酸的质量转移)。因此,O-糖基化的识别被限制为直接的糖肽的分析。直接糖肽分析用于获得连接至蛋白质的糖的组成的信息,但不提供该聚糖的结构信息。此外,聚糖的组合物是分泌后聚糖合成和处理的结果。
我们的ECM蛋白的3步提取法("英国快步")6已允许在各种心血管组织的表征ECM。该组织的分馏成若干提取物是必需的,以获得简化的ECM蛋白质组如别处所讨论的6。细胞内蛋白质否则有助于蛋白质丰度的过度的动态范围会妨碍较不丰富的ECM蛋白的鉴定的提取物中。此外,细胞内蛋白质进行O型糖基化5,将复杂化ECM糖肽富集和随后的MS分析。其他作者施加类似的提取方法来表征,例如肺21和软骨组织10中 ,然而,它们不追求的糖基化的分析。糖基化的前一个分析仅着眼于glycosites的识别,需要去除蛋白质核心聚糖的,并且不能评估O-糖基化22,23。凝集素阵列和化学浓缩可供聚糖典型的评估根据他们的结合特异性生物样品ES,但这些技术不能分配聚糖类型特异性蛋白24也不能评估基化位点。
最初,我们之前的ECM蛋白的LC-MS / MS使用凝胶电泳。虽然凝胶分离是在获得简化的蛋白组分适合于LC-MS / MS分析是有用的,最新的仪器提供更快的扫描速度。因此,电泳分离步骤可以省略。这提供了一个额外的优点,因为大的ECM蛋白,其被保留在凝胶的顶部上,更有效地进行分析。然而,对于完整蛋白质的分子量信息丢失。之前PNG酶去糖基化˚F蒸发步骤可确保完全除去定期H 2 O与最小化假阴性。糖残基( 即,可变聚糖质量)干扰通过LC分离并通过MS / MS损害后续肽鉴定。有p也建议不是集中在糖基化细胞外基质蛋白的蛋白质组学分析的-去糖基化的协议。
蛋白质组学可以提供前所未有的洞察到ECM。除了提供结构支撑,安装于ECM聚糖是宿主-病原体相互作用,细胞间通讯以及免疫应答25, 即 ,器官移植后的同种异体移植物排斥是必不可少的。 Glycoproteomics将糖生物学的重要工具。
披露声明
没有。
致谢
JBB是职业建立院士在国王的英国心脏基金会中心。 MM是英国心脏基金会的高级研究员(FS / 13 /29892分之2)。这项研究是由一个卓越的主动支持(技术中心为优秀的技术公司 - COMET)奥地利研究促进机构FFG的:"卓越的血管研究中心老化 - 蒂罗尔州,VASCage"(K-项目编号843536)和NIHR生物医学研究在国王学院医院的合作基础,在盖伊和圣托马斯国民保健服务信托基金会和伦敦国王学院的中心。
材料
| Name | Company | Catalog Number | Comments |
| A. Chemicals | |||
| Acetonitrile, MS-grade (ACN, C2H3N) | Thermo Scientific | 51101 | 5.2-5.8, 6.2, 7.11, Supp 2, 3, 4 |
| Cocktail of proteinase inhibitors | Sigma-Aldrich | P8340 | 1.3, 1.4.1, 1.5.1, 1.6.1 |
| Disodium phosphate (Na2H2PO4) | Sigma-Aldrich | S7907 | 3.1 |
| Dithiotreitol (DTT, C4H10O2S2) | Sigma-Aldrich | D0632 | 4.3 |
| Ethylenediaminetetraacetic acid (EDTA, C10H16N2O8) | Sigma-Aldrich | E9884 | 1.3, 1.4.1, 1.5.1, 1.6.1 |
| Ethanol (C2H6O) | VWR | 437433T | 2.2.1 |
| Guanidine hydrochloride (GuHCl, CH6ClN3) | Sigma-Aldrich | G3272 | 1.6.1 |
| Glycerol (C3H8O3) | Acros organics | 158920025 | Suppl 1.1 |
| H2O LC-MS Cromasolv | Sigma-Aldrich | 39253-1L-R | Throughout the protocol |
| H218O | Taiyo Nippon Sanso | FO3-0027 | 3.5 |
| Hydroxylamine (HA, H3NO) | Sigma-Aldrich | 467804 | 6.4 |
| Iodoacetamide (IAA, C2H4INO) | Sigma-Aldrich | A3221 | 4.4 |
| Phosphate-buffered Saline (PBS), 10x | Lonza | 51226 | 1.3 |
| Sodium acetate (C2H3NaO2) | Sigma-Aldrich | S7545 | 1.6.1 |
| Sodium chloride (NaCl) | Sigma-Aldrich | S9888 | 1.4.1, 3.2 |
| Sodium dodecyl sulfate (SDS, NaC12H25SO4) | Sigma-Aldrich | 466143 | 1.5.1 |
| Triethylammonium bicarbonate (TEAB, C7H17NO3) | Sigma-Aldrich | 11268 | 4.7, 6.1 |
| Trifluoroacetic acid (TFA, C2HF3O2) | Sigma-Aldrich | T62200 | 4.8, 5.2-5.8, 7.11, Supp 2, 3 |
| Thiourea (CH4N2S) | Sigma-Aldrich | T8656 | 4.2 |
| Tris-hydrochloride (Tris-HCl, NH11C4O3[HCl]) | Sigma-Aldrich | T3253 | 1.4.1, Suppl 1. |
| Urea (CH4N2O) | Sigma-Aldrich | U1250 | 4.2 |
| Name | Company | Catalog Number | Comments |
| B. Enzymes | |||
| α2-3,6,8,9-Neuraminidase (Sialidase) | EDM Millipore | 362280 (KP0012) | 3.1 |
| β1,4-Galactosidase | EDM Millipore | 362280 (KP0004) | 3.1 |
| β-N-Acetylglucosaminidase | EDM Millipore | 362280 (KP0013) | 3.1 |
| Chondroitinase ABC | Sigma-Aldrich | C3667 | 3.1 |
| Endo-α-N-acetylgalactosaminidase (O-glycosidase) | EDM Millipore | 362280 (KP0011) | 3.1 |
| Heparinase II | Sigma-Aldrich | H6512 | 3.1 |
| Keratanase | Sigma-Aldrich | G6920 | 3.1 |
| PNGase-F (N-Glycosidase F) | EDM Millipore | 362280 (KP0001) | 3.5 |
| Trypsin | Thermo Scientific | 90057 | 4.7 |
| Name | Company | Catalog Number | Comments |
| C. Reagent kits | |||
| 30 kDa MWCO spin filters | Amicon, Millipore | 10256744 | 5.9, Suppl 2 |
| Macro SpinColumn C-18, 96-Well Plate | Harvard Apparatus | 74-5657 | 5.1 |
| NuPAGE Novex BisTris Acrylamide Gels | Thermo-Scientific | NP0322PK2 | Suppl 1 |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23227 | 2.1.3 |
| ProteoExtract Glycopeptide Enrichment Kit | Merk Millipore | 72103 | 7 |
| Tandem mass tag 0 (TMT0) | Thermo Scientific | 900067 | 6.2, 6.3 |
| Name | Company | Catalog Number | Comments |
| D. Equipment and software | |||
| Acclaim PepMap100 C18 Trap, 5 mm x 300 µm, 5 µm, 100 Å | Thermo Scientific | 160454 | Suppl 3, 4 |
| Acclaim PepMap100 C18, 50 cm x 75 µm, 3 µm, 100 Å | Thermo Scientific | 164570 | Suppl 3 |
| Byonic Search Engine | Protein Metrics | Version 2.9.30 | Suppl 5 |
| Dionex UltiMate 3000 RSLCnano | Thermo Scientific | n/a | Suppl 3, 4 |
| EASY-Spray Ion Source | Thermo Scientific | ES081 | Suppl 4 |
| EASY-Spray PepMap RSLC C18, 50 cm x 75 µm, 2 μm, 100 Å | Thermo Scientific | ES803 | Suppl 4 |
| Mascot Search Engine | Matrix Science | Version 2.3.01 | Suppl 3 |
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | IQLAAEGAAPFADBMBHQ | Suppl 4 |
| Proteome Discoverer Software | Thermo Scientific | Version 2.1.1.21 | Suppl 3, 5 |
| Picoview Nanospray Source | New Objective | 550 | Suppl 3 |
| Q Exactive HF Mass Spectrometer | Thermo Scientific | IQLAAEGAAPFALGMBFZ | Suppl 3 |
| Savant SpeedVac Concentrator | Thermo Scientific | SPD131DDA | 2.2.2, 3.4, 4.6, 5.7, 6.5, 7.11 |
| Scaffold Software | Proteome Software | Version 4.3.2 | Suppl 3 |
参考文献
- Porter, K. E., Turner, N. A. Cardiac fibroblasts: at the heart of myocardial remodeling. Pharmacol. Ther. 123 (2), 255-278 (2009).
- Barallobre-Barreiro, J., et al. Proteomics analysis of cardiac extracellular matrix remodeling in a porcine model of ischemia/reperfusion injury. Circulation. 125 (6), 789-802 (2012).
- Barallobre-Barreiro, J., et al. Glycoproteomics reveals decorin peptides with anti-myostatin activity in human atrial fibrillation. Circulation. 134 (11), 817-832 (2016).
- Lynch, M., Barallobre-Barreiro, J., Jahangiri, M., Mayr, M. Vascular proteomics in metabolic and cardiovascular diseases. J. Intern. Med. 280 (4), 325-338 (2016).
- Varki, A., Lowe, J. B., Varki, A. Biological Roles of Glycans. Essentials of glycobiology. 2nd ed. , (2009).
- Barallobre-Barreiro, J., Lynch, M., Yin, X., Mayr, M. Systems biology - opportunities and challenges: The application of proteomics to study the cardiovascular extracellular matrix. Cardiovasc. Res. , (2016).
- Agnetti, G., Husberg, C., Van Eyk, J. E. Divide and conquer: the application of organelle proteomics to heart failure. Circ. Res. 108 (4), 512-526 (2011).
- Mason, R. M., Mayes, R. W. Extraction of cartilage protein-polysaccharides with inorganic salt solutions. Biochem. J. 131 (13), 535-540 (1973).
- Vogel, K. G., Peters, J. A. Isolation of proteoglycans from tendon. Methods. Mol. Biol. 171, 9-17 (2001).
- Wilson, R., et al. Comprehensive profiling of cartilage extracellular matrix formation and maturation using sequential extraction and label-free quantitative proteomics. Mol. Cell. Proteomics. 9 (6), 1296-1313 (2010).
- Barallobre-Barreiro, J., et al. Extracellular matrix remodeling in response to venous hypertension: proteomics of human varicose veins. Cardiovasc. Res. 110 (3), 419-430 (2016).
- Didangelos, A., Yin, X., Mandal, K., Baumert, M., Jahangiri, M., Mayr, M. Proteomics characterization of extracellular space components in the human aorta. Mol. Cell. Proteomics. 9 (9), 2048-2062 (2010).
- Didangelos, A., et al. Extracellular matrix composition and remodeling in human abdominal aortic aneurysms: a proteomics approach. Mol. Cell. Proteomics. 10 (8), (2011).
- Grandoch, M., et al. Loss of biglycan enhances thrombin generation in apolipoprotein E-deficient mice: Implications for inflammation and atherosclerosis. Arterioscler Thromb. Vasc. Biol. 36 (5), e41-e50 (2016).
- Yin, X., Bern, M., Xing, Q., Ho, J., Viner, R., Mayr, M. Glycoproteomic analysis of the secretome of human endothelial cells. Mol. Cell. Proteomics. 12 (4), 956-978 (2013).
- Parker, B. L., et al. Quantitative N-linked glycoproteomics of myocardial ischemia and reperfusion injury reveals early remodeling in the extracellular environment. Mol. Cell. Proteomics. 10 (8), (2011).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Anal. Biochem. 150 (1), 76-85 (1985).
- Pepinsky, R. B. Selective precipitation of proteins from guanidine hydrochloride-containing solutions with ethanol. Anal. Biochem. 195 (1), 177-181 (1991).
- de Castro-Brás, L. E., et al. Texas 3-step decellularization protocol: looking at the cardiac extracellular matrix. J. Proteomics. 86, 43-52 (2013).
- Naba, A., Clauser, K. R., Hynes, R. O. Enrichment of extracellular matrix proteins from tissues and digestion into peptides for mass spectrometry analysis. J Vis Exp. (101), e53057 (2015).
- Decaris, M. L., et al. Proteomic analysis of altered extracellular matrix turnover in bleomycin-induced pulmonary fibrosis. Mol. Cell. Proteomics. 13 (7), 1741-1752 (2014).
- Zhang, H., Li, X. J., Martin, D. B., Aebersold, R. Identification and quantification of N-linked glycoproteins using hydrazide chemistry, stable isotope labeling and mass spectrometry. Nat. Biotechnol. 21 (6), 660-666 (2003).
- Parker, B. L., et al. Site-specific glycan-peptide analysis for determination of N-glycoproteome heterogeneity. J. Proteome Res. 12 (12), 5791-5800 (2013).
- Li, Y., et al. Simultaneous analysis of glycosylated and sialylated prostate-specific antigen revealing differential distribution of glycosylated prostate-specific antigen isoforms in prostate cancer tissues. Anal. Chem. 83 (1), 240-245 (2011).
- Rienks, M., Papageorgiou, A. P., Frangogiannis, N. G., Heymans, S. Myocardial extracellular matrix: an ever-changing and diverse entity. Circ. Res. 114 (5), 872-888 (2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。