Method Article
分析肿瘤基因表达因子与CorExplorer门户网站
摘要
我们介绍了CorExplorer门户网站,一种用于探索机器学习算法CorEx(相关解释)发现的肿瘤RNA测序因子的资源,并展示了如何分析与生存、数据库注释相关的因素,蛋白质-蛋白质相互作用,彼此深入了解肿瘤生物学和治疗干预。
摘要
微分基因表达分析是了解疾病状态的重要技术。机器学习算法CorEx在分析肿瘤RNA-seq中基因群的差分表达方面显示出了效用,有助于推进肿瘤学的精确化。但是,CorEx 会产生许多因素,这些因素对于分析和连接现有理解可能具有挑战性。为了方便这种连接,我们建立了一个网站,CorExplorer,允许用户以交互方式探索数据并回答与其分析相关的常见问题。我们对CorEx进行了四种肿瘤类型的RNA-seq基因表达数据的培训:卵巢、肺、黑色素瘤和结肠直肠。然后,我们将相应的生存、蛋白质-蛋白质相互作用、基因本体学 (GO) 和京都基因和基因组百科全书 (KEGG) 通路富集和热图纳入网站,以便与因子图可视化关联。在这里,我们使用示例协议来说明数据库的使用,以理解所学肿瘤因子在此外部数据上下文中的重要性。
引言
自十多年前引入以来,RNA-seq已成为测量基因表达1的无处不在的工具。这是因为它允许快速和廉价的样本的整个转录组分析。然而,RNA-seq肿瘤数据反映了一种基础生物学,它本质上是复杂的,而且往往采样不足,而数据本身是高维和嘈杂的。这对提取可靠信号提出了重大挑战。CorEx算法利用多变量相互信息来查找这种情况下微妙的模式2,3。这项技术以前曾被改编用于分析来自癌症基因组图谱(TCGA)的卵巢肿瘤RNA-seq样本,在这种情况下,它似乎比更常用的分析方法4具有显著的优势。
虽然RNA-seq的使用在研究应用,包括肿瘤学中非常普遍,但这些努力并没有导致临床干预的广泛应用5。部分原因在于缺乏针对这些具体问题的用户友好型算法和软件。为了帮助弥补这一差距,我们设计了CorExplorer门户网站,使来自不同背景的研究人员能够研究CorEx机器学习算法发现的肿瘤RNA-seq样本的基因表达因子。CorExplorer 门户支持交互式可视化和查询来自几个不同肿瘤类型的因素,包括肺、结肠、黑色素瘤和卵巢6、7、8、9、 10,旨在帮助研究人员筛选数据相关性,并确定为治疗目的对患者进行分层的候选途径。
我们预计 CorExplorer 门户可能对多种类型的用户有用。该门户的设计考虑到了用户,希望了解导致公共数据库中肿瘤基因表达差异的广泛因素,并可能将单个基因表达配置文件置于具有类似肿瘤的肿瘤上下文中特征。除了此处概述的代表性协议外,CorExplorer 调查可以作为一个起点,为进一步测试提出假设、比较和对比 CorEx 在 CorExplorer 外部数据集上的发现,以及连接单个肿瘤中一个或几个基因的病理表达特征,对可能协调受影响的较大群体。最后,它可以作为用户友好的介绍,将机器学习应用于RNA-seq,适用于该领域入门人员。
研究方案
1. 探索包含感兴趣的基因的因素
- 打开 Web 浏览器并转到 http://corex.isi.edu,CorExplorer 主页。
- 在"快速链接"下的右侧,单击卵巢 (TCGA-OV)旁边的+展开按钮,查看在 TCGA 卵巢癌数据上训练的 CorEx 因子图的摘要(如图1所示)。或者,单击其他要进行比较。
- 完成因子图检查后,单击肺 (TCGA-LUAD)访问肺癌RNA-seq的CorExplorer页面。
- 使用 CorExplorer"因子图"窗口浏览感兴趣的基因的 CorEx 因子图。
- 将鼠标光标移到因子图形显示窗口上。使用鼠标滚轮或触控板放大因子图,查看图形的详细信息,例如每个因子中最重要的基因以及不同层节点之间的连接。或者,单击并拖动以移动视图区域或任何节点。
- 要查找目标基因(此处我们将使用 BRCA1),请单击因子图窗口顶部的"基因"下拉菜单。键入"BRCA1"以在下拉列表中选择它,然后按Return使视图缩放到因子 26,这是 BRCA1 与因子关联性最大的因素。
- 将鼠标重新定位到图形显示屏上并滚动以缩小以查看级别 2 节点 L2_8 及其与因子 26 相邻因素的相关因子。请注意,仅显示权重大于Min 链接权重滑块上指示的阈值的基因。
- 要查看与因子相关的所有基因,请单击 L1_26 节点,然后选择在弹出窗口中加载其他基因。当出现"完成"一词时,关闭弹出窗口。
- 现在返回因子图形窗口上方的标题部分,抓取并拖动Min 链接权重修改器。现在,当链接权重滑块向下移动到 0.05 时,因子 L1_26 中的其他基因(包括 BRCA2)将以重量顺序显示。或者,通过抓取和拖动来重新定位节点,以改善布局。
- 通过在生存窗口中查询,确定患者相对于该因子的分层如何影响生存。
- 在生存窗口中,取消按p-val排序,然后在"单因子"下拉菜单中选择因子 26,以显示因子 26 的生存曲线。
- 向下滚动生存图,显示沿 x 轴处于危险中的患者数量。
- 通过在注释窗口中查询查找与生物功能的关联。
- 在注释窗口中,要按因子编号而不是错误发现率 (FDR) 对因子下拉菜单进行排序,请取消选中FDR 排序。
- 滚动并单击以在注释窗口下拉列表中选择因子 26 以显示因子的丰富注释。
- 向下滚动注释列表,直到DNA 修复可见,然后单击它,立即看到图形显示屏上以黄色突出显示的相关基因。参见图 2的中间面板。
- 请注意,因子消失或出现不同的GO术语选择,根据他们是否丰富了基因与选定的注释,例如"内在凋亡信号通路响应DNA损伤。
- 通过添加具有不同功能的窗口,进一步探索因素。
- 从顶部菜单栏中,通过从"添加窗口"下拉列表中选择PPI来添加蛋白质-蛋白质交互网络 (PPI) 窗口,然后单击"添加"按钮以向显示区域添加 PPI 图形窗口。在 PPI 图形窗口中,选择因子"第 1 层:26"以显示蛋白质-蛋白质的相互作用。请注意连接的密度。
- 从顶部菜单栏(而不是PPI)中,从"添加窗口"下拉列表中选择"热图",然后单击"添加"按钮,将热图窗口添加到显示区域。在热图窗口中,选择因子"第 1 层:26"以显示基因表达模式。
- 抓取并重新定位热图窗口,以便生存窗口也可见。沿着热图的顶部,观察生存图上的橙色/蓝色/灰色条如何对应于患者风险层。结果如图2的底部所示。
- 使用 CorExplorer"因子图"窗口浏览感兴趣的基因的 CorEx 因子图。
2. 使用基因权重、存活率和注释数据过滤和解释 CorEx 因子
- 使用生存率和群集质量筛选感兴趣的因素。
- 从顶部的数据集下拉菜单中,选择TCGA_OVCA以转到 TCGA 卵巢癌 RNA-seq 的 CorExplorer 页面。
- 加载页面后,请注意,不同地层生存差异最大的因子为 114。
- 在因子图窗口的顶部,从因子下拉列表中选择"第 1 层:114"。
- 用鼠标抓取链接重量滑块并将其向上移动到 0.5。请注意,因子 114 (1609) 中的大量基因(无一个基因具有大于;0.35)表示聚类相对较弱。
- 接下来,展开生存窗口中的因子列表,并在生存窗口下拉列表中选择下一个最佳因子,因子 39,以显示其相关的生存曲线。
- 通过单击注释窗口中的因子 39 来选择因子 39。显示重要的 GO 和 KEGG 注释。
- 为了更好地了解基因在因子39中的生物学作用,使用邻域注释信息解释因子如下。
- 在因子图形窗口的顶部,在因子下拉列表中选择因子"第 1 层:39"。然后,将鼠标移到因子图窗口上并缩小以显示整个 L2_14 群集,其中有 6 个因子:14、32、39、42、52 和 82(如图3所示)。
- 要了解与 L2_14 节点相关的因子的相对重要性,请首先查看每个 L2_14 因子的生存差。在生存窗口中按p-val取消选中排序,然后依次单击每个因子编号。执行此操作时,请注意,只有因子 14、32 和 39 显示生存关联。
- 现在,从顶部菜单栏中,再次从"添加窗口"下拉列表中选择PPI。按"添加"向显示区域添加 PPI 图形窗口。在 PPI 图形窗口中,选择因子"第 1 层:52"以显示显著蛋白-蛋白质相互作用。此时的窗口示例布局如图3所示。
- 单击 PPI 窗口底部的"在 StringDB 上查看"链接以链接到 StringDB 联机数据库。从第一个屏幕单击"继续",然后选择网络图形下方的分析选项卡,获取 PPI 网络基因的在线 GO 分析。细胞组分是"MHC II类蛋白质复合物"。
- 返回到 CorExplorer 选项卡和 PPI 窗口,然后从因子下拉列表中选择因子 32。单击StringDB中的链接视图到 StringDB 分析。顶级细胞成分是"MHC I 类蛋白质复合物",与上一步中因子 52 的 II 类相反!
- 最后,返回 PPI 窗口,从顶部的因子下拉菜单中选择"第 1 层:39"。单击StringDB 中的链接视图以链接到 StringDB 分析。
- 从第一个屏幕单击"继续",然后选择网络图下方的分析选项卡,获取 PPI 网络基因的在线 GO 分析。观察,顶部分子功能是"CXCR3化学素受体结合"。
3. 使用生存和数据库注释寻找有前途的治疗组合
- 通过从数据集下拉菜单中选择TCGA_SKCM,切换到 TCGA 黑色素瘤 CorExplorer。
- 请注意,生存差异最大的因子是因子 171。通过滚动检查因子 171 注释,并注意"免疫反应"和"细胞因子介导信号通路"接近顶部(因为它们是作为顶级卵巢因子)。
- 要查找互补因素,请检查与生存相关的顶级因素及其首要注释术语。为此,请单击顶部菜单栏中的数据集概述链接,打开一个单独的选项卡,其中包含包含数据集处理详细信息的表,以及根据生存差异的 p 值汇总顶级因素。请注意,第一个非免疫因子是88。
- 返回到TCGA_SKCM浏览器选项卡。
- 在生存、注释和图形窗口中选择因子 88。前几个GO术语与"rRNA处理"和"线粒体组织"有关,确认它与免疫相关因素不同。
- 在生存窗口中,在配对因子下拉列表中,选择"88_171",看看如何提高中间阶层患者在合并171和88表达因子中的生存率。注释和生存比较如图4所示。
4. 使用搜索页面查找肿瘤类型间基因表达变异的共性和差异
- 单击CorExplorer标题返回首页。
- 单击顶部菜单栏上的"搜索"以转到允许搜索 CorExplorer 网站上所有数据集的页面。
- 在基因搜索框中,输入"FLT1"(VEGFR1),然后点击"返回"或按"搜索"。FLT1在以下因素中具有相对较高的重量:OVCA - 76、LUAD - 162、SKCM - 195 和 SKCM - 184,以及 COAD - 112 和 COAD - 74。
- 或者,在所有数据集中搜索相关的 GO 术语。在"GO 搜索"框中尝试此项,键入"血管生成"并点击"返回"或按"搜索"。除SKCM-195外,所有FLT1因子都被列为"血管生成"基因的统计富集因子——因子195确实有注释,但低于默认的10-8阈值。此和上一步的搜索结果如图5所示。
- 作为进一步的例子,在GO搜索框中,第一类"表皮生长因子受体"。只有LUAD是丰富的这个术语,一个众所周知的肺癌的分层因子。接下来,在搜索框中键入"美感"。该术语在OVCA的基因表达组中丰富,它是一个经过深入研究的分层因子。
结果
在肺癌数据集中搜索基因"BRCA1"后发现它与CorEx因子26的关系最为密切(图2)。GO术语富集这个因子被认为是非常高的,与DNA修复表现出只有1 x10-19的FDR。该选择还提请注意第二级群集 L2_8,该群集具有六个密切相关因素作为子级。在 GO 术语注释中选择"DNA 修复"或因子图的 GO 丰富下拉列表突出显示每个因子中的相关基因,因子 26 是迄今为止最多的,如预期11所示。蛋白质-蛋白质相互作用网络紧密相连,进一步支持了因子26中基因的紧密连接功能。关联的生存图表明与患者生存有可能的联系,但必须在较大的数据集中确认这一点。
从生存开始,可以解剖与特定基因表达组相关的改善生存的原因。例如,影响卵巢癌存活率的首要因素是39号,这与免疫系统相关的基因具有很强的丰富性(图3)。与同一级别 2 节点相关的其他五个因素也显示与免疫相关,但其中生存影响似乎变化很大,其中 39 个最高,52 个最低。为因子添加蛋白质-蛋白质交互窗口可显示即时交互网络,并允许链接到 StringDB12网站,以查询 PPI 网络基因的各种扩增。通过依次对每个 L2_14 因子执行此操作,人们发现 PPI 网络基因的 StringDB 扩充为与生存的关联提供了以下可能的解释。因子32包含构成主要组织相容性复合物(MHC)I类蛋白质复合物的基因,由细胞毒性T淋巴细胞识别。因子39对应于细胞因子信号和CXCR3受体结合,与CD8+T淋巴细胞相关。这两个因素似乎给表现出相对高表达相应基因的患者带来显著的生存优势。细胞毒性CD8+T淋巴细胞主要负责抗肿瘤免疫。另一方面,因子52由MHC II类复合物中蛋白质的基因编码组成,这些基因主要由CD4+T帮助细胞识别,而不是直接由细胞毒性T淋巴细胞识别。其余的L2_14因子反映了广义的免疫系统激活,不区分两种类型的淋巴细胞种群。一种特定于细胞毒性T淋巴细胞识别MCH I类细胞抗原的生存关联符合我们对一般抗肿瘤免疫和其他癌症(如黑色素瘤13、14)的理解。
门户网站支持发现具有互补功能的因子对,这些因子可能建议有效的肿瘤特异性组合疗法。可以扫描数据集概览,查找显示与生存相关但具有不同 GO 扩充性的因素。对于黑色素瘤(TCGA_SKCM;图4),它看到,最大的生存因子171是免疫相关的,而因子88下榜显示富集基因与线粒体组织有关。事实上,这已被建议作为黑色素瘤15的目标。将生存窗口添加到 CorExplorer 页面后,可以分别使用因子对将分层与每个因子的分层进行比较,表明两组有利的基因表达模式都表现出比两组更好的生存趋势单单因素。然而,顶层似乎没有改善,这表明免疫疗法可能只是一些患者的最佳选择。
肿瘤之间的共性和差异可以通过在数据集中搜索基因或GO术语来发现(图5)。例如,FLT1(又名VEGFR1)是一个经过深入研究的亲血管生成标记16,17。当它被放入搜索栏时,所有的肿瘤都有FLT1起主要作用的因素。相反,当GO术语"血管生成"被输入到搜索页面上时,6个FLT1组中有5个出现这种扩充。除SKCM-195外,所有FLT1因子均列为"血管生成"基因的统计富集。事实上,第六个因素确实具有注释,但低于默认的 10-8 阈值。当因子列表中的权重用于替代富集计算器,例如,基因集浓缩分析(GSEA)18时,第六因子也明显富集于"血管生成"基因。
检查热图以确保基因表达模式具有足够的质量以支持生物学解释非常重要。显示强烈明显变异的热图可能表现出因子基因从低到高或更复杂的模式的协调表达,有些基因的表达低,与其他有高的基因相关(图6)。高质量分组的一个关键标志是存在几个基因,这些基因作为因子评分的函数,表达有平滑的变化。因子热图显示根据因子分数排序的样本,因此应该有从左到右的平滑渐变。但是,这可能无法以至少两种不同的方式发生。最常见的情况是,相关性可能非常嘈杂(图5C),令人怀疑任何有关生存和/或生物功能的推论的鲁棒性和实用性。此外,仅在少数样本中发生的模式可能不符合 CorEx 算法假定的三种表达式状态的模型,从而导致对样本的误导性分类(图 5D右侧)。
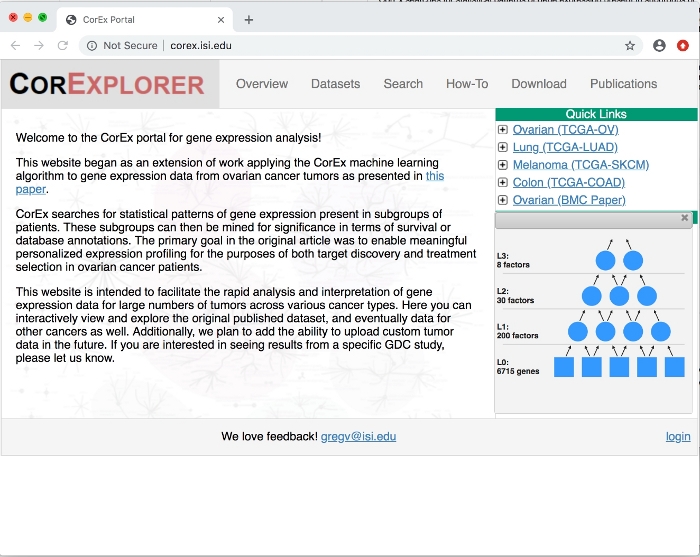
图1:CorExplorer头版。点击"快速链接"下卵巢癌旁边的"+"后,将显示因子图详细信息。 CorEx 分层模型由底层的输入变量(本例中为基因表达)和较高层中推断的潜在因子组成。请点击此处查看此图的较大版本。
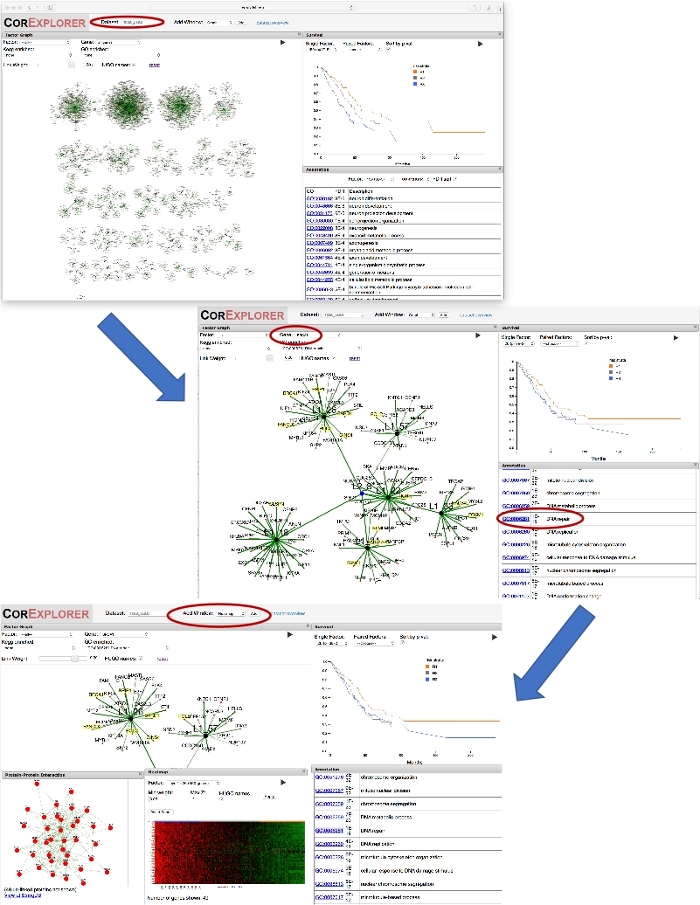
图2:使用基因名称指导探索。该图显示了一系列屏幕截图,其中说明了对与 BRCA1 密切相关的 CorEx 肺癌因素的探索。首先,在因子图的Gene下拉框中选择"BRCA1"会导致图形视图放大 BRCA1 权重最大的因子。缩小位帧第二层节点 L2_8 将该因子连接到其他相关节点。生存和注释可以比较:点击GO术语DNA修复突出显示注释基因。添加了 PPI 窗口,以显示因子中基因的网络交互。使用"添加窗口"按钮添加热图可显示表达模式与生存的关联,这表明DNA修复基因表达的增加可能与存活率下降有关。请点击此处查看此图的较大版本。
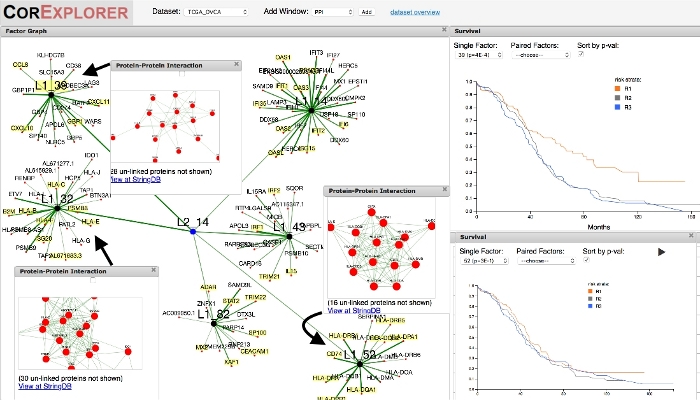
图3:利用临床数据(生存)指导探索。探索卵巢癌的首要生存相关因素(39)揭示了相邻因素之间的有趣关系。在因子图中选择因子 39 并稍微缩小后,与因子 39 关联的第二层因子被认为具有其他五个相关因子。额外的生存窗口允许直接比较相关的生存差异。因素39和32都显示出正生存相关性,而因子52则不显著。蛋白质-蛋白质相互作用网络都有明确的定义。链接到StringDB允许比较GO注释(未显示):因子39与细胞毒性CD8+T淋巴细胞活化相关的细胞因子信号网络相关,因子32由MHC I类抗原呈现蛋白为主,触发识别这种淋巴细胞;然而,邻近因素由其他免疫系统成分(如CD4+辅助体T细胞)主导,没有生存相关性。请点击此处查看此图的较大版本。

图4:探索顶级生存因子表明潜在的治疗组合。主页菜单栏上的"数据集"链接指向按 p 值排序的生存因子的简明表,以及顶部 GO 注释(未显示)。利用这些信息治疗黑色素瘤,免疫功能因子171与线粒体组织因子88的组合似乎具有互补性。该图显示每个因子的注释窗口并排显示,以便对其进行对比。患者的存活曲线由两个因素单独或一起分层,表明该组合比单独两个因素增加生存差异。请点击此处查看此图的较大版本。
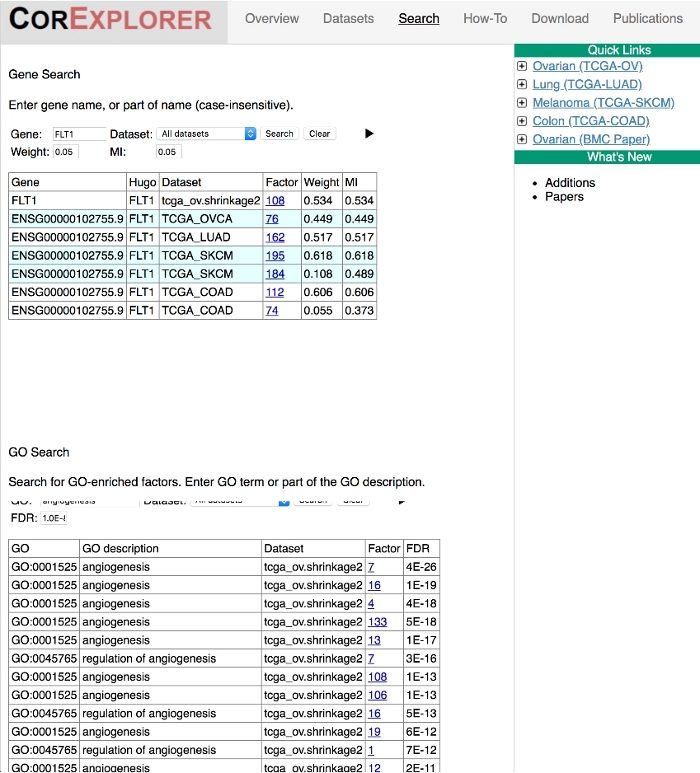
图 5:搜索页面有助于泛癌分析。可以使用主页的"搜索"链接在所有数据集中搜索基因或 GO 生物过程术语。该图显示了基因FLT1和GO术语"血管生成"的搜索结果。结果显示FLT1在癌症中带有"血管生成"一词的因子中的存在。请点击此处查看此图的较大版本。
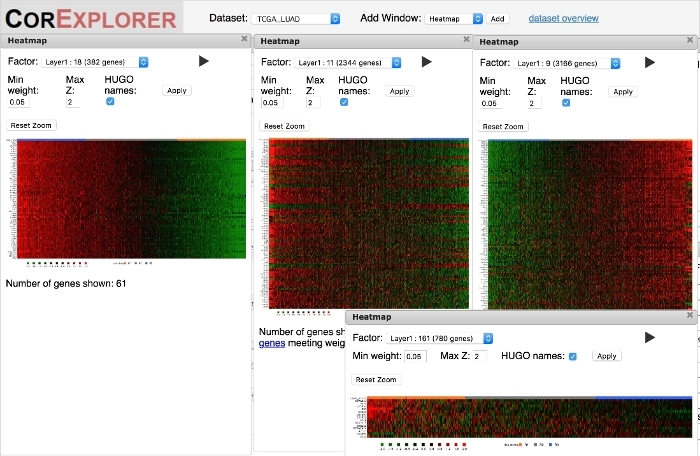
图6:热图可用于根据因子评分对基因和样本之间的相关性进行定性评估。当患者在热图中按因子评分排序时,通过平滑的分级显示高质量的基因表达关系。因子 18 的最左侧热图就是一个例子。这些模式还可以包括上下表达式的复杂特征,如因子 11 的中间大热图。较低的质量模式有时显示一组患者的表达突然变化,如右侧因子 9 热图或右侧因子 161 热图中的简单非常嘈杂的相关性。请点击此处查看此图的较大版本。
讨论
我们介绍了CorExplorer网站,这是一个可公开访问的Web服务器,用于交互式探索通过CorEx算法从肿瘤RNA-seq中学到的最大相关基因表达因子。我们已经展示了该网站如何被用来根据肿瘤基因表达对患者进行分层,以及这种分层如何与生物功能和生存相对应。
其他用于RNA-seq分析的Web服务器已经建成。肿瘤的差分和共表达分析可以检查和综合与其他数据类型在cbioPortal19,20。服务器 GenePattern21、Mev22和 Morpheus23集成了已建立的聚类技术,如主组件分析 (PCA)、kmeans 或自组织映射 (SOM)。更多的创新工作包括基于自动规则生成分类器的CamurWeb24和TACCO25,后者实现随机森林分类器和套索。此处使用的 CorEx 算法优化了多变量信息,以便找到解释数据模式的因素的层次结构。与通过PCA4找到的线性全局因子相比,非线性和分层因子学习似乎具有更好的可解释性。此外,该技术对样本信号的细粒度分析允许与更常用的广泛亚型进行精确的肿瘤比较。重叠和分层因子分析的这种组合使 CorExplorer 与大多数其他方法不同,并且需要新的可视化和汇总工具。
CorExplorer 因子分析的一个关键部分是,它不仅能够探索多个因子,而且能够探索位于重叠层次结构中的信息性基因模式的 100 多个因子。CorExplorer 有助于挖掘这些生物和临床关联的无数因素,并允许对单个肿瘤进行非常详细的表征。对如此众多因素的无监督学习意味着并非全部与疾病生物学相关。在这种情况下,必须使用注释或已知基因来拉出感兴趣的因素,或搜索与临床数据相关的因素,如生存。因此,CorExplorer 允许用户实现这个非常重要的筛选步骤。肿瘤中因子基因模式的存在甚至可能建议一种个性化肿瘤治疗的方法。此外,每个肿瘤的因子分数的多重性,允许发现潜在的有用的治疗组合。
有时,对于与生存高度相关的因素,没有出现显著的 GO 注释。虽然这可能是由于噪声或采样数据不足,还有其他可能的原因,如集群规模太小,无法记录显著的富集分数,或组是单一基因的"篮子"从不同的途径没有连贯的生物协会。此外,一类不同于KEGG和GO生物过程的注释,例如细胞隔间,可能是适当的。这些可以通过链接到 StringDB 来访问,如协议中所示。CorExplorer网站上的基因本体浓缩分析目前没有考虑到基因权重的一个因素,不过这很可能在不远的将来得到纠正。请注意,在"添加窗口"下提供了一个基因列表选项,允许下载完整的因子基因列表,以便使用外部工具进行进一步分析。
就网站而言,CorEx 在每个数据集上运行了五次,并保留了导致最大总相关性的运行。对多次运行的结果进行统计表示可能更具信息性,是未来工作的目标。此外,服务器上可用的肿瘤类型集相当小,但我们预计会根据用户兴趣随着时间的推移而扩展。
如上所述,CorExplorer 将 CorEx RNA-seq 因子关系以及临床和数据库信息可视化,从而支持各种不同的询问模式。我们希望,这一工具将引导进一步的工作,利用RNA-seq分析的力量,发现和临床应用在肿瘤学。
披露声明
提交人宣称,他们没有相互竞争的经济利益。
致谢
GV 得到了 DARPA 奖 W911NF-16-0575 的支持。
材料
| Name | Company | Catalog Number | Comments |
| Public server for CorExplorer website | USC | http://corex.isi.edu | Intel Xeon E5-2690 4-core 2.6 GHz, 8GB RAM. Backend architecture is LAMP: Linux, Apache, MySQL, PHP. |
| Web browser | Google/Apple | Chrome/Safari | Verified web browsers. |
参考文献
- Petryszak, R., et al. The RNASeq-er API-a gateway to systematically updated analysis of public RNA-seq data. Bioinformatics. 33, 2218-2220 (2017).
- Steeg, G. V., Galstyan, A. Maximally Informative Hierarchical Representations of High-Dimensional Data. Proceedings of the Eighteenth International Conference on Artificial Intelligence and Statistics (AISTATS). , (2015).
- Ver Steeg, G., Galstyan, A. Discovering structure in high-dimensional data through correlation explanation. Advances in Neural Information Processing Systems. , (2014).
- Pepke, S., Ver Steeg, G. Comprehensive discovery of subsample gene expression components by information explanation: therapeutic implications in cancer. BMC medical Genomics. 10, 12 (2017).
- Byron, S. A., Van Keuren-Jensen, K. R., Engelthaler, D. M., Carpten, J. D., Craig, D. W. Translating RNA sequencing into clinical diagnostics: opportunities and challenges. Nature Reviews Genetics. 17, 257 (2016).
- Cancer Genome Atlas Research Network. Comprehensive molecular profiling of lung adenocarcinoma. Nature. 511, 543 (2014).
- Cancer Genome Atlas Network. Comprehensive molecular characterization of human colon and rectal cancer. Nature. 487, 330 (2012).
- Akbani, R., et al. Genomic classification of cutaneous melanoma. Cell. 161, 1681-1696 (2015).
- Cancer Genome Atlas Research Network. Integrated genomic analyses of ovarian carcinoma. Nature. 474, 609 (2011).
- Grossman, R. L., et al. Toward a shared vision for cancer genomic data. New England Journal of Medicine. 375, 1109-1112 (2016).
- Moynahan, M. E., Chiu, J. W., Koller, B. H., Jasin, M. Brca1 controls homology-directed DNA repair. Molecular Cell. 4, 511-518 (1999).
- Szklarczyk, D., et al. STRING v11: protein–protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets. Nucleic Acids Research. 47, 607-613 (2018).
- Durgeau, A., Virk, Y., Corgnac, S., Mami-Chouaib, F. Recent advances in targeting CD8 T-cell immunity for more effective cancer immunotherapy. Frontiers in Immunology. 9, 14 (2018).
- Sato, E., et al. Intraepithelial CD8+ tumor-infiltrating lymphocytes and a high CD8+/regulatory T cell ratio are associated with favorable prognosis in ovarian cancer. Proceedings of the National Academy of Sciences of the United States of America. 102, 18538-18543 (2005).
- De Moura, M. B., et al. Mitochondrial respiration-an important therapeutic target in melanoma. PLoS One. 7, 40690 (2012).
- Folkman, J., Merler, E., Abernathy, C., Williams, G. Isolation of a tumor factor responsible for angiogenesis. Journal of Experimental Medicine. 133, 275-288 (1971).
- Takahashi, S. Vascular endothelial growth factor (VEGF), VEGF receptors and their inhibitors for antiangiogenic tumor therapy. Biological and Pharmaceutical Bulletin. 34, 1785-1788 (2011).
- Subramanian, A., et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proceedings of the National Academy of Sciences of the United States of America. 102, 15545-15550 (2005).
- Cerami, E., et al. The cBio Cancer Genomics Portal: An Open Platform for Exploring Multidimensional Cancer Genomics Data. Cancer Discovery. 2, 401-404 (2012).
- Gao, J., et al. Integrative Analysis of Complex Cancer Genomics and Clinical Profiles Using the cBioPortal. Science Signalling. 6, 1 (2013).
- Reich, M., et al. GenePattern 2.0. Nature Genetics. 38, 500 (2006).
- Wang, Y. E., Kutnetsov, L., Partensky, A., Farid, J., Quackenbush, J. WebMeV: A Cloud Platform for Analyzing and Visualizing Cancer Genomic Data. Cancer Research. 77, 11-14 (2017).
- . Morpheus Available from: https://software.broadinstitute.org/morpheus (2019)
- Weitschek, E., Lauro, S. D., Cappelli, E., Bertolazzi, P., Felici, G. CamurWeb: a classification software and a large knowledge base for gene expression data of cancer. BMC Bioinformatics. 19, 354 (2018).
- Chou, P. -. H., et al. tACCo, a Database Connecting transcriptome Alterations, pathway Alterations and Clinical outcomes in Cancers. Scientific Reports. 9, 3877 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。