Method Article
使用 HiBiT CRISPR 细胞系对靶向蛋白质降解化合物进行高通量细胞分析
摘要
该协议描述了活细胞中蛋白质降解动力学的定量发光检测,这些活细胞已使用CRISPR / Cas9进行工程设计,以表达融合到目标蛋白质的无抗体内源性蛋白质检测标签。包括计算和获取定量降解参数、速率、Dmax、DC 50 和 Dmax50 的详细说明。
摘要
靶向蛋白质降解化合物,包括分子胶或蛋白水解靶向嵌合体,是小分子药物发现中令人兴奋的新治疗方式。这类化合物通过接近泛素化并最终通过泛素-蛋白酶体途径(UPP)降解靶蛋白所需的靶蛋白和E3连接酶机械蛋白来诱导蛋白质降解。然而,鉴于实现降解所需的细胞途径的复杂性,以高通量方式分析靶蛋白降解仍然极具挑战性。在这里,我们提出了一种基于使用11个氨基酸HiBiT标签对靶蛋白进行CRISPR / Cas9内源性标记的协议和筛选策略,该标签与LgBiT蛋白的高亲和力互补,以产生发光蛋白。这些具有内源性标签的CRISPR靶向细胞系可用于通过使用基于发光板的阅读器监测发光信号,在实时、动力学活细胞或终点裂解模式下测量化合物诱导的降解。在这里,我们概述了不同形式的推荐筛选方案,并描述了速率,Dmax,DC 50,Dmax50的关键降解参数的计算,以及细胞活力测定的多重分析。这些方法能够快速发现和分类早期化合物,同时保持相关细胞背景中靶蛋白的内源性表达和调节,从而可以有效优化先导治疗化合物。
引言
靶向蛋白降解已成为小分子药物发现中增长最快的领域之一,这得益于免疫调节分子胶化合物(例如IMiD)在癌症治疗中的治疗成功,以及靶向嵌合体化合物1,2,3,4,5,6,7,8的蛋白水解早期临床试验数据,9,10,11,12.靶向蛋白质降解化合物通过将目标蛋白与E3连接酶机械蛋白1,2,3,4,5,6,7,8,9,10,11,12接近而起作用.这种化合物诱导的靶蛋白募集到E3连接酶中,通过泛素蛋白酶体途径(UPP)导致靶蛋白泛素化和降解1,2,3,4,5,6,7,8,9,10,11,12.从历史上看,小分子药物发现筛选计划依赖于初始生化测定来评估活性并对化合物进行排序。然而,这对靶向蛋白质降解剂提出了重大挑战,其最终活性,即通过蛋白酶体降解,取决于一系列细胞事件1,2,4,5,6,11,12,13,14,15,16,17,18. 成功靶标降解所需的蛋白质复合物的多种途径和复杂性需要细胞测定方法对初始化合物进行早期筛选和分类。目前,严重缺乏在细胞环境中以高通量方式监测靶蛋白降解的技术的可用性14。在这里,我们将介绍使用CRISPR / Cas9内源性标记的HiBiT靶细胞系18,19,20进行实时动力学活细胞或终点裂解降解活性评估的协议,以监测用降解剂化合物10,11,18,19处理后通过发光测量监测目标蛋白的损失。
为了实现治疗靶点的成功降解并扩大可成药蛋白质组,已经出现了许多方法和类型的降解剂,它们可以靶向广泛的蛋白质进行破坏,包括位于质膜或质膜,溶酶体,线粒体膜,细胞质和细胞核中的蛋白质21-57。研究最广泛的两类化合物是分子胶和靶向 cimeras2,4,5,6,7,12,26 的蛋白质。分子胶是单价的,因此通常尺寸较小,并且在与E3连接酶组分2,12,26结合时促进与靶蛋白的新型蛋白质:蛋白质相互作用界面。它们最常见的降解剂与大脑(CRBN)E3连接酶成分2,12,26,55,56,57结合。最近,利用其他E3连接酶机制(如DCAF1558,59,60和CDK / Cyclin募集到DDB145)的令人兴奋的新例子表明了这类化合物的扩展潜力。相比之下,PROTAC是较大的二价分子,由靶标结合配体(通常是抑制剂)组成,通过化学接头桥接到E3连接酶手柄1,3,4,5,7,13。因此,这些化合物能够直接结合E3连接酶和靶蛋白1,3,4,5,7,13。许多蛋白质已被证明通过这些二价分子降解,最常用的E3连接酶手柄招募CRBN或Von Hippel Lindau(VHL)1,3,4,5,7,13。然而,在靶向蛋白水解设计的嵌合体中,用于E3连接酶募集的可用手柄数量正在迅速增长,扩大了这类化合物的能力,有可能降解不同的靶标类别以及增强细胞或组织类型的特异性24,48,61,62.结合结合对目标蛋白的最低要求,即使具有边际亲和力,降解化合物也有望扩大可药物蛋白质组。
表征蛋白质损失的细胞动力学以及处理后潜在的蛋白质回收对于了解降解化合物的功能和功效至关重要。虽然可以使用蛋白质印迹抗体测定或质谱法研究相关细胞系统中的内源性蛋白质水平变化,但这些方法难以适应高通量筛选形式,定量能力有限,或能够测量许多时间点的动力学变化14。为了应对这些挑战,我们开发了一种基于板的细胞发光系统,用于监测内源性蛋白质水平的变化,该系统利用11个氨基酸标签HiBiT的CRISPR / Cas9基因组插入到任何关键降解靶标18,19,20的位点。该肽与其结合伴侣LgBiT具有高亲和力,在其底物18,19,20,63存在下产生明亮的发光,从而使这些标记的内源性蛋白质在细胞或裂解物中发光18,19,20,63.用光度计仪器测量的相对光单位(RLU)与标记的靶蛋白水平18,19,20,63成正比。随着稳定的荧光素酶底物的发展,可以在24-48小时的时间范围内进行实时动力学蛋白质水平测量18,53,64。这允许在任何给定化合物浓度下确定任何给定目标的完整降解曲线,包括初始降解速率、降解最大值(Dmax)和化合物处理后回收率的定量分析18,53。然而,如果筛选大型降解化合物库,终点分析也可以很容易地以384孔格式在不同的药物浓度和指定时间进行。
本手稿中介绍的方案代表了靶向蛋白质降解化合物的细胞筛选策略,适用于所有类型的降解剂。然而,HiBiT CRISPR细胞系与这些方案的使用不仅限于蛋白质降解,而是用于监测任何内源性靶蛋白水平的通用工具,这些靶蛋白水平可以在处理后进行调节,以研究化合物甚至抗性机制的影响20,65,66。这些基于发光的检测方法的先决条件是CRISPR内源性标记的HiBiT靶细胞系,这是至关重要的,因为它能够进行灵敏的发光检测,同时仍保持内源性靶表达和天然启动子调节18,19,20。在利用CRISRP / Cas9插入基因组标签方面取得了重大进展,特别是在可扩展性20和高检测灵敏度方面,包括CRISPR池或具有杂合或纯合等位基因插入的克隆18,19,20。在细胞中使用HiBiT或其他报告基因融合的外源表达代替内源性标记是可能的,但使用具有蛋白质过表达的系统应非常谨慎14,18。这些可以导致理解真正的化合物效力和蛋白质回收动力学的伪影14,18,包括靶标降解后激活的潜在转录反馈环。此外,低效力的早期化合物可能会被遗漏,并在筛选中表现为假阴性。由于蛋白质损失可能由化合物诱导的毒性和细胞死亡引起,因此此处描述的方案包含强烈推荐但可选的细胞活力发光或荧光测定与降解方案配对。该方案有两个主要部分,裂解终点和活细胞动力学筛选。在每个部分中,都包括用于终点或动力学格式的多重细胞活力测量的选项。监测标记的内源性蛋白的变化需要与细胞中的LgBiT互补。因此,动力学筛选部分引用了引入此方法的重要方案,该协议可以通过瞬时或稳定表达来实现,并且对于执行活细胞发光测量至关重要。这里介绍的所有方法都可以对化合物进行快速排序和活性评估,从而实现早期化合物筛选工作和更快速地鉴定铅降解剂。
该协议旨在与HiBiT CRISPR细胞系一起研究降解化合物。在最近的几篇出版物18,19,20中概述了为许多靶标生成HiBiT CRISPR插入的协议。
研究方案
1. 使用裂解形式的HiBiT CRISPR靶蛋白进行终点降解研究,并可选择细胞活力荧光分析
- 哺乳动物贴壁或悬浮细胞系的制备和接种
- 通过在用于传代和细胞生长的适当细胞培养基中稀释,将细胞密度调节至 2.22 x 105/mL。
- 将细胞分配到每个实验和控制条件下至少3孔的板中。将每孔细胞悬液 90 μL(20,000 个细胞)分配到 96 孔白板中。对于 384 孔格式,将每孔细胞悬液分配 36 μL(8,000 个细胞)到 384 孔白板中。
- 化合物的制备和添加
- 在100%DMSO中以1,000倍终浓度制备连续稀释的PROTAC或降解剂测试化合物板。然后在细胞培养基中将其稀释至终浓度的10倍。向培养基中加入等体积的DMSO,用作无复合DMSO对照。
- 对于 96 孔形式,将 10 μL 的 10x 化合物和对照溶液添加到 90 μL 的细胞中。对于 384 孔格式,将 4 μL 10x 化合物和对照溶液添加到 36 μL 细胞中。
- 将板在37°C和5%CO2 的培养箱中孵育所需的时间或在最适合其生长的条件下孵育。
注意:由于这是一项终点测定,因此测试多个时间点将需要为每个时间点准备单独的降解板,如上述步骤1.1.2中所述。检测化合物介导的降解的孵育时间变化很大,也可能取决于化合物浓度。建议的初始时间点为 6 小时和 24 小时。 - 如果在没有可选细胞活力测量的情况下测量终点发光检测,请直接执行下面的步骤1.3。如果使用细胞活力测量进行多路复用,请继续执行下面的下一节 1.4。
- 细胞裂解测量
- 在HiBiT裂解测量之前,每1 mL裂解缓冲液加入20 μL裂解底物和10 μLLLLL蛋白,制备2倍裂解检测试剂。为要分析的孔数准备足够的2x检测试剂,包括额外的体积以考虑移液误差(即孔数+ 10%)。
- 将准备好的裂解检测试剂添加到细胞中。对于 96 孔形式,向含有 100 μL 细胞的每个孔中加入 100 μL 2x 裂解检测试剂。对于 384 孔形式,向含有 40 μL 细胞的每个孔中加入 40 μL 2x 裂解检测试剂。在微孔板涡旋混合器上以350rpm混合板10-20分钟。
- 在能够读取 96 或 384 孔板中发光的光度计上测量发光。
- 可选细胞活力多路复用
注意:此步骤使用市售的荧光细胞滴定仪(CTF)试剂盒执行(参见 材料表)。- 在所需终点测量前 30-40 分钟,通过将 10 μL 底物加入 2 mL 测定缓冲液中来制备 6x 细胞活力检测试剂溶液。为每个待分析孔准备足够的6x试剂,包括移液误差的额外体积(即孔数+ 10%)。
- 将准备好的试剂加入孔中。对于 96 孔形式,向每个已经含有 100 μL 体积的孔中加入 20 μL 6x 试剂。对于 384 孔形式,向含有 40 μL 细胞的每个孔中加入 8 μL 6x 试剂。在微孔板涡旋混合器上短暂混合,然后将板在37°C培养箱中孵育30分钟。
- 在所需的测量终点(即,处理后6或24小时,步骤1.2.3),在能够以96或384孔格式读取荧光(380-400nmEx / 505nmEm)的仪器上测量荧光。
- 每 1 mL 裂解缓冲液加入 20 μL 裂解底物和 10 μL LGgBiT 蛋白,制备 2 倍裂解检测试剂。为要分析的孔数准备足够的 2x 检测试剂,包括额外的体积以考虑移液误差(例如,孔数 + 10%)。
- 将制备好的裂解检测试剂加入孔中。对于 96 孔格式,向每个已含有 120 μL 体积的孔中加入 120 μL 2x 裂解检测试剂。对于 384 孔形式,向每个已经含有 48 μL 体积的孔中加入 48 μL 2x 裂解检测试剂。在微孔板涡旋混合器上混合板10-20分钟。
- 在能够读取 96 或 384 孔板发光的光度计上测量发光。
- 降解和细胞活力的定量
- 平均 DMSO 控件在测量时间点的相对光单位 (RLU)。使用此值作为靶标的基线蛋白质水平,通过将在同一时间点测试的所有其他处理标准化为该值来计算降解分数。例如,如果DMSO对照孔在6小时时的平均RLU为10,000,而给定化合物处理在6小时时的平均RLU为5,000,则降解分数将计算为5,000÷10,000 = 0.5(公式1)。
等式1: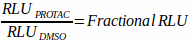
- 确定分数 RLU 的降级百分比:
等式 2: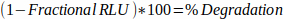
- 绘制特定时间点的分数RLU或降解百分比,以对化合物的活性进行排名。
- 或者,通过将所有处理的值与DMSO对照进行比较,分析细胞活力测定测量的相对荧光单位(RFU)数据。如果观察到相对于DMSO对照的任何处理的RFU显着下降,则可以将降解数据另外归一化为细胞活力测定数据,以确定蛋白质水平相对于细胞活力损失的变化。
- 平均 DMSO 控件在测量时间点的相对光单位 (RLU)。使用此值作为靶标的基线蛋白质水平,通过将在同一时间点测试的所有其他处理标准化为该值来计算降解分数。例如,如果DMSO对照孔在6小时时的平均RLU为10,000,而给定化合物处理在6小时时的平均RLU为5,000,则降解分数将计算为5,000÷10,000 = 0.5(公式1)。
2. HiBiT CRISPR靶蛋白的实时动力学降解和可选的细胞活力发光测定
注意:进行动力学筛选和降解的能力需要LgBiT蛋白在细胞中的共表达,这在前面已经描述过18,19,63。这可以通过瞬时转染LgBiT载体,使用BacMam LgBiT或将HiBiT CRISPR插入到LgBiT稳定细胞系中来实现。
- 贴壁细胞系的电镀。
- 通过抽吸从细胞烧瓶中取出培养基,用DPBS洗涤细胞,用0.05%胰蛋白酶-EDTA解离细胞,并使细胞从烧瓶底部解离。对于悬浮细胞系,请继续执行第 2.2 节。
- 使用含血清的细胞培养基中和胰蛋白酶,混合以收集和重悬细胞,并将细胞悬液转移到锥形管中。
- 以 125 x g 离心细胞 5 分钟。弃去细胞培养基并重悬于等体积的新鲜细胞培养基中。
- 将细胞接种到测定板中,每个实验和对照条件至少一式三份孔。对于 96 孔格式计数以估计细胞密度,在测定培养基中将密度调整为 2 x 105 个细胞/mL,并在 96 孔板中每孔分配 100 μL(20,000 个细胞)。对于 384 孔格式计数以估计细胞密度,在测定培养基中将密度调整为 4.44 x 105 个细胞/mL,每孔分配 18 μL(8,000 个细胞)。
- 将板在37°C,5%CO2 下孵育过夜或在最适合其生长的条件下孵育。
- 悬浮细胞电镀
- 在补充有 10% FBS 和 1x 恩杜拉嗪的 CO 2 非依赖性培养基中将细胞密度调节至2.22 x 105 个细胞/mL(原液试剂的 1:100 稀释度)。
- 将细胞板放入测定板中,每个实验和对照条件至少3个孔。对于 96 孔形式,每孔分配 90 μL(20,000 个细胞)。对于 384 孔形式,每孔分配 36 μL(8,000 个细胞)。
注意:对于具有低信号到背景(S:B)发光的悬浮细胞系,例如,当使用CRISPR池而不是克隆时,可以通过增加接种的细胞数量来增加发光,96孔格式高达100,000个细胞/孔,或384孔格式的40,000个细胞/孔。
- 使用表达LgBiT的HiBiT CRISPR细胞进行动力学降解测定
- 对于已经含有恩杜拉嗪的悬浮细胞,包括在2.2.的电镀步骤中,直接进入步骤2.3.3。对于贴壁细胞系,制备纳米格洛恩杜拉嗪溶液。对于 96 孔形式,通过将储备试剂 1:100 稀释到补充有 10% FBS 的CO 2 非依赖性培养基中来制备 1x 恩杜拉嗪溶液。对于 384 孔形式,通过将储备试剂 1:50 稀释到补充有 10% FBS 的CO 2 非依赖性培养基中来制备 2x 恩杜拉嗪溶液。
- 将恩杜拉嗪溶液添加到贴壁细胞的每个孔中。对于 96 孔格式的吸出培养基,加入 90 μL 的 1x 恩杜拉嗪溶液。对于 384 孔格式,将 18 μL 2x 恩杜拉嗪溶液加入 18 μL 细胞中。不要吸出培养基,因为降解测定是在384孔形式的培养基和CO2 独立培养基的50:50混合物中进行的。
- 将含有恩杜拉嗪的悬浮液或贴壁细胞板在37°C和5%CO 2的培养箱中孵育2.5 小时,以使发光平衡。
- 在 CO2 非依赖性培养基中制备 10 倍浓度的测试 PROTAC 滴定,并向 96 孔板的每个孔中加入 10 μL,或向 384 孔板的每个孔中加入 4 μL。对于功效未知的化合物,建议最高点的最终浓度为1-10μM作为起点。
- 在预平衡至37°C的光度计中收集0-48小时的发光动力学测量值。可以为每个实验定制测量的时间增量,但推荐的初始实验是每5-15分钟进行一次发光测量,持续24小时或所需的时间。
- 最终动力学测量后的可选细胞活力同孔多重分析
注意:该测定使用市售的CellTiter-Glo(CTG)试剂盒进行(参见 材料表)。- 将CTG试剂平衡至室温。
- 在动力学分析的最后一个时间点进行降解测量后,每孔板加入 100 μL(96 孔板)或 40 μL(384 孔板)试剂,并在 500-700 rpm(96 孔板)或微孔板涡旋混合器(384 孔板)上混合 5 分钟。
- 将板在室温下孵育30分钟,以允许细胞裂解和HiBiT信号的淬灭。
- 按照制造商的建议测量光度计上的总发光。
- 动力学降解曲线的定量
- 使用收集的动力学发光测量值,将每个PROTAC浓度的原始RLU标准化为每个时间点的重复平均DMSO条件,以解释游离呋喃嗪浓度随时间的变化。使用公式1计算分数RLU。
等式1: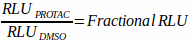
- 根据退化曲线,使用公式2将单分量指数衰减模型拟合到每条曲线的初始退化部分,直到数据达到平台的点。
注意:从拟合中排除前几个数据点可能会有所帮助,因为在观察到退化之前可能会有短暂的滞后。
等式 2: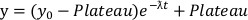
- 根据公式2,确定参数ƛ(代表降解速率常数)和平台(表示剩余蛋白质的最低量)。
- 计算Dmax,这是降解蛋白质的最大分数量,计算为1-平台。
- 绘制每种浓度的PROTAC的Dmax,以确定与时间无关的降解效力曲线。
- 确定 2.3.5 中图的 Dmax50 值以分析化合物的功效。
注意:要确定特定时间点的DC50,请绘制所选时间每种浓度的计算降解百分比。这可以指定为 DC 50 t=4 h 或 DC50 t=12 h。
- 使用收集的动力学发光测量值,将每个PROTAC浓度的原始RLU标准化为每个时间点的重复平均DMSO条件,以解释游离呋喃嗪浓度随时间的变化。使用公式1计算分数RLU。
结果
为了证明单浓度终点裂解降解分析,几种CDK靶蛋白;CDK2、CDK4、CDK7 和 CDK10 在 HEK293 细胞的 C 末端用 HiBiT 内源性标记,并用 1 μM 浓度的基于大脑的泛激酶 PROTAC、TL12-18654 处理(图 1A)。在不同时间点测量CDK蛋白的水平,并确定相对于DMSO对照的RLU分数(图1A)。每种CDK蛋白在响应化合物处理和不同时间点时表现出不同程度的降解(图1A)。为了了解CDK蛋白在蛋白质损失方面如何直接相互比较, 将图1A 中的分数RLU计算为总降解百分比,并在图 1B中的每个时间点绘制图。这表明,即使在早期时间点,即2或4小时,一些CDK家族成员也表现出高水平的退化,并随着时间的推移继续呈上升趋势(图1B)。
为了证明动力学降解分析,每个BET家族成员蛋白;BRD2、BRD3和BRD4在稳定表达LgBiT蛋白18的HEK293细胞的N末端用HiBiT内源性标记。然后用三种不同浓度的泛BET PROTACs处理这些;基于大脑的dBET650(图2A)和基于VHL的ARV-77141(图2B)。在24小时内收集动力学测量值,从每种浓度的曲线中,BET家族成员反应的差异显而易见。BRD2 在降解后化合物处理后启动更快速恢复反应的能力(图 2A,B)之前已在其他泛 BET PROTAC 中观察到,并且可能是由于与降解过程竞争的转录反馈反应18。
终点和动力学分析都可以通过完整的化合物剂量反应治疗来完成。图3所示是用四种不同分子胶化合物2,26,55,57稳定表达LgBiT蛋白的Ikaros/IKZF1-HiBiT CRISPR Jurkat细胞的动力学剂量反应降解曲线;来那度胺(图3A)、伊比度胺(CC-220)(图3B)、沙利度胺(图3C)和泊马度胺(图3D)。这些降解剂在化合物之间以及整个浓度系列中的降解响应方面表现出显着差异(图3)。
为了定量评估降解情况并对图3中的化合物进行排序,使用剂量反应曲线来计算关键降解参数,包括降解速率(图4A)、Dmax(图4B)和Dmax50值(图4B)。这些分析表明,伊比多胺(CC-220)和泊马度胺具有非常相似的快速初始降解速率(图4A),但伊比多胺(CC-220)具有最高的效力,如以前在正交研究中观察到的那样55,57(图4B)。由于伊比多米德表现出如此高的效力,并且所有测试的浓度都显示出大于50%的降解,因此为伊比多米德获得的Dmax50值代表了基于精确拟合数据的局限性的估计值。从图3C,D和图4B中的图表中可以看出,来那度胺和沙利度胺都不会降解Ikaros/IKZF1靶标,以达到测试的最高浓度。由于使用沙利度胺观察到的降解很少,降解迹线无法准确地拟合到指数衰减模型中,因此,该处理的降解速率无法量化。对于最有效的降解剂,伊比利亚多胺(CC-220)55,57(图4B)。细胞活力多重测定显示,对于测试浓度,细胞活力没有损失(图4C)。
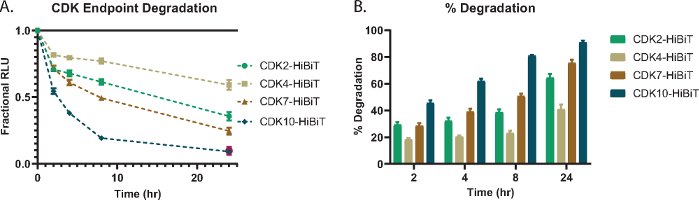
图 1:泛激酶 PROTAC 的 CDK 终点降解和毒性,TL12-18654。(A)选择一组通过CRISPR / Cas9与C末端的HiBiT融合的内源性CDK靶蛋白,并在2小时,4小时,8小时和24小时处理中用1μM TL12-186 PROTAC54 评估降解。值表示为相对于在每个时间点测量的 DMSO 控件的小数 RLU。误差线表示 3 次技术重复的平均值的 SD。(B)CDK靶蛋白组的降解百分比,计算自(A)表示在2,4,8和24小时时间点观察到的每个家族成员的降解量。误差线表示 3 次技术重复的平均值的 SD。 请点击此处查看此图的大图。
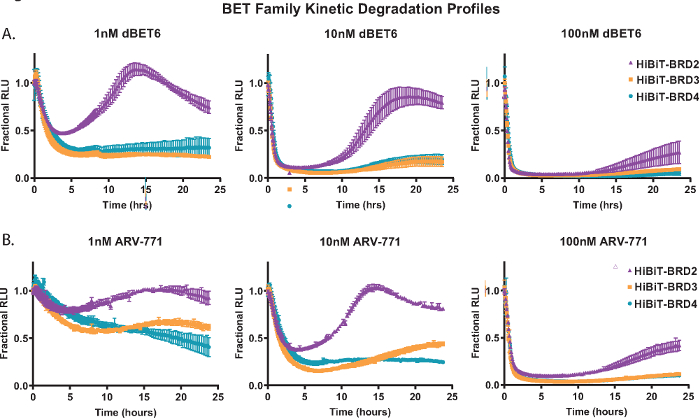
图 2:使用 BET 降解剂 dBET650 和 ARV-77141 分析 BET 家族成员的动力学降解选择性。内源性BET家族成员BRD2,BRD3和BRD4的动力学降解曲线,通过CRISPR / Cas9在N末端用HiBiT标记,处理单浓度为1 nM(左),10 nM(中)或100 nM(右)dBET650 (A)或ARV-77141 (B) PROTAC。值表示为在每个动力学时间点从 DMSO 控制计算的小数 RLU。误差线表示 4 次技术重复的平均值的 SD。 请点击此处查看此图的大图。
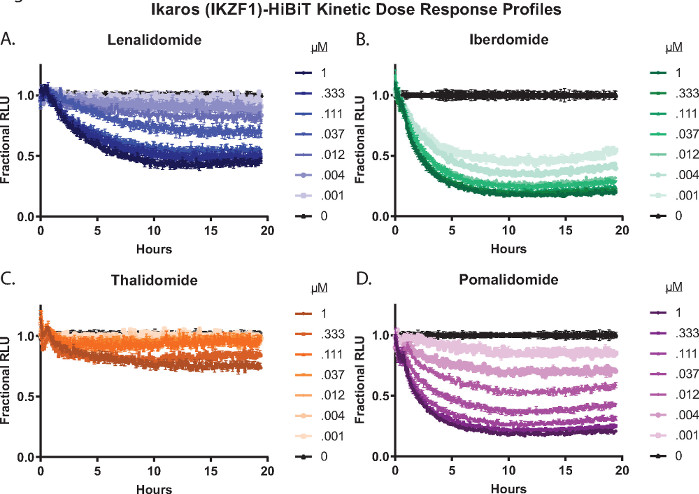
图 3:Ikaros/IKZF1-HiBiT 的活细胞动力学降解剂量反应曲线,分子胶面板2,26,55,57。稳定表达LgBiT蛋白的Jurkat细胞使用CRISPR / Cas9进行工程改造,用HiBiT肽标记Ikaros / IKZF1的C末端。用8点剂量反应浓度系列处理细胞,包括四种不同分子胶化合物2,26,55,57的DMSO:(A)来那度胺,(B)伊比那度胺(CC-220),(C)沙利度胺或(D)泊马度胺。每5分钟测量一次发光,共19.5小时。如步骤 2.4.1 中所述,将来自 (A-D) 的相对光单位 (RLU) 数据转换为分数 RLU,并绘制为时间函数。误差线表示 3 次技术重复的 SD。请点击此处查看此图的大图。
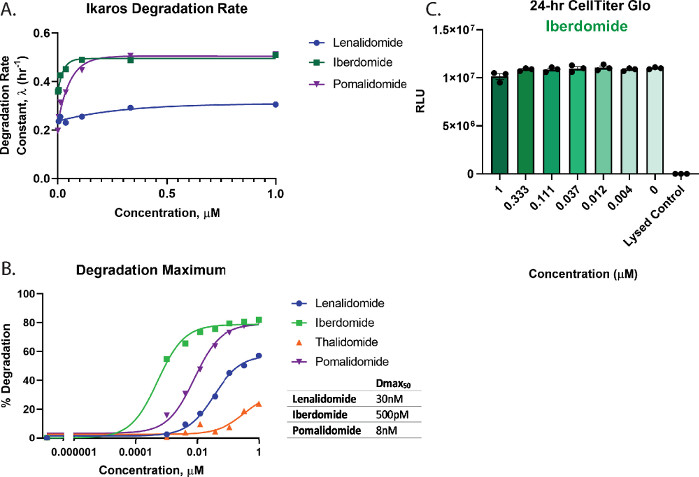
图 4:Ikaros/IKZF1-HiBiT 的降解速率和 Dmax50 计算,以及多重细胞健康测定。 使用图3中的动力学降解数据来计算定量降解参数。(A)在指示的分子胶化合物2,26,55,57的每个药物浓度下绘制降解速率和(B)降解最大值(Dmax)。(B)使用约束Hill斜率为1的剂量反应模型计算每种化合物的Dmax50值,该模型可用于对目标降解化合物进行排序。(C)在完成动力学降解测量后,使用图3B中的伊比多米(CC-220)55,57降解剂量响应进行细胞活力测定作为终点测量。误差线表示 3 次技术重复的 SD。请点击此处查看此图的大图。
讨论
我们在这里介绍了两种以终点裂解形式或活细胞动力学模式筛选降解化合物活性的方法。这些方法基于相同的发光测量原理,但提供了不同程度的细节和理解。任何一种方法的选择都可能取决于筛选目标和化合物库的大小。对于大型化合物筛选平台或主筛选以观察任何可检测到的降解,终点裂解筛选可提供灵敏高效的高通量兼容性,而其他终点方法(如蛋白质印迹或质谱)可能不切实际或难以适应14。这些筛选的起点可以用有限数量的浓度和时间点进行。推荐的初始测试浓度在100nM-10μM范围内,以解释初始降解剂具有低效力,较差的渗透性,或者在某些情况下具有高效化合物,存在钩效应。进一步建议至少测试两个不同的时间点,以确定4-6小时的早发降解和18-24小时的潜伏或持续降解。在4-6小时的时间范围内很容易观察到具有高降解效力和靶向机制的化合物,而仅在较晚的时间点观察到的降解或明显的蛋白质损失可能是由于多种机制。强烈建议在早期和晚期监测细胞活力,以便蛋白质损失可以与细胞死亡引起的损失解偶联。与任何类型的发光或荧光测定类似,文库中的化合物有可能干扰或抑制信号,因此,使用不相关的融合或替代方法来监测蛋白质水平的先导化合物的正交后续实验对于评估这些测定中RLU的损失与靶蛋白降解直接相关非常重要。
长时间以活细胞动力学形式进行筛选的能力在很大程度上依赖于测定信号到背景(S:B)。 影响S:B的因素包括靶蛋白本身的表达水平,可以跨越几个数量级,选择用于肽插入的细胞系中LgBiT表达的效率, 以及标记靶标在其各种天然复合物中互补的可用性。我们已经建立了一个一般截止要求,包括S:B为15,以成功测量使用恩杜拉嗪或维瓦嗪在动力学模式下的降解。S:B是通过测量在恩杜拉嗪或Vivazine活细胞底物存在下单独表达LgBiT的未编辑亲本细胞相对于共表达LgBiT的HiBiT编辑细胞的基线信号来确定的。Vivazine会产生更高的发光信号,但衰减速度比Endurazine快,并且可能将信号采集限制在24小时或更短时间内。此外,S:B也可能高度依赖于是使用CRISPR池还是克隆。对于更适合CRISPR / Cas9工程且具有高效率的细胞系中的靶标,编辑细胞的异质CRISPR池群可能具有足够的S:B用于动力学分析。对于更困难的细胞系中的靶标,其中通过CRISPR进行效率较低的基因组整合导致具有低S:B的池,分离CRISPR克隆对于富集编辑的群体并获得足够高的S:B以进行动力学分析可能是必要的。对于上述任何一种情况,如果恩杜拉嗪或维瓦嗪底物的 S:B 小于 15,建议进行终点裂解筛查。
为了更好地理解和表征化合物,包括使用定量参数测定降解曲线,推荐的筛选方法14,18在活细胞中进行实时动力学分析。与上面讨论的终点分析一样,初始动力学筛选可以在100nM-10μM范围内的有限数量的浓度下以高通量方式完成。在 384 孔格式中,可以在单个板上以一种浓度轻松筛选 100 多种化合物,一式三份。由此产生的降解曲线不仅可以为观察到的降解程度提供指导,还可以为降解速率、降解持续时间和蛋白质的潜在回收率提供指导14,18(图 2 和图 3)。降解曲线的形状也会产生有价值的信息。特异性和强效降解剂通常在几个小时内显示出靶蛋白的初始快速损失到平台期18,53,而其他机制(如转录反馈或化合物毒性)通常会导致蛋白质随着时间的推移而发生更线性的损失。终点裂解分析忽略了这些细节和细微差别,并且在24-48小时内进行实时分析,人们不必预测在一组新的或未知化合物中捕获真实Dmax的时间。
实时动力学还允许进行有效的剂量反应筛选,以更好地了解化合物功效、化合物浓度如何影响初始降解速率,并提供根据多个参数对化合物进行排名的可能性。降解效力的经典测量涉及基于表观降解最大值的特定时间点的 DC50 计算。相比之下,我们评估效力的动力学方法结合了每种浓度下的真实降解最大值,无论它何时在时间 18 中发生。我们将这种动力学降解效力的测量称为Dmax5018。以这种方式进行分析的化合物在较低浓度下可能更慢地引发降解,因此处理后需要更长的时间才能达到其Dmax。根据降解速率和Dmax对化合物进行排名可能特别有用。对于最有效的降解剂,这将进一步区分缓慢但有效的降解剂与快速有效的降解剂。利用HiBiT CRISPR细胞系进行裂解和活细胞动力学筛选是强大的方法,可以更全面地了解靶向蛋白质降解、化合物功能,并通过增强关键降解参数实现从初始活性评估到下游化学优化的筛选过程。
披露声明
Promega Corporation是商业所有者,通过转让HiBiT和NanoLuc技术和应用的专利。
致谢
K.M.R、S.D.M、M.U. 和 D.L.D 都是 Promega Corporation 的员工
材料
| Name | Company | Catalog Number | Comments |
| CellTiter-Glo 2.0 reagent | Promega | G9241 | Cell Viability luminescent assay |
| CellTiter-Fluor Cell Viability Assay | Promega | G6080 | Cell Viability fluorescent assay |
| CO2-independent medium | ThermoFisher | 18045-088 | Cell culture |
| DMSO | Sigma Aldrich | D2650 | For compound dilution and control |
| DPBS | Gibco | 14190 | Cell culture |
| Fetal Bovine Serum | Seradigm | 89510-194 | Cell culture |
| HEK293 LgBiT stable cell line | Promega | N2672 | For complementation with HiBiT to generate luminescence |
| HiBiT CRISPR mammalian cell line | Promega | https://www.promega.com/crispr-tpd | |
| Hygromycin B solution | Gibco | 10-687-010 | Cell culture |
| LgBiT BacMam | Promega | CS1956C01 | For complementation with HiBiT to generate luminescence |
| LgBiT Expression Vector | Promega | N2681 | For complementation with HiBiT to generate luminescence |
| Luminometer Plate Reader | Luminomenter capable of measuring luminescence and fluorescence (e.g. GloMax Discover System, Promega GM3000) | ||
| NanoGlo Endurazine live cell substrate | Promega | N2570 | Kinetic HiBiT reagent |
| NanoGlo Vivazine live cell substrate | Promega | N2580 | Kinetic HiBiT reagent |
| NanoGlo HiBiT Lytic Detection system | Promega | N3030 | Enpoint lytic HiBiT reagent |
| Opti-MEM Reduced Serum Medium, no phenol red (ThermoFisher) | ThermoFisher | 11058-021 | Cell culture |
| Tissue culture plates, white, 96 well plate | Costar | 3917 | Cell culture |
| Tissue culture plates, white, 384 well plate | Corning | 3570 | Cell culture |
| Trypsin/EDTA | Gibco | 25300 | Cell culture |
参考文献
- Burslem, G. M., Crews, C. M. Proteolysis-targeting chimeras as therapeutics and tools for biological discovery. Cell. 181 (1), 102-114 (2020).
- Chamberlain, P. P., Hamann, L. G. Development of targeted protein degradation therapeutics. Nature Chemical Biology. 15 (10), 937-944 (2019).
- Churcher, I. Protac-induced protein degradation in drug discovery: Breaking the rules or just making new ones. Journal of Medicinal Chemistry. 61 (2), 444-452 (2018).
- Ciulli, A., Farnaby, W. Protein degradation for drug discovery. Drug Discovery Today: Technologies. 31, 1-3 (2019).
- Crews, C. M. Inducing protein degradation as a therapeutic strategy. Journal of Medicinal Chemistry. 61 (2), 403-404 (2018).
- Cromm, P. M., Crews, C. M. Targeted protein degradation: from chemical biology to Drug Discovery. Cell Chemical Biology. 24 (9), 1181-1190 (2017).
- Deshaies, R. J. Protein degradation: Prime time for PROTACs. Nature Chemical Biology. 11 (9), 634-635 (2015).
- Lai, A. C., Crews, C. M. Induced protein degradation: an emerging drug discovery paradigm. Nature Reviews Drug Discovery. 16 (2), 101-114 (2017).
- Ottis, P., Crews, C. M. Proteolysis-targeting chimeras: Induced protein degradation as a therapeutic strategy. ACS Chemical Biology. 12 (4), 892-898 (2017).
- Wu, T., et al. Targeted protein degradation as a powerful research tool in basic biology and drug target discovery. Nature Structural and Molecular Biology. 27, 605-614 (2020).
- Hanan, E. J., et al. Monomeric targeted protein degraders. Journal of Medicinal Chemistry. , (2020).
- Collins, I., Wang, H., Caldwell, J. J., Chopra, R. Chemical approaches to targeted protein degradation through modulation of the ubiquitin-proteasome pathway. Biochemical Journal. 474 (7), 1127-1147 (2017).
- Carmony, K. C., Kim, K. B. PROTAC-induced proteolytic targeting. Methods in Molecular Biology. 832, 627-638 (2012).
- Daniels, D. L., Riching, K. M., Urh, M. Monitoring and deciphering protein degradation pathways inside cells. Drug Discovery Today: Technologies. 31, 61-68 (2019).
- Gu, S., Cui, D., Chen, X., Xiong, X., Zhao, Y. PROTACs: An emerging targeting technique for protein degradation in drug discovery. Bioessays. 40 (4), 1700247(2018).
- Neklesa, T. K., Winkler, J. D., Crews, C. M. Targeted protein degradation by PROTACs. Pharmacology and Therapy. 174, 138-144 (2017).
- Raina, K., Crews, C. M. Targeted protein knockdown using small molecule degraders. Current Opinion in Chemical Biology. 39, 46-53 (2017).
- Riching, K. M., et al. Quantitative live-cell kinetic degradation and mechanistic profiling of PROTAC mode of action. ACS Chemical Biology. 13 (9), 2758-2770 (2018).
- Schwinn, M. K., et al. CRISPR-mediated tagging of endogenous proteins with a luminescent peptide. ACS Chemical Biology. 13 (2), 467-474 (2018).
- Schwinn, M. K., Steffen, L. S., Zimmerman, K., Wood, K. V., Machleidt, T. A simple and scalable strategy for analysis of endogenous protein dynamics. Science Reports. 10 (1), 8953(2020).
- Bensimon, A., et al. Targeted degradation of SLC transporters reveals amenability of multi-pass transmembrane proteins to ligand-induced proteolysis. Cell Chemical Biology. 27 (6), 728-739 (2020).
- Bondeson, D. P., et al. Lessons in PROTAC design from selective degradation with a promiscuous warhead. Cell Chemical Biology. 25 (1), 78-87 (2018).
- Buckley, D. L., et al. HaloPROTACS: Use of small molecule PROTACs to induce degradation of HaloTag fusion proteins. ACS Chemical Biology. 10 (8), 1831-1837 (2015).
- Bulatov, E., Ciulli, A. Targeting Cullin-RING E3 ubiquitin ligases for drug discovery: structure, assembly and small-molecule modulation. Biochemical Journal. 467 (3), 365-386 (2015).
- Burslem, G. M., et al. The advantages of targeted protein degradation over inhibition: An RTK case study. Cell Chemical Biology. 25 (1), 67-77 (2018).
- Chamberlain, P. P., et al. Evolution of cereblon-mediated protein degradation as a therapeutic modality. ACS Medicinal Chemistry Letters. 10 (12), 1592-1602 (2019).
- Crew, A. P., et al. Identification and characterization of Von Hippel-Lindau-recruiting Proteolysis Targeting Chimeras (PROTACs) of TANK-Binding Kinase 1. Journal of Medicinal Chemistry. 61 (2), 583-598 (2018).
- DeMars, K. M., Yang, C., Castro-Rivera, C. I., Candelario-Jalil, E. Selective degradation of BET proteins with dBET1, a proteolysis-targeting chimera, potently reduces pro-inflammatory responses in lipopolysaccharide-activated microglia. Biochemical and Biophysical Research Communication. 497 (1), 410-415 (2018).
- Erb, M. A., et al. Transcription control by the ENL YEATS domain in acute leukaemia. Nature. 543 (7644), 270-274 (2017).
- Farnaby, W., et al. BAF complex vulnerabilities in cancer demonstrated via structure-based PROTAC design. Nature Chemical Biology. 15 (7), 672-680 (2019).
- Gadd, M. S., et al. Structural basis of PROTAC cooperative recognition for selective protein degradation. Nature Chemical Biology. 13 (5), 514-521 (2017).
- Gechijian, L. N., et al. Functional TRIM24 degrader via conjugation of ineffectual bromodomain and VHL ligands. Nature Chemical Biology. 14 (4), 405-412 (2018).
- Gustafson, J. L., et al. Small-Molecule-Mediated Degradation of the Androgen Receptor through Hydrophobic Tagging. Angewandte Chemie International Edition England. 54 (33), 9659-9662 (2015).
- Kerres, N., et al. Chemically induced degradation of the oncogenic transcription factor BCL6. Cell Reports. 20 (12), 2860-2875 (2017).
- Lohbeck, J., Miller, A. K. Practical synthesis of a phthalimide-based Cereblon ligand to enable PROTAC development. Bioorganic and Medicinal Chemistry Letters. 26 (21), 5260-5262 (2016).
- Lu, J., et al. Hijacking the E3 ubiquitin ligase cereblon to efficiently target BRD4. Chemical Biology. 22 (6), 755-763 (2015).
- Lu, M., et al. Discovery of a Keap1-dependent peptide PROTAC to knockdown Tau by ubiquitination-proteasome degradation pathway. Eurupean Journal of Medicinal Chemistry. 146, 251-259 (2018).
- Nabet, B., et al. The dTAG system for immediate and target-specific protein degradation. Nature Chemical Biology. 14 (5), 431-441 (2018).
- Nowak, R. P., et al. Plasticity in binding confers selectivity in ligand-induced protein degradation. Nature Chemical Biology. 14, 706-714 (2018).
- Powell, C. E., et al. Chemically induced degradation of Anaplastic Lymphoma Kinase (ALK). Journal of Medicinal Chemistry. 61 (9), 4249-4255 (2018).
- Raina, K., et al. PROTAC-induced BET protein degradation as a therapy for castration-resistant prostate cancer. Proceedings of National Academy of Science U. S. A. 113 (26), 7124-7129 (2016).
- Sakamoto, K. M., et al. Protacs: chimeric molecules that target proteins to the Skp1-Cullin-F box complex for ubiquitination and degradation. Proceeding National Academy Science. 98 (15), 8554-8559 (2001).
- Sakamoto, K. M., et al. Development of Protacs to target cancer-promoting proteins for ubiquitination and degradation. Molecular Cell Proteomics. 2 (12), 1350-1358 (2003).
- Schiedel, M., et al. Chemically induced degradation of Sirtuin 2 (Sirt2) by a proteolysis targeting chimera (PROTAC) based on sirtuin rearranging ligands (SirReals). Journal of Medicinal Chemistry. 61 (2), 482-491 (2018).
- Slabicki, M., et al. The CDK inhibitor CR8 acts as a molecular glue degrader that depletes cyclin K. Nature. , (2020).
- Smith, B. E., et al. Differential PROTAC substrate specificity dictated by orientation of recruited E3 ligase. Nature Communications. 10 (1), 131(2019).
- Sun, B., et al. BET protein proteolysis targeting chimera (PROTAC) exerts potent lethal activity against mantle cell lymphoma cells. Leukemia. 32 (2), 343-352 (2018).
- Tong, B., et al. A Nimbolide-based kinase degrader preferentially degrades oncogenic BCR-ABL. ACS Chemical Biology. 15 (7), 1788-1794 (2020).
- Winter, G. E., et al. Drug Development. Phthalimide conjugation as a strategy for in vivo target protein degradation. Science. 348 (6241), 1376-1381 (2015).
- Winter, G. E., et al. BET Bromodomain proteins function as master transcription elongation factors independent of CDK9 recruitment. Molecular Cell. 67 (1), 5-18 (2017).
- Zengerle, M., Chan, K. H., Ciulli, A. Selective Small Molecule Induced Degradation of the BET Bromodomain Protein BRD4. ACS Chemical Biology. 10 (8), 1770-1777 (2015).
- Zhang, C., et al. Proteolysis targeting chimeras (PROTACs) of anaplastic lymphoma kinase (ALK). European Journal of Medicinal Chemistry. 151, 304-314 (2018).
- Zoppi, V., et al. Iterative design and optimization of initially inactive proteolysis targeting chimeras (PROTACs) identify VZ185 as a potent, fast, and selective von Hippel-Lindau (VHL) based dual degrader probe of BRD9 and BRD7. Journal of Medicinal Chemistry. 62 (2), 699-726 (2019).
- Huang, H. T., et al. A chemoproteomic approach to query the degradable kinome using a multi-kinase degrader. Cell Chemical Biology. 25 (1), 88-99 (2018).
- Bjorklund, C. C., et al. Iberdomide (CC-220) is a potent cereblon E3 ligase modulator with antitumor and immunostimulatory activities in lenalidomide- and pomalidomide-resistant multiple myeloma cells with dysregulated CRBN. Leukemia. 34 (4), 1197-1201 (2020).
- Matyskiela, M. E., et al. SALL4 mediates teratogenicity as a thalidomide-dependent cereblon substrate. Nature Chemical Biology. 14 (10), 981-987 (2018).
- Matyskiela, M. E., et al. A cereblon modulator (CC-220) with improved degradation of Ikaros and Aiolos. Journal of Medicinal Chemistry. 61 (2), 535-542 (2018).
- Bussiere, D. E., et al. Structural basis of indisulam-mediated RBM39 recruitment to DCAF15 E3 ligase complex. Nature Chemical Biology. 16 (1), 15-23 (2020).
- Du, X., et al. Structural basis and kinetic pathway of RBM39 recruitment to DCAF15 by a sulfonamide molecular glue E7820. Structure. 27 (11), 1625-1633 (2019).
- Ting, T. C., et al. Aryl sulfonamides degrade RBM39 and RBM23 by recruitment to CRL4-DCAF15. Cell Reports. 29 (6), 1499-1510 (2019).
- Hughes, S. J., Ciulli, A. Molecular recognition of ternary complexes: a new dimension in the structure-guided design of chemical degraders. Essays in Biochemistry. 61 (5), 505-516 (2017).
- Schapira, M., Calabrese, M. F., Bullock, A. N., Crews, C. M. Targeted protein degradation: expanding the toolbox. Nature Review Drug Discovery. 18 (12), 949-963 (2019).
- Dixon, A. S., et al. NanoLuc complementation reporter optimized for accurate measurement of protein interactions in cells. ACS Chemical Biology. 11 (2), 400-408 (2016).
- Gilan, O., et al. Selective targeting of BD1 and BD2 of the BET proteins in cancer and immuno-inflammation. Science. 368 (6489), 387-394 (2020).
- Oh-Hashi, K., Furuta, E., Fujimura, K., Hirata, Y. Application of a novel HiBiT peptide tag for monitoring ATF4 protein expression in Neuro2a cells. Biochemical Biophysical Report. 12, 40-45 (2017).
- Ottis, P., et al. Cellular resistance mechanisms to targeted protein degradation converge toward impairment of the engaged ubiquitin transfer pathway. ACS Chemical Biology. 14 (10), 2215-2223 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。