需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用胺还原甲基化进行样品制备和相对定量,用于肽学研究
摘要
本文介绍了一种基于热灭活的样品制备方法,以保存内源性肽,避免死后降解,然后使用同位素标记和LC-MS进行相对定量。
摘要
肽学可以定义为生物样品中肽的定性和定量分析。其主要应用包括鉴定疾病或环境应激的肽生物标志物,鉴定神经肽,激素和生物活性细胞内肽,从蛋白质水解物中发现抗菌和营养保健肽,并可用于研究以了解蛋白水解过程。最近在样品制备,分离方法,质谱技术和与蛋白质测序相关的计算工具方面的进步有助于增加已鉴定的肽数量和所表征的肽。肽研究经常分析细胞中自然产生的肽。这里描述了一种基于热灭活的样品制备方案,其消除了蛋白酶活性,并且在温和的条件下提取,因此没有肽键裂解。此外,还显示了通过胺的还原甲基化使用稳定同位素标记的肽的相对定量。这种标记方法具有一些优点,因为试剂是市售的,与其他试剂相比价格低廉,化学稳定,并且允许在单次LC-MS运行中分析多达五个样品。
引言
"组学"科学的特点是对分子集进行深入分析,例如DNA,RNA,蛋白质,肽,代谢物等。这些生成的大规模数据集(基因组学、转录组学、蛋白质组学、肽学、代谢组学等)彻底改变了生物学,并导致了对生物过程的高级理解1。肽学一词在20世纪初 开始引入,一些作者将其称为蛋白质组学的一个分支2。然而,肽学具有独特的特殊性,其主要兴趣是研究细胞过程中自然产生的肽含量,以及这些分子的生物活性的表征3,4。
最初,生物活性肽研究仅限于通过Edman降解和放射免疫测定的神经肽和激素肽。然而,这些技术不允许进行全局分析,这取决于高浓度中每种肽的分离,抗体产生的时间,以及交叉反应性的可能性5。
只有在液相色谱耦合质谱(LC-MS)和基因组项目取得多项进展后,肽学分析才成为可能,这些项目为蛋白质组学/肽学研究提供了全面的数据池6,7。此外,需要建立针对肽酶的特定肽提取方案,因为第一批分析大脑样本中全球神经肽的研究表明,检测受到蛋白质大规模降解的影响,蛋白质在死后1分钟后主要发生在该组织中。这些肽片段的存在掩盖了神经肽信号,并不代表 体内的肽组。这个问题主要通过使用微波照射对蛋白酶进行快速加热灭活来解决,这大大减少了这些伪影片段的存在,不仅允许鉴定神经肽片段,而且揭示了来自胞质,线粒体和核蛋白的一组肽的存在,与降解6,8,9不同。
这些方法论程序允许肽组扩展到众所周知的神经肽之外,其中主要由蛋白酶体作用产生的数百种细胞内肽已在酵母10,斑马鱼11,啮齿动物组织12和人类细胞中鉴定出来13。数十种这些细胞内肽已被广泛证明具有生物学和药理活性14,15。此外,这些肽可用作疾病生物标志物,并可能具有临床意义,如颅内囊性动脉瘤患者的脑脊液16所示。
目前,除了肽序列的鉴定外,还可以通过质谱法获得绝对和相对定量的数据。在绝对定量中,将生物样品中的肽水平与合成标准品进行比较,而在相对定量中,比较两个或多个样品中的肽水平17。可以使用以下方法进行相对定量:1)"无标签"18;2) 体内 代谢标记或3)化学标记。最后两个是基于使用掺入肽中的稳定同位素形式19,20。在无标记分析中,通过考虑LC-MS18期间的信号强度(光谱计数)来估计肽水平。然而,同位素标记可以获得更准确的肽相对水平。
许多肽研究使用丁酸三甲基铵(TMAB)标记试剂作为化学标记,最近,胺的还原甲基化(RMA)与氘化和非氘代形式的甲醛和氰基硼氢化钠试剂已被使用11,21,22。然而,TMAB标签不是市售的,合成过程非常费力。另一方面,在RMA中,试剂是市售的,与其他标记相比便宜,程序易于执行,并且标记的肽稳定23,24。
RMA的使用涉及通过允许肽与甲醛反应来形成希夫碱,然后通过氰基硼氢化物进行还原反应。该反应引起N端和赖氨酸侧链和单甲基化N端脯氨酸上的游离氨基的二甲基化。脯氨酸残基在N端通常很少见,几乎所有在N端具有游离胺的肽都标有两个甲基23,24,25。
研究方案
以下肽提取和还原甲基化程序改编自先前发表的程序24,25,26,27。该协议遵循国家动物实验控制委员会(CONCEA)的指导方针,并得到圣保罗州立大学生物科学研究所动物使用伦理委员会(CEUA)的批准。协议步骤如图 1 所示。
注意:在超纯水中准备所有水溶液。
1. 肽提取
- 细胞培养
- 在含有15%胎牛血清和1%青霉素的Dulbecco改性Eagle培养基中,在37°C下在5%CO 2 下培养SHSY5Y细胞。
- 每个样品使用2-3个板。将细胞培养至100%汇合。
- 预定处理后,用磷酸盐缓冲盐水洗涤细胞两次。接下来,加入10mL磷酸盐缓冲盐水,刮擦细胞并收集到15mL管中。
- 以800× g 离心5分钟,除去上清液。将沉淀重悬于1mL超纯化去离子水中,温度为80°C。
- 将管的内容物(细胞裂解物)转移到2mL微量离心管中。
- 动物组织(主要是神经组织):
- 用MS 222(100 mg / L)的致死剂量麻醉野生型雄性成年 Danio rerio (斑马鱼),并立即对其进行8秒的微波辐射以灭活肽酶和蛋白酶。
注:可以使用家用型微波炉。900 W微波在全功率下使用8至10秒。使用的微波必须能够在10秒内将大脑温度提高到>80°C。通过将组织置于微波中的相同位置,样品之间加热的再现性也将受益。 - 热灭活后,将整个大脑收集在2mL微量离心管中,并在-80°C下冷冻直至分析。
- 将组织样品重悬于80°C的1mL超纯去离子水中。 使用探针使用30脉冲(4 Hz)1秒对组织进行超声处理。
注意:对于肝脏和肾脏组织,使用机械均质器以10,000-30,000 rpm的速度运行20秒。对于肌肉组织,使用瓷坩埚和研杵在液氮中研磨组织。以下步骤与细胞裂解物或匀浆组织相同。
- 用MS 222(100 mg / L)的致死剂量麻醉野生型雄性成年 Danio rerio (斑马鱼),并立即对其进行8秒的微波辐射以灭活肽酶和蛋白酶。
- 将细胞裂解物或匀浆组织在80°C下孵育20分钟。接下来,在冰上冷却10-30分钟。
- 每1 mL样品体积加入10μL1M HCl储备溶液,以获得10mM的最终浓度。通过涡旋混合20秒,并在冰上进一步孵育15分钟。
注意:在酸化之前,请确保样品完全冷却,以避免在高温下酸化引起的肽键断裂。 - 在4°C下以12,000× g 离心细胞裂解物或匀浆组织15分钟。将上清液收集在低结合蛋白微量离心管中,并将其储存在-80°C。
- 通过加水清洁超滤设备(10 kDa截止过滤器),并以2300× g 离心3分钟。再重复此步骤两次。
- 将上清液置于预洗的10 kDa截止过滤器中,并在4°C下以2,300×g离心50分钟在再冷藏离心机中离心。流出物代表肽提取物。
- 根据制造商的说明,使用乙腈(ACN)和三氟乙酸(TFA)溶液在反相净化柱上对样品进行脱盐,如下所述:
- 用1 mL 100%ACN平衡色谱柱。
- 用1 mL 5%ACN溶液和0.1%TFA洗涤色谱柱。
- 在色谱柱中加载样品的完整体积。
- 用1 mL 5%ACN溶液和0.1%TFA洗涤色谱柱
- 在蛋白质低结合微量离心管中,用1.8 mL 100%ACN和0.15%TFA溶液洗脱柱中的肽。
- 在真空离心机中将样品完全干燥。设置有机溶剂的浓缩方法和温度在30°C。 在显示屏上监控浓缩时间。
- 将样品储存在−80°C,直到下一步。
2. 用荧光胺定量肽
注意:如前所述,肽的量可以使用pH值为6.8的荧光胺估计11,28。该方法包括荧光胺分子附着在赖氨酸(K)残基和/或肽的N端中存在的伯胺上。该反应在pH 6.8下进行,以保证荧光胺仅与肽的氨基反应,而不与游离氨基酸反应。荧光胺是通过使用荧光计在激发波长为370nm和发射波长为480nm处测量的。
- 制备不同浓度的标准肽(0.05,0.1,0.15,0.2,0.3,0.5和0.7μg/ μL),并将等分试样储存在-20°C。
注意:建议使用肽5A(LTLRTKL),因为它具有已知的组成和浓度。 - 在丙酮中制备荧光胺储备溶液(0.3mg / mL)。在微量离心管(1 mL)中快速等分试样,使用parafilm密封,并在黑暗中储存在-20°C。
- 在pH 6.8下制备0.2M磷酸盐缓冲液(PB)。
注意:通过向250毫升水中加入0.1 M磷酸盐缓冲液pH 6.8(26.85 mL Na2HPO3 1M)和0.1 M磷酸盐缓冲液pH 6.8(23.15 mL NaH2PO3 1M)来制备0.2 M PB - 将肽样品重悬于100-200μL超纯水中。
- 将2.5μL标准肽浓度和样品移液到白色96孔板上,一式三份进行荧光测定。加入25μL0.2M磷酸盐缓冲液。
- 用多通道移液器加入12.5μL荧光素。在轨道旋转器振荡器上轻轻匀浆1分钟。
- 接下来,用多通道移液器加入110μL水以停止反应。
注意:将荧光胺储备溶液和超纯水转移到两个储液槽中,用多通道移液器移液这些溶液。 - 在光谱荧光计上调整以下读数参数:从顶部读取样品,激发波长为370 nm,发射波长为480 nm。
- 读取光谱荧光计上的板。
3. 胺标记的还原甲基化
注意:这种同位素标记方法基于胺基团与氘化和非氘代形式的甲醛和氰基硼氢化试剂的二甲基化。该反应的最终产物在每个可用标记位点(赖氨酸或N-末端)向每个肽的最终质量中加入28 Da,30 Da,32 Da,34 Da或36 Da。该反应在用在MS光谱中观察到的不同形式标记的肽中产生m / z差异(表1)。
注意:应使用适当的安全设备来处理这些化合物,并应注意尽量减少接触。使用甲醛和氰基硼氢化钠试剂的程序应在通风橱中进行,因为它们毒性很大(包括称量氰基硼氢化钠)。在淬火反应和酸化过程中,可能产生有毒气体(氰化氢)。
- 在手术当天用超纯水从储备或试剂中准备以下新鲜溶液:
- 将37%甲醛(CH2O)的储备稀释至4%。
- 将20%甲醛氘代(CD2O)的储备稀释至4%。
- 将20%氘化C13甲醛(13CD2O)的储备稀释至4%。
- 准备0.6 M NaBH3CN。
- 准备0.6 M NaBD3CN。
- 准备1%碳酸氢铵的溶液。
- 准备5%甲酸溶液。
注意:关于肽的相对定量,由于可以根据所使用的标记数量执行不同的实验方案,因此在化学标记过程中需要注意。建议将标签的小等分试样与要标记的相应样品分开,以减少在样品管中添加错误试剂时人为错误的可能性。
- 准备含有高达25μg肽的每个样品。样品不得含有Tris或碳酸氢铵。
注:对于100 μL体积中的每个样品,下面描述的量足够。
在通风橱中继续执行步骤3.3-3.8。 - 向样品中加入1 M TEAB的1/10体积 (溶液的最终浓度为100mM的TEAB)。用pH指示纸检查pH值;它必须介于 5-8 之间。如果需要,用HCl或NaOH进行调整。
- 根据既定的标记方案加入4μL非氘代,氘代甲醛或C13 氘代甲醛。通过涡旋混合5秒。
- 根据既定的标记方案加入4μLNaBH3CN(0.6 M)或NaBD3CN(0.6 M)。通过涡旋混合5秒。
- 在室温下在通风橱中孵育2小时,每30分钟混合一次。
- 重复步骤 3.4 和 3.5。将样品在通风橱中室温孵育过夜。
- 加入16μL碳酸氢铵(1%)并通过涡旋混合。将样品放在冰上,加入8μL甲酸(5%),并通过涡旋混合5 s。
- 将样品混合,将pH值调节至2-4,并在反相净化柱上对组合样品进行脱盐,如步骤1.8中所述。
- 在真空离心机中将样品完全干燥。设置有机溶剂的浓缩方法和温度在30°C。 在显示屏上监控浓缩时间。
- 将样品储存在-20°C。
4. 液相色谱和质谱
- 使用纳米HPLC系统与与甲基标签兼容的MS仪器进行LC-MS分析。
注:各种MS仪器都与RMA标签兼容。与Orbitrap耦合的nanoHPLC系统通常用于通过纳米电喷雾离子源进行LC-MS分析。首先,将样品加载到前列中,肽在分析柱中分离。肽的洗脱在90分钟内使用5%-45%乙腈的线性梯度在0.1%甲酸中进行,流量为200nL / min。质谱仪设置为在数据相关模式下工作。每次全扫描以10-30 eV,2.3 KV的强度获得,然后选择十个最高峰进行碰撞诱导解离(CID)碎片化。在离子阱上将注入时间设置为100 ms,傅里叶变换(FT)-MS注入在m/z 300-1800下以1000 ms 30,000的分辨率固定。至少使用 5000 个计数和 70 s 的动态排除来执行碎片扫描。
5. 肽的相对定量
注:MS光谱在质谱仪软件中进行分析。在MS光谱中鉴定具有不同标签的标记肽的峰组。相对定量是通过每个同位素峰的强度计算的。将每个治疗组与相应的对照组进行比较。
- 右键单击原始样本文件以打开频谱分析软件。将保留时间 (RT) 和 MS 光谱 (EM) 色谱图分别加载到顶部和底部的两个选项卡中。
- 在软件工具栏中的 "显示" 和" 质量 "选项图标上依次单击鼠标右键,然后将质量精度设置为四位小数。
- 将鼠标光标放在 RT 选项卡上的任意位置。查找要分析的相应离子的保留时间,然后单击鼠标右键。所选时间的 MS 光谱将自动显示在 EM 选项卡中。
- 将鼠标光标放在 EM 选项卡上的任意位置。寻找要分析的离子。
- 右键单击并按住这些离子附近的左侧相邻区域。然后,在所需范围内向右拖动鼠标以缩放感兴趣区域。
- 将鼠标保持在EM选项卡上,然后单击键盘右侧或左侧箭头以定义要分析的离子范围。
- 将鼠标光标再次定位在所需时间间隔的开头的 RT 选项卡上。
- 右键单击并拖动鼠标,直到所选时间值。离开按钮。离子的累积强度将自动显示在EM选项卡上。
- 在电子表格上收集 m/z、z 和离子强度数据。
注意:未添加甲基的每种肽的单同位素质量由以下公式计算:
质量未修饰肽 = (m/z a x z) - (C a x T) - (1.008 x z)
m/ zais是观察到的具有不同标签组合(a = 1,2,3,4或5,对应于样品数)的每种肽的单同位素峰的质量电荷值。
z 是充电状态。
Ca是一对甲基的同位素质量:
当a=1时,Ca = 28.0313(两个CH3基团的净添加到伯胺中)
对于 a=2,Ca = 30.0439 对于两个 CHD2 组
对于 a=3,Ca = 32.0564 对于两个 CD2H 组
对于 a=4,Ca= 34.0690 对于两个 CD3 组
当 a=5 时,Ca = 36.0757 对于两个 13 个 CD3 组
T 是掺入肽中的甲基对数。当使用五个标签时,这可以从以下公式计算: T=z*(m/z5 - m/z1)/8。对于含有单一伯胺的肽,因此仅用两个甲基标记,当使用相邻标记时,MS光谱上存在峰重叠。每个标记肽的峰值强度可以使用Tashima和Fricker25描述的方程进行校正。
6. 肽鉴定
- 为了鉴定肽,使用数据库搜索引擎分析MS / MS数据29,30。
- 要使用诱饵融合方法计算误发现率 (FDR),请搜索诱饵数据库。
注:一般使用的搜索参数无酶特异性;前驱体质量公差设定为15-50 ppm;片段离子质量公差为0.5 Da;可变修饰:来自Lys残基和肽同位素甲基化标记(L1(+28),L2(+30),L3(+32),L4(+34)和L5(+36))的N端的反应胺,氧化蛋氨酸(+15.99 Da)和乙酰化(+42.01 Da)。 - 然后,按局部置信度的平均值对肽进行排序,以选择最佳光谱进行注释,并按FDR≤5%过滤它们。
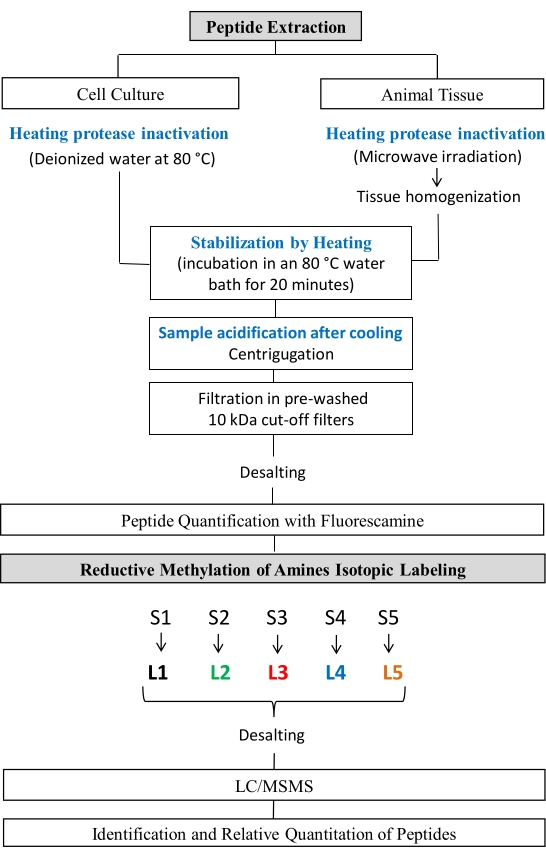
图1:肽研究工作流程。 肽提取和胺还原甲基化的步骤。 请点击此处查看此图的放大版本。
结果
从质谱仪上进行的运行中获得的结果存储在原始数据文件中,可以在质谱仪软件中打开。在MS光谱中,可以根据所使用的标记方案观察代表标记肽的峰组,范围从2-5个标记。例如,在 图2中,在色谱时间内检测到的峰对在实验中表示,其中在同一次运行中,在两个不同的样品中仅使用了两个同位素标记。 图3 显示了在每次LC-MS运行中使用3个和4个不同标?...
讨论
在大多数肽学研究中,毫无疑问,关键步骤之一是应仔细进行的样品制备,以避免在死后几分钟后存在蛋白酶产生的肽片段。对从非微波样品制备的脑提取物的初步研究表明,10 kDa微滤液中存在大量的蛋白质片段。已经描述了避免蛋白质降解肽光谱的不同方法:聚焦微波照射动物牺牲6,8,冷冻器解剖,然后煮沸提取缓冲液31, ...
披露声明
不存在相互竞争的经济利益。
致谢
本文所述技术的开发和使用得到了巴西国家研究委员会420811/2018-4(LMC)赠款的支持;圣保罗国家保护基金会(www.fapesp.br)赠款2019/16023-6(LMC),2019/17433-3(LOF)和21/01286-1(MEME)。资助者在研究设计,数据收集和分析,发表决定或准备文章方面没有任何作用。
材料
| Name | Company | Catalog Number | Comments |
| 10 kDa cut-off filters | Merck Millipore | UFC801024 | Amicon Ultra-4, PLGC Ultracel-PL Membrane, 10 kDa |
| Acetone | Sigma-Aldrich | 179124 | |
| Acetonitrile | Sigma-Aldrich | 1000291000 | |
| Ammonium bicarbonate | Sigma-Aldrich | 11213 | |
| analytical column (EASY-Column) | EASY-Column | (SC200) | 10 cm, ID75 µm, 3 µm, C18-A2 |
| Ethyl 3-aminobenzoate methanesulfonate | Sigma-Aldrich | E10521 | MS-222 |
| Fluorescamine | Sigma-Aldrich | F9015 | |
| Formaldehyde solution | Sigma-Aldrich | 252549 | |
| Formaldehyde-13C, d2, solution | Sigma-Aldrich | 596388 | |
| Formaldehyde-d2 solution | Sigma-Aldrich | 492620 | |
| Formic acid | Sigma-Aldrich | 33015 | |
| Fume hood | Quimis | Q216 | |
| Hydrochloric acid - HCl | Sigma-Aldrich | 258148 | |
| LoBind-Protein retention tubes | Eppendorf | EP0030108116-100EA | |
| LTQ-Orbitrap Velos | Thermo Fisher Scientific | LTQ Velos | |
| Microwave oven | Panasonic | NN-ST67HSRU | |
| n Easy-nLC II nanoHPLC | Thermo Fisher Scientific | LC140 | |
| PEAKS Studio | Bioinformatics Solutions Inc. | VERSION 8.5 | |
| Phosphate-buffered saline | Invitrogen | 3002 | tablets |
| precolumn (EASY-Column) | Thermo Fisher Scientific | (SC001) | 2 cm, ID100 µm, 5 µm, C18-A1 |
| Refrigerated centrifuge | Hermle | Z326K | for conical tubes |
| Refrigerated centrifuge | Vision | VS15000CFNII | for microtubes |
| Reversed-phase cleanup columns (Oasis HLB 1 cc Cartridge) | Waters | 186000383 | Oasis HLB 1 cc Cartridge |
| Sodium cyanoborodeuteride - NaBD3CN | Sigma-Aldrich | 190020 | |
| Sodium cyanoborohydride - NaBH3CN | Sigma-Aldrich | 156159 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S9763 | NOTE: 0.2 M PB= 0.1 M phosphate buffer pH 6.8 (26.85 mL of Na2HPO3 1M) plus 0.1 M phosphate buffer pH 6.8 (23.15 mL of NaH2PO3 1M) to 250 ml of water |
| Sodium phosphate monobasic | Sigma-Aldrich | S3139 | |
| Sonicator | Qsonica | Q55-110 | |
| Standard peptide | Proteimax | amino acid sequence: LTLRTKL | |
| Triethylammonium buffer - TEAB 1 M | Sigma-Aldrich | T7408 | |
| Trifluoroacetic acid - TFA | Sigma-Aldrich | T6508 | |
| Ultra purified water | Milli-Q | Direct-Q 3UV | |
| Vacuum centrifuge | GeneVac | MiVac DNA concentrator | |
| Water bath | Cientec | 266 | |
| Xcalibur Software | ThermoFisher Scientific | OPTON-30965 |
参考文献
- Kandpal, R., Saviola, B., Felton, J. The era of 'omics unlimited. Biotechniques. 46 (5), 354-355 (2009).
- Farrokhi, N., Whitelegge, J. P., Brusslan, J. A. Plant peptides and peptidomics. Plant Biotechnology Journal. 6 (2), 105-134 (2008).
- Schulz-Knappe, P., Schrader, M., Zucht, H. D. The peptidomics concept. Combinatorial Chemistry & High Throughput Screening. 8 (8), 697-704 (2005).
- Dallas, D. C., et al. Current peptidomics: applications, purification, identification, quantification, and functional analysis. Proteomics. 15 (5-6), 1026-1038 (2015).
- Chard, T. An introduction to radioimmunoassay and related techniques (3rd Ed). FEBS Letters. 238 (1), 223 (1988).
- Svensson, M., Sköld, K., Svenningsson, P., Andren, P. E. Peptidomics-based discovery of novel neuropeptides. Journal of Proteome Research. 2 (2), 213-219 (2003).
- Baggerman, G., et al. Peptidomics. Journal of Chromatography B. 803, 3-16 (2004).
- Theodorsson, E., Stenfors, C., Mathe, A. A. Microwave irradiation increases recovery of neuropeptides from brain tissues. Peptides. 11, 1191-1197 (1990).
- Che, F. Y., Lim, J., Pan, H., Biswas, R., Fricker, L. D. Quantitative neuropeptidomics of microwave-irradiated mouse brain and pituitary. Molecular & Cellular Proteomics. 4, 1391-1405 (2005).
- Dasgupta, S., et al. Analysis of the yeast peptidome and comparison with the human peptidome. PLoS One. 11 (9), 0163312 (2016).
- Teixeira, C. M. M., Correa, C. N., Iwai, L. K., Ferro, E. S., Castro, L. M. Characterization of Intracellular Peptides from Zebrafish (Danio rerio) Brain. Zebrafish. 16 (3), 240-251 (2019).
- Fricker, L. D. Analysis of mouse brain peptides using mass spectrometry-based peptidomics: implications for novel functions ranging from non-classical neuropeptides to microproteins. Molecular BioSystems. 6 (8), 1355-1365 (2010).
- Gelman, J. S., Sironi, J., Castro, L. M., Ferro, E. S., Fricker, L. D. Peptidomic analysis of human cell lines. Journal of Proteome Research. 10 (4), 1583-1592 (2011).
- De Araujo, C. B., et al. Intracellular peptides in cell biology and pharmacology. Biomolecules. 9, 150 (2019).
- Gewehr, M. C. F., Silverio, R., Rosa-Neto, J. C., Lira, F. S., Reckziegel, P., Ferro, E. S. Peptides from natural or rationally designed sources can be used in overweight, obesity, and type 2 diabetes therapies. Molecules. 25 (5), 1093 (2020).
- Sakaya, G. R., et al. Peptidomic profiling of cerebrospinal fluid from patients with intracranial saccular aneurysms. Journal of Proteomics. 240 (3), 104188 (2021).
- Fricker, L. Quantitative peptidomics: General considerations. Methods in Molecular Biology. 1719, 121-140 (2018).
- Southey, B. R., et al. Comparing label-free quantitative peptidomics approaches to characterize diurnal variation of peptides in the rat suprachiasmatic nucleus. Analytical Chemistry. 86 (1), 443-452 (2014).
- Chen, X., Wei, S., Ji, Y., Guo, X., Yang, F. Quantitative proteomics using SILAC: Principles, applications, and developments. Proteomics. 15 (18), 3175-3192 (2015).
- Boonen, K., et al. Quantitative peptidomics with isotopic and isobaric tags. Methods in Molecular Biology. 1719, 141-159 (2018).
- Gewehr, M. C. F., et al. The relevance of thimet oligopeptidase in the regulation of energy metabolism and diet-induced obesity. Biomolecules. 10 (2), 321 (2020).
- Fiametti, L. O., Correa, C. N., Castro, L. M. Peptide profile of zebrafish brain in a 6-OHDA-induced Parkinson model. Zebrafish. 18 (1), 55-65 (2021).
- Boersema, P. J., Raijmakers, R., Lemeer, S., Mohammed, S., Heck, A. J. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics. Nature Protocols. 4 (4), 484-494 (2009).
- Dasgupta, S., Castro, L. M., Tashima, A. K., Fricker, L. Quantitative peptidomics using reductive methylation of amines. Methods in Molecular Biology. 1719, 161-174 (2018).
- Tashima, A. K., Fricker, L. D. Quantitative peptidomics with five-plex reductive methylation labels. Journal of the American Society for Mass Spectrometry. 29 (5), 866-878 (2018).
- Che, F. Y., et al. Optimization of neuropeptide extraction from the mouse hypothalamus. Journal of Proteome Research. 6 (12), 4667-4676 (2007).
- Lyons, P. J., Fricker, L. D. Peptidomic approaches to study proteolytic activity. Current Protocols in Protein Science. , 13 (2011).
- Udenfriend, S., et al. Fluorescamine: a reagent for assay of amino acids, peptides, proteins, and primary amines in the picomole range. Science. 178 (4063), 871-872 (1972).
- Ma, B., et al. PEAKS: powerful software for peptide de novo sequencing by tandem mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (20), 2337-2342 (2003).
- Zhang, J., et al. PEAKS DB: de novo sequencing assisted database search for sensitive and accurate peptide identification. Molecular & Cellular Proteomics. 11 (4), 1-8 (2012).
- Sturm, R. M., Dowell, J. A., Li, L. Rat brain neuropeptidomics: tissue collection, protease inhibition, neuropeptide extraction, and mass spectrometric analysis. Methods in Molecular Biology. 615, 217-226 (2010).
- Fricker, L. D. Limitations of mass spectrometry-based peptidomic approaches. Journal of the American Society for Mass Spectrometry. 26 (12), 1981-1991 (2015).
- Ross, , et al. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents. Molecular & Cellular Proteomics. 3, 1154-1169 (2004).
- Thompson, A., et al. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75, 1895-1904 (2003).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。