A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הכנה לדוגמה וכמות יחסית באמצעות מתילציה מופחתת של אמינים למחקרי פפטידומית
In This Article
Summary
מאמר זה מתאר שיטת הכנה לדוגמה המבוססת על אי-פעולת חום כדי לשמר פפטידים אנדוגני הימנעות השפלה לאחר המוות, ואחריו כמות יחסית באמצעות תיוג איזוטופי בתוספת LC-MS.
Abstract
פפטידומיקה יכולה להיות מוגדרת כניתוח איכותי וכמותי של פפטידים במדגם ביולוגי. היישומים העיקריים שלה כוללים זיהוי סמנים ביולוגיים פפטיד של מחלות או מתח סביבתי, זיהוי נוירופפטידים, הורמונים, פפטידים תאיים ביואקטיביים, גילוי פפטידים מיקרוביאליים ו nutraceutical מחלבון הידרוליזטים, וניתן להשתמש בהם במחקרים כדי להבין את התהליכים הפרוטאוליטיים. ההתקדמות האחרונה בהכנת מדגם, שיטות הפרדה, טכניקות ספקטרומטריית מסה וכלים חישוביים הקשורים לרצף חלבונים תרמה לעלייה במספר הפפטידים והפפטידום המזוהים המאופיינים. מחקרים פפטידום לעתים קרובות לנתח פפטידים שנוצרים באופן טבעי בתאים. כאן, פרוטוקול הכנה מדגם המבוסס על אי-פעילות חום מתואר, אשר מבטל פעילות פרוטאז, ומיצוי עם תנאים קלים, כך שאין מחשוף קשרים פפטיד. בנוסף, כמות יחסית של פפטידים באמצעות תיוג איזוטופ יציב על ידי מתילציה מופחתת של אמינים מוצג גם. שיטת תיוג זו יש כמה יתרונות כמו ריאגנטים זמינים מסחרית, זול בהשוואה לאחרים, יציב מבחינה כימית, ומאפשר ניתוח של עד חמש דגימות בריצה אחת LC-MS.
Introduction
מדעי "Omics" מאופיינים בניתוח עמוק של ערכת מולקולות, כגון DNA, RNA, חלבונים, פפטידים, מטבוליטים וכו '. ערכות נתונים בקנה מידה גדול אלה (גנומיקה, תמלול, פרוטאומיה, פפטידומיקה, מטבולומיה וכו ') חוללו מהפכה בביולוגיה והובילו להבנה מתקדמת של תהליכים ביולוגיים1. המונח פפטידומיקה החל להיות מוצג בתחילת המאה ה -20, וכמה מחברים התייחסו אליו כענף של פרוטאומיקס2. עם זאת, לפפטידוזיקה יש פרטים ברורים, כאשר האינטרס העיקרי הוא לחקור את תוכן הפפטידים שנוצר באופן טבעי במהלך תהליכים תאיים, כמו גם את אפיון הפעילות הביולוגית של מולקולות אלה3,4.
בתחילה, מחקרי פפטיד ביואקטיביים הוגבלו נוירופפטידים ופפטידים הורמונליים באמצעות השפלה אדמן ורדיוימונואסאי. עם זאת, טכניקות אלה אינן מאפשרות ניתוח גלובלי, בהתאם לבידוד של כל פפטיד בריכוזים גבוהים, זמן ליצירת נוגדנים, מלבד אפשרות תגובה צולבת5.
ניתוח פפטידומיקה התאפשר רק לאחר מספר התקדמות בכרומטוגרפיה נוזלית בשילוב ספקטרומטריית מסה (LC-MS) ופרויקטים של גנום שסיפקו מאגרי נתונים מקיפים למחקרי פרוטאומיקה/פפטידומיקה6,7. יתר על כן, פרוטוקול מיצוי פפטיד ספציפי עבור peptidomes היה צריך להיות הוקם כי המחקרים הראשונים שניתחו neuropeptides ברחבי העולם בדגימות המוח הראו כי זיהוי הושפע השפלה מסיבית של חלבונים, אשר מתרחשים בעיקר ברקמה זו לאחר 1 דקות לאחר המוות. נוכחותם של שברי פפטיד אלה הסתירו את אות הנוירופפטיד ולא ייצגו את הפפטידום ב- vivo. בעיה זו נפתרה בעיקר עם היישום של חימום מהיר של פעילות פרוטאזים באמצעות הקרנת מיקרוגל, אשר הפחית באופן דרסטי את נוכחותם של שברי חפץ אלה ואפשר לא רק זיהוי של שברי נוירופפטיד, אלא חשף את נוכחותם של קבוצה של פפטידים מחלבונים ציטוזוליים, מיטוכונדריאליים וגרעין, שונים של degradome6,8,9.
הליכים מתודולוגיים אלה אפשרו הרחבה של הפפטידום מעבר לנוירופפטידים הידועים, שם זוהו מאות פפטידים תאיים שנוצרו בעיקר על ידי פעולת הפרוטאוזומים בשמרים10, זברה11, רקמות מכרסמים12 ותאים אנושיים13. עשרות פפטידים תאיים אלה הוכחו בהרחבה כבעלי פעילויות ביולוגיות ופרמקולוגיות כאחד14,15. יתר על כן, פפטידים אלה יכולים לשמש סמנים ביולוגיים של מחלות ואולי יש משמעות קלינית, כפי שהוכח בנוזל השדרתי מחולים עם מפרצת תוך גולגולתית16.
נכון לעכשיו, בנוסף לזיהוי רצפי פפטיד, ניתן באמצעות ספקטרומטריית מסה להשיג נתונים של כמות מוחלטת ויחסית. בכמות המוחלטת, רמות הפפטיד במדגם ביולוגי מושוות לתקנים סינתטיים, ואילו בכמות היחסית, רמות הפפטיד משווות בין שתי דגימות או יותר17. כמות יחסית יכולה להתבצע באמצעות הגישות הבאות: 1) "תווית חינם"18; 2) בסימון מטבולי vivo או 3) תיוג כימי. שני האחרונים מבוססים על שימוש בצורות איזוטופיות יציבות המשולבות בפפטידים19,20. בניתוח ללא תוויות, רמות הפפטיד מוערכות על ידי התחשבות בעוצמת האות (ספירות ספקטרליות) במהלך LC-MS18. עם זאת, תיוג איזוטופי יכול להשיג רמות יחסיות מדויקות יותר של פפטידים.
מחקרים פפטידום רבים השתמשו טרימתילאמוניום butyrate (TMAB) תיוג ריאגנטים כמו תיוג כימי, ולאחרונה, מתילציה רדוקטיבית של אמינים (RMA) עם צורות deuteed ולא deuteed של פורמלדהיד ונתרן cyanoborohydride ריאגנטים שימשו11,21,222. עם זאת, תוויות TMAB אינן זמינות מסחרית, ותהליך הסינתזה הוא מייגע מאוד. מצד שני, ב RMA, ריאגנטים זמינים מסחרית, זול בהשוואה תוויות אחרות, ההליך הוא פשוט לביצוע, ואת פפטידים מסומנים יציבים23,24.
השימוש ב- RMA כרוך בהקמת בסיס שיף על ידי מתן אפשרות לפפטידים להגיב עם פורמלדהיד, ואחריו תגובת הפחתה באמצעות ציאנובורוהידריד. תגובה זו גורמת דימתילציה של קבוצות אמינו חינם על N-מסופים ושרשראות צד ליצין ו monomethylates N-מסוף prolines. איך שאריות פרולין הם לעתים קרובות נדיר על N-מסוף, כמעט כל הפפטידים עם אמינים חינם על N-טרמינל מסומנים עם שתי קבוצות מתיל23,24,25.
Protocol
ההליך הבא להפקת פפטיד ומתילציה מופחתת הותאם מהליכים שפורסמו בעבר24,25,26,27. פרוטוקול זה פעל בהתאם להנחיות המועצה הלאומית לבקרת ניסויים בבעלי חיים (CONCEA) ואושר על ידי ועדת האתיקה לשימוש בבעלי חיים (CEUA) במכון הביו-מדעי של אוניברסיטת סאו פאולו. שלבי הפרוטוקול מוצגים באיור 1.
הערה: הכינו את כל הפתרונות המימיים במים אולטרה-תכשירים.
1. מיצוי פפטיד
- תרבית תאים
- טפח תאי SHSY5Y בצלחת של 15 ס"מ ב-37 °C (5% CO2 ) במדיום הנשר המותאם של Dulbecco המכיל 15% סרום בקר עוברי ו-1% פניצילין-סטרפטומיצין.
- יש להשתמש ב-2-3 צלחות לכל דגימה. להגדיל את התאים ל 100% מפגש.
- לאחר הטיפול המיועד, לשטוף את התאים פעמיים עם תמיסת מלח חוצץ פוספט. לאחר מכן, להוסיף 10 מ"ל של תמיסת מלח חוצץ פוספט, לגרד את התאים ולאסוף לתוך צינור 15 מ"ל.
- צנטריפוגטה ב 800 x g במשך 5 דקות ולהסיר את supernatant. resuspend הכדור ב 1 מ"ל של מים דה-יוניזציה מטוהרים במיוחד ב 80 °C (80 °F).
- העבר את התוכן של הצינור (ליסאט סלולרי) לצינור מיקרופוגה 2 מ"ל.
- רקמות בעלי חיים (בעיקר רקמת עצבים):
- מרטים סוג בר זכר מבוגר דניו rerio (דג זברה) עם מנה קטלנית של MS 222 (100 מ"ג / ליטר) ומיד כפוף אותו 8 s של קרינת מיקרוגל כדי לאטום פפטידאז ופרוטאז.
הערה: ניתן להשתמש בתנור מיקרוגל ביתי. מיקרוגל 900 W שימש עבור 8 עד 10 s במלוא העוצמה. המיקרוגל המשמש חייב להיות מסוגל להעלות את טמפרטורת המוח > 80 °C (50 °F) בתוך 10 מעלות צלזיוס. רבייה בחימום בין דגימות תרוויח גם על ידי הצבת הרקמה באותו מיקום במיקרוגל. - לאחר אי-פעילות חום, לאסוף את כל המוח בצינור microfuge 2 מ"ל ולהקפיא ב -80 °C (70 °F) עד ניתוח.
- resuspend דגימת הרקמה ב 1 מ"ל של מים דה-יונים מטוהרים במיוחד ב 80 °C (80 °F). Sonicate הרקמה עם בדיקה באמצעות 30 פולסים (4 הרץ) של 1 s.
הערות: עבור רקמות הכבד והכליות, השתמש homogenizer מכני ב 10,000-30,000 סל"ד עבור 20 s. עבור רקמות שרירים, לטחון את הרקמה בחנקן נוזלי באמצעות כור היתוך חרסינה ועלי. השלבים הבאים זהים עבור ליסאט הסלולר או רקמה הומוגנית.
- מרטים סוג בר זכר מבוגר דניו rerio (דג זברה) עם מנה קטלנית של MS 222 (100 מ"ג / ליטר) ומיד כפוף אותו 8 s של קרינת מיקרוגל כדי לאטום פפטידאז ופרוטאז.
- לדגור על הרקמה הסלולר ליסאט או הומוגני ב 80 °C (70 °F) במשך 20 דקות. לאחר מכן, לקרר אותו על קרח במשך 10-30 דקות.
- הוסף 10 μL של פתרון מלאי HCl 1 M עבור כל 1 מ"ל של נפח מדגם כדי להשיג ריכוז סופי של 10 mM. מערבבים על ידי מערבולת במשך 20 s ודגרה נוספת על קרח במשך 15 דקות.
הערה: לפני החמצה, ודא המדגם מקורר לחלוטין כדי למנוע שבירת קשרים פפטיד הנגרמת על ידי החמצה בטמפרטורות גבוהות. - צנטריפוגות ליסאט הסלולר או רקמה הומוגנית ב 12,000 x גרם ב 4 °C (70 °F) במשך 15 דקות. לאסוף את supernatant בצינורות microcentrifuge חלבון מחייב נמוך ולאחסן אותו ב -80 °C (70 °F).
- נקה את התקני הסינון האולטרה-סינון (מסנני ניתוק של 10 kDa) על-ידי הוספת מים וצנטריפוגות ב- 2300 x g למשך 3 דקות. חזור על שלב זה פעמיים נוספות.
- מניחים את הסופר-נט במסננים חתוךים 10 kDa שנשטפו מראש וצנטריפוגה ב-2,300 x גרם ב-4 מעלות צלזיוס למשך 50 דקות בצנטריפוגה מחודשת. הזרימה דרך מייצגת את תמצית הפפטיד.
- התפלת הדגימות בעמודות ניקוי שלב הפוך בהתאם להוראות היצרן באמצעות פתרונות acetonitrile (ACN) וחומצה טריפלואורואצטית (TFA) כמתואר להלן:
- שפל את העמודה עם 1 מ"ל של 100% ACN.
- לשטוף את העמודה עם פתרון 1 מ"ל של 5% ACN עם 0.1% TFA.
- טען את אמצעי האחסון המלא של הדוגמה בעמודה.
- לשטוף את העמודה עם פתרון 1 mL של 5% ACN עם 0.1% TFA
- יש לאחל את הפפטידים מהטור עם פתרון של 1.8 מ"ל של 100% ACN עם 0.15% TFA בצינורות מיקרוצנטריפוגה בעלי קשירה נמוכה של חלבון.
- ייבשו את הדגימה לחלוטין בצנטריפוגת ואקום. הגדר את שיטת הריכוז עבור ממיסים אורגניים וטמפרטורה ב 30 °C (50 °F). זמן הריכוז מנוטר בתצוגה.
- אחסן את הדגימות ב- −80°C עד לשלב הבא.
2. כימות פפטיד עם פלואורסאמין
הערה: ניתן להעריך את כמות הפפטיד באמצעות פלואורסאמין ב- pH 6.8 כפי שתואר בעבר11,28. שיטה זו מורכבת מחיבור של מולקולת פלואורסאמין לאמינים העיקריים הקיימים בשאריות ליסן (K) ו/או N-terminal של פפטידים. התגובה מבוצעת ב- pH 6.8 כדי להבטיח כי פלואורסצמין מגיב רק עם קבוצות האמינו של הפפטידים ולא עם חומצות אמינו חופשיות. הפלואורסאמין נמדד באמצעות ספקטרופלואורומטר אורך גל עירור של 370 ננומטר ואורך גל פליטה של 480 ננומטר.
- הכן ריכוזים שונים של הפפטיד הסטנדרטי (0.05, 0.1, 0.15, 0.2, 0.3, 0.5 ו 0.7 מיקרוגרם / μL) ולאחסן את aliquots ב -20 °C (50 °F).
הערה: פפטיד 5A (LTLRTKL) הוא הציע שכן יש לו הרכב ידוע וריכוז. - הכן פתרון מלאי פלואורסאמין (0.3 מ"ג /מ"ל) באצטון. Aliquot במהירות צינורות microcentrifuge (1 מ"ל), לאטום באמצעות parafilm, ולאחסן ב -20 °C (50 °F) בחושך.
- הכן 0.2 M חוצץ פוספט (PB) ב pH 6.8.
הערה: הכן 0.2 M PB על ידי הוספת 0.1 M חוצץ פוספט pH 6.8 (26.85 מ"ל של Na2HPO3 1M) ו 0.1 M חוצץ פוספט pH 6.8 (23.15 מ"ל של NaH2PO3 1M) עד 250 מ"ל של מים - resuspend דגימות פפטיד ב 100-200 μL של מים מטוהרים במיוחד.
- Pipette 2.5 μL של ריכוזי פפטיד סטנדרטיים ודגימות על צלחת לבן 96 באר עבור תוחם פלואורסצנטי במשולש. הוסף 25 μL של 0.2 M מאגר פוספט.
- הוסף 12.5 μL של פלואורסאמין עם פיפטה רב ערוצית. הומוגניזציה בעדינות במשך דקה אחת על שייקר המסובב מסלולית.
- לאחר מכן, להוסיף 110 μL של מים עם pipette רב ערוצית כדי לעצור את התגובה.
הערה: העבר את תמיסה מלאי פלואורסצמין ומים אולטרה-דור לשני מאגרים כדי pipette פתרונות אלה עם pipette רב ערוצי. - התאם את פרמטרי הקריאה הבאים בספקטרופלואורומטר: קרא את הדגימות מלמעלה, אורך גל עירור ב-370 ננומטר ואורך גל פליטה ב-480 ננומטר.
- קרא את הצלחת על הספקטרופלואורומטר.
3. מתילציה מופחתת של תיוג אמינים
הערה: שיטת תיוג איזוטופית זו מבוססת על דימתילציה של קבוצות אמין עם צורות deuterated ולא deuteed של פורמלדהיד ונתרן ציאנובורוהידריד ריאגנטים. התוצר הסופי של תגובה זו מוסיף 28 Da, 30 Da, 32 Da, 34 Da, או 36 Da למסה הסופית של כל פפטיד בכל אתר תיוג זמין (ליסין או N-מסוף). תגובה זו מייצרת הבדל m/z בפפטידים המסומנים בצורות שונות שנצפו בספקטרום MS (טבלה 1).
זהירות: יש להשתמש בציוד בטיחות מתאים לטיפול בתרכובות אלה, ויש לנקוט משנה זהירות כדי למזער את החשיפה. נהלים עם פורמלדהיד ונתרן ציאנובורוהידריד ריאגנטים צריכים להתבצע במכסה אדים כי הם רעילים מאוד (כולל שקלול נתרן ציאנובורוהידריד). במהלך תגובת מרווה והחמצה, גז רעיל (מימן ציאניד) עשוי להיווצר.
- הכן את הפתרונות הטריים הבאים מהמלאי או ריאגנטים ביום ההליך במים אולטרה-דור:
- לדלל את המניה של 37% פורמלדהיד (CH2O) ל 4%.
- לדלל את המניה של 20% פורמלדהיד Deuterated (CD2O) ל 4%.
- לדלל את המניה של 20% C13 פורמלדהיד (13CD2O) עד 4%.
- הכן 0.6 M NabH3CN.
- הכן 0.6 M NaBD3CN.
- הכן פתרון של 1% אמוניום ביקרבונט.
- הכן פתרון 5% חומצה פורמית.
הערה: לגבי הכמות היחסית של פפטידים, כמו תוכניות ניסיוניות שונות ניתן לבצע בהתאם למספר התוויות בשימוש, תשומת לב נדרשת במהלך הליך תיוג כימי. מומלץ להפריד aliquots קטן של תוויות במדפים נפרדים עם הדגימות המתאימות להיות מתויג כדי להפחית את הפוטנציאל לטעות אנוש בהוספת ריאגנט שגוי צינורות מדגם.
- הכן כל דגימה המכילה עד 25 מיקרוגרם של הפפטיד. אסור שהדגימות יכילו טריס או אמוניום ביקרבונט.
הערה: הכמויות המתוארות להלן מספיקות עבור כל דגימה בנפח של 100 μL.
המשך עם שלבים 3.3-3.8 בברדס אדים. - הוסף נפח 1 /10 של 1 M TEAB לדגימות (ריכוז סופי של הפתרון 100 מ"מ של TEAB). בדוק את ה- pH עם נייר מחוון pH; זה חייב להיות בין 5-8. התאימו עם HCl או NaOH במידת הצורך.
- הוסף 4 μL של פורמלדהיד לא מפורסלם, מפורסמת לא מפורסמת או C13 מפורמלדהיד שהוקמה. מערבבים במשך 5 s על ידי מערבולת.
- הוסף 4 μL של NaBH3CN (0.6 M) או NaBD3CN (0.6 M) בהתאם לתוכנית התוויות שנקבעה. מערבבים במשך 5 s על ידי מערבולת.
- דגירה במכסה אדים למשך 2 שעות בטמפרטורת החדר, ומערבבים כל 30 דקות.
- חזור על שלבים 3.4 ו- 3.5. לדגור את הדגימות בשכונת אדים לילה בטמפרטורת החדר.
- הוסיפו 16 מיקרו-אל של אמוניום ביקרבונט (1%) וערבבו על ידי מערבולת. מניחים את המדגם על קרח, מוסיפים 8 מיקרו-אל של חומצה פורמית (5%), ומערבבים על ידי מערבולת במשך 5 s.
- שלב את הדגימות, התאם את ה- pH ל- 2-4 והתפל את הדגימות המשולבות בעמודות ניקוי שלב הפוך כפי שתואר קודם לכן בשלב 1.8.
- ייבשו את הדגימה לחלוטין בצנטריפוגת ואקום. הגדר את שיטת הריכוז עבור ממיסים אורגניים וטמפרטורה ב 30 °C (50 °F). זמן הריכוז מנוטר בתצוגה.
- לאחסן את הדגימות ב -20 °C (50 °F).
4. כרומטוגרפיה נוזלית וספקטרומטריית מסה
- בצע ניתוח LC-MS באמצעות מערכת nanoHPLC בשילוב למכשיר MS תואם תגי מתיל.
הערה: מגוון מכשירי MS תואמים לתיוג RMA. מערכת nanoHPLC בשילוב Orbitrap משמש בדרך כלל לביצוע ניתוח LC-MS באמצעות מקור יון nanoelectrospray. ראשית, המדגם נטען לתוך precolumn ופפטידים מופרדים בעמודה אנליטית. ההשתלטות של פפטידים מתבצעת באמצעות שיפוע ליניארי של 5%-45% acetonitrile, ב 0.1% חומצה פורמית, במהלך 90 דקות, עם זרימה של 200 nL / min. ספקטרומטר המסה מוגדר לפעול במצב תלוי נתונים. כל סריקה מלאה נרכשת בעוצמה של 10-30 eV, 2.3 Kv, ולאחר מכן עשר הפסגות הגבוהות ביותר נבחרות עבור פיצול דיסוציאציה הנגרמת על ידי התנגשות (CID). זמן ההזרקה מוגדר על מלכודת היונים ב 100 אלפיות שנייה, ואת פורייה Transform (FT)-MS הזרקה קבוע עם רזולוציה של 1000 ms 30,000 ב m / z 300-1800. ספירה מינימלית של 5000 והחרגה דינאמית של 70 s משמשת לביצוע סריקת פיצול.
5. כמות יחסית של פפטידים
הערה: ספקטרום הטרחה של טרה-ס מנותחים בתוכנת ספקטרומטר המסה. קבוצות שיא של פפטידים עם תוויות שונות מזוהות בספקטרום MS. הכמות היחסית מחושבת על ידי עוצמת כל פסגה מונואיזוטופית. כל קבוצה מטופלת מושוות לקבוצת הביקורת המתאימה.
- לחץ פעמיים על קובץ הדגימה הגולמי כדי לפתוח את תוכנת ניתוח הספקטרום. טען את הכרומטוגרמה של זמן השמירה (RT) ו- MS (EM) בשתי כרטיסיות, עליונה ולמטה, בהתאמה.
- לחץ באמצעות לחצן העכבר הימני פעם אחת ברצף בסמלים אפשרויות תצוגה ומסה בסרגל הכלים של התוכנה והגדר את דיוק המוני לארבעה מקומות עשרוניים.
- מקם את סמן העכבר במקום כלשהו בכרטיסיה RT. חפש את זמן השמירה של היון המתאים לניתוח ולחץ על לחצן העכבר הימני. ספקטרום MS של הזמן שנבחר יוצג באופן אוטומטי בכרטיסיה EM.
- מקם את סמן העכבר במקום כלשהו בכרטיסיה EM. חפש את היונים שיש לנתח.
- לחץ והחזק באמצעות לחצן העכבר הימני באזור סמוך משמאל ליד יונים אלה. לאחר מכן, גרור את העכבר ימינה בטווח הרצוי כדי להגדיל את אזור העניין.
- שמור על העכבר ממוקם בכרטיסיה EM ולחץ על חצי לוח המקשים ימינה או שמאלה כדי להגדיר את טווח היונים שיש לנתח.
- מקם שוב את סמן העכבר בכרטיסיה RT בתחילת מרווח הזמן הרצוי.
- לחץ באמצעות לחצן העכבר הימני וגרור אותו עד לערך הזמן שנבחר. תשאיר את הכפתור. העוצמה המצטברת של היונים תוצג באופן אוטומטי בכרטיסיה EM.
- אסוף את נתוני עוצמת ה- m/z, z ו- ion בגיליון אלקטרוני.
הערה: המסה המונואיזוטופית של כל פפטיד ללא תוספת קבוצות מתיל מחושבת מהנוסחה הבאה:
מסה של פפטיד לא שודר = (m/z a x z) - (C a x T) - (1.008 x z)
m/zais המסה שנצפתה כדי לטעון ערך עבור הפסגה המונואיזוטופית עבור כל פפטיד המסומן בשילובים שונים של תגים (a =1, 2, 3, 4 או 5, המתאים למספר המדגם).
z הוא מצב חיוב.
Ca היא המסה המונואיזוטופית של זוג קבוצות מתיל:
עבור a= 1, Ca = 28.0313 (התוספת נטו של שתי קבוצות CH3 לאמין הראשי)
עבור a=2, Ca = 30.0439 עבור שתי קבוצות CHD2
עבור a=3, Ca = 32.0564 עבור שתי קבוצות CD2H
עבור a=4, Ca = 34.0690 עבור שתי קבוצות CD3
עבור a=5, Ca = 36.0757 עבור שתי קבוצות CD3 של 13
T הוא מספר הזוגות של קבוצות מתיל המשולבות בפפטיד. ניתן לחשב זאת מהנוסחה הבאה כאשר נעשה שימוש בחמישה תגים: T =z*(m/z5 - m/z1)/8. עבור פפטידים המכילים אמין ראשי יחיד ולכן מסומנים עם שתי קבוצות מתיל בלבד להציג חפיפות שיא על ספקטרום MS כאשר תוויות סמוכות משמשות. ניתן לתקן את עוצמת השיא של כל פפטיד שכותרתו באמצעות המשוואות המתוארות על ידי Tashima ו- Fricker25.
6. זיהוי פפטיד
- כדי לזהות פפטידים, נתח את נתוני MS/MS באמצעות מנוע חיפוש של מסד נתונים29,30.
- כדי לחשב את קצב הגילוי השגוי (FDR) באמצעות שיטת היתוך הפיתיון, חפש במסד נתונים של פיתיון.
הערה: פרמטרי החיפוש המשמשים בדרך כלל אינם ספציפיים לאנזים; עמידות המונית מבשר להגדיר 15-50 ppm; סובלנות מסה יון שבר של 0.5 Da; שינויים משתנים: אמינים תגובתיים משאריות ליס ו- N-terminus של תוויות מתילציה איזוטופיות פפטידים (L1 (+28), L2 (+30), L3 (+32), L4 (+34) ו- L5 (+36)), מתיונין מחומצן (+15.99 Da) ואצטילציה (+42.01 Da). - לאחר מכן, מיין את הפפטידים לפי ממוצע הביטחון המקומי שלהם כדי לבחור את הספקטרום הטוב ביותר כדי ביאור ולסנן אותם על ידי FDR ≤5%.
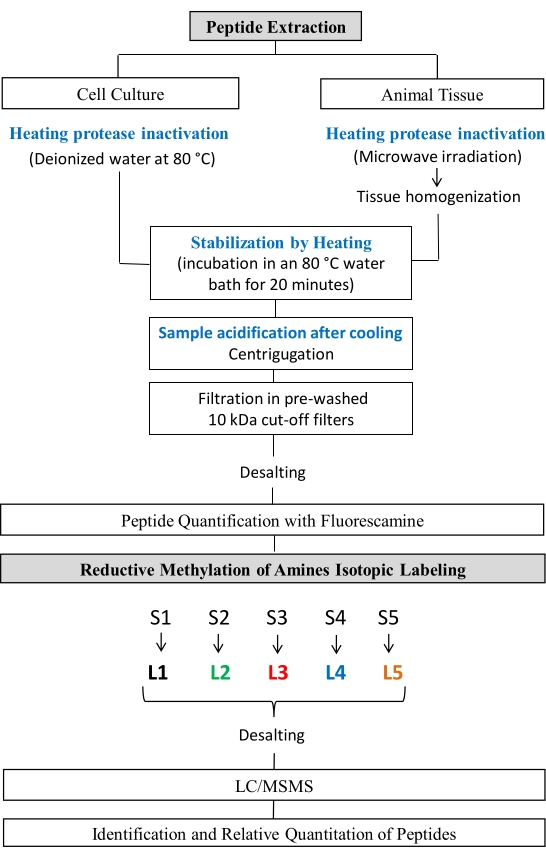
איור 1: זרימת עבודה של לימודי פפטידום. שלבים של מיצוי פפטיד ומתילציה מופחתת של אמינים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
תוצאות
התוצאות המתקבלות מהריצות שבוצעו בספקטרומטר המסה מאוחסנות בקבצי נתונים גולמיים שניתן לפתוח בתוכנת ספקטרומטר המסה. בספקטרום MS, ניתן לצפות בקבוצות שיא המייצגות פפטידים מסומנים על פי ערכת התיוג המשמשת, החל 2-5 תוויות. לדוגמה, באיור 2, זוגות פסגות שזוהו בזמן כרומטוגרפי מיוצגים ?...
Discussion
ברוב המחקרים פפטידום, אחד הצעדים הקריטיים הוא, ללא ספק, הכנת המדגם כי צריך להתבצע בקפידה כדי למנוע נוכחות של שברי פפטיד שנוצר על ידי פרוטאזים לאחר כמה דקות לאחר המוות. המחקרים הראשוניים על תמציות מוח שהוכנו מדגימות שאינן במיקרוגל הראו מספר רב של שברי חלבון להיות נוכחים 10-kDa microfiltrates. גישות ש?...
Disclosures
אין אינטרסים פיננסיים מתחרים קיימים.
Acknowledgements
הפיתוח והשימוש בטכניקות המתוארות כאן נתמכו על ידי מענק המועצה הלאומית הברזילאית 420811/2018-4 (LMC); Fundação de Amparo à Pesquisa do Estado de São Paulo (www.fapesp.br) grants 2019/16023-6 (LMC), 2019/17433-3 (LOF) ו-21/01286-1 (MEME). למממנים לא היה כל תפקיד בעיצוב המחקר, איסוף וניתוח הנתונים, ההחלטה לפרסם או הכנת המאמר.
Materials
| Name | Company | Catalog Number | Comments |
| 10 kDa cut-off filters | Merck Millipore | UFC801024 | Amicon Ultra-4, PLGC Ultracel-PL Membrane, 10 kDa |
| Acetone | Sigma-Aldrich | 179124 | |
| Acetonitrile | Sigma-Aldrich | 1000291000 | |
| Ammonium bicarbonate | Sigma-Aldrich | 11213 | |
| analytical column (EASY-Column) | EASY-Column | (SC200) | 10 cm, ID75 µm, 3 µm, C18-A2 |
| Ethyl 3-aminobenzoate methanesulfonate | Sigma-Aldrich | E10521 | MS-222 |
| Fluorescamine | Sigma-Aldrich | F9015 | |
| Formaldehyde solution | Sigma-Aldrich | 252549 | |
| Formaldehyde-13C, d2, solution | Sigma-Aldrich | 596388 | |
| Formaldehyde-d2 solution | Sigma-Aldrich | 492620 | |
| Formic acid | Sigma-Aldrich | 33015 | |
| Fume hood | Quimis | Q216 | |
| Hydrochloric acid - HCl | Sigma-Aldrich | 258148 | |
| LoBind-Protein retention tubes | Eppendorf | EP0030108116-100EA | |
| LTQ-Orbitrap Velos | Thermo Fisher Scientific | LTQ Velos | |
| Microwave oven | Panasonic | NN-ST67HSRU | |
| n Easy-nLC II nanoHPLC | Thermo Fisher Scientific | LC140 | |
| PEAKS Studio | Bioinformatics Solutions Inc. | VERSION 8.5 | |
| Phosphate-buffered saline | Invitrogen | 3002 | tablets |
| precolumn (EASY-Column) | Thermo Fisher Scientific | (SC001) | 2 cm, ID100 µm, 5 µm, C18-A1 |
| Refrigerated centrifuge | Hermle | Z326K | for conical tubes |
| Refrigerated centrifuge | Vision | VS15000CFNII | for microtubes |
| Reversed-phase cleanup columns (Oasis HLB 1 cc Cartridge) | Waters | 186000383 | Oasis HLB 1 cc Cartridge |
| Sodium cyanoborodeuteride - NaBD3CN | Sigma-Aldrich | 190020 | |
| Sodium cyanoborohydride - NaBH3CN | Sigma-Aldrich | 156159 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S9763 | NOTE: 0.2 M PB= 0.1 M phosphate buffer pH 6.8 (26.85 mL of Na2HPO3 1M) plus 0.1 M phosphate buffer pH 6.8 (23.15 mL of NaH2PO3 1M) to 250 ml of water |
| Sodium phosphate monobasic | Sigma-Aldrich | S3139 | |
| Sonicator | Qsonica | Q55-110 | |
| Standard peptide | Proteimax | amino acid sequence: LTLRTKL | |
| Triethylammonium buffer - TEAB 1 M | Sigma-Aldrich | T7408 | |
| Trifluoroacetic acid - TFA | Sigma-Aldrich | T6508 | |
| Ultra purified water | Milli-Q | Direct-Q 3UV | |
| Vacuum centrifuge | GeneVac | MiVac DNA concentrator | |
| Water bath | Cientec | 266 | |
| Xcalibur Software | ThermoFisher Scientific | OPTON-30965 |
References
- Kandpal, R., Saviola, B., Felton, J. The era of 'omics unlimited. Biotechniques. 46 (5), 354-355 (2009).
- Farrokhi, N., Whitelegge, J. P., Brusslan, J. A. Plant peptides and peptidomics. Plant Biotechnology Journal. 6 (2), 105-134 (2008).
- Schulz-Knappe, P., Schrader, M., Zucht, H. D. The peptidomics concept. Combinatorial Chemistry & High Throughput Screening. 8 (8), 697-704 (2005).
- Dallas, D. C., et al. Current peptidomics: applications, purification, identification, quantification, and functional analysis. Proteomics. 15 (5-6), 1026-1038 (2015).
- Chard, T. An introduction to radioimmunoassay and related techniques (3rd Ed). FEBS Letters. 238 (1), 223 (1988).
- Svensson, M., Sköld, K., Svenningsson, P., Andren, P. E. Peptidomics-based discovery of novel neuropeptides. Journal of Proteome Research. 2 (2), 213-219 (2003).
- Baggerman, G., et al. Peptidomics. Journal of Chromatography B. 803, 3-16 (2004).
- Theodorsson, E., Stenfors, C., Mathe, A. A. Microwave irradiation increases recovery of neuropeptides from brain tissues. Peptides. 11, 1191-1197 (1990).
- Che, F. Y., Lim, J., Pan, H., Biswas, R., Fricker, L. D. Quantitative neuropeptidomics of microwave-irradiated mouse brain and pituitary. Molecular & Cellular Proteomics. 4, 1391-1405 (2005).
- Dasgupta, S., et al. Analysis of the yeast peptidome and comparison with the human peptidome. PLoS One. 11 (9), 0163312 (2016).
- Teixeira, C. M. M., Correa, C. N., Iwai, L. K., Ferro, E. S., Castro, L. M. Characterization of Intracellular Peptides from Zebrafish (Danio rerio) Brain. Zebrafish. 16 (3), 240-251 (2019).
- Fricker, L. D. Analysis of mouse brain peptides using mass spectrometry-based peptidomics: implications for novel functions ranging from non-classical neuropeptides to microproteins. Molecular BioSystems. 6 (8), 1355-1365 (2010).
- Gelman, J. S., Sironi, J., Castro, L. M., Ferro, E. S., Fricker, L. D. Peptidomic analysis of human cell lines. Journal of Proteome Research. 10 (4), 1583-1592 (2011).
- De Araujo, C. B., et al. Intracellular peptides in cell biology and pharmacology. Biomolecules. 9, 150 (2019).
- Gewehr, M. C. F., Silverio, R., Rosa-Neto, J. C., Lira, F. S., Reckziegel, P., Ferro, E. S. Peptides from natural or rationally designed sources can be used in overweight, obesity, and type 2 diabetes therapies. Molecules. 25 (5), 1093 (2020).
- Sakaya, G. R., et al. Peptidomic profiling of cerebrospinal fluid from patients with intracranial saccular aneurysms. Journal of Proteomics. 240 (3), 104188 (2021).
- Fricker, L. Quantitative peptidomics: General considerations. Methods in Molecular Biology. 1719, 121-140 (2018).
- Southey, B. R., et al. Comparing label-free quantitative peptidomics approaches to characterize diurnal variation of peptides in the rat suprachiasmatic nucleus. Analytical Chemistry. 86 (1), 443-452 (2014).
- Chen, X., Wei, S., Ji, Y., Guo, X., Yang, F. Quantitative proteomics using SILAC: Principles, applications, and developments. Proteomics. 15 (18), 3175-3192 (2015).
- Boonen, K., et al. Quantitative peptidomics with isotopic and isobaric tags. Methods in Molecular Biology. 1719, 141-159 (2018).
- Gewehr, M. C. F., et al. The relevance of thimet oligopeptidase in the regulation of energy metabolism and diet-induced obesity. Biomolecules. 10 (2), 321 (2020).
- Fiametti, L. O., Correa, C. N., Castro, L. M. Peptide profile of zebrafish brain in a 6-OHDA-induced Parkinson model. Zebrafish. 18 (1), 55-65 (2021).
- Boersema, P. J., Raijmakers, R., Lemeer, S., Mohammed, S., Heck, A. J. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics. Nature Protocols. 4 (4), 484-494 (2009).
- Dasgupta, S., Castro, L. M., Tashima, A. K., Fricker, L. Quantitative peptidomics using reductive methylation of amines. Methods in Molecular Biology. 1719, 161-174 (2018).
- Tashima, A. K., Fricker, L. D. Quantitative peptidomics with five-plex reductive methylation labels. Journal of the American Society for Mass Spectrometry. 29 (5), 866-878 (2018).
- Che, F. Y., et al. Optimization of neuropeptide extraction from the mouse hypothalamus. Journal of Proteome Research. 6 (12), 4667-4676 (2007).
- Lyons, P. J., Fricker, L. D. Peptidomic approaches to study proteolytic activity. Current Protocols in Protein Science. , 13 (2011).
- Udenfriend, S., et al. Fluorescamine: a reagent for assay of amino acids, peptides, proteins, and primary amines in the picomole range. Science. 178 (4063), 871-872 (1972).
- Ma, B., et al. PEAKS: powerful software for peptide de novo sequencing by tandem mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (20), 2337-2342 (2003).
- Zhang, J., et al. PEAKS DB: de novo sequencing assisted database search for sensitive and accurate peptide identification. Molecular & Cellular Proteomics. 11 (4), 1-8 (2012).
- Sturm, R. M., Dowell, J. A., Li, L. Rat brain neuropeptidomics: tissue collection, protease inhibition, neuropeptide extraction, and mass spectrometric analysis. Methods in Molecular Biology. 615, 217-226 (2010).
- Fricker, L. D. Limitations of mass spectrometry-based peptidomic approaches. Journal of the American Society for Mass Spectrometry. 26 (12), 1981-1991 (2015).
- Ross, , et al. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents. Molecular & Cellular Proteomics. 3, 1154-1169 (2004).
- Thompson, A., et al. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75, 1895-1904 (2003).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved