Method Article
Hallo-C: eine Methode, um die dreidimensionale Architektur des Genoms Study.
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Erratum Notice
Zusammenfassung
Die Hallo-C-Methode ermöglicht unvoreingenommene, genomweite Identifizierung von Chromatin-Wechselwirkungen (1). Hallo-C Paare Ligationssysteme und massiv parallele Sequenzierung. Die resultierenden Daten können verwendet werden, um genomische Architektur auf mehreren Skalen zu studieren: die ersten Ergebnisse identifizierten Features wie Chromosom Gebiete, Segregation von offenen und geschlossenen Chromatin und Chromatin-Struktur an der Megabasen.
Zusammenfassung
Die dreidimensionale Faltung von Chromosomen compartmentalizes das Genom und und kann ferner funktionelle Elemente, wie Promotoren und Enhancer, in enger räumlicher Nähe 2-6 zu bringen. Die Entzifferung der Beziehung zwischen Chromosom Organisation und Genom-Aktivität wird in das Verständnis genomische Prozesse wie Transkription und Replikation unterstützen. Allerdings ist wenig darüber, wie Chromosomen falten bekannt. Mikroskopie ist nicht in der Lage, eine große Zahl von Loci gleichzeitig oder in hoher Auflösung zu unterscheiden. Bislang erforderte die Detektion von chromosomalen Interaktionen mit Chromosom Konformation capture (3C) und seine spätere Anpassungen der Wahl aus einer Reihe von Ziel-Loci, so dass genomweite Studien unmöglich 7-10.
Wir entwickelten Hallo-C, eine Erweiterung von 3C, die in der Lage zu identifizieren langreichweitige Wechselwirkungen in eine unvoreingenommene, genomweiter ist. In Hallo-C werden die Zellen mit Formaldehyd fixiert, wodurch die Interaktion loci miteinander zu durch kovalente DNA-Protein-Quervernetzungen gebunden zu sein. Wenn die DNA anschließend mit einem Restriktionsenzym fragmentiert, so bleiben diese loci verbunden. Ein biotinylierter Rückstand wird aufgenommen, als die 5 'Überhänge in. nächstes blunt-end Ligation unter verdünnten Bedingungen, die Ligation Ereignisse zwischen vernetzten DNA-Fragmente zu Gunsten durchgeführt gefüllt sind. Dies resultiert in einer genomweiten Bibliothek Ligationsprodukte, entsprechend Paare von Fragmenten, die ursprünglich in unmittelbarer Nähe zu einander in den Zellkern wurden. Jeder Ligationsprodukt ist mit Biotin auf dem Gelände der Kreuzung markiert. Die Bibliothek ist geschert, und die Kreuzungen sind mit Streptavidin-Beads-down gezogen. Das gereinigte Kreuzungen können anschließend analysiert mit Hilfe eines High-Throughput-Sequenzer, die sich in einem Katalog von wechselwirkenden Fragmente werden.
Direkte Analyse des resultierenden Kontakt Matrix zeigt zahlreiche Merkmale der genomischen Organisation, wie das Vorhandensein des Chromosoms Territorien und die bevorzugte Verband von kleinen Gen-reiche Chromosomen. Korrelationsanalyse kann, um den Kontakt Matrix angewendet werden, die zeigen, dass das menschliche Genom in zwei Abteilungen getrennt ist: ein weniger dicht gepackt Compartment mit offenen, zugänglichen und aktives Chromatin und einem dichteren Compartment mit geschlossenen, unzugänglich und inaktiven Chromatin Regionen. Schließlich ergab Ensemble Analyse der Kontakt-Matrix, mit theoretischen Ableitungen und Computersimulationen gekoppelt, dass bei der Megabasen Hallo-C Funktionen in Einklang mit einer fraktalen globule Konformation zeigt.
Protokoll
Diese Methode wurde in der Forschung in gemeldet verwendet Lieberman-Aiden et al., Science 326, 289-293 (2009) .
I. Vernetzung, Verdauung, Kennzeichnung von DNA-Enden, und Blunt-end Ligation
- Hallo-C beginnt mit Vernetzung von Zellen, die wie ein roter Faden von allen 3C-basierten Methoden. Um zu beginnen, zwischen 2 x 10 7 und 2,5 x 10 7 Säugetierzellen wachsen, entweder Anhänger oder in Suspension und vernetzen die Zellen. (Einzelheiten zur Vernetzung von Zellen finden Sie unter: 11
- Lyse der Zellen in 550 ul lysisbuffer (500 ul 10 mM Tris-HCl pH 8,0, 10 mM NaCl, 0,2% Igepal CA-630 und 50 ul-Protease-Inhibitoren) mit einem Homogenisator. Drehen Sie das Chromatin bei 5.000 Umdrehungen pro Minute und waschen das Pellet zweimal mit 500 ul 1x NEBuffer 2.
- Resuspendieren Chromatin in 1x NEBuffer 2, Aliquot in 5 nummerierten Röhrchen und fügen 1x NEBuffer 2 bis ein Endvolumen von 362 ul. Müssen 38 ul 1% SDS, vorsichtig mischen und bei 65 ° C 10 Minuten inkubieren. Die Röhrchen wieder auf Eis sofort nach der Inkubation.
- Quench der SDS, indem 44 ul Triton X-100 und vorsichtig mischen. Digest das Chromatin durch Zugabe von 400 Einheiten HindIII und bei 37 ° C über Nacht beim Drehen.
- Die nächsten Schritte sind Hallo-C spezifisch und umfassen Markierung der DNA-Enden mit Biotin und darstellende blunt-end Ligation von vernetzten Fragmenten. Dieser Schritt ermöglicht es Ligation Kreuzungen später gereinigt werden. Tube 1 sollte nicht unterziehen die Biotinylierung Schritt und sollte statt getrennt gehalten werden und dienen als 3C-Steuerung, um die Verdauung zu gewährleisten und Ligation Bedingungen waren optimal.
- Um in das Restriktionsfragment Überhänge füllen und markieren Sie die DNA-Enden mit Biotin in den verbleibenden 4 Röhren, fügen 1,5 ul 10 mM dATP, 1,5 ul 10 mM dGTP, 1,5 ul 10 mM dTTP, 37,5 ul 0,4 mM Biotin-14-dCTP und 10 pl 5U/μl Klenow zu Röhren 2-5. Vorsichtig mischen und Inkubation für 45 Minuten bei 37 ° C.
- Die Röhrchen auf Eis. Zur Inaktivierung der Enzyme, fügen Sie 86 ul 10% SDS, Rohre 1-5. Die Röhrchen bei 65 ° C für genau 30 Minuten und legen sie auf Eis unmittelbar danach.
- Die Ligation wird unter extrem verdünnten Bedingungen durchgeführt, um Ligation Ereignisse zwischen vernetzten Fragmente zu begünstigen. Arbeiten auf dem Eis, hinzu 7,61 ml Ligationsmix [745 ul 10% Triton X-100, 745 ul 10x Ligationspuffer (500 mM Tris-HCl pH 7,5, 100 mM MgCl 2, 100 mM DTT), 80 ul 10 mg / ml BSA , 80 ul 100 mM ATP und 5,96 ml Wasser] auf jedem der fünf Nummern 15 ml-Tuben. Übertragen Sie jede verdaut Chromatin Mischung auf eine entsprechende 15 ml Tube.
- Für die regelmäßige 3C Ligation, mit 10 ul 1U/μl T4 DNA-Ligase zu Schlauch 1. Für blunt-end Hallo-C-Ligation mit 50 ul 1U/μl T4 DNA-Ligase zu Röhren 2-5. Mischen durch Invertieren der Röhrchen und inkubieren alle 5 Röhren für 4 Stunden bei 16 ° C.
- Crosslinks sind vertauscht und Eiweiß abgebaut wird durch Zugabe von 50 ul 10 mg / ml Proteinase K pro Röhre und Inkubation die Röhrchen über Nacht bei 65 ° C. Hinzufügen eines zusätzlichen 50 ul 10 mg / ml Proteinase K pro Röhre am nächsten Tag und auch weiterhin die Inkubation bei 65 ° C für weitere 2 Stunden.
- Kühlen Sie die Reaktionsmischungen auf Raumtemperatur erwärmt und übertragen Sie sie auf fünf 50 ml konische Röhrchen. Purify der DNA in diesen Röhren, indem Sie eine Phenolextraktion. 10 ml Phenol pH 8,0 und Wirbel für 2 Minuten. Drehen Sie das Röhrchen für 10 Minuten bei 3.500 rpm und transferieren es vorsichtig so viel von der wässrigen Phase wie möglich in ein neues 50 ml Tube.
- Die Extraktion mit Phenol pH 8,0: Chloroform (1:1) und Fällung der DNA mit Ethanol. (Einzelheiten zur DNA-Aufreinigung, siehe: 11
- Nach Zentrifugation der DNA mit Ethanol gefällt, löst jedes DNA-Pellet in 450 ul 1x TE (10 mM Tris-HCl pH 8,0, 1 mM EDTA). Übertragen Sie die DNA-Gemisch zu einem 1,7-ml-Zentrifugenröhrchen.
- Chloroform-Extraktionen: Eine weitere Runde der Reinigung ist by doing 2 Phenol durchgeführt. Add 500 ul Phenol pH 8,0: Chloroform (1:1) und Vortex für 1 Minute. Zentrifugieren für 5 Minuten bei 14.000 rpm und Transfer der wässrigen Phase in ein neues Röhrchen. Nach der zweiten Extraktion, Fällung der DNA durch Zugabe von 0,1 x Volumen von NaOAc, 2x Volumen von 100% Ethanol und Inkubation 30 Minuten bei -80 ° C.
- Nach Abzentrifugieren des ausgefallenen DNA, waschen jedes DNA-Pellet mit 70% Ethanol und resuspendieren jedes DNA-Pellet in 25 ul 1x TE. Degrade jede RNA, die vorhanden sein könnten durch Zugabe von 1 ul 1 mg / ml RNAse A pro Röhre und Inkubation die Röhrchen für 30 Minuten bei 37 ° C. Pool der Hallo-C-Gehalte von Rohren 2-5, noch halten Rohr 1 separates als 3C-Steuerung.
- Jetzt ist eine gute Gelegenheit, die Hallo-C-Markierung und Ligation Effizienz zu untersuchen. Diese Kontrollen sind ausgezeichnete Indikatoren, ob ein Hallo-C-Bibliothek geht, erfolgreich zu sein.
- Zur Überprüfung der Qualität und Quantität der Bibliotheken, run 2 ul und 6 ul Aliquots von 1:10 Verdünnungen von beiden 3C und Hallo-C-Bibliotheken auf einem 0,8% Agarosegel. (Siehe Abbildung 2a)
- Hallo-C-Kennzeichnung und Hallo-C Ligation Effizienz wird durch eine PCR-Assay verdauen überprüft. Erfolgreiche fill-in und Ligation von einer HindIII-Stelle (AAGCTT) wird eine Website erstellt für das Restriktionsenzym NheI (GCTAGC). Eine besondere Ligationsprodukt aus zwei nahe gelegenen Restriktionsfragmente gebildet wird mit PCR (wie in 3C 11 mit 0,2 ul jeder Bibliothek als Template amplifiziert. Die PCR-Produkte werden anschließend mit HindIII, NheI oder beide verdaut. Nach dem Ausführen der Proben auf einem 2% Gel kann die relative Anzahl der 3C und Hallo-C Ligation Ereignisse durch die Quantifizierung der Intensität der geschnittenen und ungeschnittenen Bands (siehe Abbildung 2B) geschätzt werden.
- Einige Fragmente werden nicht unterbunden haben: zu vermeiden, ziehen sie dann später, entfernen Biotin aus diesen unligierten endet mit der Exonuklease-Aktivität der T4 DNA-Polymerase.
- Biotin-14-dCTP bei nicht-ligierten DNA-Enden mit der Exonuklease-Aktivität der T4-DNA-Polymerase entfernt. Mix 5 ug Hallo-C-Bibliothek mit 1 ul 10 mg / ml BSA, 10 ul 10x NEBuffer 2, 1 ul 10 mM dATP, 1 ul 10 mM dGTP und 5 Units T4-DNA-Polymerase in einem Gesamtvolumen von 100 ul und inkubieren Mischung bei 12 ° C für 2 Stunden. Wenn möglich, werden mehrere 5 ug Reaktionen durchgeführt.
- Die Reaktion wird durch Zugabe von 2 ul 0,5 M EDTA pH 8,0 gestoppt.
- Chloroform (1:1)-Extraktion erfolgt gefolgt von Ethanol-Fällung: Um die DNA, ein Phenol pH 8,0 zu reinigen.
- Der Überstand wird verworfen und die DNA-Pellets werden resuspendiert und in einem Gesamtvolumen von 100 ul Wasser vereinigt.
II. Scher-und Größenauswahl
- Um die biotinylierten DNA geeignet für Hochdurchsatz-Sequenzierung, muss die DNA zu einer Größe von 300-500 Basenpaaren mit einem Covaris S2 Instrument (duty cycle 5, Stufe 5, Zyklen / Burst 200, Zeit 60 Sekunden für 4 Zyklen) geschert werden .
- So reparieren Sie die geschert DNA-Enden, fügen Sie 14 ul 10x Ligationspuffer, 14 ul 2,5 mM dNTP-Mix, 5 ul T4-DNA-Polymerase, 5 ul T4-Polynukleotid-Kinase, 1 ul Klenow-DNA-Polymerase und 1 ul Wasser. Inkubieren für 30 Minuten bei Raumtemperatur.
- Nach der Inkubation mit einem Qiagen MinElute Spalte, um die DNA nach den Empfehlungen des Herstellers zu reinigen. Eluieren der DNA zweimal mit 15 ul 1x Tris-Low-EDTA (TLE: 10 mM Tris pH 8,0, 0,1 mM EDTA). Dann fügen Sie eine dATP an das 3'-Enden der end-reparierte DNA durch Zugabe von 5 ul 10x NEBuffer2, 10 ul 1 mM dATP, 2 ul Wasser und 3 ul Klenow (exo-). Inkubieren Sie die Reaktion für 30 Minuten bei 37 ° C.
- Zur Inaktivierung des Klenow-Fragment, inkubieren Sie die Reaktionen für 20 Minuten bei 65 ° C und anschließend kühl die Reaktionen auf Eis. Mit einem Speedvac, reduzieren Sie die Reaktionsvolumina bis 20 ul.
- Anschließend laden Sie die DNA in einem 1,5% Agarosegel mit 1X TAE und führen für 3,5 Stunden bei 80-90V. Nach der Färbung des Gels mit SYBR Grün, visualisieren die DNA auf einem DarkReader. Excise DNA-Fragmente zwischen 300 und 500 Basenpaaren und reinige sie mit einem Qiagen Gel Extraction Kit mit 2-4 Spalten je nach Gewicht des Gels. Eluieren der DNA mit 50 ul 1x TLE.
- Kombinieren Sie den Eluaten aus dem QIAquick Säulen und bringen das endgültige Volumen von bis zu 300 ul mit 1x TLE. Schließlich bestimmen die DNA-Konzentration mit dem Quant-iT-Assay mit dem Qubit Fluorometer und berechnen die Gesamtmenge der DNA.
III. Biotin Pull-down-und Paired-End-Sequenzierung
- In diesem Abschnitt des Protokolls, Ligation Kreuzungen aus dem DNA-Pool gereinigt, so dass für eine effiziente Identifizierung von interagierenden Chromatin-Fragmente durch Paired-End-Sequenzierung. Führen Sie alle weiteren Schritte in DNA LoBind Röhrchen.
- Bereiten Perlen für Biotin-pull-down durch Waschen 150 pl resuspendiert magnetische Streptavidin-Beads zweimal mit 400 ul Tween-Puffer (TB: 5 mM Tris-HCl pH 8,0, 0,5 mM EDTA, 1 M NaCl, 0,05% Tween).
Diese und zukünftige wäscht bestehen aus fünf Schritten:- Add-Puffer, um die Perlen
- Übertragen Sie die Mischung in ein neues Röhrchen
- Drehen Sie die Probe für 3 Minuten bei Raumtemperatur
- Reclaim the Perlen mit einem magnetischen Partikel-Konzentrator
- Entfernen Sie den Überstand
- Resuspendieren der Beads in 300 ul 2x Nein Tween-Puffer (2x NTB: 10 mM Tris-HCl pH 8,0, 1 mM EDTA, 2 M NaCl) und verbinden sich mit 300 pl Hallo-C DNA. Lassen Sie die Biotin markiert Hallo-C DNA an die Streptavidin-Beads durch Inkubation der Mischung bei Raumtemperatur für 15 Minuten mit einer Rotation zu binden.
- Reclaim the DNA gebunden Streptavidinbeads mit der magnetischen Partikel konzentrator, und entfernen Sie den Überstand. Waschen Sie die Perlen in 400 ul 1x NTB (5 mM Tris-HCl pH 8,0, 0,5 mM EDTA, 1 M NaCl), gefolgt von 100 ul 1x Ligationspuffer gefolgt. Resuspendieren der Beads in 50 ul 1x Ligationspuffer und die Mischung in ein neues Röhrchen.
- Zur Vorbereitung der DNA für Illumina Paired End-Sequenzierung, nehmen Sie die Gesamtmenge der DNA als Input für die Biotin-pull-down, die zuvor in Schritt 2.6 wurde berechnet, verwendet, und teilen sie mit 20 um die Höhe der Hallo-C-DNA, hat Schätzungen wurde abgerissen und ist verfügbar für die Ligation. Fügen Sie 6 Picomol Illumina Paired End Adapter pro pg Hallo-C DNA für Ligation. Verwenden Sie 1200 Einheiten T4 DNA Ligase mit den Adaptern, um die DNA zu unterbinden. Inkubieren für 2 Stunden bei Raumtemperatur.
- Entfernen Sie nicht ligierten Paired End-Adapter durch Rückgewinnung der Hallo-C DNA gebunden Perlen und Waschen der Beads zweimal mit 400 ul 1x TB.
- Waschen Sie die Perlen mit 200 ul 1x NTB von 200 ul und dann 50 ul 1x NEBuffer 2 folgte. Nach der letzten Waschung, resuspendieren die Perlen in 50 ul 1x NEBuffer 2 und Transfer in ein neues Röhrchen.
- Um zu bestimmen, die Anzahl der Zyklen erforderlich, um genügend PCR-Produkt für die Sequenzierung zu generieren, Einrichtung von vier Test-PCR-Reaktionen mit 6, 9, 12 oder 15 Zyklen. (Für Details der PCR-Amplifikation finden: 12 Bestimmen Sie die optimale Zyklenzahl, indem Sie die PCR-Reaktionen auf einem 5% Polyacrylamidgel und Färbung mit SYBR Green, Sicherstellung der Abwesenheit von störenden Bänder und das Vorhandensein eines Abstrichs zwischen 400 -. 600 Basenpaare, die die Länge des geschert Produkte nach Ligation der Adapter ist.
- Amplify den Rest der Hallo-C-Bibliothek gebunden Streptavidinbeads in einer groß angelegten PCR mit der optimalen Anzahl von PCR-Zyklen. Pool der PCR-Produkte aus den einzelnen Brunnen und Rückforderung der Perlen. Halten Sie 1% der in großem Maßstab PCR-Produkt zu trennen, um auf einem Gel laufen und reinigen den Rest des PCR-Produkts mit 1,8 x Volumen AMPure Perlen nach den Empfehlungen des Herstellers.
- Eluieren der DNA mit 50 ul 1x TLE-Puffer und vergleichen Sie 1% der AMPure bead gereinigte PCR-Produkt, um die 1%-Aliquot des ursprünglichen PCR-Produkt auf einem 5% Polyacrylamidgel, die Gewährleistung der erfolgreichen Entfernung der PCR-Primer.
- Wir empfehlen auch das Klonen 1 ul der Hallo-C-Bibliothek und die Bestimmung des Produkts von etwa 100 Klone mit Sanger-Sequenzierung. Dies ermöglicht es Ihnen, die relative Zahl der ausrichtbar Hallo-C liest in der PCR-Ansatz zu bewerten. Für typische Ergebnisse, siehe Abbildung 3B.
- Sequence die Hallo-C-Bibliothek mit Illumina gepaart Ende Sequenzierung. Richten Sie jedem Ende selbständig mit Maq (http://maq.sourceforge.net/) zu interagieren Chromatin-Fragmente zu identifizieren.
IV. Vertreter Hallo-C Ergebnisse
- Die folgenden Ergebnisse werden erwartet, wenn die Hallo-C-Protokoll ist technisch gut und ausgeführt werden können berücksichtigt werden Standards der Qualitätskontrolle.
- Qualitätskontrolle Schritte sollten zeigen, dass sowohl 3C und Hallo-C-Bibliotheken als eher engen Banden größer als 10 kb laufen. Ein DNA-Abstrich deutet auf eine schlechte Ligationseffizienz. Typischerweise wird Ligationseffizienz etwas niedriger in einem Hallo-C-Bibliothek als eine 3C-Template (siehe Abbildung 2a) verglichen.
- Hallo-C-Markierung und Ligation Effizienz kann durch die Verdauung eines PCR-Produkts unter Verwendung 3C Primer geschätzt werden. 3C-Übergänge sind durch HindIII geschnitten und nicht durch NheI. Das Gegenteil gilt für Hallo-C-Übergänge wahr. Das PCR-Assay verdauen zeigt, dass 70% der Hallo-C-Fragmente durch NheI und nicht durch HindIII geschnitten, bestätigt effiziente Kennzeichnung von Kreuzungen Ligation (siehe Abbildung 2B).
- Die Analyse der sequenzierten liest sollte zeigen, dass liest aus beiden intrachromosomale und interchromosomale Interaktionen, durch die blauen und roten Linien angedeutet, ausrichten deutlich näher an HindIII Restriktionsschnittstellen zu zufällig generierten verglichen liest, grün dargestellt (siehe Abbildung 3A).
- In einem erfolgreichen Versuch dar 55% der ausrichtbar lesen Paaren interchromosomale Wechselwirkungen. Fünfzehn Prozent vertreten intrachromosomale Wechselwirkungen zwischen Fragmenten von weniger als 20 kb voneinander entfernt und 30% sind intrachromosomale lesen Paare, die mehr als 20 kb voneinander entfernt sind (siehe Abbildung 3B). Diese Verteilung kann vor Hochdurchsatz-Sequenzierung abgetastet werden, als eine Form der Qualitätskontrolle; das Klonen und die Sanger-Sequenzierung von etwa 100 Klone ist in der Regel ausreichend.
- Die Chromatin-Wechselwirkungen können visualisiert werden als Heatmap, wo die x-und y-Achsen stellen loci in genomischer Ordnung und jedes Pixel steht für die Anzahl der beobachteten Interaktionen zwischen ihnen. Typischerweise werden DNA-Fragmente, die sehr nahe beieinander liegen im linearen Genom haben die Tendenz, häufig miteinander interagieren. Dies ist in der intrachromosomale Heatmaps als prominenter diagonal (Siehe Abbildung 4A) gesehen.
- Die folgenden Ergebnisse zeigen verschiedene Möglichkeiten der Analyse der Daten auf verschiedenen Ebenen der Genom-Organisation zu enthüllen. Trägt man den Kontakt probability gegen genomische Abstand (siehe Abbildung 5A) zeigt, dass die Wahrscheinlichkeit des Kontaktes nimmt als Funktion der genomischen Abstand und erreichte schließlich ein Plateau. Bei jeder Entfernung, sind intrachromosomale Interaktionen, in die durchgezogene Linie dargestellt, angereichert gegenüber interchromosomale Interaktionen, vertreten durch die gestrichelten Linien. Dies impliziert unmittelbar das Vorhandensein des Chromosoms Territorien.
- Die Berechnung der beobachteten / erwarteten Anzahl von interchromosomale Kontakte zwischen allen Paaren von Chromosomen zeigt bevorzugte Assoziation zwischen bestimmten Chromosomenpaare. Kleine Gen-reiche Chromosomen bevorzugt miteinander interagieren, angedeutet durch die leuchtend rote Farbe (siehe Abbildung 5B).
- Individuelle Chromosomen können ebenfalls untersucht werden. Die rohen Heatmap kann mit einem erwarteten Heatmap zur Rechenschaft für die genomische Abstand zwischen den Paaren der loci, was zu einer beobachteten / erwarteten Heatmap werden. Dann kann eine Korrelationsmatrix durch Korrelation der Zeilen und Spalten der beobachteten / erwarteten Heatmaps hergestellt werden. Mit Korrelationsanalyse wird gezeigt, dass das menschliche Genom zerfällt in zwei Abteilungen. Dies wird durch die plaid-Muster in die Korrelation Heatmaps (siehe Abbildung 4A-D) dargestellt. (Details zu den Hallo-C Datenanalyse finden: 1.
- Mit Hallo-C-Daten wurden neue Einblicke in Chromatin Falten an der Megabasen gewonnen. Das klassische Modell des Polymers Kondensation schlägt vor, dass Chromatin verpackt in ein Gleichgewicht Kugel. Plotten Kontakt Wahrscheinlichkeit als Funktion des Abstandes zeigt, dass der Kontakt Wahrscheinlichkeit Skalen als ein Potenzgesetz mit genomischer Distanz, deren Steigung beträgt ca. -1 (siehe Abbildung 6A). Dies ist nicht konsistent mit dem Verhalten eines Gleichgewichts globule, aber nicht den Erwartungen für eine alternative Struktur wie ein Fraktal globule (siehe Abbildung 6B) bekannt.
- Hier sind zwei globuläre Strukturen dargestellt. Coloration entspricht Abstand von einem Endpunkt, von blau bis cyan, grün, gelb, orange und rot (siehe Abbildung 6c oben). Im Gegensatz Gleichgewicht Globuli, fehlende fraktale Kügelchen Verstrickungen. In einer fraktalen globule neigen loci, dass in der Nähe entlang der Kontur sind in der Nähe sein in 3D, was auf das Vorhandensein von monochromatischem Blöcke (siehe Abbildung 6c Mitte). Solche Blöcke sind nicht im Gleichgewicht Kugel (siehe Abbildung 6c unten) gefunden.
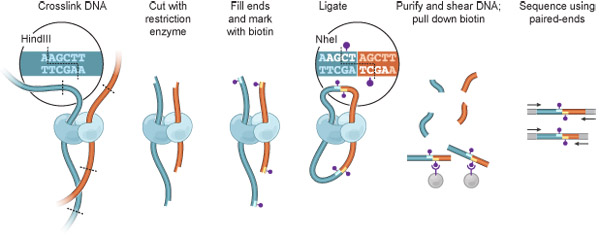
Abbildung 1. Hallo-C Überblick. Die Zellen sind mit Formaldehyd vernetzt, was in kovalenten Verbindungen zwischen räumlich benachbarten Chromatin-Segmente (DNA-Fragmente: dunkelblau, rot, Proteine, die solche Interaktionen vermitteln können, sind in hellblau und cyan dargestellt). Chromatin mit einem Restriktionsenzym verdaut wird (hier HindIII; Restriktionsstelle: gestrichelten Linie, siehe Kasten). Die daraus resultierenden klebrigen Enden mit Nukleotiden, von denen eines biotinylierten (lila Punkt) gefüllt. Ligation ist unter extrem verdünnten Bedingungen begünstigen intramolekulare Ligation Veranstaltungen durchgeführt, die HindIII-Stelle verloren geht und eine NheI-Website erstellt (kleines Bild). DNA wird gereinigt und geschert, und biotinylierten Kreuzungen sind isoliert mit Streptavidin-Beads. Interaktion Fragmente werden durch Paired-End-Sequenzierung identifiziert.

Abbildung 2. Hallo-C-Bibliothek Qualitätskontrollen. (A) Steigende Mengen eines 3C-Steuerung und ein Hallo-C-Bibliothek wurden auf einem 0,8% Agarosegel aufgelöst. Beide Bibliotheken laufen als ein eher enges Band größer als 10 kb. Typische Ligation Effizienz in einem Hallo-C-Bibliothek ist etwas niedriger als das, was in einem 3C Vorlage beobachtet und wird durch den Abstrich in die Hallo-C Fahrspuren angezeigt. (B) PCR verdauen zu kontrollieren. Ein Verknüpfungsstelle von zwei nahe gelegenen Fragmente gebildet wird verstärkt mit Standard-3C PCR-Bedingungen. Hallo-C Ligationsprodukte können von denen in herkömmlichen 3C durch Verdauung der Ligationsstelle hergestellt wurden, unterschieden werden. Hallo-C-Übergänge sind durch NheI, nicht HindIII geschnitten, die Rückseite ist für 3C Kreuzungen wahr. 70% der Hallo-C Amplikons wurden durch NheI geschnitten, bestätigt effizienten Markierung von Verknüpfungsstelle. Zwei repliziert wurden durchgeführt, um verlässliche Quantifizierung zu gewährleisten.

Abbildung 3. Hallo-C gelesen Qualitätskontrollen. (A) Liest aus Fragmenten entsprechend den beiden intrachromosomale (blau) und interchromosomale (rot) Wechselwirkungen align deutlich näher an HindIII Restriktionsschnittstellen zu zufällig generierten liest (grün) verglichen. Sowohl die intrachromosomale liest und liest interchromosomale Kurven schnell wie der Abstand von der HindIII-Stelle erhöht sinken bis zu einem Plateau in einem Abstand von ~ 500 bp erreicht wird. Dies entspricht der maximalen Fragmentgröße für die Sequenzierung verwendet. (B) In der Regel stellen 55% der ausrichtbar lesen Paaren interchromosomale Wechselwirkungen. Fünfzehn Prozent vertreten intrachromosomale Wechselwirkungen zwischen Fragmenten von weniger als 20 kbauseinander und 30% sind intrachromosomale lesen Paare, die mehr als 20 kb voneinander entfernt sind. Diese Verteilung kann vor Hochdurchsatz-Sequenzierung abgetastet werden, als eine Form der Qualitätskontrolle; das Klonen und die Sanger-Sequenzierung von etwa 100 Klone ist in der Regel ausreichend.

Abbildung 4. Korrelationsanalyse zeigt, dass der Kern in zwei Abteilungen wird getrennt. (A) Heatmap entsprechend intrachromosomale Interaktionen auf Chromosom 14. Jedes Pixel repräsentiert alle Interaktionen zwischen einem 1-Mb-Locus und ein weiteres 1-Mb-Locus; Intensität entspricht der Gesamtzahl der liest (Bereich: 0-200 liest). Teilstriche werden alle 10 Mb. Die Heatmap zeigt Unterbau in Form eines intensiven Diagonale und eine Konstellation von großen Blöcken. (Chromosom 14 akrozentrischen ist;. Kurzen Arm ist nicht gezeigt) mit dem Hallo-C Dataset, um die durchschnittliche Kontakt Wahrscheinlichkeit für ein Paar loci zu einem bestimmten genomischen Abstand berechnen, ist eine Erwartung Matrix (B) entspricht, was wäre, produziert beobachtet, wenn es keine langfristige Strukturen. Der Quotient aus diesen beiden Matrizen ist eine beobachtete / erwartete Matrix (C), wo Raubbau in blau und Bereicherung in rot [Bereich: 0,2 (blau) bis 5 (rot)] wird angezeigt. Der Block Muster wird deutlicher. Die Korrelationsmatrix (D) zeigt die Korrelation [Bereich: -1 (blau) bis 1 (rot)] zwischen den intrachromosomale Interaktion Profile von jedem Paar loci entlang Chromosom 14. Die markante Karomuster zeigt die Anwesenheit von zwei Fächern innerhalb des Chromosoms.

Abbildung 5. Das Vorhandensein und die Organisation der Chromosomen Territorien. (A) Wahrscheinlichkeit des Kontaktes nimmt als Funktion der genomischen Abstand auf Chromosom 1 und erreichte schließlich ein Plateau bei ~ 90MB (blau). Die Höhe der interchromosomale Kontakt (schwarze Striche) unterscheidet sich für verschiedene Chromosomenpaare; Loci auf Chromosom 1 sind am ehesten mit Loci auf Chromosom 10 (grün gestrichelt) und am wenigsten wahrscheinlich mit Loci auf Chromosom 21 (rot gestrichelt) interagieren interagieren. Interchromosomale Wechselwirkungen in Bezug auf intrachromosomale Interaktionen erschöpft. (B) der beobachteten / erwarteten Anzahl von interchromosomale Kontakte zwischen allen Paaren von Chromosomen. Rot bedeutet Bereicherung, und Blau Erschöpfung [Bereich: 0,5 (blau) bis 2 (rot)]. Klein, Gen-reiche Chromosomen neigen dazu, mehr mit einander interagieren.

Abbildung 6. Die lokale Verpackung von Chromatin ist konsistent mit dem Verhalten eines fraktalen globule. (A) Kontakt Wahrscheinlichkeit als Funktion der genomischen Abstand, gemittelt über das Genom (blau). Ein prominentes Potenzgesetz Skalierung zwischen 500kb und 7Mb (schraffierte Bereich) mit einer Steigung von -1,08 (in cyan dargestellt fit) zu sehen. (B) Ergebnisse der Simulation für den Kontakt Wahrscheinlichkeit als Funktion des Abstands zum Gleichgewicht (rot) und fraktalen (blau) Globuli. Die Steigung für eine fraktale Kügelchen ist sehr knapp -1 (cyan) und bestätigen unsere neuartige theoretische Vorhersage 1. Die Steigung für ein Gleichgewicht Kugel ist -3 / 2, welche Matches vor theoretischen Erwartungen. Die Steigung für die fraktale Kügelchen ähnelt der Piste in die Hallo-C Ergebnisse beobachtet, während die Neigung für ein Gleichgewicht Kugel ist nicht in der Hallo-C-Daten zu sehen. (C) Top: Eine entfaltete Polymerkette, 4000 Monomeren lang. Coloration entspricht Abstand von einem Endpunkt, von blau bis cyan, grün, gelb, orange und rot Mitte:. Typisches Beispiel für ein Fraktal globule aus unserem Ensemble gezogen. Fractal Globuli Mangel Verstrickungen. Loci, die in der Nähe entlang der Kontur sind in der Regel in der Nähe in 3D, was auf das Vorhandensein von großen monochromen Blöcke, die offensichtlich auf der Oberfläche und im Querschnitt Unten:. Ein Gleichgewicht Kugel. Die Struktur ist sehr verwickelt; loci, dass in der Nähe entlang der Kontur (ähnlicher Farbe) sind, müssen nicht in der Nähe in 3D.
Diskussion
Wir präsentieren eine Methode zur Untersuchung der 3-dimensionalen Architektur des Genoms durch die Abbildung Chromatin-Wechselwirkungen in eine unvoreingenommene, genomweite Weise. Die wichtigsten experimentellen Schritt, was diese Technologie abhebt von früheren Arbeiten - ist der Einbau von biotinylierten Nukleotiden an die Beschränkung Enden der vernetzten Fragmenten vor blunt-end Ligation. Performing dieser Schritt erfolgreich ermöglicht tiefe Sequenzierung aller Ligation Kreuzungen und gibt Hallo-C ihres Umfangs und ihrer Macht.
Die Zahl der liest, wird letztlich die Auflösung der Interaktion Karten. Hier wird eine 1 Mb Interaktion Karte für den menschlichen Genoms vorgestellt mit ~ 30 Mio. ausrichtbar liest. Um "Allzweck-" Auflösung um einen Faktor von n zu erhöhen, muss die Anzahl der Lese mit einem Faktor von n 2 erhöht werden.
Die Hallo-C-Technik kann leicht mit anderen Techniken, wie zum Beispiel Hybrid-Capture-Bibliothek nach Generation (auf bestimmte Teile des Genoms Ziel) und Chromatin-Immunopräzipitation nach Ligation (die Chromatin Umfeld der Regionen mit spezifischen Proteinen assoziiert zu untersuchen) kombiniert werden.
Offenlegungen
Danksagungen
Wir danken A. Kosmrlj für Diskussionen und Code; AP Aiden, XR Bao, M. Brenner, D. Galas, W. Gosper, A. Jaffer, A. Melnikov, A. Miele, G. Giannoukos, C. Nusbaum, AJM Walhout , L. Wood, und K. Zeldovich für Diskussionen, und L. Gaffney und B. Wong Hilfe mit Visualisierung.
Unterstützt durch eine Fannie und John Hertz-Stiftung Promotionsstipendium, ein National Defense Science and Engineering Graduate Fellowship, ein NSF Promotionsstipendium, das National Space Biomedical Research Institute, und gewähre keine. T32 HG002295 vom National Human Genome Research Institute (NHGRI) (EL); i2b2 (Informatik für die Integration von Biologie und der Bedside), der NIH-unterstützten Center for Biomedical Computing an der Brigham and Women s Hospital (LAM), Grant No. HG003143 vom NHGRI und ein Keck Foundation aufstrebenden Young Scholar Award (JD). Raw und kartiert Hallo-C-Sequenz-Daten an die GEO-Datenbank (hinterlegt worden www.ncbi.nlm.nih.gov/geo/ ), Beitritt nein. GSE18199. Weitere Visualisierungen sind verfügbar unter http://hic.umassmed.edu .
Materialien
| Name | Company | Catalog Number | Comments |
| Protease inhibitors | Sigma-Aldrich | P8340-5ml | Step 1.2 |
| biotin-14-dCTP | Invitrogen | 19518-018 | Step 1.6 |
| Klenow | New England Biolabs | M0210 | Steps 1.6 and 2.2 |
| T4 DNA ligase | Invitrogen | 15224 | Step 1.9 |
| T4 DNA polymerase | New England Biolabs | M0203 | Steps 1.17 and 2.2 |
| 10x ligation buffer | New England Biolabs | B0202 | Steps 2.2 and 3.4 |
| T4 PNK | New England Biolabs | M0201 | Step 2.2 |
| Klenow (exo-) | New England Biolabs | M0212 | Step 2.3 |
| Dynabeads MyOne Streptavin C1 Beads | Invitrogen | 650.01 | Step 3.2 |
| T4 DNA ligase HC | Enzymatics | L603-HC-L | Step 3.5 |
| Phusion HF mastermix | New England Biolabs | F531 | Step 3.8 |
| Ampure beads | Beckman Coulter Inc. | A2915 | Step 3.9 |
Referenzen
- Lieberman-Aiden, E., Van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I., Lajoie, B. R., Sabo, P. J., Dorschner, M. O., Sandstrom, R., Bernstein, B., Bender, M. A., Groudine, M., Gnirke, A., Stamatoyannopoulos, J., Mirny, L. A., Lander, E. S., Dekker, J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Kosak, S. T., Groudine, M. Form follows function: the genomic organization of cellular differentiation. Genes and Dev. 18, 1371-1384 (2004).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (2007).
- Dekker, J. Gene Regulation in the Third Dimension. Science. 319, 1793-1794 (2008).
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet. 2, 292-301 (2001).
- Sexton, T., Schober, H., Fraser, P., Gasser, S. M. Gene regulation through nuclear organization. Nat Struct and Mol Biol. 14, 1049-1055 (2007).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing Chromosome Conformation. Science. 295, 1306-1311 (2002).
- Zhao, Z., Tavoosidana, G., Sjölinder, M., Göndör, A., Mariano, P., Wang, S., Kanduri, C., Lezcano, M., Sandhu, K. S., Singh, U., Pant, V., Tiwari, V., Kurukuti, S., Ohlsson, R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347 (2006).
- Simonis, M., Klous, P., Splinter, E., Moshkin, Y., Willemsen, R., de Wit, E., van Steensel, B., de Laat, W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1348-1354 (2006).
- Dostie, J., Richmond, T. A., Arnaout, R. A., Selzer, R. R., Lee, W. L., Honan, T. A., Rubio, E. D., Krumm, A., Lamb, J., Nusbaum, C., Green, R. D., Dekker, J. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Res. 16, 1299-1309 (2006).
- Miele, A., Dekker, J. Mapping Cis- and Trans Chromatin Interaction Networks Using Chromosome Conformation Capture (3C). Methods Mol Biol. 464, 105-121 (2009).
- Maccallum, I., Przybylski, D., Gnerre, S., Burton, J., Shlyakhter, I., Gnirke, A., Malek, J., McKernan, K., Ranade, S., Shea, T. P., Williams, L., Young, S., Nusbaum, C., Jaffe, D. B. ALLPATHS 2: small genomes assembled accurately and with high continuity from short paired reads. Genome Biol . 10, R103-R103 (2009).
Erratum
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten