Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Erkennung und Genogrouping von Noroviren aus Kinder-Hocker mit der TaqMan One-Step RT-PCR
In diesem Artikel
Zusammenfassung
Ein Ein-Schritt-RT-PCR-Assay zum Nachweis und zur Genogruppe Identifizierung von Norovirus Isolate von Kindern der Stuhl, die Primer und TaqMan-Sonden spezifisch für die offenen Leserahmen 1 (ORF1)-ORF2 Übergangsregion, die am stärksten konservierte Region des Norovirus Genom verwendet beschrieben. Eine nicht-kommerzielle, kostengünstige RNA-Extraktion-Methode wird ausführlich beschrieben.
Zusammenfassung
Noroviren (NoVs) sind die führende Ursache von Ausbrüchen der sporadischen akuten Gastroenteritis weltweit bei Menschen aller Altersgruppen. Sie sind wichtige Ursachen für Hospitalisierungen bei Kindern mit einer gesundheitlichen Auswirkungen ähnlich der von Rotavirus. NoVs sind RNA-Viren von großer genetischer Vielfalt und es gibt eine kontinuierliche Auftreten neuer Stämme. Fünf Genogruppen anerkannt werden; GI und GII mit ihren vielen Genotypen und Subtypen das wichtigste für eine Infektion beim Menschen. Allerdings ist die Diagnose dieser beiden Genotypen problematisch, Verzögern Diagnose und Behandlung. 1, 2, 3
Für die RNA-Extraktion aus Stuhlproben der am häufigsten verwendete Methode ist die QIAmp Viral RNA kommerziellen Kit von Qiagen. Dieses Verfahren kombiniert die Bindungseigenschaften eines Silicagelmembran, Puffer, dass die Kontrolle RNasen und eine optimale Bindung der RNA an der Säule zusammen mit der Geschwindigkeit des MicroSpin. Diese Methode ist einfach, schnell und zuverlässig und ist Carrin wenigen Schritten, die in der Beschreibung des Herstellers detailliert sind IED.
Norovirus ist nur noch von Rotavirus als die häufigste Ursache von Durchfall. Norovirus-Diagnostik sollte in allen Studien über die Pathogenese von Durchfall als auch in Ausbrüchen oder einzelne Durchfall Fällen zur Verfügung. Derzeit jedoch Norovirus Diagnose ist auf nur wenigen Zentren aufgrund des Fehlens von einfachen Methoden der Diagnose beschränkt. Dies verzögert Diagnose und Behandlung 1, 2, 3. Darüber hinaus bedienen sich aufgrund der hohen Kosten und geregelten Transport von korrosiven Puffer innerhalb und zwischen den Ländern dieser gefertigten Bausätze wirft logistische Probleme. Als Ergebnis werden in diesem Protokoll beschreiben wir eine Alternative, wirtschaftlichen, Hausmethode die auf der ursprünglichen basiert Boom et al. Verfahren 4, die die Nukleinsäure-bindenden Eigenschaften von Siliciumdioxid-Teilchen verwendet zusammen mit den Anti-Nuklease Eigenschaften Guanidiniumthiocyanat .
et al. 5 beschrieben. Die Sequenzierung des PCR-Produkt aus der herkömmlichen PCR ermöglicht die Unterscheidung von Genotypen aus der GI und GII Genogruppen.
Protokoll
1. Stuhlproben
- Stuhlproben sollten eingefroren gelagert, um die RNA zu bewahren. Um eine 10% fäkal Federung machen, dauert ca. 0,1 g aufgetauten Stuhlprobe und füllen auf 1 ml mit PBS.
- Aliquotieren in 200 ul zu vermeiden, wiederholtes Einfrieren und Auftauen. Lagern Sie die Aliquots bei -70 ° C.
- Auftauen und zentrifugieren Aliquots bei 4.000 g für 10 min vor der Verwendung in Extraktion.
2. Vorbereitung von Silica-Partikeln für die Extraktion mit Guanidin & Silica
- Bei RT auszusetzen, 30 g Siliciumdioxid und komplett mit dH 2 O bis 250 mL.
- Nach 24 Stunden der Sedimentation, zu entfernen 215 ml des Überstands durch Sog. Fügen Sie dH 2 O bis zu 250 ml, sowie die Aussetzung der Kieselsäure Pellet durch Schütteln.
- Nach weiteren 24 Stunden wurde der Überstand entfernt durch Absaugen und der pH-Wert der Kieselsäure-Suspension auf pH 2,0 unter Verwendung von Salzsäure (HCl). Aliquot der Kieselsäuresuspension in Glas BotFlaschen und Autoklaven.
3. Vorbereitung der L6 Puffer für die Extraktion mit Guanidin & Silica
- Bei RT-Puffer vorzubereiten L6 durch Lösen von 60 g Guanidiniumthiocyanat (GuSCN) und 1,3 g Triton X-100 in 50 ml 0,1 M Tris-Hydrochlorid, pH 6,4 und 11 ml einer 0,2 M EDTA, pH 8,0 Lösung.
4. Vorbereitung der L2-Puffer für die Extraktion mit Guanidin & Silica
- Bei RT Herstellung L2-Puffer durch Auflösen von 180 g Guanidiniumthiocyanat (GuSCN) in 150 ml 0,1 M Tris-Hydrochlorid, pH 6,4.
5. Extraktionsverfahren
- In jeder Extraktion ein NoV positive Stuhlprobe, als positive Kontrolle und DEPC-behandeltem Wasser, als negative Kontrolle, einbezogen werden sollten.
- In einem Mikro-Zentrifugenröhrchen das folgende; 1 ml Puffer L6, 20 uL von Silica-Partikel, und fügen Sie 200 ul der 10% fäkal-Extrakt Suspension. Vortex kurz verlassen und bei RT für 15 min.
- Zentrifugieren Sie die Suspension bei 6000 g für 10 s und Waschen des Pellets mit 1 ml Puffer L2 zweimal, gefolgt von zwei Waschungen mit 70% Ethanol und einmal mit Aceton. Trocknen Sie das Pellet in einem trockenen Heizblock bei 56 ° C für 5 min.
- Hydrat die RNA in der Silica-Pellet durch Zugabe von 50 ul RNase-freies dH 2 0. Fügen Sie 1 ul RNasin, mischen durch Invertieren des Röhrchens und inkubieren bei 56 ° C für 15 min.
- Zentrifuge für 3 min bei voller Geschwindigkeit in einer Tischzentrifuge, und sammeln Sie 40 ul des Überstands mit der RNA.
- Quantifizierung der extrahierten RNA-Proben mit Nanodrop 2000 Spektrophotometer (Thermo Scientific). Dann bei -70 ° C gelagert bis zur Verwendung, bis zu 6 Monaten. (Hinweis: Eine bessere Möglichkeit, Gesamt-RNA intakt bewahren wandelt diese in cDNA).
6. Alternative Extraktion mit dem QIAamp Viral RNA Handelsregister RNA Kit
Vollständige Anweisungen finden Sie in der Broschüre PROV gefundenBedieneroberfläche mit dem QIAGEN-Kit. Kurz das Verfahren ist wie folgt:
- Jeweils 560 ul Puffer AVL mit den Träger-RNA in ein 1,5 ml Röhrchen, und fügen Sie 140 ul der 10% der Suspension. Mix von Puls-Vortexen für 15 Sekunden. Inkubation bei RT für 10 min.
- Fügen Sie 560 ul Ethanol (96-100%) zur Probe und mischen Sie durch Puls-Vortexen für 15 Sekunden, dann kurz zentrifugieren.
- Tragen Sie 630 ul dieser Lösung zu einem Spin-Säule in ein 2 ml Collection Tube platziert. Säule für 1 min bei 6.000 g; platzieren Sie dann den Spin-Säule in ein sauberes 2 ml Tube.
- Die Säule wird mit gebundenen RNA mit 500 ul Puffer AW1 und Zentrifuge bei 6.000 g für 1 min. Platz Säule in ein sauberes 2-ml-Collection-Tube.
- Die Säule wird mit 500 ul Puffer AW2 und Zentrifuge bei voller Geschwindigkeit (20.000 g oder 14.000 rpm) für 3 min.
- Platz Spin-Säule in ein sauberes 1,5 ml Mikro-Zentrifugenröhrchen, dann werden 40 ul DEPC-behandeltem Wasser eluieren und die RNA mit voller Geschwindigkeit. Wiederholen Sie aufce für eine abschließende 80 ul eluierte RNA.
- Quantifizierung der extrahierten RNA-Proben mit Nanodrop 2000 Spektrophotometer (Thermo Scientific). Dann bei -70 ° C gelagert bis zur Verwendung, bis zu 6 Monaten. (Hinweis: Eine bessere Möglichkeit, Gesamt-RNA intakt bewahren wandelt diese in cDNA).
7. Nachweis von Noroviren in extrahierten RNA durch Ein-Schritt-Real-Time RT-PCR mit spezifischen TaqMan-Sonden
Instrument StepOnePlus Real Time PCR Systems (Applied Biosystems).
Methodik
- Wischen und Reinigen Sie alle Arbeitsflächen, Pipetten und Zentrifugen mit RNase AWAY, um mögliche RNase-Kontaminationen zu entfernen.
- Pipettieren Die Nov GI und GII Screening Mastermixen nach Tabelle 1. Primern und TaqMan-Sonden sind in Tabelle 2 aufgeführt.
- 10 ul Aliquot von geeigneten Master-Mix zu jedem Reaktionsgefäß.
- 5 l der unbekannten Probe unverdünntRNA, DEPC-behandeltem Wasser als Negativkontrolle oder GI bzw. GII positive Kontroll-RNA, für die entsprechenden Reaktionsvertiefungen. Alle Proben sollten in doppelt durchgeführt werden. Positive Kontrollen und Standards mit Konzentration von 300 ug / ul und 500 ug / ul wurden jeweils von National Laboratory Calicivirus Center for Disease Control and Prevention (CDC) zur Verfügung gestellt.
- Zentrifugieren Sie die Reaktionsplatte bei 6.000 g für 10 sek.
- In dem Experiment Eigenschaften Bildschirm; definieren und wählen Sie eine Art von Experiment für den Lauf. Stellen Sie sicher, TaqMan-Reagenzien als Reagenzien-Typ, und dieser Pre-PCR-Amplifikation und Lese sind ausgewählt werden angezeigt.
- Ausführen 15 ul Reaktionen unter Verwendung des thermischen Profils in Tabelle 3.
- Analysieren Sie die Ergebnisse.
8. Genotypisierung von NOV GI und GII mit herkömmlichen RT-PCR und Sequenzierung
(Es ist noch nicht in diesem Video erwähnt, da sie eine häufige und weit verbreitete Methode ist.)
Instrument. Applied Biosystems StepOne StepOnePlus und Real Time PCR-Systeme mit dem Quantitec Vorlage.
Methodik
- Vor der Genotypisierung, sind die Genogruppe (GI oder GII) der Proben durch das Screening-RT-PCR oben beschrieben bestimmt. GI NoV RNA muss mit dem GI Genotypisierung Master-Mix und der GII NoV RNA mit dem GII Genotypisierung Master-Mix verstärkt werden.
- Pipettieren Die Nov GI und GII Genotypisierung Mastermixen nach Tabelle 4. Grundierungen GI SKF / SKR und G2 SKF / SKR sind in Tabelle 5 aufgeführt.
- 45 ul Aliquot des entsprechenden Master-Mix auf 0,2 ml Reaktionsgefäße.
- 5 l der DEPC-behandeltem Wasser auf jede negative Kontrolle Rohr, und 5 ul sorgfältig geprüften, Nov (GI und / oder GII) RNA positiv auf die entsprechende Reaktionsrohr.
- Ausführen 50 ul Reaktionen unter Verwendung des thermischen Profils in Tabelle 6.
- Senden Sie PCR-Produkte ContaiNing mindestens 100 ng / ul des amplifizierten Kapsid Region um Unternehmen für die Reinigung und Sequenzierung Macrogen. (9700 Große Seneca Hwy. Rockville, MD 20850 und $ 10 pro Probe pro Sequenzierung).
9. Repräsentative Ergebnisse
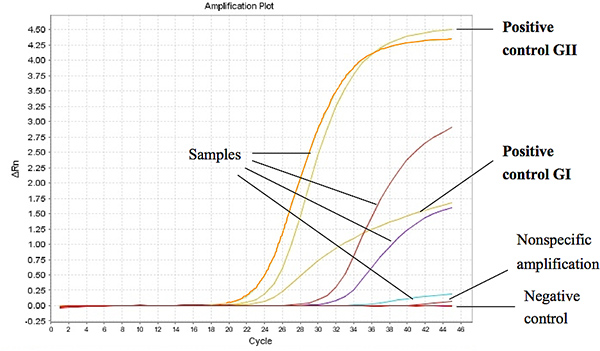
Abbildung 1 zeigt repräsentative Ergebnisse aus der TaqMan-One-Step RT-PCR-Assay-RNA, wenn sie aus Stuhlproben von Kindern extrahiert Durchfallerkrankungen eingesetzt. Der Schwellenwert (Ct) für eine positive Probe wurde bei weniger oder gleich 37 Ct für GI und GII für 39 Ct eingestellt. Die CT-Werte für die positiven Kontrollen wurde festgestellt, dass weniger als 27 und 18 des ORF1 ORF2-Knotenpunkt für GI und GII, beziehungsweise. Klicken Sie hier für eine größere Abbildung anzuzeigen .
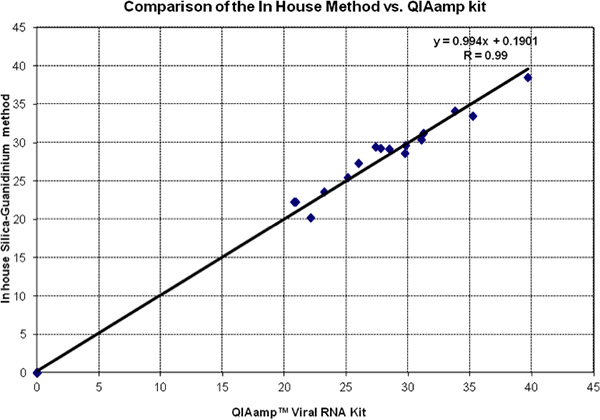
2 zeigt ein Kopf-an-Kopf-Vergleich von CT-Werte von Silica in Guanidinium-Methode und QIAmp Viral RNA Kit. Daten der beiden Tests fallen sehr nahe an der Linie Gleichheit (Steigung = 1 und Achsenabschnitt = 0). Dies wird durch die Regressionsanalyse und der Korrelationskoeffizient bestätigt. Alle negativen Kontrollen wurden ausgewertet und festgestellt, 0 sein mit beiden Methoden.
| Master Mix | Endkonzentration | Volume (ul) |
| H 2 0 PCR | 2,875 | |
| QuantiTect RT-PCR Master Mix | 1x | 6,250 |
| RT-Mix | 1x | 0,125 |
| 50 uM Primer Cog R | 1 uM | 0,250 |
| 50 uM Primer Cog F | 1uM | 0,250 |
| 10 uM Ringsonde | 1 uM | 0.125/0.250 * |
| 10,00 |
* 0,250 ul jeder Sonde wurde für GI-Tests verwendet, während 0,125 ul jeder Sonde für GII Tests verwendet wurde.
Tabelle 1. Qiagen QuantiTect Master-Mix für das Screening GI und GII (Trujillo et al., 2006). 7
| Name | Geno-Gruppe | Verwenden | Sequenz (5 'nach 3') |
| Cog 1R | GI | Grundierung | CTT CGC AGA Katze Katze Katze TYA C |
| Cog 1F | GI | Grundierung | CGY TGG ATG CGN TTY CAT GA |
| Cog 2R | GII | Grundierung | TCG ACG CCA TCT TCA TTC ACA |
| Cog 2F | GII | Grundierung | Auto gar BCN ATG TTY AGR TGG ATG AG |
| Ring 1A | GI | Sonde | FAM-AGA TYG CGA TCY CCT GTC CA-BHQ-1 |
| Ring 1B | GI | Sonde | FAM-AGA TCG CGG TCT CCT GTC CA-BHQ-1 |
| Ring2-TP | GII | Sonde | FAM-TGG GAG GGC GAT CGC AAT-CT-BHQ-1 |
Tabelle 2. Primer und Sonden für quantitative Echtzeit-RT-PCR für Genogruppen I, II (Kageyama et al. Verwendet,2003). 8
| Schritt | Temp (° C) | Zeit (min) | |
| 1 | 50 | 30:00 | cDNA-Synthese |
| 2 | 95 | 15.00 | HotStart Taq-Polymerase Aktivierung |
| 3 | 95 | 00.15 | |
| 60 | 01.00 | 45X-Zyklen |
Tabelle3. Thermal-Profil für Ein-Schritt-TaqMan real time RT-PCR (Trujillo et al., 2006). 7
| Master Mix | Endkonzentration | Volume (ul) |
| H 2 0 PCR | 29,50 | |
| D5 Qiagen RT-PCR-Puffer | 1x | 10,00 |
| 10 mM dNTP-Mix | 0,4 mM | 2,00 |
| Enzym-Mix | 2,00 | |
| 40 U / ul RNAsin | 20 U / ul | 0,50 |
| 10 uM SKF | 0,1 uM | 0,50 |
| SKR 10 uM | 0,1 uM | 0,50 |
| 45,00 |
Tabelle 4. Qiagen One-Step RT-PCR Master-Mix für die Genotypisierung GI und GII.
| Name | Geno-Gruppe | Verwenden | Sequenz (5 'nach 3') |
| G1SKF | GI | Grundierung | 5'-CTG CCC GAA TTY GTA AAT GA - 3 |
| G1SKR | GI | Grundierung | 5'-CCA CCA ACC CAR TTR TAC A - '3 |
| G2SKF | GII | Grundierung | 5'-CNT GGG AGG GCA GCG ATC A - 3 |
| G2SKR | GII | Grundierung | 5'-GCA CCN CCR CCR TRH TTR TA CAT-3 |
Tabelle 5. Primer für die Amplifikation der Region C der Capsid Region (Kojima et al., 2002). 5
| Schritt | Temp (° C) | Zeit (min) | |
| 1 | 60 | 30:00 | cDNA-Synthese |
| 2 | 96 | 15.00 | HotStart Taq-Polymerase Aktivierung |
| 3 | 94 | 00.030 | |
| 52 | 01.00 | 40X-Zyklen | |
| 72 | 00.30 | ||
| 4 | 72 | 10.00 | Glühen |
Tabelle 6. Thermische Profil der TaqMan-One-Step RT-PCR.
Access restricted. Please log in or start a trial to view this content.
Diskussion
Mit dem wirtschaftlichen Inhouse-Verfahren zur Isolierung von Nukleinsäure aus Stuhlproben, erhalten wir gleiche Ergebnisse wie mit dem kommerziellen QIAmp Viral RNA Kit von Qiagen, und zusammen mit der TaqMan RT-PCR in unserem Labor entwickelt können wir erkennen ein breites Spektrum von NOV Genotypen aus der GI und GII Genogruppe. Eine aktuelle Publikation der Region C-Protokoll berichtet Genotypisierung Raten von 78% 6. Da die Vielfalt der zeitgenössischen NoV Stämme hat sich in den vergangenen Jahren ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Autoren danken der National Laboratory Calicivirus Center for Disease Control and Prevention (CDC) für die freundliche Spende von einer Standard-und Kontrolle Positives für Nov danken und Laboratorien der School of Public Health an der Johns Hopkins für die Bereitstellung der Reagenzien.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| Guanidinisothiocyanat | Sigma-Adrich | G9277 | |
| Tris-HCl | Sigma-Aldrich | T5941 | |
| EDTA | Sigma-Aldrich | E5134 | |
| Kieselerde | Sigma-Aldrich | S5631 | |
| Triton X-100 | BDH Chemicals | 14530 | |
| Diethylpyrocarbonat | Sigma-Aldrich | D-5758 | |
| QIAamp Viral RNA Mini Kit (250) | QIAGEN | 52906 | |
| QuantiTec Probe RT-PCR-Kit (200) | QIAGEN | 204443 | |
| Qiagen One Step RT-PCR Kit (200) | QIAGEN | 210212 | |
| RNase-Inhibitor 2000 Einheiten | A.Biosystems | N808-0119 | 2000 unids / Fläschchen |
| Non-Stick-freie Rnae Mikrozentrifugenröhrchen | Ambion | AM12450 | |
| UltraPure Agarose 1000 | Invitrogen | 16550-100 |
Referenzen
- Medici, M. Molecular epidemiology of Norovirus infections in sporadic cases of viral gastroenteritis among children in Northern Italy. L. Medical Virology. 78, (2006).
- Vidal, R. Novel recombinant Norovirus causing outbreaks of gastroenteritis in Santiago, Chile. J. Clinica Microbiology. 4, (2006).
- Xavier, M. Detection of caliciviruses associated with acute infantile gastroenteritis in Salvador, an urban center in Northeast Brazil. Braz. J. Med. Biol. Res. 42, (2009).
- Boom, R. Rapid and simple method for purification of nucleic acids. J. Clin. Microbiol. 28, 495-503 (1990).
- Kojima, S. Genogroup-specific PCR primers for detection of Norwalk-like viruses. J. Virol. Methods. 100, 107-114 (2002).
- Mattison, K. Multicenter comparison of two norovirus ORF2-based genotyping protocols. J. Clin. Microbiol. 47, 3927-3932 (2009).
- Trujillo, A. A. Use of TaqMan real-time reverse transcription-PCR for rapid detection, quantification, and typing of norovirus. J. Clin. Microbiol. 44, 1405-1412 (2006).
- Kageyama, T. A broadly reactive and highly sensitive assay for Norwalk-like viruses on real-time quantitative RT-PCR. J. Clin. Microbiol. 41, 1548-1557 (2003).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten