Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Überwachung Equilibrium Veränderungen im RNA-Struktur von "peroxidativer" und "Oxidativer 'Hydroxylradikal Footprinting
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt, wie die Mg (II)-abhängige Bildung von RNA Tertiärstruktur durch zwei Methoden der Hydroxyl-Radikal footprinting quantifizieren.
Zusammenfassung
RNA-Moleküle spielen eine wesentliche Rolle in der Biologie. Neben der Übertragung genetischer Information, kann RNA in einzigartige Tertiärstrukturen Erfüllung einer bestimmten biologischen Rolle als Regulierer, Bindemittel oder Katalysator zu falten. Informationen über tertiäre Bildung Kontakt ist unerlässlich, um die Funktion von RNA-Molekülen zu verstehen. Hydroxyl-Radikale (• OH) sind einzigartig Sonden der Struktur von Nukleinsäuren aufgrund ihrer hohen Reaktivität und geringen Größe. 1 Wenn ein footprinting Sonde verwendet, map Hydroxylradikale das Lösungsmittel zugängliche Oberfläche des Phosphodiester-Rückgrat der DNA 1 und RNA 2 mit so fein wie single nucleotide Auflösung. Hydroxyl-Radikal footprinting können die Nukleotide innerhalb einer intermolekularen Kontaktfläche, in DNA-Protein-1 und RNA-Protein-Komplexe z. B. zu identifizieren. Equilibrium 3 und 4 kinetische Übergänge können durch die Durchführung Hydroxylradikal Footprinting als Funktion einer Lö bestimmt werdenauf variable oder Zeit, bzw.. Ein wesentliches Merkmal des Fußabdrucks ist, dass eine begrenzte Exposition mit der Sonde (zB "Single-Hit-Kinetik") führt in die einheitliche Probenahme jedes Nukleotid des Polymers. 5
In diesem Video-Artikel verwenden wir die P4-P6-Domäne des Tetrahymena Ribozym RNA Probenvorbereitung und die Bestimmung eines Mg (II)-vermittelte Faltung Isothermen zeigen. Wir beschreiben die Verwendung der bekannten Hydroxyl-Radikal footprinting Protokoll, das H 2 O 2 (wir nennen dies den "peroxidative"-Protokoll) und eine wertvolle, aber nicht allgemein bekannt, dass alternative natürlich gelösten O 2 verwendet (wir nennen so bedarf dies der ' oxidative 'Protokoll). Eine Übersicht über die Datenreduktion, Transformation und Analyse-Verfahren vorgestellt.
Protokoll
1. Vorbereitung der Reagenzien Footprinting
- Bereiten Sie einen 10x Reaktionspuffer mit 100 mM Natriumcacodylat, 1 mM EDTA und 1 M KCl. Den pH-Wert auf 7,4. Filtern Sie die Puffer unter Verwendung eines 0,2 uM Acetat-Filter-Gerät (Nalgene). Bemerkung: nicht pipet RNA direkt in 10x Puffer.
- Bereiten Sie die Titrationsreaktion Mix für jede Reaktion, wie in Tabelle 1 angegeben. Das Volumen der Titration Mix (1x Puffer und Mg (II) in der gewünschten Konzentration) sollte 90 ul werden, bevor man 10 &mgr; l des RNA in 1x Puffer.
- Bereiten Sie eine RNAse T1 Verdauung Puffer 6.63M Urea, 20 mM Natriumcitrat, 1mM EDTA, 0,25 ug / ul tRNA, 0,025% Xylencyanol und 0,025% Bromphenolblau. Dieser Puffer kann bei 4 ° C gelagert werden bis zu 6 Monaten.
- Die Fenton-Reaktion erfordert frisch zubereiteten wässrigen Lösungen von 250 mM Fe (NH 4) 2 (SO 4) 2, 250 mM EDTA und 500 mM Natrium-L-Ascorbat. Die Konzentrationen festgestellt in Kapitel 10,5 sind optimal für equilibrium-Experimente in Puffer wie Natriumcacodylat, die nicht abzufangen ist Radikalen und diverse zweiwertigen Ionen-Konzentrationen.
- Reagenzien für die peroxidative (1.5.1) und oxidative (1.5.2) footprinting Reaktion.
- Die peroxidative Fenton Reaktion erfordert frisch zubereiteten wässrigen Lösungen von 100 mM Fe-EDTA-Lösung und 50 mM Natrium-L-Ascorbat und 1,5% H 2 O 2. Die Fe-EDTA-Lösung wird durch Mischen von Fe (NH 4) 2 (SO 4) 2 mit EDTA bereit, das 1,1-fache über EDTA über Eisen haben.
- Kurz vor Eröffnung des oxidativen Spaltung (3.4.2), erstellen Sie die Fenton-Reaktion-Mix durch die Kombination von 2 ul Fe (NH 4) 2 (SO 4) 2 (250 mM), 2,2 ul EDTA (250 mM), 62,5 ul Natrium-L-Ascorbat (500 mM), 22,5 ul 10x Reaktionspuffer und 135,5 ul H 2 O auf die Endkonzentrationen im footprinting Reaktionsgemisch (3.4.2) von 0 zu erreichen.10 mM, 0,11 mM, und 6,61 mM.
2. RNA-Präparation für Footprinting Experiment
- RNA kann durch Standard-in vitro-Transkription der DNA-Vorlagen 6 hergestellt werden oder, wenn kürzer als 50 Nukleotide, erworben (z. B. bei Integrated DNA Technologies, www.idtdna.com) werden. Die empfohlenen Labor Sauberkeit Verfahren zur Probe Integrität der RNA während Footprinting Experimente zu erhalten. 7
- Dephosphorylierung 8 von in vitro transkribierte RNA ist notwendig, vor 32 P Kennzeichnung der 5 'Ende der RNA. 10 pmol RNA werden durch kinasing endmarkiert. 9 Die Lösung sollte nach erfolgter Inaktivierung der Phosphatase bewölkt.
- Purify radioaktiv markierte RNA mit denaturierende Gelelektrophorese. Die RNA wird durch zwei anschließende Gel Extraktion mit 0,3 M Natriumacetat (pH 5,5) erholt. RNA wird durch Mischen von 0,5 ml wässrige Lösung mit 1 ml Ethanol und anschließender ausgefälltInkubation für 1 Minute auf Trockeneis. Spin Probe bei 4 ° C, 12500 rpm für 0,5 Stunden. Überstand verwerfen und waschen RNA-Pellet mit 70% Ethanol. Entfernen Sie nach dem zusätzlichen Zentrifugationsüberstand und trocken Pellet im Vakuum.
- Lösen Sie die 32 P-markierten RNA-Proben in 330 ul 1x Buffer.Pipet 30 ul der RNA-Lösung in einem Reaktionsrohr, Niederschlag mit Ethanol und trockene RNA im Vakuum; halten dieses Rohr für die Erzeugung der Referenz Leiter (Schritt 3.5). Denaturieren gepuffert RNA-Lösung durch Erhitzen bei 95 ° C für 2 min. Kühlen Sie die Proben auf Raumtemperatur für 15 min und geben einen schnellen Spin zur Senkung der Kondensate auf den Deckeln der Rohre wieder in Lösung.
3. Die Footprinting Experiment
- Die Elektrophorese Kamm bestimmt die maximale Anzahl der Datenpunkte pro analytischen Gel. Wir verwenden eine 30 gut kämmen. Die Referenz Leiter belegt 2 Brunnen und die ungespaltenen Kontrolle ein. Bereiten 27 Reaktionsgefäße. Die endgültige Mg (II) concentrations sollten gleichmäßig auf einer logarithmischen Skala Spanning Größenordnungen um die Titration Mittelpunkt angeordnet werden.
- Separat inkubieren RNA und Mg (II)-Lösungen bei 50 ° C für 5 min. Mix 10 ul RNA-Lösung mit 90 ul der entsprechenden Menge an Mg (II) in 1x Puffer auf ein Endvolumen von 100 ul zu erreichen. Inkubieren Sie die Lösungen für 30 min bei 50 ° C.
- Lassen Sie die Lösungen bei 25 ° C für 1 Stunde ausgleichen, während Faltung auftritt.
- Peroxidativer (3.4.1) oder oxidative (3.4.2) Hydroxylradikal footprinting Reaktion.
- Starten Sie den peroxidative Reaktion, indem Tröpfchen von 2 ul Fe-EDTA (100 mM), 2 ul Natrium-L-Ascorbat (50 mM) und 2 ul H 2 O 2 (1,5%) von einander an der Spitze in der Reaktion abgetrennt Röhrchen mit der RNA-Lösung und starten Sie die footprinting Reaktion durch kräftiges Mischen. Stoppen der Reaktion nach 15 Sekunden durch Zugabe von 300 ul kaltem absolutem Ethanol. Drehen Sie den Rohren 3-5 mal. Niederschlag, Wasch-undtrockene Pellet, wie in Schritt 2.4.
- Starten Sie die oxidative Hydroxylradikal footprintingreaction durch Zugabe von 5 ul der frisch zubereiteten Fenton-Reaktion-Mix (1,6). Inkubieren für 30 min bei 25 ° C. Add 300 pl kaltem absolutem Ethanol, um die Reaktion zugeben und durch Drehen der Rohre 3-5 mal stillen. Niederschlag, waschen und trocknen das Pellet wie in Schritt 2.4.
- Generieren Sie eine RNase T1 verdauen Referenzprobe nach üblichen Verfahren 10 mit der Lösung in Schritt 1.3 vorbereitet.
- Lösen RNA-Pellets in 8 ul Gel Loading Dye II (Ambion) und bestätigen RNA wird durch die Verwendung der Geiger-Zähler resuspendiert.
- Bereiten Sie eine Denaturierung, 8% Polyacrylamid Sequenzierungsgel nach Standardprotokollen. 11 Legen Proben, darunter zwei Referenzen und eine Kontrollgruppe. Separate RNA-Fragmente bei 60 W - 75 W für 2,5 Stunden. Expose getrockneten Gel zu einer Speicherung Leuchtschirm über Nacht. Scan Leuchtschirm mit einem Imaging-System für filmlosen Autoradiographie, z. B.. Sturm 865 (GE Healthcare) oder Taifun (GE Healthcare). Transfer-Gel-Image-Datei auf Ihren Computer.
4. Data Analysis
- Das Hydroxyl-Radikal Reaktivität jedes Nukleotid ist durch Quantifizierung der Intensität jeder Band auf der analytischen Gel bestimmt. Downloaden, installieren und öffnen SAFA, ein einfach zu Open-Source-Software für Single-Band-Montage und Quantifizierung verwenden. 12, 13 Befolgen Sie die Bedienungsanleitung beachten. Kurz gesagt, laden Sie die RNA-Sequenz als. Txt-Datei, gefolgt von der Gel-Bild als. Gel-Datei. Passen Bandenintensitäten, definieren die Gassen, wählen Sie einen Anker Spur, und führen Sie ein Gel Ausrichtung. Zuordnung der Banden zu Nukleotiden tritt in Bezug auf RNase T1 Verdauung Leiter. Quantifizierung der Band Integrale und die Normalisierung / colorplot Funktion zu normalisieren und weisen potenziell invariant Rückstände. Speichern Sie die Ausgabe als. Txt-Datei.
- SAFA gibt eine Tabelle mit Spalten repräsentieren Spuren auf dem Gel und Zeilen, die die inintegrierte Band Dichte der einzelnen Banden, die die RNA-Fragment. Zunächst identifizieren die Schutzgebiete, die eine merkliche Veränderung in Lösungsmittelzugänglichkeit Display durch einen Vergleich der Schutz-Profil aus der kein Mg (II)-Probe mit dem Profil des Endpunktes Mg (II)-Konzentration (50 mM in unserem Beispiel) abgeleitet. Je niedriger der Wert, desto mehr schützten die Nukleotid ist gegen den Angriff von Hydroxyl-Radikalen und umgekehrt.
- Create Falten Isothermen aus den normalisierten Band Integrale von einzelnen oder Gruppen von Nukleotiden im Vergleich zu Mg (II)-Konzentration. Die Isothermen sind individuell auf fraktionelle Sättigung durch fi = L + (U - L) skaliert •
 wobei f die integrierte Dichte der Band (s) bezeichnet, die analysiert werden, stellen L-und U die untere und obere Grenzen für die Übergangs-und
wobei f die integrierte Dichte der Band (s) bezeichnet, die analysiert werden, stellen L-und U die untere und obere Grenzen für die Übergangs-und 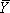 ist fraktionelle Sättigung. 14 Die Daten sind fit mit einem nicht-linearen lOsten Quadrat-Analyse-Programm (Wir verwenden entweder Origin (OriginLab) oder GraphPad (GraphPad Software, Inc.).), um die Hill-Gleichung (1),
ist fraktionelle Sättigung. 14 Die Daten sind fit mit einem nicht-linearen lOsten Quadrat-Analyse-Programm (Wir verwenden entweder Origin (OriginLab) oder GraphPad (GraphPad Software, Inc.).), um die Hill-Gleichung (1), 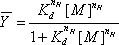
wo K d ist das Gleichgewicht Dissoziationskonstante, [M] entspricht der Konzentration der Variablen, die die Faltung Reaktion vermittelt, Mg (II) in diesem Beispiel, und n H ist der Hill-Koeffizient. Dieses Verfahren skaliert die Umstellung auf fraktionelle Sättigung bestimmt seinen Mittelpunkt und bietet eine phänomenologische Test, ob ein Übergang sigmoidal. Isothermen von Sequenziergelen (Abbildung 3A), die von SAFA (Abbildung 3B) wurden analysiert und fit wie oben beschrieben abgeleitet sind in Abbildung 3C gezeigt. Die fraktionelle Sättigung Isothermen in Abbildung 3 dargestellt wurden durch Skalierung in einer Tabellenkalkulation erstellt die Werte von SAFA gegen die besten passen Ober-und Untergrenzen (U-und L) mit der Gleichung Bruchteil generiert = value / (U - L) - L / (U - L).
5. Repräsentative Ergebnisse:
Abbildung 3 zeigt repräsentative Ergebnisse aus P4-P6-RNA • OH Footprinting Experimente. Das Gel-Bild (links) zeigt an, dass a) Hintergrund Spaltung minimal ist (rechte Spur), b) single nucleotide Zuordnung ist möglich durch gut definierte Banden in den T1 Spur (RNase T1 spaltet bei jedem G), und c) das Hydroxyl-Radikal induzierten Fragmentierung der RNA ist weit über Hintergrund. Der Übergang von niedrigen zu hohen Mg (II) wird mit abnehmender integrierte Band Dichte der einzelnen und Gruppen von Bändern auf die Bildung von RNA Tertiärstruktur angeschlossen. Einzel-oder Gruppen von aufeinander folgenden Nukleotid deren Spaltung Änderungen mitverwendet werden als "Schutz" bezeichnet. Die • OH-Schutz Merkmal der Mg (II)-vermittelte Faltung der P4-P6-RNA in engem Zusammenhang mit dem Lösungsmittel unzugänglichen Regionen des Moleküls in seine Kristallstruktur beobachtet entsprechen. 15
Gefaltete RNA-Moleküle c ein sehr unterschiedlichem Ausmaß (dh, wie Hintergrund schließen die Band Dichten Rückgang) des tertiären Berührungsschutz, die nicht mit klaren Struktur-oder dynamisches Phänomen wurden korreliert. So werden einige RNA-Moleküle zeigen, gut definierte strukturelle Übergänge, wie in Abb. 3 gezeigt, und manche nicht. Band Intensitäten quantifiziert und durch SAFA 12 Analyse (Abbildung 3B) normiert. Die Ausgabe ist eine "thermische" Plot Visualisierung der relativen Schutz gegen • OH. Der Farbübergang beschreibt die Zugänglichkeit zu ändern, auf Mg (II) hinaus. Weiß auf Rot oder Blau zeigt mehr zugänglich oder mehrere geschützte Nukleotide, jeweils. Jedes Grad der Schattierung wird mit einem numerischen Wert, der als Schutz-Kurve (Abbildung 3C) aufgetragen werden können und von einem Binding-Modell wie der Hill-Gleichung (1) angeschlossen. Die Gleichgewichts-Dissoziationskonstante mit Schutz 153-155 angegliedert ist etwa doppelt so hoch wie der entsprechende Wert des Schutzes 163-164.
NHALT ">
Abbildung 1. Hydroxylradikal Produktions-und P4-P6 RNA-Faltung. A) Fe (II) katalysiert die Bildung von Hydroxylradikalen aus Wasserstoffperoxid. Ascorbat reduziert Fe (III) zurück zu Eisen-II. B) In Abwesenheit von Mg (II), ist kein P4-P6 Tertiärstruktur gebildet, so dass Hydroxyl-Radikale zu durchdringen und zu spalten alle zugänglich Backbone-Positionen. Die Zugabe der Mg (II) initiiert Faltung des P4-P6, so dass nur das Lösungsmittel zugänglich Rückgrat durch die Hydroxyl-Radikale gespalten werden.
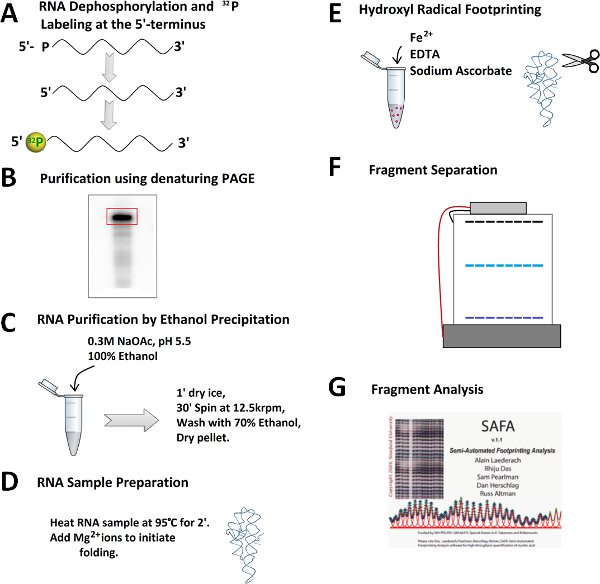
Abbildung 2. Für einen Hydroxyl-Radikal footprinting Experiment Outline. A) Dephophorylation und 32 P-5'-Ende Markierung von RNA. B) Reinigung von 32 RNA auf einem denaturierenden Polyacrylamidgel P-markierte. C) Exzision von RNA-Band, anschließende RNA-Extraktion und Ethanolfällung. D) Vorfaltung undFaltung der RNA. E) Die Zugabe von frisch zubereiteten Fenton Reaktionsgemisch zur Erzeugung der Hydroxyl-Radikale. F) RNA-Fragment Trennung durch denaturierende Polyacrylamid-Gelelektrophorese. G) Quantifizierung von RNA-Fragmenten durch SAFA-Software.
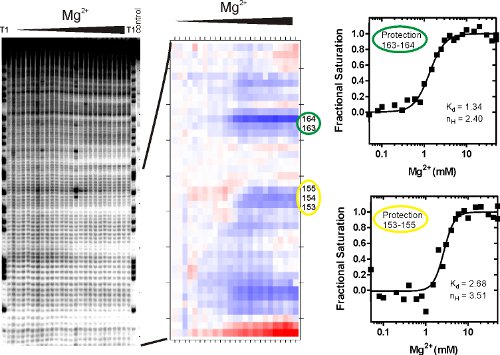
Abbildung 3. Analyse von RNA-Fragmenten nach peroxidative Fenton footprinting Reaktion. (A) Die RNA wurde zu Hydroxyl-Radikalen ausgesetzt und die Spaltprodukte wurden gelöst mit denaturierenden Polyacrylamid-Gelelektrophorese (PAGE). Das Bild zeigt peroxidative Spaltprodukte mit zunehmender Konzentration von Mg (II). Reference und Kontrolle Gassen beschriftet sind. Diese Image-Datei ist der Eingang für das SAFA-Programm, das die Lautstärke der einzelnen Bänder quantifiziert. (B) Die "thermische" Verschwörung von SAFA generiert. (C) eine Tabelle mit Werten über integrierte Band Dichte an Nukleotid-Nummer Ausgabe von SAFA wird durch ein verbindliches Modell wie der Hill equati analysiertauf (1). Regionen des Schutzes wurden nach der Erhöhung der Band integralen Dichte als Funktion der Mg (II)-Konzentration gewählt. Die K d ist die Mg (II)-Konzentration bei der die Hälfte der RNA auf der Baustelle überwacht gefaltet ist. Der Hill-Koeffizient, n H, ist ein Maß für die Steigung der Kurve, die Auskunft über die Bindung Kooperativität und bietet eine niedrigere Schätzung für die Zahl der Magnesium-Ionen in Falten von diesem besonderen Ort beteiligt.
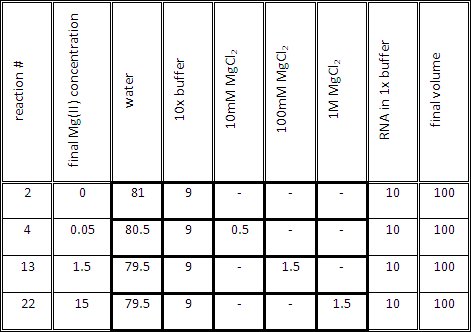
Tabelle 1. Repräsentativen Mengen zur Erzeugung von RNA Proben mit verschiedenen Mg (II)-Konzentrationen.
Access restricted. Please log in or start a trial to view this content.
Diskussion
Hydroxyl-Radikal Footprinting ist ein wertvolles Instrument, um das Lösungsmittel zugängliche Oberfläche von Nukleinsäuren zu beurteilen. Qualitative und quantitative Bildung der Tertiärstruktur 14 kann als eine Funktion von Parametern wie Ionen-Art und Konzentration, pH, Temperatur, bindende Proteine oder Falten Co-Faktoren zu beachten. Die überzeugende Kombination aus einem geradlinig und kostengünstige Protokoll und die daraus resultierende Lösungsmittel Zugänglichkeit und Falten Information...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde durch Zuschüsse aus dem National Institute of Health RO1-GM085130 und National Science Foundation MCB0929394 unterstützt. Wir danken Dr. Marion Schmidt für ihre Gastfreundschaft und für die Erlaubnis zu filmen in ihrem Labor.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Name | Company | Cat# | |
| Sodium Cacodylate(Caution! Toxic) | Sigma-Aldrich | C4945-25g | |
| EDTA (0.5 M) | Ambion | AM9260G | |
| DEPC treated water | Ambion | AM9915G | |
| Sodium Acetate (3 M) | Ambion | AM9740 | |
| MgCl2 (1 M) | Ambion | AM9530G | |
| Urea | Ambion | AM9902 | |
| Sodium Citrate | Sigma-Aldrich | W302600 | |
| tRNA | Sigma-Aldrich | R-7876 | |
| Sodium-L-ascorbate | Sigma-Aldrich | A7631-25g | |
| Fe(NH4)2(SO4)2 . 6 H2O | Sigma-Aldrich | F1543-500g | |
| RNase T1 | Fermentas | EN0541 | |
| Hydrogen Peroxide (30%) | Sigma-Aldrich | 349887 |
Referenzen
- Tullius, T. D., Dombroski, B. A. Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein. Proc. Natl. Acad. Sci. U.S.A. 83, 5469-5473 (1986).
- Celander, D. W., Cech, T. R. Iron(II)-ethylenediaminetetraacetic acid catalyzed cleavage of RNA and DNA oligonucleotides: similar reactivity toward single- and double-stranded forms. Biochemistry. 29, 1355-1361 (1990).
- Celander, D. W., Cech, T. R. Visualizing the higher order folding of a catalytic RNA molecule. Science. 251, 401-407 (1991).
- Sclavi, B., Sullivan, M., Chance, M. R., Brenowitz, M., Woodson, S. A. RNA folding at millisecond intervals by synchrotron hydroxyl radical footprinting. Science. 279, 1940-1943 (1998).
- Tullius, T. D., Dombroski, B. A., Churchill, M., Kam, L. Hydroxyl radical footprinting: a high-resolution method for mapping protein-DNA contacts. Methods. Enzym. 155, 537-558 (1987).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic. Acids. Res. 15, 8783-8798 (1987).
- Schlatterer, J. C., Brenowitz, M. Complementing global measures of RNA folding with local reports of backbone solvent accessibility by time resolved hydroxyl radical footprinting. Methods. 49, 142-147 (2009).
- Sambrook, J., Russell, D. W. Molecular Cloning: A Laboratory Manual. , 3 ed, Cold Spring Harbor Laboratory Press: Cold Spring Harbor. New York. (2001).
- Zaug, A. J., Grosshans, C. A., Cech, T. R. Sequence-specific endoribonuclease activity of the Tetrahymena ribozyme: enhanced cleavage of certain oligonucleotide substrates that form mismatched ribozyme-substrate complexes. Biochemistry. 27, 8924-8931 (1988).
- Knapp, G. Enzymatic approaches to probing of RNA secondary and tertiary structure. Methods. Enzym. 180, 192-212 (1989).
- Slatko, B. E., Albright, L. M. Denaturing gel electrophoresis for sequencing. Curr. Protoc. Mol. Biol. Chapter 7, Unit 7.6-Unit 7.6 (2001).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. SAFA: semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. RNA. 11, 344-354 (2005).
- Simmons, K., Martin, J. S., Shcherbakova, I., Laederach, A. Rapid quantification and analysis of kinetic *OH radical footprinting data using SAFA. Methods. Enzym. 468, 47-66 (2009).
- Uchida, T., He, Q., Ralston, C. Y., Brenowitz, M., Chance, M. R. Linkage of monovalent and divalent ion binding in the folding of the P4-P6 domain of the Tetrahymena ribozyme. Biochemistry. 41, 5799-5806 (2002).
- Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., Kundrot, C. E., Cech, T. R., Doudna, J. A. Crystal structure of a group I ribozyme domain: principles of RNA packing. Science. 273, 1678-1685 (1996).
- Petri, V., Brenowitz, M. Quantitative nucleic acids footprinting: thermodynamic and kinetic approaches. Curr. Opin. Biotechnol. 8, 36-44 (1997).
- Takamoto, K., Das, R., He, Q., Doniach, S., Brenowitz, M., Herschlag, D., Chance, M. R. Principles of RNA compaction: insights from the equilibrium folding pathway of the P4-P6 RNA domain in monovalent cations. J. Mol. Biol. 343, 1195-1206 (2004).
- Brenowitz, M., Senear, D. F., Shea, M. A., Ackers, G. K. Quantitative DNase footprint titration: a method for studying protein-DNA interactions. Methods. Enzym. 130, 132-181 (1986).
- Shcherbakova, I., Mitra, S. Hydroxyl-radical footprinting to probe equilibrium changes in RNA tertiary structure. Methods. Enzym. 468, 31-46 (2009).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr. Opinion. Chem. Biol. 10, 430-436 (2006).
- McGinnis, J. L., Duncan, C. D., Weeks, K. M. High-throughput SHAPE and hydroxyl radical analysis of RNA structure and ribonucleoprotein assembly. Methods. Enzym. 468, 67-89 (2009).
- Jonikas, M. A., Radmer, R. J., Laederach, A., Das, R., Pearlman, S., Herschlag, D., Altman, R. B. Coarse-grained modeling of large RNA molecules with knowledge-based potentials and structural filters. RNA. 15, 189-199 (2009).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten