Method Article
Chromatin Isolierung von RNA-Reinigung (Chirp)
In diesem Artikel
Zusammenfassung
Chirp ist eine neuartige und schnelle Technik, um genomische Bindungsstellen der langen nicht-kodierenden RNAs (lncRNAs) abzubilden. Die Methode nutzt die Spezifität der anti-sense-Oligonukleotide Fliesen um die Auszählung von lncRNA-gebundene genomische Stellen zu ermöglichen.
Zusammenfassung
Lange-kodierende RNAs sind wichtige Regulatoren der Chromatin Staaten für wichtige biologische Prozesse wie Dosiskompensation, Imprinting und Entwicklungsstörungen Genexpression 1,2,3,4,5,6,7. Die jüngste Entdeckung von Tausenden von lncRNAs in Verbindung mit spezifischen Modifikation Chromatin-Komplexe wie Polycomb Repressive Complex 2 (PRC2) vermittelt, dass Histon H3 Lysin 27 Trimethylierung (H3K27me3), breite Rollen für zahlreiche lncRNAs bei der Verwaltung von Chromatin-Staaten in einer Gen-spezifischen schlägt Mode 8,9. Während einige lncRNAs gedacht werden, um in cis auf benachbarte Gene arbeiten, arbeiten andere lncRNAs in trans zu weit entfernten Gene regulieren. Zum Beispiel sind Drosophila lncRNAs roX1 und roX2 bind zahlreichen Regionen auf dem X-Chromosom der männlichen Zellen, und entscheidend für Dosiskompensation 10,11. Allerdings sind die genauen Standorte von ihren Bindungsstellen nicht mit hoher Auflösung bekannt. In ähnlicher Weise können menschliche lncRNA HOTAIR PRC2 Belegung auf h beeinflussenundreds von Genen genomweiten 3,12,13, aber wie Spezifität erreicht wird, ist unklar. LncRNAs kann auch als Modulgerüsten dienen dazu, die Montage von mehreren Protein-Komplexe zu rekrutieren. Die klassische trans-agierende RNA-Gerüst ist die TERC RNA, die als Vorlage und Gerüst für die Telomerase-Komplex 14 dient; HOTAIR kann auch als Gerüst für PRC2 und einem H3K4 Demethylase Komplex 13 zu dienen.
Frühere Studien Mapping RNA-Belegung am Chromatin haben gezeigt, wesentliche Einblicke 15,16, aber nur an einem einzigen Gen-Locus zu einer Zeit. Die Belegung der meisten Websites lncRNAs sind nicht bekannt, und die Rollen von lncRNAs in Chromatin Regulation wurden zumeist aus den indirekten Auswirkungen der lncRNA Störung abgeleitet. So wie Chromatin-Immunopräzipitation von Microarray-oder tief-Sequenzierung (ChIP-Chip oder Chip-seq bezeichnet) verfolgt hat, unser Verständnis von Protein-DNA-Wechselwirkungen im genomischen Maßstab verbessert, hier haben wir veranschaulichen eine recenTLY veröffentlichten Strategie zu lange RNA-Belegung genomweiten bei hoher Auflösung 17 Karte. Chromatin-Komplex, der durch Antisense-Oligonukleotide Tiling, der dann eine Karte der genomischen Bindestellen bei einer Auflösung von mehreren hundert Basen mit: Diese Methode, Chromatin Isolierung von RNA-Reinigung (Chirp) (Abbildung 1), basiert auf Affinität Erfassung von Soll-lncRNA Basis hoher Empfindlichkeit und geringem Hintergrund. Chirp ist auf viele lncRNAs, weil das Design der Affinitäts-Sonden ist einfach aufgrund der RNA-Sequenz und erfordert keine Kenntnis der RNA-Struktur oder funktionelle Domänen.
Protokoll
1. OEM Anwendungen
Design Anti-sense DNA Fliesen Sonden für die selektive Abrufen von Ziel-RNA von Chirp.
- Design-Antisense-Oligo Probes mit dem Online-Sonde Designer bei singlemoleculefish.com 18.
- Verwenden Sie diese Parameter: Anzahl der Sonden = 1 Sonde / 100 bp Länge von RNA; 2) Ziel GC% = 45, 3) Oligonukleotid-Länge = 20; 4) Abstand Länge = 60-80. Brechen RNA in Segmente, wenn zu lange für den Konstrukteur. Lassen Sie Regionen von Wiederholungen oder weitgehende Homologie.
- Auftrag Antisense-DNA-Sonden mit BiotinTEG am 3'-Ende Ende.
- Markierungs-Sonden entsprechend ihrer Position entlang der RNA. Trennen Sie sie in zwei Pools, so dass die "geraden" Pool enthält alle Sonden Nummerierung 2, 4, 6, usw. und die "ungeraden" Pool enthält Sonden Nummerierung 1, 3, 5, usw. verdünnen Pool von Sonden bis 100 uM Konzentration und Lagerung bei -20 ° C.
- Alle Experimente sind mit handelsüblicherBeide Pools, die als interne Kontrollen für einander zu dienen. Echte RNA-abhängige Signal vorhanden wäre von beiden Pools, während Sonden-spezifischen Geräusche wäre einzigartig für jeden Pool. Dies gilt sowohl für CHIRP qPCR-und Chirp-seq.
2. Ernten Sie die Zellen
Sammeln Zellen, die für die Chirp-Experiment verwendet werden.
- Kultivieren von Zellen in Gewebekulturplatten oder Kolben bis zur Konfluenz. Spülen mit phosphatgepufferter Kochsalzlösung (PBS) einmal und trypsinieren. Quench mit Trypsin> 2x Volumen von Medien, Pipette auf und ab, um Zellen zu verdrängen und resuspendieren in einzelne Zell-Suspension. Übertragen Sie alle Medien und resuspendierten Zellen in 50 ml Falcon-Röhrchen. 20 Millionen Zellen sind in der Regel ausreichend für ein CHIRP Probe.
- Spin-Zellen bei 800RCF für 4 min. Aspirieren Medien und resuspendieren 40 Millionen Zellen in 40 ml PBS, Rohre verbinden, wenn nötig. Spin-Zellen bei 800RCF für 4 min. Dekantieren PBS, sorgfältig auf einem Winkel die verbleibende Flüssigkeit abzusaugen.
3. Cross-Link-Cells und Sammeln Zellpellet
Crosslink gesammelten Zellen mit Glutaraldehyd zu RNA-Chromatin-Wechselwirkungen zu erhalten und bereiten Zellpellet.
- Führen Sie alle Schritte bei Raumtemperatur.
- Bereiten Sie 1% Glutaraldehyd in PBS Raumtemperatur. Bereiten Sie 10 ml pro 10 Millionen Zellen (0,4 ml 25% Glutaraldehyd Lager + 9,6 ml PBS). Glutaraldehyd muss frisch verwendet werden.
- Tippen Sie unten auf Falcon-Röhrchen, um Pellets zu entfernen. Resuspendieren Zellpellet in 1% Glutaraldehyd, beginnend mit einem kleinen Volumen zu Klumpen zu vermeiden, dann von oben bis zur vollen Lautstärke. Umkehren, um zu mischen. Crosslink für 10min bei Raumtemperatur auf einer End-to-End-Schüttler oder Rotator.
- Quench die Vernetzungsreaktion mit 1:10 Volumen von 1,25 M Glycin bei Raumtemperatur für 5 min.
- Drehen am 2000RCF für 5 min. Absaugen des Überstandes und Waschen Pellet mit 20 ml PBS gekühlt einmal drehende 2000RCF für 5 min.
- Aspirieren und resuspendieren Sie das wveraschten, vernetzten Pellet mit 1 ml gekühltem PBS pro 20 Millionen Zellen. Übertragen Sie jede ml in ein Eppendorf-Röhrchen und drehen sich mit 2000RCF für 3 min bei 4 C. Entfernen Sie so viel wie möglich mit PBS Pipettenspitze vorsichtig.
- Flash-frieren der Zellpellets in flüssigem Stickstoff und lagern bei -80 ° C auf unbestimmte Zeit.
4. Cell Lysis
Lyse vernetzten Zellen zu Zelllysat vorzubereiten.
- Eingefrorene Zellpellets bei Raumtemperatur. Tippen schwer zu lösen und das Zellpellet zu mischen. Drehen Sie das Pellet bei 2000RCF für 3 min bei 4 ° C Mit einem scharfen 10 pl Pipettenspitze, um alle verbleibenden PBS zu entfernen.
- Auf einer elektronischen Waage (Genauigkeit 1 mg) Tara die Masse eines leeren Eppendorf-Röhrchen (unsere Rohre wiegen 1,060 Gramm sehr konsequent). Wiegen Sie jedes Pellet und notieren ihr Gewicht. Eine volle 15 cm Schale aus vernetztem HeLa-Zellen in der Regel wiegt 100 mg.
- Supplement Lysis Buffer (10x die Masse der Pellets, z. B. 1 ml für 100 mg) mit fresh Protease-Inhibitor, PMSF und Superase-in (siehe beigefügte Liste Puffer). Gut mischen.
- Fügen 10X Volumen ergänzt Lysispuffer in jedes Röhrchen und das Pellet. Für kleine Pellets <25 mg, in 250 ul ergänzt Lysepuffer resuspendieren. Die Aussetzung sollte glatt sein. Falls nicht, teilen Suspension in 500 ul Aliquots und verwenden Sie einen motorisierten Pellet-Mixer zu brechen Klumpen. Fahren Sie direkt mit Beschallung.
5. Ultraschallbehandlung
Shear DNA durch Beschallung vernetzten Zelllysaten.
- Beschallen Zelllysat in Bioruptor in 15 ml Falcon-Röhrchen. Mit <1,5 ml Lysat in jedem Röhrchen und für eine schnellere Beschallung, beschallen nicht mehr als zwei Rohre zu einer Zeit.
- Mit Ultraschall in einem 4 ° C Wasserbad bei höchster Stufe mit 30 Sekunden ein, 45 Sekunden AUS Pulspausen. Überprüfen Sie Lysat alle 30 min. Weiter Ultraschallbehandlung, bis das Zell-Lysat nicht mehr trübe ist. Dies kann so wenig wie 30 Minuten und nicht weniger als 4 Stunden. Die Anzahlvon Rohren, wird das Probenvolumen, die Badtemperatur und die Dauer der Beschallung Zeit beeinflussen, wie lange der Prozess dauert. Tubes wird wahrscheinlich mit unterschiedlichen Geschwindigkeiten beschallen, so bündeln sie zusammen alle 30 Minuten und weiterzuverbreiten in Original-Röhren, um die Homogenität zu gewährleisten. Hinweis: Glutaraldehyd-vernetzten Zellen nehmen deutlich länger als Formaldehyd-Äquivalente beschallen.
- Wenn Lysat klar macht, übertragen Sie 5 ul Lysat in ein frisches Eppendorf-Röhrchen. In 90 ul DNA-Protease K (PK) Buffer (siehe Liste Puffer) und 5 ul PK. Vortex zu mischen und kurz abzentrifugieren. Inkubieren für 45 min bei 50 ° C
- Extraktion von DNA mit Qiagen PCR Purification Kit. Eluieren DNA in 30 ul Qiagen Elutionspuffer (EB) und überprüfen DNA-Größe auf 1% Agarosegel. Wenn Großteil der DNA-Abstrich ist 100-500 bp, Beschallung ist abgeschlossen. Wenn nicht, weiter beschallen.
- Zentrifuge beschallten Proben 16100RCF für 10 min bei 4 ° C Kombinieren Sie Überstände, Aliquot in 1 ml Proben-und Flash-Freeze in flüssiger Gasdrucn. Lagerung bei -80 ° C.
6. CHIRP
Hybridisieren biotinylierten DNA-Sonden an die RNA zu isolieren und gebundenen Chromatin.
- Auftauen Rohre Chromatin bei Raumtemperatur.
- Bereiten Hybridisierungspuffer (siehe Liste Puffer, für das 2 ml pro ml Chromatin). Vortex mischen.
- Für eine typische CHIRP Probe mit 1 ml Lysat, entfernen Sie 10 ul für die RNA-INPUT und 10 ul für DNA-Input und-ort in Eppendorf-Röhrchen. Halten auf Eis bis zur weiteren Verwendung.
- 1 ml Chromatin zu 15 ml Falcon Röhrchen. 2 ml Hybridisierungspuffer in jedes Röhrchen. Für Gesamtvolumen <1,5 ml, Eppendorf-Röhrchen verwenden.
- Auftauen Sonden bei Raumtemperatur. Nanodrop Sonden an Menge nach, wenn Sie es nicht in eine lange Zeit verwendet haben (100 uM Sonden sollten spec ~ 500-600 ng / ul mit Einzelstrang-DNA-Einstellung). Fügen Sie entsprechende Volumen von Sonden auf bestimmte Rohre (100 pmol Sonde pro 1 ml Chromatin, 1 ul 100 pmol / ul Sonde pro 1 ml Chromatin).Gut mischen. Bei 37 ° C für 4 Stunden unter Schütteln.
- Mit 20 min übrigen für die Hybridisierung, bereiten Sie die C-1 magnetische Kügelchen (gelagert bei 4 ° C). Verwenden Sie 100 ul pro 100 pmol von Sonden. Waschen mit 1 ml Lysispuffer unsupplementierten dreimal mit dem Dynamag-2 Magnetstreifen zu trennen Perlen aus dem Puffer.
- Resuspendieren Sie Perlen in ursprünglichen Volumens Lysispuffer; Ergänzung mit frischen PMSF, PI und Superase-in. Nach 4 Stunden Hybridisierungsreaktion abgeschlossen ist, werden 100 ul Kügelchen in jedes Röhrchen. Gut mischen. Bei 37 ° C für 30 Minuten unter Schütteln.
- Bereiten Waschpuffer (5 ml pro Probe). Vortex mischen. Pre-warm bis 37 ° C. In PMSF vor der Verwendung.
- Waschen Kügelchen mit 1 ml Waschpuffer fünfmal. Auf dem ersten Waschgang, verwenden Dynamag-15 Magnetstreifen zu trennen Perlen, dekantieren und Resuspension in 1 ml Waschpuffer. Übertragen Volumen auf 1,5 ml Eppendorf-Röhrchen. Bei 37 ° C unter Schütteln für 5 min.
- Bei nachfolgenden Wäschen, Spin-down jedes Röhrchen auf einem Minizentrifuge, Setzen Sie auf Probe Dynamag-2 Magnetstreifen für 1 min. Dekantieren, reinigen Sie Tropfen mit einem Kimwipe, in 1 ml Waschpuffer resuspendieren. Bei 37 ° C unter Schütteln für 5 min. Wiederholen Sie dies für insgesamt fünf Wäschen.
- Endlich waschen, resuspendieren Perlen gut. Nehmen Sie 100 ul und beiseite stellen für RNA-Isolierung. Reserve für 900 ul DNA-Fraktion. Legen Sie alle Rohre auf Dynamag-2 Magnetstreifen und Waschpuffer zu entfernen. Drehen Sie alle Röhrchen nach unten kurz, legen Sie sie auf Magnetstreifen. Entfernen Sie das letzte Stück der Waschpuffer vollständig mit einem scharfen 10 pl Pipettenspitze.
7. RNA-Isolierung
Auszug RNA-Fraktion von Chirp Proben mittels qRT-PCR zu quantifizieren.
- Nehmen Sie 100 ul Wulst Proben und 10 ul RNA INPUT Probe. In 85 ul RNA-PK-Puffer pH 7,0 zu Input-RNA. Resuspendieren Sie Perlen in 95 ul RNA-PK-Puffer pH 7,0. Geben Sie 5 ul Proteinease K und inkubieren bei 50 ° C für 45 min mit Ende-zu-Ende zittern.
- Kurz Spin-Down alle Rohre undkochen Proben für 10 min auf Heizblock bei 95 ° C
- Entspannen Proben auf Eis, 500 ul TRIzol zugeben und kräftig vortexen für 10 sek. Inkubieren bei Raumtemperatur für 10 min. Bei -80 ° C oder fahren Sie mit Schritt 4 fort.
- Geben Sie 100 ul Chloroform zu TRIzol behandelten Proben. Vortex kräftig für 10 sek. Drehen am 16100RCF auf einer Tischzentrifuge für 15 min bei 4 ° C
- Nehmen Sie ~ 400 ul wässrige Überstand, die Vermeidung organischer und Schnittstelle.
- Fügen Sie 600 ul (1,5 Volumen) 100% Ethanol und gut mischen. Spin Probe durch MIRNeasy Mini-Säulen. 1x waschen mit RWT (MIRNeasy Mini Kit), 2x mit RPE pro Protokoll des Herstellers. Man eluiert mit 30 ul Nuklease-freiem H 2 O (NFH 2 O).
- Behandeln Sie die RNA-Eluats mit DNA-frei pro Protokoll des Herstellers. Nachdem die Reaktion abgeschlossen ist, erwärmen die Probe für 15 Minuten bei 65 ° C zur vollständigen Inaktivierung jegliches verbleibende DNase.
- Verwenden Sie 1 ul RNA isolieren pro Vertiefung für qRT-PCR-Analyse zu bestätigen lncRNARetrieval. GAPDH wird oft als negative Kontrolle verwendet.
8. DNA-Isolierung
Auszug DNA-Fraktion von Chirp Proben durch Sequenzierung identifizieren oder zu quantifizieren, indem qPCR.
- Bereiten DNA Elutionspuffer (siehe Liste Puffer), 150 ul pro Probe, einschließlich DNA-INPUT.
- Fügen Sie 1 0μL RNase A (10 mg / mL) und 10 ul RNase H (10 U / ul) pro ml DNA Elution Buffer und Wirbel zu mischen.
- Resuspendieren jede Probe von Kügelchen in 150 ul DNA-Elutionspuffer mit RNasen. (INPUT resuspendieren DNA in 140 ul) bei 37 ° C für 30 min unter Schütteln inkubieren.
- Separate Perlen und Überstand auf Dynamag-2 Magnetstreifen. Überstand entfernen und hinzufügen, um beschrifteten Röhrchen.
- Bereiten Sie ein zweites Aliquot der DNA-Elutionspuffer mit 10 ul RNase A (10 mg / mL) und RNaseH (10 U / ul) genau wie in 8.2 getan). Fügen Sie 150 ul zu jeder Probe (einschließlich DNA-Input), inkubieren, Überstand entfernen. Sammeln Sie alle Überstand (Schulterd sein ~ 300 ul).
- In 15 ul PK zu jeder Probe. Inkubieren bei 50 ° C für 45 Minuten unter Schütteln.
- Pre-Spin-down-Gelb-Phase-Lock-Gel-Tuben (5PRIME). Transfer-DNA-Proben zu Phase-Lock-Gel-Tuben, und 300 ul PhOH: Chloroform: Isoamylalkohol pro Probe. Gebrauch kräftig schütteln, 10 min, und der Spin-Down auf einer Tischzentrifuge bei 16100RCF für 5 min bei 4 ° C Nehmen wässrigen von der Spitze (~ 300 ul). Add 3 ul GlycoBlue, 30 ul NaOAc und 900 ul 100% EtOH. Gut mischen und bei -20 ° C über Nacht.
- Spin Proben 16100RCF für 30 min bei 4 ° C
- Überstand vorsichtig dekantieren. 1 ml 70% EtOH und Wirbel zu mischen. Drehen am 16100RCF für 5 min. Überstand entfernen mittels einer Pipette. Luft für 1min trocknen. Resuspendieren in 30 ul EB.
- DNA-Proben sind bereit für die Analyse von qPCR oder die Zubereitung von Hochdurchsatz-Sequenzierung Bibliotheken pro Illumina-Protokoll.
10. Repräsentative Ergebnisse
1 zeigt die CHIRP Workflow. Eine erfolgreiche CHIRP Experiment typischerweise bereichert Ziel-RNA signifikant gegenüber nicht-spezifische RNAs. 2 zeigt Anreicherung von humanen Telomerase-RNA (TERC) aus HeLa-Zellen über GAPDH, ein reichlich zelluläre RNA, die als negative Kontrolle dient. Mehrheit der TERC RNAs (~ 88%) in der Zelle vorhanden, wurden von der Durchführung CHIRP gezogen, während nur 0,46% der GAPDH-RNA wurde abgerufen, was zeigt, einem Anreicherungsfaktor von ~ 200 fache. Unspezifische Sonden wie Sonden Targeting LacZ-RNA, die nicht in Säugetierzellen (2) ausgedrückt wird, können als zusätzliche negative Kontrollen verwendet werden.
DNA-Regionen zu erwarten, um das Ziel zu binden lncRNA werden typischerweise über negativen Bereichen, wenn sie durch qPCR gemessen angereichert. 3 zeigt qPCR Validierung von vier HOTAIR-gebundenen Plattformen primäre humane Vorhautfibroblasten, dass wir durch Ausführen CHIRP-seq in der gleichen Zelllinie bestimmt, während TERC und GAPDH DNA Seiten an sichrve als negative Kontrolle Regionen. Sowohl "gerade" und "ungerade"-Sonde setzt erbrachten vergleichbare Anreicherung erwartet HOTAIR-gebundenen Seiten über Regionen negativ, ein Kennzeichen der wahren lncRNA-Bindungsstellen.
Hochdurchsatz-Sequenzierung von Chirp angereicherte DNA ergibt sich eine globale Karte der lncRNA-Bindungsstellen. Die Drosophila lncRNA roX2 ist bekannt, mit dem X-Chromosom in einer Weise, die für die Dosierung Kompensation erforderlich ist interagieren. 4 zeigt roX2 Bindungsprofil über einen Abschnitt des X-Chromosoms. Sowohl "gerade" und "ungerade" Proben wurden sequenziert und ihre einzigartige Geräusche wurden eliminiert, um eine Spur von sich überlappenden Signale zu erzeugen. Jede "Spitze" hier zeigt eine starke Seite von roX2 bindend. Die komplette Liste der Spur und roX2 Zielgene in Chu et al. 2011 17.
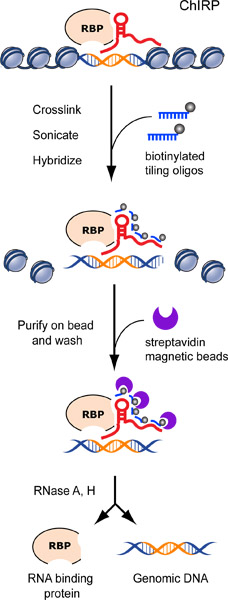
Abbildung 1. Ablaufschema des Chirp-VerfahDure. Chromatin ist lncRNA vernetzt: Proteinaddukte in vivo biotinylierten Sonden hybridisiert Fliesen zu lncRNA zielen, und Chromatin-Komplexe werden unter Verwendung von magnetischen Streptavidin-Kügelchen, gefolgt von strengen Wäschen.. Wir eluieren lncRNA gebundene DNA oder Proteinen mit einem Cocktail von RNase A und H. Eine mutmaßliche lncRNA Bindungssequenz orange schematisiert wird. Zuvor in Chu et al. 2011. 17
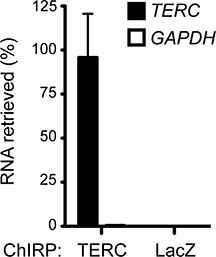
Abbildung 2. CHIRP bereichert für den menschlichen TERC RNA. TERC-asDNA Sonden abrufen ~ 88% der zellulären RNA TERC und GAPDH nicht nachweisbar. LacZ-asDNA Sonden werden als negative Kontrollen verwendet und Abrufen weder RNAs. Mittel + SD dargestellt. Zuvor in Chu et al. 2011. 17
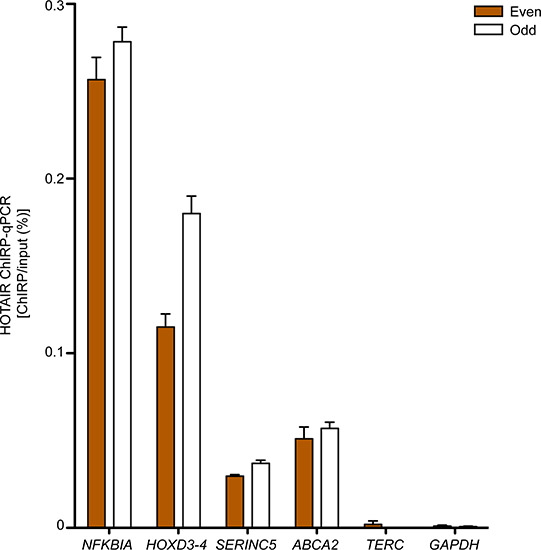
Abbildung 3. HOTAIR CHIRP-qPCR in primären humanen fürEskin Fibroblasten. NFKBIA, HOXD3-4, SERINC5 und ABCA2 sind Bereiche, die mit HOTAIR interagieren. TERC und GAPDH diente als negative Kontrollen. Mittel + SD dargestellt. Zuvor in Chu et al. 2011. 17
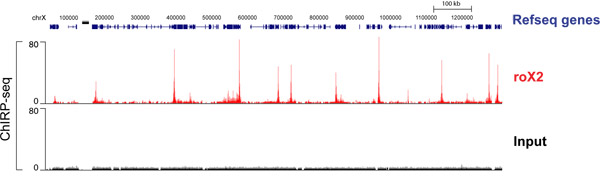
Abbildung 4. CHIRP-seq Daten von roX2 RNA in Sl2 Drosophila-Zellen. "Selbst" und "ungerade" wurden getrennt sequenziert; ihre Daten zusammenführen, um gemeinsam nur Spitzen in beide reflektieren. Das fusionierte Bahn dargestellt. Zuvor in Chu et al. 2011. 17
Diskussion
Hier beschrieben CHIRP-seq, ein Verfahren zur Zuordnung in vivo lncRNA Bindungsstellen genomweiten. Die wesentlichen Parameter für Erfolg sind die Split-Pools von Fliesen Oligonukleotid-Sonden und Glutaraldehyd-Vernetzung. Das Design der Sonden-Affinität ist einfach angesichts der RNA-Sequenz und erfordert keine Vorkenntnisse in der RNA-Struktur oder funktionellen Domänen. Unser Erfolg mit roX2, TERC und HOTAIR - drei ziemlich unterschiedlichen RNAs in zwei Arten - legt nahe, dass CHIRP-seq wahrscheinlich verallgemeinerbar zu viele lncRNAs ist. Wie bei allen Experimenten werden Pflege und angemessene Kontrollen erforderlich, um die Ergebnisse zu interpretieren. Verschiedene lncRNA kann Titration von Bedingungen, und vernünftige Änderung der Bedingungen, wie zB Auswahl von verschiedenen Affinitätssonden oder Vernetzer erfordern, kann auf unterschiedliche Aspekte der RNA-Chromatin-Wechselwirkungen. Wie Chip-seq sind nicht alle Bindungsereignisse unbedingt funktional, und weitere Studien sind erforderlich, um die biologischen Folgen der RNA-o ermittelnccupancy auf Chromatin. Dennoch sehen wir viele interessante Anwendungen dieser Technologie für Forscher anderer Chromatin-assoziierten lncRNAs, die jetzt in die Tausende 8,9. So wie ChIP-seq hat die Tür für genomweite Untersuchungen der DNA-Protein-Interaktionen eröffnet, kann CHIRP-seq Studien des "RNA Interaktom" enthüllen viele neue Wege der Biologie.
Offenlegungen
C. Chu Chang und HY sind als Erfinder auf einer Patentanmeldung auf dieser Methode benannt.
Danksagungen
Wir danken T. Hung, MC. Tsai, O. Manor, E. Segal, M. Kuroda, T. Swigut, und I. Shestopalov für Diskussionen. Unterstützt von der Agentur für Wissenschaft, Technologie und Forschung von Singapur (CC), NIH R01-R01-und CA118750 HG004361 (HYC) und California Institute for Regenerative Medicine (HYC). HYC ist ein Early Career Wissenschaftler des Howard Hughes Medical Institute.
Materialien
| Name | Company | Catalog Number | Comments |
| Buffer List: | |||
| Dissolve a pellet of complete protease inhibitor in 1 ml water as 50x stock. Make 100 mM PMSF in isopropanol (100x stock). Superase-in is used as 200x stock. Store all at -20 °C. | |||
| Lysis Buffer: | |||
| 50 mM Tris-Cl pH 7.0 10 mM EDTA 1% SDS Always add PMSF, P.I. and Superase-in fresh before use except when washing beads | |||
| Proteinase K Buffer (for DNA) | |||
| 100 mM NaCl 10 mM TrisCl pH 8.0 (For RNA use pH 7.0) 1 mM EDTA 0.5% SDS Add 5% by volume Proteainse K (Ambion AM2546 20 mg/ml) fresh before use | |||
| Hybridization Buffer | |||
| 750 mM NaCl 1% SDS 50 mM Tris-Cl pH 7.0 1 mM EDTA 15% formamide (store in the dark at 4 °C) Always add PMSF, P.I. and Superase-in fresh before use | |||
| Wash Buffer | |||
| 2x NaCl and Sodium citrate (SSC) (diluted from 20x SSC Invitrogen stock) 0.5% SDS Always add PMSF fresh before use | |||
| DNA elution Buffer | |||
| 50 mM NaHCO3 1% SDS | |||
| Table of specific reagents and equipment: | |||
| Glutaraldehyde (EM grade) | Sigma-Aldrich | G5882-10x10ml | |
| Motorized pellet mixer | VWR international | V8185-904 | |
| Protease inhibitor | Roche Group | 11873580001 | |
| PMSF | Sigma-Aldrich | 78830 | |
| Superase-in | Ambion | AM2696 | |
| Bioruptor | Diagenode | UCD-200 | |
| Falcon tubes (for sonication) | Corning | 430790 | |
| Proteinase K | Ambion | AM2546 | |
| PCR purification kit | Qiagen | 28106 | |
| C-1 magnetic beads | Invitrogen | 65002 | |
| PMSF | Sigma-Aldrich | P7626-25G | |
| DynaMag-15 magnet | Invitrogen | 123-01D | |
| DynaMag-2 magnet | Invitrogen | 123-21D | |
| MIRNeasy mini kit | Qiagen | 217004 | |
| Rnase H | Epicentre Biotechnologies | R0601K | |
| Rnase A | Sigma-Aldrich | R4875-100MG | |
| Phase Lock Gel Heavy | 5 PRIME | 2302810 | |
| Trizol | Invitrogen | 15596-018 | |
| Phenol:chloroform:Isoamyl | Invitrogen | 15593-031 | |
| Chloroform | Ricca | RSOC0020-1C | |
| GlycoBlue | Ambion | AM9515 | |
| Glycine | JT Baker | 4057-06 | |
| PBS, pH 7.4 | Invitrogen | 10010-049 | |
| Elution Buffer (EB) | Qiagen | 19086 | |
| 20x SSC | Invitrogen | 15557-036 | |
| 10% SDS | Invitrogen | 15553-027 | |
| DNA-free | Ambion | AM1906 | |
| Buffer kit | Ambion | AM9010 | |
| Formamide | Invitrogen | 15515-026 | |
Referenzen
- Koziol, M. J., Rinn, J. L. RNA traffic control of chromatin complexes. Curr. Opin. Genet. Dev. 20, 142-148 (2010).
- Mercer, T. R., Dinger, M. E., Mattick, J. S. Long non-coding RNAs: insights into functions. Nat. Rev. Genet. 10, 155-159 (2009).
- Rinn, J. L. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell. 129, 1311-1323 (2007).
- Zhao, J., Sun, B. K., Erwin, J. A., Song, J. J., Lee, J. T. Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome. Science. 322, 750-756 (2008).
- Kelley, R. L. Epigenetic spreading of the Drosophila dosage compensation complex from roX RNA genes into flanking chromatin. Cell. 98, 513-522 (1999).
- Pandey, R. R. Kcnq1ot1 antisense noncoding RNA mediates lineage-specific transcriptional silencing through chromatin-level regulation. Mol. Cell. 32, 232-246 (2008).
- Wang, K. C. A long noncoding RNA maintains active chromatin to coordinate homeotic gene expression. Nature. 472, 120-124 (2011).
- Khalil, A. M. Many human large intergenic noncoding RNAs associate with chromatin-modifying complexes and affect gene expression. Proc. Natl. Acad. Sci. U.S.A. 106, 11667-11672 (2009).
- Zhao, J. Genome-wide identification of polycomb-associated RNAs by RIP-seq. Mol. Cell. 40, 939-953 (2010).
- Meller, V. H., Wu, K. H., Roman, G., Kuroda, M. I., Davis, R. L. roX1 RNA paints the X chromosome of male Drosophila and is regulated by the dosage compensation system. Cell. 88, 445-457 (1997).
- Franke, A., Baker, B. S. The rox1 and rox2 RNAs are essential components of the compensasome, which mediates dosage compensation in Drosophila. Mol. Cell. 4, 117-122 (1999).
- Gupta, R. A. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature. 464, 1071-1076 (2010).
- Tsai, M. C. Long noncoding RNA as modular scaffold of histone modification complexes. Science. 329, 689-693 (2010).
- Zappulla, D. C., Cech, T. R. RNA as a flexible scaffold for proteins: yeast telomerase and beyond. Cold Spring Harb. Symp. Quant. Biol.. 71, 217-224 (2006).
- Nagano, T. The Air noncoding RNA epigenetically silences transcription by targeting G9a to chromatin. Science. 322, 1717-1720 (2008).
- Carter, D., Chakalova, L., Osborne, C. S., Dai, Y. F., Fraser, P. Long-range chromatin regulatory interactions in vivo. Nature. 32, 623-626 (2002).
- Chu, C., Qu, K., Zhong, F. L., Artandi, S. E., Chang, H. Y. Genomic Maps of Long Noncoding RNA Occupancy Reveal Principles of RNA-Chromatin Interactions. Mol. Cell. , (2011).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5, 877-879 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten