Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Selektive Aufnahme von 5-Hydroxymethylcytosin aus genomischer DNA
In diesem Artikel
Zusammenfassung
Beschrieben wird ein zweistufiges Verfahren unter Verwendung Kennzeichnung β-Glucosyltransferase (β-GT), um ein Azid-Glukose zu 5-HMC übertragen, gefolgt von Klickchemie einen Linker Biotin für die einfache und dichteunabhängigen Anreicherung zu übertragen. Dieses effiziente und spezifische Kennzeichnung Methode ermöglicht die Anreicherung von 5-HMC mit extrem niedrigen Hintergrund und hohem Durchsatz epigenomischen Mapping über Next-Generation-Sequenzierung.
Zusammenfassung
5-Methylcytosin (5-mC) bildet ~ 2-8% der gesamten Cytosine in humanen genomischen DNA und beeinflusst ein breites Spektrum an biologischen Funktionen, einschließlich Genexpression, die Aufrechterhaltung der Genom Integrität, elterliche Prägung, X-Chromosom-Inaktivierung, die Regulierung des Entwicklung, Alterung und Krebs 1. Kürzlich wurde die Anwesenheit eines oxidierten 5-mC, 5-Hydroxymethylcytosin (5-HMC), in Säugerzellen entdeckt, insbesondere in embryonalen Stamm (ES)-Zellen und neuronalen Zellen 2-4. 5-HMC wird durch Oxidation von 5-mC katalysiert durch TET Familie Eisen (II) / α-Ketoglutarat-abhängige Dioxygenase 2, 3 erzeugt. 5-HMC wird vorgeschlagen, bei der Aufrechterhaltung der embryonalen Stamm (mES) Zelle, normale Hämatopoiese und Tumore und Zygote Entwicklung 2, 5-10 beteiligt sein. Zum besseren Verständnis der Funktion von 5-HMC, ist eine zuverlässige und einfache Sequenzierung von wesentlicher Bedeutung. Traditionelle Bisulfit-Sequenzierung kann nicht unterscheiden, 5-HMC von 5-mC 11 12.
Hier beschreiben wir eine einfache Zwei-Schritt-Verfahren zur selektiven chemischen Kennzeichnung der 5-HMC. Im ersten Markierungsschritt wird 5-HMC in genomischer DNA mit einem 6-Azid-Glucose katalysiert durch β-GT, eine Glucosyltransferase aus Bakteriophagen T4 bezeichnet, in einer Weise, die das 6-Azid-Glucose überträgt 5-HMC vom modifiziertes Cofaktor, UDP-6-N3-Glc (6-N3UDPG). Im zweiten Schritt, Biotinylierung wird ein Disulfid Biotin Linker an die Azidgruppe durch Klickchemie befestigt. Beide Schritte sind hoch spezifisch und effizient, was zu vervollständigen Kennzeichnung unabhängig von der Fülle der 5-HMC in der genomischen Regionen und geben extrem geringe Hintergrund. Nach Biotinylierung von 5-HMC, die 5-HMC-enthaltende DNA-Fragmente werden dann selektiv eingefangenmit Streptavidin-Kügelchen in einer Dichte-unabhängige Weise. Die daraus resultierenden 5-HMC-angereicherte DNA-Fragmente konnten für nachgelagerte Analysen, einschließlich der nächsten Generation Sequenzierung verwendet werden.
Unsere selektive Markierung und Capture-Protokoll überträgt hohe Empfindlichkeit auf jede Quelle der genomischen DNA mit variablem / diverse 5-HMC Häufigkeiten. Obwohl der Hauptzweck dieses Protokolls ist seine nachgeordneten Anwendung (dh. Der nächsten Generation Sequenzierung Karte aus dem 5-HMC Verteilung im Genom), ist es kompatibel mit Einzel-Molekül-, real-time SMRT (DNA)-Sequenzierung, das ist der Lage ist, single-base Auflösung Sequenzierung von 5-HMC.
Protokoll
Ein. Genomic DNA-Fragmentierung
Fragment genomischer DNA unter Verwendung von Ultraschallbehandlung auf einen gewünschten Größenbereich für das Genom-weite Sequenzierplattform geeignet. (Wir gehen normalerweise in ~ 300 bp beschallen.) Überprüfen Sie die Größenverteilung der fragmentierten genomischen DNA auf einem 1% Agarosegel (Abbildung 1).
2. DNA-Präparation
Bestimmen Sie die Ausgangs-DNA Mengen auf die Fülle der 5-HMC in genomischer DNA. Da 5-HMC Ebenen sind in den einzelnen Gewebearten hängen Ausgangs-DNA Mengen auf den 5-HMC Pegeln der Abtastwerte. Siehe Tabelle 1 für Beispiele.
3. β-GT katalysierte Reaktion (Glucose-Transfer-Reaktions)
- Mischung durch Pipettieren der Mischung, wie in Tabelle 2 und inkubieren in einem 37 ° C Wasserbad für 1 Stunde.
- Nach der Inkubation bereinigen die Reaktion mit QIAquick Nucleotide Removal Kit mit 10 ug DNA proSpalte. Eluieren mit 30 ul Wasser pro Spalte und kombinieren.
4. Biotinylierungsreaktion (Klick-Chemie)
- Hinzufügen DBCO-SS-PEG3-Biotin-Konjugat-Arbeitslösung (1 mM) in der eluierten DNA-Lösung (aus Stufe 3) bis zu einer Endkonzentration von 150 pM (dh 5 ul Arbeitslösung pro 30 ul DNA-Lösung).
- Mix durch Pipettieren und inkubieren in einem 37 ° C warmen Wasserbad für 2 Stunden.
- Reinigen Sie die Reaktion mit QIAquick Nucleotide Removal Kit. Die ideale insgesamt Elutionsvolumen beträgt 100 ul.
- Quantifizierung der gewonnenen DNA Menge mit Mikrolitermaßstab Spektralphotometer (zB., NanoDrop).
5. Erfassen von 5-HMC-haltige DNA
- Waschen 50 ul Dynabeads MyOne Streptavidin C1 3-mal mit 1 ml 1X B & W-Puffer nach den Anweisungen des Herstellers. Trennen Sie die Perlen mit einem Magnetfuß.
- Hinzufügen identischen Volumen von 2X B & W-Puffer zu dem wiedergewonnenen biotinylierten DNA (100 ul) zu den gewaschenen Perlen.
- Inkubieren für 15 min bei Raumtemperatur unter leichtem Drehung auf einem Rotator.
- Trennen Sie die Perlen mit einem Magnetfuß und waschen die Kügelchen 3-mal mit 1 ml 1X B & W-Puffer.
- Eluieren der DNA durch Inkubation der Kügelchen in 100 ul frisch hergestellten 50 mM DTT für 2 Stunden bei Raumtemperatur unter leichtem Drehung auf einem Rotator.
- Trennen Sie die Perlen mit einem Magnetfuß. Absaugen Elutionsmittel und Last auf eine Bio-Micro Spin 6 Kolonne nach der Herstellung aus, damit die DTT entfernen. Die Ziel-DNA in der Lösung bekommen.
- Reinige das eluierte DNA aus dem vorherigen Schritt von Qiagen MinElute PCR Purification Kit und eluieren DNA in 10 ul Puffer EB. Quantifizierung DNA mit Qubit Fluorometer oder NanoDrop, wenn die Konzentration höher als 20 ng / ul. Die DNA ist bereit für nachgeschaltete genomweite Sequenzierung Bibliothek Vorbereitung.
6. Repräsentative Ergebnisse
Wenn die Qualität of genomische DNA ist hoch, sind typische Ausbeuten der Gewinnung nach dem β-GT und Biotinylierung Reaktionen ~ 60-70%. Allerdings variieren die Gefangennahme Effizienz deutlich mit verschiedenen Gewebearten abhängig von den 5-HMC Ebenen der Proben. Typischerweise ist der Abscheidegrad für Gehirn genomische DNA ~ 4-9%, und in einigen extremen Fällen kann der Wirkungsgrad bis zu 12%. Für ES-Zellen, ist die durchschnittliche Einfangeffizienz ~ 2-4%, im Gegensatz zu ~ 0,5% für neurale Stammzellen. Die geringste Effizienz bisher gesehen war für genomische DNA von Krebszellen. Alle angereicherte DNA ist bereit für Standard der nächsten Generation Bibliothek Vorbereitung Protokolle. Darüber hinaus kann das eingefangene DNA auch als Matrize für die Echtzeit-PCR, um die Anreicherung von einigen Fragmenten im Vergleich zum Ausgangs-DNA, erkennen, ob die entsprechenden Primer vorhanden sind, verwendet werden.
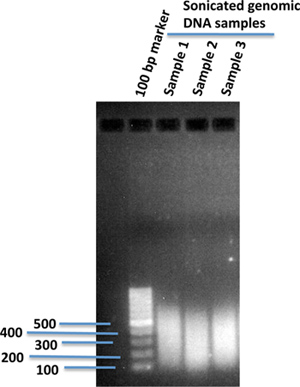
Abbildung 1. Beschallte humanen genomischen DNA-Fragmente in1% Agarosegel. 10 ug genomischer DNA aus humanen iPS Zellen in 120 ul TE Puffer 1X isoliert wurde beschallt Verwendung einer Ultraschallbehandlung Vorrichtung (Covaris). Nach der Beschallung wurden 2 ul der beschallten DNA auf 1% Agarosegel geladen mit 100 bp DNA-Marker, um die Größen der DNA-Fragmente beschallte vergleichen.
| Komponente | Volumen | Endkonzentration |
| Wasser | _ Ul | |
| 10 X β-GT Reaction Buffer | 2 ul | X 1 |
| Bis zu 10 ug genomische DNA | _ Ul | Bis zu 500 ng / ul |
| UDP-6-N 3-Glc (3 mM) | 0,67 ul | 100 uM |
| β-GT (40 uM) | 1 ul | 2 uM |
| Gesamtvolumen | 20 ul |
i) Für das Tissue genomische DNA (high 5-HMC-Gehalt> 0,1%)
| Komponente | Volumen | Endkonzentration |
| Wasser | _ Ul | |
| 10 X β-GT Reaction Buffer | 10 ul | X 1 |
| Bis zu 20 ug genomische DNA | _ Ul | Bis zu 500 ng / ul |
| UDP-6-N3-Glc (3 mM) | 1,33 ul | 100 uM |
| β-GT (40 uM) | 2 ul | 2 uM |
| Gesamtvolumen | 40 ul |
ii) für die Stammzellenforschung genomische DNA (Median 5-HMC-Gehalt ~ 0,05%)
| Komponente | Volumen | Endkonzentration |
| Wasser | _ Ul | |
| 10 X β-GT Reaction Buffer | 10 ul | X 1 |
| Bis zu 50 ug genomische DNA | _ Ul | Bis zu 500 ng / ul |
| UDP-6-N3-Glc (3 mM) | 3,33 ul | 100 uM |
| β-GT (40 uM) | 5 ul | 2 uM |
| Gesamtvolumen | 100 ul |
iii) Für Krebszelle genomische DNA (low 5-HMC-Gehalt ~ 0,01%)
Tabelle 1. Beispiele für Mengen von Input-DNA und Kennzeichnung Reaktionen unter Verwendung der Proben mit verschiedenen 5-HMC Ebenen durch die selektive chemische Markierung Methode.
| Probe | 5-HMC-Ebene | Ausgangs-DNA (ug) | Erholung nach Kennzeichnung (Eingang an Kügelchen) (ug) | Rückgewinnungsausbeute | Pull-Down-DNA (ng) | Pull-Down-Ausbeute |
| Adulten Maus Kleinhirns | 0,4% | 10 | 7,5 | 75% | 236 | 3,1% |
| Postnatalen Tag 7 Maus Kleinhirns | 0,1% | 11 | 9 | 82% | 140 | 1,6% |
| Maus-ES-Zellen E14 | 0,05% | 60 | 42 | 70% | 350 | 0,8% |
Tabelle 2. Repräsentative Ergebnisse aus Maus Hirngewebe und ES-Zellen.
Diskussion
5-Hydroxymethylcytosin (5-HMC) ist ein kürzlich identifizierten epigenetische Veränderung in erheblichen Mengen in bestimmten Säugetieren Zelltypen. Das hier vorgestellte Verfahren ist für die Bestimmung der genomweiten Verteilung der 5-HMC. Wir verwenden T4 Bakteriophagen β-glucosyltransferase zur Erzeugung eines manipulierten Glucose-Rest enthaltend eine Azidgruppe auf die Hydroxylgruppe von 5-HMC übertragen. Die Azidgruppe kann chemisch mit Biotin werden zur Feststellung, Affinitätsanreicherung und Sequenzieru...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Studie wurde zum Teil durch die National Institutes of Health (GM071440 nach CH und NS051630/MH076090/MH078972 PJ) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name | Firma | Katalog # | Kommentar |
| Reagenzien | |||
| 5M Natriumchlorid (NaCl) | Promega | V4221 | |
| 0,5 M pH 8,0 Ethylendiamintetraessigsäure (EDTA) | Promega | V4231 | |
| 1M Trizma Basis (Tris) pH7.5 | Invitrogen | 15567-027) | |
| HEPES 1M, pH-Wert 7,4 | Invitrogen | 15630 | |
| Magnesiumchlorid (MgCl 2) 1M | Ambion | AM9530G | |
| Dimethylsulfoxid (DMSO) | Sigma | D8418 | |
| Tween 20 | Fisher BioReagents | BP337-100 | |
| DBCO-SS-PEG3-Biotin-Konjugat | Klick-Chemie-Tools | A112P3 | |
| 1,4-Dithiothreitol, Reinstwasser (DTT) Superpure | Invitrogen | 15508-013 | |
| QIAquick Nucleotide Removal Kit | Qiagen | 28304 | |
| Micro Bio-Spin 6 Spalte | Bio-Rad | 732-6222 | |
| Dynabeads MyOne | Invitrogen | 650-01 | |
| Streptavidin C1 | |||
| Qiagen MinElute PCR Purification Kit | Qiagen | 28004 | |
| UltraPure Agarose | Invitrogen | 16500500 | |
| UDP-6-N 3-Glucose | Aktive Motif | 55013 | |
| Enzym | |||
| β-glucosyltransferase (β-GT) | New England Biolab | M0357 | |
| Ausrüstung | |||
| Beschallung Gerät | Covaris | ||
| Desktop-Zentrifuge | |||
| Wasserbad | Fisher Scientific | ||
| Gel laufende Apparatur | Bio-Rad | ||
| NanoDrop1000 | Thermo Scientific | ||
| Labquake Rohr Shaker | Barnstead | ||
| Labquake Rohr Shaker | Thermolyne | ||
| Magnetic Separation Ständer | Promega | Z5342 | |
| Qubit 2,0 Fluorometer | Invitrogen | ||
| Reagenz Setup 10 X β-GT Reaktionspuffer (500 mM HEPES pH 7,9, 250 mM MgCl 2) 2 X Binding und Waschen (B & W)-Puffer (10 mM Tris pH 7,5, 1 mM EDTA, 2 M NaCl, 0,02% Tween 20) . |
Referenzen
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. , 245-254 (2003).
- Ito, S. Role of Tet proteins in 5mC to 5hmC conversion, ES-cell self-renewal and inner cell mass specification. Nature. 466, 1129-1133 (2010).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Kriaucionis, S., Heintz, N. The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain. Science. 324, 929-930 (2009).
- Ko, M. Impaired hydroxylation of 5-methylcytosine in myeloid cancers with mutant TET2. Nature. 468, 839-843 (2010).
- Koh, K. P. Tet1 and tet2 regulate 5-hydroxymethylcytosine production and cell lineage specification in mouse embryonic stem cells. Cell Stem Cell. 8, 200-213 (2011).
- Iqbal, K., Jin, S. G., Pfeifer, G. P., Szabo, P. E. Reprogramming of the paternal genome upon fertilization involves genome-wide oxidation of 5-methylcytosine. Proceedings of the National Academy of Sciences of the United States of America. 108, 3642-3647 (2011).
- Wossidlo, M. 5-Hydroxymethylcytosine in the mammalian zygote is linked with epigenetic reprogramming. Nat. Commun. 2, 241 (2011).
- Gu, T. P. The role of Tet3 DNA dioxygenase in epigenetic reprogramming by oocytes. Nature. 477, 606-610 (2011).
- Dawlaty, M. M. Tet1 is dispensable for maintaining pluripotency and its loss is compatible with embryonic and postnatal development. Cell Stem Cell. 9, 166-175 (2011).
- Huang, Y. The behaviour of 5-hydroxymethylcytosine in bisulfite sequencing. PLoS One. 5, e8888 (2010).
- Song, C. X. Selective chemical labeling reveals the genome-wide distribution of 5-hydroxymethylcytosine. Nat. Biotechnol. 29, 68-72 (2011).
- Pastor, W. A. Genome-wide mapping of 5-hydroxymethylcytosine in embryonic stem cells. Nature. 473, 394-397 (2011).
- Matarese, F., Pau, C. a. r. r. i. l. l. o. -. d. e. S. a. n. t. a., E, ., Stunnenberg, H. G. 5-Hydroxymethylcytosine: a new kid on the epigenetic block. Mol. Syst. Biol. 7, 562 (2011).
- Szwagierczak, A., Bultmann, S., Schmidt, C. S., Spada, F., Leonhardt, H. Sensitive enzymatic quantification of 5-hydroxymethylcytosine in genomic DNA. Nucleic Acids Res. 38, 181 (2010).
- Terragni, J., Bitinaite, J., Zheng, Y., Pradhan, S. Biochemical characterization of recombinant β-glucosyltransferase and analysis of global 5-hydroxymethylcytosine in unique genomes. Biochemistry. , (2012).
- Rusmintratip, V., Sowers, L. C. An unexpectedly high excision capacity for mispaired 5-hydroxymethyluracil in human cell extracts. Proc. Natl. Acad. Sci. U.S.A. 97, 14183-14187 (2000).
- Globisch, D. Tissue distribution of 5-hydroxymethylcytosine and search for active demethylation intermediates. PLoS One. 5, e15367 (2010).
- Yildirim, O. Mbd3/NURD Complex Regulates Expression of 5-Hydroxymethylcytosine Marked Genes in Embryonic Stem Cells. Cell. 147, 1498-1510 (2011).
- Szulwach, K. E. Integrating 5-hydroxymethylcytosine into the epigenomic landscape of human embryonic stem cells. PLoS Genet. 7, e1002154 (2011).
- Szulwach, K. E. 5-hmC-mediated epigenetic dynamics during postnatal neurodevelopment and aging. Nat. Neurosci. 14, 1607-1616 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten