Method Article
Halbautomatische Biopanning bakterielle Anzeige Bibliotheken für Peptid Affinität Reagenz Entdeckung und Analyse der daraus resultierenden Isolate
In diesem Artikel
Zusammenfassung
Biopanning bakterielle Anzeige Bibliotheken ist eine bewährte Technik für Entdeckung von Peptid Affinität Reagenzien, eine robuste Alternative zu Antikörpern. Die halbautomatische Sortiermethode hierin wurde gestrafft, Biopanning um das Auftreten von Fehlalarmen zu verringern. Hier zeigen wir den Denkprozess und Techniken Bewertung der Kandidaten und nachgelagerte Analyse zu minimieren.
Zusammenfassung
Biopanning bakterielle Anzeige Bibliotheken ist eine bewährte Technik für Peptid Affinität Reagenz Entdeckung für die Anerkennung von biotischen und abiotischen Zielen. Peptid Affinität Reagenzien eignet sich für ähnliche Anwendungen, Antikörper, einschließlich der Erkennung und Therapie, sondern sind robuster und in extremen Umgebungen mehr durchführen. Spezifische Anreicherung von Peptid-Capture Agenten zu einem Protein-Ziel von Interesse wird verstärkt mit semi-automatischen Sortierverfahren, die Bindung zu verbessern und Schritte zu waschen und daher verringern das Auftreten von falsch positiven Bindemittel. Eine halbautomatische Sortierung ist beschrieben hierin für den Einsatz mit einer kommerziellen automatisierte magnetisch aktivierte Zelle Sortierung Gerät mit einer ungezwungenen bakterielle Anzeige sortieren Bibliothek zufällige 15-Mer Peptide zum Ausdruck zu bringen. Mit geringfügigen Änderungen, diese Methoden sind erweiterbar auf andere automatische Geräte, andere Sortierung Bibliotheken und anderen Organismen. Ein primäres Ziel dieser Arbeit ist eine umfassende Methodik und erläutern den gedanklichen Prozess angewendet in der Analyse und den daraus resultierenden Pool der Kandidaten zu minimieren. Diese Techniken umfassen die Analyse auf Zelle Bindung mit Fluoreszenz-aktivierte Zelle sortieren (FACS), um Affinität und Spezifität bei der Sortierung und beim Vergleich der einzelner Kandidaten zu beurteilen und die Analyse der Peptid-Sequenzen, um Trends zu erkennen und Konsensus-Sequenzen für das Verständnis und potenziell Verbesserung der Affinität zu und Spezifität für das Ziel von Interesse.
Einleitung
Biopanning ist eine bewährte Technik für Affinität Reagenz Entdeckung, mit bakteriellen Bibliotheken eine günstige Quelle für Peptid Affinität Reagenzien 1,2,3,4,5, 6,7,8,9,10,11,12,13,14, 15,16,17,18,19,20,21,22,23, 24. In ähnlicher Weise Phagen anzeigen 25,26 und Hefe Anzeige 27,28, die Peptide auf der Zelloberfläche ausgesetzt sind und stehen den biotischen und abiotischen Ziele mit dieser Interaktionen binden angetrieben durch das Peptidrückgrat und die einzigartigen Eigenschaften der Aminosäure-Seitenketten 29. Im Gegensatz zu den Phagen-Display die Bakterien selbst sind selbstreplizierend und enthalten alle genetische Material für Anzeige ohne Elution von gebundenen Phagen und Reinfektion der Zellen erforderlich. Im Gegensatz zu Hefe Display ist bakterielle Anzeige schneller aufgrund der Rapid (ca. 20 min 30) Verdopplungszeit von Escherichia coli. Die daraus resultierenden Peptid Affinität Reagenzien hergestellt werden können-Zellen für den Einsatz in einer Vielzahl von Plattformen 16,17,26 sensing und haben Potenzial für den Einsatz als lebendem Material wenn angezeigt auf- 2 Zelle , 31 , 32. verglichen mit monoklonalen Antikörpern für die Anerkennung der spezifischen Ziele, Peptide sind viel kleiner und flexibler, und Peptid-Display-Bibliotheken können leicht manipuliert werden, auf DNA-Ebene zu vermeiden, bestimmte Aminosäuren wie Cystein 33 . Peptide werden zunehmend ausgebeutet für Therapeutik 34,35,36 und andere Anwendungen, wo Antikörper in der Regel aufgrund ihrer einfachen Entdeckung, reproduzierbare Synthetik (off-Zellen genutzt werden würde ) Produktion, und überlegene Leistung in extremen Umgebungen, bietet eine funktionale Reagenz auch nach Exposition gegenüber hohen Temperaturen oder langfristige Lagerung ohne Kühlung 37,38. Die Fähigkeit, die Kühlkette für die Lagerung von Reagenzien zu überwinden ist von besonderem Interesse für das Department of Defense, zum Beispiel, die in extremen Umgebungen arbeiten muss. Thermischer Stabilität war daher eine wünschenswerte Eigenschaft für die Affinität Reagenzien die gewählten Ziele Beispiele in dieser Handschrift zu erkennen.
Vor kurzem, Biopanning bakterielle Anzeige Bibliotheken geworden noch unkompliziert und zuverlässig mit dem Einsatz von teilautomatisierten magnetisch aktivierte Zelle sortieren (MACS oder MCS) Methoden 9,14,39. Diese Methoden wurden nachgewiesen für biologische Ziele mit mehreren ähnlichen Plattformen mit verschiedenen Vorteilen, auch eine handelsübliche Sortierung automatisiert magnetisch aktivierte Zelle sortieren (AutoMACS oder AutoMCS) Instrument (siehe Tabelle der Verwenden Sie Materialien) 1,14,15 und mehr spezialisierte Geräte, die die Probe in eine Einweg-Patrone 7,9 oder Chip 39, eine wertvolle Spezifikation für umschließen mit Organismen oder Toxine, die Biosafety Level 2 (BSL-2) oder höhere7sind. Diese semi-automatischen Methoden könnte erweitert werden, um sortieren für Peptide, die in der Lage, an abiotischen Materialien zu binden, wenn die Materialien magnetisch sind oder die Möglichkeit haben, auf eine magnetische Wulst und für die Verwendung mit anderen Organismen (z. B. anaerobe Bakterien) beschichtet werden, wenn die Sortierung und Wachstumsbedingungen sind verändert. Zusätzlich zum Sortieren bakterielle Anzeige Bibliotheken, wurde das kommerzielle AutoMCS Instrument hier auch teilweise für die ersten Schritte der Entdeckung des menschlichen Single-Kette Antikörperfragmente angezeigt auf der Oberfläche der Hefe, gefolgt von weiteren Isolierung verwendet Verwendung von fluoreszierenden aktiviert Zelle sortieren (FACS) 40. Die Hauptvorteile, semi-Automatisierung werden verringert, Sortierung, Zeit und Mühe und mehr robust und reproduzierbare Beseitigung der ungebundenen Zellen aus der magnetischen Beads während Waschen Schritte, führt zu weniger Fehlalarme, weniger erforderliche Sortier runden und weniger komplizierte Analyse der Downstream- 9,14. Im Falle der kommerziellen AutoMCS Instrument, es gibt mehrere vorinstallierte Programme, die möglicherweise optimal für verschiedene Anwendungen, wie z. B. negative Vorsortierung (Erschöpfung), Priorisierung von Reinheit, endgültigen Probenvolumen zu minimieren und die Erhöhung oder abnehmende Empfindlichkeit für die Isolierung von seltenen Zellen 41.
Der Schwerpunkt dieser Arbeit ist die Methodik zur Biopanning eine bakterielle Anzeigebibliothek mit dem im Handel erhältlichen AutoMCS Instrument zeigen und erläutern den gedanklichen Prozess angewendet in der Analyse und Minimierung den daraus resultierenden Pool der Kandidaten in Bestellung, Ausweitung auf andere Anwendungen zu erleichtern. Die hier verwendete Bibliothek (siehe Tabelle der Materialien) 12,13 enthält ca. 109- 1011 einzigartige 15-Mer Peptide angezeigt über eine modifizierte Version des äußeren Bakterienmembran Proteins OmpX in eine ungezwungene Natur an der Zelloberfläche, die voraussichtlich für das freie Peptid in Lösung wenn repräsentativ synthetisch hergestellt. Dieses Protein Gerüst enthält zusätzlich eine Positivkontrolle P2X Peptid am C-Terminus für die Überwachung der Ausdruck über Bindung zur YPet Mona. Jedoch sollte eine gekaufte oder neuartige Bibliothek mit geeigneten Vielfalt und anderen Eigenschaften, die notwendig für die jeweilige Anwendung gewünscht in ähnlicher Weise mit diesem Biopanning Verfahren arbeiten, obwohl einige geringfügige Änderungen erforderlich sein können. Eine schematische Darstellung dieser Biopanning-Protokoll ist in Abbildung 1dargestellt. Darüber hinaus könnte das Protokoll angepasst werden andere automatisierte magnetische Sortierung Plattformen und anderen Sortierstrategien. Magnetische Handsortierung mit einem Benchtop-Magneten erfolgreich demonstriert, wenn auch mit mehr kompliziert und zeitaufwändig nachgelagerte Analyse erforderlich, durch Fehlalarme 14erhöht, und dennoch bietet eine alternative Möglichkeit, die AutoMCS Schritte, wenn keine automatisierte magnetische Zelle Sortierung Gerät steht zur Verfügung.
Protokoll
1. Biopanning bakterielle Anzeige Bibliotheken verwenden autoMCS
Hinweis: Dieses AutoMCS-basierte Sortierung Protokoll wurde zuvor beschriebenen 14und eine gründlichere "erweiterte Protokoll für neue Benutzer" gibt es in der ergänzenden Materialien für diese Handschrift für weitere Einzelheiten, einschließlich einer Erläuterung der Potenzial Änderungen. Wenn eine AutoMCS-Gerät für die Sortierung nicht verfügbar ist, siehe die zusätzliche Protokoll von Sarkes Et Al. 2015 da eine manuelle Sortieren Protokoll auch darin beschrieben wird 14arbeiten. Das AutoMCS-Protokoll vorausgesetzt, hier wurde weiter angepasst zusätzlichen negativen sortieren Schritte für verbesserte Spezifität 1,15. Eine schematische Darstellung dieser Biopanning-Protokoll ist in Abbildung 1dargestellt.
- Negativen Sortierung Bindemittel voraussichtlich mit magnetischen Beads Kreuzreaktion zu entfernen
- 500 mL Luria Brühe Miller (LB) mit geeigneten Antibiotika mit ca. 1 x 1011Zellen der verschiedenen bakteriellen anzeigen sortieren Bibliothek zu impfen (siehe Tabelle der Materialien).
Hinweis: Die bakterielle Peptid Bibliothek verwendet hier (siehe Tabelle der Materialien) enthält ca. 109- 1011Einzelelemente 8,12 und LB mit 25 µg/mL Chloramphenicol (LB Cm25) gewachsen ist. Der Rest dieses Protokolls wird davon ausgehen, dass diese Bibliothek verwendet wird. - Inkubation bei 37 ° C mit Schütteln bei 225 u/min, bis die Kultur eines OD600 von 0,5 erreicht - 0,55. Induzieren Sie Peptid Ausdruck mit 0,04 % w/V L-Arabinose durch Verdünnung eine 4 % Lager 1: 100, schütteln bei 225 u/min für 45 min bei 37 ° C.
- Ort induzierten Kultur auf Eis. Zentrifugieren Sie ca. 2 x 1011 Zellen bei 6000 X g für 20 min bei 4 ° C. Entfernen Sie überstand und Aufschwemmen Sie Zellen in 1,5 mL PBS durch sanft schütteln.
Hinweis: Ein OD600 von 1,0 entspricht ca. 1 x 109 Zellen/mL für E. Coli.
Hinweis: Als dies tun nicht VORTEX kann die Zellen lysiert; Aufschwemmen Sie per hand oder kurz schütteln bei ≤ 225 u/min, 4° C. - Übertragung von Zellen auf Microcentrifuge Tube(n) und Spin bei 6.000 x g für 5 min bei RT oder 4 ° C. Entfernen Sie überstand. Aufschwemmen der Zelle Pellet in 1 mL Phosphat gepufferte Kochsalzlösung (PBS) mit 0,5 % Rinderserumalbumin (BSA; PBS-B).
- Waschen Sie 300 µL Streptavidin beschichteten Perlen (ca. 3 x 109 Perlen, siehe Tabelle der Materialien) in 1 mL PBS-B und Zentrifuge für 5 min bei ≥5, 000 X g (bei RT). Legen Sie Rohr in einem Bank-Top Magnetpartikel-Separator. Entfernen Sie vorsichtig überstand, das Pellet zu vermeiden.
- Aufzuwirbeln Sie die Perlen in der Zelle plus PBS-B Mischung vorbereitet über. Inkubieren Sie bei 4° C auf einer rotierenden Plattform für 45 min. Ort Proben auf Eis.
- Schalten Sie ein AutoMCS Instrument, um das System zu initialisieren. Prime Linien mithilfe des Herstellers ausführen und waschen Puffer (siehe Tabelle der Materialien) durch die Auswahl "Wash Now" am unteren Rand des Bildschirms im Menü "Trennung" dann "Spülen" und "Run".
- Entlüften Sie das System mit PBS-B, mit PBS-B als Waschpuffer und Puffer läuft in den nächsten Schritten. Wählen Sie Menü "Trennung" aus der oberen Navigationsleiste und wählen Sie "Jetzt waschen". Wählen Sie "Spülen" und "Ausführen", um das System mit frischem PBS-B. prime
- Übertragen Sie Zelle und Perle Mischung aus der Inkubationsschritt auf eine 15 mL konische Rohr. Diese Röhre in das Musterfeld und leer 15 mL konische Röhrchen in den Schlitzen in positive und negative Selektion des Regales vorgekühlt (4 ° C) (siehe Tabelle der Materialien). Instrumentenplattform Rack aufsetzen.
- Weisen Sie eine Trennung-Programm für jede Probe auf dem Gestell (bis zu 5 Proben kann in einem Arbeitsgang sortiert werden). Fügen Sie einen "Spülen" Schritt zwischen jeder Probe und nach der letzten Probe. Wählen Sie "Ausführen", um die Zelle Trennung zu starten. Wählen Sie "OK" zu bestätigen, dass genügend Puffer verfügbar ist.
Hinweis: Das "Posselds"-Trennung-Programm eignet sich gut für negative sortieren und positive Sortierung Runde 1 und "Posseld" wird bevorzugt für die Sortierung runden 2-4, aber beide Programme erfolgreich verwendet worden für alle Negative und positive Sortierung 14 Runden ,15 und andere Programme können für eine bestimmte Anwendung besser erweisen (beschrieben in Diskussion). - Wenn das Programm abgeschlossen ist, entfernen Sie den Ständer und behalten Sie den entsprechenden Bruchteil. Hier, für eine negative Art behalten Sie negative Brüche (mit Zellen ohne Perlen). Auf Eis legen.
- Verwenden Sie den negativen Art Bruch in seiner Gesamtheit 1 L LB Cm25 mit 0,2 % w/V D-Glucose zu impfen. Wachsen Sie über Nacht bei 37 ° C, bei 225 u/min schütteln.
- Vor dem Ausschalten des AutoMCS Gerätes ändern Sie die Puffer laufen und Waschen des Herstellers ausführen und waschen Puffer (siehe Tabelle der Materialien). Wählen Sie "Jetzt waschen", dann "Spülen" und "Run". Wenn Sie fertig sind, sicher sein, dass es 70 % Ethanol in der Dekontamination-Linie, dann drücken Sie die "Power-Symbol" in der oberen rechten Ecke des Bildschirms und wählen Sie "Ja".
- Wenn das Herunterfahren des Systems beendet ist (die Flaschen werden lila), schalten Sie die Maschine.
- Verwenden Sie am nächsten Tag, die Übernachtung Kultur, Gefrierschrank Aktien mit ca. 1 x 1011Zellen pro Vial in LB mit 15 % Glycerin machen. Zusätzlich oder alternativ verwenden diese Kultur im nächsten Schritt: zusätzliche negative sortieren (Abschnitt 1.2) oder die erste Runde der Positivsortierung (Abschnitt 1.3).
- 500 mL Luria Brühe Miller (LB) mit geeigneten Antibiotika mit ca. 1 x 1011Zellen der verschiedenen bakteriellen anzeigen sortieren Bibliothek zu impfen (siehe Tabelle der Materialien).
- Negativen Sortierung Bindemittel voraussichtlich Kreuzreaktion mit anderen Zielen des Interesses zu entfernen
Hinweis: Abschnitt 1.2 kann übersprungen werden, wenn keine weiteren negativen sortieren benötigt oder gewünscht ist. In diesem Fall fahren Sie mit Abschnitt 1.3. Restliche Bindung an alle unerwünschten Ziele durch FACS beurteilt werden kann, wie in Abschnitt 2: FACS Analyse der Sortierung Runden.- Wiederholen Sie für negative Sortierung gegen ein bestimmtes Protein-Ziel, wie ein ähnliches Protein mit Potenzial, auch binden, um die gefundenen Peptide 1,15, Wachstum und Induktion Schritte 1.1.1 - 1.1.3, angefangen bei einem gefrorenen bestand von Streptavidin-abgereicherte Bibliothek aus Schritt 1.1.15 oder die Nacht Kultur aus Schritt 1.1.12 (mit OD600 zu schätzen und impfen mit 1 x 1011 Zellen).
- Übertragung von Zellen auf Microcentrifuge Tube(n) und Spin bei 6.000 x g für 5 min bei RT oder 4 ° C. Entfernen Sie überstand. Aufschwemmen Sie Zelle Pellet in 1 mL PBS mit 600 nM biotinylierte kreuzreaktiven Protein Ziel. Bei 4 ° C für 45 Minuten auf einer rotierenden Plattform inkubieren.
Hinweis: 600 nM ist eine empfohlene Ausgangspunkt Konzentration, die geändert werden kann, wenn ein bekannter Vorteil beobachtet wird. Biotinylierungen der Protein-Ziel erreicht werden kann und quantifizierte mit den Reagenzien in der Tabelle der Materialien vorgeschlagen. - In der Zwischenzeit, 100 µL Streptavidin beschichteten Perlen (ca. 1 x 109 Perlen) in 1 mL PBS-B und Zentrifuge für 5 min bei ≥ 5.000 x g (bei RT) waschen. Legen Sie Rohr in eine Bank Top magnetische Partikelabscheider und sorgfältig entfernen überstand, das Pellet zu vermeiden.
- Zentrifugieren Sie die Ziel-gebundenen Zellen bei 6.000 x g für 5 min (RT oder 4° C), entfernen Sie überstand und Aufschwemmen der Zelle Pellet in 1 mL PBS-B. Aufzuwirbeln Sie Perlen im gesamten Volumen der gewaschenen Zellen gebunden zu kreuzreaktiven Protein Ziel. Bei 4° C auf einer rotierenden Plattform für 30 min inkubieren.
- Legen Sie die Probe auf Eis und führen Sie die Schritte 1.1.7 - 1.1.15 für eine negative Art entsprechende Gefrierschrank Aktien machen. Abschnitt 1.3 für eine positive Art, wenn alle gewünschten negativen Sortierung erreicht worden ist, oder wiederholen Abschnitt 1.2 auf negative Art gegen eine weitere potenzielle kreuzreaktiven Protein weiter.
- Runde 1 Positive Art
Hinweis: Runde 1 Positivsortierung gegen ein bestimmtes Protein Ziel ist ähnlich wie eine negative Art gegen eine zielgruppenspezifische Sortierung (siehe 1.2), außer, dass der Wulst-haltigen, positive Bruch beibehalten wird.- Impfen Sie 500 mL LB Cm25 mit ca. 1 x 1011Zellen der verschiedenen bakteriellen anzeigen sortieren Bibliothek wurden Streptavidin-erschöpft und über Nacht (von Schritt 1.1.12) oder (aus Schritt 1.1.15) eingefroren und aufgetaut auf Eis angebaut, oder das schon Weitere erschöpft von Bindemitteln zu anderen kreuzreaktiven Protein-Targets (aus Schritt 1.2.5), wie gewünscht.
- Inkubation bei 37 ° C mit Schütteln bei 225 u/min, bis die Kultur eines OD600 von 0,5 erreicht - 0,55. Induzieren Sie Peptid Ausdruck mit 0,04 % w/V L-Arabinose durch Verdünnung eine 4 % Lager 1: 100, schütteln bei 225 u/min für 45 min bei 37 ° C.
- Ort induzierten Kultur auf Eis. Zentrifugieren Sie ca. 2 x 1011 Zellen bei 6.000 x g für 20 min bei 4 ° C. Entfernen Sie überstand und Aufschwemmen Sie Zellen in 1,5 mL PBS durch sanft schütteln (per hand oder kurz schütteln bei ≤ 225 u/min, 4° C).
- Übertragung von Zellen auf Microcentrifuge Tube(n) und Spin bei 6.000 x g für 5 min bei RT oder 4 ° C. Entfernen Sie überstand.
- Aufschwemmen Sie Zelle Pellet in 1 mL PBS mit 600 nM biotinylierte Protein Ziel von Interesse. Bei 4 ° C für 45 Minuten auf einer rotierenden Plattform inkubieren.
Hinweis: 600 nM ist eine empfohlene Ausgangspunkt Konzentration, die geändert werden kann, wenn ein bekannter Vorteil beobachtet wird. Biotinylierungen der Protein-Ziel erreicht werden kann und quantifizierte mit den Reagenzien in der Tabelle der Materialien vorgeschlagen. - In der Zwischenzeit, 100 µL Streptavidin beschichteten Perlen (ca. 1 x 109 Perlen) in 1 mL PBS-B und Zentrifuge für 5 min bei ≥ 5.000 x g (bei RT) waschen. Legen Sie Rohr in einem Bank-Top Magnetpartikel-Separator, und entfernen Sie vorsichtig des Überstands, das Pellet zu vermeiden.
- Bei der Inkubation mit Ziel abgeschlossen ist, Zentrifugieren der Ziel-gebundenen Zellen bei 6.000 x g für 5 min (RT oder 4° C), um alle ungebundenen Zielprotein zu entfernen. Entfernen Sie überstand und Aufschwemmen der Zelle Pellet in 1 mL PBS-B. Aufzuwirbeln Sie Perlen im gesamten Volumen der gewaschenen Zellen gebunden an Eiweiß Ziel. Inkubieren Sie bei 4° C auf einer rotierenden Plattform für 30 min auf Eis.
- Schalten Sie ein AutoMCS Instrument, um das System zu initialisieren. Prime Linien mithilfe des Herstellers ausführen und waschen Puffer (siehe Tabelle der Materialien) durch die Auswahl "Wash Now" am unteren Rand des Bildschirms im Menü Trennung dann "Spülen" und "Run".
- Entlüften Sie das System mit PBS-B, mit PBS-B als Waschpuffer und Puffer läuft in den nächsten Schritten. Wählen Sie Menü Trennung der oberen Navigationsleiste und waschen jetzt. Wählen Sie spülen und Run muss das System mit frischem PBS-B.
- Übertragen Sie Zelle und Perle Mischung aus Inkubationsschritt oben auf eine 15 mL konische Rohr, das Rohr mit einer zusätzlichen 500 µl PBS-B spülen und Bündelung der Wäsche mit der Probe. Diese Röhre in das Musterfeld und leer 15 mL konische Röhrchen in den Schlitzen in positive und negative Selektion des Regales vorgekühlt (4 ° C) (siehe Tabelle der Materialien). Instrumentenplattform Rack aufsetzen.
- Weisen Sie eine Trennung-Programm für jede Probe auf dem Gestell (bis zu 5 Proben kann in einem Arbeitsgang sortiert werden). Fügen Sie einen "Spülen" Schritt zwischen jeder Probe und nach der letzten Probe. Wählen Sie "Ausführen", um die Zelle Trennung zu starten. Wählen Sie "OK" oder "Weiter" bestätigen Sie, dass genügend Puffer verfügbar ist.
Hinweis: Das "Posselds"-Trennung-Programm eignet sich gut für negative sortieren und positive Sortierung Runde 1 und "Posseld" wird bevorzugt für die Sortierung runden 2-4, aber beide Programme erfolgreich verwendet worden für alle Negative und positive Sortierung 14 runden , 15 und andere Programme können für eine bestimmte Anwendung besser erweisen (beschrieben in Diskussion). - Wenn das Programm abgeschlossen ist, entfernen Sie den Ständer und behalten Sie den entsprechenden Bruchteil. Hier, für eine positive Art behalten Sie positive Brüche (mit Zellen und Perlen). Auf Eis legen.
- Vor dem Ausschalten des AutoMCS Gerätes ändern Sie die Puffer laufen und Waschen des Herstellers ausführen und waschen Puffer (siehe Tabelle der Materialien). Wählen Sie "Jetzt waschen", dann "Spülen" und "Run". Wenn Sie fertig sind, sicher sein, dass es 70 % Ethanol in der Dekontamination-Linie, dann drücken Sie die "Power-Symbol" in der oberen rechten Ecke des Bildschirms und wählen Sie "Ja".
- Wenn das Herunterfahren des Systems beendet ist (die Flaschen werden lila), schalten Sie die Maschine.
- Verwenden Sie die positive Art Bruch in seiner Gesamtheit 1 L LB Cm25 mit 0,2 % w/V D-Glucose zu impfen. Wachsen Sie über Nacht bei 37 ° C, bei 225 u/min schütteln.
- Am nächsten Tag, verwenden Sie die Übernachtung Kultur in LB, enthält 15 % Glycerin zu Gefrierschrank Aktien und/oder weiter positive Sortierung Runde 2 in Abschnitt 1.4.
- Weitere Positive Sortierung runden
Hinweis: In der Regel werden vier Sortierung runden empfohlen, obwohl drei Sortierung Runden in der Regel ausreichend 14,15 sind. Beim stoppen Sortierung unterstützt wird beschrieben die FACS-Analyse der Sortierung Runden in Abschnitt 2. Konzentrationen von Ziel- und magnetischer Wulst Volumen verringern mit jeder nachfolgenden positive Sortierung Runde.- Mit der Nacht Kultur aus der Vorrunde Sortierung (oder einen gefrorenen Zelle bestand aus dieser Runde), 5 mL LB Cm25 mit 100 µl Zellen impfen (01:50 Verdünnung). Inkubation bei 37° C mit Schütteln bei 225 u/min, bis die Kultur eines OD600 von 0,5 erreicht - 0,55.
- Induzieren Sie Peptid Ausdruck mit 0,04 % w/V L-Arabinose, schütteln bei 225 u/min für 45 min bei 37° C. Ort induzierte Zellen auf Eis.
Hinweis: Diese induzierten Kultur kann auch beurteilen, Bindungsaffinität und Spezifität für die Sortierung Runde, die, der es abstammt, wie in Abschnitt 2 unten beschrieben verwendet werden. - Zentrifugieren Sie 1 x 10-8 -Zellen bei 6.000 x g für 5 min bei RT oder 4 ° C. Entfernen Sie überstand und Aufschwemmen Zelle Pellet in 50 µL PBS mit halber Konzentration von biotinylierte Protein Ziel von Interesse für die Vorrunde der Sortierung verwendet (also 300 nM für Runde 2, 150 nM für Runde 3 und 75 nM für Runde 4 aus unseren vorgeschlagenen (Ausgangspunkt). 45 min auf Eis (oder bei 4 ° C einer rotierenden Plattform) inkubieren.
- Währenddessen waschen Streptavidin beschichteten Perlen (15 µL für Runde 2, 8 µL für Runde 3 und 4 µL für Runde 4) in 1 mL PBS-B und Zentrifuge für 5 min bei ≥ 5.000 x g (bei RT). Legen Sie Rohr in eine Bank Top magnetische Partikelabscheider und sorgfältig entfernen überstand, das Pellet zu vermeiden. Aufschwemmen Perlen in 50 µL PBS-B.
- Nach 45 min Inkubation mit Ziel im Schritt 1.4.3, Zentrifugieren der Zellen bei 6.000 x g für 5 min bei RT oder 4° C, überstand entfernen und die Zellen in den 50 µL gewaschen Perlen aus 1.4.4 Aufschwemmen. Halten Sie die Probe auf Eis und führen Sie die Schritte 1.3.7 - 1.3.13. für eine positive Art.
- Eine 5 mL-Kultur der LB Cm25 ergänzt mit 0,2 % w/V D-Glucose mit dem gesamten positiven, Perle-haltigen Bruch aus der Art zu impfen.
- Am nächsten Tag, verwenden Sie die Übernachtung Kultur Gefrierschrank Bestände in LB, enthält 15 % Glycerin und/oder den nächsten Positivsortierung Runde weiterhin, zu Schritt 1.4.1 zurückkehren.
Hinweis: Die induzierte Kultur von 1.4.2 Bindungsaffinität und Spezifität bewerten kann wie in Abschnitt 2 beschrieben mit Hilfe bestimmen, wann man aufhören sortieren. Wenn zwei Sortierung Runden in Folge zeigen ähnliche Bindungsaffinität oder eine spätere Runde zeigt Bindungsaffinität für das Ziel von Interesse verringert, dadurch ein wünschenswertes platziert um Sortierung zu stoppen. Nach der letzten Runde Rückkehr zu Schritt 1.4.1 aber halt auf 1.4.2.
(2) FACS Analyse des Sortierens runden
- Verwendung der induzierten Zellen aus Schritt 1.4.2 für jede Sortierung Runde oder identische Kulturen, 5 µL induzierte Zellen hinzufügen 25 µL der einzelnen der folgenden Lösungen für verbindliche Beurteilung vorbereitet (siehe auch Tabelle Materialien): PBS allein; PBS mit 150 nM YPet Mona (YPet 12,13, Positivkontrolle für Ausdruck), falls vorhanden; 900 nM Protein Ziel konjugiert zu einem Fluoreszenzfarbstoff, z. B. Amin Reaktivfarbstoffen mit Emission/Anregung bei 493 nm⁄518 nm (Ziel-488); 900 nM kreuzreaktiven Protein-Zielen verwendet für die negative Sortierung beschriftet mit der gleichen Fluoreszenzfarbstoff (Cross-Reactive Protein-488); und 900 nM Streptavidin-R-Phycoerythrin (SAPE). Inkubieren Sie für 45 min auf Eis.
Hinweis: Wenn weiterhin direkt von Schritt 1.4.2, dieser Schritt wird immer getan werden am Tag nach der Biopanning für diese Runde abgeschlossen wurde, da die unten ausgewählten Bibliothek über Nacht gewachsen war dann subkultiviert und induzierte am folgenden Tag. Kulturen angebaut und induziert in ähnlicher Weise aus gefrorenen Sortierung Runde Beständen können auch verwendet werden; in diesem Fall können alle Runden getestet und in einem einzigen Experiment verglichen. - Zentrifuge Zellen bei 6.000 x g für 5 min bei RT oder 4° C. Entfernen Sie überstand und lagern Sie Proben auf Eis.
Hinweis: Die Zelle Pellets können auf Eis gelagert werden, bis alle Proben zur Analyse bereit. - Schalten Sie die FACS-Instrument, öffnen Sie die Software, Inbetriebnahme des Systems und kalibrieren Sie Gerät nach Herstellerangaben Anweisungen 43,44.
- Innerhalb der FACS-Software klicken Sie auf "Administrator" und klicken Sie auf den gewünschten Ordner oder erstellen Sie einen "neuen Ordner". Klicken Sie auf "Neues Experiment" Symbol oben links auf dem Bildschirm. Klicken Sie rechts zum "Experiment umbenennen". Im Rahmen dieses Experiments klicken Sie auf "Globale Arbeitsblätter".
- Klicken Sie auf das Symbol "Dot-Plot" und klicken Sie auf dem globalen Arbeitsblatt Arbeitsblatt zu diesem Streudiagramm erstellen. Im Dot-Plot-Diagramm selbst, klicken Sie unter Wählen Sie die "Inspektor" Symbol oben links auf dem Bildschirm und die Achsen zur Biexponential Anzeige durch Auswahl die Kästchen neben "Y-Achse" und "X-Achse" im geöffneten Fenster einstellen. Schließen Sie das Biexponential Display-Fenster durch Klicken auf das X.
- Erstellen Sie eine "neue Röhre" durch Klicken auf das Symbol oben links auf dem Bildschirm und benennen Sie die Probe mit entsprechenden Informationen durch Rechtsklick das Probe-Symbol unter "globale Arbeitsblatt" und wählen Sie "umbenennen". Das Exemplar zu erweitern, indem Sie auf das Pluszeichen (+) Zeichen, dann klicken Sie doppelt auf das Symbol, Rohr, rechten Maustaste darauf, und erneut wählen Sie "umbenennen" um die Probe zu beschreiben.
- Verwenden Sie dieses Dot-Plot-Diagramm mit Standard SSC-A Vs FSC-A eine negative Kontrollprobe in PBS allein ausführen durch Doppelklick auf das Rohr oder den Pfeil daneben auswählen. Kurz vor Ausführen des Beispiels, Aufschwemmen der Zelle Pellet in 500 µL Eis kalt FACS laufen Puffer und Transfer Probe zu einem FACS Rohr (siehe Tabelle der Materialien), von pipettieren und streichen den Schlauch gut mischen. Legen Sie die resuspendierte Zellen auf die Injektion Probenröhrchen (SIT) des Instruments. Presse "erwerben Daten" mit "Events to Display" bei 30.000-50.000 und "Ereignisse zu Datensatz" 10.000, Durchfluss bei niedrigen (starten; bei Bedarf zu erhöhen), und "Sitzen bündig" ausgewählt.
Hinweis: Geeignete Geschwindigkeit (niedrig, Mittel oder hoch) ist die Geschwindigkeit, mit der die Anzahl der Ereignisse/s etwa zwischen 200 und 2000. Nutzen Sie dieses Angebot für alle Schritte. - Passen Sie beim Erwerb Photomultiplier (PMT) Rohr Spannungsgrenze bei Bedarf wählen Sie die "Schwelle" Registerkarte "klicken Sie auf an und ändern Sie den"Wert". Passen Sie Spannung für vorwärts- und seitliche Streuung (FSC und SSC an), so dass negative Kontrollzellen in PBS allein etwas unterhalb der Mitte fallen.
Hinweis: Zusätzliche Parameter (z. B. SSC) können im Register "Schwelle" über das Symbol "hinzufügen" hinzugefügt werden. In der Regel PMT Spannungen von etwa 700 V bis 1000V für SSC und FSC arbeiten gut für E. Coli. - Wenn die Probe richtig liest, einstellen der Durchflussmenge anzeigen 200-2000 Veranstaltungen/s ein und drücken "Record Data". Wenn Sie fertig sind aufzeichnen, SIT Tube entnehmen und auf Eis legen. Wählen Sie das Symbol "Polygon Tor" und verwenden Sie die Maus ein Tor um den Großteil der Zellpopulation in das Streudiagramm zu ziehen. Rechtsklick auf Dot-Plot, und wählen Sie "Zeigen Bevölkerung Hierarchie".
- Verwenden Sie dieses Tor "P1" als Eltern durch Klicken auf "P1" in der Bevölkerung-Hierarchie. Erstellen Sie einen neuen Dot-Plot nach Schritt 2.5. Jede Achse einen Rechtsklick und ändern der Y-Achse FITC-a und der X-Achse in FSC-A. Tor der negativen Kontrolle (PBS allein) verwenden das Symbol "Polygon Tor" mit dem Tor so eng wie möglich um die obere und die linke Seite der Bevölkerung.
Hinweis: Überprüfen Sie die Bevölkerung Hierarchie um sicherzustellen, dass das P1-Tor ein Elternteil von der P2-Tor ist. Wenn es nicht ist, erscheint er im Einklang mit dem P1-Tor und "Alle Termine" werden das übergeordnete Element; das Tor zu löschen und neu erstellen nach dem Schritt 2.10. - Wählen Sie "P2" in der Bevölkerung-Hierarchie, Rechtsklick und "invert Tor". Klicken Sie rechts auf den Dot-Plot mit P2-Tor, und wählen Sie "Populationen zeigen". Wählen Sie die Bevölkerungen "P2" und "Nicht P2" für die Anzeige (weniger als 1 % der Bevölkerung sollte draußen vor dem Tor fallen).
- Die Dot-Plot mit P2 Tor einen Rechtsklick und wählen Sie "Create Statistics View". Statistik-Ansicht-Fenster einen Rechtsklick und wählen Sie "Bearbeiten-Statistik-Ansicht". Klicken Sie auf die Registerkarte "Bevölkerung" und hinzufügen oder Entfernen von Bevölkerungsgruppen, wie gewünscht, einschließlich der Grundgesamtheit, P1 (P2, und nicht P2 bereits angezeigt werden sollen). Klicken Sie auf der Registerkarte "Statistik" und "FITC-A Median" und andere Statistiken von Interesse. Schließen Sie das Fenster mit OK.
- Erstellen Sie eine ähnliche Dot Plot Diagramm für PE-A Vs FSC-A und Tor mit PBS allein Probe (früheren Grundstücke können kopiert und verändert werden). Mithilfe dieser Tabelle alle Proben (einschließlich der Negativkontrolle Zellen inkubiert mit jedes Ziel Fluorophore beschriftet) auf diese Weise etwa 10.000 Veranstaltungen für jede Aufnahme ausführen.
Hinweis: Die Zellen, fallen draußen vor dem Tor nach der Inkubation mit dem Target-488-Protein, die YPet positive Kontrolle usw. sind Bindemittel, das Protein, und der Wert von "Nicht PX" (wobei X die Anzahl der geschlossenen Bevölkerung für das Diagramm ist) sollte als % gebunden aufgenommen werden. Bei SAPE als Negativkontrolle Verwendung des PE-A Vs FSC-A Plots. Der Median Fluoreszenzintensität (MFI) P1 sollten auch gespeichert werden, da dies Auskunft über % verbindlich gibt, um das Ausmaß der Bindung in relativer Hinsicht zeigen und robuster im Beisein von Ausreißern 45 ist. Mediane Fluoreszenzintensität kann normalisierte (nMFI) dividiert MFI jedes Peptid von MFI ein Gerüst nur negativ-Kontrolle (mit keine N-terminale Peptid) oder ein Klon mit einer Peptidsequenz, die nicht das Ziel von Interesse, bindet nach Inkubation mit dem gleichen Ziel auf der gleichen Fluorophor 14konjugiert. Wenn diese negativ-Kontrolle-Zellen nicht verfügbar sind, können uninduced Zellen inkubiert mit dem gleichen Ziel und mit der gleichen Fluorophore beschriftet auch für die Normalisierung verwendet werden.
3. die Sequenzanalyse von potentiellen Kandidaten und Bewertung von verbindlichen Affinität
- Peptid-Sequenz-Bestimmung
- Auswählen und Dutzende oder Hunderte von Bakterienkolonien aus der letzten Runde(n) der Biopanning-Sequenz (in der Regel runden 3 und/oder 4 sind ausreichend 14).
- Analysieren Sie die Sequenz Datenanalyse mit der Makro-Datei, die wir entwickelt haben (siehe Begleitinformationen für Code, "Sub eCPX_Sequencing") speziell die Sequenzen generiert aus der bakteriellen 15mer Bibliothek in der Tabelle der aufgeführt Biopanning Materialien. Verwenden der Tabellenkalkulations-Software, die in der Tabelle der Materialien oder andere bevorzugte kompatibler Software aufgeführt.
Hinweis: Eine Reihe von Tools sind online verfügbar, die auch in der DNA-Sequenz-Analyse 47Beihilfen können. Falls gewünscht, verwenden Sie eine andere etablierte Methode für Sequenzanalyse und überspringen Sie Schritt 3.1.3.- Laden Sie die Menge der Sequenzdateien (.seq Dateien, Text-Dokumente auch Arbeit) und in einen neuen Ordner zu extrahieren. Bei Bedarf verschieben Sie oder kopieren Sie den Ordner auf der Festplatte (im Gegensatz zu einem Ordner im Netzwerk) für verbesserte Geschwindigkeit.
- Öffnen Sie ein neues Arbeitsblattfenster. Stellen Sie sicher, aktivieren Sie Makros und alle Features, wenn diese Nachrichten auftauchen.
Hinweis: Die Schritte 3 bis 6 unter müssen nur beim ersten Starten abgeschlossen werden, die das Makro verwendet wird. Für die spätere Analyse, einfach "das Makro ausführen" ab Schritt 7. - Kopieren Sie den gesamten Inhalt des Makros in der Datei "Sub eCPX_Sequencing" gefunden.
Hinweis: Die aktuelle Version mit flankierenden Sequenzen für die 15-Mer Bibliothek aufgeführt in der Tabelle der Materialien eingerichtet ist und die Aminosäuren untereinander zu übersetzen. Zusätzliche Sequenzen können durch Kopieren 5' flankierenden Sequenzen in Spalte A und 3'-flankierenden Sequenzen in Spalte B der Tabelle, bis zu 10 Sequenzen für jeden vor dem Ausführen des Makros eingegeben werden. - Wählen Sie in der leere Kalkulationstabellendatei die Registerkarte "Ansicht", dann klicken Sie doppelt auf "Makros". Wählen Sie den personal.xlb-Ordner. Wenn dies nicht verfügbar ist, zeichnen Sie ein Makro zuerst um diese einzublenden.
- Klicken Sie auf "erstellen" oder "betreten", je nachdem, was hervorgehoben werden können. Das gesamte Makro in das Modul einfügen.
- Klicken Sie auf das Speichern-Symbol oder gehen Sie auf Datei, speichern. Beenden Sie das Modul. Wenn ein Fenster erscheint, drücken Sie "OK".
- Führen Sie das Makro: gehen Sie zu dem Ordner, wo die desired.seq-Dateien befinden. Klicken Sie auf die Leiste neben dem Symbol des Ordners an der Spitze, den Speicherort des Ordners anzuzeigen. Kopieren Sie sie.
- Gehen Sie zum Fenster neue Tabelle. Wählen Sie die Registerkarte "Ansicht", dann klicken Sie doppelt auf Makros. Wählen Sie aus "eCPX_Sequencing" aus der Liste und klicken Sie auf "ausführen".
- Im Feld erscheint, fügen Sie den Speicherort der Datei kopiert Schritt 7, und drücken Sie die Eingabetaste. Das Makro sollte beginnen, durchlaufen die Sequenzen und organisieren sie in Tabellen auf verschiedenen Blättern.
- Verwenden Sie das Tabellenblatt "Zusammenfassung", um festzustellen, ob es notwendig sein wird, Trace-Dateien auf Fehler zu überprüfen und Korrekturen vornehmen, manuell (wenn Sequenzen enthalten "X" oder nicht übersetzt werden konnte).
Hinweis: Die "Übersichtstabelle" zeigt die übersetzten Peptid-Sequenzen, sortiert nach Aminosäure-Sequenz (von A bis Z), es sei denn, ein Fehler der Sequenzierung Übersetzung verhindert. Ein "X" kennzeichnet eine einzelne Aminosäure, die nicht ermittelt werden konnte.
- Sobald die Sequenzen übersetzt werden, organisiert, und Sequenzierung Fehler korrigiert, Prüfliste sortierte Peptid-Sequenz auf dem Blatt "Übersichtstabelle" für keine sich wiederholenden Sequenzen.
- Wachsen über Nacht Kulturen für sequenzierte Kolonien von Interesse (einschließlich der Wiederholungen und/oder Peptide mit spürbaren Trends im Mindestfall) in 5 mL LB Cm25 bei 37 ° C, bei 225 u/min, schütteln und Gefrierschrank Aktien am nächsten Tag in LB, enthält 15 % Glycerin, wie in Schritt speichern 1.4.7. Testen Sie die Peptide mit sich wiederholenden Sequenzen für verbindliche Affinität und Spezifität der FACS Methoden beschrieben Abschnitt 3.3 und FASTA-Format von der Sequenzliste (vollständige Liste und Wiederholungen nur) verwenden, um die Sequenzen mit bevorzugten Alignment auszurichten und Analyseprogramme, wie in Abschnitt 3.2.
- Sequenzalignment Clustal Omega, Kalign und ähnliche Programme verwenden
- Kopieren Sie Sequenzen im FASTA-Format aus der FASTA-Tabelle des Makros, oder ansonsten erstellen Sie eine Liste der FASTA-Dateien aus dem Sequenzen (z. B. aus Schritt 3.1.3) ausgerichtet werden.
Hinweis: Spalte A enthält die FASTA-Dateien in numerischer Reihenfolge von "Seq #" während Spalte B sie sortiert nach "AA-Sequenz" aus dem Tabellenblatt "Zusammenfassung" wird angezeigt. Die Liste der Peptid-Sequenzen, die Aminosäure-Sequenz sortiert wird unbrauchbare Sequenzen angezeigt, an der Spitze, wie leere Vektor (als "# leer") und diese Sequenzen in der weder die 5' oder 3' Suchkriterien gefunden wurden (als "# Wert"). Mit dieser Funktion können für einfache Aktualisierung oder Entfernung von diesen Sequenzen aus der Analyse. - Öffnen Sie Clustal Omega48 oder Kalign49 Software zu, indem Sie auf der Webseite 50,51, oder andere bevorzugte Software für Sequenzalignment. Kopieren Sie und fügen Sie der FASTA Sequenzliste ein und geben sie in das Feld unten "Sequenzen in einem beliebigen unterstützten Format". Ändern Sie "Lücke offen Strafe" zu 30 unter "weitere Optionen" in Kalign (Dies ist nicht möglich in Clustal Omega), aber ansonsten behalten Sie Standardeinstellungen bei, bevor Sie auf "Absenden".
- Sequenzalignment mit Jalview 52,53 Software direkt in Clustal Omega und Kalign online-Software von der Registerkarte "Ergebnis Zusammenfassung" über die Ausrichtung auswählen und dann auf das Symbol "Jalview" zu analysieren.
Hinweis: Falls gewünscht, oder für die Analyse der Sequenz Achsen durch Alternativprogramme erzeugt, 54 die Software separat herunterladen und kopieren und einfügen die Ausrichtung in das Analysefenster, das geöffnet wird, indem Sie auf "Datei", "Input Alignment", und " von Textbox". Dies bietet die Möglichkeit, leicht feststellen, ob eine Konsensussequenz in die Eingabesequenz Ausrichtung vorhanden ist.
- Kopieren Sie Sequenzen im FASTA-Format aus der FASTA-Tabelle des Makros, oder ansonsten erstellen Sie eine Liste der FASTA-Dateien aus dem Sequenzen (z. B. aus Schritt 3.1.3) ausgerichtet werden.
- Vergleich von Bindungsaffinität und Spezifität mit FACS
- 5 mL LB Cm25 ergänzt mit 0,2 % w/V D-Glucose mit jeder einzelnen Isolat von Interesse (aus Schritt 3.1.4 und/oder Kolonien von Interesse aus weiteren Sequenzanalyse in Abschnitt 3.2, etc.) und eine entsprechende negativ-Kontrolle (Anzeige zu impfen Gerüst nur oder ein Peptid, die Ziel, nicht bindend ist, wie beschrieben in der Anmerkung zum Schritt 2.13). Wachsen Sie über Nacht bei 37 ° C, bei 225 u/min schütteln.
- Verwenden Sie die Übernachtung Kulturen 3 mL LB Cm25 mit 60 µl Zellen impfen (01:50 Verdünnung). Inkubation bei 37° C mit Schütteln bei 225 u/min, bis die Kultur eines OD600 von 0,5 erreicht - 0,55. Induzieren Peptid Ausdruck mit 0,04 % w/V L-Arabinose plus 2 mM EDTA (zur Erleichterung der Peptid-Display 42), schütteln bei 225 u/min für 45 min bei 37 ° C.
- Ort induzierte Kulturen auf Eis. 5 µL Zellen hinzufügen für jedes Isolat 25 µL PBS allein oder mit PBS: 150 nM YPet 12,13 (Positivkontrolle für Ausdruck), falls vorhanden; 250 nM Target-488; 250 nM kreuzreaktiven benutzt-488; und 250 nM SAPE (siehe 2.1 für mehr Detail). Inkubieren Sie für 45 min auf Eis.
- Zentrifuge Zellen bei 6.000 x g für 5 min bei RT oder 4° C. Entfernen Sie vorsichtig überstand Flüssigkeit von der gegenüberliegenden Seite des Pelletofens ziehen. Speichern Sie Zelle Pellets auf Eis, bis alle Proben und sind bereit für die Analyse.
- Aufschwemmen jede Zelle Pellet in 500 µL Eis kalt FACS Puffer, ausgeführt von pipettieren und streichen den Schlauch kurz vor lesen mit einem Durchflusszytometer gut mischen (siehe Tabelle der Materialien).
Anmerkung: Siehe Schritt 2.13 merkt weitere Erläuterungen gating und Berechnung der nMFI Bindungsaffinität und Spezifität zu vergleichen. Non-Mappen oder unspezifische Bindemittel können nun von der weiteren Analyse entfernt werden, und die höchste Affinität Bindemittel sollte von Sequenzalignment nach Abschnitt 3.2, weitere Trends suchen, die vielleicht in der ersten sequenziert verpasst neu bewertet werden Bevölkerung. Abschnitten 3.2 und 3.3 wiederholt werden kann, je nach Bedarf als neue Trends und Konsensus-Sequenzen sind vermerkt. Diese Analyse kann mehr zufällige Sequenzen enthalten, wenn Trends nicht gesehen werden.
Ergebnisse
Mit dem Einsatz von automatisierten magnetisch aktivierte Zelle sortieren (AutoMCS) anstatt manuelle magnetischen Zellsortierung sind falsche negative Kandidaten während Biopanning bakterielle Anzeige Bibliotheken 9,14deutlich reduziert. Negativen sortieren Schritte werden auch zugesetzt werden je nach Bedarf, unspezifische Bindemittel auf das Ziel des Interesses zu reduzieren, und es wird empfohlen, mindestens um eine negative Art gegen die magnetische hinzuzufügen sich vorne Perlen. Diese negative Selektion gegen die magnetische Beads selbst wurde abgeschlossen, bevor vier Runden des Biopanning der gewählten bakterielle Bibliothek für schützende Antigen (PA) Bindemittel, anzeigen, wie in Abbildung 2gezeigt, bevor zusätzliche negative Auslese gegen Rivax und vier Runden der positiven Selektion für Abrax, wie in Abbildung 3dargestellt. Die hier verwendeten Perlen sind konjugiert zu Streptavidin für die Erfassung von biotinylierte Proteine, so dass unbeabsichtigte Trennung der Peptide binden an Streptavidin selbst ein Anliegen ist und wird mit FACS mit überwacht Streptavidin-konjugiert, Phycoerythrin (Abbildung 3 , SAPE). Zumindest sollte Bindung Streptavidin für jede viel versprechende einzelnen Kandidaten getestet werden, bei der Bindung an das Zielprotein Beurteilung. Die prozentuale Bindung und nMFI für SAPE sollte niedrige Werte in allen Sortierung Runden. Die Bindung an Streptavidin kann etwas mit jeder Sortierung Runde erhöhen wie in Abbildung 3, da die Bevölkerung immer wieder die Streptavidin beschichteten magnetische Beads nach jeder Sortierung Runde ausgesetzt ist. Wenn die Ebene Hintergrund Streptavidin Bindung an nachgelagerte Analyse problematisch wird, konnte weitere negative Sortierung gegen magnetische Beads im Anschluss an die positive Sortierung Runde durchgeführt werden in dem Streptavidin Bindung Bevölkerung in erhöht Bestellung, downstream Screening der Fehlalarme zu reduzieren. Wenn Proteine oder Materialien zur Verfügung stehen speziell mit nachgeschalteter Nutzung des Peptids für eine bestimmte Anwendung kollidieren könnten, oder sollen die Kreuzreaktion mit Affinität Reagenzien für das Ziel aufgrund von strukturellen und/oder Sequenzähnlichkeit, weitere negative Sortierung gegen das Protein oder Material empfohlen. Ein Beispiel ist eine negative Sortierung gegen Rivax vor Isolation von Abrax Bindung Peptide, aufgrund der strukturellen Ähnlichkeit und Sequenzhomologie der beiden Proteine 1,15. Wiederum kann kreuzreaktiven binden an Proteine für Negative sortieren Schritte bei der Analyse der Sortierung Runden mit FACS (Abbildung 3Rivax-488) überwacht werden. Im besten Fall sind Prozent Bindung und nMFI niedrig, wie in diesem Beispiel.
Wenn verfügbar, kann eine feste Positivkontrolle Peptid, wie das P2X-Peptid am C-Terminus des Display Gerüstes produziert durch die bakterielle Anzeigebibliothek verwendet in die repräsentativen Ergebnisse hier helfen festzustellen, ob Fehlen verbindlicher Affinität in der FACS-Assay ist wegen des Mangels an Ausdruck der Anzeige Gerüst selbst, anstatt Mangel an Affinität von den Peptide(s) für das Ziel. In Abbildung 2 und Abbildung 3, YPet Mona Bindung an diese P2X Peptid wird überwacht und zeigt, dass die ersten Runden des Sortierens schlecht das Gerüst zum Ausdruck bringen. Dies dürfte vor allem aufgrund der hohen Frequenz des Stop-Codons in N-terminale Peptide in der zufälligen Bibliothek, was bedeutet, dass das Gerüst selbst nicht produziert wurde. Andere Effekte können zusätzlich beitragen, z. B. dem Vorhandensein von Peptiden mit unerwarteten toxischen Wirkungen auf E. Colioder verminderte Wachstum oder Peptid Display-Preise aus anderen Gründen (z. B. Mutationen im Bakteriengenom). Zugabe von EDTA während der Induktion Ausdruck während dieser ersten Runden des Sortierens 42verbessern kann, aber noch nicht getestet. Bindung an YPet Mona in jeder Biopanning ist durch Runde 3 Bevölkerung erheblich verbessert. Abbildung 2 , Abbildung 3vergleicht, ergibt sich auch, dass Ausdruck Ebene verbessert werden kann, indem man Induktionszeit bis 90 min (wie in Abbildung 2YPet Mona) anstatt 45 min (wie in Abbildung 3YPet Mona), Obwohl 45 min in der Regel ausreichend ist. Beachten Sie, dass die nMFI zur YPet Mona zur YPet Mona verbindliche Ebene der Negativkontrolle Zellen normalisiert wird, also Werte nahe 1,0 typisch sind, wenn Ausdruck akzeptabel ist. Normalisierung der YPet Mona MFI zu PBS allein MFI aus derselben Probe ist auch eine gültige Möglichkeit, relative Ausdruck Niveaus zu vergleichen, aber viel höhere Werte gibt (Vergleiche Abbildung 2 und Abbildung 3 mit ergibt sich aus Sarkes Et Al. 2015- 15).
Bereicherung der Bibliothek für Peptide binden an das spezifische Ziel von Interesse ist in der Regel innerhalb von drei positive Sortierung Runden erreicht, aber weiterhin eine vierte Runde der Sortierung kann zum Vorteil gereichen, wie in Abbildung 2 gezeigt und Abbildung 3 . Hier Prozent gebunden Zellen und nMFI weiter steigen von rund 3 bis Runde 4 für Beispiel Ziele, PA und Abrax, die in Abbildung 2 rot boxed sind und Abbildung 3, beziehungsweise. Die höchste Affinität Peptid Sequenzen möglicherweise bereits in Runde 3 vorhanden aber werden voraussichtlich weiter in Runde 4, die in Down-Auswahl von potentiellen Kandidaten 14Beihilfen zu bereichern. Analyse von Sequenzen von Runde 4 neigt dazu, sich wiederholenden Sequenzen offenbaren, und diese Sequenzen wiederholen sind ein ausgezeichneter Ausgangspunkt für Affinität und Spezifität Analyse und Konsens Sequenz Bestimmung. Das eCPX_Sequencing-Makro als Ergänzung zu diesem Manuskript hilft dieser ersten Analyse optimieren wie in Abbildung 4dargestellt. Beginn mit einem Ordner von .seq oder Text-Dateien, die DNA-Sequenz liegt zwischen gewählten 5' und 3'-Sequenzen ist übersetzt, organisiert von Aminosäure-Sequenz und hinsichtlich der Anzahl der einzelnen Aminosäureresten in jedes Peptid analysiert. Die sortierte Liste der isolierten Peptid-Sequenzen, wie gezeigt auf die Übersichtstabelle Blatt Screenshot in Abbildung 4, lässt sich leicht feststellen, welche Sequenzen wiederholen und auf welcher Frequenz. Die Zellen in dieser Tabelle mit sich wiederholenden Sequenzen sind in verschiedenen Farben für einfachere Analyse beschrieben. Es wird empfohlen, diese sich wiederholenden Sequenzen für Konsensus-Sequenzen und andere Trends zu überprüfen. Dies wird unterstützt durch die Sequenz Alignment-Software wie Kalign und Clustal Omega und Analyse kann durchgeführt werden, auf die gesamte Sequenz Ausgabe (einschließlich wiederholte und nicht wiederholte Sequenzen an die Frequenz, die sie erschienen) und auf der Liste unten ausgewählt, der Wiederholung von Sequenzen (mit oder ohne ihre relative Häufigkeit). Repräsentative Ergebnisse für PA und Abrax wiederholte Sequenzen, ohne die Frequenz zu berücksichtigen, sind in Abbildung 5A gezeigt und 5 b, beziehungsweise. Beachten Sie, dass in Abbildung 5A für die PA-Ziel, mehrere der einzelnen sich wiederholenden Sequenzen selbst enthalten die Konsensussequenz WFCFTC (oder eine ähnliche Abfolge), rot, die ermittelt wurde unterstrichen, indem Sie eine Kalign Jalview Softwareanalyse zuweisen Sequenzalignment der sich wiederholenden Sequenzen. Dieser Konsens war als WXCFTC, zuvor entschlossen, PA verbindlichen Konsens, der Vertrauen in die Sortierung Methode 9bietet. In Abbildung 5 b, wo sich wiederholenden Sequenzen für das Abrax Ziel in der gleichen Weise analysiert wurden, war das Ergebnis ganz anders. Erstens gab es nur fünf Sequenzen, die wiederholt in Runde 4 der Biopanning für Abrax Bindemittel, im Gegensatz zu den fünfzehn sich wiederholenden Sequenzen isoliert von Runde 4 Biopanning für PA-Bindemittel (obwohl 44 % mehr Kolonien auch für PA sequenziert wurden). Zweitens, keiner der fünf sich wiederholenden Sequenzen enthalten die vielversprechendste "Konsens" Reihenfolge (FWAWF, unterstrich in lila), obgleich Kandidat AX-A15 enthaltenen die Reihenfolge, die am besten abgestimmt (FWDTWF). Weitere Analysen, einschließlich verbindliche Affinität und Spezifität Bestimmung, dazu beigetragen, den Konsens der FWDTWF aus den Top-fünf Kandidaten für Abrax Bindung in Abbildung 5, bestimmt zu bieten, wie nachstehend erläutert.
Eine repräsentative Kolonie von einzelnen sich wiederholenden Sequenz kann weiter auf Zelle Affinität und Spezifität mit FACS, wie in Abbildung 6A für die sich wiederholenden Sequenzen von Biopanning für Abrax Bindung Peptide analysiert werden. Hier ist es klar, dass alle fünf sich wiederholenden Sequenzen höheren Affinität für Abrax, das beabsichtigte Ziel, als Rivax, das strukturell ähnliche Protein verwendet für negative sortieren oder Streptavidin, die während der Sortierung aufgrund der magnetischen Beads verwendet haben für vorhanden ist Biopanning. Dies steht im Einklang mit den bereits vielversprechende Ergebnisse für Affinität und Spezifität der Sortierung Runden gezeigt in Abbildung 3, wo es klar ist, dass Bereicherung für die Bindung an das beabsichtigte Ziel, Abrax, steigt mit jeder Runde des Biopanning beim Affinität für das ähnliche Protein, Rivax, ist nicht. Jedoch wurde eine zufriedenstellende Konsensussequenz nicht bestimmt für die Abrax Art mit der Wiederholung von Runde 4 allein Sequenzen (Abbildung 5 b und über Diskussion). Rückkehr zu den 100 sequenzierten Kolonien von Runde 4, wurde jedoch vom Auge festgestellt wenn auf der Suche nach Sequenzen ähnlich wie die beste Übereinstimmung für die vorhergesagten Konsens, dass dieser einen zusätzlichen Kandidaten, AX-A12, FWDTWF Sequenz enthalten, die in notiert wurde isolieren AX-A15 , und dass sich ein weiterer Kandidat, AX-A14, einen ähnlichen Ablauf, DWNTWF. Diese und andere Sequenzen, die Ähnlichkeiten mit den sich wiederholenden Sequenzen enthalten, zeigen Ähnlichkeiten mit anderen nicht wiederholenden Sequenzen oder wurden nach dem Zufallsprinzip ausgewählt wurden durch FACS analysiert für verbindliche Affinität und Spezifität. Die getestet werden in Sarkes Et Al. 2016 1 und die Top 5 Bindemittel aus dieser Analyse sind in Abbildung 6 b, mit der Konsensussequenz entschlossen, FWDTWF, dargestellt in Abbildung 5gezeigt.
Eine ähnliche Analyse verbindliche Affinität für sich wiederholende Peptid-Sequenzen in Runde 4 der Biopanning für Peptide, die von der PA-Ziel erkannt ergab, dass die beste Bindemittel als Rang nach dem Verhältnis des PA-488 nMFI:SAPE nMFI WXCFTC Konsens oder eine ähnliche enthalten Sequenz (Tabelle 1). Mit Hilfe dieses Verhältnis, die Sequenzen in diejenigen, die den Konsens zu enthalten und diejenigen, die nicht selbstorganisiert. Top-Kandidaten, alle enthaltenden Sequenzen im Zusammenhang mit dem Konsens, ebenso zu den Methoden, die oben beschrieben für Abrax in Abbildung 6, analysiert wurden wie in Sarkes Et Al. 2015 14dargestellt. Diese Ergebnisse zeigen, dass für einige Ziele, Analyse von Peptiden mit sich wiederholenden Sequenzen möglicherweise geeignete Bindemittel mit signifikante Affinität und Spezifität für ein gewähltes Ziel zu erhalten, und eine Konsensussequenz bestimmen, die möglicherweise zu sein, allein, für die Bindung. Beachten Sie jedoch, dass die Anzahl der Wiederholungen nicht der relative Bindungsaffinität für das Ziel von Interesse (Tabelle 1 notwendigerweise). Wenn die Analyse sich wiederholender Sequenzen allein nicht ausreichen, um ein Muster zu bestimmen ist, ist es hilfreich, abwechselnd Sequenzanalyse und Bindung Analyse um den Pool der Kandidaten einzugrenzen und die Trends bei den Kandidaten mit höherer Affinität zu überdenken . Auch wenn ein Trend beobachtet wird, aus der Analyse der sich wiederholenden Sequenzen allein, können zusätzliche nicht wiederholenden Sequenzen die gleichen Trends oder andere Trends bei der weiteren Untersuchung zu demonstrieren.

Abbildung 1: Schematische Biopanning Protokoll für bakterielle Anzeige Bibliotheken. Wie hier dargestellt, Biopanning bakterielle Anzeige Bibliotheken ist ein zyklischer Prozess. Jede Zelle in der bakteriellen Display-Bibliothek enthält (siehe Tabelle der Materialien) Plasmid-DNA, die beibehalten werden, solange die Zellen in der Gegenwart das Antibiotikum angebaut für die das Plasmid ein Resistenz-Gen (Choloramphenicol in diesem Fall enthält). Jedes Plasmid kodiert auch das Display-Gerüst-Protein, das eine zufällige Peptid an der Außenseite der Zelle macht. Da jede Zelle nur eine einzige Plasmid-DNA-Sequenz enthalten sein sollte, sollte jede Zelle nur eine einzige Peptid an mehreren Standorten in der Zellmembran angezeigt werden. Je nach Grad der Bibliothek Vielfalt zu Beginn des Biopanning und nach jeder Sortierung Runde die DNA-Sequenz kann oder möglicherweise nicht eindeutig von Zelle zu Zelle. Biopanning beginnt mit Wachstum und Induktion von der Zellbibliothek anzuzeigenden einzelnen Peptide auf ihre äußere Membran. Diese Peptide können dann mit einem Zielprotein interagieren (die biotinylierte oder sonst für die Aufnahme markiert). Für magnetische aktiviert Zelle sortieren (MCS) ungebundenen Protein wird entfernt und die Zellen sind mit magnetischen Beads, die mit einem Capture-Protein (Streptavidin in diesem Beispiel hat eine starke Interaktion mit Biotin) beschichtet werden inkubiert. Die ganze Kultur ist dann ein Magnet für gebundene Zellen aus ungebundenen Zellen trennen ausgesetzt. Mit Hilfe einer automatisierten magnetische Sortieranlage wird bevorzugt für Zellseparation, Fehlalarme zu reduzieren. Hier sehen Sie eine positive Art, in der die Zellen, die sind verpflichtet und Co eluieren mit magnetischen Beads beibehalten werden. Für eine negative Art, die wir in der Regel vorne durchführen, ist der Prozess gleich, außer, dass die Zellen, die aus der magnetischen Beads gewaschen werden stattdessen beibehalten werden. Die Bibliothek Bruchteil von Interesse, gebunden (positive Sortierung) oder ungebundene (negative Art), ist über Nacht gewachsen und in die nächste Runde der Sortierung verwendet. In jeder nachfolgenden Runde des positiven Biopanning muss eine Abnahme der Konzentration und Perle Zielvolumen Stringenz zu verbessern. Nach jeder Runde des Biopanning sind Affinität und Spezifität der Peptide für die Protein-Ziel anhand Fluoreszenz-aktivierte Zelle sortieren (FACS) um zu bestimmen, wann man aufhören Biopanning und Untersuchung der einzelne Kandidaten zu starten. Bei Bedarf werden DNA-Sequenzierung und Peptid-Sequenz-Analyse durchgeführt. Diese Analyseschritte möglicherweise selbst ein zyklischer Prozess, besonders für die Aufspürung von Trends in den einzelnen Isolate nach der letzten Runde des Biopanning. An dieser Stelle werden Peptid Sequenz Trends und/oder Konsens korreliert mit verbindlichen Affinität und Spezifität. Bitte klicken Sie hier für eine größere Version dieser Figur.
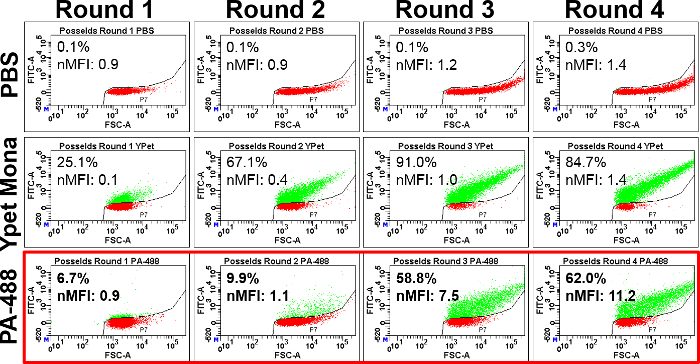
Abbildung 2: Beispieldaten für FACS Analyse des Sortierens Runden: PA Ziel. Verbindliche Affinität von FACS ist nach 4 Runden Biopanning (positive Sortierung) der gewählten bakterielle anzeigen Bibliothek für Peptide binden an das Ziel, schützendes Antigen (PA) gezeigt. Dies erfolgte nach der ersten Durchführung einer negativen Art gegen die Streptavidin beschichteten magnetischen Kügelchen. Für verbindliche Beurteilung wurden Zellen induziert mit 0,4 % L-Arabinose für 90 min vor der Inkubation mit Ziel und Steuerungslösungen und durch FACS Analyse. Verbindliche Affinität Analyse für das beabsichtigte Ziel ist rot boxed. Sehen Sie hier Streudiagrammen FITC-A vs FSC-A und Werte für Prozent Zellen gebunden (im Vergleich zu den gated Negativkontrolle mit PBS allein bebrütet) und mittlere Fluoreszenzintensität (nMFI, verglichen mit Peptid-freie Negativkontrolle mit inkubiert normalisiert das gleiche Fluorophore beschriftet Protein) induzierte Zellen, die für 45 min mit inkubiert wurden: PBS-Puffer allein 150 nM YPet Mona (Positivkontrolle für Peptid-Ausdruck) oder 250 nM PA-488 (Ziel beschriftet). Beachten Sie die zunehmende Anreicherung in verbindliche Affinität für das Ziel des Interesses nach jeder Runde der Biopanning. Im Gegensatz zu Abbildung 3wurden alle AutoMCS Zelle Trennungen mit Programm Posselds abgeschlossen. Ein Teil dieser Daten kann auch in Punktdiagrammen FITC-H vs FSC-H Sarkes Et Al. 2015 14 zum Vergleich auf der FITC-A Vs FSC-A Grundstücke hier gezeigten visualisiert werden. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: Beispieldaten für FACS Analyse des Sortierens Runden: Abrax Ziel. Ähnlich wie Abbildung 2gezeigt, hier ist vergleichende Bindungsaffinität für ein komplettes Biopanning-Experiment, wie durch FACS bestimmt. Die bakterielle Anzeigebibliothek unterzogen wurde negativ Sortierung gegen die Streptavidin beschichteten magnetische Beads, wie in Abbildung 2, aber wurde dann eine weitere Runde der negativen Sortierung gegen ein homologen Protein mit ähnlicher Struktur unterzogen und Funktion, Rivax, vor der Durchführung von 4 Runden des positiven Biopanning für Peptide binden an das beabsichtigte Ziel, Abrax. Für verbindliche Beurteilung wurden Zellen induziert mit 0,4 % L-Arabinose für 45 min vor der Bindung an Ziel, positive Kontrolle und Negativkontrollen und Lesung am FACS. Verbindliche Affinität für das beabsichtigte Ziel ist rot boxed. Sehen Sie hier Streudiagrammen FITC-A vs FSC-A (oder PE-A Vs FSC-A bei SAPE) Werte für Prozent Zellen gebunden (im Vergleich zu den gated Negativkontrolle mit PBS allein bebrütet) und nMFI der induzierten Zellen, die für 45 min mit inkubiert wurden : PBS-Puffer allein 150 nM YPet Mona (Positivkontrolle für Peptid-Ausdruck), 250 nM Abrax-488 (beschriftete Protein Ziel), 250 nM Rivax-488 (beschriftete potenzielles Ziel kreuzreaktiven Protein) oder 250 nM SAPE (negativ-Kontrolle für die direkte Bindung an Streptavidin beschichteten magnetische Beads). Beachten Sie die zunehmende Anreicherung in Bindungsaffinität für das Ziel von Interesse, Abrax, nach jeder Runde der Biopanning, und geringe Bindungsaffinität für das strukturell ähnliche Protein, Rivax. Im Gegensatz zu Abbildung 2positive Biopanning Runde 1 mit Hilfe durchgeführt wurde AutoMCS Posselds Programm während nachfolgende Sortierung Runden vollendet mit Programm Posseld, wie es für Biopanning in dieser Handschrift. Diese Daten können auch in Punktdiagrammen FITC-H vs FSC-H Sarkes Et Al. 2016 15 zum Vergleich auf der FITC-A Vs FSC-A Grundstücke hier gezeigten visualisiert werden. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4: Beispiel Sequenzanalyse Ausgabe mit "eCPX_Sequencing" Makro. Dargestellt ist ein Screenshot von 48 sequenzierten Kolonien von Runde 4 der Biopanning der gewählten bakterielle Anzeigebibliothek für PA Bindemittel mit der Posselds-Methode. Das Tabellenblatt "Zusammenfassung" wird hier angezeigt. Beachten Sie, dass die Kolonien in der alphabetischen Reihenfolge der angegebenen Dateinamen während der Sequenzierung gezählt wurden und die Felder rund um Sequenzen, die mindestens einmal wiederholt in verschiedenen Farben für einfacher Datenanalyse beschrieben werden. Das eCPX_Sequencing-Makro wird als zusätzlicher Code-Datei bereitgestellt. Bitte klicken Sie hier für eine größere Version dieser Figur.
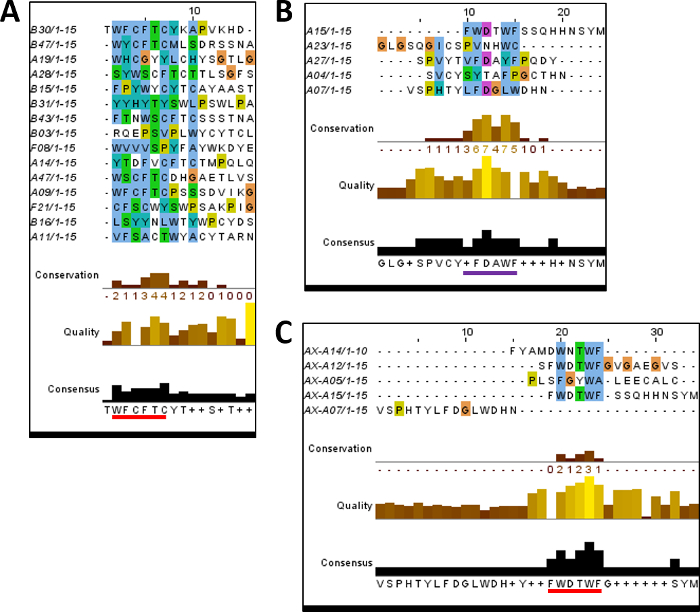
Abbildung 5: Sequence Alignment und Konsens Bestimmung für zwei unterschiedliche Protein-Targets. Alle hier gezeigten Bilder wurden mit Jalview-Software mit ClustalX Färbung nach dem Ausrichten mit Kalign Online-Analyse-Software 51Sequenzen erzeugt. (A) Ausrichtung sich wiederholender Sequenzen von PA Biopanning Runde 4 (entspricht Tabelle 1). (B) Ausrichtung sich wiederholender Sequenzen von Abrax Biopanning Runde 4 (entspricht Abbildung 6A). (C) Ausrichtung der Top 5 Kandidaten von Abrax Biopanning Runde 4, FACS Analyse der einzelnen Kandidaten bestimmt (entspricht Abbildung 6 b). Die rote Linie unterstreicht die Konsensussequenz in A und C, während die violette Linie b den Vorläufer der Konsensussequenz für Abrax verbindlich bei sich wiederholenden Sequenzen nur, der Prüfung ermittelten Hervorhebung das Potenzial unterstreicht Bindungsaffinität zu testen müssen verwenden FACS, bevor ein Konsens in einigen Fällen ermittelt werden kann. Ähnliche Ausrichtungen erzeugt mit verschiedenen Analyse-Software ist in den Sarkes Et Al. 2015 14 und Sarkes Et Al. 2016 1ersichtlich. Bitte klicken Sie hier für eine größere Version dieser Figur.
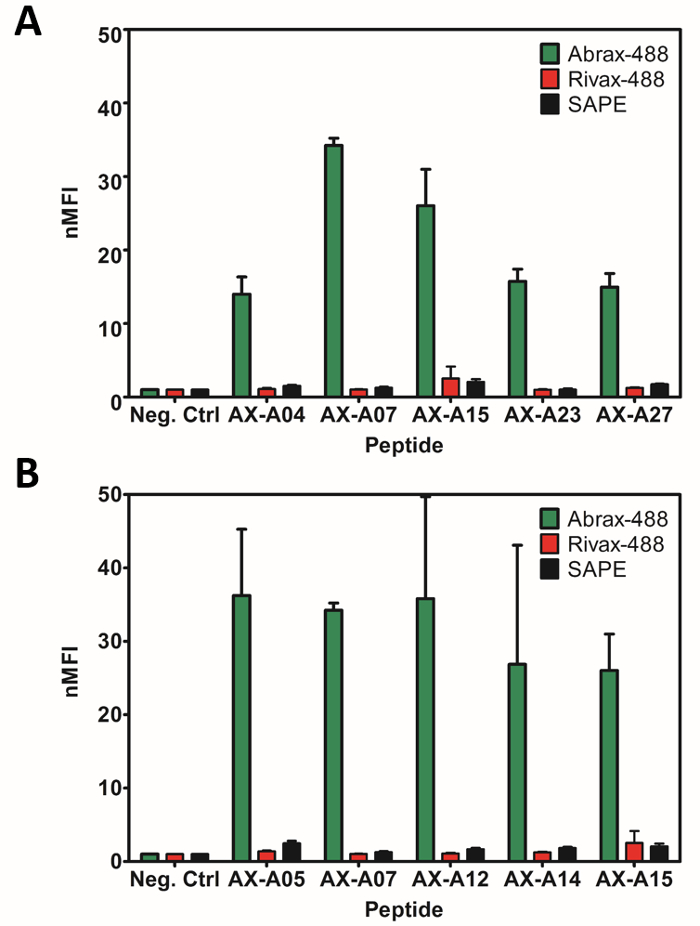
Abbildung 6: Beispiel Bindungsaffinität und Spezifität für die einzelnen Kandidaten mit FACS analysiert. Hier wird der normalisierten Median Fluoreszenzintensität (nMFI) mit FACS (A) sich wiederholende Sequenzen und (B) die Top 5-Kandidaten als bestimmt durch vergleichende Bindungsaffinität für das Ziel, Abrax, von Runde 4 des Biopanning bestimmt. Die Daten stellt die durchschnittliche (Balken) und Standardabweichung (Fehlerbalken) von drei unabhängigen Experimenten replizieren. Beachten Sie, dass alle Kandidaten gezeigt, ganz spezifisch für Abrax (Abrax-488, grüner Balken) über ein strukturell ähnlichen Protein, Rivax (Rivax-488, rote Balken), und die Streptavidin Negativkontrolle (SAPE, schwarze Balken). Obwohl zwei der sich wiederholenden Sequenzen in einer (AX-07 und AX-15) auch unter den Top 5-Kandidaten in B waren, möglicherweise Vergleich der Ergebnisse, die in A und B zeigt, dass die Analyse sich wiederholender Sequenzen allein nicht ausreichen, um die höchste Affinität Peptide zu isolieren. Vorliegenden Daten wurde von Sarkes Et Al. 2016 1angepasst. Bitte klicken Sie hier für eine größere Version dieser Figur.

Tabelle 1: Verhältnis der spezifischen Bindung an unspezifischen Bindung für die einzelnen Kandidaten wiederholen. In diesem Beispiel werden die Anzahl der wiederholten Sequenzen und das Verhältnis von PA (Ziel) nMFI, SAPE (Negativkontrolle) nMFI für die wiederholten Kandidat Sequenzen aus PA Biopanning Runde 4 angezeigt. Diese Daten wurde relative PA nMFI:SAPE nMFI Verhältnis sortiert und analysiert auf das Vorhandensein von den bekannten WXCFTC Konsens oder eine ähnliche Sequenz, die in Fettschrift dargestellt und unterstrichen. Beachten Sie, dass die Sequenzen selbst zu organisieren, so dass die Kandidaten mit dem Konsens das höchste PA nMFI:SAPE nMFI Verhältnis haben und daher zu, genauer gesagt mit dem Ziel interagieren. Die dargestellten Ergebnisse sind aus einem einzigen FACS-Experiment. Peptide mit niedrigsten Affinität für das Ziel wurden ausgeschlossen, aus der weitere Wiederholungen und Analyse, die in Sarkes Et Al. 2015 14veröffentlicht wurden.
Suppplemental Datei 1: Bitte klicken Sie hier, um diese Datei herunterladen.
Suppplemental Datei 2: Bitte klicken Sie hier, um diese Datei herunterladen.
Diskussion
Biopanning bakterielle Anzeige Bibliotheken wurde ein erfolgreicher Ansatz zur Isolation der Affinität Reagenzien und Peptide bieten eine sinnvolle Alternative zu Antikörpern für die Anerkennung, wenn bestimmte Kriterien wie Stabilität in extremen Umgebungen erforderlich sind. Biopanning dieser Bibliotheken mit den AutoMCS Methoden beschrieben hier wurde gestrafft, und eine handelsübliche AutoMCS Plattform erstreckt sich diese Technologie auf einem viel breiteren Publikum. Im Vergleich zu traditionellen abwechselnd MCS/FACS Sortierverfahren für bakterielle Anzeige Bibliotheken, AutoMCS Methoden sind weniger teuer, weil sie keine Investitionen in ein teurer FACS-Instrument in der Lage, Sortierung und isolieren die gebundenen Zellen benötigen, anstatt die Art der Durchflusszytometer, die hier verwendet wird, kann das nur Analyse 14. Die entscheidenden Schritte für erfolgreiche Biopanning von AutoMCS gehören Trennung Programmauswahl, negative gegen potenzielle Kreuz-Reaktionspartner reduzieren Fehlalarme, Überwachung Bibliothek Anreicherung mit FACS um zu bestimmen, wann man aufhören muss Biopanning, Sortierung und intelligente Analyse der Kandidatenpool umständlich Screening von Hunderten von Kandidaten zu vermeiden.
Die hier beschriebenen Beispiele zeigen erfolgreiche Biopanning der gewählten bakterielle Anzeigebibliothek für zwei separate Ziele, PA und Abrax, wenn auch mit leicht unterschiedlichen Auswertungsstrategien aufgrund von Unterschieden in der Peptid-Sequenzen für jeden Pool von Kandidaten nach vier Runden der Biopanning. PA war es einfacher bei Sequenzanalysen zeigen klare Trends aus dem Peptid-Sequenzen, die in 144 Kolonien mindestens einmal wiederholt. Dies allein war genug, um eine Konsensussequenz für das PA-Ziel zu bestimmen, und diese Kolonien, die den Konsens enthalten oder eine ähnliche Reihenfolge wurden die besten Kandidaten in Bezug auf das Verhältnis der Bindung an die PA gegenüber Bindung an die Streptavidin-negativ-Kontrolle. Jedoch wird dies nicht immer der Fall sein. Für das Ziel Abrax Sequenzierung 100 Kolonien führte zu fünf sich wiederholenden Sequenzen, aber der Trend in diesen Sequenzen war weniger klar, als eine Tendenz zu aromatischen Aminosäurereste und Asparaginsäure oder Asparagin enthalten. Der vorhergesagte "Konsens" aus dem sich wiederholenden Sequenzen nur hätte produziert und getestet für Affinität zu Abrax, aber da keine übergeordnete Sequenzen, dass genaue Konsensussequenz enthalten, wir wieder die Liste der sequenzierten Kolonien und beobachtet, dass andere Kandidaten, die mehr Trends mit einander gemein hatte. In der Tat enthalten zwei Kolonien eine ähnliche Abfolge, die "Konsens" aus den Wiederholungen allein, (FWDTWF statt FWAWF), die den gleichen Trend von Tryptophan, Phenylalanin und Asparaginsäure Rückstände, aber mit unterschiedlichen Abstand hatte. Diese Peptide gehörte zu einer sich wiederholenden Sequenz, zählte nicht. Diese zwei Sequenzen, sowie eine dritte Sequenz enthält eine ähnliche Variante wurden unter die Top 5-Bindemittel in Bezug auf die Affinität zu Abrax getestet. Insgesamt Top Bindemittel, AX-A05, auch ähnliche Trends angezeigt. Die Ergebnisse für Abrax zeigen, dass ein Ansatz der wechselt mehrmals zwischen Sequenzanalyse und Affinität und Spezifität Analyse manchmal erforderlich, abhängig von der gewählten Ziel werden. Die Ergebnisse für das Abrax Ziel zeigen auch das Niveau der Spezifität, die über ein sehr ähnliches Protein (Rivax 1,15) erreicht werden kann.
Die AutoMCS-Programme für unsere Studien ausgewählt wurden Posselds und Posseld, die sowohl für positive Selektion von beschrifteten Zielzellen mit niedriger Frequenz, weniger als 5 % der ursprünglichen Bevölkerung sind. In beiden Fällen gehen Zellen durch magnetische zweispaltig. Im Falle des Posselds-Programms durchlaufen jedoch Zellen langsamer die erste Spalte Belichtung Zeit 41zu erhöhen. Neben den Programmbeschreibungen des Herstellers des kommerziellen AutoMCS Gerätes (siehe Materialtabelle) 41, diese Programme wurden nach einer anfänglichen Vergleich mehrere potenzielle Programme ausgewählt. Jedes Programm Fähigkeit zu vermeiden Fehlalarme aus einer homogenen Kultur der Negativkontrolle Zellen herausziehen und bekannten Bindemittel erfolgreich zu einem Ziel von Interesse von einer Mischkultur Negativkontrolle Zellen gespickt mit einer abnehmenden zu isolieren Prozentsatz der bekannten Bindemittel wurden verglichen. Wenn erheblich von den hier beschriebenen Anwendungen abweichen, könnte ein ähnlicher Vergleich hilfreich bei der Programmauswahl für die neue Anwendung sein. Jedoch veröffentlichte bisher Ergebnisse Vergleich dieser beiden Programme (Posseld und Posselds) für die Isolierung von Peptid Affinität Reagenzien für PA ergab, dass unter Verwendung eines dieser Programme für alle vier Runden des Biopanning zur Isolierung von Peptiden mit ähnlichen führte Affinität und Spezifität für PA und Bestimmung des WXCFTC Konsens. Die Hauptunterschiede waren diese Posseld mit seiner schnelleren Durchfluss, schneller sortiert und verbindliche Affinität für das Ziel lag daher nach nur zwei Runden der Biopanning, während mit Posselds zu mehr einzigartigen Sequenzen nach vier Runden der Biopanning führte 14. daraus lernen, die Strategie wurde leicht verändert als Biopanning für Abrax Bindung Peptide, beide Vorteile zu integrieren: Posselds wurde für Runde 1 verwendet, um mehr Belichtungszeit bei diesem wichtigen Schritt zu ermöglichen, wo Vielfalt aber dann am höchsten, ist, die Programm wurde für die schnellere Isolierung der höchsten Affinität Bindemittel während runden 2-4- 1,15auf Posseld umgestellt. Dies schien ideal für Abrax, zumal die nMFI und prozentuale Bindung waren beide höher für Abrax Runde 4 im Vergleich zu PA Runde 4 mit beiden Programmen sortieren Sortierung (Abbildung 2 und Abbildung 3 und Sarkes Et Al. 2015 ( 14), aber ein direkter Vergleich mit einer Strategie im Vergleich zu den anderen mit dem gleichen Ziel wäre für ein aussagekräftiger Vergleich erforderlich. Welches Programm am besten für eine bestimmte Anwendung ist möglicherweise hängen die unterschiedlichen Zielgruppen, die Sortier-Bibliothek verwendet und das Niveau der Peptid-Ausdruck, der bei jeder Änderung an den hier beschriebenen Bedingungen erreicht wird.
Nicht diskutiert hier sind mögliche Ergebnisse aus Biopanning mit abiotischen Materialien, aber wir haben auch die gleiche bakterielle Anzeigebibliothek für Biopanning gegen Masse Aluminium 2verwendet. Dieser Studie erfolgte nicht mittels AutoMCS, aber ähnliche Studien mit AutoMCS, wenn das Material zur Verfügung steht, oder könnte beschichtet oder, eine magnetische Perle konjugiert erreicht werden könnte. In der Bulk-Aluminium-Studie war einzelne Aminosäure-Analyse und Modellierung der Sekundärstruktur hilfreich für das Verständnis, was der Bindungsaffinität der isolierten Peptide, fuhr, da keine Konsensussequenz entschlossen 2war. Auch mit biologischen Zielen ist dies möglicherweise zu verstehen, was treibt die Bindungsaffinität für einige Ziele, weshalb die Kalkulationstabelle generiert aus dem "eCPX_Sequencing" Makro enthält eine Registerkarte mit einzelnen Rückstände Frequenzanalyse erforderlich. Wenn keine sich wiederholenden Sequenzen zusätzlich zu den Mangel an Konsens gelten und Rückstände Frequenz kein Muster zeigt, jedoch möglicherweise andere Arten der Analyse erforderlich. Darüber hinaus kann weitere Sortierung Runden zu vermeiden, eine viel größere Anzahl von Kandidaten für Down-Auswahl derjenigen mit ausreichender Affinität zum gewünschten Ziel screening erforderlich.
Mit der zunehmenden Verfügbarkeit der Sequenzierung der nächsten Generation konnte eine gründlichere Untersuchung die aussichtsreichsten Kandidaten vor dem Testen Affinität zu ermitteln und bestimmen das Ausmaß der Bereicherung nach jeder Runde des Biopanning durchgeführt werden. Weiter zu den verbindlichen Analyseschritt Sequenzen müssen jedoch durch Klonen neu erstellt werden wenn sie nicht hoch genug Frequenz leicht mit Routine Kolonie Sequenzierung der Dutzende oder Hunderte von Kolonien zu isolieren sind. Da diese Technologie fortschreitet, immer weniger teuer und mehr Routine, und vor allem mit einer Option für die Kolonie Isolierung, es wahrscheinlich wird sein der beste Weg, um Biopanning Daten zu analysieren. Es kann zur Aufklärung der Art und Weise einzelnen Sequenzen und die gesamte Bibliothek führen, entwickeln. Darüber hinaus können die Bakterien in der Sortierung Bibliothek im Laufe der Zeit mutieren die bias könnte Sortierung in Richtung Zellen mit schneller Wachstumsraten oder Bakterien, die genomische Proteine binden ein Material auch ohne Gerüst und Peptid Anzeigeausdruck, für overexpress Instanz. Aus diesem Grund einmal viel versprechende Kandidaten entstehen, lohnt es sich das Plasmid DNA zu isolieren, Forscherkollegen frischen E. Coli und Peptid-Bindung über FACS bestätigen. Falls das Peptid in einem zellfreien Format verwenden, sollten Affinität auch für das freie Peptid bewertet werden. Zum Beispiel wurden Peptid Affinität Reagenzien durch bakterielle Anzeige für das Ziel SEB entdeckt synthetisch aus Zelle und analysiert von eher traditionellen Methoden wie Enzym-linked Immunosorbant Assay (ELISA), und auf (über FACS) und off-Zellen produziert (per ELISA) Affinität wurden verglichen mit der K-d-s durch beide Methoden 7bestimmt.
Obwohl die Stärke der Wechselwirkung Biotin-Streptavidin es eine ideale Strategie für die Aufnahme von Protein-Ziel und gebundene Peptide macht, kann dieses Verfahren für andere Strategien der Aufnahme geändert werden. Direkte Kopplung der magnetischen Beads wie Epoxy und Carbonsäure Perlen-Protein ist erfolgreich gewesen und einen verbindliche Schritt entfernt. Allerdings behindern Direktanschluss möglichen Bindungsstellen auf spezifische oder unspezifische Weise, je nach Anlage-Strategie. Ein zusätzlicher Vorteil bei der Verwendung von Streptavidin Perlen ist, dass weniger stabiles Protein Ziele können frisch biotinylierte in 30 min oder gespeicherte eingefroren, wenn benötigt, während die Perlen sich längere Inkubation mit dem Protein zur Vernetzung erfordern tendenziell und sind, in der Regel bei 2 bis 8° c gelagert Die empfohlene Start Konzentration der biotinylierte Protein Ziel hier, 600 nM, für mehrere Ziele erfolgreich gewesen, aber es kann möglich sein, Peptid-Capture-Reagenzien mit erhöhte Affinität zu isolieren, wenn diese Konzentration reduziert wird. Darüber hinaus kann diese Methode erweitert und für andere bakterielle Anzeige-Bibliotheken und andere Organismen modifiziert. Beispielsweise können diese Schritte in der Abwesenheit von Sauerstoff für die Verwendung mit Anaerobier oder in anderen Umgebungen für den Einsatz mit Extremophilen Peptide mit einzigartigen Eigenschaften zu isolieren durchgeführt werden. Die Peptide und/oder Konsens bestimmt für ein bestimmtes Ziel von Interesse kann potenziell verwendet auf Zelle als ein lebendiges Material, oder synthetisch hergestellten aus Zelle. Synthetisch hergestellte Peptide können weiter gereift werden, für die Entwicklung von noch höheren Affinität und robuster Protein katalysiert Capture (PCC) Agenten für Sensoren oder für andere Anwendungen, wo Antikörper würden in der Regel für die Bindung verwendet werden, oder Anerkennung 38,55. Molecular Modelling kann auch verwendet werden, um zu bestimmen, bindend, Lage des Peptids mit ihrem Ziel vor kurzem für die Abrax Bindung Peptide und Konsens durchgeführt wurde in Abbildung 5 1aufgeführt. Insgesamt Biopanning bakterielle Anzeige Bibliotheken für Peptid Affinität Reagenzien ist eine schnelle und unkomplizierte Alternative zu die Produktion von Antikörpern für die Anerkennung und Nachweis von Protein-Targets und dieser halbautomatischen Biopanning Ansatz zuverlässige Ergebnisse mit weitreichenden Anwendungen produziert.
Offenlegungen
Die Autoren haben nichts preisgeben
Danksagungen
Die Autoren möchten die Unterstützung der US-Army Research Laboratory (ARL) Postdoctoral Fellowship-Programm, von Oak Ridge verbundene Universitäten durch einen Vertrag mit ARL, verwaltet durch die Ernennung von Dr. Justin P. Jahnke anerkennen. Verbleibenden Mittel der Sensoren und Elektron Geräte Direktion ARL sorgte. Die Autoren möchten auch Daugherty Labor an UCSB vielen Dank, dass die bakterielle Anzeigebibliothek und Information zum Klonen YPet Mona Reagenz, Dr. Bryn Adams für Unterstützung beim Klonen der YPet Mona-Reagenz bei ARL, Dr. Joshua Kogot für seine erste Arbeit auf und Ausbildung in Biopanning bakterielle Anzeige Bibliotheken, Alena Ruhe Edgewood chemischer und biologischer Zentrum für den Austausch von Plasmiden verwendet, um auszudrücken und zu reinigen Abrax und Rivax Proteine, Brandi Dorsey für den technischen Support für die Isolierung von PA Bindung Peptiden, Qin Guo für den technischen Support von Vorversuchen zur Isolation von Abrax Bindung Peptide und Dr. Jessica Terrell für hilfreiche Gespräche über diese Handschrift.
Materialien
| Name | Company | Catalog Number | Comments |
| autoMACS Pro Separator | Miltenyi Biotec | 130-092-545 | Automated cell separator for use with magnetic beads |
| Luria Broth, Miller (LB) | Fisher Scientific | BP1426-500 | Growth medium |
| Chloramphenicol | Fisher BioReagents | BP904-100 | Antibiotic |
| Agar | Fisher Scientific | BP2641-500 | Thickening/geling agent |
| eCPX 3.0 Bacterial Display Peptide Library | Cytomx Therapeutics | N/A; http://cytomx.com/contact/ | On-cell peptide display library. Provided by collaborators at USCB. |
| L-arabinose | Sigma | A3256-500G | Sugar, for induction of protein expression |
| Phosphate Buffered Saline pH 7.4 (PBS) | Amresco | 0780-10L | Buffer |
| EZ-Link Sulfo-NHS-LC-Biotin, No-Weigh | Thermo Scientific | PI21327 | Adds biotin molecule to primary amines |
| Pierce Biotin Quantitation Kit | Thermo Scientific | PI28005 | Ensures biotin molecule is covalently bound to protein |
| Dynabeads MyOne Strepavidin T1 Beads | Invitrogen | 65601 | Magnetic beads for capture of biotin-congugated proteins |
| Bovine Serum Albumin (BSA) | Sigma | A9647-100G | Protein, used as blocking agent for non-specific binding |
| D-glucose | Sigma | G8270-1KG | Sugar, for growth and impeding inducible protein expression |
| Ypet Mona | UCSB and ARL | N/A | Positive control for monitoring expression of eCPX 3.0. Produced by authors and collaborators; see references 12 and 13. |
| Dylight 488 NHS Ester | Thermo Scientific | 46403 | For conjugating protein target to fluorophore |
| Streptavidin, R-Phycoerythrin conjugate (SAPE) | Thermo Scientific | S866 | Negative control for monitoring non-specific binding to streptavidin |
| BD FACSFlow | Becton, Dickinson, and Company | 342003 | Buffer for running FACS instrument |
| autoMACS Columns | Miltenyi Biotech | 130-021-101 | For MACS Separation |
| autoMACS Running Buffer – MACS Separation Buffer | Miltenyi Biotech | 130-091-221 | For autoMACS instrument maintenance. Hazard: contains 0.09% azide. |
| autoMACS Pro Washing Solution | Miltenyi Biotech | 130-092-987 | For autoMACS instrument maintenance |
| 70% Ethanol | VWR | E505-4L | Decontamination of autoMACS |
| Glycerol | Sigma | G6279-1L | For bacterial freezer stocks |
| Chill 15 rack | Miltenyi Biotec | 130-092-952 | For MACS Separation |
| BD FACSCanto II | Becton, Dickinson, and Company | 744810; http://www.bdbiosciences.com/us/instruments/research/cell-analyzers/bd-facscanto-ii/m/744810/overview | For assessment of peptide binding to target |
| BD FACSDiva Software | Becton, Dickinson, and Company | 659523 | For assessment of peptide binding to target |
| Dynabeads MPC-S magnetic particle concentrator | Thermo Scientific | A13346 | Bench top cell separator for use with magnetic beads |
| Colony Sequencing | Genewiz | N/A; https://www.genewiz.com/ | DNA and Colony Sequencing Services |
| Excel | Microsoft | 323021400; https://www.microsoftstore.com/store/msusa/en_US/pdp/Excel-2016/productID.323021400 | For use with sequence analysis macro |
| Anthrax Protective Antigen (PA) | List Biological Laboratories, Inc. | 171 | Target protein used as example for representative results. |
| Abrax | ECBC and ARL | N/A | Target protein used as example for representative results. Produced by authors and collaborators; see references 1 and 15. |
| Rivax | ECBC and ARL | N/A | Target protein used as example for representative results. Produced by authors and collaborators; see references 1 and 15. |
Referenzen
- Sarkes, D. A., Hurley, M. M., Stratis-Cullum, D. N. Unraveling the Roots of Selectivity of Peptide Affinity Reagents for Structurally Similar Ribosomal Inactivating Protein Derivatives. Molecules. 21 (11), 1504 (2016).
- Adams, B. L., Finch, A. S., Hurley, M. M., Sarkes, D. A., Stratis-Cullum, D. N. Genetically Engineered Peptides for Inorganics: Study of an Unconstrained Bacterial Display Technology and Bulk Aluminum Alloy. Adv. Mater. 25 (33), 4585-4591 (2013).
- Bessette, P. H., Rice, J. J., Daugherty, P. S. Rapid isolation of high-affinity protein binding peptides using bacterial display. Protein Eng. Des. Sel. 17 (10), 731-739 (2004).
- Daugherty, P. S. Protein engineering with bacterial display. Curr. Opin. Struct. Biol. 17 (4), 474-480 (2007).
- Getz, J. A., Schoep, T. D., Daugherty, P. S. Peptide Discovery Using Bacterial Display and Flow Cytometry. Methods Enzymol. 503, 75 (2012).
- Kenrick, S. A., Daugherty, P. S. Bacterial display enables efficient and quantitative peptide affinity maturation. Protein Eng. Des. Sel. 23, 9-17 (2010).
- Kogot, J. M., et al. Screening and characterization of anti-SEB peptides using a bacterial display library and microfluidic magnetic sorting. J. Mol. Recognit. 27 (12), 739-745 (2014).
- Kogot, J. M., Pennington, J. M., Sarkes, D. A., Stratis-Cullum, D. N., Pellegrino, P. M. . Population Enrichment and Isolation with Magnetic Sorting, DTIC Document No. ARL-TN-0452. Report No. ARL-TN-0452. , (2011).
- Kogot, J. M. Screening of peptide libraries against protective antigen of Bacillus anthracis in a disposable microfluidic cartridge. PLOS ONE. 6 (11), e26925 (2011).
- Little, L. E., Dane, K. Y., Daugherty, P. S., Healy, K. E., Schaffer, D. V. Exploiting bacterial peptide display technology to engineer biomaterials for neural stem cell culture. Biomaterials. 32 (6), 1484-1494 (2011).
- Pennington, J. M., Kogot, J. M., Sarkes, D. A., Pellegrino, P. M., Stratis-Cullum, D. N. Isolation and characterization of anti-SEB peptides using magnetic sorting and bacterial peptide display library technology. SPIE Defense, Security, and Sensing. 83581Z-1-83581Z-6 International Society for Optics and Photonics. , (2012).
- Rice, J. J., Daugherty, P. S. Directed evolution of a biterminal bacterial display scaffold enhances the display of diverse peptides. Protein Eng. Des. Sel. 21 (7), 435-442 (2008).
- Rice, J. J., Schohn, A., Bessette, P. H., Boulware, K. T., Daugherty, P. S. Bacterial display using circularly permuted outer membrane protein OmpX yields high affinity peptide ligands. Protein Sci. 15 (4), 825-836 (2006).
- Sarkes, D. A., Dorsey, B. L., Finch, A. S., Stratis-Cullum, D. N. Method for Discovery of Peptide Reagents Using a Commercial Magnetic Separation Platform and Bacterial Cell Surface Display Technology. J. Anal. Bioanal. Tech. 6 (4), 1 (2015).
- Sarkes, D. A. Rapid discovery of peptide capture candidates with demonstrated specificity for structurally similar toxins. SPIE Commercial+ Scientific Sensing and Imaging. 986305-1-986305-10 International Society for Optics and Photonics. , (2016).
- Stratis-Cullum, D., Kogot, J. M., Sarkes, D. A., Val-Addo, I., Pellegrino, P. M., Pramatarova, L. Chapter 30, Bacterial display peptides for use in biosensing applications. On Biomimetics. , 629-642 (2011).
- Stratis-Cullum, D. N., Finch, A. S. Current Trends in Ubiquitous Biosensing. J. Anal. Bioanal. Tech. S7 (009), (2013).
- Rockberg, J., Löfblom, J., Hjelm, B., Uhlén, M., Ståhl, S. Epitope mapping of antibodies using bacterial surface display. Nature Methods. 5 (12), 1039-1045 (2008).
- Francisco, J. A., Campbell, R., Iverson, B. L., Georgiou, G. Production and fluorescence-activated cell sorting of Escherichia coli expressing a functional antibody fragment on the external surface. Proceedings of the National Academy of Sciences. 90 (22), 10444-10448 (1993).
- Georgiou, G. Display of heterologous proteins on the surface of microorganisms: from the screening of combinatorial libraries to live recombinant vaccines. Nat. Biotechnol. 15 (1), 29-34 (1997).
- Löfblom, J., Wernérus, H., Ståhl, S. Fine affinity discrimination by normalized fluorescence activated cell sorting in staphylococcal surface display. FEMS Microbiol. Lett. 248 (2), 189-198 (2005).
- Kjærgaard, K., Sørensen, J. K., Schembri, M. A., Klemm, P. Sequestration of zinc oxide by fimbrial designer chelators. Appl. Environ. Microbiol. 66 (1), 10-14 (2000).
- Kishimoto, J., Fukuma, Y., Mizuno, A., Nemoto, T. K. Identification of the pentapeptide constituting a dominant epitope common to all eukaryotic heat shock protein 90 molecular chaperones. Cell stress & chaperones. 10 (4), 296-311 (2005).
- Westerlund-Wikström, B. Peptide display on bacterial flagella: principles and applications. International journal of medical microbiology. 290 (3), 223-230 (2000).
- Gaskin, D. J., Starck, K., Turner, N. A., Vulfson, E. N. Phage display combinatorial libraries of short peptides: ligand selection for protein purification. Enzyme Microb. Technol. 28 (9), 766-772 (2001).
- Goldman, E. R. Phage-displayed peptides as biosensor reagents. J. Mol. Recognit. 13 (6), 382-387 (2000).
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat. Biotechnol. 15 (6), 553-557 (1997).
- Cherf, G. M., Cochran, J. R. Applications of Yeast Surface Display for Protein Engineering. Yeast Surface Display: Methods, Protocols, and Applications. , 155-175 (2015).
- London, N., Movshovitz-Attias, D., Schueler-Furman, O. The structural basis of peptide-protein binding strategies. Structure. 18 (2), 188-199 (2010).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli physiology in Luria-Bertani broth. J. Bacteriol. 189 (23), 8746-8749 (2007).
- Jahnke, J. P., Terrell, J. L., Smith, A. M., Cheng, X., Stratis-Cullum, D. N. Influences of Adhesion Variability on the "Living" Dynamics of Filamentous Bacteria in Microfluidic Channels. Molecules. 21 (8), 985 (2016).
- Chen, A. Y., et al. Synthesis and patterning of tunable multiscale materials with engineered cells. Nature materials. 13 (5), 515-523 (2014).
- Löfblom, J., Frejd, F. Y., Ståhl, S. Non-immunoglobulin based protein scaffolds. Current opinion in biotechnology. 22 (6), 843-848 (2011).
- Ladner, R. C., Sato, A. K., Gorzelany, J., de Souza, M. Phage display-derived peptides as therapeutic alternatives to antibodies. Drug Discov. Today. 9 (12), 525-529 (2004).
- Pini, A., Falciani, C., Bracci, L. Branched peptides as therapeutics. Current Protein and Peptide Science. 9 (5), 468-477 (2008).
- Gongora-Benitez, M., Tulla-Puche, J., Albericio, F. Multifaceted roles of disulfide bonds. Peptides as therapeutics. Chemical Reviews. 114 (2), 901-926 (2013).
- Coppock, M. B., et al. Peptide-based protein capture agents with high affinity, selectivity, and stability as antibody replacements in biodetection assays. SPIE Sensing Technology+ Applications. 910711-1-910711-6 International Society for Optics and Photonics. , (2014).
- Coppock, M. B. Protein Catalyzed Capture Agents with Tailored Performance for In Vitro and In Vivo Applications. Peptide Science. , (2016).
- Bessette, P. H., Hu, X., Soh, H. T., Daugherty, P. S. Microfluidic library screening for mapping antibody epitopes. Anal. Chem. 79 (5), 2174-2178 (2007).
- Chao, G. Isolating and engineering human antibodies using yeast surface display. Nat. Protoc. 1 (2), 755-768 (2006).
- Miltenyi Biotec GmbH. . autoMACS™ Pro Separator User Manual, Version 1.1. , (2007).
- Henriques, S. T., et al. A novel quantitative kinase assay using bacterial surface display and flow cytometry. PLOS ONE. 8 (11), e80474 (2013).
- . . BD FACSCanto II Operator Course Workbook. , (2007).
- . . BD FACSCanto II Instructions For Use. , (2007).
- Chan, L. Y., Yim, E. K., Choo, A. B. Normalized Median Fluorescence: An Alternative Flow Cytometry Analysis Method for Tracking Human Embryonic Stem Cell States During Differentiation. Tissue Eng. Part C Methods. 19 (2), 156-165 (2012).
- . DNA Sequencing Services Available from: https://www.genewiz.com/ (2017)
- Honegger, A., PluÈckthun, A. Yet another numbering scheme for immunoglobulin variable domains: an automatic modeling and analysis tool. Journal of molecular biology. 309 (3), 657-670 (2001).
- Sievers, F., et al. Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega. Mol. Syst. Biol. 7 (1), 539 (2011).
- Lassmann, T., Sonnhammer, E. L. Kalign-an accurate and fast multiple sequence alignment algorithm. BMC bioinformatics. 6 (1), 298 (2005).
- Clamp, M., Cuff, J., Searle, S. M., Barton, G. J. The jalview java alignment editor. Bioinformatics. 20 (3), 426-427 (2004).
- Waterhouse, A. M., Procter, J. B., Martin, D. M., Clamp, M., Barton, G. J. Jalview Version 2-a multiple sequence alignment editor and analysis workbench. Bioinformatics. 25 (9), 1189-1191 (2009).
- Farrow, B., et al. A chemically synthesized capture agent enables the selective, sensitive, and robust electrochemical detection of anthrax protective antigen. ACS Nano. 7 (10), 9452-9460 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten