Method Article
Biochemische und strukturelle Charakterisierung von Kohlenhydrat Transport Substrat-Bindeprotein SP0092
In diesem Artikel
Zusammenfassung
Eine optimierte Protokoll für die Durchführung einer umfangreichen biochemischen und strukturelle Charakterisierung von ein Kohlenhydrat-Substrat-Bindeprotein von Streptococcus Pneumoniae wird vorgestellt.
Zusammenfassung
Entwicklung von neuen Antibiotika und Impfstoffe für Streptococcus Pneumoniae (Pneumokokken) sind notwendig, um den raschen Anstieg der multiplen Resistenzen zu stoppen. Kohlenhydrat-Substrat-bindende Proteine (SBPs) stellen praktikable Ziele für die Entwicklung von Protein-basierten Impfstoffen und neue Antibiotika wegen ihrer extrazellulären Lokalisierung und die Zentralität des Kohlenhydrat-Import für Pneumokokken Stoffwechsel, bzw.. Hier beschriebene ist ein rationalisierter integrierte Protokoll, eine umfassende Charakterisierung der SP0092, die aus der Pneumokokken und andere Bakterien auf andere Kohlenhydrat SBPs erweiterbar durchzuführen. Dieses Verfahren kann helfen, die Struktur-basierte Design-Inhibitoren für diese Klasse von Proteinen. Präsentiert im ersten Teil dieser Handschrift sind Protokolle für die biochemische Analyse durch thermische Verschiebung Assay, Multi Winkel Lichtstreuung (MALS) und Größe Ausgrenzung Chromatography (SEC), die die Stabilität optimieren und Homogenität der Probe gerichtet Kristallisation Studien und so erhöhen die Wahrscheinlichkeit des Erfolgs. Der zweite Teil dieses Verfahrens beschreibt die Charakterisierung der SBP Kristalle mit einer abstimmbaren Wellenlänge anomale Beugung Synchrotron Strahlrohr und Datenprotokolle Sammlung für Messdaten, die verwendet werden können, das kristallisierte Protein lösen Struktur.
Einleitung
Streptococcus Pneumoniae (Pneumokokken) ist ein Gram-positiven Bakterium mit Wohnsitz asymptomatisch in den oberen Luftwegen der menschlichen Atemwege mit der Fähigkeit, normalerweise sterilen Nischen verursacht Otitis, Lungenentzündung, Sepsis, Blutvergiftung, Migration und Meningitis1,2. Pneumokokken-Infektion ist übrigens die häufigste Ursache für ambulant erworbener Pneumonie, die zu einer klinischen und wirtschaftlichen Belastung weltweit3,4beiträgt. Antibiotika-resistente Stämme von S. Pneumoniae rund um den Globus ausgebreitet haben und obwohl eine sieben-Valent und ein 13-Valent Pneumokokken Protein konjugierte Impfstoff geholfen haben reduzieren die Rate von Antibiotikaresistenzen, Ersatz-Stämme aus Impfstoff sind entstanden und führten zu erhöhten Anforderungen für die Forschung in der Entwicklung von neuen Therapien gegen Pneumokokken-Erkrankungen5,6,7,8.
Die Pneumokokken hängt von Zucker aus den Host als CO2-Quelle9,10importiert; in der Tat wendet es 30 % seiner Import Maschinen für den Transport von 32 verschiedenen Kohlenhydrate11,12,13. Diese Importeure sind mindestens acht ABC-Transportern13. In ABC-Transporter spielen die extrazelluläre SBPs eine wesentliche Rolle bei der Bestimmung der Spezifität für die Liganden und fester Membran-Transporter für die Aufnahme in die Zelle zu präsentieren. SBPs repräsentieren gültige Ziele für die Gestaltung von neuen Impfstoffen und Antibiotika, weil sie Oberflächenproteine und ihre entscheidende Rolle bei der zellulären Prozesse sind.
Ziel Protein Charakterisierung und detaillierte Beschreibung der strukturellen Merkmale wie Liganden Taschen und domänenübergreifendes Flexibilität bieten ein nützliches Werkzeug für Struktur-based Drug Design14,15. Röntgen-Kristallographie ist die Methode der Wahl für die strukturelle Charakterisierung von Proteinen in der Nähe von atomarer Auflösung, aber die Kristallisation ist unberechenbar, zeitaufwendig und nicht immer erfolgreich. Systematische Methoden haben die Erfolgsquote verbessert und wichtige Faktoren sind die Sample-Qualität und Stabilität. Die Erfolgsquote bei der Kristallisation wird durch das Protein chemische Eigenschaften und Probe Vorbereitung Methode beeinflusst. Die Wirkung dieser werden bewertet und durch biochemische Charakterisierung16,17informiert.
Eine weitere Komplikation für Struktur-basiertes Design ist das Problem der kristallographischen Phase, die angegangen werden müssen. Sobald mehr Protein-Strukturen vorliegen haben, können viele Strukturen durch die molekulare Ersetzungsmethode erfordern eine homologe Struktur18behoben werden. Wie SBPs eine flexible Domänenstruktur präsentieren, könnte molekulare Ersatz auch anspruchsvolle19. Ist ein strukturelles Modell, das ähnlich genug, um das Zielprotein ist nicht verfügbar, kann eine Reihe von Techniken verwendet werden, um die experimentelle Phasenabgleich20zu erhalten. Unter diesen Einzel-Wellenlänge anomale Dispersion (SAD) Methode hat als die wichtigste Technik entstanden und wurde ausgiebig genutzt, um die Phase Problem21zu lösen. Die Verwendung der traurig-Methode hat weiter mit Verbesserungen bei der Hardware und Software sowie Daten-Sammlung-Strategien ermöglichen die Erkennung und Nutzung von schwachen anomale Signale zur schrittweisen22,23, 24. Darüber hinaus Fortschritte in der direkten Methoden für die Lösung von Strukturen der Makromoleküle, die in der Vergangenheit Beugung Daten zu atomarer Auflösung benötigt, jetzt genutzt werden kann, durch die Kombination von z. B. stereochemische wissen, wie im Programm implementiert ARCIMOLDO25. Eine nützliche Überprüfung der Methoden zur Lösung des Problems der Phase in der Kristallographie wird von Taylor26gegeben.
Hier präsentieren wir eine rationalisierte Protokoll zur Charakterisierung von Kohlenhydrat Transport SBP, SP0092 von S. Pneumoniae, biochemische und strukturelle Techniken (Abbildung 1) zu integrieren. Dieses schrittweise Protokoll bietet ein nützliches Beispiel Test Case von Strategien, die Erfolgsquote der strukturelle Studien über SBPs im Allgemeinen zu verbessern, die in allen Königreichen des Lebens zu finden sind. Insbesondere das Protokoll unterstreicht die Bedeutung der Charakterisierung der stabilsten Oligomeren Zustand des Proteins in Lösung in eine schnelle und effektive Methode, und erlaubt die Identifikation der besten Arten, für Kristallisation Experimente zu folgen. Zwar gibt es über 500 SBP-Strukturen in der Protein Data Bank27berichtet, kann molekulare Ersatz aufgrund der inhärenten flexible Natur der beiden Domänen α/β, verbunden durch ein Scharnier Region19schwierig sein. So beschreibt der zweite Teil des Protokolls mit der traurigen Methode für Ausstieg aus gebundenen Metall-Ionen, die häufig in SBPs sowie die Einbeziehung von Selenomethionin und Verwendung von Selen (Se) ist in traurige Ausstieg.
Protokoll
Hinweis: die kodierende Sequenz, für die das Signalpeptid gelöscht, wird im Anschluss an eine standard-Fusion-Protokoll pOPINF Vektor geklont; das native Protein wird als eine His-Tag-Fusion in Escherichia coli BL21 Rosetta Zellen ausgedrückt 28 , 29. die Variante mit der Bezeichnung Selenomethionin drückt sich nach Standardmethoden laut Hersteller 30. Die rekombinante SBP ist wie zuvor gereinigt beschrieben 31 , 32.
1. biochemische Charakterisierung
- thermische Verschiebung Assay
- bereiten 48 Pufferlösungen mit einem pH-Wert von 4.0 bis zu 9,5 und NaCl-Konzentration von 0 bis 0,5 M und 40 µL in eine 96-Well-Platte zu verzichten, wie in beschrieben Tabelle 1 33.
- verleihen jedem gut 5 µL der SBP Lösung in 1-2 mg/mL Konzentration.
- Verleihen jedem gut 5 µL 20 x fluoreszierende Fleck für Proteine (siehe Tabelle der Materialien).
- Mischen die Lösungen durch pipettieren, Dichtplatte mit einer transparenten Klebefolie und spin für 2 min bei 112 X g bei Raumtemperatur.
- Führen Sie das Experiment auf einer Echtzeit-Maschine: Rampen-Temperatur von 3 ° C/min von 4 bis 99 ° C mit einem 10 s Haltezeit eingestellt, und lesen Sie der Fluoreszenz jeder 0,5 ° C mit Erregung bei 483 nm und Emission bei 568 nm.
- Analysieren die Fluoreszenz-Emission-Kurven für jeden Puffer Zustand, die Schmelztemperatur (T m) als das Minimum der Ableitung zu identifizieren.
Hinweis: Diese Prozedur gibt einen guten Überblick über die besten Pufferlösung zur Verbesserung der Proteinstabilität Unterstützung. Wählen Sie den pH-Bereich und die Salzkonzentration auf der Grundlage der Puffer Bedingung mit der höchsten T-m und Hinzufügen von 2,5 % (V/V) Glycerin und 0,5 mM tris(2-carboxyethyl) Phosphin (DÄMMUND) zu definieren, die Pufferlösung für die folgenden Schritte (SEC-Puffer).
- MALS und analytische SEC
- führen Sie eine zweispaltige Volumen Waschen der abgepackten Gel Filtration Spalte mit entgastem gefiltertes Wasser (verwenden Sie eine breite Palette Molekulargewicht 24 mL Spalte).
- Verbinden Sie die Spalte mit der Lichtstreuung-Detektor und die Spalte mit dem ermittelten SEC-Puffer von der thermischen Shift-Test equilibrate.
- Injizieren 100 µL der gereinigten SBP bei 5 mg/mL in das äquilibriert SEC Spalte und Flow eine Spalte Volumen der SEC-Puffer.
- Überprüfen Sie die Elution Chromatogramm für einzelne oder mehrere Gipfel und die Streuung Daten mit Hilfe der Analysesoftware, Erlangung der molaren Masse und der Polydispersität Index für alle Tierarten.
Hinweis: Für jeden Peak als Oligomere Art entspricht, es ist vorteilhaft für die Polydispersität prüfen Index berechnet als molare Masse auf Masse/molare Masse gewichtete Anzahl der Moleküle (Mw/Mn) gewichtet. Ein Polydispersität Wert nahe bei 1 zeigt einen Prozess Höhepunkt.
Hinweis: Sobald die Oligomerisierung Arten definiert werden, es empfiehlt sich, ihre Abhängigkeit von Proteinkonzentration, studieren wie höhere Konzentrationen größer Oligomer Bildung begünstigen könnte. - Führen Sie mehrere analytische SEC läuft; 100 µL der gereinigten SBP bei steigenden Konzentrationen (0,1 - 10 mg/mL) in die äquilibriert Gel Filtration Spalte (verwenden Sie eine breite Palette Molekulargewicht 5 mL Spalte) zu injizieren und fließen eine Spalte Volumen von SEC-Puffer jedesmal.
- Der Elution Chromatogramm für einzelne oder mehrere Gipfel prüfen und analysieren die Intensitäten der Absorption bei 280 nm der Kurven, verschiedene Start Proteinkonzentrationen entspricht. Wenn die relative Intensität der verschiedenen Peaks konstant bleiben, ist keine inter Konvertierung zwischen den Oligomeren Arten vorhanden. Variation in der relativen Intensitäten bei verschiedenen Konzentrationen ist ein Indiz für den Wechsel zwischen den verschiedenen Oligomeren Spezies, die Konzentration des Proteins abhängig ist.
Hinweis: Dieser Schritt Charakterisierung ist von grundlegender Bedeutung zu definieren, welche Arten für Kristallisation besser geeignet sind. In der Tat unterschiedliche Prozess Arten sind anfälliger für kristallisieren, wenn sie in einem stetigen Oligomere Zustand in einer definierten Konzentration sind.
2. Protein-Vorbereitung und Kristallisation
- präparative SEC
- nach einer Spalte Volumen waschen mit entgastem gefiltertes Wasser, equilibrate mit SEC-Puffer die präparativen vorverpackte Gel Filtration Spalte (zahlreiche molekulare Gewicht 120 mL Spalte).
- Injizieren 1-5 mL gereinigtes SBP bei 5-50 mg/mL in der äquilibriert Gel Filtration Spalte und Strömung eine Spalte Volumen von SEC-Puffer.
- Der Spalte Durchflussverfahren in 1 mL Fraktionen sammeln.
- Bündeln die Fraktionen entsprechend zu einer einzigen Monodisperse Spitze; drehen Sie die Probe in 15 mL Zentrifugation Filter bei 1.500 g und die Konzentration zu quantifizieren, bis die gewünschte Konzentration erreicht ist (in der Regel 50-100 mg/mL für SP0092).
- Kristallisation
- bestimmen die optimale Konzentrationsbereich für die Kristallisation Experiment mit der Pre-Kristallisation zu testen, wie vom Hersteller beschrieben:
- Dispense 0,5 - 1,0 mL eines jeden der vier Kristallisation Test Lösungen in einem anderen Behälter einer 24-Well-Sitzung fallen Kristallisation Platte.
- Bereiten die Kristallisation Tropfen durch das Mischen von 1 µL Proteinlösung mit 1 µL Reservoir-Lösung, die Dichtplatte und Inkubation bei Raumtemperatur für nicht weniger als 1 h (zuverlässigere Ergebnisse nach der Inkubation über Nacht).
- Prüfen Sie die Drop-Qualität für jede Konzentration mit einem 10-fach Vergrößerung Mikroskop.
Hinweis: Testen Sie mindestens drei verschiedene Proteinkonzentration: (I) die meisten der verbleibenden klare Tropfen zeigen, dass das Protein zu verdünnen für Kristallisation; (II) die Anwesenheit des schweren Niederschlags in der Mehrzahl der Tropfen impliziert eine übermäßig hohe Konzentration; und (III) eine ausgewogene Auftreten von klaren Tropfen und Niederschlag (besser, wenn das Licht) ist ein guter Indikator, dass die getestete Konzentration günstig für die Kristallisation ist.
- Bereiten die 96-Well-Sitzung Drop Kristallisation Platten; 100 µL der verschiedenen kommerziellen Kristallisation Lösung in jedem Behälter gut 34 , 35 , zu verzichten 36 , 37.
- verzichten 100 nL Protein Probe bei der zuvor definierten Konzentration (Schritt 1.1.6) in alle Kristallisation Drop Brunnen mit einem Robotersystem Kristallisation.
- Verzichten 100 nL des verschiedenen Reservoirs Lösungen für die entsprechenden Kristallisation fallen Brunnen mit Protein verzichtet in Schritt 2.2.3 mischen. Die Kristallisation Dichtplatte, um Verdunstung zu vermeiden und ermöglichen die Gleichgewichtherstellung der Kristallisation Tropfen mit dem Reservoir.
- Überprüfen Sie die Tropfen in regelmäßigen Abständen (anfangs alle 1-2 Tage, später wöchentlich) mit einem 10 X (mindestens) Vergrößerung Mikroskop Kristallbildung und Wachstum bewerten.
Hinweis: Fällt ein automatisiertes imaging System für Protein Kristallisation verwendet werden, für die kann Visualisierung und Erfassung.
- bestimmen die optimale Konzentrationsbereich für die Kristallisation Experiment mit der Pre-Kristallisation zu testen, wie vom Hersteller beschrieben:
- Kristall Montage
- bereiten die Kryoprotektivum Lösung durch Zugabe von 25 % (V/V) Glycerin (Endkonzentration) zur Kristallisation Bedingung (also als Ersatz für 25 % des Wassers in der ersten Gemisch) 38.
- Füllen einen Schaum Dewar mit flüssigem Stickstoff und Gehäuse Musterteil ein Uni-Puck in der Dewar. Flüssigem Stickstoff Temperatur abkühlen lassen.
- Ausschneiden und entfernen die Abdichtung mit Klebeband von der Kristallisation Platte über die Tropfen, wo die Kristalle gebildet werden.
- Geben Sie einen Tropfen von 1 µL Kryoprotektivum Lösung auf eine Coverslide in der Nähe des Ziel-Drop positioniert.
- Übertragen die ausgewählten Kristall aus dem ursprünglichen Drop auf das Kryoprotektivum Lösung Tropfen mit einer Nylon-Cryo-Schleife auf eine Wirbelsäule Standardbasis montiert auf einem magnetischen Zauberstab 39 , 40.
- Den Kristall aus dem Kryoprotektivum Drop schnell zu übertragen, zu flüssigem Stickstoff, indem die Schleife in die erste leere Position von der Uni-Puck-Probenhalter.
- (Von der Abdichtung Band geschnittenen Schritt) zu wiederholen, bis alle gewünschten Kristalle sind geerntet und in der Uni-Puck-Probenhalter gespeichert.
- Ort der Uni-Puck basieren auf den Uni-Puck mit dem Puck Zauberstab und nehmen den Puck in das Strahlrohr (unter flüssigem Stickstoff Bedingungen).
Vorsicht: Extrem niedrige Temperatur! - Verwenden die Cryo-Zange und den Puck Dewar laden Tool der Uni laden-Puck(s) in das Strahlrohr Probe Wechsler Roboter Dewar. Der Uni-Puck-Probenhalter löst sich vom verlassen der Schleife Probenhalter aufrecht in der Probe Roboter Basis Deckel Dewar und damit der flüssige Stickstoff ausgesetzt und der Probe Wechsler Roboter zugänglich.
- Kristall Charakterisierung
Hinweis: die geernteten Kristalle können dann gesiebt, Beugung Qualität verwenden entweder eine Labor Röntgenquelle oder eine Röntgenquelle Synchrotronstrahlung (SR). Makromolekularen Kristallographie (MX) Beamlines bieten eine einstellbaren Energiequelle um anomale Beugung für Struktur-Lösung zu nutzen. Im folgenden Abschnitt eine Reihe von Arbeitsanweisungen in Übereinstimmung mit den experimentellen Fähigkeiten von Diamond Light Source MX Beamlines vorgeschlagen, aber diese Richtlinien können auch MX Beamlines an anderen Synchrotrone weltweit angepasst werden.- Als ein erster Schritt ist es sinnvoll, entweder bestätigen oder anomalen Streusignal im Kristall zu identifizieren. Dies kann bequem erreicht werden, durch die Messung eine Fluoreszenz Röntgenspektrum aus Kristall. Zunächst sicherstellen, dass die Röntgenenergie das Strahlrohr ist hoch genug, um alle Elemente in der Regel erwartet für makromolekulare Kristalle begeistern eingestellt (14 keV oder höher).
- Verwenden die Strahlrohr-Steuerungs-Software, die Stichprobe der Probe Wechsler Roboter montiert werden; die Schleife wird automatisch zentriert in der Röntgenstrahl und Zentrierung der Kristall kann manuell über die Steuerungssoftware Strahlrohr bestätigt werden.
- Aufzeichnen ein Röntgenspektrum Fluoreszenz mit dem Strahlrohr-Steuerungs-Software: der Benutzer wählt eine Belichtungszeit und initiiert die Messung (Fluoreszenz/Fluoreszenz-Spektrum-Einstellung, → laufen, Abb. 2A). Die Steuerungssoftware Strahlrohr positioniert automatisch das Röntgen Fluoreszenz-Detektor im Ort und bestimmt die minimale Vorfall x-ray Flux um eine lesbare Signal auf der Fluoreszenz-Detektor zu erhalten. Das erworbene Spektrum wird dann analysiert in einer automatisierten Weise mit Emission Spitzen ausgestattet, um natürlich vorkommende biologische Elemente. Diese Routinen sind in das Strahlrohr Control Software grafische Benutzeroberfläche (GUI) - GDA (www.opengda.org) an unserer Firma und an Synchrotrons in ganz Europa 41 , 42.
- , Wenn geeignete Elemente identifiziert werden, die für eine anomale Beugung Experiment ausgenutzt werden kann, führen Sie einen x-ray Absorption Rand Energie Scan ermitteln die optimale Wellenlänge für anomale Beugung Datenerhebung aus dem Crystal: auf das Strahlrohr Steuerungs-Software, wählen Sie das Element und den Scan zu initiieren (Fluoreszenz/Fluoreszenz-Scan-Einstellung, → Atom Namen → laufen, Abb. 2 b).
Hinweis: Für ein einzelnes anomale Beugung-Experiment, dem Höhepunkt der Absorptionskante dient die anomale Streuung zu maximieren. Experimentelle Überlegungen zur Durchführung einer Multi-Wellenlänge anomale Beugung Experiments wurden bereits detaillierte 43. - Zu bestimmen, die Kristall-Einheit Zellenparameter, die Symmetrie und die Beugungsgrenze, Messen drei x-ray Diffraction Muster in 45° Abständen mit der Oszillation-Methode (Data Collection/Screening → laufen aktuelle, Abbildung 2). Die Strahlrohr-Steuerungs-Software bietet dem Anwender mit einer Auswahl der Oszillation Winkel und Belichtungszeit und die Möglichkeit, die prozentuale Übertragung der Röntgenstrahl festzulegen. Ein standard-Quarz-Screening Oszillation Winkel von 0,5 °, eine Belichtungszeit von 0,5 s und 5 % Röntgentransmission Strahl empfohlen. Die gemessenen Beugung-Bilder werden automatisch analysiert, indem die EDNA-Pipeline und wieder eine Reihe von Strategien für die Sammlung von einem vollständigen Datensatz 22.
- X-ray Datensammlung
Hinweis: der Benutzer kann auswählen, sammeln Daten im standard Oszillation oder inverse Strahl Modus, wodurch Friedel Kumpels schließen in Zeit und Strahlendosis und mit etwa der gleichen Absorptionsverhalten aufgezeichnet werden ermöglicht eine genauere Messung der anomalen Unterschiede. Letzteres ist nützlich, besonders wenn kleine anomale Unterschiede dürften und/oder die Proben Strahlung empfindlich sind bei der Durchführung einer anomalen Beugung-Experiment. Überlegungen über die besten Daten-Sammlung-Strategien verwenden wurden umfassend überarbeitete 22 , 23 , 44 , 45 , 46 , 47.- mit dem Strahlrohr Steuerungssoftware importieren Daten Auflistung Parameter wie das Strategieprogramm, das zur Verfügung stellt:
i. Startposition der ω-Drehachse
Ii. Oszillation Breite der ω-Achse Drehung Winkel für jedes Bild Beugung
Iii. Belichtungszeit für jedes Bild Beugung
iv. Anzahl der Bilder für ein komplettes Dataset (indirekt Definition des gesamten ω-Achse Drehwinkels für die gesamte Datensammlung)
v. Prozentsatz der Dämpfung der Röntgenstrahl auf Überbelichtung und Strahlung Schäden zu vermeiden
Hinweis: an unserem Institut für ausgeführt wird die Datensammlung, die Software-Pipelines für die automatisierte Verarbeitung der gesammelten Daten etabliert sind: (i) die Beugung Daten sinken (indiziert, integrierte und skalierte) von der xia2-Pipeline, erzeugt die Reflexion Mtz-Dateien für den Benutzer als Input für Ausstieg und Struktur Lösung/Verfeinerung 48. (Ii) Wenn ein signifikantes anomales Signal erkannt wird während der Datenanalyse, eine erste schnelle automatisierte Struktur Lösung Pipeline (Fast_ep) mit SHELX Versuche, die die schweren Atom-Unterkonstruktion durch experimentelle Ausstieg, eine stufenweise Elektron Dichte Karte soweit möglich. Eine zweite umfassendere Struktur Lösung Pipeline verpflichtet sich automatisch versuchen zu lösen und die Structu bauenRe mit unabhängigen Software-Suiten 49 , 50 , 51 , 52; in Fällen, wo dies erfolgreich ist, wird der Benutzer mit einer anfänglichen Modell und Elektron Dichtekarte bereitgestellt werden. Dies bildet die Grundlage für den Benutzer Verfeinerung und bestätigen das Modell mit der kristallographischen Softwarepakete Wahl.
- mit dem Strahlrohr Steuerungssoftware importieren Daten Auflistung Parameter wie das Strategieprogramm, das zur Verfügung stellt:
Ergebnisse
Dieses integrierte Protokoll nachgewiesen wurde, um mit vier erfolgreich zu sein (zwei veröffentlicht und zwei unveröffentlichte Strukturen) von sechs Kohlenhydrat Protein Verbindlichkeit von Pneumokokken32,53bisher analysiert. In diesem Abschnitt präsentieren wir Ihnen die biochemische und strukturelle Charakterisierung von SP0092 als ein repräsentatives Ergebnis, strukturelle Studien der SBPs im Allgemeinen zu führen.
Nachdem die SBP-SP0092 zum Ausdruck gebracht und gereinigt wie zuvor32definiert wurde, wurde gereinigte Protein für Puffer-Stabilität über eine thermische Verschiebung Assay analysiert: SP0092 weist eine verstärkte Tm bei pH 6,5 und in Gegenwart von NaCl im Bereich 0 - 0,2 M Konzentrationsbereich (Abb. 3A). Vor diesem Hintergrund wurde die Pufferlösung für die nachfolgenden Schritte definiert als: 0,02 M MES pH 6,5, 0,2 M NaCl, 2,5 % (V/V) Glycerin, 0,5 mM DÄMMUND. Die absolute molare Masse der verschiedenen Oligomerisierung Zustände des SP0092 wurde gemessen, indem MALS gekoppelt an SEC ein Molekulargewicht von 187.2, 140,8 und 97,0 49,4 kDa Messung entspricht tetrameres, Trimere dimeres und Monomeren Arten bzw. () Abb. 3 b). Die SEC-Profil bei verschiedenen Protein Verdünnungen ergab, dass die Oligomerisierung durch erhöhte Proteinkonzentration ausgelöst wird, was darauf hindeutet, dass die größeren Oligomere stabiler bei höheren Konzentrationen als in der Regel gewöhnt sind Kristallisation. In der Tat, gereinigten größeren Oligomere Sorte gerichtet zur Kristallisation Studien erfolgreich produzierten Proteinkristallen zwar das Monomer Arten nicht.
Die optimierte Kristalle aus dem einheimischen gewonnen und Se-Methionin beschriftet Formen der SP0092 zeichneten sich durch Röntgenbeugung. Messung der Röntgen-Fluoreszenz von diesen Kristallen enthüllt in beiden Fällen Emissionen Gipfel für Zn wird an das Protein gebunden, während Se nur für die Se-Methionin Kristalle wie erwartet erkannt wurde. Später wurden x-ray Absorption Scans an den Se und Zn Kanten durchgeführt die vorausgesetzt direkten experimentelle Daten die einfallende Röntgen-Wellenlänge zu den jeweiligen x-ray Absorption Rändern des Zn oder Se Tune in den Kristallen, die anomale Signal zu maximieren, aus den daraus resultierenden Daten (Abbildung 4A–B) erhältlich.
Nach drei Beugungsmuster bei niedrigen Transmission zu messen, wurde ein komplettes Set für anomales Daten erhalten mit Hilfe der Daten-Sammlung-Strategie vorgeschlagen von EDNA (Abbildung 4). Das anomale anliegende Signal löst die automatisierte Phasenabgleich Pipeline an das Strahlrohr an die Unterkonstruktion zu bestimmen und anhand der experimentellen Anfangsphase abgeleitet, produziert erste Karten und Modelle, die dann verfeinert und validiert werden kann (Abbildung 4 ).
Zusammenfassend lässt sich sagen werden potenzielle Fallstricke der Technik in erster Linie auf Kristall-Verfügbarkeit und Qualität zentriert. Optimierung der Pufferbedingungen Proteinstabilität zu verbessern sowie Identifizierung von den am besten geeigneten Oligomere Zustand des Proteins zu verwenden, wenn mehr als ein Oligomer in Lösung identifiziert wird, kann das Risiko eines Ausfalls bei der Kristallisation reduzieren. Bühne. Nutzung den Einsatz von gebundenen Metall-Ionen in einem frühen Stadium erkannt Struktur Lösung beschleunigen und vermeiden unnötige Produktion von Selenomethionin Protein beschriftet, wenn molekulare Ersatzmethoden Versagen.
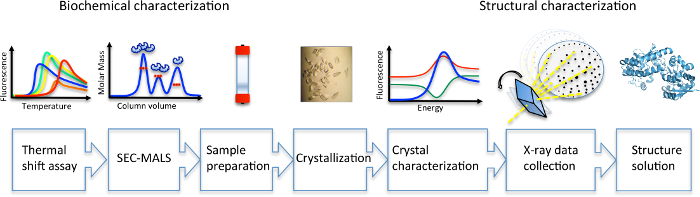
Abbildung 1: Diagramm des Workflows für biochemische und strukturelle Charakterisierung von Kohlenhydraten SBPs. Bitte klicken Sie hier für eine größere Version dieser Figur.
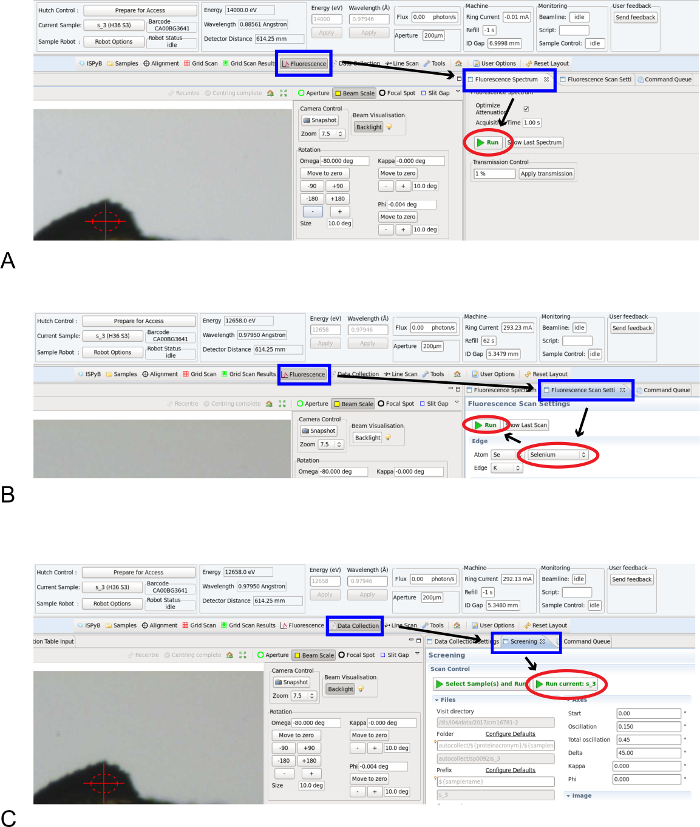
Abbildung 2: Kristall Charakterisierung in GDA. (A) Screenshot der Registerkarte Röntgen-Fluoreszenz Kontrolle der GDA Strahlrohr Steuerungssoftware. (B) Screenshot der Registerkarte der x-ray für Energie Rand-Scannen in GDA. (C) Screenshot von der Registerkarte Daten Sammlung Kristall Screening in GDA. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3. Biochemische Charakterisierung der SP009239-491. (A) 3D-Oberfläche Graphen Plotten die Schmelztemperatur der SP009239-491 als eine Funktion des pH-Wertes und NaCl-Konzentration der Pufferlösung. (B) SEC und MALS Ergebnisse für SP009239-491. Absorption bei 280 nm ist in blau dargestellt. Die Molmassen der verschiedenen Oligomerisierung Staaten werden in rot angezeigt. Zentrale (B) wurde von32geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
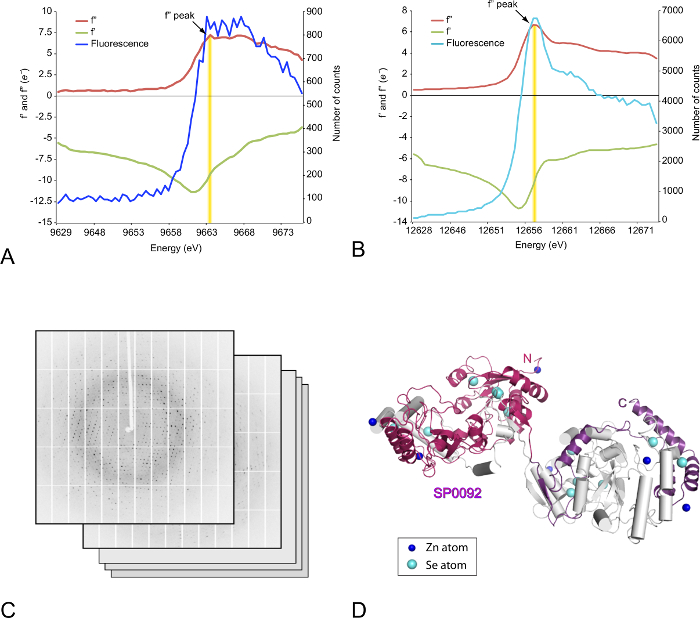
Abbildung 4. Strukturelle Charakterisierung von SP009239-491. (A) und (B) x-ray Absorption Energie am Rande Zn und Se für SP0092-39-491 -Kristalle zu scannen. Die gemessenen Fluoreszenz von Zn und Se mit Kristallen erscheint in blau und Cyan, die berechneten f' und f "anomale Streuung Brüche sind in grün und rot, jeweils. (C) Beispiele für x-ray Diffraction Muster von SP009239-491 Kristalle gesammelt. (D) Cartoon Darstellung der SP009239-491 Dimer Struktur. Ein Protomer ist weiß eingefärbt und den anderen Magenta (Rückstände 39-366) und violett (Rückstände 367-491), und die anomale Streuung Atome werden als blau und Cyan Kugeln für Zn und Se, bzw. angezeigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
| 0.1 M Zitronensäure pH 4.0 | 0.1 M Zitronensäure pH 4,5 | 0,1 M Phosphat pH 5,0 | 0,1 M Citrat pH 5,5 | 0,1 M Bis-Tris pH 6 | 0,1 M ADA pH 6,5 | 0,1 M MOPS pH 7 | 0,1 M HEPES pH 7,5 | 0,1 M Imidazol pH 8.0 | 0,1 M Tris pH 8,5 | 0,1 M CHES pH 9 | 0,1 M CHES pH 9.5 | |
| NaCl 0 M | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL |
Tabelle 1. Komposition für thermische Verschiebung Assay-Puffer.
Diskussion
In diesem Papier wir beschreiben und eine integrierte Protokoll für biochemische und strukturelle Charakterisierung von Kohlenhydrat SBPs mit besonderem Schwerpunkt auf Proteine von S. Pneumoniaezu validieren. Dennoch kann dies als Standardverfahren für die Analyse von anderen SBPs von verschiedenen Organismen und auch andere unabhängige lösliche Proteine verwendet werden.
Der erste Teil des Protokolls konzentriert sich auf die biochemischen Informationen über Proteinstabilität und quartäre Struktur, die bei der Vorbereitung der Proteinproben für Kristallisation ausgenutzt werden können. Im Bereich thermische Verschiebung Assays beschreiben wir nur den pH-Wert und NaCl-Konzentration-Variationen zu den allgemeinen Charakter dieses Verfahrens zu erhalten. Trotz dieser, viele andere Pufferbedingungen können getestet werden in ähnlicher Weise, z. B. einschließlich chemische Verbindung, die als ein stabilisierendes Additiv verwendet: insbesondere die tatsächliche Ligand(s), die an einem bestimmten SBP binden sind besonders wirksam bei der Erhöhung der Tm von wenigen Grad Celsius31. In einigen Fällen können die denaturierenden Kurven schlecht definiert werden, durch low-Signal oder eine hohe Fluoreszenz-Hintergrund, hervorgerufen durch Proteinaggregation oder partielle Entfaltung. Um dies zu vermeiden, kann ein Protein: Farbstoff Titration durchgeführt werden, um die unklaren denaturierenden Kurven zu optimieren. Wenn keine Besserung erreicht wird, empfiehlt sich die screening-verschiedene Zusatzstoffe, die die Stabilität des Proteins zu verbessern, können und geeignete Bildschirme wurden zuvor beschriebenen54.
In der Regel die meisten SBPs sind Monomere in ihrer natürlichen Umgebung, aber wie hier gezeigt, dass Multimerization in den höheren Konzentrationen Kristallisation Experimente auftreten kann, so ist die Oligomerisierung Verhalten Charakterisierung von MALS und SEC zur Verfügung gestellt wesentlich für den günstigsten stabile Monodisperse Oligomerisierung stand für Kristallisation zu bewerten. Dennoch ist es schwer vorherzusagen, die Wirkung von verschiedenen Chemikalien in die Kristallisation Bedingung auf die Oligomerisierung Verhalten der Proteine enthalten. Wenn die SEC und MALS Prüfung umfangreiche Ansammlung von der Protein-Probe zeigt, empfehlen wir die folgenden zur Verringerung der Wahrscheinlichkeit dieses auftritt: verwenden Sie frische Eiweiß-Beispiel (nicht einfrieren aufgetaut) und erweitern die durchgeführte mit thermischen Stabilisierung-Analyse Shift-Assays, Tests möglich Zusatzstoffe und milde Reinigungsmittel als letztes Mittel, um Aggregation zu minimieren. In diesem Papier stellen wir grundlegende Richtlinien für die Kristallisation mit Hochdurchsatz-dünnbesetzte Matrix kommerzielle Kristallisation Screening auf um den allgemeinen Charakter dieses Protokolls zu erhalten. Jedoch erhalten hochauflösende Röntgenbeugung Proteinkristallen möglicherweise iterative Feinabstimmung um die Kristallisation Bedingungen bezüglich Fällungsmittel Konzentration, pH-Wert, Zusatz von chemischen Zusätzen, unterschiedliche Temperaturen, zu optimieren und andere Faktoren, die sich verändernde Gleichgewicht Dynamik zwischen den Kristall Tropfen und Stausee16,17.
Der zweite Teil des Protokolls beschreibt die Charakterisierung von Proteinkristallen um die optimale Strategie für x-ray Diffraction Datenerfassung mit einem besonderen Schwerpunkt auf den Erwerb der anomale Daten für traurige Ausstieg zu definieren. Auch wenn SBPs eine ähnliche allgemeine Architektur pflegen (und es viele hinterlegte 3D Strukturen potenziell nutzbare gibt als Ausgangspunkt Modelle), Ausstieg dieser Proteine durch die molekulare Ersetzungsmethode ist nicht immer einfach aufgrund der Variabilität von der Elemente der Sekundärstruktur und die innere Flexibilität dieser Proteine. Daher, wir schlagen die traurige Methode und betonen, dass diese Proteine bereits intrinsisch Metalle oder in der Tat unspezifische Bindung von Metallen aus der Kristallisation Pufferbedingungen gebunden haben kann die eine Palette von anomalen Interferenzgitters Elemente wie anbieten kann eine Standard-Schritt in unserer allgemeinen Protokolls.
Zusammenfassend, definiert dieses Protokoll einen standard geführte Workflow Verfahren ermöglicht die detaillierte Beschreibung der biochemischen und strukturellen Merkmale der SBPs, die ausgenutzt werden können, um die Struktur Entschlossenheit Erfolgsquote zu erhöhen, als auch zu beschleunigen die strukturelle Charakterisierung der SBPs im Allgemeinen.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir erkennen OPPF-UK für Hilfe beim Klonen, Gemma Harris für SEC-MALLS und die Wissenschaftler Beamlines I03 und I04 bei Diamond Light Source.
Materialien
| Name | Company | Catalog Number | Comments |
| SelenoMethionine Medium Complete | Molecular Dimensions | MD12-500 | Based on a synthetic M9 minimal media supplemented with glucose, vitamins and amino acids with the exception of L-methionine. Other equivalent products are commercially available by other companies. |
| MicroAmp Optical 96-Well plate | Applied Biosystems | 4306737 | The Applied Biosystems MicroAmp Optical 96-Well Reaction Plate with Barcode is optimized to provide unmatched temperature accuracy and uniformity for fast, efficient PCR amplification. This plate, constructed from a single rigid piece of polypropylene in a 96-well format, is compatible with Applied Biosystems® 96-Well Real-Time PCR systems and thermal cyclers. |
| SYPRO Orange | Molecular Probes | S6651 | SYPRO Orange Protein Gel Stain is a sensitive, ready-to-use fluorescent stain for proteins in 1D gels. Quite universal and well-established protein dye for hydrophobic regions. |
| MicroAmp Optical Adhesive film | Applied Biosystems | 4311971 | The Applied Biosystems MicroAmp Optical Adhesive Film reduces the chance of well-to-well contamination and sample evaporation when applied to a microplate. It is ideal for optical measurement, because it gives low background. |
| 7500 Fast Real-time PCR System | Applied Biosystems | 4362143 | The Applied Biosystems 7500 Fast Real-Time PCR System enables standard 96-well format high speed thermal cycling, significantly reducing your run time for quantitative real-time PCR applications, delivering results in about 30 minutes. The Upgrade Kit is available to upgrade a standard 7500 Real-Time PCR System to the Fast Configuration via a field service installation. Other RT-PCR machine can be used. Data export not easy for the old data analysis software. |
| Superdex 200 increase 10/300 GL column | GE Healthcare | 28990944 | Superdex 200 Increase 10/300 GL is a versatile, prepacked column for size exclusion chromatography in small-scale (mg) preparative purification as well as for characterization and analysis of proteins with molecular weights between 10 000 and 600 000, such as antibodies. Optimal separation ideal for high resolution biophysical techniques. |
| DAWN HELEOS II | Wyatt | DAWN HELEOS II is the premier Multi-Angle static Light Scattering (MALS) detector for absolute characterization of the molar mass and size of macromolecules and nanoparticles in solution. The DAWN offers the highest sensitivity, the widest range of molecular weight, size and concentration, and the largest selection of configurations and optional modules for enhanced capabiliites. Other MALS detecting systems from other companies apart from Wyatt have not been tested, so no additional feedback can be provided. | |
| Superdex 200 5/150 GL | GE Healthcare | 28906561 | Superdex 200 are prepacked size exclusion chromatography columns for high-resolution small-scale preparative and analytical separations of biomolecules. Superdex 200 has a separation range for molecules with molecular weights between 10 000 and 600 000. The peak separation is not as optimal as for the "increase" version but this model is ideal for the standard day by day use. |
| HiLoad 16/600 Superdex 200 pg | GE Healthcare | 28989335 | HiLoad 16/600 Superdex 200 prep grade are prepacked XK columns designed for high-resolution preparative gel filtration chromatography. |
| 24 Well "Big" Sitting Drop Crystallization Plate | MiTeGen | XQ-P-24S-A | The 24 well "big" crystallization plate is used mainly for protein crystal screening by sitting drop vapor diffusion techniques, and for crystallization condition optimization. It has quite large reservoir well and sample container, which favor manual handling and big sized protein crystal growth. In addition, its flat surfaces are easily to be sealed by transparent tape or cover slips. |
| PCT Pre-Crystallization Test | Hampton | HR2-140 | The PCT Pre-Crystallization Test is used to determine the appropriate protein concentration for crystallization screening. |
| 96 Well CrystalQuick | Greiner bio-one | 6098xx | Square-well plates have three crystallization wells per reservoir, making it possible to test 288 samples per plate. Generally used only for the inital screening because their squared edge make crystals fishing difficult. |
| Uni-Puck | Molecular Dimensions | MD7-601 | The Universal V1-Puck (Uni-puck) is a sample pin storage and shipping container for use with the majority of automated sample mounting systems worldwide - includes the ACTOR, SAM and CATS systems amongst others (Diamond, Soleil, SPring-8, Photon Factory, CLSI and across the USA) |
| Standard Foam Dewar | Molecular Dimensions | MD7-35 | 5.7" diameter by 2.8" deep. 800mL capacity. |
| Mounted CryoLoop - 20 micron | Hampton | HR4-955 | Mounted CryoLoops with 20 micron diameter nylon. These nylon loops are bonded to hollow, stainless steel MicroTubes™ that are used to mount, freeze, and secure the crystal during cryocrystallographic procedures and X-ray data collection. Different sizes exist and they can be adapted in lenght. They are quite versatile tools. |
| CryoWand | Molecular Dimensions | MD7-411 | |
| Puck dewar loading tool | Molecular Dimensions | MD7-607 | This tool is used to separate uni-pucks to load them into the robot dewar. It consists of two pieces: a Teflon tube part and a metal rod part. |
Referenzen
- Austrian, R. Prevention of pneumococcal infection by immunization with capsular polysaccharides of Streptococcus pneumoniae: current status of polyvalent vaccines. J Infect Dis. 136, S38-S42 (1977).
- Bogaert, D., De Groot, R., Hermans, P. W. Streptococcus pneumoniae colonisation: the key to pneumococcal disease. Lancet Infect Dis. 4 (3), 144-154 (2004).
- van Mens, S. P., et al. Longitudinal analysis of pneumococcal antibodies during community-acquired pneumonia reveals a much higher involvement of Streptococcus pneumoniae than estimated by conventional methods alone. Clin Vaccine Immunol. 18 (5), 796-801 (2011).
- Weycker, D., Strutton, D., Edelsberg, J., Sato, R., Jackson, L. A. Clinical and economic burden of pneumococcal disease in older US adults. Vaccine. 28 (31), 4955-4960 (2010).
- Doern, G. V. Antimicrobial use and the emergence of antimicrobial resistance with Streptococcus pneumoniae in the United States. Clin Infect Dis. 33, S187-S192 (2001).
- Vasoo, S., et al. Increasing antibiotic resistance in Streptococcus pneumoniae colonizing children attending day-care centres in Singapore. Respirology. 16 (8), 1241-1248 (2011).
- Black, S., et al. Efficacy, safety and immunogenicity of heptavalent pneumococcal conjugate vaccine in children. Northern California Kaiser Permanente Vaccine Study Center Group. Pediatr Infect Dis J. 19 (3), 187-195 (2000).
- Hanage, W. P., et al. Diversity and antibiotic resistance among nonvaccine serotypes of Streptococcus pneumoniae carriage isolates in the post-heptavalent conjugate vaccine era. J Infect Dis. 195 (3), 347-352 (2007).
- Burnaugh, A. M., Frantz, L. J., King, S. J. Growth of Streptococcus pneumoniae on human glycoconjugates is dependent upon the sequential activity of bacterial exoglycosidases. J Bacteriol. 190 (1), 221-230 (2008).
- King, S. J. Pneumococcal modification of host sugars: a major contributor to colonization of the human airway?. Mol Oral Microbiol. 25 (1), 15-24 (2010).
- Tettelin, H., et al. Complete genome sequence of a virulent isolate of Streptococcus pneumoniae. Science. 293 (5529), 498-506 (2001).
- Buckwalter, C. M. Pneumococcal carbohydrate transport: food for thought. Trends Microbiol. 20 (11), 517-522 (2012).
- Bidossi, A., et al. A functional genomics approach to establish the complement of carbohydrate transporters in Streptococcus pneumoniae. PLoS One. 7 (3), e33320 (2012).
- Zheng, X., Gan, L., Wang, E., Wang, J. Pocket-based drug design: exploring pocket space. AAPS J. 15 (1), 228-241 (2013).
- Singh, S., Malik, B. K., Sharma, D. K. Molecular drug targets and structure based drug design: A holistic approach. Bioinformation. 1 (8), 314-320 (2006).
- Dessau, M. A., Modis, Y. Protein crystallization for X-ray crystallography. J Vis Exp. (47), (2011).
- Bergfors, T. . Protein crystallization: techniques, strategies, and tips. A laboratory manual. , (1999).
- Scapin, G. Molecular replacement then and now. Acta Crystallogr D Biol Crystallogr. 69 (Pt 11), 2266-2275 (2013).
- Mao, B., Pear, M. R., McCammon, J. A., Quiocho, F. A. Hinge-bending in L-arabinose-binding protein. The "Venus's-flytrap" model. J Biol Chem. 257 (3), 1131-1133 (1982).
- McCoy, A. J., Read, R. J. Experimental phasing: best practice and pitfalls. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 458-469 (2010).
- Dodson, E. Is it jolly SAD?. Acta Crystallogr D Biol Crystallogr. 59 (Pt 11), 1958-1965 (2003).
- Incardona, M. F., et al. EDNA: a framework for plugin-based applications applied to X-ray experiment online data analysis. J Synchrotron Radiat. 16 (Pt 6), 872-879 (2009).
- Popov, A. N., Bourenkov, G. P. Choice of data-collection parameters based on statistic modelling. Acta Crystallogr D Biol Crystallogr. 59 (Pt 7), 1145-1153 (2003).
- Terwilliger, T. C., et al. Can I solve my structure by SAD phasing? Planning an experiment, scaling data and evaluating the useful anomalous correlation and anomalous signal. Acta Crystallogr D Struct Biol. 72 (Pt 3), 359-374 (2016).
- Millan, C., Sammito, M., Uson, I. Macromolecular ab initio phasing enforcing secondary and tertiary structure. IUCrJ. 2 (Pt 1), 95-105 (2015).
- Taylor, G. L. Introduction to phasing. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 325-338 (2010).
- Berman, H. M., et al. The Protein Data Bank. Nucleic Acids Res. 28 (1), 235-242 (2000).
- Bird, L. E., et al. Green fluorescent protein-based expression screening of membrane proteins in Escherichia coli. J Vis Exp. (95), e52357 (2015).
- Berrow, N. S., et al. A versatile ligation-independent cloning method suitable for high-throughput expression screening applications. Nucleic Acids Res. 35 (6), e45 (2007).
- Hendrickson, W. A., Horton, J. R., LeMaster, D. M. Selenomethionyl proteins produced for analysis by multiwavelength anomalous diffraction (MAD): a vehicle for direct determination of three-dimensional structure. EMBO J. 9 (5), 1665-1672 (1990).
- Culurgioni, S., Harris, G., Singh, A. K., King, S. J., Walsh, M. A. Structural Basis for Regulation and Specificity of Fructooligosaccharide Import in Streptococcus pneumoniae. Structure. , (2016).
- Culurgioni, S., Tang, M., Walsh, M. A. Structural characterization of the Streptococcus pneumoniae carbohydrate substrate-binding protein SP0092. Acta Crystallogr F Struct Biol Commun. 73 (Pt 1), 54-61 (2017).
- Ericsson, U. B., Hallberg, B. M., Detitta, G. T., Dekker, N., Nordlund, P. Thermofluor-based high-throughput stability optimization of proteins for structural studies. Anal Biochem. 357 (2), 289-298 (2006).
- Wooh, J. W., Kidd, R. D., Martin, J. L., Kobe, B. Comparison of three commercial sparse-matrix crystallization screens. Acta Crystallogr D Biol Crystallogr. 59 (Pt 4), 769-772 (2003).
- Gorrec, F. Protein crystallization screens developed at the MRC Laboratory of Molecular Biology. Drug Discov Today. 21 (5), 819-825 (2016).
- Jancarik, J., Scott, W. G., Milligan, D. L., Koshland, D. E., Kim, S. H. Crystallization and preliminary X-ray diffraction study of the ligand-binding domain of the bacterial chemotaxis-mediating aspartate receptor of Salmonella typhimurium. J Mol Biol. 221 (1), 31-34 (1991).
- Newman, J., et al. Towards rationalization of crystallization screening for small- to medium-sized academic laboratories: the PACT/JCSG+ strategy. Acta Crystallogr D Biol Crystallogr. 61 (Pt 10), 1426-1431 (2005).
- Garman, E. F., Mitchell, E. P. Glycerol concentrations required for cryoprotection of 50 typical protein crystallization solutions. Journal of Applied Crystallography. 29 (5), 584-587 (1996).
- Teng, T. -. Y. Mounting of crystals for macromolecular crystallography in a free-standing thin film. Journal of Applied Crystallography. 23 (5), 387-391 (1990).
- Cipriani, F., et al. Automation of sample mounting for macromolecular crystallography. Acta Crystallogr D Biol Crystallogr. 62 (Pt 10), 1251-1259 (2006).
- Gabadinho, J., et al. MxCuBE: a synchrotron beamline control environment customized for macromolecular crystallography experiments. J Synchrotron Radiat. 17 (5), 700-707 (2010).
- Leonard, G. A., et al. Online collection and analysis of X-ray fluorescence spectra on the macromolecular crystallography beamlines of the ESRF. Journal of Applied Crystallography. 42 (2), 333-335 (2009).
- Walsh, M. A., Evans, G., Sanishvili, R., Dementieva, I., Joachimiak, A. MAD data collection - current trends. Acta Crystallogr D Biol Crystallogr. 55 (Pt 10), 1726-1732 (1999).
- Dauter, Z. Data-collection strategies. Acta Crystallogr D Biol Crystallogr. 55 (Pt 10), 1703-1717 (1999).
- Finke, A. D., et al. Advanced Crystallographic Data Collection Protocols for Experimental Phasing. Methods Mol Biol. 1320, 175-191 (2016).
- Gonzalez, A. Optimizing data collection for structure determination. Acta Crystallogr D Biol Crystallogr. 59 (Pt 11), 1935-1942 (2003).
- Leslie, A. G. The integration of macromolecular diffraction data. Acta Crystallogr D Biol Crystallogr. 62 (Pt 1), 48-57 (2006).
- Winter, G., Lobley, C. M., Prince, S. M. Decision making in xia2. Acta Crystallogr D Biol Crystallogr. 69 (Pt 7), 1260-1273 (2013).
- Sheldrick, G. M. Experimental phasing with SHELXC/D/E: combining chain tracing with density modification. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 479-485 (2010).
- Skubak, P., Pannu, N. S. Automatic protein structure solution from weak X-ray data. Nat Commun. 4, 2777 (2013).
- Terwilliger, T. C., et al. Decision-making in structure solution using Bayesian estimates of map quality: the PHENIX AutoSol wizard. Acta Crystallogr D Biol Crystallogr. 65 (Pt 6), 582-601 (2009).
- Vonrhein, C., Blanc, E., Roversi, P., Bricogne, G. Automated structure solution with autoSHARP. Methods Mol Biol. 364, 215-230 (2007).
- Culurgioni, S., Harris, G., Singh, A. K., King, S. J., Walsh, M. A. Structural Basis for Regulation and Specificity of Fructooligosaccharide Import in Streptococcus pneumoniae. Structure. 25 (1), 79-93 (2017).
- Boivin, S., Kozak, S., Meijers, R. Optimization of protein purification and characterization using Thermofluor screens. Protein Expression and Purification. 91 (2), 192-206 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten