Method Article
Messung der Interaktionen von Kugelsternhaufen und filamentösen Proteinen Kernresonanzspektroskopie (NMR) und Microscale Thermophorese (MST)
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen ein Protokoll für die Herstellung und Reinigung von Proteinen, die gekennzeichnet sind mit stabilen Isotopen und anschließende Charakterisierung von Protein-Protein-Wechselwirkungen mit Spektroskopie der Kernspinresonanz (NMR) und MicroScale Thermophorese (MST) Experimente.
Zusammenfassung
Filamentöse Proteine wie Vimentin bieten Organisation innerhalb der Zellen durch die Bereitstellung einer strukturellen Gerüst mit Websites, die mit Plakin Proteine binden wiederholt. Hier ist ein Protokoll zur Erfassung und Messung solcher Wechselwirkungen beschrieben mit kugelförmigen Plakin Wiederholung Bereich der Envoplakin und die spiralförmige Spule Vimentin. Dies bietet eine Grundlage zur Feststellung, ob ein Protein Vimentin (oder ähnliche filamentöse Proteine) bindet und für die Messung der Affinität der Interaktion. Die kugelförmige Protein des Interesses ist beschriftet mit 15N und titriert mit Vimentin Protein in Lösung. Eine zweidimensionale NMR-Spektrum wird erworben, Interaktionen zu erkennen, durch die Beobachtung von Veränderungen der Peakform oder chemische Veränderungen und Auswirkungen der Lösungsbedingungen einschließlich Salzgehalte, die Vimentin quartäre Struktur beeinflussen zu erhellen. Wenn das Protein des Interesses den filamentösen Liganden bindet, wird die Bindung Interaktion durch MST mit der gereinigten Proteine quantifiziert. Der Ansatz ist eine einfache Möglichkeit zur Feststellung, ob ein Protein des Interesses ein Filament bindet und für die Beurteilung, wie Änderungen, wie Mutationen oder Lösungsbedingungen, beeinflussen die Interaktion.
Einleitung
Wechselwirkungen zwischen Proteinen ermöglichen die Bildung von molekularen Maschinen, die Ordnung in den Zellen zu schaffen. Die einzelnen Interaktionen sind oft schwach sondern in der Regel zur multivalente komplexe, die kooperative und dynamisch reguliert werden können. Sensible Assays, die atomarer Auflösung und quantitative Informationen über solche komplexen Interaktionen bieten genügen, um Mechanismen ableiten und Interventionen wie Drogen wie Moleküle zu entwerfen. NMR-Spektroskopie ist eine effiziente Methode zur Erlangung dieser Informationen über Protein-Interaktionen und auch für schnelle Screening für Liganden, einschließlich derer, die schwach1binden verwendet wird. Die NMR Methoden können kategorisiert werden, in denen, die sind Protein beobachten oder Liganden zu beobachten. Dieses Manuskript verwendet das erste Verfahren, in dem ein Spektrum an ein Protein, das vergleichsweise gering (in der Regel unter 20 kDa) ist mit der Bezeichnung Stall-Isotop wird erworben und die unbeschriftete Liganden titriert. Die beschrifteten Rückstände wird dadurch die Interaktion zugeordnet werden, in günstigen Fällen beteiligt. Einmal die komplexen Formen, gibt es Änderungen in der chemischen Umgebungen von interagierenden Rückstände, die sich als Änderungen in der chemischen Verschiebung manifestieren und Form ihrer NMR-Signale. Das Ausmaß dieser Veränderungen korreliert mit dem Grad der Einbeziehung dieser Gruppen in der Interaktion. Chemische Verschiebung Störungen (CSPs) können gemessen werden, durch den Vergleich einer Reihe von NMR-Spektren des Proteins in Abwesenheit und Anwesenheit von unterschiedlichen Mengen des Liganden gesammelt. Für größere Liganden oder komplexe Interaktionen kann die Änderung der Peak-Form oder Intensität gemessen werden Interaktionen ableiten.
Das am weitesten verbreitete 2D Experiment zur Erfassung Ligand Interaktionen ist die 15N-Heteronuclear einzelnen Quanten Korrelation (HSQC) Experiment2. Dies erfordert, dass ein Protein gleichmäßig mit 15N, bezeichnet werden, die in der Regel erzielt wird, indem zum Ausdruck zu bringen als Affinität-Tags Versionen in E. Coli Bakterienkulturen in 15N angereicherte Medien gewachsen. Bindung ist offensichtlich, wenn die HSQC-Spektren, gesammelt während der Titration überlagert werden, enthüllt Peak Änderungen für einen Teil der Rückstände in der Komplexbildung einbezogen. Die Interaktion kann im schnellen Austausch Regime auftreten, wo die freie und Liganden gesättigt Zustandssignalen in einer Bevölkerung im Durchschnitt Spitze zusammenbrechen. Alternativ sind bei langsamen Austausch zwischen den Staaten, beide Signale mit integrale beobachtet, die ihre relativen Mengen darstellen. Während NMR linienförmiges Analyse verwendet werden kann, um die verbindliche Affinitäten in einigen Fällen zu schätzen Methoden wie MST haben auch praktisch bewährt und bieten Kreuzvalidierung echte Interaktionen.
Das Beispiel ist von zwei Proteinen innerhalb Desmosomes gefunden. Sie vermitteln Kreuzungen zwischen Zelloberflächen und dem Zytoskelett und vermitteln multivalente Interaktionen zwischen Zelle Adhäsion Maschinen und fortgeschrittene Filamente, die Integrität der Haut und Herz Gewebe und widerstehen der Scherkräfte. Krankheiten können entstehen, wenn Desmosomal Proteine wie z. B. Desmoplakin oder Vimentin durch Mutationen oder Autoantikörper gefährdet sind, führt zu der Zell-Zell-Verbindungen, Destabilisierung und deren Wechselwirkungen daher von entscheidender Bedeutung3 sind. Die strukturelle Basis der Ligand-Bindung durch Desmosomal Proteine kann durch NMR-Spektroskopie charakterisiert werden, während die Wechselwirkungen von MST quantifiziert werden können. Hierin wurden Methoden charakterisieren die Wechselwirkungen zwischen Plakin repeat Domänen (PRDs) die oft als Tandem-Sets vorhanden sind, die grundlegenden Rillen anbieten und Vimentin, eine intermediate Filament, das durch eine saure Oberfläche angeboten durch die spiralförmige interagiert Bundle4. Diese komplexe bilden sich an der Zellmembran wo sie Anker für fortgeschrittene Filamente der Zelle Cytoskelett, Desmosomes, die eine Verbindung zu benachbarten Zellen bilden so ein Netzwerk von Klebeverbindungen, die durch ein Gewebe ausstrahlt.
Protokoll
(1) rekombinante Proteinexpression
-
Ausdruck von Envoplakin PRD (E-PRD) und Vimentin 99-249 (VimRod)
- E. Coli BL21(DE3) Zellen mit dem Plasmid enthält das gewünschte gen zu verwandeln. Die Zellen auf Agarplatten mit 100 µg/mL Ampicillin zu verbreiten. Über Nacht inkubieren Sie die Platten bei 37 ° C.
- Wählen Sie eine einzelne Kolonie und impfen Sie 20 mL Super Brühe (TB), enthält 100 µg/mL Ampicillin, wählen für das Plasmid zu. Wachsen Sie die Kultur bei 37 ° C mit schütteln (180 u/min) über Nacht.
- Übertragen Sie die gesamten 20 mL Kultur auf 1 L des TB mit 50 µg/mL Ampicillin. Inkubation die Kultur bei 37 ° C mit Schütteln bei 180 u/min bis die OD600 = 0,6-0,8.
- Reduzieren Sie die Temperatur auf 18 ° C und induzieren Sie Protein-Expression durch Zugabe von Isopropyl β-D-1-Thiogalactopyranoside (IPTG), eine Endkonzentration von 1 mM zu. Weiter Inkubation bei 18 ° C mit Schütteln bei 160 u/min über Nacht Proteinexpression ermöglichen.
- Ernte der Zellen durch Zentrifugieren der Kultur bei 8.000 x g für 15 min. Dekantieren und den Überstand verwerfen.
- Waschen der geernteten Zellen durch resuspending der Zelle Pellet in ca. 40 mL von Phosphat gepufferte Kochsalzlösung (PBS: 20 mM Phosphatpuffer, pH 7,4, 120 mM NaCl). Übertragen Sie die Wiederfreisetzung auf eine 50 mL-Tube. Zentrifuge wieder bei 8.000 x g für 15 Minuten.
- Dekantieren und den Überstand verwerfen. Sofort beginnen Sie die Reinigung-Protokoll oder frieren Sie die Zelle Pellets bei-20 ° C zur späteren Verwendung ein.
-
Expression von isotopisch beschriftete Protein
- Verwandeln Sie E. Coli BL21(DE3) Zellen zu, und bereiten Sie eine 20 mL Starterkultur wie in 1.1.1-1.1.2.
- Übertragen Sie die gesamten 20 mL Kultur auf 1 L des angereicherten TB mit einer zusätzlichen 4,0 g Tryptone, 5,0 g NaCl und 100 µg/mL Ampicillin. Inkubation die Kultur bei 37 ° C mit Schütteln bei 160 u/min bis die OD600 = 1,6 – 1,9.
- 1 L Kultur durch Zentrifugation bei 8000 X g für 15 min. Dekantieren ernten und den Überstand verwerfen.
- Waschen Sie die Zelle Pellet durch sanft resuspending in ca. 40 mL PBS und übertragen Sie die Wiederfreisetzung auf eine 50 mL-Tube.
- Zentrifugieren Sie wieder bei 8.000 x g für 15 min. Dekantieren und verwerfen Sie den überstand.
- Aufzuwirbeln Sie die Zelle-Pellets in 20 mL M9 minimal Medien (Tabelle 1) und die Übertragung auf den Rest des 950 mL M9 minimal Medien enthält 100 µg/mL Ampicillin.
- Fügen Sie 50 mL Filter sterilisiert Nährstoff-Mix (Tabellen 2 und 3).
- Akklimatisieren Sie die Kultur auf 18 ° C für 30 min bevor Sie hinzufügen eine Endkonzentration von 1 mM IPTG.
- Über Nacht bei 18 ° C mit Schütteln bei 160 u/min inkubieren.
- Ernten Sie die Zellen wie in Abschnitt 1.1.4-1.1.6.
(2) immobilisiert Metall Affinität Chromatographie (IMAC) Reinigung des VimRod und des E-PRD
-
Reinigung des His6-Tags VimRod
- Aufschwemmen der Zelle Pellet in 5 mL/g von PBS, eine Protease Inhibitor cocktail fehlen EDTA enthalten. Homogenisieren Sie mit 12 Schlägen in einem Dounce Gewebe Homogenisator, die Zelle Lyse im folgenden Schritt zu verbessern.
- Auf dem Eis beschallen die Zellsuspension auf einen Impuls von 1 s/1 s aus, 80 % Amplitude für eine Gesamtmenge von 1,5 min. wiederholen die Beschallung zwei zusätzliche Zeiten, wirbelnden sanft auf Eis zwischen läuft, um eine Überhitzung zu verhindern.
- Zentrifugieren Sie die Probe auf 75.000 x g für 45 min. Dekantieren und Filtern des Überstands mit einer Spritze Filter (0,45 µm).
- Equilibrate eine 5 mL-IMAC-Säule mit 5 Spalte Volumen (CV) der Bindung Puffer (20 mM HEPES, pH 7.5, 500 mM NaCl, 10 mM Imidazol) bei einer Durchflussmenge von 1 mL/min mit einem schnellen Protein Flüssigkeitschromatographie (FPLC) System.
- Laden Sie die gefilterten Überstand auf die Säule mit einer Durchflussrate von 0,5 mL/min.
- Waschen Sie die Spalte mit 5 CV von waschen Puffer (20 mM HEPES, pH 7.5, 500 mM NaCl, 50 mM Imidazol) bei einer Durchflussmenge von 1 mL/min.
- Das Protein mit 3 eluieren CV der Elution Puffer (20 mM HEPES, pH 7.5, 500 mM NaCl, 350 mM Imidazol) bei einer Fließgeschwindigkeit von 0,5 mL/min sammeln 1,5 mL Fraktionen. Falls vorhanden, wählen Sie den Up-Flow Elution Modus um die Konzentration der eluierten Proteins zu erhöhen.
- Identifizieren Sie die Fraktionen aus der FPLC Chromatogramm, die enthalten des Proteins des Interesses durch SDS-PAGE und standard-Methoden verwenden, um die Protein-Konzentration-5messen.
- Bündeln Sie und konzentrieren Sie die Elution Brüche mit den höchsten Mengen an Protein mit einem zentrifugalen Ultrafiltration Gerät (MWCO 3 kDa, 5 mL), 2 mL. Zentrifugieren Sie bei 21.000 x g jeder Niederschlag zu entfernen und durch einen 0,22-μm-Filter übergeben.
- Eine 120 mL Größe Ausgrenzung (S) Chromatographiesäule mit 2 CV S Puffer (20 mM HEPES, 150 mM NaCl, pH 7.5, 0,5 mM DÄMMUND) bei einer Durchflussmenge von 1 mL/min mit einem FPLC Equilibrate.
- Das konzentrierte Protein aus 2.1.9 auf die Säule zu injizieren und eluieren mit 1 CV S-Puffer mit einer Durchflussrate von 0,5 mL/min, 1 mL Fraktionen zu sammeln.
- Identifizieren Sie die Brüche, enthält das Protein des Interesses als vor.
- Bündeln Sie die Fraktionen, die die höchsten Mengen Eiweiß enthalten.
- Lagerung bei 4 ° C für den kurzfristigen Einsatz oder 20 % und bei-80 ° C in kleinen Aliquote Glycerin hinzufügen.
-
Reinigung des E-PRD Protein mit dem His6 Tag entfernt
- Folgen Sie den Schritten im 2.1.1-2.1.8 His6 tagged E-PRD Protein zu reinigen. Die Peak-Fraktionen bündeln und Konzentration des Proteins zu bestimmen.
- Fügen Sie Tabak Ätzen Virus (TEV) Protease (1 mg/mL) bei 2 µL/mg gepoolten Protein. Transfer zur Dialyse (6 kDa) Schläuche und Dialyse in S Puffer über Nacht bei 4 ° c Dieser Schritt ermöglicht es Spaltung des His6 Tags und Entfernung von Imidazol, die Bindung an das Ni-NTA-Harz im nächsten Schritt stören wird.
- Equilibrate 5 mL Ni-NTA-Harz in einer Schwerkraft-Spalte mit 3 CV S Puffer. Abfluss überschüssigen Puffer aus dem Harz.
- Gießen Sie das gespalten E-PRD-Protein auf das Harz und inkubieren Sie für 1 Stunde auf eine rockende Plattform, uncleaved His6 tagged E-PRD und gespalten His6 Tag zu binden. Der TEV-Protease ist auch His6 markiert und wird das Harz zu binden. Sammeln der Durchströmung, die enthält die Tag-freie E-PRD. Waschen des Harzes mit 2 CV S Puffer, um sicherzustellen, dass alle E-PRD wird wiederhergestellt.
- E-PRD in der Durchströmung zu 2 mL mit einem zentrifugalen Ultrafiltration Gerät (MWCO 3 kDa, 5 mL) zu konzentrieren. Zentrifugieren Sie bei 21 000 x g jeder Niederschlag zu entfernen und durch einen 0,22-μm-Filter übergeben.
- Eine 120 mL S Spalte mit 2 CV S Puffer (20 mM HEPES, 150 mM NaCl, pH 7.5, 0,5 mM DÄMMUND für MST oder 20 mM Tris-HCl, 1 mM DTT, pH 7 für NMR) mit einer Durchflussrate von 1 mL/min mit einem FPLC Equilibrate.
- Das konzentrierte E-PRD-Protein aus 2.2.5 auf die Säule zu injizieren und eluieren mit 1 CV S-Puffer mit einer Durchflussrate von 0,5 mL/min, 1 mL Fraktionen zu sammeln.
- Identifizieren Sie die Brüche, enthält das Protein des Interesses als vor.
- Bündeln Sie die Fraktionen, die die höchsten Mengen Eiweiß enthalten.
- Lagerung bei 4 ° C für den kurzfristigen Einsatz oder 20 % und bei-80 ° C in kleinen Aliquote Glycerin hinzufügen.
(3) NMR Methoden
- NMR-Probenvorbereitung
- 15N-mit der Bezeichnung Wildtyp oder R1914E E-PRD Protein wie zuvor beschrieben, mit 20 mM Tris-HCl, 1 mM DTT, pH-Wert 7 als die S-Zwischenspeicher für Schritt 2.2.6 zu reinigen. Protein Stammlösungen reichen in der Regel von 0,3 bis 1 mM mit einem Volumen von etwa 1 mL.
Bemerkung: Protein kann auf > 100 µM mit einem MWCO 3 kDa, 5 mL zentrifugale Ultrafiltration Gerät bringen die Konzentration in einem geeigneten Bereich für die Probenvorbereitung. - Eine Probe der unbeschrifteten VimRod Protein mit 20 mM Tris-HCl, 1 mM DTT, pH-Wert 7 als die S-Zwischenspeicher für Schritt 2.1.10 zu reinigen.
- Fügen Sie in einem Endvolumen von 500 µL Wildtyp oder mutierte E-PRD-Protein eine Endkonzentration von 100µM, Deuteriumoxid (D2O), eine Endkonzentration von 10 % (V/V) und DSS (4,4-dimethyl-4-silapentane-1-sulfonic Säure), eine Endkonzentration von 20 µM. Bring Das Probenvolumen bis zu 500 µL mit 20 mM Tris-HCl, 1 mM DTT, pH 7. Eine repräsentative Stichprobe Vorbereitung ist in Tabelle 4beschrieben.
Hinweis: 0 ppm Resonanz des DSS wird verwendet, um die 1H chemische Verschiebungen zu kalibrieren, sowie für die indirekte Referenzierung der 15N chemischen Verschiebungen von der Protein-6. D2O wird für das Deuterium-Sperre-Signal verwendet, um das Spektrometer auf ein konstantes Magnetfeld net zu halten. - Eine zweite Probe von E-PRD, D2O und DSS wie im vorherigen Schritt machen und eine Endkonzentration von 50 µM vor einer Klage das Volumen bis zu 500 µL VimRod hinzufügen.
- Übertragen Sie die 500 µL Proben auf einen 5 mm breiten NMR Röhrchen für das Experiment.
- 15N-mit der Bezeichnung Wildtyp oder R1914E E-PRD Protein wie zuvor beschrieben, mit 20 mM Tris-HCl, 1 mM DTT, pH-Wert 7 als die S-Zwischenspeicher für Schritt 2.2.6 zu reinigen. Protein Stammlösungen reichen in der Regel von 0,3 bis 1 mM mit einem Volumen von etwa 1 mL.
- NMR-Versuchsaufbau
- Schalten Sie den Luftstrom mit dem Auswerfen Befehl "Ej"; Dies öffnet die Probe vom Magneten. Nun legen Sie die Probe in ein Spinner auf den Magneten durch die Öffnung und setzen Sie mit dem Befehl "Ij". Warten Sie, bis die Probe setzt sich im Inneren des Magneten, bevor Sie fortfahren.
- Erstellen Sie ein neues Dataset mithilfe des Befehls "Edc" und laden Sie standard 1H NMR Parameter wählen Sie Experiment "ZGPR" (Abbildung 1). NAME, EXPNO (Experimentnummer) und PROCNO (verarbeiteten Daten Ordnernummer) Felder ausfüllen. Wählen Sie das Lösungsmittel im Feld "Set Lösungsmittel" und klicken Sie auf "Ausführen 'Getprosol'" standard Probehead und Lösungsmittel (Prosol) Parameter zu lesen.
- Verriegeln Sie die Probe auf die deuterierte Lösungsmittel, z. B. D2O, mit Befehl "Sperren" und warten Sie, bis es fertig ist pauschal und Sperre erreicht.
- Korrigieren Sie die Resonanzfrequenz des Magneten durch tuning der Probe mit dem automatischen tuning-Befehl "Atma". Überwachen Sie die Wobble-Kurve, bis die automatische Abstimmung abgeschlossen ist.
- Shim das Magnetfeld mit TOPSHIM (Befehl "Topshim"). Shim ist Anpassungen, Magnetfeld, Gleichmäßigkeit um die Probe zu erreichen. Es empfiehlt sich, die Shim-Werte mit dem Befehl "Wsh" speichern und lesen Sie sie mit "Rsh" vor Topshim, wenn die gleiche oder ähnliche Beispiele verwenden.
- Passen Sie die Empfänger-Verstärkung mit "Rga"-Befehl, um maximale Signal-Rausch-Verhältnis zu erreichen.
- Legen Sie der Mitte des Spektrums auf der Wasser-Resonanz-Offset (o1) und legen Sie die 90-Grad-Proton-Puls (p1) bei hoher Leistung mit "calibo1p1".
- Sammeln der Proton-Spektrum mit der Null gehen "Zg" Befehl und Prozess mit "Efp" umfasst exponentielle Vermehrung ("Em"), die freie Induktion Decay (FID) Einbeziehung Linie zu erweitern, "ft" Fourier-Transformation der FID und "Pk" Phase anwenden Korrektur.
- Gelten Sie die automatische Phase Korrektur "Apk" und die automatische Basislinienkorrektur "Absn" über das Polynom ohne Integrationsmöglichkeit.
- Erstellen Sie ein neues Dataset (wie in 3.2.2) für das SOFAST HMBC-Experiment durch Auswahl von "SFHMQC3GPPH" im Experiment.
- Kopie P1 und O1 von Proton Spektrum optimiert und P1 abhängigen Impulse mit Befehl "Getprosol 1H p1 plw1", wo p1 ist der optimierte P1-Wert und plw1 ist die Leistungsstufe P1 zu füllen.
- Optimieren Sie die CNST54-konstante um den Offset für Amid chemische Verschiebung festzulegen und CNST55, die Bandbreite zu definieren, um den Spektralbereichen von Interesse zu erfassen, wodurch den Empfänger Gewinn sein optimiert (Abbildung 2). Um diese Parameter zu wählen, extrahieren Sie die erste FID (free Induction Decay) aus dem zweidimensionalen Spektrum zu und suchen Sie das beobachtete Signal, sie zu definieren. Darüber hinaus variieren Sie die Entspannung Verzögerung (D1), Anzahl der Scans (NS) und Dummy-Scans (DS) akzeptabel Signalempfindlichkeit mit Befehl "gs", erhalten die ermöglicht gehen und scannen, um die Qualität der Daten in Echtzeit zu überwachen.
- Notieren Sie die Spektren mit Null Go "Zg".
- NMR-Daten-Verarbeitung
- Festlegen der Verarbeitungsparameter auf die Größe des direkten F2 (1H) und indirekte F1 (15N) Dimensionen des Spektrums mit "SI F2 = 2048, F1 = 512" mit optionalen lineare Vorhersage in indirekte Dimension (Abbildung 3).
- Wählen Sie "QSINE" als die Fensterfunktion und geben Sie eine Sinus-Glocke-Verschiebung (SSB) 2, das zweidimensionale Spektrum zu verarbeiten.
- Geben Sie den Befehl "Xfb" zur Verarbeitung der Daten in beide Richtungen mit Fenster-Funktion und Fourier-Transformation.
- Verwenden Sie den Befehl "apk2d" automatische Phasenkorrektur in beide Richtungen durchführen. Wenn der automatische Prozess kein zufriedenstellendes Niveau der Phasenkorrektur erreicht, FIDs mit dem Befehl "Rser" extrahieren Phase Werte von 1D Verarbeitung zu berechnen und auf die 2D Daten anwenden.
- Korrigieren Sie die Grundlinie mit der automatischen Grundlinie Korrekturfunktion "abs2" für 2D Daten. Dies gilt eine Polynomfunktion zwischen der ppm-Werte in den Verarbeitungsparametern definiert und erzeugt ein 2D Spektrum zur weiteren Analyse.
- Falls Serienverarbeitung zum Vergleich von Interaktionsdaten mit einem anderen Molekül durchführen, speichern Sie die Verarbeitungsparameter mit dem Befehl "Wpar" und erinnern sie mit "aufgerufenen". Auf diese Weise können alle Datensätze mit gleichen Parametern und Variationen verarbeitet werden werden nicht eingeführt werden, aufgrund der Verarbeitung unterschieden.
- NMR-Daten-Analyse
- Geben Sie die Befehl "pp" um mit der Spitze pflücken beginnen.
- Definieren Sie die ppm Bereich und mindestens Intensitätsmaximum/Anzahl der Peaks anhand der erwarteten Gipfel (Abbildung 4). Klicken Sie auf "OK" und überprüfen Sie die Ergebnisse durch Sichtkontrolle. Bei Bedarf erneut führen Sie aus, der Prozess bis Ergebnisse zufriedenstellend sind anhand von Spektren Qualität.
- Erzeugen Sie eine Peaklist mit dem Befehl "pp".
Hinweis: Dieses Peaklist enthält Daten Höhe/Peak Intensität Informationen standardmäßig und zur anschließenden Spektren exportiert werden kann und von anderen Programmen gelesen werden kann. - Beobachten Sie Veränderungen in der Peak-Intensitäten oder Bewegung in chemischen Verschiebungen in den Protein-HSQC--Spektren, die Interaktion mit einem anderen Molekül angeben. Wenn die interagierende Moleküle groß ist, erwarten Sie Kürzungen in Spitzenzeiten Intensitäten zusammen mit Verschwinden der einige Gipfel.
- Importieren Sie die Peak-Liste auf den nächsten Datensatz durch Klicken auf die Registerkarte "Peaks" und "importieren" auswählen mit der rechten Maustaste im Fenster "Gipfel".
- Visualisieren Sie die Gipfeln über das Spektrum und bei Bedarf an eine neue Position verschieben. Klicken Sie auf "Zurücksetzen Intensitäten" für "komplette Table", eine Peaklist für das Spektrum mit Intensitäten (Abbildung 5) zu generieren. Dieser Gipfel-Liste wird über die Positionsinformation von gespeicherten Gipfel Liste durchführen.
- Exportieren Sie die Peak-Listen aus verschiedenen Datasets zu einem Tabellenkalkulationsprogramm oder anderen mathematischen Programm zur Analyse indem Sie die Funktion "Exportieren" auswählen.
- Berechnen Sie die Veränderung der Intensitäten der Peak mit der Funktion "Peak-Intensität in komplexen Spektrum/Peak-Intensität im Protein-Spektrum" für jeden Peak. Werte können in prozentuale Veränderung durch Multiplikation der 100 konvertiert werden. Beachten Sie, dass Peak Bände auch nützliche obwohl Peak Intensitäten einfacher sind zu messen für Gipfeln, die nah beieinander liegen, da in der Regel für Proteine mit einer hohen Dichte der relativ breiten Gipfel der Fall ist.
4. MicroScale Thermophorese (MST)
-
Vorbereitung der Liganden Protein E-PRD
- Tauschen Sie den Liganden in einer MST-kompatibel-Puffer von dialyzing bis zu 800 μl des Proteins in einem 3,5 kDa Mini-Dialyse-Einheit in 1 L 20 mm HEPES, pH 7,5, 10 mM NaCl, unter ständigem Rühren langsam bei 4 ° C über Nacht ausgesetzt aus.
- Die Liganden mit einer zentrifugalen Ultrafiltration-Einheit (3 kDa MWCO) durch Zentrifugieren bei 14 000 x g für 10 min. Transfer konzentrierten Proteins in ein sauberes Röhrchen zu konzentrieren.
- Die Liganden bei 21.000 x g für 10 min Zentrifugieren und sorgfältig übertragen des Überstands auf eine neue Röhre, jede ausgefälltes Eiweiß zu entfernen. Bestimmen die Konzentration des Liganden mit der Absorption bei 280 nm und dem Liganden Aussterben Koeffizienten. Fügen Sie 10 % Tween-20, eine Endkonzentration von 0,015 % zu geben. Tween-20 ergänzt die Testpuffer Adsorption zu den Kapillaren zu verhindern. Die endgültige Testpuffer, die für die MST-Experimente verwendet wird ist 20 mM HEPES, pH 7,5, 10 mM NaCl, 0,015 % Tween-20.
-
Vorbereitung der Farbstoff-markierten Ziel Protein VimRod
- Bereiten Sie den rot-Tris-NTA-Farbstoff durch Zugabe von 50 μL 1 x PBS-T mit der rot-Tris-NTA zu einer Konzentration von 5 μM geliefert. 2 μl-Aliquots in 200 μl Tuben und Lagerung bei-20 ° C.
- Verdünnen Sie das Zielprotein, 0,34 μM mit Testpuffer. Ein 2 μL aliquoten rot-Tris-NTA Farbstoff 58 μL des Ziels hinzu und 30 min bei Raumtemperatur inkubieren. Die Endkonzentration von Farbstoff-markierten Ziel ist 0,33 μM. Das markierte Ziel bei 21.000 x g für 10 min Zentrifugieren und sorgfältig übertragen des Überstands auf eine neue Röhre, jede Niederschlag zu entfernen. Der rot-Tris-NTA-Farbstoff bindet an Proteine durch die His6-Tag und hat eine verbindliche Dissoziationskonstante (K-D) im Sub-nanomolaren Bereich. Es ist effektiv zu 100 % an das Zielprotein gebunden, so dass keine weitere Reinigung erforderlich ist.
- Schalten Sie die MST-Instrument, und öffnen Sie die Steuerungs-Software. Wählen Sie die rote für die rot-Tris-NTA-Farbstoff. Schalten Sie den Temperaturregler auf 25 ° C.
- Wählen Sie den Pretest zu validieren, die Kennzeichnung des Ziels und Aggregation oder Adsorption an die Küvetten suchen. Eine Bewertung dieser Parameter wird automatisch bereitgestellt.
- Mix-17 μL Testpuffer und 3 μL Zielprotein und Mix von pipettieren. Füllen Sie zwei standard Kapillaren durch Eintauchen in das verwässerte Ziel und die Erstellung von Flüssigkeit in der Mitte der Kapillare. Platzieren Sie Kapillaren in das Fach und in die Instrumente. Starten Sie die Messung.
- Überprüfen Sie die Ergebnisse, die auf der Suche nach ein ausreichendes Maß an Fluoreszenz und keine Anzeichen der Adsorption (Verzerrung in der Kapillarleitung Form) oder Aggregation (Verzerrungen in der MST-Ablaufverfolgung), die in der Softwareanalyse gekennzeichnet werden. Wenn die Ergebnisse positiv sind an den Liganden Schritte, wenn keine andere Puffer muss ausprobiert werden oder die Höhe der Tween-20 kann erhöht werden, bis 0,05 % oder höher.
-
Vorbereitung der Baureihe E-PRD Liganden zweifache Verdünnung
- Bereiten Sie eine Verdünnungsreihe durch Kennzeichnung 16, 200 μL Tuben von 1-16.
- Fügen Sie 17 μL des Liganden bei 1,17 x höhere Konzentration als die maximale Liganden-Konzentration Tube1 wollte. Dieser Band ist doppelt so viel benötigt (8,5 μL) wie eine aliquote dann an das nächste Rohr hintereinander übertragen werden. Der letzte Band des Assays ist 10 μL so 8,5 μL 1,17 X Konzentration des Liganden auf 1 X durch Zugabe von 1,5 μL des Zielproteins, VimRod verdünnt werden. Diese maximale Konzentration gewählt sollte mindestens 20 Mal den geschätzten KD -Wert sein.
- Tubes2-16 mit einer neuen Pipettenspitze für jedes aliquoten fügen Sie 8,5 μL der Testpuffer hinzu. Wiederverwendung von Pipettenspitzen beeinflussen die Genauigkeit (für die Beratung über genaues Pipettieren siehe Herstellerangaben). Übertragen Sie 8,5 μL des Liganden von Tube1 an Tube2 langsam loslassen die Lösung in der Assay-Puffer ohne Luftblasen zu generieren. Mischen Sie die Liganden und Puffer durch Pipettieren oben und unten mindestens 6 mal wieder ohne Luftblasen erzeugen. E-PRD in der konzentriertesten Röhre 1,5 mM und die beschrifteten VimRod war an eine Endkonzentration von 50 nM.
- 8.5 μL Liganden von Tube2 auf Tube3 übertragen und mischen mit dem Assay-Puffer. Die serielle Verdünnung zu wiederholen, bis alle Röhren Liganden hinzugefügt haben. 8.5 μL aus Tube16 zu verwerfen, so dass alle Röhren 8,5 μL der Liganden in einer Reihe von zwei-Fach-Verdünnungen enthalten.
-
Vorbereitung des verbindliche Reaktion und MST-Experiments
- Jedes der Rohre 1,5 μL der beschrifteten Zielprotein hinzu und mischen Sie leicht durch Pipettieren nach oben und unten, wobei Sie darauf achten, um Luftblasen zu vermeiden. 15 min inkubieren.
- Wählen Sie den Expertenmodus zum verbindlichen Test und geben Sie die Parameter für eine serielle Verdünnungsreihen. Stellen Sie sicher, Temperaturregler eingestellt auf 25 ° C, die Erregerleistung wird voraussichtlich 40 % und die MST macht, Medium. Diese Parameter müssen für andere Bindungspartner untersuchten optimiert werden.
- Füllen Sie die Kapillaren mit der Bindung Reaktionen und legen in die Kapillare Fach. Laden Sie das Fach in das Gerät, warten Sie, bis die Temperatur auf 25 ° C wieder und starten Sie die Messung.
-
Datenanalyse
- Öffnen Sie die Bindung Assay-Datei in die Affinität-Analyse-Software. Überprüfen Sie die Kapillare Scans; Fluoreszenz-Ebenen sollte nicht mehr als 10 % vom Durchschnitt abweichen, keine Aggregation oder Adsorption sollte erkannt werden, wie im Abschnitt 3.6 beschrieben.
- Wählen Sie die MST-Analyse auf der rechten Seite und ziehen Sie die Test-Daten in die Analyse-Set. Es gibt mehrere Durchläufe des gleichen Ziel-Ligand verbindliche Tests unter identischen Bedingungen können sie durch Fallenlassen in den gleichen Satz zusammengeführt werden. Alternativ kann jeder Ausführung in Analyse unabhängig fallen gelassen werden.
- Wechseln Sie zur Registerkarte Dosis Antwort passen die Daten grafisch darstellen. Wählen Sie die K-D -Modell, wenn eine einzelne Bindungsstelle erwartet wird. Das Hill-Modell ist auch eine Option für mehrere Bindungsstellen mit kooperatives Verhalten. Ausreißer-Punkte, die Qualitätskontrolle nicht bestanden haben können in diesem Stadium die Passform entfernt. Merge-Sets mit mehreren Tests gemittelt werden und Fehler als die Standardabweichung berechnet.
- Öffnen Sie die letzte Registerkarte und vergleichen Sie die Ergebnisse zwischen allen Parzellen in einem einzigen Diagramm. Rufen Sie passende Ergebnisse für jede Kurve aus der Tabelle, die generiert wird. Auf Wunsch exportieren Sie die Daten oder angepasste Kurven zu anderen Präsentation oder Analyse-Software.
Ergebnisse
Die E-PRD-Domäne (Rückstände 1822-2014 in pProEX-HTC kloniert) des menschlichen Envoplakin Gens und der VimRod-Domäne (Rückstände in pET21a kloniert 99-249) des menschlichen Vimentin4 ausgedrückt mit His6 Tags und gereinigt wurden. Abbildung 6 und Abbildung 7 zeigen die Reinheit des VimRod (18.8 kDa) und E-PRD (21,8 kDa) erhalten von dieser Methode der Proteinreinigung. Die Entfernung von der His6 Tag aus dem E-PRD-Konstrukt wesentlich für die MST-Experimente ist, da das VimRod-Protein wird mit einer His6 Tag verbindlich Färbung gekennzeichnet und jede E-PRD Beibehaltung seiner His6 Tag kann an den Qualifikationsturnieren für die Bindung des Farbstoffes. Die zweite Spalte der IMAC nach Spaltung des Tags mit TEV Protease entfernt der TEV-Protease, die gespalten Tag und uncleaved His6-E-PRD, die blieb. Die letzte Polierstufe der Reinigung ist Größe Ausgrenzung Chromatographie. Obwohl beide Proteine von ähnlicher Größe, elutes die VimRod aus der Spalte bei einem 51 mL, während E-PRD Elution Peak bei 72 mL zentriert ist, wo ein Protein-Monomer dieser Größe erwarten würde. Die scheinbare Zunahme der Größe der VimRod ist wahrscheinlich wegen der seine Eigenschaften als eine filamentöse lange Rute geformte Protein wie analytisch Ultrazentrifugen Experimente zeigten, dass VimRod Monomere4war. Geringere Erträge des Proteins ergeben sich aus den Kulturen angebaut in M9 als jene aus reichen Brühe aufgrund einer geringeren Menge an Zellen in den minimalen Medien produziert wird. Das erste Wachstum der größeren Starterkulturen für M9 Vorbereitungen in TB ermöglicht Verbesserung der Zelle Renditen und gleichzeitig das Ausmaß der 15N Kennzeichnung notwendig für die NMR-Experimente.
15N -1H HSQCs wurden für den Wildtyp und R1914E-Mutante von E-PRD in Anwesenheit oder Abwesenheit von VimRod (Abb. 8A-8D) erworben. Das Spektrum der E-PRD in Abbildung 8A zeigt die erwartete Anzahl von gut gelöst Gipfeln, bezeichnend für ein richtig gefaltetes Protein. In Anwesenheit von VimRod (Abb. 8 b) zeigt das Spektrum umfassenden Produktpalette erweitern und Gipfel verschwinden, Bindung zwischen E-PRD und VimRod entspricht. Diese Bindung ist durch Mutation des R1914E als belegt durch Vergleich von Abbildung 8 und 8 Dverloren. Kleine Änderung wird im Spektrum bei Zusatz von VimRod, die R1914E mutierten zeigt einen Mangel an Bindung zwischen diesem mutierten E-PRD und VimRod beobachtet. E-PRD Peak Intensitäten in die Anwesenheit/Abwesenheit der VimRod wurden verglichen und als die relative Peak Intensitäten in Abbildung 8E, was darauf das Spektrum hindeutet der Peak Verbreiterung in der E-PRD-Komplex dargestellt. R1914E-Mutante von E-PRD (nicht dargestellt) erhalten ca. 97 % der Peaks bei 20 % oder höherer Peak Intensität in Anwesenheit des VimRod im Vergleich zu etwa 20 % für den Wildtyp (Abbildung 8E). Diese steht für einen Verlust der Funktion Punkt Mutant mit zusätzlichen Mutanten mit mittleren Wirkungen auch mit Studium4gewesen.
Zu validieren und die Bindung von VimRod und E-PRD MST-Analyse mit His6-VimRod beschriftet mit rot-Tris-NTA Leuchtstofffärbung als Ziel quantitate gemischt mit abnehmender Konzentration des Liganden E-PRD von 1,28 mM um 39,1 nM durchgeführt wurden. Drei verbindliche Titrationen wurden durchgeführt und die Ergebnisse werden gemittelt und in Abbildung 9gezeigt. Die Daten wurden mit einem standard-Modell ein-Standort-Ligand-Bindung passen und gab eine K-D von 25,7 ± 2,1 μm. Bewertung der Bindung zwischen VimRod und E-PRD durch Oberflächenplasmonenresonanz gab einen ähnlichen KD -Wert von 19,1 ± 1,3 μm4.

Abbildung 1: Screenshot des Setups des NMR-Experimentes. Abgebildete Fenster dient ein standard Experiment so einrichten, dass ein HSQC--Dataset zu sammeln. Testparameter eingelesen sind neben dem Experiment. Das ZGPR-Experiment gezeigt wird als ein erstes Experiment gewählt, die Standard- und Lösungsmittel abhängigen Proton-Parameter zu laden. Der Titel Fenster dient zur Eingabe experimentelle Daten für Zwecke der Aufzeichnungen. Um das HSQC-Spektrum zu sammeln ist das ZGPR-Experiment mit SFHMQC3GPPH ersetzt. Bitte klicken Sie hier für eine größere Version dieser Figur.
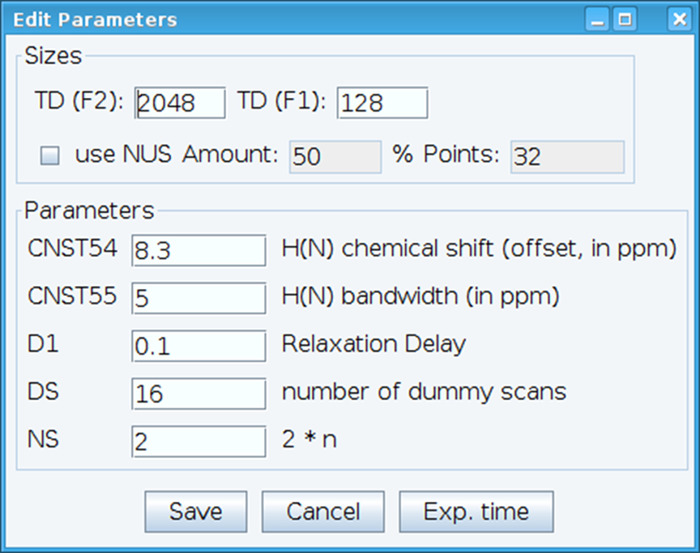
Abbildung 2: Einstellen der NMR Versuchsparameter. Abgebildete Fenster dient zur Eingabe der Grundparameter für die NMR-Pulsfolge um das Signal zu optimieren. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3: NMR Datenverarbeitung. Parameter für die Bearbeitung jeder der zwei Dimensionen des NMR-Spektrums verwendet werden angezeigt, mit Pfeilen, die diejenigen, die in der Regel angepasst sind. Bitte klicken Sie hier für eine größere Version dieser Figur.
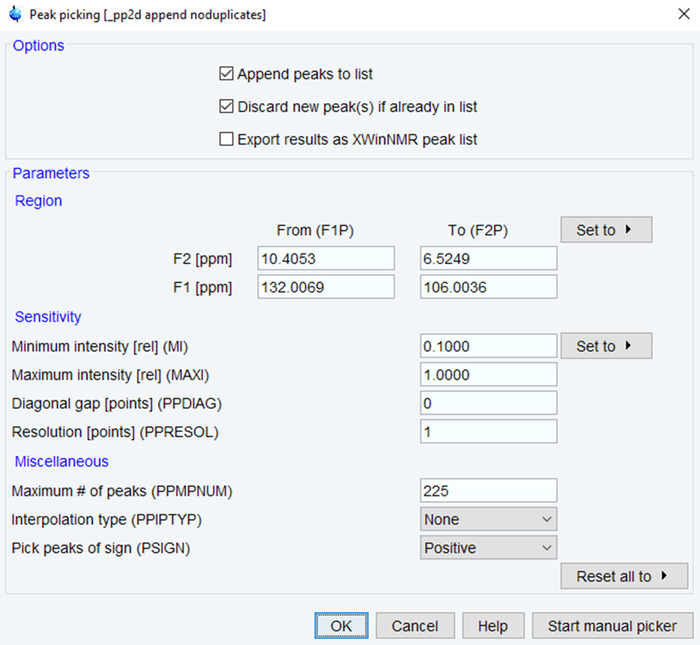
Abbildung 4: Parameter für NMR-Peak Picking. Die Parameter für die Kommissionierung NMR Gipfeln in der verarbeiteten NMR-Spektrum werden mit typischen Werten angezeigt. Passen Sie die ppm-Bereich, die Intensität und die Anzahl der Spitzen, die Spektren zu optimieren. Bitte klicken Sie hier für eine größere Version dieser Figur.
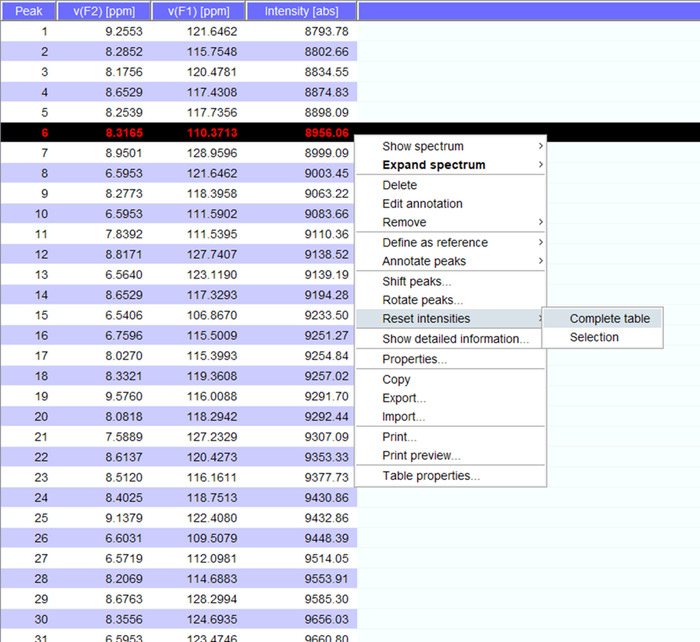
Abbildung 5: Vertreter Peaklist mit Intensitäten. Jede Spitze, die in der NMR-Spektrum aufgenommen wird erhält eine Nummer und seinem 1H und 15N chemische Verschiebungen und Signalstärke angezeigt. Dieser Peaklist kann dann verwendet werden, Spektren erwarb die Anwesenheit/Abwesenheit von einem interagierenden Partner zu vergleichen. Bitte klicken Sie hier für eine größere Version dieser Figur.
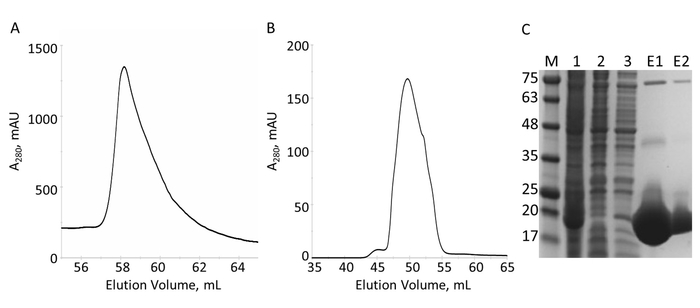
Abbildung 6: Reinigung von His6 getaggt VimRod von IMAC und S. A. Das Chromatogramm für die Elution aus der Spalte "IMAC" zeigt einen großen Höhepunkt des VimRod. B. das Chromatogramm für die Elution aus der Spalte "S" zeigt einen großen Höhepunkt. C. SDS-PAGE der Fraktionen im Laufe der Reinigung gesammelt: MW-Standards mit der MW im kDa auf der linken Seite des Gel (M), Zelle lysate (1), IMAC Durchfluss (2), waschen (3), gepoolte Elution (E1), gebündelt S Elution (E2) angegeben. Bands bei höheren Molmassen in Bahnen E1 und E2 sind Oligomere des reinen VimRod durch western-Blot (Daten nicht gezeigt) bestätigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
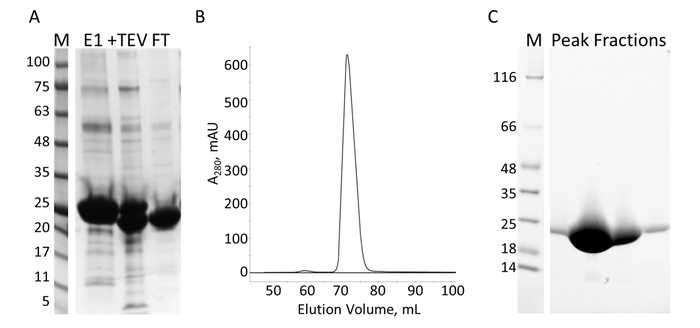
Abbildung 7: Reinigung des E-PRD von IMAC und S. A. SDS-PAGE der IMAC Reinigung zeigt das Molekulargewicht Standards mit den MW in kDa angegeben, auf der linken Seite das Gel (M) und das Eluat aus der ersten IMAC-Spalte (E1), der TEV Spaltung Produkte (+ TEV), und der Durchfluss aus der zweiten Spalte der IMAC (FT). (B) der Chromatograph aus der Spalte "S" zeigt einen großen Höhepunkt. C. SDS-PAGE Molekulargewicht Normen (M) und die Fraktionen von S-Spitze. Bitte klicken Sie hier für eine größere Version dieser Figur.
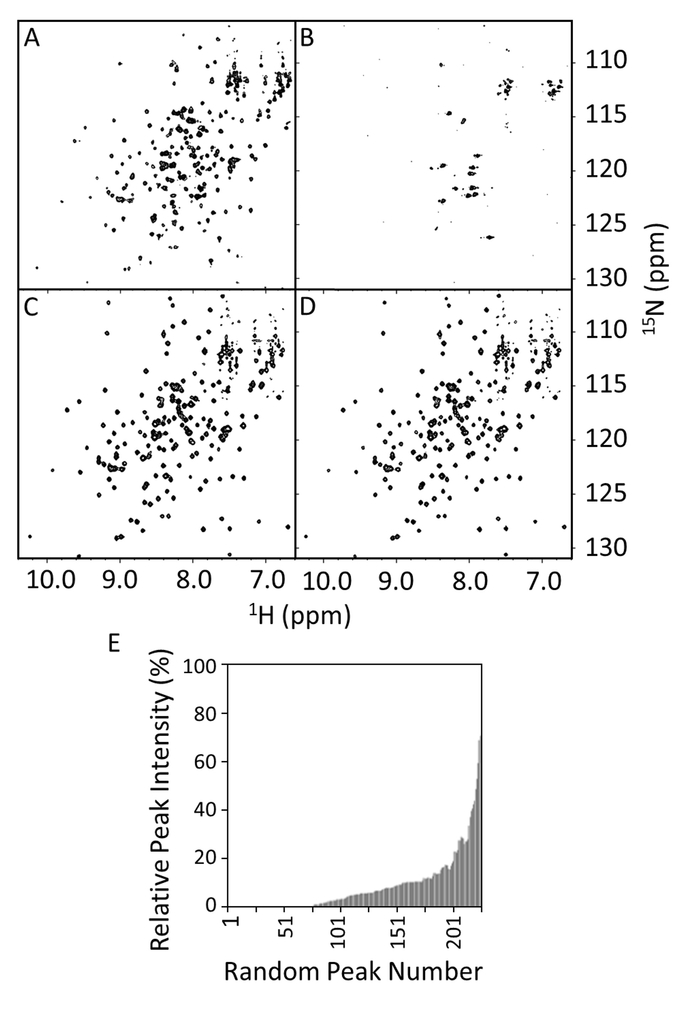
Abbildung 8: HSQC-Spektren von Wildtyp und R1914E-Mutante von E-PRD in Anwesenheit und Abwesenheit von VimRod. Die HSQC-Spektren zeigen Wildtyp E-PRD (100 µM) in 20 mM Tris-HCl, 150 mM NaCl, 1 mM DTT, pH-Wert 7 in Abwesenheit (A) oder die Anwesenheit von 50 µM VimRod (B). Panels, C und D sind die HSQC-Spektren der R1914E Mutant (100µM) in das Fehlen oder Vorhandensein von 50 µM VimRod, beziehungsweise. Im E relative 1H -15N Gipfel sind Intensitäten der E-PRD mit oder ohne VimRod Bindung in Abhängigkeit von der Höchstzahl angezeigt, die willkürlich zugeordnet und nicht anhand der Reihenfolge Position. Diese Werte können verwendet werden, um eine Bedeutung Cutoff für Peak Intensität Reduktion bei Zusatz eines Liganden zu definieren. Wenn Zuordnungen zur Verfügung stehen, können signifikante Werte oft gesehen werden, ein Bindungsbereich zuordnen. Bitte klicken Sie hier für eine größere Version dieser Figur.
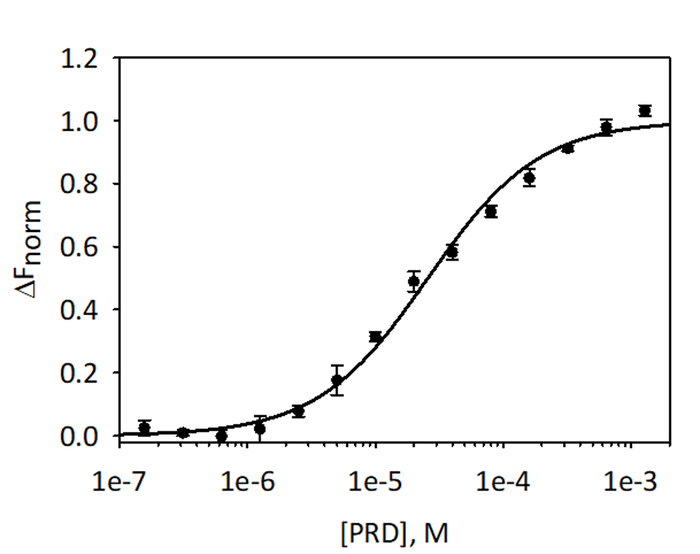
Abbildung 9: Bindung von E-PRD, VimRod. E-PRD wurde in einer Reihe von zwei-Fach-Verdünnungen von 1,28 mM um 39,1 verdünnt nM und vor der Ausführung MST-Analyse mit beschrifteten VimRod inkubiert. Daten aus drei unabhängigen Tests wurden kombiniert. Die Daten wurden zu einem K-D -Modell geben eine K-D von 25,7 μM mit einem KD Vertrauen von ± 2,1 μm passen. Bitte klicken Sie hier für eine größere Version dieser Figur.
| Reagenz | Menge |
| Natriumphosphat, Diabas-(wasserfrei) | 6,2 % |
| Kalium-Phosphat, monobasic (wasserfrei) | 3,2 % |
| Natrium-Chlorid | 0,5 g |
| H2O | Bis zu 950 mL |
Tabelle 1. M9 Medien zur isotopischen Kennzeichnung.
| Reagenz | Menge |
| 15 NH4Cl | 1,0 g |
| Glukose (oder 13C-Glukose) | 2,0 g |
| 1 M MgSO4 | 2 mL |
| 50 mM CaCl2 | 4 mL |
| 20 mg/mL Thiamin | 1,0 mL |
| 3 mM FeCl3 | 400 ΜL |
| Metall-Mix (Tabelle 3) | 500 ΜL |
| H2O | Bis zu 50 mL |
Tabelle 2. Nährstoff-Mix für die Ergänzung der M9 Medien.
| Reagenz | Menge |
| 4 mM ZnSO4 | 323 mg |
| 1 mM MnSO4 | 75,5 mg |
| 4,7 mM H3BO3 | 145 mg |
| 0,7 mM CuSO4 | 55,9 mg |
| H2O | Bis zu 500 mL |
Tabelle 3. Metall-Mix Ergänzung zur Anreicherung des MT-Nährstoffen-Mix.
| Probe | 1 mM E-PRD in Puffer A1 (µL) | 1mM VimRod in Puffer A (µL) | Puffer (µL) | 200 µM DSS in D2O (µL) | Puffer B2 (µL) | Gesamtvolumen (µL) |
| E-PRD allein | 50 | 0 | 50 | 50 | 350 | 500 |
| E-PRD + VimRod | 50 | 50 | 0 | 50 | 350 | 500 |
| 1 Puffer A: 20 mM Tris-HCl, 1 mM DTT, pH 7 | ||||||
| 2 Puffer B: 23 mM Tris-HCl, 1,14 mM DTT, pH 7 | ||||||
Tabelle 4. NMR-Probenvorbereitung.
Diskussion
2D 15N gelöst NMR-Experiment ist eines der am häufigsten verwendeten Methoden, um zu zeigen, wie zwei Molekülen interagieren. Es ist die am meisten informationsreichen-Methode, die beide Partner Signale, während eine Titration Experiment in Lösung Zustand kontinuierlich überwacht werden kann. Obwohl in der Regel bei großen komplexen qualitative, die Methode auch in günstigen Fällen einsetzbar, um verbindliche Affinitäten zu messen wo NMR-Signale in hochauflösende Spektren nachverfolgt werden können. Wo Aufgaben bequem erfolgen können wie bei vielen Proteinen unter 20 kDa groß, auch die Bindungsstellen zugeordnet werden können. Ergänzende Tests wie MST quantitative Informationen über Interaktionen in Lösung und erfordern weniger Protein in unbeschrifteten Staaten. Vergleich der mutierten binden von Daten ist nützlich für die Kontrollen, um sicherzustellen, dass Interaktionen durch NMR-Linie Erweiterung belegt echt sind und keine Artefakte, z. B. Aggregation oder Viskosität Änderungen.
Protein-Expression
Prozessoptimierung Ausdruck reduziert die Menge der Arbeit intensiv Proteinproduktion. Dieser Optimierungsprozess gehört die Identifizierung der eine entsprechende Belastung von E. Coli für die rekombinante Expression des Proteins. Sorte bevorzugt hängt von Faktoren einschließlich der Art des Vektors im Einsatz, und genauer gesagt, die ultimative Stabilität des rekombinanten Proteins Seins ausgedrückt7. Das Risiko der Verschlechterung des heterologen Proteins durch endogene E. Coli Proteasen reduziert werden, durch Verwendung von Protease mangelhaft E. Coli wie der Stamm BL21. Für Gene, die seltene Codons enthält kann eine Belastung wie BL21-CodonPlus (DE3) RIPL vorzuziehen. Diese Sorte kombiniert die Protease mangelhaft Natur der BL21 Belastung mit zusätzliche endogenen Exemplare des seltenen Codons tRNAs für Arginin, Prolin, Isoleucin und Leucin. Alternativ können seltene Codons die Überexpression gefährden können vermieden werden, durch die Bestellung ein Codon-optimierte Konstrukt aus kommerziellen Quellen. Viele Stämme von E. Coli sind für rekombinante Genexpression, jeweils für die Umgehung eines bestimmten Problems während Ausdruck7optimiert. Bei dieser Studie erstellt der standard Protease mangelhaft Stamm BL21(DE3) ausreichende Mengen an löslichen Proteinen für die anschliessende Reinigung und Analyse.
Proteinreinigung
Die Reinigung Protokoll für ein gegebenes Protein ist oft einzigartig in dem Sinne, dass jedes Protein stabil und unter verschiedenen Bedingungen wie Temperatur, Salzgehalt oder pH-Wert löslich bleibt. Die allgemeine Wirksamkeit der Reinigung durch Affinitätschromatographie ist auch empfindlich auf die Konzentration von Medikamentenfreisetzende Arten wie Imidazol in verschiedenen Schritten während des Reinigungsprozesses. In dieser Arbeit wurden kritische Pufferbedingungen für IMAC pH-Wert für die E-PRD und Imidazol-Konzentration für die VimRod. Ein pH-Wert von 7,5 bedurfte, um die Ausfällung von E-PRD nach anfänglichen Elution aus der Spalte "IMAC" zu vermeiden. Für die IMAC-Reinigung von VimRod Erhöhung der Konzentration von Imidazol von 30 bis 50 mM in der Spalte Waschschritt zeigte sich eine deutliche Verbesserung in der Reinheit der endgültigen Elution Fraktionen. Für die Elution Schritt Erhöhung der Konzentration von Imidazol von 250 bis 350 mM auch erwies sich um die Ausbeute an die endgültige Elution zu verbessern. Erste Versuche, mit 250 mM Imidazol Protein eluieren führte zu unvollständigen Elution von VimRod wie durch einen abschließenden 1 M Imidazol-Streifen der Spalte (Daten nicht gezeigt) offenbart. Erhöhung der Imidazol-Konzentration bis 350 mM für die Elution reichte aus, um all das Protein gebunden in die Spalte wiederherzustellen. S kann einen doppelten Zweck dienen, weil es als ein Polierstufe für Proteinreinigung während gleichzeitig ausführen Buffer Tausch wirkt. Buffer Tausch ist ein entscheidender Schritt für die anschließende Bindung Analyse, da sie die Imidazol verwendet, um His6-markierte Protein eluieren entfernt. Es dient auch als eine Gelegenheit zu ändern Bedingungen wie Salzkonzentration oder pH, die die Wirksamkeit bestimmter nachgeschaltete Techniken oder Assays auswirken können. Protein, das thermische Verschiebung (PTS) verwendet werden, kann um optimale Puffer für nachgeschaltete Assays, vor allem für diejenigen, die stabile Protein für identifizieren längere Zeit bei Zimmertemperatur8,9.
Analyse der Bindung
Protein, das frisch zubereitet wird ist entscheidend für genaue verbindliche Assays, obwohl gefrorenen Protein kann auch verwendet werden, solange die Ergebnisse verglichen werden. Filamentöse Proteine wie Vimentin Multimerize Salz und pH-Wert abhängige Weise, und somit die Lösung bedingen müssen optimiert werden und die Oligomere Zustand durch eine Methode wie SEC10,11, dynamisches Licht Streuung geschätzt 12 oder Analytische Ultrazentrifugation13,14,15. NMR-Spektroskopie ist gut geeignet zur Messung der Ligand Interaktionen der kleinen Proteine in atomarer Auflösung. Jedoch wenn ein Protein mit größeren Molekül interagiert, langsamer Tumbling folgt, und dies zu Verlust von Signalen, die verbindlich bestätigen kann führt, obwohl dies nicht unbedingt der Fall erlauben Zuordnung Bindungsstellen, die auch die Zuweisung von mindestens erfordern würde Rückgrat Resonanzen. In diesem Szenario lassen NMR-Experimente nicht Identifikation des Standortes Interaktion. Daher directed Website Mutagenesis angewendet wird, um die kritischen Rückstände für die Bindung benötigt zu identifizieren. Solche Mutanten aufweisen Signalverlust daher nicht. In diesem Protokoll eine mutierte Form mit einer Substitution an Position 1914 behält die Peak-Intensitäten in Anwesenheit von VimRod und Störung der Interaktion von E-PRD und VimRod bestätigt. Zuordnung der Rückgrat und Sidechain Resonanzen würde dieser Ansatz Wert hinzufügen, zumal die Struktur für das kostenlose E-PRD durch Röntgen-Kristallographie4gelöst worden ist. Zukünftige Anwendungen der NMR Charakterisierung der komplexen Wechselwirkungen zwischen größeren Molekülen gehören und profitieren von extrem hohen Bereich Magnete und die Verwendung von anderen beobachtbaren Gruppen wie z. B. 13C beschriftet und trifluor Methylgruppen als Reporter.
MST hat eine Reihe von Vorteilen für das Studium bindende Wechselwirkungen16. Die Bindungspartner sind frei in Lösung und nicht immobilisiert. Analyse der Qualität der Proben ist die Software mit Qualitätskontrolle der Aggregation, die Adsorption an den Kapillaren oder unzureichende fluoreszierende Kennzeichnung des Zielmoleküls eingebaut. Kleine Mengen des Ziels dienen in der Regel, die Konzentration der das markierte Ziel ist in der Regel zwischen 20-50 nM in einer 10-20 μl Volumen/Reaktion. Dieses Protokoll verwendet sehr kleine Reaktion Volumen (10 μl), die Konzentration des Liganden zu maximieren, die in der Titrationen ermöglichen schwach bindender Wechselwirkungen gekennzeichnet werden erreicht werden kann. Dies erfordert genaues pipettieren und Sorgfalt zu vermeiden, Einführung von Luftblasen beim noch gründlich mischen. Ausreichende Vermischung ist entscheidend für präzise, konsistente Fluoreszenz-Messungen entlang der Satz von Verdünnungsreihen. Die Tween-20 in der MST-Experimenten wurde reduziert von einer Norm 0,05 bis 0,015 %, senken die Tendenz zu Bläschen zu schaffen und zu verbessern, mischen.
Der Farbstoff rot-Tris-NTA ermöglicht schnell, einfach und bequem Eindringmittel kennzeichnen jedes Protein, das eine seiner Tag. Die Kennzeichnung ist in nur 30 Minuten wirksam abgeschlossen und ist sehr eng, so dass kein Farbstoff Deinstallation erforderlich ist. An Aminosäurereste des Proteins, die die Liganden Bindungseigenschaften beeinflussen könnten, werden keine Änderungen vorgenommen. Ein Nachteil ist, dass nur das Protein zu etikettierenden einen His6 Tag haben sollte. Dies erforderte die Spaltung des Tags aus der Liganden-Protein, E-PRD und Entfernen von Tags und uncleaved E-PRD mit einem zweiten Schritt der IMAC-Spalte. Wenn möglich, die Liganden Protein sollten bereit sein, ohne den Einsatz eines seiner Tag. Alternativ können Proteine kovalent mit einem Fluorophor durch Amin Ankopplung an Lysin Rückstände oder Thiol Ankopplung an Cystein Rückstände beschriftet werden. Allerdings muss darauf geachtet werden, wenn mit solchen Systemen seit der kovalenten Befestigung ein Fluorophor elektrostatische oder polare Bindung Interaktionen unter Berufung auf Lysin oder Cystein Rückstände beeinträchtigen kann. Die Quantifizierung von verbindlichen Affinität zwischen dem VimRod und E-PRD von MST war ungewöhnlich Konzentration salzempfindlich. Dieses Problem wurde durch die dialyzing zunächst das Ziel und die Liganden in der gleichen Charge von Testpuffer gemindert. Sättigung der MST verbindliche Kurve konnte jedoch nicht erreicht werden, bei der Durchführung der MST-Assay im Beisein von 150 mM NaCl aufgrund des komplexen Verhaltens von VimRod. Zuverlässige und vollständige Daten wurden ermittelt, sobald die Konzentration von NaCl, 10 mM, so dass genaue Berechnung des K-Dgesenkt wurde. Daher sind sorgfältige Optimierung der Lösungsbedingungen und Vergleich mit ergänzenden Tests empfohlen, robuste Ergebnisse zu erzielen. Darüber hinaus kann MST verwendet werden, um das Salz Abhängigkeit für einen bestimmten Umgang zu quantifizieren, stöchiometrische Eigenschaften von Protein-Interaktionen, Monitor Proteinfaltung und Sonde in Enzym Kinetik17zu quantifizieren.
Offenlegungen
Die Autoren Interessenkonflikte keine.
Danksagungen
Dieses Projekt wurde von NSERC RGPIN-2018-04994, Campus Alberta Innovationsprogramm (RCP-12-002 C) und Alberta Prion Research Institute unterstützt / Alberta innoviert Bio Solutions (201600018), verliehen an M.O und Genom-Kanada und Canada Foundation for Innovation-Finanzhilfen der Metabolomik Innovation Centre (TMIC) und NANUC.
Materialien
| Name | Company | Catalog Number | Comments |
| Monolith NT.115, includes control and analysis software | NanoTemper Technologies | MO-G008 | Instrument for microscal thermophoresis |
| His-Tag Labeling Kit RED-Tris-NTA | NanoTemper Technologies | MO-L008 | RED-tris-NTA dye for MST |
| Standard capillaries | NanoTemper Technologies | MO-K022 | capillaries for MST |
| HisTrap HP, 5 mL | GE Healthcare | 17524801 | IMAC column |
| HisPur Ni-NTA resin, 100 mL | Thermo Fisher Scientific | 88222 | IMAC resin |
| HiLoad 16/600 Superdex 75 pg | GE Healthcare | 28989333 | SEC column |
| TEV protease | Sigma Aldrich | T4455 | cleavage of his tag from E-PRD |
| BL21(DE3) Competent E. coli | New England Biolabs | C2527H | cells for protein expression |
| cOmplete Protease Inhibitor Cocktail Tablets EDTA-Free | Sigma Aldrich | 11873580001 | protease inhibitors for protein purification by IMAC |
| TCEP, Tris(2-carboxyethyl)phosphine hydrochloride | Sigma Aldrich | C4706 | reducing agent for protein purification |
| Reagents (HEPES, NaCl, etc) | Sigma Aldrich | various | Preparation of media and buffers |
| Ammonium chloride (15N, 99%) | Cambridge Isotope Laboratories | NLM-467 | isotope labelling for NMR |
| D2O, Deuterium Oxide (D, 99.8%) | Cambridge Isotope Laboratories | DLM-2259 | NMR sample preparation |
| DSS, Sodium 2,2-dimethyl-2-silapentane-5-sulfonate-D6 (D,98%) | Cambridge Isotope Laboratories | DLM-8206 | reference for NMR |
| Precision 5 mm NMR Tubes, 7” long | SJM/Deuterotubes | BOROECO-5-7 | NMR tubes |
| NMR spectrometer (14.1 Tesla) | Bruker | acquisition of NMR data | |
| TCI 5mm z-PFG cryogenic probe | Bruker | acquisition of NMR data | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Bruker TopSpin 4.0.1 | Bruker | processing of NMR data | |
| MO.Control | NanoTemper Technologies | included with Monlith NT.115 | |
| MO.Affinity Analysis | NanoTemper Technologies | included with Monlith NT.115 |
Referenzen
- Vinogradova, O., Qin, J. NMR as a unique tool in assessment and complex determination of weak protein-protein interactions. Topics in Current Chemistry. 326, 35-45 (2012).
- McKercher, M. A., Wuttke, D. S. NMR Chemical Shift Mapping of SH2 Peptide Interactions. Methods in Molecular Biology. 1555, 269-290 (2017).
- Al-Jassar, C., Bikker, H., Overduin, M., Chidgey, M. Mechanistic Basis of Desmosome-Targeted Diseases. Journal of Molecular Biology. 425 (21), 4006-4022 (2013).
- Fogl, C., et al. Mechanism of intermediate filament recognition by plakin repeat domains revealed by envoplakin targeting of vimentin. Nature Communications. 7, (2016).
- Walker, J. M. . The Protein Protocols Handbook. , (2002).
- Wishart, D. S., et al. 1H, 13C and 15N chemical shift referencing in biomolecular NMR. J. Biomol. NMR. 6, 135-140 (1995).
- Casali, N. Escherichia coli Host Strains. E. coli Plasmid Vectors. , 27-48 (2003).
- Ericsson, U. B., Hallberg, B. M., DeTitta, G. T., Dekker, N., Nordlund, P. Thermofluor-based high-throughput stability optimization of proteins for structural studies. Analytical Biochemistry. 357 (2), 289-298 (2006).
- Kozak, S., et al. Optimization of protein samples for NMR using thermal shift assays. Journal of Biomolecular Nmr. 64, 281-289 (2016).
- Burgess, R. R. A brief practical review of size exclusion chromatography: Rules of thumb, limitations, and troubleshooting. Protein Expression and Purification. , (2018).
- Kunji, E. R. S., Harding, M., Butler, P. J. G., Akamine, P. Determination of the molecular mass and dimensions of membrane proteins by size exclusion chromatography. Methods. 46 (2), 62-72 (2008).
- Stetefeld, J., McKenna, S. A., Patel, T. R. Dynamic light scattering: a practical guide and applications in biomedical sciences. Biophysical Reviews. 8 (4), 409-427 (2016).
- Patel, T. R., Winzor, D. J., Scott, D. J. Analytical ultracentrifugation: A versatile tool for the characterisation of macromolecular complexes in solution. Methods. 95, 55-61 (2016).
- Pearson, J. Z., Cole, J. L., et al. Chapter One - Next-Generation AUC Adds a Spectral Dimension: Development of Multiwavelength Detectors for the Analytical Ultracentrifuge. Methods in Enzymology. 562, 1-26 (2015).
- Gorbet, G. E., Pearson, J. Z., Demeler, A. K., Cölfen, H., Demeler, B., Cole, J. L. Chapter Two - Next-Generation AUC: Analysis of Multiwavelength Analytical Ultracentrifugation Data. Methods in Enzymology. 562, 27-47 (2015).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nature Communications. 1, 100 (2010).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten