Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Beurteilung der zellulären Immunantwort der Fruchtfliege Drosophila Melanogaster, mit einem In-Vivo-Phagozytose-Assay
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt einen in-vivo Phagozytose Assay in Erwachsenen Drosophila Melanogaster , Phagozyten Anerkennung und Räumung von mikrobiellen Infektionen zu quantifizieren.
Zusammenfassung
Bei allen Tieren bietet die angeborene Immunität eine sofortige und robuste Verteidigung gegen ein breites Spektrum von Krankheitserregern. Humorale und zelluläre Immunantwort sind die Hauptzweige der angeborenen Immunität, und viele der Faktoren, die zur Regelung dieser Reaktionen sind evolutionär konserviert zwischen Wirbellosen und Säugetiere. Phagozytose, die zentraler Bestandteil der zellulären angeborenen Immunität erfolgt durch spezialisierte Zellen des Immunsystems. Die Fruchtfliege Drosophila Melanogaster, entstanden als ein leistungsfähiges genetisches Modell, die molekularen Mechanismen und physiologischen Auswirkungen der Phagozytose in ganze Tiere zu untersuchen. Hier zeigen wir einen Injektion-basierte in-vivo Phagozytose Assay um die Partikel Aufnahme und Zerstörung von Blutkörperchen Drosophila , Hemocytes zu quantifizieren. Das Verfahren ermöglicht Forschern genau steuern die Partikelkonzentration und Dosis, macht es möglich, hoch reproduzierbare Ergebnisse in kürzester Zeit zu erhalten. Das Experiment ist quantitativ, einfach durchzuführen, und Bildschirm für Host Faktoren dieser Einfluss Erreger Anerkennung, Aufnahme und Abstand angewendet werden können.
Einleitung
Angeborenen Immunabwehr bilden die erste Verteidigungslinie gegen pathogene Keime. Diese Reaktionen können funktionell humorale und zelluläre angeborene Immunität unterteilt werden, die beide durch Keimbahn-kodierte Muster Anerkennung Rezeptoren (PRRs), die Pathogen-assoziierte molekulare Muster (PAMPs)1Sinn vermittelt werden. Ein Großteil der Signalwege und Effektor Mechanismen der angeborenen Immunität sind bei Säugetieren und Wirbellose Tiere, wie z. B. der Fadenwurm Caenorhabditis Elegans und der Fruchtfliege Drosophila Melanogaster2konserviert. Die Fruchtfliege ist ein leistungsfähiges System Host Verteidigung gegen infektiöse Mikroorganismen3studieren entstanden. Drosophila ist genetisch gefügig, einfach und kostengünstig in Laboratorien, aufgezogen und hat eine kurze Generationszeit. Darüber hinaus weist der Fruchtfliege hoch effiziente Abwehr gegen ein Array von Mikroben, die Prüfung der Host Immunität gegen virale, bakterielle, Pilze oder parasitäre Erreger aktivieren.
Drosophila Immunologen haben historisch verwendet vorwärts genetischer Bildschirme, genomweiten RNA-vermittelte Interferenz (RNAi screening von Insekten Zelllinien und bereits vorhandene mutierte Fliege Stämme um angeborene Immunität – führt zu untersuchen) die Identifizierung und Charakterisierung der mehrere evolutionär konservierte humorale Immunsystem Wege4,5,6,7,8. Die angeborene humorale Immunantwort ist wohl, das beste gekennzeichnet Immunabwehr bei Fruchtfliegen. Nach Infektion führt die humorale Antwort zu die Produktion und die systemische Freisetzung von antimikrobiellen Peptid (AMP) Moleküle in die Hämolymphe, das Blut gleichwertig in Insekten. Verstärker sind von hoch konservierte Maut und Imd Signalwege produziert. Der Maut-Pfad ist homolog zu Säugetieren TLR/IL-1R-Rezeptor Signalgebung und der Imd-Pfad ist homolog zu Säugetieren Tumor-Nekrose-Faktor-Alpha-Signalisierung. In Drosophila, Maut Signalisierung wird induziert durch Gram-positiven Bakterien, Pilze und Drosophila X Virus6,9,10 und Imd Signalisierung wird induziert durch gramnegative Bakterien11 ,12.
Zelluläre Immunität, bestehend aus Kapselung, Melanization und Phagozytose invasive Erreger durch spezialisierte Blutkörperchen genannt Hemocytes13durchgeführt. Es gibt drei Klassen von Hemocytes in der Taufliege: Kristall Zellen, Lamellocytes und Plasmatocytes13. Kristall-Zellen, die machen 5 % der zirkulierenden Hemocytes in Larven frei ProPhenoloxidase (d) Enzyme führt zu Melanization Erreger und Wirt Gewebe an Wunde Stellen. Lamellocytes, die normalerweise nicht in gesunden Embryonen oder Larven gefunden werden, sind adhärente Zellen, die Fremdkörper zu Kapseln. Diese Zellen induziert werden, beim Pupariation oder beim Fliegen Wespe Eier in die Larven hinterlegt sind. Phagocytic Plasmatocytes, die 95 % der zirkulierenden Hemocytes bei Larven und alle restlichen Hemocytes bei Erwachsenen ausmachen, spielen eine Rolle im Gewebe Umbau während der Entwicklung und vor allem dienen als die wichtigsten Effektor-Zelle von Drosophila zelluläre Immunität.
Phagozytose eine unmittelbare und entscheidende Linie der angeborenen Immunabwehr; Mikroben, die die Host epitheliale Barriere zu durchbrechen sind schnell verschlungen und eliminiert durch phagocytic Blutzellen (für eine umfassende Überprüfung der Zellbiologie der Phagozytose siehe Referenz 14). Dieser Prozess wird eingeleitet, wenn Keimbahn-codierte Mustererkennung Rezeptoren (PRRs) auf Hemocytes Erreger erkennen molekulare Muster (PAMPs) von Mikroben verbunden. Nach der Bindung an ihre Ziele, initiieren PRRs Signalisierung Kaskaden, die zur Bildung von Scheinfüßchen durch Aktin Zytoskelett Umgestaltung führen. Die Scheinfüßchen umgeben die Mikrobe, die anschließend verschlungen und in einer im Entstehen begriffenen Organell, das Phagosom verinnerlicht. Mikroben sind zerstört, als die Phagosom das Phagosom Reifung durchläuft wenn das Phagosom Opfer von in Richtung des Inneren der Hemocyte Menschenhandel ist und durch eine Reihe von Wechselwirkungen mit Lysosomen säuert. In Vitro und Zelle haben Studium der Biologie in Säugetierzellen Primary maßgeblich bei der Identifizierung und Charakterisierung von Faktoren, die Phagozytose, wie Säugetier-Fc-Gamma-Rezeptor und C3b Rezeptoren15,16zu regulieren. Dennoch sind die Fähigkeit zum Ausführen von großen Bildschirmen oder in vivo Studien in Säugetier-Systeme beschränkt.
Hier präsentieren wir Ihnen einen in-vivo Assay für Phagozytose bei Erwachsenen Fruchtfliegen, basiert auf einem Verfahren erstmals im Labor von David Schneider in 200017. Das Schneider-Labor zeigte, dass festsitzende Hemocytes gruppierten entlang der Abdominal-dorsalen Gefäß leicht Polystyrol-Kügelchen und Bakterien phagozytieren. Um Phagozytose zu visualisieren, fliegen mit Gewebekulturen beschrifteten Partikel (z. B. E. Coli mit Fluorescein erfolgt (E. Coli -FITC) gekennzeichnet), injiziert sind für 30 Minuten zu Hemocytes Zeit, um die Partikel zu verschlingen inkubiert und dann mit Trypan blau, die die Fluoreszenz der Partikel nicht phagozytiertes während der Inkubationszeit stillt injiziert. Fliegen dorsalen Gefäße werden dann abgebildet mit einem inversen Fluoreszenz-Mikroskop. Diese Samen-Papier mit ein relativ einfaches Experiment nachgewiesen, dass Hemocytes Bakterien phagozytieren und Latex Perlen, dass bakterielle Phagozytose durch die Injektion von Pre gehemmt werden können mit Latex Perlen fliegt und fliegt ohne zelluläre und humorale Immunantworten sind auch anfällig für E. Coli. Der Test in diesem Bericht vorgestellten baut auf der Arbeit von Schneider-Lab, in-vivo Phagozytose durch Messung der Fluoreszenzintensität von Partikeln durch dorsale Schiff verbundenen Hemocytes verschlungen zu quantifizieren.
Ähnlich wie bei dem Ansatz Säugetier-Systeme, Drosophila Genetiker ursprünglich verwendet genomweite in-vitro-RNAi-Bildschirme um zu identifizieren, die für die zelluläre Immunantwort18,19,20 erforderlich ,21,22,23. Jedoch ermöglichte die Entwicklung des Erwachsenen in-vivo Phagozytose Assays Follow-up-Experimente bereitwillig in ganze Tiere, so dass Forscher die biologischen überprüfen die Rolle der Faktoren, die in in-vitro-Studien durchgeführt werden. Dies war der Fall mit dem transmembrane Rezeptor Esser, die zunächst als ein bakterielle Rezeptor in einer RNAi-Bildschirm mit S224 Zellen identifiziert und dann später gezeigt, um Escherichia coli (E. Coli),, Enterococcus vermitteln Faecalis, und Staphylococcus Aureus (S. Aureus) Phagozytose in Erwachsene25.
Unser Labor beschäftigt die in-vivo Phagozytose Assay in vorwärts genetischer Bildschirme und genomweite Assoziationsstudien (unter Verwendung der Drosophila genetische Referenz Panel (DGRP)), um neuartige Gene zu identifizieren, die Phagozytose in Erwachsenen Hemocytes zu regulieren. Diese Studien führten bis hin zur Charakterisierung der Rezeptoren PGRP-SC1A und PGRP-SA26, die intrazelluläre Vesikel Handel Protein Rab1427, die Glutamat-Transporter Polyphem28und RNA-bindende Protein Fox-129.
Wir gehen davon aus, dass zukünftige Bildschirme unter Einbeziehung der in-vivo Phagozytose zur Identifizierung von zusätzlichen Gene führen könnte, die wichtig für die zelluläre Immunantwort bei Drosophilasind. Bildschirme mit vollständig sequenziert Inzuchtlinien, z. B. die DGRP oder die Drosophila synthetische Bevölkerung Ressource (DSPR), können natürliche Varianten die Phagozytose oder Hemocyte Entwicklung zu identifizieren. Darüber hinaus könnte die Technik angenommen in andere Arten von Drosophila oder verwendet, um Bildschirm neue Community-Ressourcen, wie z. B. die Sammlung von 250 Drosophila -Arten gepflegt durch die nationalen Drosophila Arten Lager Center (NDSSC ) an der Cornell University. Diese Experimente durchgeführt werden können mit Gewebekulturen beschriftet bakterielle oder Pilzinfektionen-Wand Bioparticles, die im Handel erhältlich sind oder können mit einer beliebigen Anzahl von bakterielle oder Pilzinfektionen Arten – vorausgesetzt, dass die Mikrobe fluoreszierenden Marker drückt durchgeführt werden .
Protokoll
1. bereiten Sie Fluorescein Partikel zur Injektion
- 10 mg im Handel erhältlichen, Hitze getötet Bakterien rekonstruieren Partikel mit Fluorescein gekennzeichnet (siehe Tabelle der Materialien) Lager Konzentration von 10 mg/mL durch Zugabe von 990 µL steriler 1 X PBS und 10 µL 50 mM Natriumazid. Vortex mischen.
- In Einweg-8 µL-Aliquots in 0,2 mL Röhrchen teilen und speichern in einer dunklen Box bei 4 ° C, Lichtempfindlichkeit verbunden zu minimieren.
Hinweis: Konservierungsmittel Natriumazid ist optional und kann weggelassen werden, wenn Aktien 10 mg/mL mit 1 mL sterilen 1 X PBS vorgenommen werden regelmÄÑig und bei-20 ° c gelagert
- In Einweg-8 µL-Aliquots in 0,2 mL Röhrchen teilen und speichern in einer dunklen Box bei 4 ° C, Lichtempfindlichkeit verbunden zu minimieren.
- Eine 10 mL-Lösung 5 % Lebensmittelfarbe in 1 X PBS durch Mischen von 500 µL Spritze gefiltert grüne Lebensmittel Färbung und 9,5 mL sterilen 1 X PBS zu machen.
- Waschen Sie Partikel vor der Injektion, Natriumazid zu entfernen. Mix-42 µL steriler 1 x PBS und 8 µL von 10 mg/mL in einer 1,7 mL Tube. Zentrifugieren Sie bei max. Geschwindigkeit 2,5 min bei Raumtemperatur.
- Entfernen Sie den Überstand zu, fügen Sie 50 µL 1 x PBS und Zentrifuge mit max. Geschwindigkeit 2,5 min bei Raumtemperatur.
- Wiederholen Sie die Schritte 1.3 und 1.3.1 2 X, für eine Gesamtmenge von 3 Wäschen.
- Verwerfen Sie nach der letzten Wäsche überstand und erneut aussetzen Sie Partikel bis 1,6 mg/mL in 50 µL 5 % Lebensmittelfarbe in 1 X PBS.
- Wickeln Sie das Rohr in Alufolie um vor Licht zu schützen. Lagerung bei 4 ° C, nach 1 Woche entsorgen.
2. bereiten Sie die Injektion Station und fliegen
- Vorbereiten der Injektion-Pad. Bis zu injizieren pad 4 Genotypen fliegen gleichzeitig Verwendung Labor Band eine rechteckige CO2 Fliege aufteilen in 4 Abschnitte. Bezeichnen Sie auf der Bank in der Nähe des Mikroskops Bereiche, die Fläschchen zu platzieren, wenn fliegen haben auf dem Pad (eine für jede Ecke des Pads) ausgekleidet.
- Fläschchen mit Alter abgestimmt, 4-7 Tage vorbereiten-alt, fliegen für die Injektion. Für jede Belastung zu prüfenden transfer 5 Rüden und 5 Hündinnen in ein frisches, beschrifteten Fläschchen des Gargutes fliegen und halten bei 25 ° C.
- Vorbereiten den pneumatischen Injektor (siehe Tabelle der Materialien) indem das Instrument auf einen 100 ms (kurze Bursts von Gas-Druck, die Flüssigkeit – ermöglicht die Lieferung von Sub-Nanoliter Bände zu vertreiben) TIMED -Modus.
- Bereiten Sie den Objektträger. Schneiden Sie 1,5-Zoll-Streifen Isolierband, in eine Schleife mit der klebenden Seite nach außen Falten Sie und legen Sie auf einen beschrifteten Objektträger.
3. bereiten Sie Glas Kapillare Nadeln
- Ziehen Sie Glas-Nadeln (dünne Wand Glaskapillaren) mit einer Nadel Abziehvorrichtung ab.
- Halten Sie die Nadel unter dem Mikroskop mit einem Mikrometer und brechen Sie den Tipp #5 feine Spitze Edelstahl Pinzette. 100 µm-Tipps sind ausreichend, um die Fliege Nagelhaut zu durchbohren, bei gleichzeitiger Minimierung der Verwundung.
- Messen Sie das Volumen der Flüssigkeit, die in jede Fliege injiziert wird. Laden Sie die Nadel mit sterilen 5 % Lebensmittelfarbe in 1 X PBS und vertreiben Sie die Flüssigkeit auf einen Tropfen von Mineralöl auf einer Bühne Mikrometer 0,01 mm.
Hinweis: Wenn das flüssige Tröpfchen kugelförmig ist, ist das Volumen in Picoliters (Größe)3/1910 berechnet. 30 eine Nadel mit einem Durchmesser von 100 µm wird ausgeworfen ~ 2 nL in 100 ms.
(4) injizieren fliegen
- Pipette 10 µL von 1,6 mg/mL Partikel auf einem kleinen Platz des Parafilm.
- Ziehen Sie die Flüssigkeit in die Nadel und montieren in der Einspritzdüse (siehe Tabelle der Materialien).
- Betäuben Sie fliegen mit CO2 zu und richten sie in ihrem ausgewiesenen Bereich auf das Flypad mit der ventralen Seite nach oben und die Köpfe auf der Vorderseite des Pads ausgerichtet. Fläschchen in den entsprechenden Bereichen auf der Bank Platz.
- Fliegen in der oberen Ecke des Bauches mit 5, 100 ms-Pumpen von Flüssigkeiten (~ 10 nL gesamt) zu injizieren.
- Übertragen Sie die injizierten fliegen auf die geeignetsten Fläschchen, notieren Sie sich die Zeit auf dem Fläschchen. Halten Sie bei 25 ° C.
- Laden Sie eine neue Nadel mit 0,4 % Trypan blau Lösung.
- Legen Sie die pneumatische Injektor auf GATED, ermöglicht einen konstanten Luftstrom um die Flüssigkeit aus der Nadel schieben.
- Fliegen zu betäuben, nachdem sie für 30 Minuten ausgeruht haben und Spritzen mit Trypan blau, bis der Bauch voll und aufgetrieben ist.
Hinweis: Bei der Prüfung von Phagosom Reifung mit Partikeln beschriftet mit einem pH-Sensitive Farbstoff fliegen für 1 h ruhen lassen und nicht mit Trypan blau vor Montage fliegen injizieren. - Montieren Sie fliegen auf Objektträger mit Isolierband, Bauchseite nach unten. Schieben Sie die Flügel auf die Seite der Fliege und sichern Sie sie auf das Band. Auch, schieben Sie vorsichtig den Kopf in die Band um sicherzustellen, dass die Fliege nicht verschoben werden.
- Sofort fahren Sie mit Schritt 5.
(5) Bildgebung fliegen
- Bild fliegt, eine zu einem Zeitpunkt, bei 25 X oder 32 X Vergrößerung mit einem invertierten Fluoreszenzmikroskop angebracht, eine Digitalkamera und einen Computer (siehe Tabelle der Materialien). Konzentrieren Sie sich auf der dorsalen Gefäß der Fliege mit Computer-Software für die digitale Kamera.
- Notieren Sie die Belichtungszeit und Vergrößerung zwischen Experimente.
Hinweis: Verlust des Genotypen eine potenzielle Fehlerquelle ist, wenn mehrere Stämme in einer einzigen Sitzung zu fotografieren. Um zu vermeiden, falsche Etikettierung fliegen, notieren Sie die Bildnummer des ersten und letzten fliegen Fotos für jeden Genotyp.
(6) zu quantifizieren und die Normalisierung der Fluoreszenz
- Öffnen Sie die Software, und öffnen Sie ein Bild zu einem Zeitpunkt.
- Messen der Fluoreszenzintensität des dorsalen Schiffes. Zeichnen Sie ein Polygon um den dorsalen Gefäß. Wählen Sie messen und zeichnen Sie der Fluoreszenzintensität innerhalb des Polygons auf.
- Die Hintergrund-Fluoreszenz-Intensität zu bestimmen. Kopieren Sie das erste Polygon und verschieben Sie sie in einen Bereich angrenzend an der dorsalen Gefäß der Fliege. Wählen Sie messen und aufzeichnen Fluoreszenzintensität von den Hintergrundbereich.
- Die dorsalen Gefäß Fluoreszenz durch die Hintergrundfluoreszenz zu normalisieren:
Dorsalen Gefäß ÷ Hintergrund. - Berechnen Sie die durchschnittliche dorsalen Gefäß Fluoreszenzintensität alle fliegen in einem Stamm normalisiert.
- Wiederhole das Experiment 2 weitere Male.
- Verwendung ein Student der ungepaarten t-Test, um die mittlere relative Fluoreszenz-Intensitäten der Steuerung fliegen vergleichen und testen Sie fliegt aus den drei Experimenten. Berechnen der Effektgröße mit Hilfe der Formel: Cohens d = (M1- M2) ÷ SDgebündelt, wo M1 ist der Mittelwert der Genotyp 1 und M2 ist der Mittelwert der Genotyp 2 &
SD gepoolte =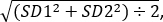
wo SD1 ist die Standardabweichung der Genotyp 1 und SD2 ist die Standardabweichung der Genotyp 2.
Ergebnisse
Abbildung 1Azeigt eine schematische Darstellung des in-vivo Phagozytose Assays mit Fluorescein-markierten Partikeln. Fliegen sind berittene Bauchseite nach unten auf ein Stück Isolierband und die ersten beiden Segmente des Abdomens, wo der dorsale Gefäß befindet, ist deutlich sichtbar (Abb. 1 b). Hauptquellen der experimentellen Fehler entstehen bei der Injektion und imaging-Schritte des Verfahrens (Abbild...
Diskussion
Handelsübliche, Eindringmittel beschrifteten Partikel werden zur Phagozytose im Allgemeinen (0,2 µm Carboxylat-modifizierte Mikrosphären) oder Phagozytose von Mikroben (Eindringmittel beschriftet Hitze- oder chemisch tote Bakterien oder Hefe) zu beurteilen. Um Phagosom Reifung zu beurteilen, können Forscher wählen Sie Partikel beschriftet mit einem pH-Sensitive-Farbstoff, der fluoresziert, wenn der pH-Wert von neutral zu sauer, wie in der Phagolysosome abnimmt. Alternativ um die ersten Schritte der Phag...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren danken Dr. Beth Gonzalez und Dr. Aprajita Garg für Unterstützung bei der Durchführung der Phagozytose in-vivo-Experimente. Ein NSF UMD ADVANCE Seed Grant und UMD NIH T32 Ausbildung Zuschüsse, Zelle und Molekularbiologie (CMB) und der Wirt-Pathogen Interaktionen (HPI) finanziert diese Arbeit.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.2μm Red Fluorescent Carboxylate Modified FluoSpheres | Invitrogen | F8810 | Fluorescently-labeled latex beads to test general phagocytic capacity of phagocytes. (~580/~605 nm) Inject a 1:20 dilution in PBS with 5% dye. |
| 5430-10 PicoNozzle Kit | World Precision Instruments | 5430-10 | Holder for 1.0mm pipette |
| E. coli (K-12 Strain) BioParticles, Alexa Fluor 488 conjugate | Invitrogen | E13231 | Killed E. coli labeled with Alexa Fluor 488. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~495/~519 nm) |
| E. coli (K-12 Strain) BioParticles, Alexa Fluor 594 conjugate | Invitrogen | E23370 | Killed E. coli labeled with Alexa Fluor 594. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~590/~617 nm) |
| E. coli (K-12 Strain) BioParticles, Fluorescein conjugate | Invitrogen | E2861 | Killed E. coli labeled with FITC (Fluorescein). Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~494/~518 nm) |
| E. coli (K-12 Strain) BioParticles, Texas Red conjugate | Invitrogen | E2863 | Killed E. coli labeled with Texas Red. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~595/~615 nm) |
| E. coli (K-12 Strain) BioParticles, Texas Red conjugate | Invitrogen | E2863 | Killed E. coli labeled with Texas Red. Use to test phagocyte recogntion and uptake of gram-negative bacteria. (~595/~615 nm) |
| Needle Pipette Puller | David Kopf Instruments | Model 725 | |
| pHrodo Red E. coli BioParticles Conjugate for Phagocytosis | Invitrogen | P35361 | Killed E. coli labeled with pHrodo Red. Use to test phagocyte reconition, uptake, and phagosome maturation of gram-negative bacteria. (~560/~585 nm). No need to quench with Trypan Blue. |
| pHrodo Red S. aureus BioParticles Conjugate for Phagocytosis | Invitrogen | A10010 | Killed S. aureus labeled with pHrodo Red. Use to test phagocyte reconition, uptake, and phagosome maturation of gram-positve bacteria. (~560/~585 nm). No need to quench with Trypan Blue. |
| Pneumatic PicoPump PV820 | World Precision Instruments | SYS-PV820 | The World Precision Instruments Pneumatic PicoPump PV820 uses differential pressures to hold liquid in the glass needle between injections. The user manually controls short bursts of gas pressure to expel the liquid – allowing delivery of sub-nanoliter volumes. The amount of liquid delivered depends on two main variables – the size of the glass needle opening and the amount of time injection pressure is applied. set the instrument to 100 ms “TIMED” mode. |
| S. aureus (Wood Strain without protein A) BioParticles, Alexa Fluor 488 conjugate | Invitrogen | S23371 | Killed S. aureus labeled with Alexa Fluor 488. Use to test phagocyte recogntion and uptake of gram-positive bacteria. (~495/~519 nm) |
| S. aureus (Wood Strain without protein A) BioParticles, Alexa Fluor 594 conjugate | Invitrogen | S23372 | Killed S. aureus labeled with Alexa Fluor 594. Use to test phagocyte recogntion and uptake of gram-positive bacteria. (~590/~617 nm) |
| S. aureus (Wood Strain without protein A) BioParticles, Fluorescein conjugate | Invitrogen | E2851 | Killed S. aureus labeled with FITC (Fluorescein). Use to test phagocyte recogntion and uptake of gram-positive bacteria. (~494/~518 nm) |
| Thin Wall Glass Capillaries | World Precision Instruments | TW100F-3 | Needles for injection. OD = 1.0 mm |
| Trypan Blue Solution (0.4%) | Sigma | T8154 | Used to quench extracellular fluorescence of Fluorescein, Alexa Fluor, or Texas Red labeled particles. |
| ZEISS SteREO Microscope (Discovery.V8) | Zeiss | SteREO Discovery.V8 | Inverted fluorescence microscope for imaging flies. Use a digital camera (example: AxioCam HC camera) and the accompanying software (example: AxioVision 4.7 software) to take pictures. |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Alexa Fluor 488 conjugate | Invitrogen | Z23373 | Killed labeled with Alexa Fluor 488. Use to test phagocyte recogntion and uptake of yeast. (~495/~519 nm) |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Alexa Fluor 594 conjugate | Invitrogen | Z23374 | Killed labeled with Alexa Fluor 594. Use to test phagocyte recogntion and uptake of yeast. (~590/~617 nm) |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Fluorescein conjugate | Invitrogen | Z2841 | Killed labeled with FITC (Fluorescein). Use to test phagocyte recogntion and uptake of yeast. (~494/~518 nm) |
| Zymosan A (Saccharomyces cerevisiae) BioParticles, Texas Red | Invitrogen | Z2843 | Killed labeled with Texas Red. Use to test phagocyte recogntion and uptake of yeast. (~595/~615 nm) |
Referenzen
- Akira, S., Uematsu, S., Takeuchi, O. Pathogen recognition and innate immunity. Cell. 124 (4), 783-801 (2006).
- Kim, D. Studying host-pathogen interactions and innate immunity in Caenorhabditis elegans. Disease Models & Mechanisms. 1 (4-5), 205-208 (2008).
- Lemaitre, B., Hoffmann, J. The host defense of Drosophila melanogaster. Annual Review Immunology. 25, 697-743 (2007).
- Wu, L. P., Choe, K. M., Lu, Y., Anderson, K. V. Drosophila Immunity: Genes on the Third Chromosome Required for the Response to Bacterial Infection. Genetics. 159 (1), 189-199 (2001).
- De Gregorio, E., Spellman, P. T., Tzou, P., Rubin, G. M., Lemaitre, B. The Toll and Imd pathways are the major regulators of the immune response in Drosophila. EMBO Journal. 21 (11), 2568-2579 (2002).
- Michel, T., Reichhart, J. M., Hoffmann, J. A., Royet, J. Drosophila Toll is activated by Gram-positive bacteria through a circulating peptidoglycan recognition protein. Nature. 414 (6865), 756-759 (2001).
- Choe, K. M., Werner, T., Stoven, S., Hultmark, D., Anderson, K. V. Requirement for a peptidoglycan recognition protein (PGRP) in Relish activation and antibacterial immune responses in Drosophila. Science. 296 (5566), 359-362 (2002).
- Wu, J., Randle, K. E., Wu, L. P. ird1 is a Vps15 homologue important for antibacterial immune responses in Drosophila. Cellular Microbiology. 9 (4), 1073-1085 (2007).
- Lemaitre, B., Nicolas, E., Michaut, L., Reichhart, J. M., Hoffmann, J. A. The dorsoventral regulatory gene cassette spatzle/Toll/cactus controls the potent antifungal response in Drosophila adults. Cell. 86 (6), 973-983 (1996).
- Zambon, R. A., Nandakumar, M., Vakharia, V. N., Wu, L. P. The Toll pathway is important for an antiviral response in Drosophila. Proceedings of the National Academy of Science of the United States of America. 102 (20), 7257-7262 (2005).
- Lemaitre, B., et al. A recessive mutation, immune deficiency (imd), defines two distinct control pathways in the Drosophila host defense. Proceedings of the National Academy of Science of the United States of America. 92 (21), 9465-9469 (1995).
- Leulier, F., Rodriguez, A., Khush, R. S., Abrams, J. M., Lemaitre, B. The Drosophila caspase Dredd is required to resist gram-negative bacterial infection. EMBO Reports. 1 (4), 353-358 (2000).
- Meister, M., Lagueux, M. Drosophilablood cells. Cellular Microbiology. 5 (9), 573-580 (2003).
- Flannagan, R. S., Jaumouille, V., Grinstein, S. The cell biology of phagocytosis. Annual Review Pathology. 7, 61-98 (2012).
- Anderson, R. A., Sando, G. N. Cloning and expression of cDNA encoding human lysosomal acid lipase/cholesteryl ester hydrolase. Similarities to gastric and lingual lipases. Journal of Biological Chemistry. 266 (33), 22479-22484 (1991).
- Ross, G. D., Reed, W., Dalzell, J. G., Becker, S. E., Hogg, N. Macrophage cytoskeleton association with CR3 and CR4 regulates receptor mobility and phagocytosis of iC3b-opsonized erythrocytes. Journal of Leukocyte Biology. 51 (2), 109-117 (1992).
- Elrod-Erickson, M., Mishra, S., Schneider, D. S. Interactions between the cellular and humoral immune responses in Drosophila. Current Biology. 10, 781-784 (2000).
- Ramet, M., Pearson, A. M., Manfruelli, P., Li, X., Koziel, H., Gobel, V. Drosophila Scavenger Receptor CI Is a Pattern Recognition Receptor for Bacteria. Immunity. 15 (6), 1027-1038 (2001).
- Ramet, M., Manfruelli, P., Pearson, A. M., Mathey-Prevot, B., Ezekowitz, R. A. Functional genomic analysis of phagocytosis and identification of a Drosophila receptor for E. coli. Nature. 416 (6881), 644-648 (2002).
- Philips, J. A., Rubin, E. J., Perrimon, N. Drosophila RNAi screen reveals CD36 family member required for mycobacterial infection. Science. 309, 1251-1253 (2005).
- Agaisse, H., Burrack, L. S., Philips, J. A., Rubin, E. J., Perrimon, N., Higgins, D. E. Genome-wide RNAi screen for host factors required for intracellular bacterial infection. Science. 309 (5738), 1248-1251 (2005).
- Stuart, L. M., et al. Response to Staphylococcus aureus requires CD36-mediated phagocytosis triggered by the COOH-terminal cytoplasmic domain. Journal of Cell Biology. 170 (3), 477-485 (2005).
- Stroschein-Stevenson, S. L., Foley, E., O’Farrell, P. H., Johnson, A. D. Identification of Drosophila gene products required for phagocytosis of Candida albicans. PLoS Biology. 4 (1), e4 (2006).
- Kocks, C., et al. Eater, a transmembrane protein mediating phagocytosis of bacterial pathogens in Drosophila. Cell. 123 (2), 335-346 (2005).
- Nehme, N. T., et al. Relative roles of the cellular and humoral responses in the Drosophila host defense against three gram-positive bacterial infections. PLoS One. 6 (3), e14743 (2011).
- Garver, L. S., Wu, J., Wu, L. P. The peptidoglycan recognition protein PGRP-SC1a is essential for Toll signaling and phagocytosis of Staphylococcus aureus in Drosophila. Proceedings of the National Academy of Science of the United States of America. 103 (3), 660-665 (2006).
- Garg, A., Wu, L. P. Drosophila Rab14 mediates phagocytosis in the immune response to Staphylococcus aureus. Cellular Microbiology. 16 (2), 296-310 (2014).
- Gonzalez, E. A., Garg, A., Tang, J., Nazario-Toole, A. E., Wu, L. P. A glutamate-dependent redox system in blood cells is integral for phagocytosis in Drosophila melanogaster. Current Biology. 23 (22), 2319-2324 (2013).
- Nazario-Toole, A. E., Robalino, J., Okrah, K., Corrada-Bravo, H., Mount, S. M., Wu, L. P. The Splicing Factor RNA-Binding Fox Protein 1 Mediates the Cellular Immune Response in Drosophila melanogaster. Journal of Immunology (Baltimore, Md: 1950). 201 (4), 1154-1164 (2018).
- Guille, M. . Molecular Methods in Developmental Biology: Xenopus and Zebrafish. , (1999).
- Koundakjian, E. J., Cowan, D. M., Hardy, R. W., Becker, A. H. The Zuker collection: a resource for the analysis of autosomal gene function in Drosophila melanogaster. Genetics. 167 (1), 203-206 (2004).
- Horn, L., Leips, J., Starz-Gaiano, M. Phagocytic ability declines with age in adult Drosophila hemocytes. Aging Cell. 13 (4), 719-728 (2014).
- Brennan, C. A., Delaney, J. R., Schneider, D. S., Anderson, K. V. Psidin is required in Drosophila blood cells for both phagocytic degradation and immune activation of the fat body. Current Biology. 17 (1), 67-72 (2007).
- Akbar, M. A., Tracy, C., Kahr, W. H., Kramer, H. The full-of-bacteria gene is required for phagosome maturation during immune defense in Drosophila. Journal of Cell Biology. 192 (3), 383-390 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten