Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eine Bioinformatik-Pipeline zur genauen und effizienten Analyse der MicroRNA-Transkriptome in Pflanzen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Eine Bioinformatik-Pipeline, nämlich miRDeep-P2 (kurz miRDP2), mit aktualisierten pflanzen-miRNA-Kriterien und einem überarbeiteten Algorithmus, könnte microRNA-Transkriptome in Pflanzen genau und effizient analysieren, insbesondere für Arten mit komplexen und großen Genomen.
Zusammenfassung
MicroRNAs (miRNAs) sind 20- bis 24-Nukleotid (nt) endogene kleine RNAs (sRNAs), die in Pflanzen und Tieren, die eine starke Rolle bei der Regulierung der Genexpression auf posttranskriptionaler Ebene spielen, weitreichend vorhanden sind. Die Sequenzierung von sRNA-Bibliotheken nach NEXT Generation Sequencing (NGS)-Methoden wurde in den letzten zehn Jahren häufig eingesetzt, um miRNA-Transkriptome zu identifizieren und zu analysieren, was zu einer schnellen Zunahme der miRNA-Entdeckung führte. Zwei große Herausforderungen ergeben sich jedoch in der pflanzlichen miRNA-Anmerkung aufgrund der zunehmenden Tiefe sequenzierter sRNA-Bibliotheken sowie der Größe und Komplexität von Pflanzengenomen. Erstens werden viele andere Arten von sRNAs, insbesondere kurze störende RNAs (siRNAs) aus sRNA-Bibliotheken, von vielen Rechenwerkzeugen fälschlicherweise als miRNAs bezeichnet. Zweitens wird es zu einem extrem zeitaufwändigen Prozess zur Analyse von miRNA-Transkriptomen bei Pflanzenarten mit großen und komplexen Genomen. Um diese Herausforderungen zu meistern, haben wir vor kurzem miRDeep-P (ein beliebtes Tool für miRNA-Transkriptomanalysen) auf miRDeep-P2 (kurz miRDP2) aktualisiert, indem wir eine neue Filterstrategie einsetzen, den Bewertungsalgorithmus überarbeiten und die neu aktualisierte Anlage miRNA Anmerkungskriterien. Wir testeten miRDP2 an sequenzierten sRNA-Populationen in fünf repräsentativen Pflanzen mit zunehmender genomischer Komplexität, einschließlich Arabidopsis, Reis, Tomaten, Mais und Weizen. Die Ergebnisse deuten darauf hin, dass miRDP2 diese Aufgaben mit sehr hoher Effizienz verarbeitet hat. Darüber hinaus übertraf miRDP2 andere Vorhersagetools in Bezug auf Empfindlichkeit und Genauigkeit. Zusammengenommen zeigen unsere Ergebnisse miRDP2 als schnelles und genaues Werkzeug zur Analyse von pflanzlichen miRNA-Transkriptomen, daher ein nützliches Werkzeug, um der Community dabei zu helfen, miRNAs in Pflanzen besser zu kommentieren.
Einleitung
Eine der spannendsten Entdeckungen der letzten zwei Jahrzehnte in der Biologie ist die sich ausbreitende Rolle von sRNA-Arten bei der Regulierung verschiedener Funktionen des Genoms1. Insbesondere stellen miRNAs eine wichtige Klasse von 20- bis 24-nt sRNAs in Eukaryoten dar und fungieren hauptsächlich auf posttranskriptionärer Ebene als prominente Genregulatoren während der gesamten Lebenszyklusentwicklungsphase sowie in Stimulus- und Stressreaktionen2,3. Bei Pflanzen entstehen miRNAs aus primären Transkripten, sogenannten pri-miRNAs, die in der Regel durch RNA-Polymerase II als individuelle Transkriptionseinheiten4,5transkribiert werden. Verarbeitet durch evolutionär konservierte zelluläre Maschinen (Drosha RNase III bei Tieren, DICER-like in Pflanzen) werden pri-miRNAs in die unmittelbaren miRNA-Vorstufen, pre-miRNAs, eingeschnitten, die Sequenzen enthalten, die intramolekulare Stammschleifenstrukturenbilden 6,7. Pre-miRNAs werden dann zu doppelsträngigen Zwischenprodukten verarbeitet, nämlich miRNA-Duplexen, bestehend aus dem funktionellen Strang, der reifen miRNA und dem seltener funktionellen Partner miRNA*2,8. Nach dem Einladen in den RNA-induzierten Silencing-Komplex (RISC) konnten die reifen miRNAs ihre mRNA-Ziele anhand der Sequenzkompleärität erkennen, was zu einer negativen Regulatorischen Funktion2,8führte. miRNAs könnten entweder ihre Zieltranskripte destabilisieren oder Zielübersetzungen verhindern, aber die frühere Art und Weise wird in den Anlagen8,9dominiert.
Seit der zufälligen Entdeckung der ersten miRNA in der Nematode Caenorhabditis elegans10,11wurde viel Forschung zur miRNA-Identifikation und ihrer funktionellen Analyse, insbesondere nach der Verfügbarkeit der NGS-Methode, durchgeführt. Die breite Anwendung der NGS-Methode hat die Nutzung von Rechenwerkzeugen, die entwickelt wurden, um die einzigartige Eigenschaft von miRNAs zu erfassen, wie die Stammschleifenstruktur von Vorläufern und ihre bevorzugte Akkumulation von Sequenzlesungen auf ausgereifter miRNA und miRNA* stark gefördert. Als Ergebnis haben die Forscher bemerkenswerte Erfolge bei der Identifizierung von miRNAs in verschiedenen Arten erzielt. Basierend auf einem zuvor beschriebenen Wahrscheinlichkeitsmodell12entwickelten wir miRDeep-P13, das erste Rechenwerkzeug zur Erkennung von pflanzlichen miRNAs aus NGS-Daten. miRDeep-P zielte speziell darauf ab, die Herausforderungen der Dekodierung von pflanzlichen miRNAs mit variablerer Vorläuferlänge und großen paralogusfarbenen Familien13,14,15zu meistern. Nach seiner Veröffentlichung wurde dieses Programm tausende Male heruntergeladen und verwendet, um miRNA-Transkriptome bei mehr als 40 Pflanzenarten16zu kommentieren. Angetrieben von NGS-basierten Tools wie miRDeep-P, hat die Anzahl der registrierten miRNAs im öffentlichen miRNA-Repository miRBase17, wo derzeit über 38.000 miRNA-Elemente gehostet werden (Release 22.1), im Vergleich zu nur 500 MIRNA-Elementen (Release 2.0) im Jahr 200818erhöht.
Allerdings sind zwei neue Herausforderungen aus der pflanzlichen miRNA-Anmerkung entstanden. Erstens haben hohe Verhältnisse von Falsch-Positiven die Qualität der pflanzlichen miRNA-Anmerkungen16,19 aus folgenden Gründen stark beeinflusst: 1) eine Flut von endogenen kurz störenden RNAs (siRNAs) aus NGS sRNA-Bibliotheken wurde fälschlicherweise als miRNAs bezeichnet, da keine strengen miRNA-Anmerkungskriterien vorliegen; 2) Für Arten ohne a priori miRNA-Informationen sind falsch positive Vorhersagen, die auf NGS-Daten basieren, schwer zu eliminieren. Am Beispiel von miRBase fanden Taylor et al.20 ein Drittel der pflanzlichen miRNA-Einträge im öffentlichen Endlager21 (Release 21) ohne überzeugende Belege und sogar drei Viertel der pflanzlichen miRNA-Familien waren fragwürdig. Zweitens wird es zu einem extrem zeitaufwändigen Prozess zur Vorhersage von pflanzlichen miRNAs mit großen und komplexen Genomen16. Um diese Herausforderungen zu meistern, haben wir miRDeep-P aktualisiert, indem wir eine neue Filterstrategie hinzugefügt, den Bewertungsalgorithmus überarbeitet und neue Kriterien für die pflanzliche miRNA-Anmerkung integriert und die neue Version miRDP2 veröffentlicht haben. Darüber hinaus haben wir miRDP2 mit NGS sRNA-Datensätzen mit allmählich steigenden Genomgrößen getestet: Arabidopsis, Reis, Tomaten, Mais und Weizen. Im Vergleich zu anderen fünf weit verbreiteten Werkzeugen und seiner alten Version analysierte miRDP2 diese sRNA-Daten und analysierte miRNA-Transkriptome schneller mit verbesserter Genauigkeit und Empfindlichkeit.
Inhalt des miRDP2-Pakets
Das miRDP2-Paket besteht aus sechs dokumentierten Perl-Skripten, die sequenziell vom vorbereiteten Bash-Skript ausgeführt werden sollen. Von den sechs Skripten werden drei (convert_bowtie_to_blast.pl, filter_alignments.plund excise_candidate.pl) von miRDeep-P geerbt. Die anderen Skripts werden ab der Originalversion geändert. Die Funktionen der sechs Skripte werden im Folgenden beschrieben:
preprocess_reads.pl filtert Eingabelesevorgänge, einschließlich Lesevorgänge, die zu lang oder zu kurz sind (<19 nt oder >25 nt), und liest korreliert mit Rfam ncRNA-Sequenzen sowie Lesevorgänge mit RPM (Reads Per Million) kleiner als 5. Das Skript ruft dann Lesevorgänge ab, die mit bekannten miRNA-Reifensequenzen korreliert sind. Die Eingabedateien sind Originallesevorgänge im FASTA/FASTQ-Format und bowtie2-Ausgabe von Lesekarten, die zu miRNA- und ncRNA-Sequenzen mapping.
Die Formel für die Berechnung von RPM lautet wie folgt:
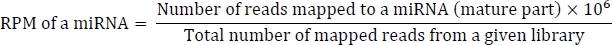
convert_bowtie_to_blast.pl ändert das Bowtie-Format in ein BLAST-parsed-Format. BLAST-parsed Format ist ein benutzerdefiniertes tabellarisches separates Format, das vom Standard-NCBI BLASToutput-Format abgeleitet wird.
filter_alignments.pl filtert die Ausrichtungen von Tiefensequenzierungslesungen zu einem Genom. Es filtert Partielle Ausrichtungen sowie mehrstimmige Lesevorgänge (benutzerspezifische Frequenzabschaltung). Die Basiseingabe ist eine Datei im BLAST-parsed Format.
excise_candidate.pl schneidet potenzielle Vorläufersequenzen aus einer Referenzsequenz unter Verwendung ausgerichteter Lesevorgänge als Richtlinien aus. Die Basiseingabe ist eine Datei im BLAST-parsed-Format und eine FASTA-Datei. Die Ausgabe sind alle potenziellen Vorläufersequenzen im FASTA-Format.
mod-miRDP.pl benötigt zwei Eingabedateien, Signaturdatei und Strukturdatei, die aus dem kernigen miRDeep-P-Algorithmus geändert wird, indem das Bewertungssystem mit pflanzenspezifischen Parametern geändert wird. Die Eingabedateien sind dot-bracket Precursor-Strukturdatei und liest VerteilungSignatur-Datei.
mod-rm_redundant_meet_plant.pl benötigt drei Eingabedateien: chromosome_length, Vorläufer und original_prediction, die von mod-miRDP.pl generiert werden. Es generiert zwei Ausgabedateien, nicht redundante vorhergesagte Datei und vorhergesagte Datei gefiltert durch neu aktualisierte plant miRNA Kriterien. Details zum Format der Ausgabedatei werden in Abschnitt 1.4 beschrieben.
Protokoll
1. Installation und Prüfung
- Download erforderliche Abhängigkeiten: Bowtie222 und RNAfold23. Kompilierte Pakete werden empfohlen.
- Laden Sie Bowtie2, ein Lesemapping-Tool, von seiner Homepage herunter (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml).
- Laden Sie RNAfold, ein Werkzeug des Vienna-Pakets zur Vorhersage der sekundären RNA-Struktur, von http://www.tbi.univie.ac.at/~ivo/RNA/herunter.
- Stellen Sie vor der Installation von miRDP2 sicher, dass diese beiden Abhängigkeiten ordnungsgemäß installiert sind, und passen Sie die bash-Umgebungsdatei (z. B. .bashrc) an, um einen korrekten PATH für diese beiden Abhängigkeiten festzulegen.
HINWEIS: Andere Mapping-Tools wie Bowtie24 sind ebenfalls für miRDP2 geeignet; entweder Bowtie oder Bowtie2 können nach Version 1.1.3 verwendet werden.
- Um das miRDP2-Paket herunterzuladen, gehen Sie zu https://sourceforge.net/projects/mirdp2/files/latest_version/ und holen Sie die Tarball-Dateien ab.
- Stellen Sie vor der Installation von miRDP2 sicher, dass Sich Perl im PATH befindet. Um miRDP2 zu installieren, extrahieren Sie den gesamten Inhalt der heruntergeladenen Tarball-Datei in einem Ordner (Befehlszeilen wie in 1.4.2), und legen Sie dann den Ordnerpfad in den PATH fest.
HINWEIS: Für die Ausführung von miRDP2 wird ein Computer oder Computerknoten mit mindestens 8 GB RAM und 100 GB Speicher empfohlen. - Testen Sie die MiRDP2-Pipeline.
- Um zu testen, ob miRDP2 ordnungsgemäß installiert wurde, verwenden Sie die Testdaten und die erwartete Ausgabe in https://sourceforge.net/projects/mirdp2/files/TestData/. Testdaten enthalten eine formatierte GSM-Sequenzierungsdatei und eine Arabidopsis thaliana Genomdatei.
- Verschieben Sie alle heruntergeladenen Dateien in das aktuelle Arbeitsverzeichnis:
mv miRDP2-v*.tar.gz TestData.tar.gz ncRNA_rfam.tar.gz
cd - Extrahieren Sie die komprimierten Tarball-Dateien:
tar –xvzf miRDP2-v*.tar.gz
tar –xvzf TestData.tar.gz
tar –xvzf ncRNA_rfam.tar.gz - Erstellen Sie den Arabidopsis Genom Referenzindex:
bowtie2-build -f ./TestData/TAIR10_genome.fa ./TestData/TAIR10_genome - Erstellen Sie den ncRNA-Referenzindex:
bowtie2-build -f ./ncRNA_rfam.fa ./1.1.3/script/index/rfam_index - Führen Sie die miRDP2-Pipeline aus:
bash ./1.1.3/miRDP2-v1.1.3_pipeline.bash –g ./TestData/TAIR10_genome.fa -i ./ TestData/TAIR 10_genome –f ./TestData/GSM2094927.fa –o .
HINWEIS: Die verwendeten Linux-Befehle sind fett und kursiv, mit Befehlszeilenoptionen in Kursivschrift. *gibt die Version von miRDP2 an (die aktuelle Version ist 1.1.3). Der Bowtie2-Build-Befehl sollte etwa 10 Minuten dauern, und die miRDP2-Pipeline sollte innerhalb weniger Minuten fertig sein.
- Überprüfen Sie die Testergebnisse.
- Beachten Sie, dass ein Ordner mit dem Namen 'GSM2094927-15-0-10' automatisch in generiert wird, der alle Zwischendateien und Ergebnisse enthält.
- Prüfen Sie, ob die tab-getrennte Ausgabedatei GSM2094927-15-0-10_filter_P_prediction, die endgültige Ausgabe der vorhergesagten miRNAs, Spalten enthält, die Chromosomen-ID, Strangrichtung, repräsentative Lese-ID, Vorläufer-ID, reife miRNA-Position, Vorläufer Position, ausgereifte Sequenz und Vorläufersequenz. Beachten Sie die zusätzliche Bettdatei, die aus dieser Datei abgeleitet wurde, um eine weitere Analyse zu erleichtern.
- Überprüfen Sie die Datei "progress_log", die Informationen über abgeschlossene Schritte enthält, und die Dateien "script_log" und "script_err", die Programmausgabe und Warnungen enthalten.
HINWEIS: Derzeit haben wir miRDP2 auf zwei Linux-Plattformen getestet, einschließlich CentOS Release 6.5 auf einem Cluster-Server und Cygwin 2.6.0 auf DEM PC-Windows-System, und miRDP2 sollte auf ähnlichen Systemen funktionieren, die Perl unterstützen.
2. Identifizieren neuartiger miRNAs
- Stellen Sie vor dem Ausführen der Pipeline sicher, dass die Eingabelesevorgänge im richtigen Format vorverarbeitet werden.
HINWEIS: Die neue Version 1.1.3 von miRDP2 kann originale FASTQ-Formatdateien als Eingaben akzeptieren, obwohl der Prozess der Formatierung von Lesevorgängen wie in früheren Versionen durchgeführt wird.- Entfernen Sie zunächst Adapter von den 5' und 3' Enden der tiefen Sequenzierungslesungen (falls vorhanden).
- Zweitens analysieren Sie die Deep-Sequenzierungs-Lesevorgänge im FASTA-Format.
- Drittens: Entfernen Sie Redundanz, sodass Lesevorgänge mit identischer Reihenfolge mit einem einzigen und eindeutigen FASTA-Eintrag dargestellt werden.
- Stellen Sie schließlich sicher, dass alle FASTA-Bezeichner eindeutig sind. Jeder Sequenzbezeichner muss mit einem "_x" und einer ganzzahligen Datei enden, die die Kopiernummer der genauen Sequenz angibt, die in den Deep-Sequenzing-Datasets abgerufen wurde. Eine Möglichkeit, einen eindeutigen FASTA-Bezeichner sicherzustellen, besteht darin, eine laufende Nummer in die ID aufzunehmen. Als Referenz siehe die Datei GSM2094927.fa in den Testdaten (https://sourceforge.net/projects/mirdp2/files/TestData/).
- Beispiele für korrekt formatierte Lesevorgänge finden Sie im Folgenden:
>read0_x29909
TTTGGATTGAAGGCTCTA
>read1_x36974
TTCCACAGCTTTCTTGAACTG
>read2_x32635
TTCCACAGCTTTCTTGAACTT
- Erstellen Sie Referenzindizes.
- Um Zeit zu sparen, laden Sie Bowtie2-Indexdateien von der iGenomes-Website (https://support.illumina.com/sequencing/sequencing_software/igenome.html) herunter, wenn die Genomsequenzen der interessierten Arten indiziert wurden. Andernfalls indizieren Benutzer Referenzsequenzen und behalten die Indexdatei für eine Weile, bis das Projekt abgeschlossen ist, da die Genomsequenz möglicherweise neu indiziert werden muss. Einzelheiten zum Indexieren einer Genomreferenz sind im Bowtie2-Handbuch enthalten (http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml).
- Ein weiterer Nicht-miRNA-ncRNA-Index wird ebenfalls benötigt, um laute Sequenzen aus anderen nicht-kodierenden RNA-Fragmenten herauszufiltern. Die Datei ist eine Sammlung von ncRNA-Hauptsequenzen von Rfam, einschließlich rRNA, tRNA, snRNA und snoRNA. Um diesen Index zu erstellen, lesen Sie bitte Teil 1.4, da der Index korrekt platziert und benannt werden sollte, d. h. /script/index/rfam_index.
- Führen Sie miRDP2 aus.
- Um miRDP2 zum Erkennen neuer miRNAs aus deep sequencing-Daten zu verwenden, führen Sie das Bash-Skript im Paket aus, um die Analysepipeline zu starten (Ein Beispiel finden Sie in Schritt 1.4):
/miRDP2-v*.*_pipeline.bash –g -i -f -o
wobei * die Version des Pipeline-Bash-Skripts angibt. Es gibt drei Parameter, die geändert werden können: 1) die Anzahl der verschiedenen Positionen, denen ein Leseort zugeordnet werden könnte, 2) die Nichtübereinstimmungsnummer für das Ausführen von bowtie2 und 3) den Schwellenwert für RPM (Reads Per Million). Ändern Sie diese mit den Optionen –L, -M und –R. Eine ausführliche Erläuterung finden Sie in Abschnitt 3.1.
- Um miRDP2 zum Erkennen neuer miRNAs aus deep sequencing-Daten zu verwenden, führen Sie das Bash-Skript im Paket aus, um die Analysepipeline zu starten (Ein Beispiel finden Sie in Schritt 1.4):
- Überprüfen Sie die miRDP2-Ausgänge.
- Beachten Sie, dass der Ausgabeordner automatisch unter generiert wird und den Namen '-15-0-10'; Die letzten 3 Zahlen geben die Werte (in diesem Fall Standard) für die Parameter 1, 2 und 3 an. Die Datei _filter_P_prediction enthält Informationen über die endgültigen vorhergesagten miRNAs, die die neu aktualisierten pflanzlichen miRNA-Anmerkungskriterien erfüllen. Details zum Format der Ausgabedatei werden in Teil 1.4 beschrieben.
3. Änderungen und Vorsicht mit miRDP2
- Parameter, die geändert werden können
- Verwenden Sie die Option '-L', um die Begrenzung festzulegen, wie viele Positionen ein Leseort zugeordnet werden kann (Parameter 1). Die Lesezuordnung zu zu vielen Standorten ist möglicherweise mit Wiederholungssequenzen verknüpft und kann wahrscheinlich nicht mit miRNAs verknüpft werden. Die Standardeinstellung ist 15. Bei bestimmten Arten kann der erste Parameter manuell erhöht werden, um sich an die Genomlandschaft anzupassen, wenn es miRNA-Familien mit vielen Mitgliedern gibt.
- Verwenden Sie die Option '-M', um die zulässigen Nichtübereinstimmungen für Bowtie festzulegen (Parameter 2). Die Standardeinstellung ist 0.
- Verwenden Sie die Option '-R', um den Schwellenwert für Lesevorgänge festzulegen, die möglicherweise reifen miRNAs entsprechen (Parameter 3). Um den Zeitverbrauch und False-Positiv wertet, werden Filter nach Drehzahl gelesen. Nur Lesevorgänge, die einen bestimmten Drehzahlschwellenwert überschreiten, können reife Sequenzen von miRNAs und nicht Hintergrundrauschen darstellen und würden zur weiteren Analyse aufbewahrt. Die Standardeinstellung ist 10 RPM.
- Beachten Sie, dass das Ändern dieser Parameter die Leistung und den Zeitverbrauch beeinträchtigen kann. Im Allgemeinen würde eine Erhöhung der Parameter 1 und 2 und eine Abnahme von Parameter 3 ein weniger strenges Ergebnis und eine längere Laufzeit erzeugen und umgekehrt.
- Redundanz und miRNA*
- Beachten Sie, dass die Ausgabe-miRNAs von miRDP2 von den bekannten miRNAs abweichen können. Wir fanden heraus, dass dies hauptsächlich auf einen von zwei Gründen zurückzuführen ist: Heterogenität der reifen miRNAs oder die relative Fülle von miRNA und miRNA*. Wir fanden heraus, dass dies keine Auswirkungen auf die optimale Längenauswahl von Vorläufern und die Profilierung bekannter miRNA-Gene hat.
Ergebnisse
Die miRNA-Annotationspipeline miRDP2, die hier beschrieben wird, wird auf 10 öffentliche sRNA-seq-Bibliotheken von 5 Pflanzenarten mit allmählich erhöhter Genomlänge angewendet, einschließlich Arabidopsis thaliana, Oryza sativa (Reis), Solanum lycopersicum (Tomate), Zea mays (Mais) und Triticum aestivum (Weizen) (Abbildung 1A). Insgesamt werden für jede Spezies 2 repräsentative sRNA-Bibliotheken aus verschiedenen Geweben (i...
Diskussion
Mit dem Aufkommen von NGS wurde eine große Anzahl von miRNA-Loci aus einer ständig wachsenden Menge an sRNA-Sequenzierungsdaten in verschiedenen Artenidentifiziert 29,30. In der zentralen Community-Datenbank miRBase21haben sich die hinterlegten miRNA-Artikel in den letzten zehn Jahren fast 100 Mal erhöht. Im Vergleich zu miRNAs bei Tieren haben pflanzen-miRNAs jedoch viele einzigartige Merkmale, die die Identifizierung/Anmerkung kompliz...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde von der Beijing Academy of Agriculture and Forestry Sciences (KJCX201917, KJCX20180425 und KJCX20180204) an XY und National Natural Science Foundation of China (31621001) bis LL unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Computer/computing node | N/A | N/A | Perl is required; at least 8 GB RAM and 100 GB storage are recommended |
Referenzen
- Ghildiyal, M., Zamore, P. D. Small silencing RNAs: an expanding universe. Nature Reviews Genetics. 10 (2), 94-108 (2009).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Moran, Y., Agron, M., Praher, D., Technau, U. The evolutionary origin of plant and animal microRNAs. Nature Ecology Evolution. 1 (3), 27 (2017).
- Xie, Z., et al. Expression of Arabidopsis MIRNA genes. Plant Physiology. 138 (4), 2145-2154 (2005).
- Zhao, X., Zhang, H., Li, L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics. 101 (3), 187-194 (2013).
- Bologna, N. G., Mateos, J. L., Bresso, E. G., Palatnik, J. F. A loop-to-base processing mechanism underlies the biogenesis of plant microRNAs miR319 and miR159. EMBO JOURNAL. 28 (23), 3646-3656 (2009).
- Rogers, K., Chen, X. Biogenesis, turnover, and mode of action of plant microRNAs. Plant Cell. 25 (7), 2383-2399 (2013).
- Voinnet, O. Origin, biogenesis, and activity of plant microRNAs. Cell. 136 (4), 669-687 (2009).
- Iwakawa, H. O., Tomari, Y. The Functions of MicroRNAs: mRNA Decay and Translational Repression. Trends in Cell Biology. 25 (11), 651-665 (2015).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75 (5), 855-862 (1993).
- Friedlander, M. R., et al. Discovering microRNAs from deep sequencing data using miRDeep. Nature Biotechnology. 26 (4), 407-415 (2008).
- Yang, X., Li, L. miRDeep-P: a computational tool for analyzing the microRNA transcriptome in plants. Bioinformatics. 27 (18), 2614-2615 (2011).
- Meyers, B. C., et al. Criteria for annotation of plant MicroRNAs. Plant Cell. 20 (12), 3186-3190 (2008).
- Yang, X., Zhang, H., Li, L. Global analysis of gene-level microRNA expression in Arabidopsis using deep sequencing data. Genomics. 98 (1), 40-46 (2011).
- Kuang, Z., Wang, Y., Li, L., Yang, X. miRDeep-P2: accurate and fast analysis of the microRNA transcriptome in plants. Bioinformatics. , (2018).
- Kozomara, A., Birgaoanu, M., Griffiths-Jones, S. miRBase: from microRNA sequences to function. Nucleic Acids Research. 47 (1), 155-162 (2019).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucleic Acids Research. 36, 154-158 (2008).
- Axtell, M. J., Meyers, B. C. Revisiting Criteria for Plant MicroRNA Annotation in the Era of Big Data. Plant Cell. 30 (2), 272-284 (2018).
- Taylor, R. S., Tarver, J. E., Hiscock, S. J., Donoghue, P. C. Evolutionary history of plant microRNAs. Trends in Plant Science. 19 (3), 175-182 (2014).
- Kozomara, A., Griffiths-Jones, S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 42, 68-73 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, 26 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), 25 (2009).
- An, J., Lai, J., Sajjanhar, A., Lehman, M. L., Nelson, C. C. miRPlant: an integrated tool for identification of plant miRNA from RNA sequencing data. BMC Bioinformatics. 15, 275 (2014).
- Lei, J., Sun, Y. miR-PREFeR: an accurate, fast and easy-to-use plant miRNA prediction tool using small RNA-Seq data. Bioinformatics. 30 (19), 2837-2839 (2014).
- Evers, M., Huttner, M., Dueck, A., Meister, G., Engelmann, J. C. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics. 16, 370 (2015).
- Mathelier, A., Carbone, A. MIReNA: finding microRNAs with high accuracy and no learning at genome scale and from deep sequencing data. Bioinformatics. 26 (18), 2226-2234 (2010).
- Zhu, Q. H., et al. A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains. Genome Research. 18 (9), 1456-1465 (2008).
- Fahlgren, N., et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana. Plant Cell. 22 (4), 1074-1089 (2010).
- Fromm, B., et al. A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome. Annual Review of Genetics. 49, 213-242 (2015).
- Blevins, T., et al. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding de novo DNA methylation in Arabidopsis. Elife. 4, 09591 (2015).
- Zhai, J., et al. A One Precursor One siRNA Model for Pol IV-Dependent siRNA Biogenesis. Cell. 163 (2), 445-455 (2015).
- Werner, S., Wollmann, H., Schneeberger, K., Weigel, D. Structure determinants for accurate processing of miR172a in Arabidopsis thaliana. Current Biology. 20 (1), 42-48 (2010).
- Mateos, J. L., Bologna, N. G., Chorostecki, U., Palatnik, J. F. Identification of microRNA processing determinants by random mutagenesis of Arabidopsis MIR172a precursor. Current Biology. 20 (1), 49-54 (2010).
- Vitsios, D. M., et al. Mirnovo: genome-free prediction of microRNAs from small RNA sequencing data and single-cells using decision forests. Nucleic Acids Research. 45 (21), 177 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten