Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Herstellung von multifunktionalen Mikrokapseln auf Seidenbasis, die mit DNA-Plasmiden beladen sind, die für RNA, Aptamere und Riboschalter kodieren
In diesem Artikel
Zusammenfassung
Das Protokoll beschreibt die Bildung robuster und biokompatibler DNA-beladener Mikrokapseln als gemultiplexte In-vitro-Biosensoren , die in der Lage sind, mehrere Liganden zu verfolgen.
Zusammenfassung
Wir stellen ein Protokoll für die Herstellung von DNA-beladenen Seidenfibroin-Mikrokapseln über die Layer-by-Layer (LbL) Assemblierungsmethode auf sphärischen Opferkernen vor. Nach Adsorption einer Hauptschicht und DNA-Plasmiden wurde die Bildung robuster Mikrokapseln durch die Induktion von β-Blättern in der Seidensekundärstruktur während der akuten Dehydratisierung einer einzelnen Seidenschicht erleichtert. Daher erfolgte die Schichtung über mehrfache Wasserstoffbrückenbindungen und hydrophobe Wechselwirkungen. Nach der Adsorption von mehrschichtigen Hüllen können die Kern-Schale-Strukturen mit Goldnanopartikeln (AuNPs) und/oder Antikörpern (IgG) weiter funktionalisiert werden, um sie für die Fernerkundung und/oder den gezielten Transport zu verwenden. Die Einstellung mehrerer Schlüsselparameter während der sequentiellen Abscheidung von wichtigen Makromolekülen auf Siliziumdioxidkernen, wie z. B. das Vorhandensein eines Polymerprimers, die Konzentration von DNA und Seidenprotein sowie eine Reihe von adsorbierten Schichten, führte zu biokompatiblen, DNA-beladenen Mikrokapseln mit variabler Permeabilität und DNA-Beladung. Nach der Auflösung von Siliziumdioxidkernen zeigte das Protokoll die Bildung von hohlen und robusten Mikrokapseln mit DNA-Plasmiden, die an der Innenfläche der Kapselmembran immobilisiert waren. Durch die Schaffung einer selektiv permeablen biokompatiblen Membran zwischen den DNA-Plasmiden und der äußeren Umgebung blieb die DNA während der Langzeitlagerung erhalten und spielte eine wichtige Rolle bei der verbesserten Output-Antwort von räumlich begrenzten Plasmiden. Die Aktivität von DNA-Templates und ihre Zugänglichkeit wurden während in vitro Transkriptions- und Translationsreaktionen (zellfreie Systeme) getestet. DNA-Plasmide, die für RNA-Light-up-Aptamere und Riboschalter kodieren, wurden erfolgreich mit entsprechenden Analyten aktiviert, wie bei der Lokalisierung von fluoreszenzmarkierten RNA-Transkripten oder GFPa1-Proteinen in den Schalenmembranen sichtbar wurde.
Einleitung
Das Gebiet der synthetischen Biologie bietet einzigartige Möglichkeiten, Sensorfähigkeiten zu entwickeln, indem natürliche Mechanismen genutzt werden, die von Mikroorganismen entwickelt wurden, um ihre Umwelt und potenzielle Bedrohungen zu überwachen. Wichtig ist, dass diese Sensormechanismen in der Regel mit einer Reaktion verbunden sind, die diese Mikroorganismen vor schädlicher Exposition schützt und die Genexpression reguliert, um negative Auswirkungen zu mildern oder die Aufnahme giftiger Materialien zu verhindern. Es wurden erhebliche Anstrengungen unternommen, um diese Mikroorganismen so zu verändern, dass sie Ganzzellsensoren herstellen, die diese natürlichen Reaktionen nutzen, sie aber umleiten, um neue Ziele zu erkennen und/oder ein messbares Signal zu erzeugen, das zu Quantifizierungszwecken gemessen werden kann (typischerweise Fluoreszenz)1,2. Gegenwärtig deuten Bedenken hinsichtlich der Verwendung genetisch veränderter Mikroorganismen (GVO), insbesondere bei der Freisetzung in der Umwelt oder im menschlichen Körper, aufgrund des Austritts ganzer Zellen oder eines Teils ihres genetischen Materials, selbst wenn es in einer Polymermatrix eingekapselt ist, darauf hin, dass alternative Wege zur Nutzung dieser Sensoransätze erforderlich sind3.
Ein wirksamer Ansatz, um die Vorteile der mikroorganismenbasierten Sensorik zu nutzen, ohne sich Gedanken über den Einsatz von GVO machen zu müssen, ist die Verwendung von In-vitro-Transkriptions-/Translationssystemen (IVTT). Aus praktischer Sicht bestehen IVTT-Systeme aus einer Mischung, die die meisten Zellbestandteile in einem aktiven Zustand enthält, die auf unterschiedliche Weise aus den Zellen "extrahiert" wurden,z. B. durch Beschallung, Perlenschlagen oder andere 4. Das Endprodukt dieses Prozesses ist ein biochemisches Reaktionsgemisch, das bereits für die Transkription und Translation optimiert ist und zum Testen verschiedener Sensoren in einem "offenen Gefäß"-Format verwendet werden kann, ohne die Einschränkungen, die mit der Verwendung ganzer Zellen verbunden sind (Membrandiffusion, Transformationseffizienz, Zelltoxizität usw.). Wichtig ist, dass verschiedene Sensorkomponenten quantitativ hinzugefügt und ihre Wirkung durch verschiedene optische und spektrometrische Techniken untersucht werden können, wie wir gezeigt haben5. Es wurde festgestellt, dass die Leistung von IVTT-Systemen inkonsistent sein kann. Neuere Studien haben jedoch Ansätze zur Standardisierung ihrer Präparation und Charakterisierung gezeigt, was bei der Untersuchung ihrer Leistung im Sensordesign eine große Hilfe ist6. In jüngster Zeit wurden viele Beispiele für IVTT-Systeme demonstriert, die zur Herstellung papierbasierter Assays durch die Gefriertrocknung ihrer Komponenten in Papiermatrizes verwendet werden, einschließlich der Detektion von Schwermetallionen, Drogen, Quorum-Sensorelementen und anderen 7,8,9. Ein spannender Anwendungsbereich für IVTT-basierte Sensoren ist ihr Einsatz in Sensoranwendungen in verschiedenen Arten von Umgebungen, einschließlich Boden, Wasser und dem menschlichen Körper. Um diese IVTT-Systeme in diesen anspruchsvollen Umgebungen einsetzen zu können, muss ein Kapselungsansatz implementiert werden, um die IVTT-Komponenten einzudämmen und sie vor Verschlechterung zu schützen.
Die gebräuchlichsten Verkapselungsansätze für IVTT-Systeme umfassen die Verwendung von Lipidkapseln, Mizellen, Polymersomen und anderen dicht geschlossenen Mikrobehältern10,11,12. Ein Nachteil dieses Ansatzes ist die Notwendigkeit, entweder passive oder aktive Mechanismen für den Transport von Materialien in und aus den Behältern zu integrieren, um die Kommunikation mit der externen Umgebung zu ermöglichen und Sensorfunktionen bereitzustellen. Um einige dieser Probleme zu überwinden, berichtet die Studie hier über eine Methode, die einen einfachen, aber effektiven Ansatz zur Verkapselung der Kodierungsmaterialien für verschiedene Sensordesigns bietet, die in IVTT-Systemen ausgedrückt werden sollen. Dieser Ansatz basiert auf der Verwendung der Layer-by-Layer (LbL)-Abscheidung eines Biopolymers in Gegenwart der interessierenden Plasmide, um hohle Mikrokapseln mit hoher Porosität herzustellen, die es dem geschützten genetischen Material ermöglichen, mit den verschiedenen Komponenten des IVTT der Wahl zu interagieren. Die Studie zeigte, dass verkapselte Plasmide die Transkription und Translation steuern können, wenn sie innerhalb dieser polymeren Matrix aktiviert werden, wie die Reaktion eines Plasmid-kodierten Aptamers und eines Riboschalters auf ihre entsprechenden Ziele zeigt. Zusätzlich schützt diese LbL-Beschichtung die Plasmide monatelang ohne besondere Lagerbedingungen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Konstruktion des Plasmidvektors.
- Konstruieren eines Plasmidvektors (pSALv-RS-GFPa1, 3,4 kb) durch Amplifikation der kodierenden Sequenz eines Theophyllin-Riboschalters (ThyRS) gekoppelt mit GFPa1 aus pJ201:23976-RS-GFPa1-Vektor (entworfen und erzeugt von DNA2.0) und Insertion in den E . coli-Expressionsvektor pSAL13. Verwenden Sie vorwärts (5'-CGTGGTACCGGTGATACCAGCATCGTCTTGATG-3') und umgekehrt (5'-CGTGCTCAGCTTAAGCCAGCTCGTAG-3') Primer, um die kodierende Sequenz von ThyRS gekoppelt mit GFPa1 zu amplifizieren und eine PCR-Reaktion in 50 μl Volumen unter Verwendung der DNA-Polymerase gemäß dem Protokoll des Herstellersdurchzuführen 14.
- Bereiten Sie ein 1%iges Agarose-Gel aus 0,5 g Agarose, 50 ml TAE-Puffer (40 mM Trisacetat, 1 mM EDTA, pH 8,0) und 3 μl DNA-Färbung vor.
- Mischen Sie 5 μL aliquot des PCR-amplifizierten Produkts mit 5 μL RNase/DNase-freiem Wasser und 2 μL 6x Gelladefarbstoff und analysieren Sie es mittels Agarose-Gelelektrophorese. Laden Sie eine DNA-Leiter (0,1-10,0 kb) als Referenz. Lassen Sie das Gel bei 120 V laufen, bis die Färbelinie fast den Boden des Gels erreicht hat.
- Visualisieren Sie die DNA-Fragmente mit einem UV-Transilluminator-Bildgebungssystem, um die richtige Größe von DNA15 zu überprüfen.
- Reinigen Sie das PCR-Produkt mit einem PCR-Aufreinigungskit gemäß dem Protokoll des Herstellers16.
- Das PCR-Produkt und der pSAL-Expressionsvektor werden mit KpnI- und BlpI-Restriktionsenzymen in einer 15-μl-Reaktion aufgeschlossen, die 10 μl PCR-Produkt oder Plasmidvektor (Konzentration 20-50 ng/μl), 1,5 μl 10x Enzympuffer, 1 μl jedes Enzyms und 1,5 μl RNase/DNase-freies Wasser enthält, bei 37 °C für 2 h.
- Fügen Sie der Reaktionsmischung 3 μl 6x Gelladefarbstoff hinzu und trennen Sie die aufgeschlossenen Fragmente auf einem 1%igen Agarosegel, wie in den Schritten 1.3 bis 1.5 beschrieben.
- Reinigen Sie die DNA-Fragmente mit einem Gelextraktionskit gemäß dem Protokoll des Herstellers16.
- Ligatieren Sie das verdaute PCR-Produkt in einen verdauten linearisierten Plasmidvektor, pSAL, unter Verwendung der T4-DNA-Ligase und des supplementierten Ligasepuffers in einer 10-μl-Reaktion, die 3-20 fmol des verdauten Vektors, 9-60 fmol des verdauten PCR-Produkts, 2 μl Ligasepuffer, 1 μl (1 Einheit) T4-DNA-Ligase und DNase/RNase-freies Wasser enthält. Inkubieren Sie die Ligationsreaktion bei 25 °C für 3 h.
Anmerkungen: Stellen Sie sicher, dass der Gesamt-DNA-Gehalt im Reaktionsgemisch 0,01-0,1 μg beträgt. - Transformieren Sie E. coli DH5α-kompetente Zellen mit 10 ng des Ligationsreaktionsgemisches gemäß dem Protokoll des Herstellers17.
- Die transformierten Zellen werden bei 37 °C über Nacht auf LB-Agarplatten gezüchtet, die mit Ampicillin (100 μg/ml) angereichert sind.
- Nehmen Sie 3-4 Bakterienkolonien von der Platte und übertragen Sie jede von ihnen aseptisch in 5 ml LB-Medien, die mit Ampicillin (100 μg/ml) angereichert sind. Die Kulturen werden über Nacht bei 37 °C in einem Schüttelinkubator bei 225 U/min gezüchtet.
- Pelletieren Sie die Kulturen über Nacht durch Zentrifugieren bei 11 x g für 3 min bei Raumtemperatur.
- Verwenden Sie ein Aufreinigungskit, um die Plasmide gemäß dem Protokoll des Herstellers16 zu reinigen.
- Verifizieren Sie die Sequenzen der gereinigten Plasmide durch DNA-Sequenzierung. Die Plasmidkarte und die Sequenz des resultierenden Konstrukts (pSALv-RS-GFPa1) sind in Abbildung 1 dargestellt.
2. DNA-Aufreinigung im großen Maßstab.
- Der Plasmidvektor pSALv-RS-GFPa1 (3,4 kb) (kodiert für Theophyllin-Riboswitch gekoppelt mit GFPa1-Reportergen) oder pET28c-F30-2xBroccoli (5,4 kb) (kodiert für Brokkoli-Aptamer) wird gemäß dem Protokoll des Herstellers in E. coli DH5α-kompetente Zellen transformiert17.
- Die transformierten Zellen werden bei 37 °C über Nacht auf LB-Agar-Platten gezüchtet, die mit Ampicillin (100 μg/ml) für Zellen, die mit pSALv-RS-GFPa1 transformiert wurden, oder Kanamycin (50 μg/ml) für Zellen, die mit pET28c-F30-2x Brokkoli transformiert wurden, ergänzt werden.
- Nehmen Sie 3-4 Bakterienkolonien von der Platte und übertragen Sie jede Kolonie aseptisch in 5 ml LB-Medien, die mit einem geeigneten Antibiotikum (100 μg/ml Ampicillin oder 50 μg/ml Kanamycin) ergänzt werden. Die Kulturen werden über Nacht bei 37 °C in einem Schüttelinkubator bei 225 U/min gezüchtet.
- Verwenden Sie 3 ml der Übernachtkultur, um in 150 ml LB zu inokulieren, die mit einem geeigneten Antibiotikum (100 μg/ml Ampicillin oder 50 μg/ml Kanamycin) ergänzt werden, und züchten Sie die Kulturen über Nacht bei 37 °C in einem Schüttelinkubator bei 225 U/min.
- Die Zellen werden durch Zentrifugation bei ≥3400 x g für 10 min bei 4 °C pelletiert.
- Verwenden Sie ein Aufreinigungskit, um die Plasmide gemäß dem Protokoll des Herstellers16 zu reinigen.
- Die DNA wird mit 0,5 ml reinem DNase/RNase-freiem Wasser eluiert. Messen Sie die DNA-Konzentration und bereiten Sie 1 ml DNA-Stammlösungen (100 ng/μl) vor. Lagern Sie die Röhrchen mit DNA bis zur weiteren Verwendung bei 4 °C.
3. Extraktion von Seidenfibroin und Aufbereitung der Ausgangsmaterialien.
- Bereiten Sie eine wässrige Lösung von rekonstituiertem Seidenfibroin (SF)-Protein aus Bombyx mori-Seidenraupenkokons gemäß dem an anderer Stelle ausführlich beschriebenen Verfahren vor, das 10 % der Silk-LiBr-Lösung18 ausmacht.
- Bestimmen Sie die Endkonzentration der wässrigen SF-Lösung. Pipettieren Sie 0,5 ml Seidenlösung in eine 60-mm-Petrischale, lassen Sie sie bei 60 °C trocknen und messen Sie das Gewicht des trockenen Seidenfilms. Teilen Sie das Trockengewicht durch 0,5 ml, um den Gewichtsprozentsatz pro Volumen zu berechnen.
- Verdünnen Sie die konzentrierte Seidenlösung mit DNase/RNase-freiem destilliertem Wasser, indem Sie langsam Wasser über eine serologische Pipette hinzufügen, um eine Endkonzentration von 1 mg/ml zu erhalten. Lagern Sie die Lösung für die spätere Verwendung bei 4 °C.
- Fluoreszenzmarkiertes Seidenfibroin mit einem Antikörpermarkierungskit herstellen. Verwenden Sie 1 ml 2 mg/ml Seidenfibroinlösung, um die N-terminalen α-Aminogruppen des Proteins mit einem NHS-Ester-aktivierten derivativen Farbstoff gemäß dem Protokoll des Herstellers zu koppeln19.
- Bereiten Sie 50 ml wässrige Lösung aus Polyethylenimin (PEI) mit einer Konzentration von 6 mg/ml vor und stellen Sie den pH-Wert mit HCl (1 M) auf 4 ein. Filtern Sie die Lösung durch eine sterile 0,2 μm Membran. Die Lagerung ist bei Raumbedingungen über Monate möglich.
- Bereiten Sie SiO2-Kerne vor. Pipettieren Sie 300 μlSiO2-Partikel in ein 2-ml-Mikrozentrifugenröhrchen. Waschen Sie die Mikropartikel zweimal mit 1 ml DNase/RNase-freiem Wasser durch Zentrifugieren bei 0,2 x g für 1 Minute.
4. Führen Sie eine schichtweise Abscheidung einer Hauptschicht, von DNA-Plasmiden und Seidenschichten durch.
- Um die PEI-Prime-Schicht auf den SiO2-Mikropartikeln abzuscheiden, wird 1 ml PEI-Lösung in das abgeschleuderte Pellet aus Schritt 3.6 gegeben und das Gemisch bei Umgebungsbedingungen auf einem Thermomischer bei 800 U/min 15 min lang gerührt. Waschen Sie die Partikel viermal mit 1 ml DNase/RNase-freiem deionisiertem Wasser durch Zentrifugation bei 0,2 x g für 1 min.
- Um die Abscheidung der DNA-Schicht durchzuführen, wird 1 ml der wässrigen Lösung von DNA-Plasmiden aus Schritt 2.7 zu den PEI-grundierten Mikropartikeln gegeben und das Gemisch bei 4 °C auf einem Thermomischer bei 800 U/min 15 min lang vorsichtig gerührt. Um Mikrokapseln mit unterschiedlichen DNA-Beladungen herzustellen, stellen Sie die Konzentration der DNA-Plasmide von 50-200 ng/μl mit DNase/RNase-freiem destilliertem Wasser ein und verwenden Sie 1 ml dieser Lösungen, um die DNA abzuscheiden. Sammeln Sie die Mikropartikel durch Zentrifugation bei 0,2 x g für 1 min.
- Markieren Sie die Röhrchen für DNA-Plasmide, die für Theophyllin-Riboschalter kodieren, gekoppelt mit GFPa1 als ThyRS-GFPa1, und für DNA-Plasmide, die für Brokkoli-Aptamer als BrocApter kodieren.
Anmerkungen: Bewahren Sie die Mikrozentrifugenröhrchen mit DNA auf Eis auf. - Entfernen Sie vorsichtig den Überstand und waschen Sie die Mikropartikel viermal mit 1 ml DNase/RNase-freiem destilliertem Wasser, wobei Sie den Überstand jedes Mal nach der Zentrifugation bei 0,2 x g für 1 Minute verwerfen. Führen Sie alle Experimente bei Raumtemperatur (RT) durch, sofern nicht anders angegeben.
- Um die Abscheidung der Seidenfibroinschicht durchzuführen, wird 1 ml der rekonstituierten wässrigen SF-Lösung aus Schritt 3.3 zu den DNA-adsorbierten Mikropartikeln gegeben, vorsichtig vortexiert und das Gemisch bei 10 °C auf dem Thermomischer bei 750 U/min für 15 min gerührt. Sammeln Sie die Mikropartikel durch Zentrifugieren bei 0,2 x g für 1 min bei 4 °C, entfernen Sie den Überstand und waschen Sie sie dann einmal mit 1 ml DNase/RNase-freiem destilliertem Wasser. Wiederholen Sie die Zentrifugation und entsorgen Sie den Überstand.
Anmerkungen: Halten Sie die Seidenlösung während des Experiments auf Eis, um eine temperaturbedingte Gelierung zu vermeiden. - Behandeln Sie die Partikel nach und nach mit Methanol, um die Bildung von β-Faltblättern in der Seidenproteinstruktur zu induzieren. Fügen Sie zuerst 0,5 ml DNase/RNase-destilliertes Wasser hinzu, wirbeln Sie das Mikrozentrifugenröhrchen und fügen Sie dann 0,5 ml 100%iges Methanol hinzu. Schütteln Sie die Partikel auf dem Thermomischer bei 10 °C 5 min lang vorsichtig. Sammeln Sie die Partikel durch Zentrifugieren bei 0,2 x g für 1 min. Entfernen Sie den Überstand.
- Behandeln Sie die Partikel mit Methanol, um die Bildung von β-Blättern zu fördern und eine starke physikalische Adsorption der Seidenschicht zu gewährleisten. Fügen Sie 1 ml 100%iges Methanol hinzu. Schütteln Sie die Partikel auf dem Thermomischer bei 750 U/min für 10 min bei 10 °C vorsichtig.
- Sammeln Sie die Partikel durch Zentrifugieren bei 0,2 x g für 1 min bei 4 °C und waschen Sie sie jedes Mal zweimal mit 1 ml DNase/RNase-freiem destilliertem Wasser, wobei Sie den Überstand verwerfen und vor der nächsten Zentrifugation vorsichtig wirbeln.
- Wiederholen Sie die Schritte 4.5-4.8 20 Mal, um mehrschichtige Kern-Schale-Strukturen aus Seide zu erhalten. Für den letzten Abscheidungsschritt verwenden Sie fluoreszenzmarkierte Seide aus Schritt 3.4 (Silk-DyLight550, 1 ml).
- Führen Sie den letzten Waschschritt durch und bewahren Sie die Mikropartikel in 1 ml DNase/RNase-freiem destilliertem Wasser bei Umgebungsbedingungen auf.
Anmerkungen: Um eine Aggregation von Partikeln während der Abscheidung von Seidenschichten zu vermeiden, führen Sie eine Sichtprüfung der Partikelsuspension durch und pipettieren Sie sie mit einer 1-ml-Pipettenspitze auf und ab, um eine homogene Partikelverteilung zu fördern. - Berechnen Sie die Anzahl der DNA-Plasmidkopien, die in jeder Mikrokapsel eingekapselt sind, N-DNA mit Gleichung 1:
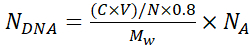 (1)
(1)
Wobei N = 6,769 × 1011 - die Anzahl der SiO2-Kerne, die für die Kapselung verwendet werden. Berechnen Sie sie anhand einer Standardkurve für bekannte Konzentrationen von Siliziumdioxidpartikeln unter Verwendung von Verdünnungsreihen und Absorption A 320 bei λ =320 nm;
C- Anfangskonzentration der DNA, die für die Adsorption verwendet wird
V- das DNA-Volumen, das für die Adsorption verwendet wird
0,8- DNA-Adsorptionseffizienz auf den Kernen
Mw- Molekulargewicht des DNA-Plasmids
NA- Avogadro-Zahl (6.022 × 1023)
5. Auflösung der Kerne zur Gewinnung von Seidenmikrokapseln.
- Bereiten Sie eine 8%ige Flusssäure (HF)-Lösung mit einem pH-Wert von 5,5 vor, indem Sie die Stammlösung (48%) mit destilliertem Wasser verdünnen. Besorge dir ein 50-ml-Zentrifugenröhrchen. Pipettieren Sie vorsichtig 5 ml HF und fügen Sie 25 ml destilliertes Wasser hinzu, um eine 8%ige HF-Lösung zu erhalten.
VORSICHT: HF ist eine stark ätzende Säure und kann schwere Verbrennungen im Gewebe verursachen. Bei der Handhabung und Verwendung von HF für die Experimente ist äußerste Vorsicht geboten. Halten Sie sich an die Standardarbeitsanweisungen (SOP) für die ordnungsgemäße Verwendung und Handhabung von HF, die von der Organisation entwickelt wurden, um unerwünschte Unfälle durch Verschütten zu vermeiden. Verwenden Sie keine Glasbehälter, um HF-Säure zu verdünnen. Verwenden Sie die chemische Haube, um diesen Schritt des Protokolls durchzuführen. - Lösen Sie SiO-2-Kerne auf, indem Sie 1,5 ml 8%ige HF-Lösung zu pelletierten Kern-Schale-Mikropartikeln aus Schritt 4.10 hinzufügen. Sanft wirbeln und lassen Sie die Kerne über Nacht bei Umgebungsbedingungen mit leichtem Schütteln bei 450 U/min auflösen.
Anmerkungen: Um das Verschütten von HF zu vermeiden, verwenden Sie Pfropfband, um das Mikrozentrifugenröhrchen zu versiegeln. Verwenden Sie eine chemische Haube, um diesen Schritt des Protokolls durchzuführen. - Bereiten Sie einen 2-Liter-Glasbecher vor, der mit 2 l deionisiertem Wasser gefüllt ist. Übertragen Sie die Mikrokapsellösung in Dialysegeräte (50 kDa MWCO) und dialysieren Sie sie gegen deionisiertes Wasser mit einem wiederholten Wasserwechsel alle 3 Stunden für die nächsten 3 Tage.
Anmerkungen: Sammeln Sie den Überstand während der ersten drei Wasserwechsel und entsorgen Sie die Lösung gemäß dem festgelegten Protokoll für gefährliche Abfälle. - Verwenden Sie eine 1-ml-Pipette, um die Suspension von Dialysegeräten in neue 2-ml-Mikrozentrifugenröhrchen zu übertragen, um die Mikrokapseln zu sammeln.
Anmerkungen: Lagern Sie die wässrigen Lösungen von Mikrokapseln mehrere Jahre bei Raumbedingungen.
6. Bildgebung von Seidenfibroin-Mikrokapseln mit konfokalem Laser-Scanning-Mikroskop (CLSM).
- Führen Sie eine DNA-Färbung mit einem DNA-Farbstoff durch.
- Übertragen Sie 300 μl hohle Seidenfibroin-Mikrokapseln in ein frisches 1-ml-Mikrozentrifugenröhrchen. Fügen Sie 500 μl RNase/DNase-freies destilliertes Wasser hinzu.
- 5 μL des DNA-Färbefarbstoffs zugeben, kurz vortexieren und bei RT 2 h lichtgeschützt inkubieren.
- Führen Sie vier Waschschritte durch, indem Sie bei 0,1 x g für jeweils 20 Minuten bei 4 °C zentrifugieren, wobei vorsichtig 400 μl des Überstands entfernt und mit 400 μl RNase/DNase-freiem destilliertem Wasser aufgefüllt werden.
- Führen Sie die Abbildung von Seidenkapseln auf invertierten konfokalen Systemen durch, die mit drei Hauptlasern (405 nm, 488 nm, 561 nm) ausgestattet sind, und verwenden Sie ein 100-faches Ölimmersionsobjektiv (NA 1,49). Übertragen Sie 100 μl der Kapselprobe in eine einzige Vertiefung mit 8-Well-Objektträgern, lassen Sie die Kapseln vor der Bildgebung 20-30 Minuten sedimentieren.
HINWEIS: Farbstoffe reagieren sehr empfindlich auf Photobleiche. Schützen Sie die Proben, indem Sie die Objektträger mit Alufolie abdecken.
7. Abschätzung der Permeabilität von Mikrohohlkapseln mittels Molekulargewichts-Cut-off-Methode (MWCO).
- Bereiten Sie jeweils 2 ml FITC-markierte Dextran-Fluorophorlösungen (20 μM, diH 2O) unterschiedlicher Mw (4 kDa, 20 kDa, 40 kDa, 70 kDa, 150 kDa,250kDa, 500 kDa und 2 MDa) vor.
- Pipettieren Sie 100 μl der Kapselsuspension in eine einzelne Vertiefung eines Objektträgers aus Kammerglas. Analysieren Sie jedes Mikrokapseldesign (Konzentration von PEI, Anzahl der DNA-Plasmide, Konzentration von Seidenfibroin und Anzahl der Schichten) separat.
- Fügen Sie jeder Vertiefung 300 μl der spezifischen Fluorophorlösung hinzu, beginnend mit dem niedrigsten Mw bis zum höchsten, so dass jede Vertiefung der spezifischen Fluorophorlösung entspricht. Mischen Sie durch Auf- und Abpipettieren und lassen Sie die Mischung 1 h bei RT inkubieren, bis die Diffusion der Fluorophorlösungen ein Gleichgewicht erreicht.
- Übertragen Sie den Objektträger in ein konfokales Laser-Scanning-Mikroskop (CLSM) und bilden Sie jede Vertiefung mit einem 100-fachen Ölimmersionsobjektiv bei Anregung λ = 488 nm ab.
- Identifizieren Sie den interessierenden Bereich, indem Sie die Fokusebene anpassen, um sicherzustellen, dass die Kapseln in Form von Kreisen mit dem größten Durchmesser erscheinen. Dies geschieht in der Regel, wenn die Proben näher am Boden des Bohrlochs betrachtet werden, wenn Kapseln aufgrund der Schwerkraft sedimentieren.
- Sammeln Sie mehrere Bilder von Mikrokapselproben, indem Sie den Objektträger in XY-Richtung bewegen. Nehmen Sie Bilder auf, um bis zu 100-150 Kapseln pro Probe zu berücksichtigen.
- Verwenden Sie die ImageJ-Software, um die Permeabilität der Kapselmembran in jeder MW-Fluorophorlösung zu analysieren, indem Siedie Fluoreszenzintensitäten innerhalb und außerhalb der Kapseln vergleichen. Zeichnen Sie dazu eine Region of Interest (ROI) in Form eines Kreises, um den Umfang der Kapsel zu umreißen, und klicken Sie auf Analysieren/Messen , um die Fluoreszenzintensität im Inneren zu messen. Tabellieren Sie die Daten in einer Tabelle. Führen Sie diesen Vorgang für jede Mikrokapsel für insgesamt 200-300 Kapseln durch.
- Beurteilen Sie die äußere Fluoreszenzintensität auf die gleiche Weise, indem Sie den ROI skizzieren und die Intensität außerhalb der Kapseln messen. Führen Sie 3-5 Messungen für die statistische Analyse durch.
- Um eine statistische Analyse durchzuführen, vergleichen Sie die Fluoreszenzintensitäten innerhalb und außerhalb der Kapseln mit einem gepaarten t-Test (p < 0,05).
- Verwenden Sie die Umrechnungstabelle 2 , um die Permeabilität von Mikrokapseln basierend auf den hydrodynamischen Radien für FITC-Dextran mit der Variablen Mw abzuschätzen.
8. In-vitro-Aktivierung von synthetischem Theophyllin-Riboswitch in Seidenmikrokapseln
- Bereiten Sie 1 ml Theophyllin-Stammlösung (100 mM, DMSO) vor. Bereiten Sie das E. coli S30-Extraktsystem für zirkuläre DNA vor, indem Sie die Komponenten 40 Minuten lang auf Eis auftauen.
- Besorgen Sie sich ein 0,5-ml-DNase/RNase-freies Mikrozentrifugenröhrchen. Führen Sie eine In-vitro-Transkriptions- /Translationsreaktion durch, kombinieren Sie die zellfreien Komponenten mit einer Probe von Mikrokapseln in der folgenden Reihenfolge (50 μl Gesamtvolumen): S30-Vormischung ohne Aminosäuren (20 μl); S30-Extrakt, kreisförmig (15 μL); vollständige Aminosäuremischung (5 μL); hohle Mikrokapseln mit ThyRS-GFPa1-Plasmiden aus Schritt 4.10 (9 μL); und Theophyllin, 100 mM DMSO (1 μL).
Anmerkungen: Nachdem Sie alle Komponenten hinzugefügt haben, wirbeln Sie das Röhrchen kurz vor und sammeln Sie die Probe während einer kurzen Zentrifugation bei 0,2 × g für einige Sekunden. - Inkubieren Sie das Röhrchen bei 30 °C für 4 h und überprüfen Sie die Fluoreszenz an einem Plattenleser mit Anregung bei λ = 488 nm und Emission für GFP/FITC-Filter (510 nm ± 20 nm).
- Bebilden Sie die Kapseln auf einem beliebigen LCSM-System mit 488-nm- und 561-nm-Lasern. Erzielen Sie Bilder in bester Qualität mit einem 100-fachen Ölimmersionsobjektiv und 8-Well-Objektträgern.
9. In-vitro-Aktivierung von Brokkoli-Aptamer in Seiden-Mikrokapseln
- Bereiten Sie 1 ml Stammlösung des Farbstoffs DFHBI-1T (30 μM, diH2O) vor. Bereiten Sie das zellfreie Systemreaktionskit PURE (Proteinsynthese mit rekombinanten Elementen) vor, indem Sie die Komponenten 40 Minuten lang auf Eis auftauen.
- Besorgen Sie sich ein 0,5-ml-DNase/RNase-freies Mikrozentrifugenröhrchen. Durchführung einer In-vitro-Transkriptionsreaktion durch Kombination zellfreier Reaktionskomponenten mit einer Probe von Mikrokapseln in der folgenden Reihenfolge (50 μl Gesamtvolumen): Lösung A (20 μl); Lösung B (15 μL); hohle Mikrokapseln mit BrocApt-Plasmiden aus Schritt 4.10 (14 μL); und DFHBI-1T-Farbstoff (1 μL).
Anmerkungen: Nachdem Sie alle Komponenten hinzugefügt haben, wirbeln Sie das Röhrchen kurz vor und sammeln Sie die Probe während einer kurzen Zentrifugation bei 0,2 × g für einige Sekunden. - Inkubieren Sie das Röhrchen bei 37 °C für 6 h und überprüfen Sie die Fluoreszenz an einem Plattenleser mit Anregung bei λex = 470 nm und Emission bei λem = 510 nm ± 20 nm.
- Bebilden Sie die Kapseln auf einem beliebigen LCSM-System mit 488-nm- und 561-nm-Lasern. Erzielen Sie Bilder in bester Qualität mit einem 100-fachen Ölimmersionsobjektiv und 8-Well-Objektträgern mit Deckglas.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Hier befasst sich die Studie mit der Funktionalität von DNA-Templates, die nach der Verkapselung in Seidenproteinkapseln für verschiedene Sensordesigns (zwei Arten von RNA-regulierten Transkriptions-/Translationselementen) kodieren. Mikrokapseln wurden durch Schicht-für-Schicht-Montage (LbL) der Schlüsselkomponenten hergestellt: Eine Hauptschicht, DNA-Plasmide, die für Sensordesigns kodieren, und Seidenfibroin-Biopolymer (Abbildung 2). Die schichtweise Abscheidung von Makromolekülen er...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Selektiv permeable Hydrogel-Mikrokapseln, die mit verschiedenen Arten von DNA-kodierten Sensordesigns beladen sind, können nach diesem Protokoll hergestellt werden. Eine der Besonderheiten des LbL-Ansatzes ist die Fähigkeit, die Komplexität von Mikrokapseln während der Bottom-up-Assemblierung maßzuschneidern, die in der Regel mit der Adsorption molekularer Spezies an Opfervorlagen beginnt. Durch sorgfältige Anpassung der Konzentrationen der Ausgangskomponenten, der pH-Bedingungen und der Anzahl der Schichten könne...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die hier dargestellten Ansichten und Meinungen sind die der Autoren und repräsentieren nicht unbedingt die Ansichten des DoD oder seiner Komponenten
Danksagungen
Diese Arbeit wurde durch das LRIR 16RH3003J-Stipendium des Air Force Office of Scientific Research sowie durch das Programm Synthetic Biology for Military Environments Applied Research for the Advancement of S&T Priorities (ARAP) des U.S. Office of the Under Secretary of Defense for Research and Engineering unterstützt.
Die Plasmidvektorsequenz für ThyRS (pSALv-RS-GFPa1, 3,4 kb) wurde großzügigerweise von Dr. J. Gallivan zur Verfügung gestellt. Seidenraupenkokons von Bombyx mori wurden großzügig von Dr. D.L. Kaplan von der Tufts University, MA, gespendet.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| (Z)-4-(3,5-difluoro-4-hydroxybenzylidene)-2-methyl-1-(2,2,2-trifluoroethyl)-1H-imidazol-5(4 H)-one (DFHBI-1T) | Lucerna | DFHBI-1T | |
| 5x T4 DNA Ligase Buffer | ThermoFisher Scientific | 46300-018 | |
| 6x Blue Gel Loading Dye | New England BioLabs | B7021S | |
| 96-well plates, black circular | Corning | 3601 | |
| Agarose | Sigma-Aldrich | A9539 | BioReagent, for molecular biology, low EEO |
| Ampicillin sodium salt | Sigma-Aldrich | A0166 | powder or crystals, BioReagent, suitable for cell culture |
| BlpI restriction enzymes | New England BioLabs | R0585S | |
| Corning Disposable Vacuum Filter/Storage Systems | FisherScientific | 09-761-1 | |
| Dimethyl sulfoxide, DMSO | Sigma-Aldrich | 472301 | ACS reagent, ≥99.9% |
| DNA Plasmid, pET28c-F30-2x Broccoli (5.4 kb), BrocApt. | Addgene | Plasmid #66788 | |
| DyLightTM550 Antibody Labeling kit (Invitrogen) | ThermoFisher Scientific | 84530 | |
| E. coli S30 extract system for circular DNA | Promega | L1020 | |
| Falcon Conical centrifuge tubes, 15 mL | FisherScientific | 14-959-53A | |
| Falcon Conical centrifuge tubes, 50 mL | 14-432-22 | ||
| Fisherbrand Microcentrifuge tubes, 1.5 mL | FisherScientific | 05-408-129 | |
| Hydrofluoric acid, HF | Sigma-Aldrich | 695068 | ACS reagent, 48% |
| Kanamycin sulfate | Sigma-Aldrich | 60615 | mixture of Kanamycin A (main component) and Kanamycin B and C |
| KpnI restriction enzymes | New England BioLabs | R0142S | |
| LB agar plate supplemented with 100 µg/mL ampicillin | Sigma-Aldrich | L5667 | pre-poured agar plates with 100 µg/mL ampicillin |
| LB agar plate supplemented with 50 µg/mL kanamycin | Sigma-Aldrich | L0543 | pre-poured agar plates with 50 µg/mL kanamycin |
| LB broth (Lennox grade) | Sigma-Aldrich | L3022 | |
| Lithium bromide, LiBr | Sigma-Aldrich | 213225 | ReagentPlus, ≥99% |
| Max Efficiency DH5-α competent E. coli strain | ThermoFisher Scientific | 18258012 | |
| Methanol | MilliporeSigma | 322415 | anhydrous, 99.8% |
| MilliQ-water | EMD MilliPore | Milli-Q Reference Water Purification System | |
| MinElute PCR Purification Kit | Qiagen | 28004 | |
| N-(3-Dimethylaminopropyl)-N′-ethylcarbodiimide hydrochloride, EDC | Sigma-Aldrich | E1769 | |
| PBS (phosphate buffered saline) | ThermoFisher Scientific | 10010023 | 1x PBS, pH 7.4 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530S | |
| Polyethylenimine, branched | Sigma-Aldrich | 408727 | average Mw ~25,000 |
| PURExpress In Vitro Protein Synthesis Kit | New England BioLabs | E6800S | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| Quick-Load 2-Log DNA Ladder (0.1-10.0 kb) | New England BioLabs | N0469S | |
| SiO? silica microspheres, 4.0 µm | Polysciences, Inc. | 24331-15 | 10% aqueous solution |
| Slide-A-Lyzer G2 Dialysis Cassettes, 3.5K MWCO, 15 mL | ThermoFisher Scientific | 87724 | |
| Sodium carbonate, Na?CO? | Sigma-Aldrich | 222321 | ACS reagent, anhydrous, ≥99.5%, powder |
| Spectrum Spectra/Por Float-A-Lyzer G2 Dialysis Devices | FisherScientific | 08-607-008 | Spectrum G235058 |
| SYBR Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase (5 U/µL) | ThermoFisher Scientific | EL0011 | |
| Theophylline | Sigma-Aldrich | T1633 | anhydrous, ≥99%, powder |
| Tris Acetate-EDTA buffer (TAE buffer) | Sigma-Aldrich | T6025 | Contains 40 mM Tris-acetate and 1 mM EDTA, pH 8.3. |
| UltraPure DNase/RNase-Free Distilled Water | FisherScientific | 10-977-023 | |
| ZymoPURE II Plasmid MaxiPrep kit | ZymoResearch | D4202 |
Referenzen
- Slomovic, S., Pardee, K., Collins, J. J. Synthetic biology devices for in vitro and in vivo diagnostics. Proceedings of the National Academy of Sciences of the United States of America. 112 (47), 14429-14435 (2015).
- Harbaugh, S. V., Goodson, M. S., Dillon, K., Zabarnick, S., Kelley-Loughnane, N. Riboswitch-based reversible dual-color sensor. ACS Synthetic Biology. 6 (5), 766-781 (2017).
- König, H., Frank, D., Heil, R., Coenen, C. Synthetic genomics and synthetic biology applications between hopes and concerns. Current Genomics. 14 (1), 11-24 (2013).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: An expanded repertoire of applications. Nature Reviews Genetics. 21, 151-170 (2020).
- Chushak, Y., et al. Characterization of synthetic riboswitch in cell-free protein expression systems. RNA Biology. , 1-12 (2021).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Grӓwe, A., et al. A paper-based, cell-free biosensor system for the detection of heavy metals and date rape drugs. PLoS One. 14 (3), 0210940(2019).
- Lin, X., et al. Portable environment-signal detection biosensors with cell-free synthetic biosystems. RSC Advances. 10 (64), 39261-39265 (2020).
- Caschera, F., Lee, J. W., Ho, K. K. Y., Liu, A. P., Jewett, M. C. Cell-free compartmentalized protein synthesis inside double emulsion templated liposomes with in vitro synthesized and assembled ribosomes. Chemical Communications. 52 (31), 5467-5469 (2016).
- Niederholtmeyer, H., Chaggan, C., Devaraj, N. K. Communication and quorum sensing in non-living mimics of eukaryotic cells. Nature Communications. 9, 5027(2018).
- Timin, A. S., Gould, D. J., Sukkhorukov, G. B. Multi-layer microcapsules: Fresh insights and new applications. Expert Opinion on Drug Delivery. 14 (5), 583-587 (2017).
- Bomati, E. K., Haley, J. E., Noel, J. P., Deheyn, D. D. Spectral and structural comparison between bright and dim green fluorescent proteins in Amphioxus. Scientific Reports. 4, 5469(2014).
- Frey, B. Amplification of Genomic DNA by PCR. Molecular Diagnosis of Infectious Diseases. Methods in Molecular Medicine. Reischl, U. 13, Humana Press. Totowa, NJ. 143-156 (1998).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. Journal of Visualized Experiments. (62), e3923(2012).
- Zhou, Y., et al. Rapid regeneration and reuse of silica columns from PCR purification and gel extraction kits. Scientific Reports. 8, 12870(2018).
- Kostylev, M., Otwell, A. E., Richardson, R. E., Suzuki, Y. Cloning should be simple: Escherichia coli DH5α-mediated assembly of multiple DNA fragments with short end homologies. PLoS One. 10 (9), 0137466(2015).
- Rockwood, D. N., et al. Materials fabrication from Bombyx mori silk fibroin. Nature Protocols. 6 (10), 1612-1631 (2011).
- Drachuk, I., et al. Silk macromolecules with amino acid-Poly(Ethylene Glycol) grafts for controlling layer-by-layer encapsulation and aggregation of recombinant bacterial cells. ACS Nano. 9 (2), 1219-1235 (2015).
- Antipov, A. A., Sukhorukov, G. B. Polyelectrolyte multilayer capsules as vehicles with tunable permeability. Advances in Colloid and Interface Science. 111 (1-2), 49-61 (2004).
- Drachuk, I., Harbaugh, S., Chávez, J. L., Kelley-Loughnane, N. Improving the activity of DNA-encoded sensing elements through confinement in silk microcapsules. ACS Applied Materials & Interfaces. 12 (43), 48329-48339 (2020).
- Melnikov, S., Ben-Shem, A., Garreau de Loubresse, N., Jenner, L., Yusupova, G., Yusupov, M. Structural basis for the inhibition of the eukaryotic ribosome. Nature Structural & Molecular Biology. 19 (6), 560-567 (2012).
- Zhao, S., et al. The future of layer-by-layer assembly: A tribute to ACS Nano associate editor Helmuth Möhwald. ACS Nano. 13 (6), 6151-6169 (2019).
- Main, K. H. S., Provan, J. I., Haynes, P. J., Wells, G., Hartley, J. A., Pyne, A. L. B. Atomic force microscopy-A tool for structural and translational DNA research. APL Bioengineering. 5, 031504(2021).
- Riera, R., Feiner-Gracia, N., Fornaguera, C., Cascante, A., Borrós, S., Albertazzi, L. Tracking the DNA complexation state of pBAE polyplexes in cells with super resolution microscopy. Nanoscale. 11 (38), 17869-17877 (2019).
- Bilokapic, S., Strauss, M., Halic, M. Cryo-EM of nucleosome core particle interactions in trans. Scientific Reports. 8, 7046(2018).
- Pritchard, E. M., Dennis, P. B., Omenetto, F., Naik, R. R., Kaplan, D. L. Physical and chemical aspects of stabilization of compounds in silk. Biopolymers. 97 (6), 479-498 (2012).
- Fritz, B. R., Jamil, O. K., Jewett, M. C. Implications of macromolecular crowding and reducing conditions for in vitro ribosome construction. Nucleic Acids Research. 43 (9), 4774-4784 (2015).
- Ge, X., Luo, D., Xu, J. Cell-free protein expression under macromolecular crowding conditions. PLoS One. 6 (12), 28707(2011).
- Cawte, A. D., Unrau, P. J., Rueda, D. S. Live cell imaging of single RNA molecules with fluorogenic mango II arrays. Nature Communications. 11, 1283(2020).
- Chen, X., et al. Visualizing RNA dynamics in live cells with bright and stable fluorescent RNAs. Nature Biotechnology. 37 (11), 1287-1293 (2019).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten