Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Skalierbare Isolierung und Reinigung extrazellulärer Vesikel aus Escherichia coli und anderen Bakterien
In diesem Artikel
Zusammenfassung
Bakterien sezernieren nanometergroße extrazelluläre Vesikel (EVs), die bioaktive biologische Moleküle tragen. Die EV-Forschung konzentriert sich auf das Verständnis ihrer Biogenese, ihrer Rolle bei Mikroben-Mikroben- und Wirt-Mikroben-Interaktionen und Krankheiten sowie ihrer potenziellen therapeutischen Anwendungen. Ein Workflow für die skalierbare Isolierung von EVs aus verschiedenen Bakterien wird vorgestellt, um die Standardisierung der EV-Forschung zu erleichtern.
Zusammenfassung
Verschiedene Bakterienarten sezernieren ~20-300 nm extrazelluläre Vesikel (EVs), die aus Lipiden, Proteinen, Nukleinsäuren, Glykanen und anderen Molekülen bestehen, die von den Elternzellen stammen. EVs fungieren als intra- und interspezielle Kommunikationsvektoren und tragen gleichzeitig zur Interaktion zwischen Bakterien und Wirtsorganismen im Kontext von Infektion und Besiedlung bei. Angesichts der Vielzahl von Funktionen, die EVs in Gesundheit und Krankheit zugeschrieben werden, besteht ein wachsendes Interesse daran, EVs für In-vitro- und In-vivo-Studien zu isolieren. Es wurde die Hypothese aufgestellt, dass die Trennung von EVs basierend auf physikalischen Eigenschaften, nämlich der Größe, die Isolierung von Vesikeln aus verschiedenen Bakterienkulturen erleichtern würde.
Der Isolationsworkflow besteht aus Zentrifugation, Filtration, Ultrafiltration und Größenausschlusschromatographie (SEC) zur Isolierung von EVs aus Bakterienkulturen. Ein pumpengetriebener Tangential-Flow-Filtrationsschritt (TFF) wurde integriert, um die Skalierbarkeit zu verbessern und die Isolierung von Material aus Litern Ausgangszellkultur zu ermöglichen. Escherichia coli wurde als Modellsystem verwendet, das EV-assoziierte Nanoluciferase und nicht-EV-assoziierte mCherry als Reporterproteine exprimiert. Die Nanoluciferase wurde gezielt auf die EVs gerichtet, indem ihr N-Terminus mit Cytolysin A fusioniert wurde. Frühe Chromatographiefraktionen, die 20-100 nm EVs mit assoziiertem Cytolysin A - nanoLuc enthielten, unterschieden sich von den späteren Fraktionen, die die freien Proteine enthielten. Das Vorhandensein von EV-assoziierter Nanoluciferase wurde durch Immunogold-Markierung und Transmissionselektronenmikroskopie bestätigt. Dieser EV-Isolations-Workflow ist auf andere gramnegative und grampositive Bakterienarten anwendbar, die mit dem menschlichen Darm assoziiert sind. Zusammenfassend lässt sich sagen, dass die Kombination von Zentrifugation, Filtration, Ultrafiltration/TFF und SEC eine skalierbare Isolierung von EVs aus verschiedenen Bakterienarten ermöglicht. Die Verwendung eines standardisierten Isolationsworkflows wird vergleichende Studien mikrobieller Elektrofahrzeuge über Spezies hinweg erleichtern.
Einleitung
Extrazelluläre Vesikel (EVs) sind nanometergroße, liposomenähnliche Strukturen, die aus Lipiden, Proteinen, Glykanen und Nukleinsäuren bestehen und sowohl von prokaryotischen als auch von eukaryotischen Zellen abgesondert werden1. Seit den frühen Studien, die die Freisetzung von EVs aus gramnegativen Bakterien visualisierten2, hat die Anzahl der biologischen Funktionen, die bakteriellen EVs (20-300 nm Durchmesser) zugeschrieben werden, in den letzten Jahrzehnten stetig zugenommen. Zu ihren Funktionen gehören die Übertragung von Antibiotikaresistenz3, Biofilmbildung4, Quorum Sensing5 und Toxinabgabe6. Es besteht auch ein wachsendes Interesse an der Verwendung bakterieller EVs als Therapeutika, insbesondere in der Vakzinologie7 und Krebstherapie8.
Trotz des wachsenden Interesses an der EV-Forschung gibt es immer noch technische Herausforderungen in Bezug auf Methoden der Isolierung. Insbesondere besteht ein Bedarf an Isolationsmethoden, die reproduzierbar, skalierbar und kompatibel mit verschiedenen EV-produzierenden Organismen sind. Um einen einheitlichen Satz von Prinzipien für die Planung und Berichterstattung von EV-Isolierung und Forschungsmethoden zu schaffen, veröffentlicht und aktualisiert die International Society for Extracellular Vesicles das MISEV-Positionspapier9. Darüber hinaus bietet das EV-TRACK-Konsortium eine offene Plattform für die Berichterstattung über detaillierte Methoden zur Isolierung von Elektrofahrzeugen, die in veröffentlichten Manuskripten verwendet werden, um die Transparenz zu erhöhen10.
In diesem Protokoll wurden frühere Methoden zur Isolierung von EVs aus Säugetierzellkulturen angepasst11,12, um die Isolierung von EVs aus bakteriellen Zellkulturen zu ermöglichen. Wir haben versucht, Methoden einzusetzen, die eine EV-Isolierung von einer Vielzahl von Mikroben ermöglichen, die skalierbar sein können, und EV-Reinheit und -Ausbeute ausgleichen (wie im MISEV-Positionspapier9 diskutiert). Nach dem Entfernen von Bakterienzellen und Trümmern durch Zentrifugation und Filtration wird das Kulturmedium entweder durch Zentrifugalgeräte-Ultrafiltration (für ein Volumen von bis zu ~100 ml) oder pumpengetriebene TFF (für größere Volumina) konzentriert. Elektrofahrzeuge werden dann durch SEC mit Säulen isoliert, die für die Reinigung kleiner Elektrofahrzeuge optimiert sind.
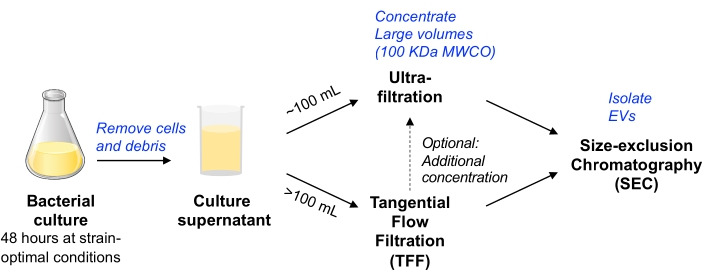
Abbildung 1: Übersicht über den Workflow für die bakterielle EV-Isolierung. Abkürzungen: EV = extrazelluläres Vesikel; TFF = tangentiale Strömungsfiltration; SEC = Größenausschlusschromatographie; MWCO = Molekulargewichtsgrenze. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ein Mauskommensalstamm von Escherichia coli (d.h. E. coli MP113) wurde als Modellorganismus verwendet und modifiziert, um EV-assoziierte Nanoluciferase durch Fusion zu Cytolysin A zu exprimieren, wie bereits berichtet14. Die hier verwendeten Methoden können mindestens bis zu mehreren Litern Bakterienkulturen verarbeiten und EV-assoziierte von nicht-EV-assoziierten Proteinen effektiv trennen. Schließlich kann diese Methode auch für andere grampositive und gramnegative Bakterienarten angewendet werden. Alle relevanten Daten der gemeldeten Experimente wurden an die EV-TRACK Knowledgebase (EV-TRACK ID: EV210211)10 übermittelt.
Access restricted. Please log in or start a trial to view this content.
Protokoll
HINWEIS: Stellen Sie sicher, dass alle Arbeiten mit Bakterien und rekombinanter DNA den besten Praktiken für die biologische Sicherheitseindämmung entsprechen, die der biologischen Sicherheitsrisikostufe jedes Stammes entsprechen. Die Arbeit sollte in Übereinstimmung mit lokalen, nationalen und internationalen Biosicherheitsvorschriften erfolgen.
1. Bakterienstämme und Kulturbedingungen
HINWEIS: In dieser Studie verwendete Bakterienstämme waren Escherichia coli MP113, Akkermansia mucinophila, Bacteroides thetaiotaomicron, Bifidobacterium breve und Bifidobacterium dentium.
- Verwenden Sie für E. coli eine sterile Schlaufe, um einzelne Kolonien in 250 bis 1.000 ml Luria-Bertani (LB) -Brühe zu impfen und aerob in einem Schüttelinkubator bei 300 U/min und 37 ° C für 48 Stunden zu inkubieren, bevor die Kultur verarbeitet wird. Für rekombinante E. coli MP1-Stämme mit p114-mCherry-Clyluc (Supplemental Method und Supplemental Figure S1) fügen Sie dem LB-Agar und der Brühe Chloramphenicol in einer Endkonzentration von 17 μg/ml hinzu.
- Für A. mucinophila, B. thetaiotaomicron, B. breve und B . dentium streifen Sie auf Brain heart infusion (BHI) Agarplatten und inkubieren anaerob in einer anaeroben Vinylkammer. Impfen Sie einzelne Kolonien in 100 ml vorreduzierte BHI-Brühe und inkubieren Sie für 48 h anaeroben.
2. Isolierung von Elektrofahrzeugen
- Klärung von Bakterienkulturmedium durch Zentrifugation und Filtration
- Die in Schritt 1 inokulierten Bakterienzellkulturen werden durch Gießen in 250 mL oder 500 mL Polypropylen-Zentrifugenflaschen gereinigt. Zentrifugieren Sie die Flaschen in einem großflächigen Rotor mit festem Winkel bei 4 °C und 5.000 × g für 15 min. Den Überstand durch vorsichtiges Eingießen in saubere Zentrifugenflaschen überführen und bei 10.000 × g erneut für 15 min zentrifugieren.
HINWEIS: Verwenden Sie die Flaschen nach biosicherheitsgerechter Reinigung und Dekontamination wieder.- Wenn nach der zweiten Zentrifugation große Pellets von Bakterienzellen vorhanden sind, wiederholen Sie die Zentrifugation in einer sauberen Flasche, um die Zellen weiter zu entfernen.
- Der Überstand wird durch Gießen in eine 0,22 μm Polyethersulfon-Vakuumfiltervorrichtung geeigneter Größe überführt. Filtern Sie, indem Sie das Filtergerät an eine Vakuumwandversorgung anschließen. Wenn die Filtrationsrate deutlich sinkt, bewegen Sie einfach das ungefilterte Material in ein neues Gerät. Lagern Sie das gefilterte Medium über Nacht bei 4 °C und setzen Sie das Protokoll auf Wunsch am nächsten Tag fort.
HINWEIS: Die oben genannten Zentrifugationen ermöglichen typischerweise die Verarbeitung von ~ 2x des angegebenen Volumens der Zellkultur durch jedes Gerät. Zum Beispiel könnte eine einzelne 500-ml-Filtervorrichtung ~ 1.000 ml vorzentrifugierte Kultur filtern. Diese Geräte werden normalerweise nicht wiederverwendet. Die Verwendung von Spritzenfiltern in diesem Schritt wird nicht ohne Optimierung empfohlen, da bei den getesteten Modellen erhebliche Verluste festgestellt wurden. Dies ist ein potenzieller Haltepunkt. - Überprüfen Sie die vollständige Entfernung der lebensfähigen Zellen an dieser Stelle, indem Sie ein Aliquot des gefilterten Überstands auf geeignete Agarplatten verteilen und sicherstellen, dass nach der Inkubation unter optimalen Bedingungen für den Bakterienstamm keine Kolonien vorhanden sind. Wenn Bakterien nachgewiesen werden, optimieren Sie das oben beschriebene Verfahren weiter, indem Sie zusätzliche Zentrifugationen und/oder Filtrationen durchführen.
- Die in Schritt 1 inokulierten Bakterienzellkulturen werden durch Gießen in 250 mL oder 500 mL Polypropylen-Zentrifugenflaschen gereinigt. Zentrifugieren Sie die Flaschen in einem großflächigen Rotor mit festem Winkel bei 4 °C und 5.000 × g für 15 min. Den Überstand durch vorsichtiges Eingießen in saubere Zentrifugenflaschen überführen und bei 10.000 × g erneut für 15 min zentrifugieren.
- Konzentration des gefilterten Mediums
- Wenn Sie mit Volumina von deutlich >100 ml arbeiten, fahren Sie mit Schritt 2.2.2 fort. Wenn Sie mit Volumina von ~ 100 ml arbeiten, laden Sie 90 mL gefiltertes Kulturmedium auf das Reservoir mit einer entsprechenden Kapazität von 100 kDa Molekulargewichts-Cutoff (MWCO) zentrifugaler Ultrafiltrationsvorrichtung unter Verwendung serologischer Pipetten. Immer mit einem passenden Ultrafiltrationsgerät balancieren und im schwingenden Schaufelrotor bei 4 °C und 2.000 × g für 15-30 min Intervalle zentrifugieren, bis das Volumen des Mediums im oberen Reservoir auf <0,5 ml konzentriert ist.
- Füllen Sie das Reservoir mit dem verbleibenden gefilterten Kulturmedium auf. Wenn Sie "aufladen", entfernen Sie den Durchfluss in der Unterseite des Geräts und balancieren Sie alle Geräte neu aus.
HINWEIS: Es wurde beobachtet, dass das maximale Volumen des gefilterten Kulturmediums, das mit diesen Geräten konzentriert werden kann, <2-fache des empfohlenen Volumens beträgt. - Wenn die Viskosität des konzentrierten Mediums im Reservoir sichtbar erhöht ist (dunkles, viskoses Material), mit phosphatgepufferter Kochsalzlösung (PBS) verdünnen und durch Zentrifugieren erneut konzentrieren, um alle Nicht-EV-Proteine zu verdünnen, die kleiner als der MWCO von 100 kDa sind.
HINWEIS: Dies ist ein potenzieller Haltepunkt. - Das konzentrierte Medium wird in ein proteinarmes Röhrchen überführt, über Nacht bei 4 °C gelagert und das Protokoll auf Wunsch am nächsten Tag fortgesetzt.
- Füllen Sie das Reservoir mit dem verbleibenden gefilterten Kulturmedium auf. Wenn Sie "aufladen", entfernen Sie den Durchfluss in der Unterseite des Geräts und balancieren Sie alle Geräte neu aus.
- Wenn Sie mit Volumina von deutlich >100 ml arbeiten, wählen Sie ein TFF-Gerät mit geeigneter Größe (100 kDa MWCO), um das zu verarbeitende Volumen aufzunehmen.
HINWEIS: Filtrationsgeräte für die Verarbeitung von 100 mL bis >1.000 mL sind kommerziell erhältlich. Die lokale Verfügbarkeit, die Kosten und die Kompatibilität mit der Pumpe und den Schläuchen/Anschlüssen bestimmen, welche Modelle am nützlichsten sind. Bis zu 2 l Kulturmedium wurden mit dem in der Materialtabelle angegebenen Gerät verarbeitet, bevor der Filter gereinigt werden musste (siehe Schritt 2.3 unten für das Reinigungsprotokoll).- Montieren Sie einen Filterkreislauf mit #16 Low-Binding/Low-Lauging-Schläuchen, 1/8-Zoll-Schlauch-Barb-zu-Luer-Adaptern, dem TFF-Gerät und einer Schlauchpumpe, wie in der ergänzenden Abbildung S2 angegeben.
HINWEIS: Führen Sie TFF in einer Biosicherheitswerkbank durch, um das Risiko einer Kontamination des EV-Präparats mit Umweltbakterien zu minimieren. - Beginnen Sie bei Raumtemperatur mit der Umwälzung des gefilterten, konditionierten Mediums bei etwa 200 ml/min (mindestens 100 ml/min). Bestimmen Sie die geeignete Drehzahl, die der gewünschten Durchflussrate entspricht, indem Sie 200 ml PBS in einen Messbehälter pumpen. Beim Umlauf von gefiltertem, konditioniertem Medium werden die Moleküle <100 kDa, die die Ultrafiltrationsmembran passieren, als Abfall in einem separaten Gefäß gesammelt.
HINWEIS: Das folgende Beispiel geht von einem Startvolumen von 2 L Kultur aus. - Fahren Sie fort, das konditionierte Medium zu zirkulieren, bis sein Volumen auf ~ 100-200 ml reduziert wurde. Wechseln Sie bei Bedarf zu kleineren Schiffen. Verdünnen Sie das 2-fache mit PBS und zirkulieren Sie weiter mit der Pumpe, wobei Sie sich auf 75-100 ml konzentrieren. 2-fach mit PBS verdünnen und weiter auf ein Endvolumen von 25 ml zirkulieren. 2-fach mit PBS verdünnen und weiter zirkulieren bis <10 ml.
- Heben Sie den Zufuhrschlauch aus dem Probenbehälter und pumpen Sie weiter, um den Filter zu reinigen und die maximale Probenmenge zu gewinnen.
HINWEIS: Dies ist ein potenzieller Haltepunkt. - Die konzentrierte Probe wird in ein konisches Röhrchen überführt und auf Wunsch über Nacht bei 4 °C gelagert. Alternativ können Sie mit dem Protokoll fortfahren.
- Bewegen Sie die konzentrierte Probe in eine 15 ml Kapazität 100 kDa MWCO zentrifugale Ultrafiltrationsvorrichtung. Zentrifugieren Sie in einem schwingenden Schaufelrotor bei 4 °C und 2.000 × g für 15-30 min Intervalle, bis das Volumen des Mediums im oberen Reservoir auf <2 ml konzentriert ist.
HINWEIS: Dies ist ein potenzieller Haltepunkt. - Das konzentrierte Medium wird in ein Röhrchen mit niedriger Proteinbindung überführt und über Nacht bei 4 °C gelagert, wobei das Protokoll auf Wunsch am nächsten Tag fortgesetzt wird.
- Montieren Sie einen Filterkreislauf mit #16 Low-Binding/Low-Lauging-Schläuchen, 1/8-Zoll-Schlauch-Barb-zu-Luer-Adaptern, dem TFF-Gerät und einer Schlauchpumpe, wie in der ergänzenden Abbildung S2 angegeben.
- Wenn Sie mit Volumina von deutlich >100 ml arbeiten, fahren Sie mit Schritt 2.2.2 fort. Wenn Sie mit Volumina von ~ 100 ml arbeiten, laden Sie 90 mL gefiltertes Kulturmedium auf das Reservoir mit einer entsprechenden Kapazität von 100 kDa Molekulargewichts-Cutoff (MWCO) zentrifugaler Ultrafiltrationsvorrichtung unter Verwendung serologischer Pipetten. Immer mit einem passenden Ultrafiltrationsgerät balancieren und im schwingenden Schaufelrotor bei 4 °C und 2.000 × g für 15-30 min Intervalle zentrifugieren, bis das Volumen des Mediums im oberen Reservoir auf <0,5 ml konzentriert ist.
- Reinigen des TFF-Geräts (optional)
HINWEIS: Die Filtrationsrate nimmt ab, wenn das TFF-Gerät während des Prozesses zu "verstopfen" beginnt (Fouling). Bei Bedarf kann die Filtervorrichtung gereinigt werden, um die Filtration weiterer Proben im selben Reinigungslauf zu erleichtern. Obwohl theoretisch möglich, wurde ein gereinigter TFF-Filter nicht für einen anderen Reinigungslauf verwendet, um Kreuzkontaminationen zu vermeiden.- Entfernen Sie zum Reinigen alle Schläuche und Kappen vom TFF-Gerät und lassen Sie die restliche Flüssigkeit ab.
- Verwenden Sie die Schlauchpumpe und den Schlauch, um sowohl die inneren als auch die äußeren Abteilungen des TFF-Geräts (d. h. über die parallelen und senkrechten Anschlüsse im in der Materialtabelle aufgeführten Modell) mit ~ 100 ml destilliertem Wasser zu fluten. Entfernen Sie alle Schläuche/Kappen und entleeren Sie das TFF-Gerät.
- Verschließen Sie die äußeren (senkrechten, filtratartigen) Anschlüsse und zirkulieren Sie 250 ml 20% Ethanol in destilliertem Wasser bei >200 ml/min für 10 min durch das innere Fach. Abtropfen lassen, mit destilliertem Wasser überfluten und wieder wie oben abtropfen lassen.
- 250 mL 0,5 N frische NaOH-Lösung für 30 min durch das Innenfach zirkulieren lassen und erneut abtropfen lassen.
- Schließen Sie alle Schläuche und Kappen wieder an die Einlass-, Auslass- und Filtratanschlüsse an, wie in der ergänzenden Abbildung S2 gezeigt, und zirkulieren Sie 0,5 N NaOH-Lösung erneut, bis ein Volumen NaOH > 1 ml/cm2 Filteroberfläche durch die Filtermembran dringt und als Filtrat/Abfall gesammelt wird.
- Spülen Sie das TFF-Gerät wie oben beschrieben mit destilliertem Wasser ab. Verwenden Sie das TFF-Gerät sofort oder fluten Sie das Gerät mit ~100 mL 20% Ethanol und lagern Sie es über Nacht bei 4 °C.
HINWEIS: Wenn Sie in Ethanol gelagert werden, achten Sie darauf, 250 ml PBS durch das Gerät zu entleeren, mit Wasser abzuspülen, abzulassen und durch das Gerät zu zirkulieren, bis ein Volumen von >1 ml / cm2 Filteroberfläche durch die Filtermembran dringt und als Filtrat / Abfall gesammelt wird, um Restethanol vor der Probenverarbeitung zu entfernen.
- Größenausschlusschromatographie (SEC)
HINWEIS: SEC wird verwendet, um die Reinheit von EVs zu erhöhen und nicht-vesikuläres Protein zu entfernen.- Verwenden Sie eine kleine SEC-Säule (10 ml Bettvolumen) für die Isolierung von Elektrofahrzeugen aus <100 ml Ausgangsmaterial und eine größere Säule (47 ml Bettvolumen) für die Isolierung von Elektrofahrzeugen aus >100 ml Ausgangsmaterial.
HINWEIS: Im folgenden Beispiel werden Bände für die größere Spalte aufgelistet, mit Volumen für die kleinere Spalte in Klammern. - Bringen Sie die SEC-Säule und PBS über mehrere Stunden auf Raumtemperatur. Stabilisieren Sie die SEC-Säule in vertikaler Position mit einem Standard-Laborständer und -halter. Alternativ können Sie handelsübliche Chromatographie-Säulenständer verwenden.
- Vor dem Anschluss an die SEC-Säule hydratisieren Sie das Probenreservoir, indem Sie 5 ml PBS durch die Fritte und in einen Abfallbehälter fließen lassen. Schrauben Sie die Einlasskappe der SEC-Säule ab, geben Sie 2 ml PBS in das Probenreservoir und verbinden Sie das Reservoir vorsichtig mit der Säule, da das PBS durch die Fritte tropft (gilt nicht für kleine SEC-Säulen).
HINWEIS: Dieser vorherige Schritt verhindert, dass Luftblasen oben in der SEC-Spalte eingeschlossen werden. Wenn Luft eingeschlossen ist, entfernen Sie den Behälter, tippen Sie auf die Säule, um die Luftblase herauszubekommen, und wiederholen Sie den Verbindungsvorgang. Für die kleinere Säule öffnen Sie einfach die Oberseite der SEC-Säule und bringen Sie den Probentrichter an. - Geben Sie 47 ml (10 ml) PBS in das Probenreservoir und öffnen Sie den Boden der SEC-Spalte. Lassen Sie den gesamten geladenen Probenpuffer zur Äquilibrierung durch die Spalte fließen. Verwerfen Sie den Durchfluss.
- Laden Sie maximal 2 ml (0,5 ml) der Probe in das Probenreservoir, verwerfen Sie den Durchfluss und lassen Sie die Probe vollständig in die Säule gelangen.
- Geben Sie sofort PBS in das Probenreservoir oder den Trichter mit einem Volumen von 14,25 ml minus dem Probenvolumen (3 ml minus Probenvolumen für die kleine Säule). Lassen Sie die Lösung durch die Säule fließen und verwerfen Sie diesen Betrag, der dem Spaltenleervolumen entspricht.
HINWEIS: Für eine typische 2-ml-Probe beträgt die Menge an PBS, die dem Probenbehälter oder Trichter hinzugefügt werden soll, 12,25 ml. - Positionieren Sie ein 2 mL Low-Binding Mikroröhrchen direkt unter der SEC-Säule. Geben Sie sofort 2 ml (0,5 ml) PBS in das Probenreservoir und lassen Sie es in die Säule gelangen. Kennzeichnen Sie diese ersten 2 ml (0,5 ml) Durchfluss als Fraktion 1. Geben Sie weiterhin 2 ml (0,5 ml) gleichzeitig in das Probenreservoir, um jede nachfolgende Fraktion zu sammeln.
HINWEIS: Die meisten bakteriellen EVs eluieren in den ersten 5 Fraktionen. Bei der Optimierung wurden die ersten 12 Fraktionen gesammelt. - Lagern Sie die Fraktionen bei 4 °C für die Kurzzeitlagerung (Tage) bzw. -80 °C für die Langzeitlagerung.
- Reinigung und Lagerung der wiederverwendbaren SEC-Säulen
HINWEIS: Die in diesem Protokoll beschriebenen SEC-Spalten können je nach Hersteller bis zu 5 Mal wiederverwendet werden. Wenn die Durchflussrate der SEC-Säulen nach <5 Anwendungen abnimmt, empfiehlt der Hersteller, die konzentrierten Proben bei 10.000 x g für 10 min zu zentrifugieren, um alle Aggregate vor SEC zu entfernen. Laden Sie dann den Überstand dieser Zentrifugation auf die SEC-Säule zur EV-Isolierung.- Um die SEC-Säule nach jedem Gebrauch zu reinigen und zu lagern, fügen Sie 2 ml (0,5 ml) 0,5 M NaOH hinzu und lassen Sie sie vollständig in die Säule gelangen. Lassen Sie 100 ml (20 ml) 20% Ethanol durch die Säule laufen und lagern Sie es bei 4 °C bis zur nächsten Verwendung. Vor der nächsten Verwendung das Ethanol wie oben auf Raumtemperatur ausgleichen und mit PBS-Puffer austauschen, indem Sie weitere 150 ml (30 ml) PBS durch die Säule laufen lassen.
- Um die SEC-Säule nach jedem Gebrauch zu reinigen und sofort wiederzuverwenden, fügen Sie 2 ml (0,5 ml) 0,5 M NaOH hinzu und lassen Sie sie vollständig in die Säule eindringen. Lassen Sie ungefähr 150 ml (30 ml) PBS-Puffer laufen, um NaOH wegzuspülen. Wenn der pH-Wert von Eluat gleich PBS (~ 7) ist, kann eine neue Probe geladen werden.
- Verwenden Sie eine kleine SEC-Säule (10 ml Bettvolumen) für die Isolierung von Elektrofahrzeugen aus <100 ml Ausgangsmaterial und eine größere Säule (47 ml Bettvolumen) für die Isolierung von Elektrofahrzeugen aus >100 ml Ausgangsmaterial.
3. Qualitätskontrolle der EV-Vorbereitung
- Sterilitätsprüfung
HINWEIS: Da diese EVs aus Bakterienkulturen stammen, ist es wichtig, die Sterilität vor der nachgeschalteten Verwendung sicherzustellen.- Erhalten Sie 100 μL (20 μL) der Fraktionen, die in Assays verwendet werden sollen, und inokulieren Sie 3 ml des Mediums, das zum Züchten der Quellbakterien verwendet wird. Kultur unter den jeweils optimalen Bedingungen für mindestens 3 Tage und auf Trübung beobachten. Alternativ können Sie die Fraktionsproben auf Agarplatten auftragen, die das Medium enthalten, mit dem die produzierenden Bakterien gezüchtet werden, und nach Koloniebildung suchen.
HINWEIS: Wenn eine bakterielle Kontamination festgestellt wird, wird nicht empfohlen, das EV-Präparat für Experimente zu verwenden. Wiederholen Sie stattdessen die Isolierung und achten Sie darauf, das Risiko einer bakteriellen Kontamination zu minimieren, indem Sie (a) eine ausreichende Zentrifugation / Filtration des konditionierten bakteriellen Zellkulturmediums durchführen, (b) saubere Flaschen, Schläuche, Filter und Chromatographiesäulen verwenden und (c) geeignete aseptische Techniken anwenden.
- Erhalten Sie 100 μL (20 μL) der Fraktionen, die in Assays verwendet werden sollen, und inokulieren Sie 3 ml des Mediums, das zum Züchten der Quellbakterien verwendet wird. Kultur unter den jeweils optimalen Bedingungen für mindestens 3 Tage und auf Trübung beobachten. Alternativ können Sie die Fraktionsproben auf Agarplatten auftragen, die das Medium enthalten, mit dem die produzierenden Bakterien gezüchtet werden, und nach Koloniebildung suchen.
- Protein-Quantifizierung
HINWEIS: Es wurde ein hochempfindliches, fluoreszenzbasiertes Proteinquantifizierungskit verwendet (siehe Materialtabelle). Das Kit arbeitet mit einem passenden proprietären Fluorimeter bei Anregungs-/Emissionswellenlängen von 485/590 nm.- Bringen Sie alle Reagenzien, Standards und Proben auf Raumtemperatur.
- Es wird eine Mastermischung aus Proteinreagenz und Puffer hergestellt, indem 1 μL des Reagenzes zu 199 μL Puffer für jede zu untersuchende Probe und jeden zu untersuchenden Standard hinzugefügt wird. Fügen Sie mit dünnwandigen 0,5-ml-PCR-Röhrchen 10 μL Standard + 190 μL Master-Mix zu jedem Standardröhrchen hinzu.
ANMERKUNG: Um innerhalb des Bereichs des Assays zu liegen, hängt die Menge jeder Fraktion, die jedem Probenröhrchen zugesetzt werden soll, von der erwarteten Proteinausbeute der Reinigung ab. Typischerweise wurden 5 μL jeder Fraktion + 195 μL Mastermix verwendet. Das Endvolumen der Probe + Mastermischung muss 200 μL betragen. - Wirbeln Sie die Assay-Röhrchen ab und inkubieren Sie sie mindestens 15 Minuten bei Raumtemperatur im Dunkeln.
- Messen Sie die Standards auf dem entsprechenden proprietären Fluorimeter (siehe Materialtabelle), indem Sie die Option Proteinassay mit den Pfeiltasten auswählen und zur Bestätigung die GO-Taste drücken. Folgen Sie den Anweisungen auf dem Bildschirm, führen Sie jedes Standardrohr ein und drücken Sie GO.
- Setzen Sie das experimentelle Probenröhrchen ein; Drücken Sie GO , um zu lesen; und notieren Sie das angezeigte Ergebnis, d. h. die tatsächliche Proteinkonzentration im Assay-Puffer/Proben-Gemisch. Um die Proteinkonzentration in der Probe zu erhalten, verwenden Sie die Pfeiltasten, um die Option Probenkonzentration berechnen auszuwählen, drücken Sie GO und verwenden Sie die Pfeiltasten, um das Probenvolumen auszuwählen, das dem Assay-Puffer für die angegebene Probe hinzugefügt wurde. Drücken Sie GO und notieren Sie die Proteinkonzentration der Probe. Wiederholen Sie diesen Schritt für jede zu analysierende Probe.
- Partikelzählung und Größenverteilung
HINWEIS: Mikrofluidik resistive pulse sensing (MRPS) wurde verwendet, um EV-Konzentration und Größenverteilung zu quantifizieren.- Die Proben in PBS, ergänzt mit 1% Tween-20, das durch einen 0,02-μm-Spritzenfilter filtriert wurde, auf eine Proteinkonzentration von etwa 0,1 μg/ml verdünnen.
HINWEIS: Das Ziel der Verdünnung ist es, eine erwartete Partikelkonzentration im Bereich von 1010 Partikeln / ml in EV-haltigen Fraktionen zu erreichen. Die optimale Verdünnung muss möglicherweise empirisch bestimmt werden. Für spätere Fraktionen (über Fraktion 6 hinaus) werden nur wenige EVs erwartet. Daher wird die Partikelkonzentration trotz Analyse bei niedrigen Verdünnungen wahrscheinlich <1010 Partikel/ml betragen. - Legen Sie 3 μL jeder Probe mit einer Mikropipette in die Einweg-Mikrofluidikkartusche, führen Sie die Patrone in das MRPS-Instrument ein und drücken Sie den Metallknopf mit einem blau beleuchteten Rand.
- Klicken Sie in der Erfassungssoftware auf Go! und warten Sie, bis die Probe vom Gerät analysiert wurde. Erfassen Sie 1.000 bis 10.000 Partikelereignisse, um den technisch-statistischen Analysefehler zu minimieren. Klicken Sie an dieser Stelle auf Beenden und Testlauf beenden , um die Beispielerfassung abzuschließen.
HINWEIS: Zusammen mit den Rohdatendateien gibt das Gerät eine zusammenfassende Tabelle aus, in der die Partikelkonzentration in der Probe aufgeführt ist. Korrigieren Sie diesen Wert entsprechend der durchgeführten Probenverdünnung. - Laden Sie mithilfe von Analysesoftware die Rohdaten und generieren Sie benutzerdefinierte Diagramme der Größenverteilung.
- Die Proben in PBS, ergänzt mit 1% Tween-20, das durch einen 0,02-μm-Spritzenfilter filtriert wurde, auf eine Proteinkonzentration von etwa 0,1 μg/ml verdünnen.
4. EV-Speicher
- Einzelne oder gepoolte Fraktionen auf 25-50% der individuellen Fraktionsgröße (abhängig von der Größe der verwendeten Säule) in proteinarmen Bindungsröhrchen aliquotieren und bei -80 °C lagern, um Gefrier-Tau-Zyklen zu vermeiden.
HINWEIS: Verschiedene Anwendungen können kleinere oder größere Aliquots erfordern, abhängig von der erwarteten Menge, die in jedem Experiment verwendet wird. Dies muss empirisch ermittelt werden. Die nicht EV-haltigen Fraktionen können verworfen werden, wenn sie nicht für die Forschungsziele geeignet sind.
5. Transmissionselektronenmikroskopie
- Negative Färbung
- 5 μL der EV-Probe in das kohlenstoffbeschichtete Kupfergitter mit 400 Maschen geben und 10 min bei Raumtemperatur inkubieren. Die Probenseite wird mit 5 Tropfen 5 mM Tris-Puffer (pH 7,1) und anschließend mit 5 Tropfen destilliertem Wasser gewaschen.
- Probenseite mit 5 Tropfen 2% Uranylacetat färben. Wischen Sie zusätzliche Mengen an Flecken mit Filterpapier ab und lassen Sie das Rost mehrere Stunden oder über Nacht vollständig trocknen. Visualisieren Sie die Proben mit einem Elektronenmikroskop, das mit 80 kV betrieben wird.
- Immunogold-Markierung
- 10 μL der EV-Suspension auf ein Formvar/Carbon 400 Mesh-Gitter auftragen und bei Raumtemperatur 1 h inkubieren. Waschen Sie das Gitter dreimal in PBS und tragen Sie dann 4% Paraformaldehyd für 10 Minuten auf, um die Probe zu fixieren. Waschen Sie die Gitter fünfmal mit PBS.
- Blockieren Sie das Gitter mit drei PBS-Wäschen mit 0,1% Rinderserumalbumin (BSA). Tragen Sie dann 10 μL eines primären Antikörpers für 40 min bei Raumtemperatur auf (hier 1 μg/ml nluc-Antikörper). Dreimal mit PBS waschen, das 0,1% BSA enthält.
- Geben Sie 10 μL sekundären goldmarkierten Antikörper in das Gitter und inkubieren Sie für 40 min bei Raumtemperatur. Waschen Sie die Gitter dreimal mit PBS.
HINWEIS: Hier wurde ein mit 10 nm Goldnanopartikeln konjugierter Ziegen-Anti-Maus-Antikörper verwendet, nachdem er 1:10 im Blockierpuffer verdünnt wurde. Wenn die Goldmarkierung die EV-Visualisierung verdeckt, können stattdessen sekundäre Antikörper mit kleineren Goldnanopartikeln (z.B. 5 nm) verwendet werden. - Das Gitter mit 10 μL 2,5% Glutaraldehyd für 10 min bei Raumtemperatur nachfixieren. Dreimal in PBS waschen. Führen Sie eine negative Färbung mit 2% Uranylacetat (10 μL) für 15 min durch. Die Proben in 10 μL 0,5% Uranylacetat und 0,13% Methylcelluloselösung für 10 min einbetten.
- Lassen Sie die Probengitter über Nacht bei Raumtemperatur trocknen, bevor Sie sie auf dem Elektronenmikroskop abbilden.
- Bestimmen Sie in der Mikroskopaufnahmesoftware die Belichtung empirisch, um die optimale Bildqualität zu erhalten (z. B. 0,80851 s in diesem speziellen Setup), und passen Sie sie an, indem Sie diesen Wert in das Optionsfeld für die Belichtungszeit eingeben. Wählen Sie die Option 80 kV und klicken Sie auf Aufnahme starten , um das Bild aufzunehmen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Um zu beurteilen, welche SEC-Chromatographiefraktionen für EVs angereichert waren, wurde die SEC-Säule mit 2 ml E. coli MP1-konditioniertem Kulturmedium beladen, das 1.000-fach durch TFF konzentriert worden war, und sequentielle Fraktionen wurden gesammelt. Unter Verwendung von MRPS wurde festgestellt, dass die Fraktionen 1-6 die meisten EVs enthielten (Abbildung 2A). Nachfolgende Fraktionen enthielten sehr wenige EVs, die anstelle von EV-freien Proteinen bestanden (
Access restricted. Please log in or start a trial to view this content.
Diskussion
Im obigen Protokoll wird eine Methode beschrieben, die skalierbar ist und EVs zuverlässig von verschiedenen gramnegativen/positiven und aeroben/anaeroben Bakterien isoliert. Es hat mehrere potenzielle Haltepunkte während des gesamten Verfahrens, obwohl es besser ist, nicht länger als 48 Stunden zu benötigen, um EVs aus konditionierten Bakterienkulturmedien zu isolieren.
Erstens besteht es aus der Kultivierung von Bakterien, um ein konditioniertes Bakterienkulturmedium zu erzeugen. Es wurde...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben keine Interessenkonflikte zu erklären.
Danksagungen
Die oben beschriebene Forschung wurde durch NIH TL1 TR002549-03 Training Grant unterstützt. Wir danken Dr. John C. Tilton und Dr. Zachary Troyer (Case Western Reserve University) für die Erleichterung des Zugangs zum Partikelgrößenanalysegerät; Lew Brown (Spectradyne) für technische Unterstützung bei der Analyse der Partikelgrößenverteilungsdaten; Dr. David Putnam von der Cornell University für die Bereitstellung von pClyA-GFP-Plasmid14; und Dr. Mark Goulian von der University of Pennsylvania für die Bereitstellung des E. coli MP113.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.5 mL flat cap, thin-walled PCR tubes | Thermo Scientific | 3430 | it is important to use thin-walled PCR tubes to obtain accurate readings with Qubit |
| 16% Paraformaldehyde (formaldehyde) aqueous solution | Electron microscopy sciences | 15700 | |
| 250 mL Fiberlite polypropylene centrifuge bottles | ThermoFisher | 010-1495 | |
| 500 mL Fiberlite polypropylene centrifuge bottles | ThermoFisher | 010-1493 | |
| 65 mm Polypropylene Round-Bottom/Conical Bottle Adapter | Beckman Coulter | 392077 | Allows Vivacell to fit in rotor |
| Akkermansia mucinophila | ATCC | BAA-835 | |
| Amicon-15 (100 kDa MWCO) | MilliporeSigma | UFC910024 | |
| Avanti J-20 XPI centrifuge | Beckman Coulter | No longer sold by Beckman. Avanti J-26XP is closest contemporary model. | |
| Bacteroides thetaiotaomicron VPI 5482 | ATCC | 29148 | |
| Bifidobacterium breve | NCIMB | B8807 | |
| Bifidobacterium dentium | ATCC | 27678 | |
| Brain Heart infusion (BHI) broth | Himedia | M2101 | After autoclaving, Both BHI broth and agar were introduced into the anaerobic chamber, supplemented with Menadione (1 µg/L), hematin (1.2 µg/L), and L-Cysteine Hydrochloride (0.05%). They were then incubated for at least 24 h under anaerobic conditions before inoculation with the anaerobic bacterial strains. |
| C-300 microfluidics cartridge | Spectradyne | ||
| Chloramphenicol | MP Biomedicals | ICN19032105 | |
| Electron microscope | FEI company | Tecnai G2 SpiritBT | |
| Escherichia coli HST08 (Steller competent cells) | Takara | 636763 | |
| Escherichia coli MP1 | Dr. Mark Goulian (gift) | commensal bacteria derived from mouse gut | |
| Fiberlite 500 mL to 250 mL adapter | ThermoFisher | 010-0151-05 | used with Fiberlite rotor to enable 250 mL bottles to be used for smaller size of starting bacterial culture |
| Fiberlite fixed-angle centrifuge rotor | ThermoFisher | F12-6x500-LEX | fits 6 x 500 mL bottles |
| Formvar Carbon Film 400 Mesh, Copper | Electron microscopy sciences | FCF-400-CU | |
| Glutaraldehyde (EM-grade, 10% aqeous solution) | Electron microscopy sciences | 16100 | |
| Hematin | ChemCruz | 207729B | Stock solution was made in 0.2 M L-histidine solution as 1.2 mg/mL |
| Infinite M Nano+ Microplate reader | Tecan | This equibment was used to measure the mCherry fluorescence | |
| In-Fusion HD Cloning Plus | Takara | 638909 | For cloning of the PCR fragements into the PCR-lineraized vectors |
| JS-5.3 AllSpin Swinging-Bucket Rotor | Beckman Coulter | 368690 | |
| Lauria Bertani (LB) broth, Miller | Difco | 244620 | |
| L-Cysteine Hydrochloride | J.T. Baker | 2071-05 | It should be weighed and added directly to the autoclaved BHI media inside the anaerobic chamber |
| Masterflex Fitting, Polypropylene, Straight, Female Luer to Hose Barb Adapter, 1/8" ID; 25/PK | cole-parmer - special | HV-30800-08 | connection adapters for filtration tubing circuit |
| Masterflex Fitting, Polypropylene, Straight, Male Luer to Hose Barb Adapter, 1/8" ID; 25/PK | cole-parmer - special | HV-30800-24 | connection adapters for filtration tubing circuit |
| Masterflex L/S Analog Variable-Speed Console Drive, 20 to 600 rpm | Masterflex | HV-07555-00 | |
| Masterflex L/S Easy-Load Head for Precision Tubing, 4-Roller, PARA Housing, SS Rotor | Masterflex | EW-07514-10 | |
| Masterflex L/S Precision Pump Tubing, PharmaPure, L/S 16; 25 ft | Cole Palmer | EW-06435-16 | low-binding/low-leaching tubing |
| Menadione (Vitamin K3) | MP | 102259 | Stock solution was made in ethanol as 1 mg/mL |
| MIDIKROS 41.5CM 100K MPES 0.5MM FLL X FLL 1/PK | Repligen | D04-E100-05-N | TFF device we have used to filter up to 2 L of E. coli culture supernatant |
| Nano-Glo Luciferase Assay System | Promega | N1110 | This assay kit was used to measure the luminescence of the nluc reporter protein |
| NanoLuc (Nluc) Luciferase Antibody, clone 965808 | R&D Systems | MAB10026 | |
| nCS1 microfluidics resistive pulse sensing instrument | Spectradyne | ||
| nCS1 Viewer | Spectradyne | Analysis software for particle size distribution | |
| OneTaq 2x Master Mix with Standard Buffer | NEB | M0482 | DNA polymerase master mix used to perform the routine PCR reactions for colony checking |
| Protein LoBind, 2.0 mL, PCR clean tubes | Eppendorf | 30108450 | |
| Q5 High-Fidelity 2x Master Mix | NEB | M0492 | DNA polymerase master mix used to perform the PCR reactions needed for cloning |
| qEV original, 35 nm | Izon | maximal loading volume of 0.5 mL | |
| qEV rack | Izon | for use with the qEV-original SEC columns | |
| qEV-2, 35 nm | Izon | maximal loading volume of 2 mL | |
| Qubit fluorometer | ThermoFisher | Item no longer available. Closest available product is Qubit 4.0 Fluorometer (cat. No. Q33238) | |
| Qubit protein assay kit | ThermoFisher | Q33211 | Store kit at room temperature. Standards are stored at 4 °C. |
| Sorvall Lynx 4000 centrifuge | ThermoFisher | 75006580 | |
| SpectraMax i3x Microplate reader | Molecular Devices | This equipment was used to measure the nanoluciferase bioluminescence | |
| Stericup Quick-release-GP Sterile Vacuum Filtration system (150, 250, or 500 mL) | MilliporeSigma | S2GPU01RE S2GPU02RE S2GPU05RE | One or multiple filters can be used to accommodate working volumes. In our experience, you can filter twice the volume listed on the product size. |
| Uranyl acetate | Electron microscopy sciences | 22400 | |
| Vinyl anaerobic chamber | Coy Lab | ||
| Vivacell 100, 100,000 MWCO PES | Sartorius | VC1042 | |
| Whatman Anotop 10 Plus syringe filters (0.02 micron) | MilliporeSigma | WHA68093002 | to filter MRPS diluent |
Referenzen
- Yanez-Mo, M., et al. Biological properties of extracellular vesicles and their physiological functions. Journal of Extracellular Vesicles. 4, 27066(2015).
- Chatterjee, S. N., Das, J. Electron microscopic observations on the excretion of cell-wall material by Vibrio cholerae. Journal of General Microbiology. 49 (1), 1-11 (1967).
- Ciofu, O., Beveridge, T. J., Kadurugamuwa, J., Walther-Rasmussen, J., Hoiby, N. Chromosomal beta-lactamase is packaged into membrane vesicles and secreted from Pseudomonas aeruginosa. Journal of Antimicrobial Chemotherapy. 45 (1), 9-13 (2000).
- Yonezawa, H., et al. Outer membrane vesicles of Helicobacter pylori TK1402 are involved in biofilm formation. BMC Microbiology. 9, 197(2009).
- Mashburn, L. M., Whiteley, M. Membrane vesicles traffic signals and facilitate group activities in a prokaryote. Nature. 437 (7057), 422-425 (2005).
- Kato, S., Kowashi, Y., Demuth, D. R. Outer membrane-like vesicles secreted by Actinobacillus actinomycetemcomitans are enriched in leukotoxin. Microbial Pathogenesis. 32 (1), 1-13 (2002).
- Petousis-Harris, H., et al. Effectiveness of a group B outer membrane vesicle meningococcal vaccine against gonorrhoea in New Zealand: a retrospective case-control study. Lancet. 390 (10102), 1603-1610 (2017).
- Kim, O. Y., et al. Bacterial outer membrane vesicles suppress tumor by interferon-gamma-mediated antitumor response. Nature Communications. 8 (1), 626(2017).
- Thery, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. Journal of Extracellular Vesicles. 7 (1), 1535750(2018).
- Consortium, E. -T., et al. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14 (3), 228-232 (2017).
- Watson, D. C., et al. Efficient production and enhanced tumor delivery of engineered extracellular vesicles. Biomaterials. 105, 195-205 (2016).
- Watson, D. C., et al. Scalable, cGMP-compatible purification of extracellular vesicles carrying bioactive human heterodimeric IL-15/lactadherin complexes. Journal of Extracellular Vesicles. 7 (1), 1442088(2018).
- Lasaro, M., et al. Escherichia coli isolate for studying colonization of the mouse intestine and its application to two-component signaling knockouts. Journal of Bacteriology. 196 (9), 1723-1732 (2014).
- Kim, J. Y., et al. Engineered bacterial outer membrane vesicles with enhanced functionality. Journal of Molecular Biology. 380 (1), 51-66 (2008).
- Beveridge, T. J. Structures of gram-negative cell walls and their derived membrane vesicles. Journal of Bacteriology. 181 (16), 4725-4733 (1999).
- Reimer, S. L., et al. Comparative analysis of outer membrane vesicle isolation methods with an Escherichia coli tolA mutant reveals a hypervesiculating phenotype with outer-inner membrane vesicle content. Frontiers in Microbiology. 12, 628801(2021).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten