Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Abgetrennte Maisscheiden für die Lebendzell-Bildgebung der Infektion durch pilzliche Blattmais-Erreger
In diesem Artikel
Zusammenfassung
Dieses Manuskript beschreibt ein optimiertes Inokulationsprotokoll, das abgelöste Maisblatthüllen für reproduzierbare zytologische, physiologische und molekulare Studien von Maisinteraktionen mit pilzlichen Pflanzenpathogenen verwendet. Die Blattscheiden erleichtern die Echtzeitbeobachtung zellulärer Interaktionen zwischen der lebenden Pflanze und dem Pilz in nicht fixiertem Gewebe.
Zusammenfassung
Wir haben ein Protokoll optimiert, um Maisblattscheiden mit hemibiotrophen und nekrotrophen blattpathogenen Pilzen zu impfen. Die Methode ist eine Abwandlung von einer Methode, die ursprünglich auf Reisblattscheiden angewendet wurde, und ermöglicht die direkte mikroskopische Beobachtung des Pilzwachstums und der Pilzentwicklung in lebenden Pflanzenzellen. Blattscheiden, die von Maiskeimlingen mit zwei vollständig geschlüpften Blatthalsen entnommen wurden, werden mit 20 μl-Tropfen von 5 x 105 Sporen/ml Pilzsporensuspensionen beimpft und in Feuchtekammern bei 23 °C unter kontinuierlichem Fluoreszenzlicht inkubiert. Nach 24-72 h wird überschüssiges Gewebe mit einer Rasierklinge entfernt, um eine einzelne Schicht Epidermiszellen zu hinterlassen, eine optisch klare Probe, die direkt abgebildet werden kann, ohne dass eine chemische Fixierung oder Reinigung erforderlich ist. Pflanzen- und Pilzzellen bleiben für die Dauer des Experiments am Leben und Interaktionen können in Echtzeit visualisiert werden. Hüllen können gefärbt oder einer Plasmolyse unterzogen werden, um die Entwicklungszytologie und Lebensfähigkeit von Wirts- und Pathogenzellen während der Infektion und Kolonisierung zu untersuchen. Pilzstämme, die so transformiert wurden, dass sie fluoreszierende Proteine exprimieren, können auf den Hüllen inokuliert oder co-inokuliert werden, um die Auflösung zu erhöhen und die Bewertung kompetitiver oder synergistischer Interaktionen zu erleichtern. Pilzstämme, die fluoreszierende Fusionsproteine exprimieren, können verwendet werden, um die Produktion und das Targeting dieser einzelnen Proteine in planta zu verfolgen und zu quantifizieren. Inokulierte Scheidengewebe können extrahiert werden, um Nukleinsäuren, Proteine oder Metaboliten zu charakterisieren. Der Einsatz dieser Scheidenassays hat die detaillierten Untersuchungen der Mechanismen der pilzlichen Pathogenität in Mais sowie der Effektoren von Pilzproteinen und Sekundärmetaboliten, die zur Pathogenität beitragen, erheblich vorangebracht.
Einleitung
Räumliche und zeitliche Analysen auf zellulärer Ebene sind entscheidend für das Verständnis der Physiologie und Zytologie von Pilz-Pflanzen-Interaktionen. Blattgewebe, das chemischfixiert wurde 1,2,3 oder gereinigt und gefärbt 4, sowie künstliche Membranen5 wurden in der Vergangenheit verwendet, um die Zytologie der Blattpathogenentwicklung und der Wechselwirkungen zwischen Pflanzen und Pilzen zu untersuchen. Die Untersuchung von Infektionsereignissen in lebenden Wirtsgeweben in Echtzeit ohne Fixierung oder Klärung ist jedoch aufgrund technischer Probleme im Zusammenhang mit der Vorbereitung optisch transparenter Proben für die Bildgebung eine Herausforderung.
In den späten 1940er Jahren wurde ein Inokulationsprotokoll für die Inokulation mit abgelöster Blattscheide entwickelt, um die Resistenz lebender Reisepidermiszellen gegen den Reisblastenpilz Magnaporthe oryza6 hellfeldmikroskopisch zu untersuchen. In jüngerer Zeit wurden detaillierte molekulare, physiologische und zytologische Beobachtungen der Wirtsbesiedlung durch Colletotrichum- und Magnaporthe-Arten durch die Kombination modifizierter Versionen dieser Blattscheidenmethode mit Pilztransformanten, die fluoreszierende Proteine exprimieren, und leistungsstarken Live-Cell-Imaging-Protokollen, einschließlich Epifluoreszenz und konfokaler Mikroskopie, erheblich erleichtert 7,8,9,10.11,12,13.
In dieser Arbeit wird ein optimiertes Inokulationsprotokoll unter Verwendung von abgelösten Maisblattscheiden zur Beobachtung von Infektionsprozessen durch hemibiotrophe und nekrotrophe Blattpilzerreger beschrieben. Wir haben es speziell verwendet, um Colletotrichum graminicola (C. graminicola), den Erreger der Anthraknose-Blattfäule und Stängelfäule, und Stenocarpella maydis, die Diplodia-Blattfäule und Stängelfäule verursacht, zu untersuchen. Die Methode sollte jedoch auch auf andere hemibiotrophe und nekrotrophe Blattpilzpathogene anwendbar sein. Die zytologischen und physiologischen Reaktionen während der Infektions- und Kolonisationsereignisse in diesen herausgeschnittenen Blattscheiden ähneln denen in ganzen Blattspreiten12,14,15. Darüber hinaus ähnelt die hemibiotrophe Besiedlung von Scheidenepidermiszellen durch C. graminicola der Besiedlung von Stängelmarkzellen16,17. Abgelöste Hüllen zeigen eine größere Synchronizität und experimentelle Reproduzierbarkeit der Pilzpenetration und -besiedlung als Blattspreiten oder Stängelmarkgewebe 14,16,17,18. Die meisten Maissorten können für dieses Protokoll verwendet werden. Inzuchten oder Hybriden mit übermäßigen violetten Pigmenten in den Scheiden sind jedoch weniger geeignet, da die Pigmente die Bildgebung stören. Golden Jubilee Zuckermais war für unsere Studien besonders nützlich, da unbehandeltes Saatgut im Handel erhältlich ist, die Pflanzen sehr anfällig für viele Blattkrankheiten sind und im Gewächshaus gut wachsen. Die ersten Epidemien der Anthraknose-Stängelfäule in den Vereinigten Staaten führten in den 1970er Jahren zum Totalverlust der Zuckermaisernte in Indiana19,20. Diese Methode der Blattscheideninokulation kann angewendet werden, um das Pilzwachstum und die Pilzentwicklung in lebenden vs. lokal abgetöteten Pflanzenzellen direkt zu beobachten und zu quantifizieren, um Resistenzreaktionen in kompatiblen/inkompatiblen Reaktionen auf eine Pilzinfektion nachzuweisen und um Interaktionen zwischen Pilzstämmen auf derselben Scheide in Echtzeit zu testen.
Protokoll
HINWEIS: Der Workflow für die Methode ist in Abbildung 1 dargestellt.
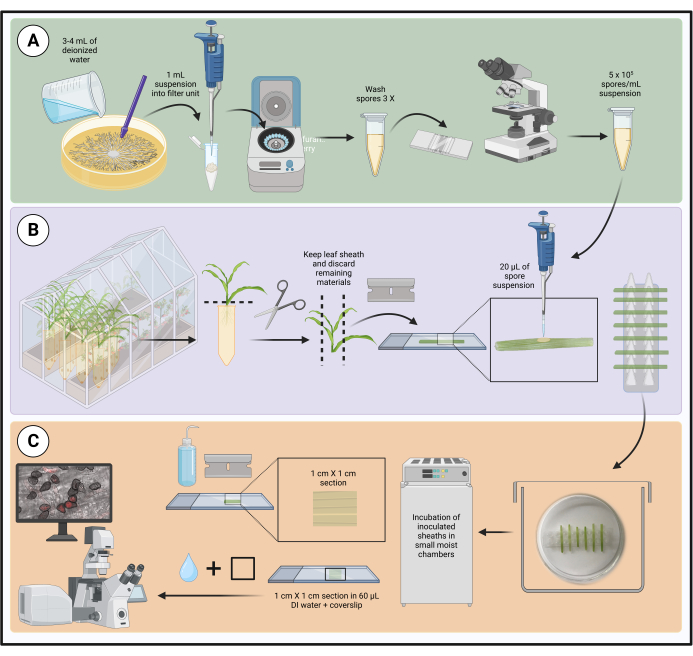
Abbildung 1: Schritte des optimierten Impfprotokolls mit abgelösten Maisblatthüllen. Die Herstellung der Sporensuspension, die Inokulation der Blattscheide und die Probenvorbereitung für die Lebendzellmikroskopie sind in grünen (A), violetten (B) bzw. orangefarbenen (C) Kästchen hervorgehoben. Erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Pflanzen- und Pilzmaterial
- Pflanzenwachstum
- Maissetzlinge (siehe Materialtabelle) im Gewächshaus (14 h hell/27 °C und 10 h dunkel/22 °C) in SC10-Behältern (siehe Materialtabelle) ziehen. Verwenden Sie ein Nährmedium, das aus drei Teilen handelsüblicher Blumenerde (siehe Materialtabelle) und zwei Teilen dampfsterilisierter Muttererde besteht.
- Gießen Sie die Setzlinge einmal täglich morgens 2 Minuten lang mit einem Überkopf-Bewässerungssystem.
- Ernten Sie Sämlinge im V2-Stadium, indem Sie sie auf Bodenebene schneiden. Das V2-Wachstumsstadium wird erreicht, wenn die Sämlinge 5 bis 10 cm hoch sind und zwei sichtbare Kragenblätter haben21.
- Wickeln Sie die Pflanzen mit einem feuchten Papiertuch ein und legen Sie sie in eine Plastiktüte, um sie zur weiteren Verarbeitung ins Labor zu transportieren.
- Pilzkulturen
- Reaktivieren Sie gelagerte Pilz-Kieselsäure-Stammkulturen unter aseptischen Bedingungen22, indem Sie etwa 50 Körnchen der befallenen Kieselsäure auf ein geeignetes Agar-Medium streuen. Um morphologische Veränderungen und den Verlust der Pathogenität zu vermeiden, werden Subkulturstämme nicht mehr als 3x nach der Reaktivierung gezüchtet. Kartoffel-Dextrose-Agar (PDA; siehe Materialtabelle) wird routinemäßig für die Kultivierung von C. graminicola verwendet, während Haferflocken-Agar (OA; siehe Materialtabelle) für S. maydis verwendet wird.
- Um PDA für die Kultivierung von Pilzbeständen vorzubereiten, suspendieren Sie 19,5 g kommerziell hergestelltes dehydriertes PDA in 500 ml deionisiertem (DI) Wasser. Um OA herzustellen, kochen Sie 36 g kommerziell hergestelltes dehydriertes OA 15-30 Minuten lang in DI-Wasser, filtern Sie durch drei Schichten Mulltuch und bringen Sie das Filtrat mit DI-Wasser auf 500 ml. Zu sterilisierende Medien autoklavieren.
- Ergänzen Sie das geschmolzene, gekühlte Medium mit einem geeigneten Antibiotikum (z. B. Hygromycin B, Geneticin), wenn Sie mit transformativen Stämmen arbeiten. Die Endkonzentrationen von Hygromycin oder Geneticin in 500 ml Medien betragen 250 μg/ml bzw. 100 μg/ml.
- Kulturen unter optimalen Bedingungen inkubieren, bis sich ein sporulierendes Myzel entwickelt hat. Colletotrichum graminicola und S. maydis gedeihen gut bei 23 °C bei kontinuierlichem Fluoreszenzlicht und Luftfeuchtigkeit. Die Sporulation erfolgt 7 Tage nach der Inokulation (dai) bei S. maydis bzw. 14 dai bei C. graminicola.
2. Impfungen der Blattscheide
- Glaswolle-Filtereinheit
- Verwenden Sie eine schwere Schere oder Schere, um die Kappe und etwa 0,5 mm vom konischen Boden eines 1,5-ml-Mikrozentrifugenröhrchens abzuschneiden (siehe Materialtabelle).
- Legen Sie ein Stück Glaswolle (ca. 0,5 cm x 0,5 cm; siehe Materialtabelle) in das geschnittene Rohr, um das mittlere Loch abzudecken, wie in Abbildung 2 dargestellt.
- Tragen Sie beim Schneiden von Glaswolle Handschuhe, da Borosilikatglas Reizungen verursachen kann.
- Vorbereitung von Blattscheiden-Stützgestellen
- Schneiden Sie eine 96-Well-PCR-Platte ohne Rand (siehe Materialtabelle) in sechs zweispaltige (8 x 2 Wells) Stützracks, wie in Abbildung 3A dargestellt.
- Drehen Sie die Zweisäulen-Racks um, so dass die konischen Böden der Wells nach oben und die flachen Böden nach unten zeigen (Abbildung 3B).
- Legen Sie jede Stütze in eine Petriplatte aus Glas, die angefeuchtetes Filterpapier enthält, und decken Sie sie mit dem Deckel ab (siehe Materialtabelle). Diese Einheit fungiert als kleine Feuchtigkeitskammer für die Hüllen.
- Inokulum-Präparat
- Legen Sie für jeden Pilzstamm eine zusammengebaute Glaswolle-Filtereinheit in ein intaktes, steriles 1,5-ml-Mikrozentrifugenröhrchen, wie in Abbildung 2 dargestellt. Letzteres fungiert als Sammelröhrchen.
- Beschriften Sie die Röhrchen.
- Entnehmen Sie Konidien aus sporulierenden Kulturen, indem Sie zuerst jede Platte mit 3-4 ml sterilem DI-Wasser fluten. In einigen Fällen kann mehr Wasser erforderlich sein, um eine Flüssigkeitsschicht auf der Kolonieoberfläche zu erzeugen. Netzmittel sind bei diesem System nicht notwendig.
- Lösen Sie die Sporen aus dem Agar, indem Sie einen sterilen Stößel mit konischer Spitze (siehe Materialtabelle) verwenden, um gleichmäßig über die gesamte Platte zu kratzen.
- 1 ml der Sporensuspension aseptisch auf eine Glaswollefiltereinheit geben und die Sporen durch Schwerkraft in das Sammelröhrchen fließen lassen.
- Die Sammelröhrchen mit den filtrierten Sporensuspensionen werden bei 3.500 x g 5 min zentrifugiert. Die Sporen sollten am Boden des Mikrozentrifugenröhrchens pelletiert werden.
HINWEIS: Das Zentrifugieren von C. graminicola-Sporen mit höheren Geschwindigkeiten als hier empfohlen führt zu einem erheblichen Verlust der Lebensfähigkeit. - Gießen Sie die Flüssigkeit in einen autoklavierbaren Behälter, fügen Sie 1 ml steriles DI-Wasser hinzu und rühren Sie vorsichtig, um die pelletierten Sporen zu resuspendieren. Zentrifugieren Sie wie in Schritt 2.3.6.
- Waschen Sie die Sporen 3x, um alle Konidialmatrix zu entfernen, die Autoinhibitoren enthalten kann, die die Keimung oder Penetration verringern könnten.
- Nach dem dritten Waschen fügen Sie 300-500 μl steriles DI-Wasser hinzu, um die Sporen für die Quantifizierung zu resuspendieren.
- Verwenden Sie ein Hämozytometer unter einem zusammengesetzten Mikroskop mit 100-facher Vergrößerung, um die Sporenkonzentration zu bestimmen. Eine Sporenfärbung ist vor der Zählung nicht notwendig.
- Bereiten Sie eine 5 x 105 Sporen/ml Suspension mit sterilem DI-Wasser vor.
HINWEIS: Die Sporensuspension von C. graminicola kann nicht länger als 4 h bei Raumtemperatur aufbewahrt werden, bevor sie schnell an Lebensfähigkeit verliert. Das Kühlen von Konidien erhöht nicht die Lebensfähigkeit.
- Scheiden-Impfungen
- Überprüfen Sie die relevanten Biosicherheitspraktiken und -verfahren, bevor Sie Pflanzen mit Pilzstämmen impfen.
- Entferne die Hülle vom ersten echten Blatt der V2-Sämlinge, indem du mit einem Daumennagel am überlappenden Rand der Scheide entlangfährst, und löse sie vorsichtig vom Trieb. Löse die Hülle von beiden Seiten des Sprosses, bevor du versuchst, sie zu entfernen.
- Schneiden Sie die geborgenen Blattscheiden in 3-5 cm große Segmente. Es ist nicht notwendig, die Hüllen vor der Impfung zu desinfizieren.
- Rollen Sie jedes Segment sehr vorsichtig ab, um die innere (adaxiale) Epidermisschicht freizulegen.
- Bereiten Sie die Hüllen für die Impfung vor und wickeln Sie die verbleibenden herausgeschnittenen Hüllen in ein feuchtes Papiertuch, um ein Austrocknen zu vermeiden.
- Geben Sie 20 μl der Pilzsporensuspension auf die Innenfläche in der Mitte des Mantelstücks, direkt über der Mittelrippe, wie in Abbildung 4 dargestellt.
- Legen Sie die inokulierten Blattscheiden waagerecht, mit der Mittelrippe nach unten, in die Halterung innerhalb einer Petriplatte aus Glas, die ein angefeuchtetes Filterpapier enthält, wie in Abbildung 5 dargestellt.
- Jedes Gestell fasst bis zu sieben Hüllen. Impfen Sie mindestens fünf Hüllen pro Stamm, um den Verlust von Replikaten zu kompensieren, der während der Probenvorbereitung oder Inkubation auftreten kann.
- Stellen Sie die kleinen Feuchtigkeitskammern in eine durchsichtige Aufbewahrungsbox, die mit angefeuchtetem Keimpapier ausgelegt ist (siehe Materialtabelle).
- Decken Sie die Schachtel mit dem Deckel ab. Die Box wird bei 23 °C mit kontinuierlicher Beleuchtung für den vorgesehenen Zeitverlauf bebrütet, der vom Pilz und den beobachteten Stadien der Pilzentwicklung abhängt. Eine Zusammenfassung des Zeitverlaufs für Maisschalen, die mit C. graminicola geimpft wurden, ist in Tabelle 1 enthalten.
- Kontrollieren Sie jeden Tag die Hüllen auf Anzeichen/Symptome von Krankheiten und halten Sie sowohl die Keim- als auch die Filterpapiere feucht. Die Blattscheiden können bis zu 6 Tage lang aufbewahrt werden, ohne dass es zu einem offensichtlichen Absterben oder Abbau der Pflanzenzellen kommt, wenn keine Inokulation durchgeführt wird.
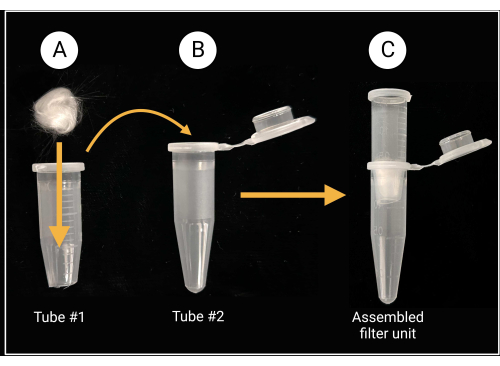
Abbildung 2: Vorbereitung der Glaswolle-Filtereinheit. (A) Eine Glaswollekugel von 0,5 cm x 0,5 cm wird in das Mikrozentrifugenröhrchen 1 gelegt, dessen konischer Boden entfernt wurde. (B-C) Das Filterrohr wird dann in das Mikrozentrifugenröhrchen 2 gelegt, um eine zusammengebaute Filtereinheit für die Herstellung der Sporensuspension zu erzeugen. Erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
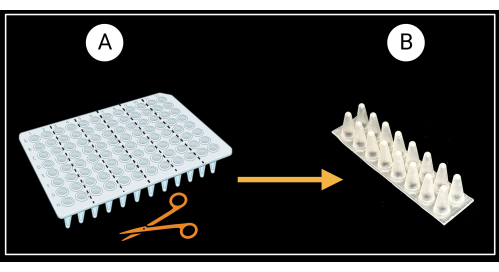
Abbildung 3: Methode zum Schneiden einer 96-Well-PCR-Platte ohne Rand. (A) PCR-Platte, geschnitten in sechs Stützracks, 8 x 2 Wells. Ein Beispiel für eine einzelne Mantelstütze ist in (B) dargestellt. Blattscheiden werden waagerecht auf die Auflage gelegt. Erstellt mit BioRender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Methode der Scheideninokulation. Einzelner Tropfen des Inokulums, der direkt auf die adaxiale Oberfläche des Mantelabschnitts aufgetragen wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
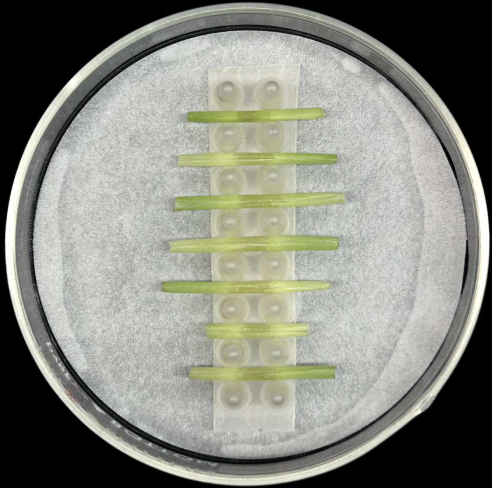
Abbildung 5: Inkubationsmethode der Scheide. Inokulierte Blatthüllen, die horizontal in einem Stützgestell in einer Petriplatte aus Glas platziert werden, die angefeuchtetes Filterpapier enthält. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Lebendzell-Mikroskopie
- Probenvorbereitung für die Mikroskopie
- Spülen Sie die Scheidenteile vorsichtig mit sterilem DI-Wasser ab, um nicht anhaftende Sporen oder oberflächliches Pilzwachstum zu entfernen.
- Legen Sie die Hülle auf einen sauberen Objektträger aus Glas (siehe Materialtabelle), wobei die adaxiale (innere) Oberfläche des Hülsenstücks nach unten und die abaxiale (äußere) Oberfläche nach oben zeigt.
- Verwende eine neue einschneidige Rasierklinge (siehe Materialtabelle), um etwa 1 cm von den Scheidenenden abzuschneiden und den größten Teil des Laminagewebes auf beiden Seiten der Mittelrippe zu entfernen.
- Halten Sie die Rasierklinge in einem Winkel von 90° zur Scheide, um Gewebe von der abaxialen Mittelrippe zu rasieren und die Epidermisschicht auf der adaxialen Oberfläche über der inokulierten Stelle freizulegen. Versuchen Sie, eine gleichmäßige Dicke beizubehalten. Sobald die Blattscheiden rasiert sind, wird ein weiteres Trimmen nicht empfohlen, da dies die Probe beschädigen kann.
- Achten Sie darauf, dass die rasierten Partien nicht größer als 1 cm x 1 cm sind. Vermeiden Sie es, während dieses Vorgangs auf die Mitte der Hülle zu drücken. Wenn Sie mit Hüllen arbeiten, die mit verschiedenen Sporensuspensionen beimpft wurden, desinfizieren Sie die Handschuhe und wechseln Sie die Rasierklinge zwischen den Proben.
- Halten Sie einen sauberen Objektträger aus Glas bereit. Heben Sie das Mantelteil von einer Kante ab und übertragen Sie es vorsichtig auf einen neuen Objektträger. Die inokulierte (adaxiale) Oberfläche der Hülle sollte sich oben befinden, der Objektivlinse am nächsten. Montieren Sie die Abschnitte vorsichtig, um Schäden an Pilzstrukturen zu vermeiden.
- Tragen Sie 60 μl steriles DI-Wasser auf die Sektion auf und fügen Sie ein 24 mm x 60 mm großes Deckglas hinzu (siehe Materialtabelle). Tun Sie dies langsam, um Luftblasen zu vermeiden.
- Wenn Sie für die Mikroskopie bereit sind, versiegeln Sie das Deckglas mit klarem Nagellack (siehe Materialtabelle), indem Sie einen kleinen Tropfen auf jede Kante geben und die Tropfen dann mit einem Nagellackpinsel verbinden, um eine nahtlose Versiegelung zu erzielen.
HINWEIS: Dieses Mantelprotokoll kann mit zytologischen Färbungen verwendet werden, z. B. 4′,6-Diamidino-2-phenylindol (DAPI), neutrales Rot oder Trypanblau, oder zur Beobachtung der Zellplasmolyse, um die Lebensfähigkeit von Pflanzenzellen zu bewerten. Bei Mantelfärbungen oder Zellplasmolyse-Assays darf das Deckglas nicht versiegelt werden. - Für Plasmolyse-Assays wird eine hypertone Lösung (0,75 M Saccharose oder 1 M Natriumchlorid) auf einen Rand des Deckglases gegeben und die Lösung mit einem gefalteten fusselfreien Seidenpapier über die Probe zum anderen Rand des Deckglases gezogen. Verwenden Sie für die Färbung eine ähnliche Methode, um die Färbelösung aufzutragen.
- Weitfeld-Mikroskopie
- Sobald die Blattscheiden auf Objektträgern montiert sind, untersuchen Sie sie mit einem Weitfeld-Lichtmikroskop mit 400-facher Vergrößerung auf Pilzbesiedelung.
- Um Pilzstrukturen im Detail zu beobachten, verwenden Sie ein Wasserimmersionsobjektiv anstelle von Öl, um eine bessere Bildqualität der Proben zu erzielen. Eine sphärische Aberration aufgrund einer Fehlanpassung des Brechungsindex kann zu einer Verschlechterung des Bildes führen. Wasserobjektive sind sowohl für Weitfeld-Lichtmikroskope als auch für konfokale Mikroskope erhältlich.
- Um die relative Menge der Besiedlung pro Hülle zu bestimmen, wird die Anzahl der Maiszellen, die von jeder Pilzeindringstelle eingedrungen sind, mit einem Weitfeld-Lichtmikroskop bei 100-facher Vergrößerung gezählt. Diese Quantifizierung ist ein geeigneter Screening-Schritt vor statistischen Analysen, bei denen Stämme, Behandlungen und/oder Entwicklungsstadien verglichen werden.
- Konfokale Laser-Scanning-Mikroskopie
- Um fluoreszierende Proteine in Maisgewebe während der Entwicklung transgener Pilzstämme zu beobachten, müssen die grundlegenden Bildaufnahmeparameter angepasst werden. Ermitteln Sie die besten Anregungs-/Emissionseinstellungen basierend auf dem ausgewählten Fluoreszenzmarker.
HINWEIS: Ein 60-faches Wasserobjektiv ist vorzuziehen, wenn fluoreszierende Fusionen mit sezernierten Proteinen analysiert werden. Moderne konfokale Mikroskope verfügen über hochkorrigierte Wasserimmersionsobjektive, um Artefakte und Formfehler zu vermeiden. - Wenn die Bildgebung fluoreszierender Proteine in lebenden Zellen beabsichtigt ist, verwenden Sie untransformierte Pilzstämme als Kontrollen für die Autofluoreszenz. Verwenden Sie die folgenden Bildaufnahmeparameter, um die Dynamik von Pilzen und Wirten auf einem inversen konfokalen Mikroskop und für das fluoreszierende mCherry-Protein zu erfassen. Stellen Sie die Laserleistung auf bis zu 5 %, 450-600 Hochspannung (HV) je nach Expressionsniveau, 1,375-fache Verstärkung, 3 % Offset, einen optischen Zoomfaktor von 1 für die Analyse von bis zu fünf Maiszellen pro Abschnitt und einen optischen Zoomfaktor von 2-5 für einzelne Hyphen ein.
HINWEIS: Hochauflösende konfokale Z-Stack-Bilder liefern dreidimensionale Daten und eine bessere Sicht auf Echtzeit-Interaktionen zwischen Wirt und Erreger. - Für die Quantifizierung von Fluoreszenzintensitäten, die den sezernierten Fusionsproteinen entsprechen, verwenden Sie eine Bildanalysesoftware des konfokalen Herstellers oder ein Bildverarbeitungsprogramm wie ImageJ, das kostenlos online heruntergeladen werden kann. In einem Handbuch finden Sie weitere Informationen zur entsprechenden Quantifizierung von Fluoreszenzsignalen.
- Um fluoreszierende Proteine in Maisgewebe während der Entwicklung transgener Pilzstämme zu beobachten, müssen die grundlegenden Bildaufnahmeparameter angepasst werden. Ermitteln Sie die besten Anregungs-/Emissionseinstellungen basierend auf dem ausgewählten Fluoreszenzmarker.
Ergebnisse
Die folgenden Beispiele beschreiben repräsentative Ergebnisse nach der Anwendung der Inokulationsmethode mit Maisblattscheiden. Diese Beispiele zeigen, wie einfach, schnell und präzise die Beobachtung und der Vergleich von Mais-Pilz-Interaktionen mit diesem optimierten Assay in Echtzeit durchgeführt werden können. Die Bildgebung lebender Zellen ermöglicht auch die Extraktion quantitativer Informationen und ist ein nützliches Werkzeug für vergleichende molekulare, zytologische und physiologische Studien. Weitere De...
Diskussion
Die hier beschriebene optimierte Blattscheideninokulationsmethode ist modifiziert von einem Originalprotokoll, das für Reisblattscheiden entwickelt wurde und auf Reisblattscheiden angewendet wurde 6,8,36. Es ermöglicht direkte, detaillierte Beobachtungen des Pilzwachstums und der Pilzentwicklung in lebenden Pflanzenzellen entweder mit Weitfeld- oder konfokaler Mikroskopie. Das Protokoll eignet sich für die Charakterisierung, ...
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben und nichts offenzulegen haben.
Danksagungen
Die Autoren danken USDA-NIFA für die finanzielle Unterstützung (Fördernummern 2018-67013-28489 und 2020-70410-32901). Alle Meinungen, Erkenntnisse, Schlussfolgerungen oder Empfehlungen, die in diesem Manuskript zum Ausdruck gebracht werden, sind ausschließlich die der Autoren und spiegeln nicht unbedingt die Ansichten des US-Landwirtschaftsministeriums wider. Wir danken der brasilianischen Gaststudentin von Science Without Borders, Mayara de Silva, für die Bilder, die in Abbildung 6A und in Abbildung 7D zu sehen sind. Wir danken auch der Abteilung für Pflanzenpathologie an der University of Kentucky für den Zugang zu den konfokalen Mikroskopen von Olympus.
Materialien
| Name | Company | Catalog Number | Comments |
| Axiocam monochrome microscope camera | ZEISS | 426560-9010-000 | Compatible with the Axioplan 2 microscope; provides low read noise and high speed for live cell imaging |
| Axioplan 2 epifluorescence microscope | ZEISS | N/A | Allows live viewing and image/video capture of biological samples |
| Benchtop centrifuge 24 X 1.5/2 mL | Thermo Fisher Scientific | 75002431 | Sorvall Legend Micro 17; max speed: 13,300 rpm (17,000 x g) |
| Falcon bacteriological Petri dish with lid | Fisher Scientific | 08-757-105 | Polystyrene material; hydrophobic surface |
| Filter paper | Fisher Scientific | 09-920-115 | Whatman grade 1 for Petri plate moist chambers |
| FV 3000 laser scanning confocal microscope | Olympus | N/A | For visualization of fungal transformants' |
| Germination paper | Anchor Paper Co. | SD7615L | 76# heavy weight for plastic box moist chambers |
| Glass Petri dishes | VWR International | 75845-542 | Type 1 class A, 33 expansion borosilicate glass; complete set (cover + bottom), for Petri plate moist chambers |
| Glass wool | Ohio Valley Specialty Chemical | 3350 | For glass-wool filter units |
| Hemocytometer/Neubauer counting chamber and cover glass | VWR International | 15170-172 | 0.1 mm chamber depth; comes with two 0.4 mm cover glasses |
| Microscope coverslips | Fisher Scientific | 12-553-457 | Borosilicate glass; 100/Pk.; 22 mm length, 22 mm width |
| Maize cultivar Golden Jubilee seeds | West Coast Seeds Ltd., Delta, BC, Canada | CN361 | Matures in 95-105 days; seed type: F1 |
| Microcentrifuge tubes | USA Scientific | 1415-2500 | 1.5 mL capacity |
| Microscope slides | Fisher Scientific | 12-550-123 | Superfrost white tab slide; 76 mm length, 25 mm width |
| Oatmeal Agar (OA) | VWR International | 255210 | Difco Oatmeal Agar, BD; 500 g |
| Nail polish | Revlon | 43671 | Clear nail polish for sealing microscope slides; color 771 Clear |
| Non-skirted 96-well PCR plate | USA Sientific | 1402-9500 | 100 uL plate volume |
| Pestle for microcentrifuge tubes | USA Scientific | 1415-5390 | Conical tip; polypropylene material |
| PlanApo 60X/1,00 WLSM water objective | Olympus | 1-UB933 | Compatible with the Olympus FV 3000 confocal microscope |
| Potato Dextrose Agar (PDA) | VWR International | 90000-758 | Difco Potato Dextrose Media, BD; 500 g |
| Pro-Mix BX | Premium Horticulture Supply Co. | N/A | Premium general-purpose growing medium formulated to provide a balance of water retention and proper drainage |
| SC10 cone-tainers | Greenhouse Megastore | CN-SS-SC-10B | 1.5 inch diameter, 8.25 inch depth, and a volume of 164 mL |
| SC10 cone-tainers tray | Greenhouse Megastore | CN-SS-SCTR98 | 24 inch length x 12 inch width x 6.75 inch height; holds up to 98 of SC10 cone-tainers |
| Single edge razor blade | Thermo Fisher Scientific | 17-989-145 | AccuTec blade; steel material; 38 mm length blade |
| Storage containers/boxes with latch closure | Target | 002-02-0405 | Clear view storage boxes for rmoist chamber; outside dimensions: 23 5/8 inch x 16 3/8 inch x 6 1/2 inch; 32 qt. capacity |
Referenzen
- Cheng, Y., Yao, J., Zhang, H., Huang, L., Kang, Z. Cytological and molecular analysis of nonhost resistance in rice to wheat powdery mildew and leaf rust pathogens. Protoplasma. 252 (4), 1167-1179 (2015).
- Hickey, E. L., Coffey, M. D. A fine-structural study of the pea downy mildew fungus Peronospora pisi in its host Pisum sativum. Canadian Journal of Botany. 55 (23), 2845-2858 (1977).
- Wharton, P. S., Julian, A. M., O'Connell, R. J. Ultrastructure of the infection of Sorghum bicolor by Colletotrichum sublineolum. Phytopathology. 91 (2), 149-158 (2001).
- Latunde-Dada, A. O., et al. Infection process and identity of the hemibiotrophic anthracnose fungus (Colletotrichum destructivum O'Gara) from cowpea (Vigna unguiculata (L) Walp.). Mycological Research. 100 (9), 1133-1141 (1996).
- Bourett, T. M., Howard, R. J. In vitro development of penetration structures in the rice blast fungus Magnaporthe grisea. Canadian Journal of Botany. 68 (2), 329-342 (1990).
- Sakamoto, M. On the new method of sheath inoculation of rice plants with blast fungus, Pyricularia oryzae Cav., for the studying of the disease-resistant nature of the plant. Bulletin of the Institute for Agricultural Research, Tōhoku University. 1 (3), 120-129 (1949).
- Buiate, E. A., et al. A comparative genomic analysis of putative pathogenicity genes in the host-specific sibling species Colletotrichum graminicola and Colletotrichum sublineola. BMC genomics. 18 (1), 1-24 (2017).
- Kankanala, P., Czymmek, K., Valent, B. Roles for rice membrane dynamics and plasmodesmata during biotrophic invasion by the blast fungus. The Plant Cell. 19 (2), 706-724 (2007).
- Khang, C. H., et al. Translocation of Magnaporthe oryzae effectors into rice cells and their subsequent cell-to-cell movement. The Plant Cell. 22 (4), 1388-1403 (2010).
- Mosquera, G., Giraldo, M. C., Khang, C. H., Coughlan, S., Valent, B. Interaction transcriptome analysis identifies Magnaporthe oryzae BAS1-4 as biotrophy-associated secreted proteins in rice blast disease. The Plant Cell. 21 (4), 1273-1290 (2009).
- Sakulkoo, W., et al. A single fungal MAP kinase controls plant cell-to-cell invasion by the rice blast fungus. Science. 359 (6382), 1399-1403 (2018).
- Torres, M. F., Cuadros, D. F., Vaillancourt, L. J. Evidence for a diffusible factor that induces susceptibility in the Colletotrichum-maize disease interaction. Molecular Plant Pathology. 15 (1), 80-93 (2014).
- Valent, B., Khang, C. H. Recent advances in rice blast effector research. Current Opinion in Plant Biology. 13 (4), 434-441 (2010).
- Mims, C. W., Vaillancourt, L. J. Ultrastructural characterization of infection and colonization of maize leaves by Colletotrichum graminicola, and by a C. graminicola pathogenicity mutant. Phytopathology. 92 (7), 803-812 (2002).
- Vargas, W. A., et al. Plant defense mechanisms are activated during biotrophic and necrotrophic development of Colletotricum graminicola in maize. Plant Physiology. 158 (3), 1342-1358 (2012).
- Venard, C., Vaillancourt, L. Colonization of fiber cells by Colletotrichum graminicola in wounded maize stalks. Phytopathology. 97 (4), 438-447 (2007).
- Venard, C., Vaillancourt, L. Penetration and colonization of unwounded maize tissues by the maize anthracnose pathogen Colletotrichum graminicola and the related nonpathogen C. sublineolum. Mycologia. 99 (3), 368-377 (2007).
- Berruyer, R., Poussier, S., Kankanala, P., Mosquera, G., Valent, B. Quantitative and qualitative influence of inoculation methods on in planta growth of rice blast fungus. Phytopathology. 96 (4), 346-355 (2006).
- Belisário, R., Robertson, A. E., Vaillancourt, L. J. Maize anthracnose stalk rot in the genomic era. Plant Disease. 106 (9), 2281-2298 (2022).
- Warren, H. L., Nicholson, R. L., Ullstrup, A. J., Sharvelle, E. G. Observations of Colletotrichum graminicola on sweet corn in Indiana. Plant Disease Reporter. 57, 143-144 (1973).
- Ritchie, S. W., Hanway, J. J., Benson, G. O. How a corn plant develops. Special Report, Iowa State University. 48, (1986).
- Tuite, J. . Plant pathological methods: fungi and bacteria. , (1969).
- Wicklow, D. T., Rogers, K. D., Dowd, P. F., Gloer, J. B. Bioactive metabolites from Stenocarpella maydis, a stalk and ear rot pathogen of maize. Fungal Biology. 115 (2), 133-142 (2011).
- Daudi, A., O'Brien, J. A. Detection of hydrogen peroxide by DAB staining in Arabidopsis leaves. Bio-protocol. 2 (18), e263-e263 (2012).
- Benhamou, R. I., Jaber, Q. Z., Herzog, I. M., Roichman, Y., Fridman, M. Fluorescent tracking of the endoplasmic reticulum in live pathogenic fungal cells. ACS Chemical Biology. 13 (12), 3325-3332 (2018).
- Lorang, J. M., et al. fluorescent protein is lighting up fungal biology. Applied and Environmental Microbiology. 67 (5), 1987-1994 (2001).
- Liu, C. Y., Zhu, J., Xie, Z. Visualizing yeast organelles with fluorescent protein markers. JoVE (Journal of Visualized Experiments). 182, e63846 (2022).
- Westermann, B., Neupert, W. Mitochondria-targeted green fluorescent proteins: convenient tools for the study of organelle biogenesis in Saccharomyces cerevisiae. Yeast. 16 (15), 1421-1427 (2000).
- Lee, A. S. The ER chaperone and signaling regulator GRP78/BiP as a monitor of endoplasmic reticulum stress. Methods. 35 (4), 373-381 (2005).
- Krishnakumar, V., et al. A maize database resource that captures tissue-specific and subcellular-localized gene expression, via fluorescent tags and confocal imaging (Maize Cell Genomics Database). Plant and Cell Physiology. 56 (1), e12-e12 (2015).
- Wu, Q., Luo, A., Zadrozny, T., Sylvester, A., Jackson, D. Fluorescent protein marker lines in maize: generation and applications. International Journal of Developmental Biology. 57 (6-7-8), 535-543 (2013).
- O'Connell, R. J., et al. Lifestyle transitions in plant pathogenic Colletotrichum fungi deciphered by genome and transcriptome analyses. Nature Genetics. 44 (9), 1060-1065 (2012).
- Torres, M. F., et al. A Colletotrichum graminicola mutant deficient in the establishment of biotrophy reveals early transcriptional events in the maize anthracnose disease interaction. BMC Genomics. 17 (202), 1-24 (2016).
- Andrie, R. M., Martinez, J. P., Ciuffetti, L. M. Development of ToxA and ToxB promoter-driven fluorescent protein expression vectors for use in filamentous ascomycetes. Mycologia. 97 (5), 1152-1161 (2005).
- Gordon, C. L., et al. Glucoamylase:: green fluorescent protein fusions to monitor protein secretion in Aspergillus niger. Microbiology. 146 (2), 415-426 (2000).
- Koga, H., Dohi, K., Nakayachi, O., Mori, M. A novel inoculation method of Magnaporthe grisea for cytological observation of the infection process using intact leaf sheaths of rice plants. Physiological and Molecular Plant Pathology. 64 (2), 67-72 (2004).
- Xavier, K. V., Pfeiffer, T., Parreira, D. F., Chopra, S., Vaillancourt, L. Aggressiveness of Colletotrichum sublineola strains from Sorghum bicolor and S. halepense to sweet sorghum variety Sugar Drip, and their impact on yield. Plant Disease. 101 (9), 1578-1587 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten