Method Article
Creación de perfiles proteoma redox tiol Uso del etiquetado de espectrometría de masas
En este artículo
Resumen
Nivel de especies reactivas del oxígeno se eleva cuando las células encuentran condiciones de estrés. Aquí se muestra el ejemplo de la tinción Diaminobencidina 3'-3 ', así como el etiquetado cysTMT y espectrometría de masas para perfilar el proteoma redox en Pseudomonas syringae Tratar las hojas de tomate.
Resumen
Pseudomonas syringae pv. Tomate DC3000 cepa no sólo causa la enfermedad mancha bacteriana en Solanum lycopersicum sino también en especies de Brassica, así como en Arabidopsis thaliana, una planta huésped genéticamente 1,2 tratable. La acumulación de especies reactivas del oxígeno (ROS) en los cotiledones inoculados con DC3000 indica un papel de ROS en la modulación de la muerte celular necrótica durante la enfermedad mancha bacteriana del tomate 3. El peróxido de hidrógeno, un componente de ROS, se produce después de la inoculación de las plantas de tomate con Pseudomonas 3. El peróxido de hidrógeno puede ser detectado mediante una tinción histoquímica 3'-3 'diaminobenzidina (DAB) 4. Tinción DAB reacciona con el peróxido de hidrógeno para producir una mancha marrón en el tejido de la hoja 4. ROS tiene un papel regulador del medio ambiente redox celular, que puede cambiar el estado redox de ciertas proteínas 5. La cisteína es un aminoácido importante sensible acambios redox. Bajo la oxidación leve, la oxidación reversible de los grupos sulfhidrilo de cisteína sirve como sensores y transductores de señal redox que regulan una variedad de procesos fisiológicos 6,7. De masas en tándem de etiquetas (TMT) los reactivos permite la identificación simultánea y multiplexado de la cuantificación de proteínas en diferentes muestras mediante espectrometría de masas 8,9. Los reactivos de cisteína-TMT (cysTMT) reactivos habilitar etiquetado selectiva y cuantificación relativa de cisteína que contiene péptidos de hasta seis muestras biológicas. Cada etiqueta cysTMT isobárica tiene la misma masa padre nominal y se compone de un grupo sulfhidrilo-reactiva, un brazo espaciador de MS-neutro y un reportero de MS / MS 10. Después de etiquetado, las muestras fueron sometidos a digestión con proteasa. Los péptidos marcados en cisteína se enriquecieron utilizando una resina que contiene anticuerpos anti-TMT. Durante MS / MS análisis, una serie de iones reportero (es decir, 126-131 Da) surgen en la región de baja masa, proporcionando información sobre la cuantificación relativa.El flujo de trabajo es eficaz para reducir la complejidad de la muestra, la mejora de rango dinámico y el estudio de las modificaciones de cisteína. Aquí presentamos el análisis de proteómica redox del tomate Pst DC3000 tratado (Río Grande) utilizando la tecnología deja cysTMT. Este método de alto rendimiento tiene el potencial de ser aplicado al estudio de otros redox reguladas procesos fisiológicos.
Protocolo
1. Las plántulas en crecimiento y bacterias Preparación
- Las plántulas se germinaron en la mezcla del suelo Metromix 500 (Empresas ICM) en una cámara de crecimiento (160 mmol fotones m -2 s -1 con un fotoperíodo de luz de 8 horas a 22 ° C y 16 horas de oscuridad a 20 ° C durante 1 semana. Relativa humedad se fijó a 70%. Las plántulas fueron regadas con agua del grifo como sea necesario para mantener el suelo se seque.
- Dos plantas de semillero de tamaño similar luego se trasplantan a 4 macetas de diámetro "que contienen la Metromix 500 del suelo y se cultivaron durante 3 semanas en las mismas condiciones que 1,1.
- Pseudomonas syringae (Pst) es inicialmente de T-sembró en B Reyes Medio (KBM) placas (por litro: 20 g de proteosa peptona, 0,1975 g de MgSO 4, 1% de glicerol, 1,5 g de K 2 HPO 4, 18 g de agar) y se cultivaron durante dos días a 28 ° C. Una sola colonia se recoge, se mezcla con 300μl Reyes líquido medio B (20 g peptona proteosa, 0,1975 g de MgSO 4 , 10 ml de glicerina, 10 ml de 100x K 2 HPO 4 acciones (1,5 g de K 2 HPO 4 en 10 ml de H 2 O), y se extendió sobre una placa de los Reyes B Medio. Esto se cultivaron durante la noche a 28 ° C.
- El inóculo se prepara bacterias raspando las bacterias cultivadas a partir de la placa KBM en 20 ml de H 2 O. Un diámetro exterior de 600 0,03 (~ 10 6 ufc / ml) se prepara en 1 l de tampón de inoculación (10 mM de MgCl 2 + 400 l Silwett-70). Tampón de inoculación menos bacterias se preparó para la solución de control.
2. La inoculación de tomate con Pst y H 2 O 2 tinción histoquímica
- Cuatro semanas de edad plantas se inocularon por inmersión en la solución por encima de la inoculación durante 30 segundos. Bolsas de plástico se coloca sobre las plantas inmediatamente después de la inoculación.
- 3'-3 'diaminobencidina (DAB) mancha de 11 se preparó (1 mg / ml de DAB en H 2 O, pH 3,8 (HCl)) tres days antes de su uso para lograr la completa solubilización. La solución se sacudió para mezclar a temperatura ambiente.
- Veinticuatro horas después del tratamiento, la hoja completamente expandida se separó, se colocaron en DAB mancha lado epidérmico, y vacío infiltrado durante 15 min. Las hojas se pusieron entonces en la oscuridad a temperatura ambiente durante una noche para la tinción.
- Las hojas se hierven en etanol al 95% durante 10 minutos, y después se mantuvo en 75% de etanol.
3. La extracción de proteínas
- Después de la cosecha, las hojas de tomate se molieron a polvo fino con nitrógeno líquido que utiliza mortero y almirez.
- Por 0,5 g de tejido, añadir 1,25 ml de Tris pH 8,8 fenol tamponado y 1,25 ml / 0,5 g de tampón de extracción (0,1 M Tris-HCl pH 8,8, 10 mM EDTA, 0,9 M de sacarosa) y luego continuar molienda durante unos minutos más en un campana extractora de humos.
- Transferir el extracto a tubo de centrífuga Oakridge (Thermo Fisher Scientific Company Nalgene) y agitar durante 2 horas a temperatura ambiente.
- Centrifugar a 5000 xgy 15 ° C durante 10 min. Transferir la fase fenol superior a un tubo limpio nuevo.
- Volver a extraer la fase acuosa se extrajo con un volumen igual de fenol tampón; agitar en un agitador durante 30 minutos. Centrifugar y transferir el extracto a un nuevo tubo Falcon.
- Repita el paso anterior, una vez más y transferir el extracto a las nuevas tubo de Oakridge.
- Precipitar fenol proteínas extraídas mediante la adición de 5 volúmenes de acetato de amonio 0,1 M en metanol al 100% (almacenado a -20 ° C). Vortex y se incuba a -20 ° C durante la noche.
- Recoger la proteína sedimento por centrifugación a 20.000 xg, 4 ° C durante 20 min.
- Lavar el pellet 2 veces con el acetato de amonio 0,1 M en metanol frío y 2 veces con 80% de acetona fría. Añadir 1,5 ml de etanol al 70% en frío para que sedimenten y suspender. Transferir a un tubo de microcentrífuga de 2 ml (EE.UU. Científico) y se centrifuga a 14.000 rpm, 4 ° C durante 20 min. Eliminar el etanol.
- Secar el precipitado brevemente en un concentrador SpeedVac y se disuelven en una proteína EXTRtampón acción, por ejemplo, la proteína ReadyPrep Extracción reactivo Kit 3 (8 M de urea, CHAPS 4%, 40 mM Tris-base, 2 M tiourea) (Bio-Rad). Centrifugar a 14.000 rpm y 20 ° C durante 30 minutos para formar un precipitado. Recoger el sobrenadante.
- Medir la concentración de proteínas utilizando una proteína de ensayo CB-X de acuerdo al manual del fabricante (Geno Technology).
4. Preparación de muestras y etiquetado de péptidos con cysTMTs
- Preparar muestra de proteína a una concentración de 2-5 mg / l. Aquí usamos 100 g de proteína por cada etiqueta de masa, 20-l 50 muestras l. Para este experimento todas las etiquetas se utilizaron 6.
- Añadir un volumen igual de tampón de alquilación (100 mM Tris-HCl pH 7,5, 200 mM de yodoacetamida) a la muestra de proteína para bloquear el grupo tiol libre. Buffer debe estar preparado fresco. La alquilación se realiza a 37 ° C durante 1 hora.
- Precipitar la proteína mediante la adición de 1 ml de acetona fría al 80% a -20 ° C durante la noche. Pellet la proteína por centrifugación a 14.000rpm, 4 ° C durante 20 min. Lavar el pellet 3 veces con 80% de acetona vórtex cada vez. Eliminar el sobrenadante y dejar secar al aire seco, mientras que en hielo.
- Resuspender el sedimento en 50 l de tampón de lisis (6 M urea, 50 mM Tris base, 1 mM EDTA). 0,5 l de 100 mM de Tris (2-carboxietil) fosfina (TCEP) se añade a continuación para reducir los enlaces disulfuro por incubación durante una hora a temperatura ambiente.
- Preparar etiquetas mediante la adición de 20 l de acetonitrilo para los tubos de reactivos para solubilizar las etiquetas. Vortex y girar hacia la parte inferior.
- Lavar TCEP la capacidad utilizando una centrífuga Microcon 3KD dispositivo de filtro (Millipore). Añadir 50 l de la muestra a la columna Microcon 3KD, y 50 l de tampón de lisis a la columna. Centrifugar durante 15 min a 10.000 xg y 4 ° C. Repita este paso para un total de tres veces. Quitar la columna y se invierte en un tubo limpio. Centrifugar durante 5 minutos a 1.000 xg y 4 ° C.
- Comprobar el pH. Si es necesario, ajustar el pH a 7,0-8,0 con HCl 1 M.
- Añadir 5 l de la cysTMReactivo T para cada muestra (El estuche de reactivos cysTMT proporciona etiquetas 20 l de hasta 500 mg de proteína. Este método utiliza 100 ug de proteína, por lo tanto, utilizar una quinta parte de la etiqueta.). Permitir que la reacción continúe durante 2 horas a temperatura ambiente.
- Las muestras se combinaron con tampón de muestra Laemmli (Bio-Rad) en una proporción de 1:1.
5. Remoción de los no reaccionó Fraccionamiento Etiqueta de muestra
- Las muestras se hirvieron durante 5 min y se separaron en un gel de poliacrilamida prefabricado 12% (Bio-Rad). El gel se lavó tres veces en intervalos de 5 minutos con filtró H 2 O y se tiñeron con azul de Coomassie (Bio-Rad) durante 1 hora. El gel fue teñido de noche a la mañana con H 2 O.
- Doce fracciones se obtuvieron de cada carril del gel. Las fracciones se determina por las concentraciones de proteínas de la banda. Las fracciones se cortan en trozos de 1 mm y se recoge en un tubo de 1,5 ml.
- Piezas de gel fueron de teñido utilizando 50% de acetonitrilo y 0,1 M de bicarbonato de amonio en H 2 O. Este paso se debe repetir hasta que el color azul se quita de piezas de gel (aproximadamente 3-4 veces, 15 min cada vez).
- De teñidos de piezas de gel se secó usando 100% de acetonitrilo (cubrir piezas de gel) durante 15 min, el sobrenadante eliminado, y las piezas se secaron utilizando un SpeedVac.
- Disolver tripsina (Promega) a 1:10 (tripsina: proteína) proporción en el mismo volumen (muestra como combinaron etiquetados) de bicarbonato de amonio 25 mM. Por ejemplo, 600 g de la muestra de proteína con un volumen de 500 l necesita 60 mg de tripsina se disolvió en 500 l de 25 mM de bicarbonato de amonio.
- Añadir la solución de tripsina a las piezas de gel y establecer en el hielo para rehidratar. Si las piezas no están cubiertos añadir 50 mM de bicarbonato de amonio. Después de la rehidratación, se incuba a 37 ° C durante 12-16 horas.
- Quitar el extracto de proteína líquido de las muestras incubadas.
- Se detiene la reacción y eliminar cualquier digerir proteína restante cubriendo piezas de gel con 5% de ácido fórmico, 50% de acetonitrilo .. Agitar piezas de gel durante 20 minutos en la sala de temperature. Transferir el sobrenadante a los tubos de muestra extraídos de proteínas de 5,7. Repita el procedimiento de extracción en dos ocasiones. Secar los péptidos extraídos utilizando el SpeedVac.
6. El enriquecimiento de la muestra cysTMT etiquetadas-péptidos
- Añadir un gradiente de anti-TMT resina para tubos de 0,5 ml de una suspensión al 50%. (Gradiente de concentración se determinó banda en gel de fraccionamiento. Si una colección fracción consiste en una banda oscura, más resina que se necesita. Fracciones con bandas más débiles requieren menos resina ya que hay menos proteína.)
- La resina se lava luego tres veces con un volumen de columna de 1x tampón con Tris (TBS) (25 mM Tris, 0,15 M NaCl, pH 7,2) (Thermo Scientific Pierce Protein Research Products).
- Añadir 200 1xTBS l para cada muestra. Las muestras se añade entonces a anti-TMT resina (Thermo Scientific Pierce Protein Research Products) y se agitó a temperatura ambiente durante 2 horas seguido de balanceo durante la noche a 4 ° C.
- Añadir la muestra a la columna (ElRMO científicos Pierce productos proteínicos de investigación).
- Lavar cada columna tres veces con 1 200 l de TBS. Esto es seguido con el lavado tres veces con CHAPS 0,05% (disuelto en 1x TBS).
- La columna se lava luego tres veces con 4 M urea en 1x TBS. Doscientos microlitros de H 2 O se utiliza entonces para lavar la columna tres veces.
- Cada muestra se eluyó tres veces con 200 l tampón de elución 50% (50% de acetonitrilo, 0,4% de TFA).
- Las muestras se secan entonces en un concentrador de vacío.
7. Masa análisis de espectrometría
- Resuspender las muestras en 12 l de 3% de acetonitrilo con 0,1% de ácido fórmico y 5 l inyectar directamente en una Eksigent nanoLC-1D columna de cromatografía líquida de alta presión (AB Sciex, EE.UU.).
- Los péptidos se separaron en un ID de Proteopep II columna C18 micras 75 x 20 cm (nuevo objetivo, EE.UU.) utilizando un gradiente de 4-60% (A: acetonitrilo 3%, 0,1% de ácido fórmico; B: 97% de acetonitrilo, 0,1% fórmico ácido)a 3 l / min durante 60 min.
- Un Thermo Scientific LTQ Orbitrap XL espectrómetro de masas se utilizó para detectar péptidos usando una tapa de 2 x 3 experimento consistente en una sola etapa MS seguido por la adquisición de tres espectros MS / MS con mayor energía C-trampa de la disociación (HCD) de la fragmentación seguido de 3 MS / MS con colisión inducida por la disociación (CID) para la identificación de proteínas. Parámetros en este modo fueron: anchura de aislamiento: 3,0 m / z; energía de colisión: 50% (10% con dos etapas). Sólo péptidos por partida doble y triplemente cargados fueron seleccionados para la fragmentación. Los parámetros dinámicos de exclusión se establece en: Repita cuenta = 1; duración de repetición = 60, el tamaño de la lista de exclusión = 500; Exclusión duración = 28. Los valores objetivo son las siguientes: EM = 5 e 5; MS / MS (HCD) = 1 e 5. Los tiempos de transferencia de iones se establece en 500 para FTMS y 300 para MS / MS (HCD). Dos microscans se requiere para los espectros de HCD.
8. Base de datos de la búsqueda y cuantificación
- Los datos adquiridos CID y HCD eran unanalizaron utilizando el software Thermo Scientific Discoverer Proteoma 1.2 (Thermo Scientific Pierce productos de la investigación de proteínas) a través de un flujo de trabajo ramificada que puso en práctica un reportero de iones de cuantificador (20 ppm de tolerancia de la masa de los fragmentos de iones) para cuantificar la relación. Un segmento aparte procesado la MS 2 espectros a través del espectro de selección, Normalizador del espectro, y los ganglios del espectro de mero.
- Los datos se buscaron después contra el motor de búsqueda SEQUEST. Una tolerancia de 20 ppm en masa se utilizó. Modificaciones estáticas se establece en el reactivo cysTMT (304,18 Da) y modificaciones dinámicas incluyó la fosforilación y la oxidación de la metionina (15,99 Da). Una base de datos personalizada de Solanum lycopersicum fue compuesta utilizando ARN de datos (con cerca de 350.000 entradas de la Universidad de Harvard) 13 se utilizó en ambos casos.
9. Los resultados representativos
Una imagen representativa de una hoja de control de la planta de tomate y una hoja de Pseudomonas es inoculadose muestra en la Figura 1. Una diferencia entre el control y tratados hojas Pseudomonas tratados se observa. Después de que las hojas se retiran y se tiñeron utilizando DAB, el proceso de tinción permite la tinción histoquímica a mostrar signos de ROS en el tejido de la hoja (Figura 2). Figura 2A es representativo de una hoja de control sin tinción. Figura 2B es representativa de una hoja tratada con tinción positiva para Pseudomonas y H 2 O 2 de producción. Un ejemplo de Proteoma Descubre salida de datos de una proteína redox diferencialmente regulada se muestra en la Figura 3. Esta proteína es un conocido ferredoxina-1 redox proteína regulada 14 y se ha demostrado que desempeñan un papel en la defensa contra Pseudomonas syringae pv tomate 15. Intensidad de pico entre el control y muestras inoculadas se utiliza para obtener la cuantificación relativa, que mostró cambios significativos en ferredoxen-1 de regulación de óxido-reducción (p <0,05). Picos de alta intensidad sugieren que esta proteína se oxida en respuesta al tratamiento patógeno. Figura 4 es un ejemplo de Proteoma Descubre salida de datos de una proteína que tiene regulación similar redox de una proteína entre un control y muestra inoculada. Los picos de intensidad similar, sugieren la presencia de disulfuro de bonos no están regulados por un cambio en el tratamiento. El método va a revolucionar cómo los científicos detectar cisteínas redox sensibles y disulfuros 10.

Figura 1. Una imagen representativa de tomate inoculado deja con solución de control (A) y Pseudomonas (B).

Figura 2. Una imagen representativa de la tinción de DAB de tomate inoculado deja con solución de control (A) y Pseudomonas (B). Las hojas fueron teñidas utilizando 3'-3 'diaminobencidina. La clorofila fue retirado de las hojas por ebullición en etanol al 95%. Coloración oscura indica la presencia de H 2 O 2. Sólo deja inoculado con cultivo de bacterias mostraron una coloración oscura.
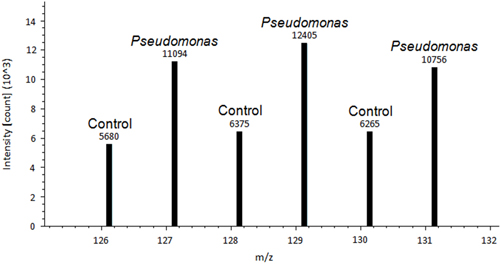
Figura 3. Un ejemplo de Proteoma Descubre salida de datos de ferredoxina-1, proteína regulada diferencialmente redox 14. Intensidad máxima por encima de cada pico se utiliza para la cuantificación absoluta. Intensidad de pico entre el control y muestras inoculadas se utilizapara llevar a cabo la cuantificación relativa.

Figura 4. Un ejemplo de Proteoma Descubre salida de datos de una proteína que tiene la intensidad de pico similar entre un control y muestra inoculada. Los picos de intensidad similar, sugieren la presencia de disulfuro de bonos no están regulados por un cambio en el tratamiento.
Discusión
Este protocolo proporciona información sobre cómo realizar la tinción de DAB, así como la cuantificación cysTMT etiquetados cisteína redox. Estos procedimientos son beneficiosos en el examen de la producción de ROS, así como el efecto sobre la regulación de proteínas cuando Solanum lycopersicum se inocula con Pseudomonas syringae. Los métodos presentados en este protocolo de proporcionar una forma de examinar las ROS en muestras de hojas enteras de una manera que cause el mínimo de daño al tejido de la hoja. El procedimiento de etiquetado proporciona una forma de examinar las proteínas reguladas potencialmente redox mediante la utilización de un método de etiquetado cisteína. Esto es beneficioso cuando se examina una etapa temprana de la respuesta al estrés.
Métodos tales como etiqueta de afinidad isótopo codificado (ICAT) y cysTMT se puede utilizar en el examen de potenciales redox proteínas reguladas en muestras biológicas. ICAT permite el etiquetado y la comparación de dos muestras de 12. Ambos métodos etiquetar cisteínas libres y se puede utilizar para la proteína quantificción 10,12. Sin embargo, el método cysTMT permite una disminución en la variación experimental, así como multiplexación 10. El número de etiquetas disponibles permite a los investigadores para incluir repeticiones o varias muestras de su diseño experimental. La función más muestras proporciona el potencial para un mayor número de proteínas identificadas. Una desventaja importante de la técnica cysTMT es que compromete la calidad general de la identificación de proteínas debido a los pasos de enriquecimiento selectivo para cysTMT etiquetados-péptidos (6.5 a 6.6). El número de péptidos para la identificación de proteínas depende en gran medida el número de residuos de cisteína en la secuencia de la proteína. Este problema se puede superar mediante la presentación de parte de la muestra antes de tríptico enriquecimiento para la identificación de masa de la proteína espectrometría.
Debido a la naturaleza del diseño experimental, así como el mecanismo de etiquetado que utiliza el método cysTMT, ciertos pasos son críticos. Durante la realización de la proteína precisiónpitation y pellets de lavado (3,9) es importante para mantener las muestras refrigerados en hielo para reducir la degradación de proteínas. Durante etiquetado cysTMT, la eliminación del reactivo reductor (4,6) es importante porque las muestras pueden someterse etiquetado inverso. Etiquetado inversa es posible si la reducción de reactivo permanece en la muestra. Si las muestras se reduce después de etiquetado, la etiqueta cysTMT se puede quitar. Una vez que las etiquetas se añaden a las muestras, el nivel de pH debe ser revisado (4,7) con el fin de tener un rendimiento óptimo de etiquetado. Además, el análisis de datos depende de lo que se requiere del investigador y el objetivo final en el uso del protocolo. Es también depende del software utilizado, ya que cada software tiene diferentes algoritmos.
Este experimento utiliza patógeno como un desencadenante de una mayor producción de especies reactivas de oxidación en el tomate, sin embargo, otras respuestas regulados redox se puede medir en consecuencia. Este diseño experimental es adaptable a otra planta y sistemas animales.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Los autores desean agradecer al Dr. Greg Martin (Universidad de Cornell) y su grupo para proporcionar la cepa DC3000, semillas de tomate, y el asesoramiento. También quisiera agradecer al Dr. Zhonglin Mou busca de ayuda con el protocolo de DAB y la División de Proteómica del Centro Interdisciplinario de Investigación UF Biotecnología para la asistencia en el desarrollo del método. El protocolo para la extracción de proteína fue modificada de Hurkman y Tanaka 16. El protocolo sobre cysTMT etiquetado, los pasos 4 a 6 fue adaptado basado en el original producto de Thermo Fisher Scientific Pierce manual de 17. Este trabajo fue financiado por la National Science Foundation (MCB 0,818,051 a S Chen).
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
| Metromix 500 | ICM Empresas | TX-500 | |
| 3,3 '-diaminobenzidina | Sigma-Aldrich | D8001 | |
| ReadyPrep extracción secuencial reactivo kit 3 | Bio-Rad | 163-2104 | |
| CB-X de ensayo de proteínas | Geno Tecnología | 786-12x | |
| cysTMT reactivos | Thermo Scientific Pierce productos proteínicos de investigación | 90071 | |
| Laemmli tampón de muestra | Bio-Rad | 161-0737 | |
| Bio-Safe Comassie (G-250 mancha) | Bio-Rad | 161-0786 | |
| Microcon 3KD columna | Millipore | 42403 | |
| Inmovilizado Anti-TMT resina | Thermo Scientific Pierce productos proteínicos de investigación | 90076 | |
| Se centrifuga la columna | Thermo Scientific Pierce productos proteínicos de investigación | 89896 | |
| Proteopep II columna C18 | Nuevo Objetivo | PFC7515-PP2-10 | |
| NanoLC-1D HPLC | AB Sciex | 90389 | |
| LTQ Orbitrap XL | Thermo Scientific | 0020137580 | |
| SpeedVac | Labconco | 7812013 | |
| Descubridor del Proteoma 1.2 del software | Thermo Scientific Pierce productos proteínicos de investigación | ||
| Tripsina | Promega | V5111 | |
| Oakridge tubo de centrífuga | Thermo Scientific Nalgene la empresa | 3139-0050 | |
| Microcentrífuga tubo (2 ml) | EE.UU. Científico | 1620-2700 | |
| 12% Mini-Protean TGX Prefabricados de Gel | Bio-Rad | 456-1043 | |
| Top of Form > Bio-Safe Coomassie StainBottom del Formulario | Bio-Rad | 161-0786 | |
| TMT kit de enriquecimiento | Thermo Scientific Pierce productos proteínicos de investigación | 90077 |
Referencias
- Almeida, N. F. A draft genome sequence of Pseudomonas syringae pv. tomato T1 reveals a type III effector repertoire significantly divergent from that of Pseudomonas syringae pv. tomato DC3000. Mol. Plant Microbe Interact. 22, 52-62 (2009).
- Preiter, K. Novel virulence gene of Pseudomonas syringae pv. tomato strain DC3000. J. Bacteriol. 187, 7805-7814 (2005).
- Apostol, I., Heinstein, P. F., Low, P. S. Rapid stimulation of an oxidative burst during elicitation of cultured plant cells: role in defense and signal transduction. Plant Physiol. 90, 109-116 (1989).
- Orozco-Cardenas, M., Ryan, C. A. Hydrogen peroxide is generated systemically in plant leaves by wounding and systemin via the octadecanoid pathway. Proc. Natl. Acad. Sci. U.S.A. 96, 6553-6557 (1999).
- Tonks, N. K. Redox redux: revisiting PTPs and the control of cell signaling. Cell. 121, 667-670 (2005).
- Alvarez, S., Wilson, G. H., Chen, S. Determination of in vivo disulfide-bonded proteins in Arabidopsis. J. Chromatogr. B. 877, 101-104 (2009).
- Apel, K., Hirt, H. Reactive oxygen species: metabolism, oxidative stress, and signal transduction. Annu. Rev. Plant Biol. 55, 373-399 (2004).
- Dayon, L. Relative quantification of proteins in human cerebrospinal fluids by MS/MS using 6- plex isobaric tags. Anal. Chem. 80, 2921-2931 (2008).
- Thompson, A. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Anal. Chem. 75, 1895-1904 (2003).
- Rosenblatt, M. A novel cysteine-reactive tandem mass tag reagent for subproteome labeling, enrichment and quantitation. , TP169 (2010).
- Clark, J. Phenotypic analysis of Arabidopsis mutants: diaminobenzidine stain for hydrogen peroxide. Cold Spring Harb. Protoc. , (2009).
- Sethuraman, M. Isotope-Coded affinity tag (ICAT) approach to redox proteomics: identification and quantification of oxidant-sensitive cysteine thiols in complex protein mixtures. J. Proteome Res. 3, 1228-1233 (2004).
- Asso, M. EPR and redox characterization of ferredoxins I and II from Desulfovibrio vulgaris Miyazaki. Biochem. Biophys. Res. Commun. 211, 198-204 (1995).
- Huang, H. e. Disease resistance to bacterial pathogens affected by the amount of ferredoxin-I proteins in plants. Mol. Plant Pathol. 8, 129-137 (2007).
- Hurkman, W., Tanaka, C. Solubilization of plant membrane proteins for analysis by two-dimensional gel electrophoresis. Plant Physiol. 81, 802-806 (1986).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados