Method Article
La detección de bacterias mediante ADNzimas fluorogénicos
En este artículo
Resumen
Recientemente hemos informado de un nuevo enfoque para la generación de sondas fluorogénicos DNAzyme que se pueden aplicar para crear un simple, "mezclar y leer" ensayo de inmunofluorescencia para la detección de bacterias. Estas sondas de ADN especiales catalizar la escisión de un cromóforo modificado con ADN-ARN sustrato quimérico en la presencia de la mezcla bruta extracelular (CEM), producida por una bacteria específica, con lo que la traducción de detección de bacterias en la generación de señales de fluorescencia. En este informe se describen los principales procedimientos experimentales que se emplea una sonda DNAzyme específica denominada "RFD-EC1" para la detección de la bacteria modelo, Escherichia coli (E. coli).
Resumen
Los brotes vinculados a transmitidas por los alimentos y la adquirida en el hospital cuenta de los agentes patógenos de millones de muertes y hospitalizaciones, así como colosales pérdidas económicas cada año. La prevención de estos brotes y la minimización del impacto de la epidemia de un lugar permanente a una demanda cada vez mayor de métodos analíticos que permitan identificar con precisión los agentes patógenos causantes en la primera etapa. Aunque hay una gran variedad de métodos eficaces para la detección de patógenos, ninguno de ellos puede satisfacer todos los siguientes cinco requisitos principales enunciados de un método de detección ideal: alta especificidad (detección de la bacteria sólo de intereses), de alta sensibilidad (capaz de detectar un precio tan bajo como una sola célula bacteriana viva), corto tiempo de los resultados (de minutos a horas), una gran sencillez de funcionamiento (sin necesidad de largos procedimientos de muestreo y el uso de equipo especializado), y la rentabilidad. Por ejemplo, los métodos clásicos microbiológicos son altamente específicos, sino que requieren un tiempo largo (days a semana) para adquirir un resultado definitivo. 1 PCR y técnicas basadas en anticuerpos ofrecen tiempos de espera (horas a días), pero requieren el uso de reactivos caros y / o equipo sofisticado. 2-4 En consecuencia, existe todavía una gran demanda para la investigación científica hacia el desarrollo de innovadores métodos de detección de bacterias que ofrecen características mejoradas en uno o más de los requisitos mencionados anteriormente. Nuestro laboratorio está interesado en examinar el potencial de ADNzimas como una nueva clase de sondas moleculares para aplicaciones de biosensores, incluyendo la detección de bacterias. 5
ADNzimas (también conocido como deoxyribozymes o enzimas de ADN) son hechos por el hombre de cadena sencilla moléculas de ADN con la capacidad de catalizar reacciones químicas. 6-8 Estas moléculas se pueden aislar de una piscina de ADN al azar vasta-secuencia (que contiene hasta 10 16 secuencias individuales) mediante un proceso conocido como "selección in vitro" Or "SELEX" (evolución sistemática de ligandos por enriquecimiento exponencial). 9-16 Estas moléculas de ADN especiales han sido ampliamente examinados en los últimos años como herramientas moleculares para aplicaciones biosensores. 6-8
Nuestro laboratorio ha establecido en los procedimientos de selección in vitro para el aislamiento de ARN-escisión ADNzimas fluorescentes (DSR,. Fig. 1) e investigó el uso de la DSR como herramientas analíticas 17-29 DSR catalizan la escisión de un sustrato de ADN-ARN quimérico en una sola ribonucleótido. cruce (R) que está flanqueado por un fluoróforo (F) y un extintor (Q). La proximidad de F y Q representa la fluorescencia mínima sustrato no escindido. Sin embargo, el evento de escisión conduce a la separación de F y Q, que está acompañado por un incremento significativo de la intensidad de fluorescencia.
Más recientemente, hemos desarrollado un método para aislar DSR para la detección de bacterias. 5 Estos DSR especiales fueron aislados a "iluminar "en la presencia de la mezcla bruta extracelular (CEM), dejada atrás por un tipo específico de bacterias en su medio ambiente o en los medios que se cultivan (fig. 1). El uso de la mezcla bruta evita el tedioso proceso de purificación y la identificación de un objetivo adecuado del microbio de interés para el desarrollo de biosensores (que podría tomar meses o años para completar). El uso de objetivos extracelulares, el procedimiento es simple ensayo, porque no hay necesidad de adoptar medidas para obtener los objetivos intracelulares.
Utilizando el método anterior, se deriva una RFD que escinde su sustrato (FS1;. Fig. 2A) sólo en presencia del CEM producida por E. E. (CEM-CE). 5 E. Este E. detección RFD, llamado RFD-EC1 (Fig. 2A), se encontró que era estrictamente sensible a CEM-CE pero no responde a la CEM de una multitud de otras bacterias (fig. 3).
Aquí se present los procedimientos experimentales clave para la creación de E. Los ensayos de detección de E. coli utilizando los resultados de RFD-EC1 y representativo.
Protocolo
1. Preparación de soluciones químicas
- 0,5 M de ácido etilendiaminotetraacético (EDTA): En un litro 2 (L) vaso de precipitados de plástico, pesa 186,1 g de EDTA (EM Science) y añadir 800 mililitros (ml) de autoclave desionizada agua destilada (ddH 2 O). Ajustar el pH a 8,0 utilizando NaOH gránulos (EM Science). Hacer que el volumen final de 1 L con ddH 2 O. Transferir la solución a botellas de vidrio, autoclave y se almacena a 4 º C.
- 10 x Tris-borato EDTA (10 x TBE; 89 mM Tris, 89 mM de ácido bórico, 2 mM EDTA, pH 7,5): Pesar 432 g de Tris-base (Bioshop Canadá) y 220 g de ácido bórico (Bioshop Canadá) , y añadir a cada una vaso de precipitados de plástico de 4 l. Medir 80 ml de EDTA 0,5 M (pH 8,0) y añadir al vaso de precipitados. Añadir ddH 2 O hasta un volumen final de 4 L. Mezclar la solución con una barra de agitación magnética hasta que los componentes están completamente disueltos. Transferir la solución a botellas de vidrio, autoclave y se almacena a 4 º C.
- 10% de desnaturalización polyacrylamide gel de acciones: Para un vaso de precipitados de plástico de 4 l, añadir 1681,7 g de urea (Bioshop Canadá), 400 ml de 10 x TBE, 1 l de acrilamida / bisacrilamida 40% (29:1) solución (Bioshop Canadá). Ajuste el volumen a 4 litros con ddH 2 O. Disolver la urea con agitación. Transferir la solución a 1 L de botellas de vidrio color ámbar y se almacena a 4 ° C. (Acrilamida ¡Precaución deben ser manejados con guantes, mascarilla, gafas y bata de laboratorio, ya que es una neurotoxina antes de la polimerización).
- 2 x tampón de carga de gel (2 x GLB): Para un vaso de precipitados de 200 ml de vidrio, añadir 44 g de urea, 8 g de sacarosa (Bioshop Canadá), 10 mg de azul de bromofenol (Bioshop Canadá), 10 mg de xylenecyanol FF (Sigma -Aldrich), 400 l de sulfato sódico al 10% de dodecil (SDS; Bioshop Canadá), y 4 ml de 10 x TBE. Ajustar el volumen final a 40 ml con ddH 2 O y disolver los sólidos con calentamiento suave (50 ° C) y agitación con una barra magnética. Transferir una alícuota mL en tubos de 1,5 mL de microcentrífuga y se almacena a 4 º C. Sobre la base de nuestraexperiencia, es necesario al calor 2 x brevemente GLB a 90 ° C antes de su uso debido a 2 x GLB solidifica bajo la condición de almacenamiento.
- 1 M Tris-HCl (pH 7,5): Pesar 12,1 g de Tris-base en un vaso de precipitados de 200 ml de vidrio. Añadir 60 ml de ddH 2 O y disolver los sólidos por agitación con un agitador magnético. Ajustar el pH a 7,5 utilizando HCl 1 M (Sigma-Aldrich). Completar el volumen a 100 ml con ddH2O y transferir a una botella de vidrio y autoclave. Almacenar a 4 ° C.
- 5 M de NaCl: En vaso de vidrio pesan 58,4 g de NaCl (Bioshop Canadá) y se disuelven con 150 ml de ddH 2 O. Ajuste el volumen a 200 ml con ddH 2 O. Transferir la solución a una botella de vidrio, autoclave y almacenar a 4 ° C.
- Tampón de elución del ADN: En un vaso de vidrio, mezclar 2 ml de 1 M Tris-HCl (pH 7,5), 8 ml de NaCl 5 M y 0,4 ml de EDTA 0,5 M (pH 8,0). Ajuste el volumen a 200 ml con ddH 2 O. Autoclave y se almacena a 4 º C.
- 2 × tampón de reacción (2 x RB; HEPES 100 mM, 300 mM NaCl, 30 mM MgCl 2): A un tubo de 50 ml Falcon (BD Falcon), añadir 1,2 g de HEPES (Bioshop Canadá), 0,88 g de NaCl y 0,30 g de MgCl 2 • 6H 2 O (Merck Chemicals) y 30 ml de ddH 2 O. Mezclar agitando suavemente hasta que los componentes están completamente disueltos. Ajustar el pH a 7,5 mediante la adición de 10 N solución de NaOH y ajustar el volumen final a 50 ml con ddH 2 O. Purificar la solución con una unidad de filtro de jeringa impulsado por (0.22 m, Millipore) y se almacena a 4 º C.
- Luria Bertani (LB) Caldo: En un vaso de precipitados, pesa 20,0 g de polvo de LB (Sigma-Aldrich) seguido por la adición de 1 l de ddH 2 O. Mezcle la solución con una barra de agitación magnética, transferir la solución a un matraz cónico. Autoclave la solución y almacenar a temperatura ambiente.
- 1,5% de agar LB: Pesar 1,5 g de agar (Bioshop Canadá) en un matraz de 250 ml y añadir 100 ml de LB líquido. Autoclave la mezcla y se almacena a temperatura ambiente.
- Placas de agar: Melt el agar LB en un horno de microondas y enfriar la solución a ~ 50 ° C. Vierta la solución en placas de Petri (Fisher Scientific) en el marco de una llama. En nuestra experiencia, 100 ml de agar LB que pueden producir placas de 5-6.
2. La construcción de RFD-EC1 y RFSS1 por plantilla mediada unión enzimática
RFD-EC1 (Fig. 2A) es la DNAzyme funciones. Consiste en la EC1 secuencia catalítica y la secuencia de sustrato FS1 (indicado por líneas negras y verde en la fig. 2A). RFSS1 (Fig. 2A) es una versión codificada de la RFD-EC1 donde está parcialmente la secuencia catalítica EC1 barajado en SS1 pero la porción FS1 permanece inalterado. RFD-EC1 y RFSS1 hicieron plantilla mediada ligación enzimática de la FS1 oligonucleótido con el oligonucleótido EC1 o SS1 en presencia de LT1 como la plantilla de la ligadura (véase el cuadro insertado en la fig. 2A). El procedimiento para llevar a cabo la reacción de ligación se proporciona a continuación.FS1 se obtuvo de Instalaciones Keck oligosíntesis la Universidad de Yale, desprotegió y se purificó mediante electroforesis en gel siguiendo un protocolo previamente establecido. 17-24 EC1, SS1 y LT1 fueron adquiridos de Integrated DNA Technologies y purificado mediante electroforesis en gel.
- Prepare una solución de 100 mM stock de FS1, EC1, SS1 y LT1 utilizando ddH 2 O. Almacenar a -20 ° C hasta su uso.
- Transferir 5 l de la solución madre FS1 a dos tubos de microcentrífuga 1,5 l marcados como tubo 1 y tubo 2. A cada tubo, añadir 38,5 l de ddH 2 O y luego 5 l de 10 x T4 polinucleótido quinasa (PNK) Un tampón de reacción (MBI Fermentas), que contiene 500 mM Tris-HCl (pH 7,6, 25 ° C), 100 mM MgCl2, 50 mM de DTT, 1.0 mM de espermidina. Mezcle cada solución con la pipeta (pipeta la solución de arriba a abajo unas cuantas veces).
- Añadir 1 l de ATP (100 mM; MBI Fermentas) y mezclar con la pipeta.
- Añadir 0,5 l de T4 polinucleótido quinasa (PNK; 10 unidades / l; Fermentas MBI) y mezclar con la pipeta. Incubar las mezclas de reacción a 37 ° C durante 30 min. Asegúrese de cubrir los tubos con papel de aluminio para reducir al mínimo photobleaching del fluoróforo.
- Sofocar la reacción por calentamiento a 90 ° C durante 5 min. Enfriar las mezclas de reacción a temperatura ambiente durante 10 minutos.
- Añadir 5 l de la EC1 y SS1 solución madre en el tubo 1 y tubo 2, respectivamente.
- Añadir 5 l de la solución madre LT1 a cada tubo, mezcla pipeteando. Calentar las mezclas de reacción a 90 ° C durante 1 min y enfriar a temperatura ambiente durante 10 minutos.
- Añadir 118 l de ddH 2 O y luego 20 l de 10 x tampón de T4 ADN ligasa (MBI Fermentas), que contiene 400 mM Tris-HCl (pH 7,8 a 25 ° C), 100 mM de MgCl 2, 100 mM de DTT, y 5 mM de ATP. Mezclar la solución con la pipeta.
- Añadir 2 l de ADN ligasa de T4 (5 unidades / l; MBI Fermentas) y mezclar por pipeteado. Incubar las mezclas de reacción a temperatura ambiente durante 1 h.
- Añadir 20 l de 3 M NaOAc (pH 7,0) a cada tubo, vórtice y girar hacia abajo. Añadir 500 l de etanol frío 100% a cada tubo, mezclar la solución con el vórtex y colocar los tubos en congelador a -20 ° C durante 30 min.
- Centrifugar la mezcla a 11.000 g durante 20 min a 4 ° C en una centrífuga refrigerada (Allegra X22-R, Beckman Coulter) y retirar con cuidado el sobrenadante con la pipeta.
- Secar el sedimento de ADN utilizando un concentrador de ADN (Savant Speedvac ADN, Thermo Scientific) durante 10 min.
- Volver a suspender las pastillas de ADN en 30 l de 1 × tampón de carga de gel (GLB), agitar brevemente y girar hacia abajo con una centrífuga de mesa (Minicentrifuge, VWR Scientific). Las muestras de DNA ligado está listo para su carga en un gel de dPAGE 10%.
3. Preparación de Gel dPAGE 10%
Los pasos siguientes se describen brevemente el aparato de electroforesis en gel y su puesta en marcha. Para mayores detalles sobre los aparatos, la configuración y manejo, por favor consulte to los protocolos publicados previamente 30,31.
- Lavar y secar dos placas de cristal, dos espaciadores 0,75 mm y un peine 16-bien. Montar placas de vidrio y los separadores con los clips y poner en posición horizontal sobre una superficie plana con placa de vidrio con muescas hacia arriba.
- Transferir 40 ml de mezcla dPAGE 10% en un vaso de precipitados de 150 ml. Añadir 40 l de tetrametiletilendiamina (TEMED; Bioshop Canadá), 400 l de 10% de APS, y mezclar por agitación con una pipeta.
- Vierta la mezcla con cuidado entre las placas y de inmediato insertar el peine. Dejar que la mezcla a polimerizar durante 10 a 20 min. La polimerización puede ser confirmado por el control de la mezcla de gel residual que queda en el vaso de precipitados.
- Una vez polimerizado, retirar el peine suavemente y enjuagar los pocillos con ddH 2 O para eliminar la solución de gel residual en los pocillos.
- Montar las placas en el aparato de electroforesis en gel con la placa sin muesca rectangular, mirando hacia fuera y colocar una placa de metal detrás de la placa dentada. El uso de la pl de metalcomió ayuda a prevenir el sobrecalentamiento, que puede romper las placas de vidrio.
- Añadir 1 x TBE a las cámaras superior e inferior del aparato. Ver para asegurar que los pozos se llenan con el tampón y el borde inferior del gel se sumergió en el buffer.
- Aplicar un 40 mA (o 750 V) actual y anterior-una duración de 10 a 15 min.
4. La purificación de Ligated RFD-EC1 y RFSS1 por Gel dPAGE 10%
- Después de la etapa de 3,7, enjuague pozos con 1 x TBE utilizando una jeringa y una aguja.
- Cargar la mezcla de ligación de RFD-EC1 y que de RFSS1 (desde 2,13) en 2 pocillos (uno para cada uno) utilizando una pipeta y puntas de gel de carga (DiaMed). Aplicar un 40 mA (750 V) corriente abajo hasta que el colorante (azul de bromofenol) es de aproximadamente 5 cm por encima del borde inferior de las placas.
- Retire las placas de vidrio del aparato de funcionamiento de gel, se acueste en una mesa de trabajo plana y retire con cuidado los separadores.
- Retirar con cuidado las placas de vidrio superior del gel y seenvolver el gel con una envoltura de plástico (tratar de evitar las arrugas y el plegamiento de los abrigos en el gel).
- Los productos ligados pueden ser visualizados, ya sea por sombreado UV (260 nm) o mediante transiluminación (360 nm), que producirá una banda de ADN visibles unos pocos centímetros por encima de EC1 o SS1 (100 pmol de EC1 o SS1 se puede utilizar como un marcador y cargado en un pozo en el paso de 4,2). Marque las bandas de ADN deseados con un marcador.
- Impuestos Especiales de la banda de ADN con hojas de afeitar estériles, cortar el gel en pequeños trozos y la transferencia a un nuevo tubo de microcentrífuga de 1,5 ml.
- Triturar las piezas de gel en el tubo de microcentrífuga estéril utilizando una punta de la pipeta (200 l tamaño de la punta).
- Añadir 500 l buffer de elución de ADN a cada tubo y cúbralos con papel de aluminio para proteger a los fluoróforos de la luz. Vortex las muestras durante 10 minutos.
- Centrifugar las muestras a 11.000 g durante 4 min a 4 ° C en una centrífuga refrigerada (Allegra X22-R, Beckman Coulter) y transferir cuidadosamente 350 l de la supernatant a un nuevo tubo de microcentrífuga 1,5 ml (si es necesario, una segunda elución se puede hacer con 350 l de tampón de elución fresco durante otros 10 minutos).
- Añadir 35 l (0,1 x del volumen de muestra) de acetato sódico 3 M (NaOAc, pH 7,0) a cada tubo, mezclar mediante agitación y girar hacia abajo. Añadir 900 uL de etanol frío 100% a cada tubo. Mezclar cada muestra por agitación a mano el tubo durante unos pocos segundos. Colocar los tubos a -20 ° C durante al menos 1 hora.
- Centrifugar las muestras a 11.000 g durante 20 min a 4 ° C en una centrífuga refrigerada y quitar cuidadosamente el sobrenadante por pipeteado.
- Añadir 100 l de etanol frío al 70% y usarlo para enjuagar suavemente toda la pared interior del tubo. Re-centrifugar a 11.000 g durante 7 min a 4 ° C. Eliminar el sobrenadante y secar el sedimento usando el concentrador de ADN durante 10 min.
- Disolver los pellets de ADN en 100 l de ddH 2 O y vórtice. Determinar la concentración de ADN basado en la absorbancia UV a 260 nm. Conservar las muestras a -20° C hasta su uso.
5. Preparación de Bacterias
- El objetivo cepa bacteriana E. coli K12 (MG1655) y las cepas de control pertinentes, en primer lugar se sembraron en placas de agar LB de las existencias de glicerol. En virtud de una llama o dentro de una cabina de seguridad biológica, toque la acción bacteriana de glicerol con una punta de pipeta estéril y extenderla suavemente sobre la superficie de la placa para no dañar el agar LB.
- Invertir las placas con rayas y se incuba a 37 ° C durante 14 h. Después de la incubación, sellar todo el perímetro de las placas con Parafilm (Pechiney Plastic Packaging) y se almacena a 4 º C. Estas placas se pueden almacenar durante un máximo de 4 semanas.
6. La preparación de mezclas de crudo extracelulares (CEM)
- Dispensar 2 ml de LB en tubos de cultivo estériles 14 mL (BD Falcon) utilizando una pistola de pipeta (Corning).
- Usando una punta de pipeta estéril, recoger una sola colonia de una placa de agar preparado en la etapa de 5,2 y la inserta en corriente alternaultura tubo. Colocar los tubos en una incubadora (New Brunswick Scientific) a 37 ° C y se agita a 250 rpm durante 14 h.
- 1% reinoculación cultura: Aplique 2 ml de LB fresco en 14 ml tubos de cultivo, y la espiga con 20 l de cultivo bacteriano preparado en el paso 6.2. Incubar los tubos a 37 ° C con agitación a 250 rpm hasta que cada solución bacteriana alcanza una DO 600 (densidad óptica medida a 600 nm) de aproximadamente 1. Para medir el diámetro exterior de 600, transferir 1 ml de cada cultura en una cubeta desechable y la absorbancia medida a 600 nm con un espectrofotómetro UV (Genesys UV 10, Thermo Scientific).
- Transferir 1 ml de cada cultivo a un nuevo tubo de microcentrífuga 1,5 ml y precipitar las células por centrifugación a 11.000 g durante 5 min a temperatura ambiente.
- Transferir el sobrenadante a un nuevo tubo de microcentrífuga de 1,5 ml y almacenar a -20 ° C si no se utiliza inmediatamente.
7. Detección mediante espectrofotómetro de fluorescencia
- Encienda el espectrofotómetro de fluorescencia (Cary Eclipse de Varian Inc.) y configurar los parámetros de adquisición de datos con excitación a 488 nm y emisión a 520 nm. Las lecturas pueden ser tomadas cada minuto durante 1 h.
- Lavar 3 cubetas de cristal de cuarzo (Varian Cary) con ddH2O, seguido de etanol al 100%. Seque las cubetas mediante el parpadeo de gas nitrógeno. Cubetas Discográfica C1 (control 1), C2 (Control 2) y T (prueba).
- Transferir 24 l de ddH2O a C1 y 24 L de la CEM-CE a C2 y T. Añadir 25 l de 2 x RB a cada cubeta y el lugar en el espectrofotómetro de fluorescencia. Comience a recoger los datos de fluorescencia de los primeros 5 minutos.
- Añadir 1 l de RFSS1 (de una solución 5 mM de reserva) a C2 y 1 l de RFD-EC1 (a partir de una solución 5 mM de reserva) a T y C1. Mezcle cada solución con la pipeta. Esto debe ser cuidadosamente iniciado de manera que las lecturas de fluorescencia no se interrumpe. Dejar que la reacción continúe durante el resto del tiempo de adquisición de 1 h.
- Ahorrarlos datos en formato de archivo de Excel, transferir los datos a un ordenador personal y los datos del proceso para crear una imagen gráfica.
8. La detección por electroforesis en gel
Las mezclas de reacción mismos preparados en el paso 7,4 puede ser utilizado para el análisis por electroforesis en gel; alternativamente nuevas reacciones se pueden preparar de manera similar y se incubaron en tubos de 1,5 ml de microcentrífuga. En cualquier caso:
- Reacciones interferentes (después de 1 h) mediante la adición de 5 l de 3 M NaOAc y 125 l de etanol al 100%. Mezclar cada solución con el vórtex y colocar los tubos en congelador a -20 ° C durante 1 h.
- Centrifugar las mezclas de reacción a 11.000 g durante 20 min a 4 ° C y retirar cuidadosamente el sobrenadante por pipeteado.
- Secar los gránulos utilizando el concentrador de ADN durante 10 min.
- Volver a suspender las pastillas en 20 l de 1 × GLB con una breve agitación. Gira por tubos muy brevemente con una centrífuga de mesa (Minicentrifuge, VWR Scientific). Estas muestras se leeny para la carga en un gel dPAGE.
- Preparar un gel de dPAGE 10% como se describe en 3.1-3.7. Cargar las muestras de reacción (del paso 8.4) en los pocillos con una pipeta y puntas de gel de carga (DiaMed). Aplicar un 40 mA (750 V) corriente abajo hasta que el colorante (azul de bromofenol) es de aproximadamente 5 cm por encima del borde inferior de las placas.
- Retire las placas de vidrio y lavar abundantemente con agua del grifo para eliminar las piezas de gel. Limpie las placas con un Kimwipe (Kimberly-Clark Professional).
- Busque en la placa de gel de fluorescencia utilizando un escáner Typhoon (Tifón 9200, el modo Variable, GE Healthcare). Analizar los datos utilizando el software de ImageQuant (Molecular Dynamics).
9. La especificidad de detección de
- Para el ensayo de especificidad bacteriana, los mismos procedimientos descritos anteriormente para el cultivo de E. coli, la preparación de su CEM y la realización de un ensayo de reacción de escisión se puede realizar para un número de diferentes cepas bacterianas tales como B. subtilis, P. Peli, Y. ruckeri, L. planturum, P. acidilactici (que se muestra en la fig. 3A).
10. La detección de los organismos unicelulares
Preparar un 1 ml E. acciones coli glicerol de 2 UFC / ml (UFC: unidades formadoras de colonias) por dilución en serie y confirmar la concentración de UFC en placas 5 Esta población debe contener 0,2 UFC/100 mL.. Almacenar a -80 ° C hasta su uso.
- Preparar 10 tubos de cultivo que contienen 2 ml de LB.
- Se inoculan cada cultivo con 100 l de 2 stock de glicerol UFC / ml y se incuba a 37 ° C con agitación a 250 rpm. El uso de la población entera de glicerol (1 ml) debe producir cultivos de 10.
- Cosecha 300 l de cada tubo de cultivo inoculado en los puntos de tiempo siguientes: 4, 8, 12, 16 y 24 h. Deje el cultivo restante a crecer durante 24 horas.
- Medir el diámetro exterior 600 y precipitar las células por centrifugación a 11.000 g durante 5 min.
- Traslado de los CEM a nuevos tubos de 1,5 mL microcentrífuga y almacenar a -20 + Dpor ejemplo, C hasta su uso.
Nota: Dado que puede ser no detectables OD 600 para las muestras recogidas en tiempo de los puntos 4, 8 y 12, permiten el cultivo restante para crecer durante 24 h con el fin de identificar los cultivos que contienen bacterias (determinado por la turbidez y la medición OD). Sólo uno o dos de los cultivos de 10 pueden contener E. E. después de la inoculación y los tubos restantes no contiene ninguna celda. - Utilice los CEM se recuperaron de cultivos positivos (almacenada a -20 ° C) en los intervalos de tiempo designados para preparar las reacciones de escisión con la RFD-EC1, y luego analizar las mezclas de reacción por electroforesis en gel de dPAGE como se describe en la Sección 8.
11. Concepto y Representante Resultados
El concepto de la explotación de un DNAzyme ARN-escindir fluorescente (RDF) para la detección bacteriana se ilustra en la figura. 1. La RFD rompe un ADN quimérico / sustrato de ARN en un vínculo único ARN (azul R) flanqueada por dos nucleótidos marcadoscon un fluoróforo (F) y un extintor (Q), respectivamente. Como una bacteria de interés (tales como E. coli) crece en medios de comunicación, se dejará una mezcla bruta extracelular (CEM). Este CEM como un todo se utiliza entonces en un experimento en selección in vitro para obtener un RFD que responda específicamente a la CEM; presumiblemente el RFD interactúa con una molécula específica (púrpura estrella) en el CEM que es una molécula de la firma de la bacteria. Cuando el CEM se añade a la solución de reacción que contiene el RFD, se dispara la actividad de ARN-escisión de la RFD. El evento escisión separa F de Q, resultando en una señal fluorescente que puede ser detectado, ya sea usando un fluorímetro o por electroforesis en gel.
La validación experimental del concepto anterior se realizó con el CEM de E. E. (CEM-CE). Se obtuvieron 3 moléculas RFD a través de selección in vitro, y el más eficiente fue designado como RFD-EC1 (Fig. 2A). 5 We probado la actividad de escisión de la RFD-EC1 (junto con una secuencia mutante llamado RFSS1) en respuesta a CEM-CE. Tanto RFD-EC1 y RFSS1 se prepararon por ligación enzimática de las porciones DNAzyme al sustrato FS1 (todas las secuencias se muestran en la fig. 2A). En el experimento de medición de fluorescencia (Fig. 2B), CEM-CE se incubó solo durante 5 min, seguido por la adición de RFD-EC1 o RFSS1, y por incubación adicional durante 55 min más. La intensidad de la fluorescencia de la solución se lee continuamente cada minuto y los datos se utilizó para calcular la fluorescencia relativa (RF; calcula como la relación de la intensidad de fluorescencia en el tiempo t vs la intensidad de fluorescencia a tiempo 0). Los valores de RF vs el tiempo de incubación se representan como la fig. 2B. Se encontró que la RFD-EC1 producido un alto nivel de señal de fluorescencia tras la adición de CEM-CE; en marcado contraste, RFSS1 no producir una señal fuerte fluorescencia. Así, el fu fluorescencia productoranction de la RFD-EC1 al ponerse en contacto CEM-CE es específica de la secuencia.
Con el fin de verificar que los aumentos de fluorescencia observados son debido a la escisión del enlace ARN, se analizaron mezclas de reacción por dPAGE. La escisión del RFD-EC1 se espera que genere dos fragmentos de ADN, un fragmento 5 'de retención del fluoróforo y un fragmento 3' de retención del extintor. Sólo uncleaved RFD-EC1 (unclv) y el fragmento 5 '(CLV) puede ser detectada por fluorescencia de imagen. El resultado dPAGE se muestra en la fig. 2C revela que la mezcla de reacción de RFD-EC1 y CEM CE-hecho producido el producto de escisión esperado, mientras que la mezcla RFSS1/CEM-EC no lo hizo.
La especificidad de la RFD-EC1 fue examinada usando CEM recogidas de varios otros gram positivas y gram negativas y los datos se muestra en la figura. 3A. Sólo la muestra que contiene CEM-CE (azul curva) produjo un aumento de la fluorescencia. La falta de reactividad cruzada con CEMde las otras bacterias indica que la RFD-EC1 es altamente selectivo para E. coli.
También se examinó el tiempo necesario para el cultivo de un solo E. E. celda con el fin de producir suficiente CEM que puede inducir la escisión de la RFD-EC1. Para este experimento, una E. muestra coli que contiene definido UFC (unidades formadoras de colonias) se diluyó adecuadamente para conseguir la concentración de 1 UFC / ml. Esto fue seguido por mezcla de 100 l de la muestra bacteriana diluida con medios de cultivo y el cultivo de ella para 4, 8, 12, 16 y 24 h. CEM se recogieron entonces para cada punto de tiempo y se ensayaron para inducir la actividad de escisión de la RFD-EC1. El resultado dPAGE se muestra en la fig. 3B indica que un tiempo de cultivo de 12 h se necesita.
Es importante señalar que el aumento inicial de pequeña señal observada en las mediciones de fluorescencia después de la adición de RFSS1 secuencia (como un control negativo) a CEM-CE (Fig. 2B; rojo Curve) o RFD-EC1 a otros CEM bacterianas (Fig. 3B; todas las curvas excepto el azul) se atribuye a la fluorescencia intrínseca del módulo FRQ (debido a la incompleta temple de F por Q). Por lo tanto, se espera que la adición de F y Q-etiquetados secuencias produciría un aumento de fluorescencia inicial. Sin embargo, sólo mezclas RFD-EC1/CEM-EC son capaces de producir un alto nivel de fluorescencia con el tiempo.

Figura 1. Esquema del ARN del clivaje de fluorescentes DNAzyme (RFD) de la sonda fluorescente que al entrar en contacto con la mezcla de crudo extracelular (CEM), producida por las células bacterianas específicas de interés. La RFD escinde un ADN quimérico / RNA substrato en un ligamiento solitario ARN (azul R) flanqueada por dos nucleótidos marcados con fluorocromo (F) y un extintor (Q), respectivamente. Antes de la división de reacción, el nivel de fluorescencia de la RFD es mínimo debido a la proximidad cercanamidad de F y P. Al escisión, Q se aparta de F, y como resultado, una señal fuerte fluorescencia se produce.
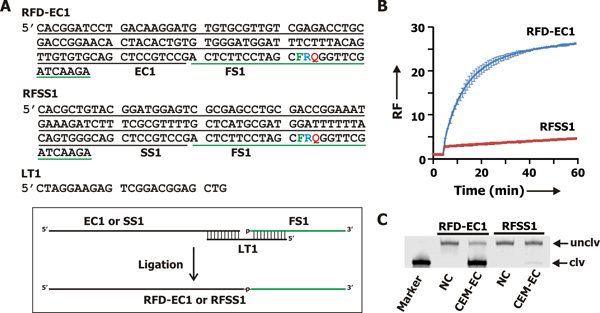
Figura 2. El E. coli sensible a RFD. (A) RFD-EC1 es la sonda DNAzyme que puede ser activado por el CEM-CE. RFSS1 es una secuencia codificada de RFD-EC1 utilizó como control. RFD-EC1 y RFSS1 fueron producidos por ligación con EC1 FS1 y SS1, respectivamente, en presencia de LT1 como la plantilla. F: fluoresceína-modificado desoxitimidina. Q: Dabcyl modificado desoxitimidina. R: ribonucleótido adenina. (B) de fluorescencia de señalización perfiles de RFD-EC1 y RFSS1 en presencia de CEM-CE. (C) dPAGE análisis de las mezclas de reacción de escisión en B (tiempo de reacción: 60 min). En la foto, una imagen de la fluorescencia del gel obtenido con dPAGE por el escáner Typhoon. Lane, Carolina del Norte: RFD-EC1 o RFSS1 en el tampón de reacción solo; Lane, CEM-EC: RFD-EC1 o RFSS1 en el buffer de reacción que contiene CEM-CE. Marcador: RFD-CE1 trató con NaOH 0,25 N, un procedimiento conocido para causar la escisión completa de ARN. unclv: uncleaved RFD-EC1. CLV: el fragmento de escisión que contiene el fluoróforo.
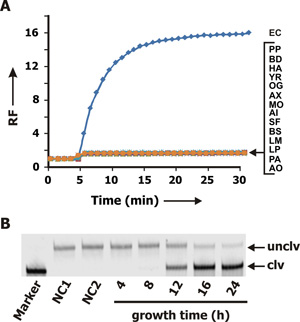
Figura 3. (A) de fluorescencia de señalización perfil de RFD-EC1 en CEM preparados a partir de diversas células bacterianas. CE: Escherichia coli-K12; PP: Pseudomonas peli; BD: Brevundimonas diminuta; HA: Hafnia alvei, YR: Yersinia ruckeri; OG: Ochrobactrum grignonese; AX: Achromobacter xylosoxidans, MO: Moraxella osloensis; AI: Acinetobacter lwoffi; SF: Serratia fonticola; BS: Bacillus subtilis, LM: Leuconostoc mesenteroides, LP: planturum Lactobacillus; PA: Pediococcus acidilactici, AO: Actinomyces orientalis. Cada muestra CEM se incubó durante 5 min seguido por la adición de RFD-EC1. (B) dPAGEanálisis de mezclas RFD-EC1/CEM-EC después de una reacción de 60 minutos. Carril NC1: RFD-EC1 en el tampón de reacción solo. Lane, NC2: RFD-EC1 en el buffer de reacción que contiene CEM-BS (el CEM preparado a partir de Bacillus subtilis). Los carriles marcados con 4, 8, 12, 16 y 24: RFD-EC1 en el tampón de reacción que contiene CEM-CE tomada a partir del cultivo bacteriano que contiene un único E. E. celular después de un período de crecimiento de 4, 8, 12, 16 y 24 h, respectivamente.
Discusión
La mayoría de los métodos de detección de bacterias comunes hoy en día son o bien lento (microbiana clásica) o técnicamente exigente (anticuerpos, PCR). Por lo tanto, creemos que la próxima generación de herramientas de detección debe atender a la velocidad y la simplicidad. Para este fin, se ha creado una ARN-escisión y la fluorescencia de señalización DNAzyme que puede utilizarse para desarrollar ensayos simples para informar la presencia de bacterias a través de la generación de una señal de fluorescencia. La sonda equipado DNAzyme, RFD-EC1, es activado por el CEM producidos durante el crecimiento de E. coli en medios de cultivo. Puesto que nuestro método utiliza crudo mezclas extracelulares de una bacteria como el objetivo de detección y evita la extracción de destino laborioso y pasos de amplificación, que puede ser utilizado para configurar muy simple ", mezclar y leer" tipo de ensayos para la detección bacteriana. El uso de nuestro DNAzyme no se limita al método de detección basado en fluorescencia. Por ejemplo, la detección colorimétrica utilizando el mismo sistema de ensayo DNAzyme cun ser diseñado usando un método ya se ha informado que explota amplificación círculo rodante en conjunción con un colorante orgánico. 32 Se prevé el uso de ADNzimas para la detección bacteriana como una atractiva vía para generar nuevos biosensores bacterianos con mayor simplicidad operativa.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
La financiación de este trabajo fue proporcionado por las Ciencias Naturales e Ingeniería de Investigación de Canadá (NSERC) y la Red de Médicos Centinelas de papel bioactivo.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo o modelo | |
| Agar | Bioshop Canadá | AGR003 | |
| Persulfato de amonio (APS) | Bioshop Canadá | AMP001 | |
| La acrilamida / bis-acrilamida (40%, 29:1) | Bioshop Canadá | ACR004 | |
| El ácido bórico | Bioshop Canadá | BOR001 | |
| Azul de bromofenol | Sigma-Aldrich | B8026 | |
| EDTA | EM Science | EXO539-1 | |
| De HCl | Sigma-Aldrich | 38281 | |
| HEPES | Bioshop Canadá | HEP001 | |
| Caldo LB | Sigma-Aldrich | L3022 | |
| MgCl 2 | EMDProductos Químicos | B10149-34 | |
| NaCl | Bioshop Canadá | SOD002 | |
| NaOAc | Merck Chemicals | SXO255-1 | |
| NaOH | Merck Chemicals | SXO590-1 | |
| SDS | Bioshop Canadá | SDS001 | |
| TEMED | Bioshop Canadá | TEM001 | |
| Tris-base | Bioshop Canadá | BST666 | |
| Tween 20 | Sigma-Aldrich | P9416 | |
| Urea | Bioshop Canadá | URE001 | |
| Xylenecyanol FF | Sigma-Aldrich | X4126 | |
| ADN concentrador | Thermo Scientific | Savant ADN SpeedVac 120 | |
| Millex filtro de la unidad | Millipore | SLGP033RS | |
| Gel puntas de carga | DiaMed | TEC200EX-K | |
| ImageQuant software | Dinámica Molecular | La versión 5.0 | |
| Kimwipes | Kimberly-Clark Professional | 34705 | |
| Mini vórtice | VWR | 58816-121 | |
| Parafilm | Pechiney Plastic Packaging | PM996 | |
| Placas de Petri | Fisher Scientific | Fisherbrand 08-757-12 | |
| Stripettor Plus (con una pipeta arma de fuego) | Corning | 07764714 | |
| Cubetas de cuarzo | Varian Inc. | 66-100216-00 | |
| Shaker / Incubadora | New Brunswick Scientific | Classic Series C24 | |
| El tifón escáner | GE Healthcare | 9200 modo Variable | |
| Centrifugar | Beckman Coulter | Allegra X22-R | |
| Espectrofotómetro UV | Thermo Scientific | GenesysUV 10 | |
| Espectrofotómetro de fluorescencia | Varian Inc. | Cary Eclipse |
Referencias
- Zourob, M., Elwary, S., Truner, A. . Principles of Bacterial Detection: Biosensors, Recognition Receptors and Microsystems. , (2008).
- Call, D. R. Challenges and Opportunities for Pathogen Detection Using DNA Microarrays. Crit. Rev. Microbiol. 31, 91-99 (2005).
- Lazcka, O., Campo, D. e. l., J, F., Muñoz, F. X. Pathogen detection: A perspective of traditional methods and biosensors. Biosens. Bioelectron. 22, 1205-1217 (2007).
- Velusamy, V. An overview of foodborne pathogen detection: In the perspective of biosensors. Biotechnol. Adv. 28, 232-254 (2010).
- Ali, M. M. Fluorogenic DNAzyme Probes as Bacterial Indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Liu, J., Cao, Z., Lu, Y. Functional Nucleic Acid Sensors. Chem. Rev. 109, 1948-1998 (2009).
- Li, Y., Lu, Y. . Functional Nucleic Acids for Analytical Applications. , (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Joyce, G. F. Forty Years of In Vitro Evolution. Angew. Chem. Int. Ed. 46, 6420-6436 (2007).
- Breaker, R. R., Joyce, G. F. A DNA enzyme that cleaves RNA. Chem. Biol. 1, 223-229 (1994).
- Cuenoud, B., Szostak, J. W. A DNA metalloenzyme with DNA ligase activity. Nature. 375, 611-614 (1995).
- Chinnapen, D. J., Sen, D. A deoxyribozyme that harnesses light to repair thymine dimers in DNA. Proc. Natl. Acad. Sci. U. S. A. 101, 65-69 (2004).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Silverman, S. K. DNA as a versatile chemical component for catalysis, encoding, and stereocontrol. Angew. Chem. Int. Ed. 49, 7180-7201 (2010).
- Mei, S. H. An efficient RNA-cleaving DNA enzyme that synchronizes catalysis with fluorescence signaling. J. Am. Chem. Soc. 125, 412-420 (2003).
- Liu, Z. Assemblage of signaling DNA enzymes with intriguing metal-ion specificities and pH dependences. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Kandadai, S. A., Li, Y. Characterization of a catalytically efficient acidic RNA-cleaving deoxyribozyme. Nucleic Acids Res. 33, 7164-7175 (2005).
- Rupcich, N. Quenching of fluorophore-labeled DNA oligonucleotides by divalent metal ions: implications for selection, design, and applications of signaling aptamers and signaling deoxyribozymes. J. Am. Chem. Soc. 128, 780-790 (2005).
- Shen, Y., Brennan, J. D., Li, Y. Characterizing the secondary structure and identifying functionally essential nucleotides of pH6DZ1, a fluorescence-signaling and RNA-cleaving deoxyribozyme. Biochemistry. 44, 12066-12076 (2005).
- Chiuman, W., Li, Y. Revitalization of six abandoned catalytic DNA species reveals a common three-way junction framework and diverse catalytic cores. J. Mol. Biol. 357, 748-754 (2006).
- Chiuman, W., Li, Y. Evolution of high-branching deoxyribozymes from a catalytic DNA with a three-way junction. Chem. Biol. 13, 1061-1069 (2006).
- Shen, Y. Catalysis and rational engineering of trans-acting pH6DZ1, an RNA-cleaving and fluorescence-signaling deoxyribozyme with a four-way junction structure. Chem BioChem. 7, 1343-1348 (2006).
- Ali, M. M., Kandadai, S. A., Li, Y. Characterization of pH3DZ1 - An RNA-cleaving deoxyribozyme with optimal activity at pH 3. Can. J. Chem. 85, 261-273 (2007).
- Chiuman, W., Li, Y. Efficient signaling platforms built from a small catalytic DNA and doubly labeled fluorogenic substrates. Nucleic Acids Res. 35, 401-405 (2007).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA MgZ based on a non-classic allosteric design. PLoS ONE. 2, e1224 (2007).
- Shen, Y. Entrapment of fluorescence signaling DNA enzymes in sol gel-derived materials for metal ion sensing. Anal. Chem. 79, 3494-3503 (2007).
- Kandadai, S. A. Characterization of an RNA-cleaving deoxyribozyme with optimal activity at pH 5. Biochemistry. 48, 7383-7391 (2009).
- Zhao, W., Brook, M. A., Li, Y. Periodic assembly of nanospecies on repetitive DNA sequences generated on gold nanoparticles by rolling circle amplification. Methods Mol. Biol. 474, 79-90 (2008).
- Navani, N. K., Mok, W. K., Li, Y. In vitro selection of protein-binding DNA aptamers as ligands for biosensing applications. Methods Mol. Biol. 504, 399-415 (2009).
- Ali, M. M., Li, Y. Colorimetric sensing by using allosteric-DNAzyme-coupled rolling circle amplification and a peptide nucleic acid-organic dye probe. Angew. Chem. Int. Ed. 48, 3512-3515 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados