Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La lignina Down-regulación de
En este artículo
Resumen
Una interferencia de ARN de doble cadena (dsRNAi) técnica se emplea para regular a la baja la coenzima A reductasa cinamoilo maíz gen (ZmCCR1) a un menor contenido de lignina de la planta. La lignina regulación a la baja de la pared celular se visualiza mediante análisis microscópicos y se cuantifica por el método de Klason. Se analizan los cambios de composición de la hemicelulosa y la celulosa cristalina.
Resumen
Para facilitar el uso de la biomasa lignocelulósica como un recurso bioenergía alternativa, durante los procesos de conversión biológica, se necesita una etapa de pretratamiento para abrir la estructura de la pared celular de la planta, el aumento de la accesibilidad de los hidratos de carbono de la pared celular. La lignina, un material polifenólico presente en muchos tipos de pared celular, se sabe que es un obstáculo significativo a la enzima de acceso. Reducción en el contenido de lignina a un nivel que no interfiere con la integridad y la defensa del sistema estructural de la planta puede ser un paso importante para reducir los costes de la producción de bioetanol. En este estudio, hemos genéticamente las reguladas uno de los genes relacionados con la biosíntesis de lignina, cinamoil-CoA reductasa (ZmCCR1) a través de una técnica de interferencia de ARN de doble cadena. El constructo ZmCCR1_RNAi se integró en el genoma del maíz utilizando el método de bombardeo de partículas. Plantas de maíz transgénicas crecieron normalmente en comparación con las plantas de control de tipo salvaje y sin enterfering con el crecimiento de la biomasa o los mecanismos de defensa, con la excepción de la visualización de color marrón-coloración en plantas transgénicas hoja mediados de los nervios, las cáscaras y tallos. Los análisis microscópicos, en conjunción con el ensayo histológico, revelaron que las fibras de esclerénquima de hojas se apretó pero la estructura y el tamaño de otros componentes principales del sistema vascular no fue alterada. El contenido de lignina en el maíz transgénico se redujo en un 7-8,7%, se aumentó el contenido de celulosa cristalina en respuesta a la reducción de la lignina, hemicelulosas y se mantuvo sin cambios. Los análisis pueden indicar que el flujo de carbono podría haber sido desplazado de la biosíntesis de la lignina para la biosíntesis de la celulosa. Este artículo delinea los procedimientos utilizados para regular a la baja el contenido de lignina en el maíz a través de la tecnología del RNAi, y los análisis de la composición de la pared celular utilizado para verificar el efecto de las modificaciones en la estructura de la pared celular.
Introducción
La producción de biocombustibles a partir de biomasa lignocelulósica es altamente deseable debido a su abundancia actual en los EE.UU. 1, y en el caso de la cosecha sostenible de los residuos agrícolas y forestales, la capacidad de no competir directamente por las tierras de cultivo utilizado para la alimentación animal y la producción de piensos. Sin embargo, a diferencia de grano de maíz, que es la principal fuente de biocombustible se está generando en los EE.UU., los materiales lignocelulósicos son significativamente más complejo y difícil de romper. Además de la hidratos de carbono de cadena larga, celulosa y hemicelulosa, que son las principales fuentes de azúcares durante la fermentación de materiales lignocelulósicos, muchos tipos de paredes de células vegetales también contienen lignina, un polímero de fenilpropanoides que proporciona fuerza, la defensa contra el ataque de patógenos, y la hidrofobicidad a las paredes celulares. Mientras que es necesario para el crecimiento vegetal y la supervivencia, la lignina también presenta una barrera significativa para la conversión enzimática con éxito de la celulosa y hemicelluperder a los azúcares solubles. Los materiales con alto contenido de lignina son generalmente materiales menos deseables, tanto para el biocombustible (a través de las vías de conversión biológica) y las industrias de pulpa y papel, debido a los impactos negativos sobre las características de procesamiento y la calidad del producto. Por lo tanto, la manipulación genética de los materiales vegetales para la reducción de la lignina en un nivel que no interfiera con la resistencia estructural de los cultivos y los sistemas de defensa podría ser importante para la reducción de los costos de producción, tanto para el biocombustible lignocelulósico y las industrias de pulpa y papel.
En el maíz (Zea mays), la lignina es covalentemente reticulado para la hemicelulosa en la pared celular primaria a través de ferulato y puentes diferulato 2. El complejo lignina-hemicelulosa se une a las microfibrillas de celulosa a través de enlaces de hidrógeno, formando una matriz compleja que confiere la integridad y la fuerza a la pared celular secundaria. La resistencia mecánica de las paredes celulares de la planta está determinada en gran medida por el tipo de Lignin subunidades 3-5. En estudios anteriores, la alteración de las proporciones de las subunidades de lignina ha mostrado ninguna tendencia clara en la digestibilidad enzimática 6-11. Sin embargo, las reducciones en el contenido de lignina en general, muestran una mejora en las conversiones de 12,13 y pueden ser una clave para el aumento de la digestibilidad de la materia vegetal por las enzimas hidrolíticas incluyendo endocellulases, celobiohidrolasas, y β-glucosidasas 14.
La ingeniería genética de regular el nivel de expresión de las transcripciones ha sido ampliamente practicada para mejorar características de los cultivos. Las técnicas avanzadas, incluyendo anti-sentido 15 y cosupresión 16 tecnologías, permiten efectiva la baja regulación de los genes diana. Completar gen knock-out también se ha conseguido utilizando construcciones de genes que codifican ARN intrón-empalmado con una estructura de horquilla 17. Además, una interferencia de ARN de doble cadena técnica (dsRNAi), es decir, un medio de expresión génica potente y eficazTor que funciona por cualquiera de focalización degradación de transcripción o traducción represión, proporciona un potente medio para inducir una amplia gama de efectos de supresión en el ARNm diana 18. Técnicas de silenciamiento génico muestran varias limitaciones. Estas técnicas no regulan con precisión el nivel de la transcripción y que podría causar efectos de silenciamiento inesperadas en otros genes homólogos.
En este método, hemos empleado el bombardeo de partículas para llevar a la dsRNAi construye en el genoma de maíz. Hasta la fecha, una gran variedad de especies de plantas se han transformado con éxito utilizando bombardeo de partículas, transformación mediada por Agrobacterium, electroporación, y los métodos de microinyección. En la transformación genética del maíz, el método de bombardeo de partículas es ventajoso con respecto a todos los otros métodos, ya que es el más eficiente. El bombardeo de partículas no depende de las bacterias, por lo que el método es libre de limitaciones biológicas, tales como el tamaño de los genes, especies de gen oigin, o el genotipo de la planta. El sistema de suministro de transgén física permite ADN de alto peso molecular y múltiples genes a ser introducidos en los genomas de plantas y en ciertos casos en los cloroplastos en alta eficiencia de transformación 19. La reducción de la lignina en el sistema vascular de la hoja de nervadura puede ser visualizado a través de microscopía electrónica de barrido (SEM) que es beneficioso para el examen de la topografía y la composición de las muestras.
En las plantas de maíz, dos de la reductasa de cinamoílo-CoA reductasa (ZmCCR1: X98083 y ZmCCR2: Y15069) se encontraron genes en el genoma del maíz 20. Cinamoil-CoA reductasa cataliza la conversión de los ésteres hidroxicinamoil-CoA reductasa en aldehídos de cinamilo. Elegimos el gen ZmCCR1 para regular a la baja esta enzima debido a que el gen se expresa en todos los tejidos lignifying. Los 523 nucleótidos en el extremo 3 'del gen ZmCCR1 se eligieron para un dsRNAi construir porque las secuencias parecían sermás diversa en comparación con los de ZmCCR2. Por lo tanto, la construcción dsRNAi ataría precisamente sólo para ZmCCR1, evitando fuera del objetivo silenciar a 21. Una construcción ZmCCR1_RNAi fue diseñado en el sistema de expresión citoplásmica ImpactVector1.1-tag (IV 1,1) que contiene el promotor específico de tejido verde, ribulosa-1, carboxilasa oxigenasa 5-bifosfato-carboxilasa/oxigenasa (RuBisCO).
Para estudiar los efectos de la dsRNAi construir en plantas transgénicas, se cuantificó el contenido de lignina. La medición de lignina Klason (insoluble en ácido) es conocido por ser más precisa en comparación con los métodos de cuantificación de lignina detergente ácido que solubilizan algunos de la lignina 22. Por lo tanto, la lignina Klason se midió en tallos de maíz transgénicas. Este procedimiento consiste en una hidrólisis ácida de dos pasos que convierte los carbohidratos poliméricos solubles en monosacáridos 23. A continuación, la biomasa hidrolizada se fraccionó en ácido Materi soluble e insolubleALS y la lignina insoluble en ácido se midió de acuerdo con estudios anteriores 23,24. Idealmente, el análisis de lignina debe incluir extracciones con agua y etanol antes de la etapa de hidrólisis, con el fin de eliminar los materiales solubles que pueden interferir con los resultados, y una combustión después de la hidrólisis del residuo de lignina para dar cuenta de cualquier ceniza presente en el residuo. Sin estos pasos, el contenido de lignina de la muestra podría ser inflado artificialmente. El método de integración global se presenta aquí, sin embargo, para nuestros experimentos hemos podido llevar a cabo estos dos pasos debido al pequeño volumen de material disponible para la prueba
También se analizaron otros dos componentes de la pared celular, la celulosa y la hemicelulosa en la lignina líneas de maíz transgénico-hacia abajo regulado. Se ha informado de que las plantas transgénicas que han sido regulados hacia abajo, ya sea en su fenilalanina amoníaco-liasa (PAL) 25, 4-cumarato: CoA ligasa (4CL) 26, o una de cinamilodeshidrogenasa l alcohol (CAD) 27 muestran un aumento de otros componentes estructurales de la pared celular. Como un primer paso en nuestros estudios, celulosa cristalina se midió utilizando el método Updegraff 28. Este método fue ideado originalmente para la determinación de la celulosa en un gran número de bacterias celulolíticas y hongos. Brevemente, las existencias de maíz molidos fueron tratados con Updegraff reactivo (ácido acético: ácido nítrico: agua) para eliminar la hemicelulosa, lignina, y xylosans. La celulosa cristalina fue completamente hidrolizada en glucosa a través de Saeman hidrólisis mediante la adición de H 2 SO 4. A continuación, la celulosa cristalina se ensayó usando el método colorimétrico de antrona 29. Para verificar si se cambiaron el contenido de hemicelulosa, los extractos de monosacáridos de tallos molidos se hidroliza usando ácido trifluoroacético, derivatizados utilizando el método del acetato de alditol y se analizaron por cromatografía de gases (GC) 30. Los procedimientos detallados para cel cristalinaanálisis de composición de contenido lulosa y de la matriz de polisacáridos se describen en Foster et al. (2010) 31.
A continuación, describimos los procedimientos utilizados para la lignina baja regulación en el maíz a través de una tecnología de RNAi, la transformación por bombardeo de partículas, y el análisis de la lignina para la deconstrucción acelerada de maíz de biomasa lignocelulósica en azúcares fermentables para los biocombustibles.
Protocolo
1. Preparación de dsRNAi construcciones utilizadas para el descenso de regulación de ZmCCR1
- Primers específicos de genes de diseño que incluyen sitios de enzimas de restricción necesarios para hacer una dsRNAi construir a ronda el gen ZmCCR1. Dos conjuntos de cebadores fueron diseñados para amplificar dos segmentos de fragmentos de ADNc ZmCCR1:. Un fragmento de 523 pb desde el nucleótido 748 hasta 1271, y un fragmento de 285 pb desde el nucleótido 986 hasta 1271 El ADNc ZmCCR1 fue proporcionada por el Instituto del Genoma de Arizona (AGI). Más detalles se describen en la Figura 1.
- Amplificar el fragmento grande por reacción en cadena de la polimerasa (PCR) de la plantilla de cDNA ZmCCR1 utilizando los ZmCCR1_748F_BglII cebadores (5'-AGATCTACATCCTCAAGTACCTGGAC-3 ') y ZmCCR1_1271R_NcoI (5'-CCATGGTTTACACAGCAGGGGAAGGT-3'). Amplificar el fragmento más pequeño (285 pb) utilizando los cebadores ZmCCR1_986F_BglII (5'-AGATCTGGAAGCAGCCGTACAAGTTC-3 ') y un ZmCCR1_1271R_SacI (5R17;-GAGCTCTTTACACAGCAGGGGAAGGT-3 ').
- Individualmente ligar los fragmentos en pGEM-T Easy siguiendo las instrucciones del fabricante.
- Realizar Mini-Prep de aislamiento de ADN plásmido de los transformantes individuales, cada uno que contiene el pGEM-T construye usando un kit de plásmido mini-preparación comercial.
- Digerir tanto el pGEM-T :: ZmCCR1 (523 pb) y ImpactVector (IV) -1,1 (vector de expresión citoplasma) con tanto BglII y NcoI.
- Se liga el fragmento grande se purificó en gel ZmCCR1 digeridos (523 pb) en el gel digerido IV-1.1 purificada.
- Se digiere el pGEM-T :: ZmCCR1 (285 pb) y IV-1.1 :: ZmCCR1 (523 pb) tanto con BglII y SacI para insertar el pequeño fragmento en el IV-1.1 :: ZmCCR1 (523 pb).
- Se liga el fragmento pequeño de gel digerido ZmCCR1 purificado (285 pb) en el gel digerido IV-1.1 purificado :: ZmCCR1 (523 pb).
- Clonar tanto 523 pb y 285 pb fragmentaciónTS a IV-1.1 para hacer que el ZmCCR1 ARNi construir, que tiene una secuencia de repetición invertida 285 pb con un espaciador 238 pb en el medio de los fragmentos de repetición invertida (véase la Figura 1).
- Transferencia de esta construcción en Escherichia coli (E. coli), crecer y realizar un aislamiento de ADN plásmido tamaño-midi de preparación para obtener el plásmido de ADN suficiente para la transformación genética del maíz.
2. Maíz Transformación Genética
- Preparación de partículas de tungsteno
- Coloque 60 mg de perlas de tungsteno (M10) en un tubo de 1,5 ml y se lava con 1 ml de etanol al 70% por agitación rotatoria durante 2 min. Incubar durante 10 minutos a 23 ° C y centrifugar a 18.894 xg durante 2 min y descartar el sobrenadante.
- Lavar 3 veces con 1 ml de etanol 100%, centrifugando durante 2 min y descartar el sobrenadante. Añadir 1 ml de 50% de glicerol estéril para llevar la concentración de micropartículas de 60 mg / ml.
- Preparación de ADN para el bombardeo
- Plel as de los 50 l (3 mg) de perlas de tungsteno preparadas en glicerol al 50% en un tubo de 1,5 ml. Añadir 5 l (1 mg) de IV-1.1 :: plásmido de ADN ARNi ZmCCR1, 50 l de CaCl2 2,5 M, y 20 l de 0,1 M espermidina. Vórtice brevemente entre cada adición de los reactivos anteriores.
- Vortex el tungsteno-ADN del grano brevemente la mezcla y se centrifuga a 18.894 xg durante 30 segundos. Retirar el sobrenadante y resuspender los gránulos en 140 l de etanol al 70%. Retirar el líquido y desecharlo. Añadir 140 l de etanol al 100%. Retirar el líquido y desecharlo.
- Añadir 48 l de etanol al 100%. Utilizar inmediatamente o almacenar en hielo durante un máximo de 4 horas antes del bombardeo.
- Bombardeo
- Coloque una callos de maíz embriogénico 3-5 cm de diámetro Hi-II (proporcionado desde el Centro de transformación de maíz, de la Universidad del Estado de Iowa) en el medio de 100 mm placas de Petri que contienen medio N6OSM 32 (como osmotium) al menos 4 horas antes de la bombardeo.
- Prepare la PSD-1000/He dispositivo de partícula de suministro de acuerdo con las instrucciones del fabricante 33.
- Esterilizar pared de la cámara con etanol al 70%. Cargue el disco de ruptura de 650 psi estéril en tapa de retención estéril. Spread 5-6 l de la solución de M10-ADN sobre la superficie de un macroportador, brevemente seco. Macroportador carga y detener pantalla en microsoporte ensamblaje de lanzamiento.
- Coloque microsoporte ensamblaje de lanzamiento y los callos de maíz en la cámara a una distancia seleccionada de la pantalla de detención (L2 = 6 cm) y cierre la puerta. Acelerar en un vacío de 27 psi en contra de una pantalla de malla de alambre.
- Pulse el botón de disparo hasta ráfagas de disco de ruptura y de la presión de helio medidor llega a cero. Suelte el botón de disparo.
- Incubar los callos bombardeados en una placa de Petri que contiene N6OSM (medio osmótico) 32 durante 16 horas en la oscuridad a 27 ° C. Romper los callos en unos diez piezas y la transferencia a N6E (medio de inducción de callo) 32 en placas de Petri y se incuba durante 5 días in la oscuridad a 27 ° C.
- Selección
- Después de 5 días en N6E, transferir los callos en un medio N6S (medio de selección) 32. Subcultura todo callos en un medio de selección, cada 30 días durante 8-12 semanas sin alterar la estructura de los callos.
- Después de aproximadamente 8-10 semanas, los sectores de rápido crecimiento blancos crecerán fuera de la no proliferación y calli madre parcialmente necrótica. Impuestos especiales de los tejidos de rápido crecimiento blancas y la subcultura a medio de selección fresco (N6S) 32 y continuar incubar que el anterior.
- Regeneración
- Transferir la callos embrionarios blanco y de crecimiento rápido en medio de regeneración 32 y se incuba como anteriormente durante 1 semana. Cambie los callos embriogénicos regenerar a un período de 16 horas luz del día y 8 horas de oscuridad a 25-27 ° C
- Transferir la regeneración dispara en el medio de enraizamiento 32 en un tubo de ensayo de vidrio después de 3-4 semanas, continúe incubar que el anterior. Después r sustancialAparece el desarrollo OOT, lávese cuidadosamente las raíces bajo el agua del grifo, y luego trasplantar las plántulas de 4 "macetas con tierra. Cubra las ollas con bolsas de plástico para mantener la humedad. Después de 2 días hacen agujeros pequeños de las bolsas de plástico. Después de 5-6 días retirar las bolsas de plástico. Continuar incubar como anteriormente para otros 5-6 días.
- Invernadero
- Transfiera las plantas de semillero en macetas de 18 "con el suelo y mantener en sol de verano completo o la luz de efecto invernadero. Las plantas regeneradas iniciales se llaman T 0, mientras que las primeras semillas pertenecen a la generación T 1.
3. Ensayo histológico
- Fijar las de maíz hoja mediados de los nervios en 5 ml de formalina al 10% tamponada neutra.
- Proceso de vacío y se infiltran con parafina en un procesador de tejidos usando un procesador de tejidos.
- Incrustar los tejidos en parafina utilizando una estación de incrustación Histocentre III.
- Retire el exceso de parafina de los bordes de una vez bloqueks se enfrían.
- Muestra en la Sección 4-5 micras con un micrótomo usando un microtomo.
- Coloque las secciones en portaobjetos de microscopio y seca en una incubadora de 56 ° C durante 2-24 horas. Haga secciones estén totalmente adheridos a la diapositiva.
- Desparafinar secciones en dos cambios de xileno durante 5 min a 23 ° C.
- Hidrato se desliza a través de dos cambios de etanol al 100% durante 2 min y dos cambios de etanol 95% durante 2 min a 23 ° C.
- Enjuague las secciones en agua corriente durante 2 min.
- Teñir con un 0,05% de toluidina O azul durante 1-2 minutos y enjuagar brevemente con ddH 2 O.
- Coloque un cubreobjetos sobre las muestras con aceite de inmersión y visualización con microscopia de luz.
- Microscopía Electrónica de Barrido (SEM)
- Fijar los de maíz hoja mediados de los nervios-seccionadas transversal en 4% de glutaraldehído y 0,1 M de tampón de fosfato de sodio (pH 7,4) a 4 ° C durante 1-2 horas.
- Brevemente enjuague las muestras en la memoria intermedia, ellos se deshidrataron en unaserie de etanol (25%, 50%, 75%, y 95%) durante 10-15 min en cada gradación y etanol al 100% durante 10 min, 3x.
- Secar los deshidratados maíz hoja mediados de nervios transversales de sección en una secadora de punto crítico que utiliza dióxido de carbono líquido como fluido de transición.
- Montar las muestras secas en los talones de aluminio mediante fichas de carbono de alto vacío
- Cubra las hoja de maíz a mediados de los nervios montados en los talones de aluminio de oro (de aproximadamente 20 nm de espesor) en un dispositivo de recubrimiento por pulverización catódica purgado con gas argón.
- Examine las muestras recubiertas en un JEOL JSM-6400V (emisor de electrones hexaboruro lantano) microscopio electrónico de barrido.
- Las imágenes digitales fueron fotografiados utilizando un software Pro Analysis (versión 3.2).
4. Lignina Klason Medición
- Molino de las muestras a través de un tamiz de 2 mm.
- Utilizar un analizador de humedad para determinar el contenido de humedad de cada muestra y registrar el valor.
- Pesar aproximadamente 1,5 g de cada muestra y se registra la masa. EXtract las muestras usando agua para la primera extracción, seguido de etanol para la segunda extracción usando un extractor automatizado disolvente (3 ciclos por extracción, ~ 14 min por ciclo) o aparato de Soxhlet (8 horas por extracción). (Nota: Este paso elimina extractivos que pueden condensarse durante la hidrólisis ácida e interferir con la medición exacta de la lignina, lo que aumenta la aparente contenido de lignina Klason.)
- Secar las muestras extraídas a 45 º C durante la noche, y luego deje que se enfríen en el desecador y pesar de nuevo.
- Establecer una incubadora a 30 ° C. Mide 0,3 g de cada uno, muestra extraída seco en tubos con tapa de rosca de alta presión (por triplicado por muestra se recomienda) y registrar los pesos a 0,1 mg. Añadir 3 ml de 72% de H 2 SO 4 a cada tubo de presión.
- Mezclar la muestra utilizando una varilla de agitación de vidrio o teflón. Deje la varilla de agitación en el tubo hasta que el agua se añade después de la incubación.
- Colocar los viales en una incubadora en síT a 30 ° C y 150 rpm durante 60 min. Después de 1 hora añadir 84 ml de agua desionizada para diluir la concentración de ácido al 4% y mezclar con la varilla de agitación. Tenga cuidado de no dejar grandes cantidades de muestra en los lados del vial por encima de la línea de agua.
- Sellar herméticamente las trabas de todos los viales y colóquelos en un bastidor de metal o de grandes vasos. Autoclave a 121 ° C utilizando un ciclo de esterilización líquido durante 1 hora. Deje que se enfríen a temperatura ambiente antes de abrir.
- Pre-ash los crisoles filtrantes en un horno a 575 ° C durante al menos 4 horas. Permita que se enfríen los crisoles en un desecador durante al menos una hora.
- Vacío filtrar la solución de cada tubo a través de un crisol separado, utilizando un adaptador de caucho para asegurar el crisol. Utilice agua desionizada para enjuagar las partículas restantes del tubo.
- Secar el residuo de lignina a 105 ° C durante un mínimo de 4 horas. Anotar el peso del crisol seco y residuos.
- Si se utiliza un 575 ° C, preEl fuego de las muestras sobre un mechero de Bunsen hasta que no haya humo o ceniza y luego colocar en el horno durante 24 horas, o si se utiliza un horno programable, no pre-ash y utilizar el siguiente programa:
- Rampa de temperatura ambiente a 105 ° C y mantener durante 12 min.
- Rampa a 250 º C a 10 º C / min y mantener durante 30 min.
- Rampa a 575 ° C a 20 ° C / min y mantener durante al menos 180 minutos.
- Retire los crisoles del horno y enfriar en un desecador. Pesar el crisol y ceniza.
- Calcular el residuo insoluble en ácido mediante la siguiente ecuación:
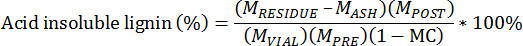
M PRE = Masa de la biomasa antes de extraer
M POSTE = Masa de la biomasa post-extraída
M VIAL = masa de la biomasa extraída añadió al vial
M RESIDUOS = Masa de resid crisol y ligninaue
M ASH = Masa del crisol y cenizas
MC = Contenido de humedad de la biomasa pre-extraído, respecto al peso total
5. Análisis de carbohidratos
- Realice la pared celular de carbohidratos análisis basado en el Foster et al. (2010) Protocolo de 31. En resumen, Preparar alcohol residuo insoluble de liofilizado material vegetal. A continuación, hidrolizar el material con ácido trifluoroacético y combinar los derivados de monosacáridos solubilizadas a sus acetatos de alditol correspondientes. Analizar estos derivados volátiles por cromatografía de gases (GC) conectado a un espectrómetro de masas cuádruple.
Resultados
Hemos demostrado una reducción en el contenido de lignina en las plantas de maíz a través de ARNi. El método de transformación por bombardeo de partículas produjo alrededor del 30% de eficiencia trnasformation. El silenciamiento de los genes de ZmCCR1 se observó consistentemente en generaciones T0-T2. Los transgénicos lignina reducida crecieron de manera similar a las plantas de maíz de tipo salvaje, excepto para la visualización de la coloración marrón en el medio de la hoja de la costilla, la cás...
Discusión
La accesibilidad de las celulasas microbianas para plantar polisacáridos de la pared celular depende en gran medida del grado en el que se asocian con polímeros fenólicos 23. La tasa de conversión de biomasa lignocelulósica en azúcar fermentable se correlaciona negativamente con el contenido de lignina depositado en las paredes celulares de las plantas secondadry. Esta correlación se atribuye a las propiedades físicas de la lignina como hidrofobicidad 24, la heterogeneidad química, y la au...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
La imagen microscópica se realizó a través de los servicios del Centro de la Universidad Estatal de Michigan de Microscopía Avanzada. Callos de maíz fue adquirido desde el Centro de Transformación de maíz de Iowa State University. Los autores desean agradecer a Jeffrey R. Weatherhead del Laboratorio de Investigación de Plantas MSU por su asistencia técnica en el análisis de carbohidratos. Esta investigación fue generosamente financiado por el Programa de Maíz de Marketing de Michigan (CMPM) y el Consorcio para la Investigación de Biotecnología Vegetal (CPBR).
Materiales
| Name | Company | Catalog Number | Comments |
| N6OSM (Osmotic medium) | Made in-house | ||
| N6E (Callus induction) | Made in-house | ||
| N6S media (Selection media) | Made in-house | ||
| Regeneration medium | Made in-house | ||
| Rooting medium | Made in-house | ||
| 10% Neutral buffered formalin (1 L) | Made in-house | ||
| Bio-Rad PSD-1000/He Particle Delivery device | Hercules, CA, United States | ||
| Zeiss PASCAL confocal laser scanning microscope | Carl Zeiss, Jena, Germany | For brightfield microscopy, the images were recorded using a Zeiss (Jena, Germany) PASCAL confocal laser scanning microscope with a 488 nm excitation mirror, a 560 nm emission filter, and a 505-530 nm emission filter. Image analysis was performed using Laser scanning microscope PASCAL LSM version 3.0 SP3 software. | |
| Excelsior ES Tissue Processor | Thermo Scientific, Pittsburgh, PA, United States | ||
| HistoCentre III Embedding Station | Thermo Scientific, Pittsburgh, PA, United States | ||
| Microtome Model Reichert 2030 | Reichert, Depew, NY, United States | ||
| Emscope Sputter Coater model SC 500 | Ashford, Kent, England | ||
| JEOL JSM-6400V Scanning Electron Microscope | JEOL Ltd., Tokyo, Japan | ||
| Fitzpatrick JT-6 Homoloid mill | Continental Process Systems, Inc., Westmont, IL | ||
| MA35 Moisture Analyzer | Sartorius | ||
| Critical point dryer, Balzers CPD | Leica Microsysstems Inc, Buffalo Grove, IL, United States | ||
| Screw-top high pressure tubes | Ace Glass, Vineland, NJ | #8648-27 | |
| Screw-top high pressure tube plugs | Ace Glass, Vineland, NJ | #5845-47 |
Referencias
- Ralph, J., Grabber, J. H., Hatfield, R. D. Lignin-ferulate cross-links in grasses - Active incorporation of ferulate polysaccharide esters into ryegrass lignins. Carbohydrate research. , 275-178 (1995).
- Park, S. -. H. . Expediting cellulosic biofuels agenda: Production of high value-low volume co-products and lignin down-regulation of bioenergy crops [Ph.D. thesis]. , (2011).
- Boerjan, W., Ralph, J., Baucher, M. Lignin biosynthesis. Annual review of plant biology. 54, 519-546 (2003).
- Gibson, L. J. The hierarchical structure and mechanics of plant materials. Journal of the Royal Society, Interface / the Royal Society. 9, 2749-2766 (2012).
- Dien, B. S., et al. Enhancing alfalfa conversion efficiencies for sugar recovery and ethanol production by altering lignin composition. Bioresource technology. , 102-6486 (2011).
- Fu, C. X., et al. Downregulation of Cinnamyl Alcohol Dehydrogenase (CAD) Leads to Improved Saccharification Efficiency in Switchgrass. Bioenerg Res. 4, 153-164 (2011).
- Grabber, J. H., Ralph, J., Hatfield, R. D., Quideau, S. p-hydroxyphenyl, guaiacyl, and syringyl lignins have similar inhibitory effects on wall degradability. Journal of agricultural and food chemistry. 45, 2530-2532 (1997).
- Li, X., et al. Lignin monomer composition affects Arabidopsis cell-wall degradability after liquid hot water pretreatment. Biotechnology for biofuels. 3, (2010).
- Mansfield, S. D., Kang, K. Y., Chapple, C. Designed for deconstruction--poplar trees altered in cell wall lignification improve the efficacy of bioethanol production. The New phytologist. 194, 91-101 (2012).
- Studer, M. H., et al. Lignin content in natural Populus variants affects sugar release. Proceedings of the National Academy of Sciences of the United States of America. 108, 6300-6305 (2011).
- Chen, F., Dixon, R. A. Lignin modification improves fermentable sugar yields for biofuel production. Nature. 25, 759-761 (2007).
- Ziebell, A., et al. Increase in 4-coumaryl alcohol units during lignification in alfalfa (Medicago sativa) alters the extractability and molecular weight of lignin. The Journal of biological chemistry. 285, 38961-38968 (2010).
- Park, S. -. H., et al. The quest for alternatives to microbial cellulase mix production: corn stover-produced heterologous multi-cellulases readily deconstruct lignocellulosic biomass into fermentable sugars. Journal of Chemical Technolog., and Biotechnology. 86, 633-641 (2011).
- Mol, J. N., et al. Regulation of plant gene expression by antisense RNA. FEBS letters. 268, 427-430 (1990).
- Adamo, A., et al. Transgene-mediated cosuppression and RNA interference enhance germ-line apoptosis in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 109, 3440-3445 (2012).
- Smith, N. A., et al. Total silencing by intron-spliced hairpin RNAs. Nature. 407, 319-320 (2000).
- Park, S. -. H., et al. Downregulation of Maize Cinnamoyl-Coenzyme A Reductase via RNA Interference Technology Causes Brown Midrib and Improves Ammonia Fiber Expansion-Pretreated Conversion into Fermentable Sugars for Biofuels. Crop Sci. 52, 2687-2701 (2012).
- Altpeter, F., et al. Particle bombardment and the genetic enhancement of crops: myths and realities. Mol Breeding. 15, 305-327 (2005).
- Pichon, M., Courbou, I., Beckert, M., Boudet, A. M., Grima-Pettenati, J. Cloning and characterization of two maize cDNAs encoding cinnamoyl-CoA reductase (CCR) and differential expression of the corresponding genes. Plant molecular biology. 38, 671-676 (1998).
- Mansoor, S., Amin, I., Hussain, M., Zafar, Y., Briddon, R. W. Engineering novel traits in plants through RNA interference. Trends in plant science. 11, 559-565 (2006).
- Hatfield, R. D., Jung, H. -. J. G., Ralph, J., Buxton, D. R., Weimer, P. J. A comparison of the insoluble residues produced by the Klason lignin and acid detergent lignin procedures. J Sci Food Agr. 65, 51-58 (1994).
- Sluiter, J. B., Ruiz, R. O., Scarlata, C. J., Sluiter, A. D., Templeton, D. W. Compositional analysis of lignocellulosic feedstocks. 1. Review and description of methods. Journal of agricultural and food chemistry. 58, 9043-9053 (2010).
- Sluiter, A., Hames, B., Ruiz, R., Scarlata, C., Sluiter, J., Templeton, D., Crocker, D. Determination of structural carbohydrates and lignin in biomass. Laboratory Analytic Procedure. , (2008).
- Bate, N. J., et al. Quantitative Relationship between Phenylalanine Ammonia-Lyase Levels and Phenylpropanoid Accumulation in Transgenic Tobacco Identifies a Rate-Determining Step in Natural Product Synthesis. Proceedings of the National Academy of Sciences of the United States of America. 91, 7608-7612 (1994).
- Hu, W. J., et al. Repression of lignin biosynthesis promotes cellulose accumulation and growth in transgenic trees. Nature. 17, 808-812 (1999).
- Lapierre, C., et al. Signatures of cinnamyl alcohol dehydrogenase deficiency in poplar lignins. Phytochemistry. 65, 313-321 (2004).
- Updegraff, D. M. Semimicro determination of cellulose in biological materials. Anal Biochem. 32, 420-424 (1969).
- Yemm, E. W., Willis, A. J. The estimation of carbohydrates in plant extracts by anthrone. The Biochemical journal. 57, 508-514 (1954).
- Filomena, A. P., Cherie, W., Geoffrey, B. F., Antony, B. Determining the polysaccharide composition of plant cell walls. Nature. 7, 1590-1607 (2012).
- Foster, C. E., Martin, T. M., Pauly, M. Comprehensive Compositional Analysis of Plant Cell Walls (Lignocellulosic biomass) Part II: Carbohydrates. J. Vis. Exp. (e1837), (2010).
- Department of Agronomy, Iowa State University. Particle bombardment of Hi II immature zygotic embryos and recovery of transgenic maize plants. , (2005).
- Cano-Delgado, A., Penfield, S., Smith, C., Catley, M., Bevan, M. Reduced cellulose synthesis invokes lignification and defense responses in Arabidopsis thaliana. The Plant journal : for cell and molecular biology. 34, 351-362 (2003).
- Boudet, A. M., Kajita, S., Grima-Pettenati, J., Goffner, D. Lignins and lignocellulosics: a better control of synthesis for new and improved uses. Trends in plant science. 8, 576-581 (2003).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados