Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Lignine régulation à la baisse de
Dans cet article
Résumé
Un double interférence ARN brin (dsRNAi) technique est employée pour réguler à la baisse la coenzyme maïs cinnamoyl A réductase (ZmCCR1) gène à faible teneur en lignine des plantes. La lignine régulation à la baisse à partir de la paroi cellulaire est visualisée par des analyses microscopiques et quantifiée par la méthode Klason. Changements dans la composition de l'hémicellulose et la cellulose cristalline sont analysés.
Résumé
Pour faciliter l'utilisation de la biomasse ligno-cellulosique en tant que ressource de bioénergie alternatif, pendant les processus de conversion biologique, une étape de prétraitement est nécessaire pour ouvrir la structure de la paroi cellulaire de la plante, en augmentant l'accessibilité des hydrates de carbone de la paroi cellulaire. La lignine, une matière polyphénolique présente dans de nombreux types de parois cellulaires, est connu pour être un obstacle important à l'accès de l'enzyme. Réduction de la teneur en lignine à un niveau qui n'interfère pas avec le système de l'intégrité structurelle et la défense de la plante peut être une étape importante pour réduire les coûts de la production de bioéthanol. Dans cette étude, nous avons génétiquement régulé à la baisse l'un des gènes liés à la biosynthèse de la lignine-,-cinnamoyl CoA réductase (ZmCCR1) par l'intermédiaire d'une technique d'interférence de l'ARN double brin. Le produit d'assemblage ZmCCR1_RNAi a été intégré dans le génome de maïs en utilisant le procédé de bombardement de particules. Plants de maïs transgéniques ont augmenté normalement par rapport à des plantes témoins de type sauvage sans enterfering à la croissance de la biomasse ou des mécanismes de défense, à l'exception de l'affichage de brun-coloration dans les plantes feuilles milieu des côtes transgéniques, les balles et les tiges. Les analyses microscopiques, en conjonction avec le test histologique, ont révélé que les fibres de sclérenchyme de feuilles ont été éclaircis mais la structure et la taille des autres principaux composants du système vasculaire n'a pas été modifiée. La teneur en lignine dans le maïs transgénique a été réduite de 7 à 8,7%, la teneur en cellulose cristalline a été augmenté en réponse à la réduction de la lignine, des hémicelluloses et est restée inchangée. Les analyses peuvent indiquer que le flux de carbone aurait été déplacé de la biosynthèse de la lignine de la cellulose biosynthèse. Cet article définit les procédures utilisées pour réguler à la baisse la teneur en lignine dans le maïs via la technologie ARNi, et la composition de la paroi cellulaire des analyses permet de vérifier l'effet des modifications sur la structure de la paroi cellulaire.
Introduction
La production de biocarburants à partir de biomasse lignocellulosique est hautement souhaitable en raison de son abondance actuelle aux États-Unis 1, et dans le cas de la récolte durable des résidus agricoles et forestiers, la capacité de ne pas concurrencer directement les terres cultivées utilisées pour l'alimentation animale et la production d'aliments. Cependant, contrairement à grains de maïs, qui est la principale source de biocarburant a généré aux Etats-Unis, les matériaux lignocellulosiques sont beaucoup plus complexes et difficiles à briser. En plus de la hydrates de carbone à longue chaîne, de la cellulose et de l'hémicellulose, qui sont les principales sources de sucres pendant la fermentation de matières ligno-cellulosiques, de nombreux types de parois de cellules végétales contiennent également de la lignine, un polymère phénylpropanoïdes qui fournit la force, à la défense contre une attaque pathogène, et l'hydrophobicité de parois cellulaires. Alors que nécessaire pour la croissance des plantes et de la survie, de la lignine présente également un obstacle important à la conversion enzymatique de la cellulose avec succès et hemicelluperdre de sucres solubles. Matériaux à haute teneur en lignine sont généralement des matériaux moins souhaitables à la fois pour le biocarburant (par des voies de conversion biologique) et les industries de pâtes et papiers en raison des impacts négatifs sur les caractéristiques de traitement et la qualité des produits. Par conséquent, la manipulation génétique du matériel végétal pour la réduction de la lignine à un niveau qui n'interfère pas avec la culture et la résistance structurelle des systèmes de défense pourrait être important pour la réduction des coûts de production à la fois pour le biocarburant lignocellulosique et les industries de pâtes et papiers.
Dans le maïs (Zea mays), de la lignine est réticulé de manière covalente à l'hémicellulose dans la paroi de la cellule primaire par l'intermédiaire de ponts férulate et diferulate 2. Le complexe lignine-hémicellulose se lie à des microfibrilles de cellulose à travers des liaisons hydrogène, la formation d'une matrice complexe qui confère une intégrité et une résistance à la paroi cellulaire secondaire. La résistance mécanique des parois cellulaires de la plante est largement déterminée par le type de lignin sous-unités 3-5. Dans des études antérieures, en modifiant les proportions de sous-unités de la lignine n'a montré aucune tendance claire sur la digestibilité enzymatique 6-11. Cependant, les réductions de la teneur en lignine présentent généralement une amélioration de la conversion et 12,13 peuvent être une clé pour augmenter la digestibilité de la matière végétale par des enzymes hydrolytiques incluant endocellulases, cellobiohydrolases, β-glucosidases et 14.
Le génie génétique pour réguler le niveau de transcrits d'expression a été largement pratiquée à améliorer les caractères des cultures. Des techniques de pointe, y compris les anti-sens 15 et co-suppression de 16 technologies, permettent une régulation efficace de gènes cibles. Gène complet knock-out ont également été réalisés en utilisant des constructions de gènes codant pour des ARN d'intron-épissé avec une structure en épingle à cheveux 17. Par ailleurs, un double brin d'ARN interférence (dsRNAi) de la technique, c'est à dire un support d'expression de gène efficace et puissantteur qui fonctionne soit en ciblant la dégradation de transcription ou de traduction répression, fournit un moyen puissant pour induire un large éventail d'effets de suppression sur l'ARNm cible 18. Techniques Gene silencing montrent plusieurs limites. Ces techniques ne règlent pas avec précision le niveau de la transcription et il pourrait provoquer des effets inattendus sur d'autres silencieux des gènes homologues.
Dans cette méthode, nous avons utilisé le bombardement de particules pour effectuer la dsRNAi construit dans le génome du maïs. À ce jour, une vaste gamme d'espèces de plantes ont été transformés avec succès en utilisant un bombardement de particules, Agrobacterium transformation médiation, l'électroporation, et les méthodes de micro-injection. Dans la transformation génétique du maïs, le procédé de bombardement de particules est avantageuse par rapport à toutes les autres méthodes car il est le plus efficace. le bombardement de particules ne dépend pas de bactéries, de sorte que le procédé est exempt de contraintes biologiques tels que la taille des espèces de gènes, le gène ouIgin, ou le génotype de la plante. Le système de délivrance d'un transgène physique permet à l'ADN de haut poids moléculaire et de multiples gènes à introduire dans le génome des plantes et dans certains cas dans les chloroplastes à haute efficacité de la transformation 19. La réduction de la lignine dans le système vasculaire de la nervure centrale des feuilles peut être visualisé par microscopie électronique à balayage (SEM) qui est bénéfique pour l'examen de la topographie et la composition des échantillons.
Dans les plants de maïs, de la réductase de deux cinnamoyl-CoA (ZmCCR1: X98083 et ZmCCR2: Y15069) gènes ont été trouvés dans le génome du maïs 20. Cinnamoyle-CoA reductase catalyse la conversion de l'ester hydroxycinnamoyl-CoA en aldéhydes cinnamyliques. Nous avons choisi le gène ZmCCR1 à réguler à la baisse cette enzyme parce que le gène est exprimé dans tous les tissus lignifying. Les 523 nucleotides à l'extrémité 3 'du gène ZmCCR1 ont été choisis pour la construction d'un dsRNAi parce que les séquences ont semblé êtreplus diversifiée par rapport à ceux de ZmCCR2. Ainsi, la construction dsRNAi lierait précisément qu'à ZmCCR1, évitant hors cible au silence 21. Produit d'assemblage ZmCCR1_RNAi a été conçu dans le système d'expression cytoplasmique ImpactVector1.1-tag (IV 1.1) contenant le vert promoteur spécifique de tissu, le ribulose-1, 5 carboxylase oxygénase-bisphosphate (RuBisCO).
Pour étudier les effets de la dsRNAi construire sur des plantes transgéniques, la teneur en lignine a été quantifiée. Le Klason (insolubles dans l'acide) mesure de la lignine est connu pour être plus précis par rapport à des méthodes de quantification de la lignine détergent acide qui solubilisent une partie de la lignine 22. Par conséquent, la lignine Klason a été mesurée dans les tiges de maïs transgéniques. Ce procédé consiste en une hydrolyse acide en deux étapes qui permet de convertir des glucides polymères solubles dans 23 monosaccharides. La biomasse hydrolysée a ensuite été fractionné en materi solubles et insolubles acidesal et la lignine insoluble de l'acide a été mesurée d'après des études antérieures 23,24. Idéalement, l'analyse de la lignine doit comprendre extractions avec de l'eau et de l'éthanol avant l'étape d'hydrolyse, afin d'éliminer les matières solubles qui peuvent interférer avec les résultats, et une combustion post-hydrolyse du résidu de lignine pour tenir compte de toutes les cendres présentes dans le résidu. Sans ces mesures, la teneur en lignine de l'échantillon pourrait être gonflé artificiellement. La méthode complète est présentée ici, mais pour nos expériences, nous n'avons pas pu effectuer ces deux étapes en raison du faible volume de matériel disponible pour les tests
Deux autres composants de la paroi cellulaire, la cellulose et l'hémicellulose ont également été analysés dans la lignine de lignes régulée à la baisse de maïs transgénique. Il a été rapporté que les plantes transgéniques qui ont été régulés à la baisse dans les deux leur phénylalanine ammoniac-lyase (PAL) 25, 4 coumarate: CoA ligase (4CL) 26, ou un cinnamyliquelcohol déshydrogénase (CAD) 27 montrent une augmentation des autres éléments structurels de la paroi cellulaire. Comme première étape dans nos études, la cellulose cristalline a été mesuré en utilisant la méthode Updegraff 28. Cette méthode a été initialement conçu pour la détermination de la cellulose dans un grand nombre de bactéries et de champignons cellulolytiques. En bref, les stocks de maïs broyées ont été traités avec Updegraff réactif (acide acétique: acide nitrique: l'eau) pour enlever l'hémicellulose, la lignine, et xylosans. La cellulose cristalline a été complètement hydrolysé en glucose via Saeman hydrolyse par addition de H 2 SO 4. La cellulose cristalline a ensuite été testée en utilisant la méthode colorimétrique à l'anthrone 29. Pour vérifier si le contenu d'hémicellulose ont été modifiées, les extraits de monosaccharides à partir de tiges broyées ont été hydrolysés à l'aide de l'acide trifluoroacétique, transformés en dérivés en utilisant la méthode de l'acétate d'alditol et ensuite analysés par chromatographie en phase gazeuse (GC) 30. Les procédures détaillées pour cel cristallineLes analyses de composition et de matrice contenu lulose polysaccharides sont décrits dans Foster et al. (2010) 31.
Ici, nous décrivons les méthodes utilisées pour la lignine régulation à la baisse dans le maïs par une technologie ARNi, particule transformation de bombardement, et l'analyse de la lignine pour la déconstruction accélérée de maïs biomasse lignocellulosique en sucres fermentescibles pour les biocarburants.
Protocole
1. Préparation de dsRNAi constructions utilisées pour la régulation à la baisse de ZmCCR1
- amorces spécifiques du gène de conception, y compris nécessaires sites d'enzymes de restriction pour faire un dsRNAi construire de knock-out du gène ZmCCR1. Deux jeux d'amorces ont été conçues pour amplifier deux segments de fragments de ZmCCR1 ADNc:. Un fragment de 523 pb du nucléotide 748 à 1271, et un pb fragment 285 de nucléotides 986-1271 Le ZmCCR1 ADNc a été fourni par l'Institut de génomique de l'Arizona (AGI). Plus de détails sont décrits dans la figure 1.
- Amplifier le grand fragment par réaction de polymérisation en chaîne (PCR) à partir du modèle ZmCCR1 ADNc en utilisant des amorces (5'-ZmCCR1_748F_BglII AGATCTACATCCTCAAGTACCTGGAC-3 ') et ZmCCR1_1271R_NcoI (5'-CCATGGTTTACACAGCAGGGGAAGGT-3'). Amplifier le plus petit fragment (285 pb) en utilisant les amorces (5'-ZmCCR1_986F_BglII AGATCTGGAAGCAGCCGTACAAGTTC-3 ') et une ZmCCR1_1271R_SacI (5R17;-GAGCTCTTTACACAGCAGGGGAAGGT-3 ').
- Ligaturer individuellement les fragments dans pGEM-T Easy en suivant les instructions du fabricant.
- Effectuer des mini-prep isolement d'ADN plasmidique à partir des transformants individuels, chacun contenant le vecteur pGEM-T construit en utilisant un mini-prep de plasmide kit commercial.
- Digérer la fois le pGEM-T :: ZmCCR1 (523 pb) et ImpactVector (IV) -1,1 (vecteur d'expression de cytoplasme) avec à la fois Ncol et BglII.
- Ligaturer le grand gel purifié ZmCCR1 fragment digéré (523 pb) dans le gel digéré IV-1.1 purifié.
- Digérer le vecteur pGEM-T :: ZmCCR1 (285 pb) et IV-1.1 :: ZmCCR1 (523 pb) à la fois avec BglII et Sacl afin d'insérer le petit fragment dans le IV-1.1 :: ZmCCR1 (523 pb).
- Ligaturer le petit gel digéré fragment ZmCCR1 purifié (285 pb) dans le gel digéré IV-1.1 purifié :: ZmCCR1 (523 pb).
- Cloner fois 523 pb et 285 pb fragmentationts en IV-1.1 pour rendre le ZmCCR1 ARNi construire, qui a une séquence de répétition inversée de 285 pb avec un 238 pb entretoise au milieu des fragments répétées inversées (voir la figure 1).
- Transférer cette construction dans Escherichia coli (E. coli), les cultiver et d'effectuer une taille plasmide isolement de l'ADN de midi-prep à obtenir suffisamment d'ADN plasmidique pour le maïs transformation génétique.
2. Maïs transformation génétique
- Préparation de particules de tungstène
- Placer 60 mg de billes de tungstène (M10) dans un tube de 1,5 ml et on lave avec 1 ml d'éthanol à 70% au vortex pendant 2 min. Incuber pendant 10 min à 23 ° C puis centrifuger à 18 894 g pendant 2 min et jeter le surnageant.
- Laver 3 fois avec 1 ml d'éthanol à 100%, la centrifugation pendant 2 min et en éliminant le surnageant. Ajouter 1 ml de glycerol stérile à 50% pour amener la concentration de microparticules à 60 mg / ml.
- Préparation de l'ADN pour le bombardement
- Place les 50 pi (3 mg) de billes de tungstène préparées dans du glycerol à 50% dans un tube de 1,5 ml. Ajouter 5 ul (1 ug) de IV-1.1 :: ZmCCR1 ARNi ADN plasmidique, 50 ul de 2,5 M CaCl 2, et 20 ul de 0,1 M de spermidine. Vortex brièvement entre chaque addition de réactifs ci-dessus.
- Vortex le tungstène bourrelet ADN mélange brièvement et centrifuger à 18 894 x g pendant 30 s. Décanter le surnageant et remettre en suspension les culots dans 140 ul d'éthanol à 70%. Retirer le liquide et le jeter. Ajouter 140 ul d'éthanol à 100%. Retirer le liquide et le jeter.
- Ajouter 48 ul d'éthanol à 100%. Utiliser immédiatement ou stocker sur la glace jusqu'à 4 heures avant le bombardement.
- Bombardement
- Placez un 3-5 cm de diamètre Salut-II maïs embryonnaire cals (fourni du centre de transformation de maïs de l'Iowa State University) au milieu de 100 mm boîtes de Petri contenant un milieu de N6OSM 32 (comme osmotium) au moins 4 heures avant le bombardement.
- Prepare dispositif de particules de livraison PSD-1000/He selon les instructions du fabricant 33.
- Stériliser paroi de la chambre avec 70% d'éthanol. Chargez stérile 650 psi disque de rupture dans le capuchon de retenue stérile. Étaler 5-6 pi de la solution M10-ADN sur la surface d'un macrosupport, brièvement sec. macrosupport de charge et l'arrêt de l'écran en microporteur ensemble de lancement.
- Placez microporteur ensemble de lancement et le maïs cals dans la chambre à une distance choisie de l'écran d'arrêt (L2 = 6 cm) et fermer la porte. Accélérer dans le vide de 27 psi contre un tamis à mailles de fil.
- Appuyez sur le bouton de tir jusqu'à rafales disque de rupture et la pression de l'hélium jauge tombe à zéro. Relâchez le bouton de tir.
- Incuber les cals bombardés dans une boîte de Pétri contenant N6OSM (milieu osmotique) 32 pendant 16 heures dans l'obscurité à 27 ° C. Cassez les cals en une dizaine de morceaux et de les transférer à N6E (milieu d'induction de cal) 32 dans des boîtes de Pétri et incuber pendant 5 jours in l'obscurité à 27 ° C.
- Sélection
- Après 5 jours sur N6E, transférer les cals sur un milieu N6S (médias de sélection) 32. Subculture tous cals sur un milieu de sélection tous les 30 jours pour les 8-12 semaines sans perturber la structure de cals.
- Après environ 8-10 semaines, blancs secteurs à croissance rapide se développeront à partir de la non-prolifération et partiellement nécrotique mère cals. Exciser les tissus à croissance rapide blancs et repiquer leur milieu de sélection frais (N6S) 32 et continuer à incuber comme ci-dessus.
- Régénération
- Transférer les cals embryonnaires blanc et en croissance rapide sur milieu de régénération 32 et incuber comme ci-dessus pendant 1 semaine. Mettez la régénération de cals embryogènes à une période de 16 heures jour et 8 h obscurité à 25-27 ° C
- Transférer la régénération tire sur le milieu d'enracinement 32 dans un tube à essai en verre au bout de 3-4 semaines, de continuer à incuber comme précédemment. Après r substantielledéveloppement oot apparaît, laver les racines soigneusement sous l'eau du robinet, puis transplanter les plantules à 4 "pots de terreau. Couvrez les pots avec des sacs de plastique pour garder humide. Après 2 jours font de petits trous les sacs en plastique. Après 5-6 jours enlever les sachets en plastique. Continuer à incuber comme ci-dessus pendant 5-6 jours.
- Effet de serre
- Transférer les plants dans des pots de 18 "avec le sol et de maintenir dans la lumière du soleil de plein été ou la lumière à effet de serre. Les plantes régénérées initiales sont appelés T 0 alors que les premières graines appartiennent à la génération T 1.
3. Essai histologique
- Fixer les feuilles de maïs nervures médianes dans 5 ml de 10% du formol tamponné neutre.
- Processus et vide infiltrent avec de la paraffine sur un processeur de tissu à l'aide d'un processeur de tissus.
- Incluez les tissus en paraffine à l'aide d'une station d'enrobage Histocentre III.
- Retirer l'excès de paraffine sur les bords une fois blocks sont refroidis.
- Section échantillon à 4-5 pm avec un microtome à l'aide d'un microtome.
- Placez sections sur des lames de microscope et sec dans un incubateur à 56 ° C pendant 2-24 h. Assurez-vous que les articles sont entièrement respectées à la diapositive.
- Déparaffiner sections dans deux bains de xylène pendant 5 min à 23 ° C.
- Hydrate glisse dans deux changements d'éthanol à 100% pendant 2 min et deux changements de l'éthanol à 95% pendant 2 min à 23 ° C.
- Rincer les sections dans l'eau du robinet pendant 2 min.
- Colorer avec 0,05% de bleu de toluidine O pendant 1-2 min et rincer brièvement avec le trou DDH 2 O.
- Placez une lamelle sur les échantillons avec de l'huile d'immersion et de visualisation en microscopie optique.
- Microscopie électronique à balayage (MEB)
- Fixer les feuilles de maïs milieu des nervures à section transversale à 4% de glutaraldéhyde et 0,1 M de tampon phosphate de sodium (pH 7,4) à 4 ° C pendant 1-2 heures.
- Rincer rapidement les échantillons dans la mémoire tampon, les déshydratées dans unsérie d'éthanol (25%, 50%, 75% et 95%) pour 10 à 15 min à chaque gradation et 100% d'éthanol pendant 10 minutes, 3 fois.
- Sécher les feuilles de maïs milieu des nervures transversales à section déshydratées dans un séchoir à point critique en utilisant du dioxyde de carbone liquide en tant que fluide de transition.
- Montez les échantillons séchés sur les talons de l'aluminium à l'aide des onglets de carbone de haute vide
- Enduire les feuilles de maïs milieu des nervures montées sur les moignons d'aluminium avec de l'or (environ 20 nm d'épaisseur) dans un dispositif de revêtement par pulvérisation cathodique purgé avec de l'argon gazeux.
- Examiner les échantillons revêtus à un JEOL JSM-6400V (émetteur d'électrons de l'hexaborure de lanthane) au microscope électronique à balayage.
- Les images numériques ont été photographiés à l'aide d'un logiciel Pro analyse (de la version 3.2).
4. Klason lignine mesure
- Mill les échantillons à travers un tamis de 2 mm.
- Utiliser un analyseur d'humidité pour déterminer la teneur en humidité de chaque échantillon et noter la valeur.
- Peser environ 1,5 g de chaque échantillon et enregistrer la masse. Extract les échantillons en utilisant de l'eau pour la première extraction, suivi par de l'éthanol pour la seconde extraction à l'aide soit d'un solvant d'extraction automatique (3 cycles par extraction, ~ 14 min par cycle) ou un appareil Soxhlet (8 h par extraction). (Remarque: Cette étape élimine extraits qui peuvent se condenser pendant l'hydrolyse acide et interférer avec la mesure de la lignine précis, l'augmentation de la teneur apparente en lignine Klason.)
- Sécher les échantillons extraits à 45 ° C pendant une nuit, puis les laisser refroidir dans le dessiccateur et peser à nouveau.
- Définir un incubateur à 30 ° C. Mesurer 0,3 g de chaque échantillon sec extrait dans des tubes à haute pression vis-top (triple par échantillon est recommandé) et enregistrer le poids à 0,1 mg près. Ajouter 3 ml de 72% de H 2 SO 4 dans chaque tube de pression.
- Mélanger l'échantillon en utilisant une tige de verre ou téflon d'agitation. Laisser la tige d'agitation dans le tube jusqu'à ce que l'eau est ajoutée après l'incubation.
- Placer les flacons dans un incubateur soit à 30 ° C et 150 rpm pendant 60 min. Après 1 heure, ajouter 84 ml d'eau désionisée pour diluer la concentration de l'acide à 4% et mélanger avec la tige d'agitation. Veillez à ne pas laisser de grandes quantités d'échantillon sur les côtés du flacon-dessus de la ligne d'eau.
- Bien sceller les bouchons sur les flacons et les placer dans un rack de métal ou de grands gobelets. Autoclave à 121 ° C en utilisant un cycle de stérilisation liquide pendant 1 heure. Laisser refroidir à température ambiante avant de l'ouvrir.
- Pré-cendre les creusets de filtrage dans un four à 575 ° C pendant au moins 4 heures. Permettre aux creusets refroidir dans un dessiccateur pendant au moins une heure.
- Filtrer la solution sous vide à partir de chaque tube à travers un creuset séparé, au moyen d'un adaptateur en caoutchouc pour fixer le creuset. Utiliser de l'eau déminéralisée pour rincer les particules restantes du tube.
- Sécher le résidu de lignine à 105 ° C pendant un minimum de 4 heures. Noter le poids du creuset sec et les résidus.
- Si vous utilisez un 575 ° C, pré-Le-feu des échantillons sur un bec Bunsen jusqu'à ce qu'il n'y a pas de fumée ou de la cendre et les placer dans le four pendant 24 heures, ou si vous utilisez un four programmable, ne pas pré-ash et utiliser le programme suivant:
- Rampe de la température ambiante à 105 ° C et maintenir pendant 12 min.
- Rampe à 250 ° C à 10 ° C / min et maintenez pendant 30 min.
- Rampe à 575 ° C à 20 ° C / min et pendant au moins 180 minutes.
- Retirez les creusets du four et laisser refroidir dans un dessiccateur. Peser le creuset et de cendre.
- Calculer le résidu insoluble dans l'acide à l'aide de l'équation suivante:
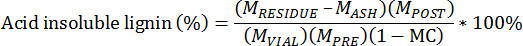
M PRE = masse de la biomasse des pré-extrait
M POST = masse de la biomasse post-extrait
M = masse du VIAL biomasse extraite ajouté à la fiole
M RÉSIDUS = masse du résidu creuset et de la lignineue
M ASH = masse du creuset et de cendre
MC = teneur en eau de la biomasse des pré-extrait, base du poids total
5. Analyse des glucides
- Effectuer la paroi cellulaire glucidique analyses sur la base du Foster et al. (2010) Protocole 31. En bref, préparer l'alcool résidu insoluble de la matière végétale lyophilisée. Ensuite, hydrolyser le matériau avec de l'acide trifluoroacétique et faire correspondre les dérivés de monosaccharides solubilisées à leurs acétates d'alditols correspondants. Analyser ces dérivés volatils par chromatographie en phase gazeuse (GC) relié à un spectromètre de masse quadruple.
Résultats
Nous avons mis en évidence une réduction de la teneur en lignine dans des plants de maïs par l'ARNi. Procédé de transformation d'un bombardement de particules a donné environ 30% d'efficacité de trnasformation. Le silence des gènes de ZmCCR1 a été systématiquement observée dans les générations T0-T2. Les transgéniques de lignine réduit ont augmenté de manière similaire à des plants de maïs de type sauvage, sauf pour l'affichage de coloration brune dans la feuille mi-côte, ba...
Discussion
L'accessibilité des cellulases microbiennes pour planter des polysaccharides de la paroi cellulaire est largement dépendante de la mesure dans laquelle ils sont associés à des polymères phénoliques 23. Le taux de biomasse lignocellulosique de conversion fermentescibles sucre est corrélée négativement avec le contenu de la lignine déposé dans secondadry parois cellulaires végétales. Cette corrélation est attribuée aux propriétés physiques de la lignine tels que l'hydrophobie 24,
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
L'imagerie microscopique a été réalisée par les services du Centre de l'Université d'État du Michigan pour la microscopie avancée. Maïs cal a été acheté à partir du Centre de transformation de maïs de l'Iowa State University. Les auteurs tiennent à remercier Jeffrey R. Weatherhead du Laboratoire MSU recherche sur les plantes pour son aide technique sur l'analyse des glucides. Cette recherche a été généreusement financé par le Programme commercialisation du maïs du Michigan (CMPM) et le Consortium pour la recherche en biotechnologie des plantes (CPBR).
matériels
| Name | Company | Catalog Number | Comments |
| N6OSM (Osmotic medium) | Made in-house | ||
| N6E (Callus induction) | Made in-house | ||
| N6S media (Selection media) | Made in-house | ||
| Regeneration medium | Made in-house | ||
| Rooting medium | Made in-house | ||
| 10% Neutral buffered formalin (1 L) | Made in-house | ||
| Bio-Rad PSD-1000/He Particle Delivery device | Hercules, CA, United States | ||
| Zeiss PASCAL confocal laser scanning microscope | Carl Zeiss, Jena, Germany | For brightfield microscopy, the images were recorded using a Zeiss (Jena, Germany) PASCAL confocal laser scanning microscope with a 488 nm excitation mirror, a 560 nm emission filter, and a 505-530 nm emission filter. Image analysis was performed using Laser scanning microscope PASCAL LSM version 3.0 SP3 software. | |
| Excelsior ES Tissue Processor | Thermo Scientific, Pittsburgh, PA, United States | ||
| HistoCentre III Embedding Station | Thermo Scientific, Pittsburgh, PA, United States | ||
| Microtome Model Reichert 2030 | Reichert, Depew, NY, United States | ||
| Emscope Sputter Coater model SC 500 | Ashford, Kent, England | ||
| JEOL JSM-6400V Scanning Electron Microscope | JEOL Ltd., Tokyo, Japan | ||
| Fitzpatrick JT-6 Homoloid mill | Continental Process Systems, Inc., Westmont, IL | ||
| MA35 Moisture Analyzer | Sartorius | ||
| Critical point dryer, Balzers CPD | Leica Microsysstems Inc, Buffalo Grove, IL, United States | ||
| Screw-top high pressure tubes | Ace Glass, Vineland, NJ | #8648-27 | |
| Screw-top high pressure tube plugs | Ace Glass, Vineland, NJ | #5845-47 |
Références
- Ralph, J., Grabber, J. H., Hatfield, R. D. Lignin-ferulate cross-links in grasses - Active incorporation of ferulate polysaccharide esters into ryegrass lignins. Carbohydrate research. , 275-178 (1995).
- Park, S. -. H. . Expediting cellulosic biofuels agenda: Production of high value-low volume co-products and lignin down-regulation of bioenergy crops [Ph.D. thesis]. , (2011).
- Boerjan, W., Ralph, J., Baucher, M. Lignin biosynthesis. Annual review of plant biology. 54, 519-546 (2003).
- Gibson, L. J. The hierarchical structure and mechanics of plant materials. Journal of the Royal Society, Interface / the Royal Society. 9, 2749-2766 (2012).
- Dien, B. S., et al. Enhancing alfalfa conversion efficiencies for sugar recovery and ethanol production by altering lignin composition. Bioresource technology. , 102-6486 (2011).
- Fu, C. X., et al. Downregulation of Cinnamyl Alcohol Dehydrogenase (CAD) Leads to Improved Saccharification Efficiency in Switchgrass. Bioenerg Res. 4, 153-164 (2011).
- Grabber, J. H., Ralph, J., Hatfield, R. D., Quideau, S. p-hydroxyphenyl, guaiacyl, and syringyl lignins have similar inhibitory effects on wall degradability. Journal of agricultural and food chemistry. 45, 2530-2532 (1997).
- Li, X., et al. Lignin monomer composition affects Arabidopsis cell-wall degradability after liquid hot water pretreatment. Biotechnology for biofuels. 3, (2010).
- Mansfield, S. D., Kang, K. Y., Chapple, C. Designed for deconstruction--poplar trees altered in cell wall lignification improve the efficacy of bioethanol production. The New phytologist. 194, 91-101 (2012).
- Studer, M. H., et al. Lignin content in natural Populus variants affects sugar release. Proceedings of the National Academy of Sciences of the United States of America. 108, 6300-6305 (2011).
- Chen, F., Dixon, R. A. Lignin modification improves fermentable sugar yields for biofuel production. Nature. 25, 759-761 (2007).
- Ziebell, A., et al. Increase in 4-coumaryl alcohol units during lignification in alfalfa (Medicago sativa) alters the extractability and molecular weight of lignin. The Journal of biological chemistry. 285, 38961-38968 (2010).
- Park, S. -. H., et al. The quest for alternatives to microbial cellulase mix production: corn stover-produced heterologous multi-cellulases readily deconstruct lignocellulosic biomass into fermentable sugars. Journal of Chemical Technolog., and Biotechnology. 86, 633-641 (2011).
- Mol, J. N., et al. Regulation of plant gene expression by antisense RNA. FEBS letters. 268, 427-430 (1990).
- Adamo, A., et al. Transgene-mediated cosuppression and RNA interference enhance germ-line apoptosis in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 109, 3440-3445 (2012).
- Smith, N. A., et al. Total silencing by intron-spliced hairpin RNAs. Nature. 407, 319-320 (2000).
- Park, S. -. H., et al. Downregulation of Maize Cinnamoyl-Coenzyme A Reductase via RNA Interference Technology Causes Brown Midrib and Improves Ammonia Fiber Expansion-Pretreated Conversion into Fermentable Sugars for Biofuels. Crop Sci. 52, 2687-2701 (2012).
- Altpeter, F., et al. Particle bombardment and the genetic enhancement of crops: myths and realities. Mol Breeding. 15, 305-327 (2005).
- Pichon, M., Courbou, I., Beckert, M., Boudet, A. M., Grima-Pettenati, J. Cloning and characterization of two maize cDNAs encoding cinnamoyl-CoA reductase (CCR) and differential expression of the corresponding genes. Plant molecular biology. 38, 671-676 (1998).
- Mansoor, S., Amin, I., Hussain, M., Zafar, Y., Briddon, R. W. Engineering novel traits in plants through RNA interference. Trends in plant science. 11, 559-565 (2006).
- Hatfield, R. D., Jung, H. -. J. G., Ralph, J., Buxton, D. R., Weimer, P. J. A comparison of the insoluble residues produced by the Klason lignin and acid detergent lignin procedures. J Sci Food Agr. 65, 51-58 (1994).
- Sluiter, J. B., Ruiz, R. O., Scarlata, C. J., Sluiter, A. D., Templeton, D. W. Compositional analysis of lignocellulosic feedstocks. 1. Review and description of methods. Journal of agricultural and food chemistry. 58, 9043-9053 (2010).
- Sluiter, A., Hames, B., Ruiz, R., Scarlata, C., Sluiter, J., Templeton, D., Crocker, D. Determination of structural carbohydrates and lignin in biomass. Laboratory Analytic Procedure. , (2008).
- Bate, N. J., et al. Quantitative Relationship between Phenylalanine Ammonia-Lyase Levels and Phenylpropanoid Accumulation in Transgenic Tobacco Identifies a Rate-Determining Step in Natural Product Synthesis. Proceedings of the National Academy of Sciences of the United States of America. 91, 7608-7612 (1994).
- Hu, W. J., et al. Repression of lignin biosynthesis promotes cellulose accumulation and growth in transgenic trees. Nature. 17, 808-812 (1999).
- Lapierre, C., et al. Signatures of cinnamyl alcohol dehydrogenase deficiency in poplar lignins. Phytochemistry. 65, 313-321 (2004).
- Updegraff, D. M. Semimicro determination of cellulose in biological materials. Anal Biochem. 32, 420-424 (1969).
- Yemm, E. W., Willis, A. J. The estimation of carbohydrates in plant extracts by anthrone. The Biochemical journal. 57, 508-514 (1954).
- Filomena, A. P., Cherie, W., Geoffrey, B. F., Antony, B. Determining the polysaccharide composition of plant cell walls. Nature. 7, 1590-1607 (2012).
- Foster, C. E., Martin, T. M., Pauly, M. Comprehensive Compositional Analysis of Plant Cell Walls (Lignocellulosic biomass) Part II: Carbohydrates. J. Vis. Exp. (e1837), (2010).
- Department of Agronomy, Iowa State University. Particle bombardment of Hi II immature zygotic embryos and recovery of transgenic maize plants. , (2005).
- Cano-Delgado, A., Penfield, S., Smith, C., Catley, M., Bevan, M. Reduced cellulose synthesis invokes lignification and defense responses in Arabidopsis thaliana. The Plant journal : for cell and molecular biology. 34, 351-362 (2003).
- Boudet, A. M., Kajita, S., Grima-Pettenati, J., Goffner, D. Lignins and lignocellulosics: a better control of synthesis for new and improved uses. Trends in plant science. 8, 576-581 (2003).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon