Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un Sistema de Cultivo Hidropónico para el Análisis Simultáneo y Sistemático de Interacciones Moleculares de Plantas / Microbios y Señalización
En este artículo
Resumen
El sistema de cocultivo hidropónico descrito soporta plantas intactas con mallas de malla metálica y las convierte en cocultivas con bacterias. El tejido de las plantas, las bacterias y las moléculas secretadas pueden ser recogidas separadamente para análisis de aguas abajo, permitiendo simultáneamente que se investiguen las respuestas moleculares tanto de los huéspedes de la planta como de los microbios o microbios que interactúan.
Resumen
Un diseño experimental que simula las interacciones naturales planta-microbio es muy importante para delinear los complejos procesos de señalización planta-microbio. Arabidopsis thaliana - Agrobacterium tumefaciens Proporciona un sistema modelo excelente para estudiar la patogénesis bacteriana y las interacciones de las plantas. Los estudios previos de las interacciones planta- Agrobacterium se han basado en gran medida en cultivos de suspensión de células de plantas, heridas artificiales de plantas o inducción artificial de factores de virulencia microbiana o defensas de plantas mediante productos químicos sintéticos. Sin embargo, estos métodos son distintos de la señalización natural in planta , donde las plantas y los microbios reconocen y responden en formas espaciales y temporales. Este trabajo presenta un sistema de cocultivo hidropónico donde las plantas intactas son soportadas por mallas de malla metálica y cocultivadas con Agrobacterium . En este sistema de cocultivación, no hay fitohormona sintética o producto químico que induzca micrLa virulencia obial o la defensa de la planta se complementa. El sistema de cocultivación hidropónica se asemeja mucho a las interacciones planta-microbios naturales y señala la homeostasis in planta . Las raíces de las plantas se pueden separar del medio que contiene Agrobacterium , y la señalización y las respuestas tanto de los huéspedes de la planta como de los microbios que interactúan pueden ser investigadas simultánea y sistemáticamente. En cualquier punto / intervalo dado, los tejidos vegetales o las bacterias se pueden cosechar por separado para diversos análisis "ómicos", demostrando la potencia y eficacia de este sistema. El sistema de cocultivación hidropónico se puede adaptar fácilmente al estudio: 1) la señalización recíproca de diversos sistemas de microbios de plantas, 2) señalización entre un huésped vegetal y múltiples especies microbianas ( es decir, consorcios o microbios microbianos), 3) cómo están implicados los nutrientes y los productos químicos En la señalización de microbios de plantas, y 4) cómo los microbios interactúan con los huéspedes de la planta y contribuyen a la tolerancia de las plantas aR estrés abióticos.
Introducción
Los microbios asociados a las plantas desempeñan un papel importante en el ciclo biogeoquímico, la biorremediación, la mitigación del cambio climático, el crecimiento y la salud de las plantas y la tolerancia de las plantas a las tensiones bióticas y abióticas. Los microorganismos interactúan con las plantas tanto directamente a través del contacto de la pared celular de la planta como indirectamente a través de la secreción y señalización química 1 , 2 , 3 . Como organismos sésiles, las plantas han desarrollado mecanismos directos e indirectos para resistir la infección por patógenos. Las defensas directas incluyen defensas estructurales y la expresión de proteínas de defensa, mientras que las defensas indirectas incluyen la producción secundaria de metabolitos de plantas y la atracción de organismos antagónicos a patógenos invasores 4 , 5 . Los exudados radiculares, secreciones, mucílagos, mucigel y lisados de las raíces alteran las propiedades físico-químicas de la rizosfera para atraer o repelerMicrobios hacia sus huéspedes 6 . La composición química de la secreción de las raíces es específica de la especie, sirviendo así como un filtro selectivo que permite que ciertos microorganismos capaces de reconocer tales compuestos florezcan en la rizosfera 6 . Por lo tanto, las especies microbianas compatibles pueden ser estimuladas para activar y mejorar sus asociaciones, ya sea en beneficio o detrimento de la planta huésped 1 .
La comprensión de las interacciones planta-microbio en la rizosfera es clave para mejorar la productividad de la planta y el funcionamiento del ecosistema, ya que la mayoría de la exposición microbiana y química ocurre en la estructura de la raíz y en la interfase suelo-aire 2 , 6 , 7 , 8 . Sin embargo, el examen de las interacciones planta-microbios subterráneas y respuestas recíprocas ha sido un desafío debido a su intrigante Compleja y dinámica y la falta de modelos experimentales adecuados con estructura de raíces naturales y morfología de plantas bajo condiciones de crecimiento controlables. Como uno de los fitopatógenos más estudiados, Agrobacterium infecta una amplia gama de plantas con importancia agrícola y hortícola, incluyendo cereza, manzana, pera, uva y rosa 9 . Agrobacterium es un organismo modelo importante para la comprensión de las interacciones planta-patógeno y es una herramienta poderosa en la transformación de plantas y la ingeniería de plantas 10 , 11 , 12 , 13 , 14 .
Las interacciones moleculares entre plantas y Agrobacterium han sido bien estudiadas durante varias décadas, y la comprensión actual de la patogenicidad de Agrobacterium es extensa 9 ,f "> 11, 15, 16. patogenicidad Agrobacterium se atribuye en gran parte a sus capacidades evolucionadas de percibir señales derivadas de plantas, dando como resultado la modulación fina de su programa de la virulencia y la comunicación de célula a célula, llamado quórum de detección 17. La El programa de virulencia de Agrobacterium está regulado por varias señales disponibles en la rizosfera e incluye dos sistemas de sistemas de dos componentes, el sistema ChvG / I y el sistema VirA / G. Las condiciones ácidas en la rizosfera activan la transcripción de chvG / I , virA / G , Y varios otros genes implicados en la patogenicidad de Agrobacterium , incluyendo virE0 , virE1 , virH1 , virH2 y genes del sistema de secreción de tipo VI (T6SS) 18. Compuestos fenólicos derivados de plantas, incluyendo acetosiringona (4'-hidroxi-3 ', 5 '-dimetoxiacetofenona), activar el VIrA / G sistema de 2 componentes a través de mecanismos de señalización de fosforilación 19 . VirA / G activa entonces todo el vir regulon, dando como resultado la transferencia e integración de un fragmento de ADN bacteriano de aproximadamente 20 kb denominado ADN de transferencia (T-DNA) desde su plásmido inductor de tumores (Ti) al núcleo de la planta 16 . T-DNA transporta genes responsables de la síntesis de las hormonas vegetales indol-3-acético (IAA) ( iaaM y iaaH ) y citoquinina ( ipt ), y una vez expresado en las células vegetales, se producen grandes cantidades de estas fitohormonas. Esto da lugar a la proliferación anormal del tejido y al desarrollo del tumor de la planta, conocida como enfermedad de la bilis de la corona, que es un problema crónico y resurgente para las plantas 9 , 11 , 20 . IAA también actúa colectivamente con ácido salicílico y ácido gamma-amino butírico para reprimir la virulencia de Agrobacterium o para reducir Agrobacteriu M quorum sensing (QS) 17 , 21 , 22 . Para contrarrestar esta represión, el T-DNA también lleva genes para la biosíntesis de opina, que activa la detección de Agrobacterium quorum para promover la patogenicidad de Agrobacterium y también sirve como fuente de nutrientes para el patógeno 22 , 23 .
A pesar de una profunda comprensión global de Agrobacterium- planta interacciones y la transferencia de T-DNA resultante en el huésped de la planta, los complejos eventos de señalización en la etapa inicial de la interacción son menos bien entendidos. Esto se debe en parte a las limitaciones de los métodos convencionales para investigar la señalización de planta de Agrobacterium . Los cultivos de suspensión de células vegetales y heridas artificiales específicas del sitio se usan comúnmente para estudiar las interacciones moleculares planta-microbio 24 ,. ef "> 26, 27 Sin embargo, las suspensiones de células carecen de morfología de la planta típica, en particular, células en suspensión planta no tienen estructuras profundas y los exudados de la raíz, que son muy importantes para la activación de la quimiotaxis microbiana y la virulencia 28, 29 El mantenimiento de la morfología de la planta. Y la estructura de la raíz se ha tratado de herir artificialmente a las plantas, lo que facilita la infección específica del sitio, lo que resulta en la detección de genes inducidos relacionados con la defensa de la planta en tejido vegetal directamente infectado 30 , 31. Sin embargo, la herida artificial es significativamente diferente de la infección patógena en la naturaleza , Particularmente como herida conduce a la acumulación de ácido jasmónico (JA), que sistemáticamente interfiere con la señalización de la planta natural y la defensa 26. Además, los productos químicos sintéticos se utilizan normalmente para inducir artificialmente las respuestas del huésped de la plantaO virulencia patógena. Aunque la suplementación de tales compuestos químicos refleja concentraciones en planta es posible, tal suplementación no explica la difusión de los exudados de raíz gradualmente en la rizosfera circundante, lo que genera un gradiente quimiotáctico detectado por los microbios 28 , 32 . Dadas las limitaciones de los enfoques convencionales para estudiar las interacciones planta-microbios, la exactitud y la profundidad de los datos obtenidos podrían ser impedidas y restrictivas, y el conocimiento generado a partir de los enfoques convencionales puede no traducir directamente in planta . Muchos aspectos de la señalización de Agrobacterium en las plantas aún no se entienden completamente, particularmente en la etapa temprana de las interacciones, cuando los síntomas de la enfermedad aún no se han desarrollado.
Para enmendar las limitaciones de los enfoques convencionales, este trabajo presenta un bajo costo, controlable, y flexible hydroponic cOcultivation sistema que permite a los investigadores a obtener una visión más profunda de la compleja de señalización y respuesta a las vías en la etapa inicial de la planta molecular microbio interacciones. La hidroponía se ha utilizado ampliamente para estudiar los nutrientes de las plantas, los exudados de las raíces, las condiciones de crecimiento y los efectos de la toxicidad metálica en las plantas 33 , 34 . Existen varias ventajas de los modelos hidropónicos, incluyendo las pequeñas exigencias espaciales, la accesibilidad de los diversos tejidos vegetales, el estricto control de las condiciones nutrientes / ambientales y el control de plagas / enfermedades. Los sistemas hidropónicos también son menos limitantes para el crecimiento de las plantas en comparación con las técnicas de placas de agar / phytoagar, que típicamente restringen el crecimiento después de 2-3 semanas. Es importante destacar que el mantenimiento de las estructuras de toda la planta facilita la secreción de raíces naturales necesarias para la quimiotaxis microbiana y la inducción de la virulencia [ 8 , 29] . El sistema descriLa cama aquí es más simple y menos laboriosa que las alternativas 33 , 34 . Utiliza menos piezas y no requiere ninguna otra herramienta que las tijeras estándar. Utiliza malla metálica (en oposición al nylon 33 ) como soporte fuerte para el crecimiento de la planta y un método simple de aireación en condiciones estériles mediante agitación para soportar el crecimiento microbiano. Además, el sistema puede utilizar malla metálica de varios tamaños para apoyar el crecimiento de la planta, que acomoda diversas especies de plantas sin restringir la anchura de sus raíces.
En el sistema de cocultivación hidropónica presentado aquí, las plantas se cultivan en un sistema hidropónico estéril donde las raíces de las plantas secretan compuestos orgánicos que apoyan el crecimiento de las bacterias inoculadas. En este sistema de cocultivación, no se suplementan productos químicos artificiales, tales como hormonas vegetales, elicitor de defensa, o productos químicos que inducen virulencia, lo que refleja la célula natural-significando la homeostasis durante las interacciones planta-microbio. Con este sistema de cocultivación hidropónica, fue posible determinar simultáneamente la expresión génica en el tejido de raíz de Col-0 de Arabidopsis thaliana tras la infección por Agrobacterium , así como la activación de genes de Agrobacterium tras la cocultivación con Arabidopsis . Se demostró además que este sistema es adecuado para estudiar la fijación de Agrobacterium a las raíces de las plantas, así como el perfil de secreto de la raíz de la planta, al cocultivar (infección) con Agrobacterium ( Figura 1 ).
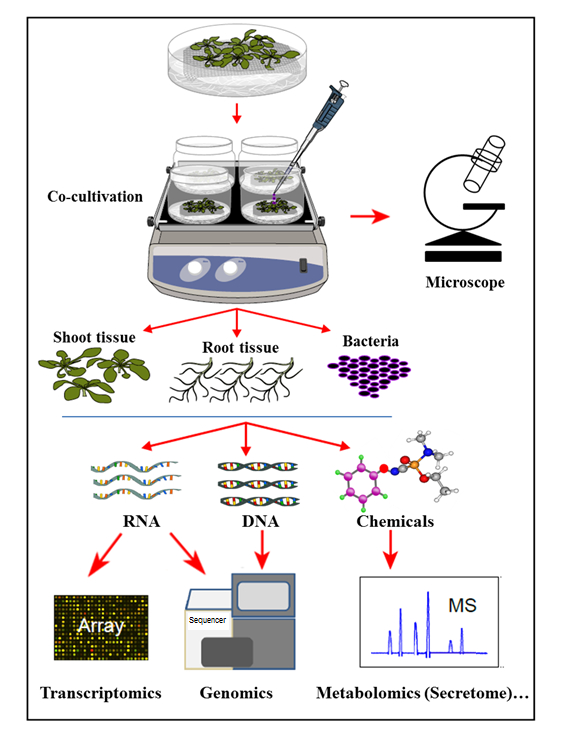
Figura 1: Visión general del sistema de cocultivación hidropónica, con análisis de muestras. Las plantas se cultivan en la parte superior de la malla (brotes por encima de la malla), con las raíces sumergidas en medio hidropónico que luego se inocula con bacterias fO el coculture. Los tejidos vegetales y las bacterias se separan para extracciones y análisis simultáneos. Esta cifra ha sido modificada a partir de la referencia 35 .
Protocolo
1. Planificación Experimental
- Determinar los objetivos específicos del experimento.
- Lea todo el protocolo a continuación. Determinar qué secciones son relevantes para los objetivos específicos y si es necesario realizar modificaciones. Vea a continuación ejemplos.
- Considere si los experimentos utilizarán plantas de A. thaliana y bacteria A. tumefaciens , como a continuación, u otra combinación planta-microbio.
NOTA: Si bien pueden usarse varias especies de plantas, el grosor de las raíces de las plantas puede requerir una malla de tamaño diferente (paso 3.3) y los parámetros de crecimiento (pasos 3.10-3.11 y 4.4) y el tamaño del tanque hidropónico y el volumen medio 4.2) también puede requerir ajuste ( Figura 2 ). Los microbios pueden inocularse fácilmente como cultivos puros, especies mixtas (consorcios), o de muestras ambientales (microbiomas), pero cualquier cambio puede afectar el protocolo, particularmente el paso 4.5. - Considera elTipos de experimentos que se utilizarán para analizar las muestras.
NOTA: La microscopía de fluorescencia (etapa 5) requiere organismos que están marcados con un constructo indicador fluorescente.
- Considere si los experimentos utilizarán plantas de A. thaliana y bacteria A. tumefaciens , como a continuación, u otra combinación planta-microbio.
- Diseñe controles apropiados. Incluyen tanques hidropónicos sin plántulas que se inoculan con bacterias de control y tanques con plántulas de control que no son inoculadas con bacterias.
- Planifique experimentos con el número apropiado de semillas y tanques hidropónicos. Consideremos los controles, las repeticiones biológicas (3 como mínimo) y las cantidades de tejido requeridas para todos los análisis.
- Determine la longitud adecuada de cocultivación para los objetivos experimentales (paso 4.8).
- Revise los pasos siguientes, según sea necesario.

Figura 2. Ejemplos de otras plantas que podrían ser cultivadas en el sistema hidropónico, apoyadas por un P Latform de la malla del metal. Compatibilidad de un conjunto de mallas metálicas para una variedad de semillas de plantas y cultivo. ( A ) De acero inoxidable tipo 304 malla weldmesh 3 × 3 malla × 0,047 "dia de alambre para Vicia faba . ( B ) De acero inoxidable tipo 304 weldmesh malla 4 × 4 × 0,035" dia de alambre para Zea mays . ( C ) Acero inoxidable tipo 304 malla de soldadura 4 × 4 malla × 0,032 "diámetro de alambre para Glycine max (soja) ( D ) Acero inoxidable tipo 304 malla de soldadura 6 × 6 malla × 0,047" diámetro de cable para Raphanus sativus rábano). ( E ) Acero inoxidable tipo 304 malla soldada 6 × 6 malla × 0,047 "diámetro para Triticum spp ( F ) Acero inoxidable tipo 304 malla de soldadura 6 × 6 malla × 0,035" dia de alambre para Cucumis sativus . Esta cifra ha sido modificada a partir de la referencia 35 .955fig2large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
2. Esterilización de superficies de semillas de plantas
- Vórtice (a alta velocidad) aproximadamente 200 semillas de A. thaliana con 500 μl de agua desionizada en un tubo de microcentrífuga. Para las especies de plantas con semillas más grandes, use un tubo grande para facilitar la esterilización superficial.
- Centrifugar esto a aproximadamente 9.000 xg durante 30 s en una microcentrífuga de mesa. Retire el agua (sobrenadante) usando una pipeta.
- Añadir 300 μl de hipoclorito de sodio al 2% a las semillas y agitar el vórtice. Dejar a temperatura ambiente durante 1 min. Para las especies de plantas con semillas más grandes, use volúmenes mayores para cubrir las semillas.
- Centrifugar a aproximadamente 9.000 xg durante 30 s en una microcentrífuga de mesa. Retire la solución de hipoclorito de sodio con una pipeta.
- Añadir 500 μl de agua estéril, desionizada a las semillas y vórtice. Centrifugar a aproximadamente 9.000 xg para30 s y retire el agua con una pipeta estéril.
- Repita el paso 2.5 un adicional de 4 veces.
- Agregue 500 μl de etanol al 70% a las semillas y agite el vórtice. Dejar a temperatura ambiente durante 1 min.
- Centrifugar a aproximadamente 9.000 xg durante 30 s en una microcentrífuga de mesa. Retirar el etanol al 70% con una pipeta.
- Repita el paso 2.5 por 5 veces más.
- Resuspender las semillas esterilizadas en 500 μl de agua estéril y ultrapura.
3. Germinación de Semillas y Cultivo Semisólido de Plantas
- Preparar medio semisólido de Murashige y Skoog (MS): 2.165 g / L MS sales basales; 10 g / l de sacarosa; 0,25 g / L de MES; Y 59 ml / L de mezcla de vitaminas B5, pH 5,75, con 4 g / l de phytoagar.
- Autoclave el medio MS. Vierta 25 ml de él en cada placa de Petri profunda estéril (100 x 25 mm 2 ).
- Para cada placa de Petri, corte un cuadrado de 90 x 90 mm de malla de acero inoxidable.
NOTA: La malla recomendada para A. thaliana < / Em> tiene un grado de 304 (grado estándar), el número de mallas (número de agujeros por pulgada lineal) de 40 x 40 y un diámetro de alambre de 0,01 "(0,0254 cm) Un cuadrado de malla mayor es requerido para tanques hidropínicos más grandes O plataformas más altas. - Doble las esquinas de cada malla cuadrada lo suficiente para que la malla encaje dentro de la placa de Petri. Doblar las esquinas en un ángulo de 90 ° con el grueso de la malla para permitir que la malla para ser apoyado por las esquinas, dejando suficiente espacio por debajo para el desarrollo de raíces ( Figura 3a ].
- Coloque los cuadrados de malla cortada en un vaso de precipitados, cubra con papel de aluminio y esterilice en autoclave con un ciclo seco de 30 min.
- Una vez que el medio se ha solidificado en las placas de Petri y la malla es estéril, coloque cada malla esterilizada cuadrada sobre el medio semisólido, con las esquinas dobladas hacia abajo.
- Empujar la malla para que las esquinas penetren en el medio y el grueso de la malla toque la superficie superior del medio (> Figura 3b).
- Use una pipeta de transferencia de 200 μl para extraer semillas individuales de A. thaliana esterilizadas en la superficie (las semillas permanecerán en la punta de la pipeta debido a la fuerza de vacío) y transferir suavemente cada semilla sobre la malla. Coloque las semillas de modo que estén espaciadas apropiadamente para las necesidades experimentales ( por ejemplo, 4 6 semillas / placa).
- Selle las placas de Petri con tapas, colocando cinta quirúrgica porosa alrededor de los bordes.
- Estratificar las semillas colocando toda la placa de Petri a 4 ° C durante 2 d en la oscuridad ( por ejemplo, envolver en papel de aluminio para simular la oscuridad.)
- Cultivar las semillas en la placa de Petri a 22-24 ° C con un fotoperiodo de 16 h durante 10-14 días ( Figura 3c ].

Figura 3: Sistema hidropónico de cocción de microbios de plantas. El pa izquierdoNel representa un diagrama de flujo que delinea los seis pasos claves en el ensamblaje y operación del sistema de co-cultivo hidropónico. El panel derecho demuestra los materiales experimentales reales, el equipo y los procedimientos operacionales para el sistema de cocultivación hidropónico cuando se estudian las interacciones de Arabidopsis-Agrobacterium . Se muestran ahora el paso de inoculación y el paso final para el muestreo de plantas o bacterias. Esta cifra ha sido modificada a partir de la referencia 35 . Haga clic aquí para ver una versión más grande de esta figura.
4. Sistema Cocultivo Hidropónico
- Preparar el medio líquido de Murashige y Skoog (MS): 2.165 g / L de sales basales de MS, 10 g / L de sacarosa, 0.25 g / L de MES y 59 mL / L de mezcla de vitaminas B5, pH 5.75.
- Autoclave el medio. Verter 18 ml de la misma en cada vaso cilíndrico estéril o en un recipiente de plástico transparente (placas de cristalización de 100 x 80 mm2) con tapas estériles.
NOTA: Presteilizar los tanques y tapas envolviéndolos en papel de aluminio y autoclave. - En una campana de flujo esterilizada, use fórceps estériles para transferir suavemente cada malla cuadrada con plántulas de 10 a 14 días desde el medio semisólido a un tanque cilíndrico hidropónico ( Figura 3d ). Selle las tapas en los tanques usando cinta quirúrgica porosa.
- Cultivar las plántulas en la malla en el tanque durante 72 h a 22-24 ° C, con un fotoperiodo de 16 h y agitando a 50 rpm para la aireación ( Figura 3e ).
NOTA: Esto permite que las plantas se adapten al medio hidropónico antes de la inoculación (cocultivación) con microorganismos. Este período de espera también permitirá que la contaminación microbiana accidental sea evidente antes de la inoculación.- Comience el siguiente paso el día anterior al final del período de incubación.
- Cultivar A. tumefaciens en medio AB(0,5% w / extracto v levadura, 0,5% w / v de triptona, 0,5% w / v de sacarosa, 50 mM MgSO4, pH 7,0) a 28 ° C con agitación durante la noche hasta que el cultivo alcanza una densidad óptica final de 1,0 a 600 Nm (DO 600 ), medido usando un espectrofotómetro, con medio AB no inoculado como blanco.
NOTA: Un OD 600 de 1,0 es equivalente a aproximadamente 10 9 células / mL. - Lavar las A. tumefaciens tres veces en un volumen igual de 0,85% de NaCl. Resuspender en un volumen igual de agua estéril doblemente destilada.
- Antes de la inoculación, examine cuidadosamente el tanque con las plántulas para asegurarse de que no haya contaminación; El medio en los tanques no contaminados debe ser claro y transparente.
- Desechar cualquier tanque con líquido turbia (contaminación) y numerar el resto de los tanques. Muestre una pequeña cantidad (20 μl) de medio hidropónico de cada tanque numerado y colóquelo en una placa de Petri llena de caldo de Luria (LB). IncubarEl plato a 28 ° C.
- Inmediatamente, añadir 50 μL de la suspensión de A. tumefaciens (aproximadamente 5 x 10 7 células, estimada a partir de la DO 600 ) en cada tanque hidropónico numerado. Coculen las plántulas de A. thaliana y A. tumefaciens a 22-24 ° C con un fotoperiodo de 16 h y agitando a 50 rpm ( Figura 3f ).
- Examine la placa LB manchada de la etapa 4.7.1. Después de 12 y 28 h de incubación para verificar la esterilidad de los tanques. Si hay alguna contaminación, no utilice el tanque numerado correspondiente (inoculado con bacterias) para otros ensayos aguas abajo.
- Si se desea, monitorear el crecimiento de A. tumefaciens a intervalos regulares retirando una muestra de medio del tanque y midiendo la OD 600 usando un espectrofotómetro con medio de un control no inoculado como blanco ( Figura 4 ).
NOTE: Los síntomas de las enfermedades de las plantas deberían ser evidentes después de 7 días ( Figura 5 ). - Separar las raíces de A. thaliana de la suspensión bacteriana levantando la placa de malla; La temporización de este paso depende de los procesos posteriores. Vea los pasos 5-8 para más detalles.
- Si utiliza raíces para análisis subsiguientes, enjuague rápidamente las raíces con agua doblemente destilada. Si usa hojas u otros tejidos, corte el tejido. Para los análisis de ARN, proceda inmediatamente al paso 7.1.
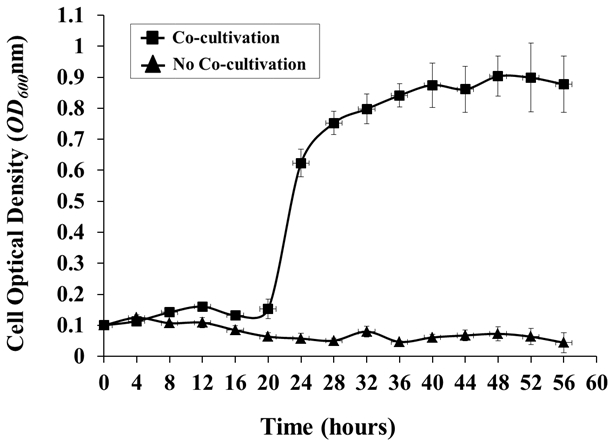
Figura 4: Crecimiento de Agrobacterium en el Sistema de Cocultivación Hidropónico. El crecimiento de Agrobacterium en presencia o ausencia de un huésped vegetal ( Arabidopsis ) se monitorizó cada 4 h. Las células de Agrobacterium se hicieron crecer en medio AB O / N, se lavaron 3x con NaCl al 0,85%, y se inocularon en el sistema hidropónico, con uSin co-cultivo de Arabidopsis , a partir de una OD 600 inicial de alrededor de 0,1. Los valores de DO 600 son los medios de tres replicaciones biológicas, con desviaciones estándar ( OD 600 de 1,0 = 1 x 10 9 células / ml).

Figura 5: Fenótipos de las plantas representativas y síntomas de la enfermedad observable durante la cocultivación. Dentro de 4 días después de la inoculación, no se observan síntomas de la enfermedad ( A ) en comparación con las plantas no inoculadas ( B ). Después de 7 d de Cocultivación (infección), se observan síntomas de enfermedad en plantas infectadas ( C ), mientras que las plantas no inoculadas permanecen sanas ( D ).
5. Microscopía de Fluorescencia
- Utilice A. tumefaciens albergando un constructo reportero para una proteína autofluorescente paraLa configuración de cocultivación en el paso 4.5 .
NOTA: Se utiliza aquí un plásmido pJP2 modificado que expresa pCherry, un derivado de dsRed 36 . - Después de un co-cultivo apropiado ( por ejemplo, 48 h) en el paso 4.8, separe las raíces secundarias individuales (ramas laterales de la raíz principal) usando tijeras estériles.
- Enjuague las raíces en agua doblemente destilada para eliminar el material suelto. Sumergir cada raíz en 30 μl de agua en un portaobjetos de microscopio y cubrir con un cubreobjetos de vidrio.
- Selle los bordes del cubreobjetos con esmalte de uñas para prevenir la deshidratación de las raíces.
- Visualice la fijación de A. tumefaciens a las raíces de A. thaliana mediante microscopía de fluorescencia.
Se utiliza un microscopio confocal con excitación de un láser de helio-neón (He-Ne) 543/594 nm para visualizar la fluorescencia roja de pCherry a 590-630 nm bajo un objetivo de lente de agua 63X invertida con una abertura numérica de 1,4 ( Figura 6).

Figura 6: Adjunto de la raíz de Agrobacterium , determinado por Microscopía Confocal. El pCherry rojo marcado fluorescencia Agrobacterium se visualizó a 590-630 nm, con la excitación de helio-neón (He-Ne) 543/594 nm láser. La visualización se llevó a cabo bajo un objetivo invertido de lente de agua 63X con una abertura numérica de 1,4. Barras graduadas = 11 μm. Haga clic aquí para ver una versión más grande de esta figura.
6. Análisis de transcripciones bacterianas
- Después de un co-cultivo apropiado ( por ejemplo, 8 h) en la etapa 4.8, separar las raíces del medio hidropónico como en el paso 4.9. Transferir 1,5 ml del medio hidropónico a un micro de 1,5 mlCentrifugar y centrifugar a 12.000 xg durante 2 minutos para sedimentar las células.
- Retirar el sobrenadante. Transferir otros 1,5 ml del medio hidropónico al mismo tubo de microcentrífuga de 1,5 ml y centrifugar a 12.000 g durante 2 minutos para sedimentar las células.
- Retirar el sobrenadante.
NOTA: El protocolo puede detenerse aquí congelando las muestras a -80 ° C hasta su uso. - Extraer el ARN de las células de A. tumefaciens usando métodos estándar 37 o un kit de aislamiento y purificación de ARN comercial adecuado con tratamiento con DNasa para evitar la contaminación del ADN.
NOTA: El protocolo puede detenerse aquí congelando alícuotas de ARN a -80 ° C hasta su uso. - Utilice el ARN aislado para una variedad de análisis usando métodos estándar, incluyendo los siguientes.
- Utilice un microarrays comercial de acuerdo con el protocolo del fabricante para detectar conjuntos de genes que se expresan diferencialmente en cocultivado versus controlcultura.
- Utilizar cuantitativa en tiempo real Reacción en cadena de la polimerasa (qRT-PCR) para detectar la expresión de genes específicos o para validar los datos de microarrays [ 38] .
7. Análisis de transcripciones de plantas
- Después de una cocultivación apropiada ( por ejemplo, 8 h) en la etapa 4.8, separar las raíces del medio hidropónico, como en la etapa 4.9, o separar otros tejidos según sea necesario. Coloque inmediatamente aproximadamente 150 mg de raíces u otro tejido vegetal en nitrógeno líquido.
NOTA: El protocolo puede detenerse aquí congelando las muestras a -80 ° C hasta su uso. - Moler las muestras congeladas a un polvo en nitrógeno líquido usando un mortero y un mazo.
- Extraer el ARN del polvo utilizando métodos estándar 39 o un kit de aislamiento y purificación de ARN comercial adecuado con tratamiento con DNasa para evitar la contaminación del ADN.
NOTA: El protocolo puede detenerse aquí congelando alícuotas de ARN en-80 ° C hasta su uso. - Utilice el ARN aislado para una variedad de análisis usando métodos estándar, incluyendo los siguientes.
- Utilice un microarrays comercial de acuerdo con el protocolo del fabricante para detectar conjuntos de genes que se expresan diferencialmente en plantas co-cultivadas versus control.
- Utilizar qRT-PCR para detectar la expresión diferencial de genes específicos o para validar los datos de microarrays [ 38] .
8. Secreome Perfilado
- Después de un co-cultivo apropiado ( por ejemplo, 72 h) en la etapa 4.8, separar las raíces del medio hidropónico como en el paso 4.9. Esterilice los 18 ml de medio hidropónico haciéndolo pasar a través de un filtro de poro de 0,2 μm en tubos cónicos de 50 ml.
- Congelar las muestras a -80 ° CO / N.
- Afloje las tapas de los tubos cónicos de 50 ml y colóquelas en una máquina de liofilización durante 36 h. Proceda a almacenar o procesar las muestras (como desDescrito a continuación) para el análisis de HPLC y la detección de compuestos.
NOTA: El protocolo se puede pausar aquí. - Vuelva a suspender cada muestra en 5 mL de agua doblemente destilada en un tubo de ensayo sellable.
- Añadir 5 ml de acetato de etilo y mezclar por inversión del tubo varias veces.
- Dejar que las fases se separen a temperatura ambiente durante 5 min.
- Transferir la parte superior (fase orgánica) a un nuevo recipiente por pipeteo. Si es necesario, agrupe múltiples fracciones orgánicas.
- Secar bajo una corriente suave de gas nitrógeno (~ 45 min).
- Se vuelve a suspender la muestra en una solución apropiada para HPLC ( por ejemplo, metanol al 100%).
- Realizar HPLC de acuerdo con los métodos estándar 40 .
- Detectar compuestos por medios apropiados.
NOTA: La Espectrometría de Masa de Tiempo de Vuelo de Ionización por Electrospray (ESI-TOF-MS) 40 se usa aquí.
Resultados
Crecimiento en el sistema de co-cultivo hidropónico
La curva de crecimiento de A. tumefaciens C58 demostró una fase de retraso significativo en las 16 h iniciales de cocultivación, seguido de un crecimiento muy estable cuando se co-cultivó con A. thaliana Col-0, hasta un máximo de DO 600 de aproximadamente 0,9 a partir de 48 h postinoculación. Por el contrario, esencialmente no se observó creci...
Discusión
Dada la naturaleza gradual de la secreción de las raíces, la concentración de los productos químicos inducidos por la virulencia producidos in planta y sus efectos sobre las interacciones dinámicas planta-microbio ocurren en gradientes espaciales y temporales. En este sistema de co-cultivo hidropónico, no se añade fitohormona sintética o sustancia química que induce la virulencia microbiana o las defensas de las plantas. Por el contrario, utilizando métodos convencionales, la adición de productos qu?...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Nos gustaría agradecer a Brian Weselowski y Alexander W. Eastman por su ayuda y útil discusión. También queremos dar las gracias a los Dres. Eugene W. Nester, Lingrui Zhang, Haitao Shen, Yuhai Cui y Greg Thorn por su ayuda, útiles discusiones y lectura crítica del manuscrito. Esta investigación fue financiada por Agricultura y Agroalimentación de Canadá, Growing Forward-AgriFlex (número RBPI 2555) y el proyecto de Cultivo de Frente II número 1670, realizado por los autores como parte de sus funciones. Este estudio también fue parcialmente financiado por el Consejo de Investigación de Ciencias Naturales e Ingeniería de Canadá (CRSNG) Discovery Grant RGPIN-2015-06052 otorgado a ZC Yuan.
Materiales
| Name | Company | Catalog Number | Comments |
| plant seeds (Arabidopsis thaliana Col-0) | Arabidopsis Biological Resource Centre | CS7000 | https://abrc.osu.edu/order-stocks |
| bacteria (Agrobacterium tumefaciens C58) | University of Washington | N/A | |
| labeled bacteria | in-house | optional, depends on downstream analyses | |
| vortex | (various) | ||
| microcentrifuge tubes | (various) | ||
| microcentrifuge | (various) | ||
| 5% sodium hypochlorite | (various) | ||
| double distilled water | (various) | ||
| autoclave | (various) | ||
| micropipette | (various) | ||
| 70% ethanol | (various) | ||
| Murashige and Skoog (MS) basal salts | Sigma-Aldrich | M5524 | |
| sucrose | (various) | ||
| MES | (various) | ||
| B5 vitamin mix | Sigma-Aldrich | G1019 | |
| phytoagar | (various) | ||
| Deep Petri dishes | (various) | ||
| stainless steel mesh | Ferrier Wire Goods Company Ltd | N/A | grade: 304; mesh count: 40 × 40; wire DIA: 0.01 |
| micropore tape, 1" | 3M | 1530-1 | |
| diurnal growth chamber | (various) | ||
| cylindrical glass tanks, 100 × 80 mm | Pyrex | 3250 | other sizes can be used, in which case liquid content may need adjustment |
| flow hood | (various) | ||
| forcepts | (various) | ||
| yeast extract | (various) | ||
| tryptone | (various) | ||
| MgSO4 | (various) | ||
| shaking incubator | (various) | ||
| spectrophotometer | (various) | ||
| NaCl | (various) | ||
| shaker | (various) | ||
| scissors | (various) | optional, depends on downstream analyses | |
| fluorescence microscope | (various) | optional, depends on downstream analyses | |
| microscope slides and cover slips | (various) | optional, depends on downstream analyses | |
| nail polish | (various) | optional, depends on downstream analyses | |
| Bacterial RNA extraction kit | (various) | optional, depends on downstream analyses | |
| plant RNA extraction kit (RNeasy Plant Mini Kit) | Qiagen | 74903 or 74904 | optional, depends on downstream analyses |
| material and equipment for qRT-PCR | (various) | optional, depends on downstream analyses | |
| material and equipment for microarray analysis | (various) | optional, depends on downstream analyses | |
| liquid nitrogen | (various) | optional, depends on downstream analyses | |
| mortar and pestle | (various) | optional, depends on downstream analyses | |
| 0.2 µm pore filter | (various) | optional, depends on downstream analyses | |
| 50 mL conical tubes | (various) | optional, depends on downstream analyses | |
| freeze dryer | (various) | optional, depends on downstream analyses | |
| sealable test tubes | (various) | optional, depends on downstream analyses | |
| ethyl acetate | (various) | optional, depends on downstream analyses | |
| nitrogen gas | (various) | optional, depends on downstream analyses | |
| material and equipment for HPLC | (various) | optional, depends on downstream analyses | |
| material and equipment for ESI-TOF-MS | (various) | optional, depends on downstream analyses |
Referencias
- Lambers, H., Mougel, C., Jaillard, B., Hinsinger, P. Plant-microbe-soil interactions in the rhizosphere: An evolutionary perspective. Plant Soil. 321 (1), 83-115 (2009).
- Philippot, L., Raaijmakers, J. M., Lemanceau, P., vander Putten, W. H. Going back to the roots: The microbial ecology of the rhizosphere. Nat. Rev. Microbiol. 11 (11), 789-799 (2013).
- Somers, E., Vanderleyden, J., Srinivasan, M. Rhizosphere bacterial signalling: A Love Parade beneath our feet. Crit. Rev. Microbiol. 30 (4), 205-240 (2004).
- Barah, P., Winge, P., Kusnierczyk, A., Tran, D. H., Bones, A. M. Molecular signatures in Arabidopsis thaliana in response to insect attack and bacterial infection. PLoS ONE. 8 (3), (2013).
- Zhang, J., Zhou, J. -. M. Plant immunity triggered by microbial molecular signatures. Mol. Plant. 3 (5), 783-793 (2010).
- Paterson, E., Gebbing, T., Abel, C., Sim, A., Telfer, G. Rhizodeposition shapes rhizosphere microbial community structure in organic soil. New Phytol. 173 (3), 600-610 (2006).
- Hartmann, A., Schmid, M., van Tuinen, D., Berg, G. Plant-driven selection of microbes. Plant Soil. 321 (1-2), 235-257 (2008).
- Micallef, S. A., Shiaris, M. P., Colon-Carmona, A. Influence of Arabidopsis thaliana accessions on rhizobacterial communities and natural variation in root exudates. J. Exp. Bot. 60 (6), 1729-1742 (2009).
- Burr, T., Otten, L. Crown gall of grape: Biology and disease management. Annu. Rev. Phytopathol. 37, 53-80 (2001).
- Clough, S. J., Bent, A. F. Floral dip: A simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. Plant J. 16 (6), 735-743 (1998).
- Binns, A. N., Costantino, P., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. The Agrobacterium oncogenes. The Rhizobiaceae. , (1998).
- Gheysen, G., Angenon, G., Van Montagu, M., Lindsey, K. Agrobacterium-mediated plant transformation: a scientifically intriguing story with significant applications. Transgenic Plant Research. , (1998).
- Valvekens, D., Von Montagu, M. V., Van Lijsebettens, M. Agrobacterium tumefaciens-mediated transformation of Arabidopsis thaliana root explants by using kanamycin selection. Proc. Natl. Acad. Sci. USA. 85 (15), 5536-5540 (1988).
- Krysan, P. J., Young, J. C., Sussman, M. R. T-DNA as an insertional mutagen in Arabidopsis. Plant Cell. 11 (12), 2283 (1999).
- Chilton, M. D., et al. Stable incorporation of plasmid DNA into higher plant cells: The molecular basis of crown gall tumorigenesis. Cell. 11 (2), 263-271 (1977).
- Gelvin, S. B. Agrobacterium in the genomics age. Plant Physiol. 150 (4), 1665-1676 (2009).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. -. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front. Plant Sci. 5, 322 (2014).
- Yuan, Z., Liu, P., Saenkham, P., Kerr, K., Nester, E. W. Transcriptome profiling and functional analysis of Agrobacterium tumefaciens reveals a general conserved response to acidic conditions (pH 5.5) and a complex acid-mediated signaling involved in Agrobacterium-plant interactions. J. Bacteriol. 190 (2), 494-507 (2008).
- Stachel, S. E., Messens, E., Van Montagu, M., Zambryski, P. Identification of the signal molecules produced by wounded plant cells that activate T-DNA transfer in Agrobacterium tumefaciens. Nature. 318 (6047), 624-629 (1985).
- Memelink, J., de Pater, B. S., Hoge, J. H. C., Schilperoort, R. A. T-DNA hormone biosynthetic genes: Phytohormones and gene expression in plants. Dev. Genet. 8 (5-6), 321-337 (1987).
- Yuan, Z. -. C., et al. The plant signal salicylic acid shuts down expression of the vir regulon and activates quormone-quenching genes in Agrobacterium. Proc. Natl. Acad. Sci. USA. 104 (28), 11790-11795 (2007).
- Yuan, Z. -. C., Haudecoeur, E., Faure, D., Kerr, K. F., Nester, E. W. Comparative transcriptome analysis of Agrobacterium tumefaciens in response to plant signal salicylic acid, indole-3-acetic acid and γ-amino butyric acid reveals signalling cross-talk and Agrobacterium-plant co-evolution. Cell. Microbiol. 10 (11), 2339-2354 (2008).
- Li, P. L., Farrand, S. K. The replicator of the nopaline-type Ti plasmid pTiC58 is a member of the repABC family and is influenced by the TraR-dependent quorum-sensing regulatory system. J. Bacteriol. 182 (1), 179-188 (2000).
- Atkinson, M. M., Huang, J., Knopp, J. A. Hypersensitivity of suspension-cultured tobacco cells to pathogenic bacteria. Phytopathology. 75 (11), 1270-1274 (1985).
- Veena, ., Jiang, H., Doerge, R. W., Gelvin, S. B. Transfer of T-DNA and Vir proteins to plant cells by Agrobacterium tumefaciens induces expression of host genes involved in mediating transformation and suppresses host defense gene expression. Plant J. 35 (2), 219-236 (2003).
- León, J., Rojo, E., Sanchez-Serrano, J. J. Wound signalling in plants. J. Exp. Bot. 52 (354), 1-9 (2001).
- Ditt, R. F., Kerr, K. F., de Figueiredo, P., Delrow, J., Comai, L., Nester, E. W. The Arabidopsis thaliana transcriptome in response to Agrobacterium tumefaciens. Mol. Plant Microbe In. 19 (6), 665-681 (2006).
- Mandimba, G., Heulin, T., Bally, R., Guckert, A., Balandreau, J. Chemotaxis of free-living nitrogen-fixing bacteria towards maize mucilage. Plant Soil. 90 (1-3), 129-139 (1986).
- Rudrappa, T., Czymmek, K. J., Pare, P. W., Bais, H. P. Root-secreted malic acid recruits beneficial soil bacteria. Plant Physiol. 148 (3), 1547-1556 (2008).
- Lee, C. W., et al. Agrobacterium tumefaciens promotes tumor induction by modulating pathogen defense in Arabidopsis thaliana. Plant Cell. 21 (9), 2948-2962 (2009).
- Reymond, P., Weber, H., Damond, M., Farmer, E. E. Differential gene expression in response to mechanical wounding and insect feeding in Arabidopsis. Plant Cell. 12 (5), 707-720 (2000).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front. Plant Sci. 5, 252 (2014).
- Alatorre-Cobos, F., et al. An improved, low-cost, hydroponic system for growing Arabidopsis and other plant species under aseptic conditions. BMC Plant Biol. 14 (1), 69 (2014).
- Conn, S. J., et al. Protocol: Optimising hydroponic growth systems for nutritional and physiological analysis of Arabidopsis thaliana and other plants. Plant Methods. 9 (1), 4 (2013).
- Nathoo, N. . Identification of putative plant defense genes using a novel hydroponic co-cultivation technique for studying plant-pathogen interaction. , (2015).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat. Biotechnol. 22 (12), 1567-1572 (2004).
- Wise, A. A., Liu, H., Binns, A. N. Nucleic acid extraction from Agrobacterium strains. Methods Mol. Bio. 343, 67-76 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1 (3), 1559-1582 (2006).
- Salter, M. G., Conlon, H. E. Extraction of plant RNA. Methods Mol. Bio. 362, 309-314 (2007).
- Bao, Y., Wang, S., Yang, X., Li, T., Xia, Y., Meng, X. Metabolomic study of the intervention effects of Shuihonghuazi Formula, a Traditional Chinese Medicinal formulae, on hepatocellular carcinoma (HCC) rats using performance HPLC/ESI-TOF-MS. J. Ethnopharmacol. 198, 468-478 (2017).
- Korves, T. M., Bergelson, J. A developmental response to pathogen infection in Arabidopsis. Plant Physiol. 133 (1), 339-347 (2003).
- Lyons, R., Rusu, A., Stiller, J., Powell, J., Manners, J. M., Kazan, K. Investigating the association between flowering time and defense in the Arabidopsis thaliana-Fusarium oxysporum interaction. PLoS ONE. 10 (6), e0127699 (2015).
- Badri, D. V., Weir, T. L., vander Lelie, D., Vivanco, J. M. Rhizosphere chemical dialogues: Plant-microbe interactions. Curr. Opin. Biotechnol. 20 (6), 642-650 (2009).
- Baerson, S. R., et al. Detoxification and transcriptome response in Arabidopsis seedlings exposed to the allelochemical benzoxazolin-2(3H)-one. J. Biol. Chem. 280 (23), 21867-21881 (2005).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados