Method Article
Asignación simultánea y la cuantificación de ribonucleótidos en el ADN mitocondrial humano
En este artículo
Resumen
Aquí describimos un método favorable para cuantificar simultáneamente y ribonucleótidos del mapa del genoma de ADN altamente intacto en la resolución del solo-nucleótido, combinando hendidura enzimática del ADN genómico con hidrólisis alcalina y posterior 5´-final secuencia.
Resumen
Establecido métodos para estimar el número de ribonucleótidos en un genoma se limitan a la cuantificación de ribonucleótidos incorporados utilizando fragmentos cortos de ADN sintéticos o de plásmidos como plantillas y luego extrapolar los resultados a toda la genoma. Por otra parte, el número de ribonucleótidos en un genoma puede estimarse usando geles alcalinos o Southern blot. Más reciente en vivo enfoques emplean secuenciación de próxima generación que permite mapeo de genoma de ribonucleótidos, proporcionando la posición e identidad de ribonucleótidos incrustados. Sin embargo, no permiten la cuantificación de la cantidad de ribonucleótidos que se incorporan en un genoma. Aquí describimos cómo simultáneamente mapear y cuantificar el número de ribonucleótidos que se incorporan a humano mitocondrial ADN en vivo por secuenciación de próxima generación. Utilizamos ADN altamente intacto e introducir secuencia específicos de doble cadena se rompe por digestión con una endonucleasa, ribonucleótidos incorporados posteriormente hidrolización con álcali. Se unen los extremos generados con adaptadores y estos fines son ordenados en una máquina de secuenciación de próxima generación. El número absoluto de ribonucleótidos puede calcularse como el número de lecturas fuera del sitio de reconocimiento por promedio de Lee en el sitio de reconocimiento para la endonucleasa específica de secuencia. Este protocolo también puede utilizarse para mapear y cuantificar los nicks gratis en ADN y permite la adaptación a otras lesiones de ADN que pueden ser procesados a 5´ OH extremos o puntas de 5´-fosfato. Además, este método puede aplicarse a cualquier organismo, dado que existe un genoma de referencia adecuados. Por lo tanto, este protocolo proporciona una herramienta importante para estudiar la replicación del ADN, procesamiento de fin de 5´, daño de la DNA y reparación del ADN.
Introducción
En una célula eucariota, la concentración de ribonucleótidos (rNTPs) es mucho mayor que la concentración de desoxirribonucleótidos (dNTPs)1. Polimerasas de la DNA discriminarán ribonucleótidos, pero esta discriminación no es perfecta y, como consecuencia, ribonucleótidos en desoxirribonucleótidos lugar de pueden ser incorporados en genomas durante la replicación del ADN. Ribonucleótidos pueden ser los nucleótidos no canónico más común incorporados en el genoma2. La mayoría de estos ribonucleótidos se elimina durante la maduración del fragmento de Okazaki Rnasa H2 ribonucleótido inició reparación de supresión (RER) o topoisomerasa 1 (revisado en la referencia3). Ribonucleótidos permanezca estable incorporados en el ADN de2,4 y pueden afectar de maneras perjudiciales y beneficiosas (revisados en ha5). Además de ser capaces de actuar como señales positivas, por ejemplo en acoplamiento tipo interruptor en Schizosaccharomyces pombe6 y marca la cadena naciente de ADN durante el desajuste repair (MMR)7,8, ribonucleótidos afectan la 9 de la estructura y estabilidad del ADN circundante debido al grupo 2´-hidroxilo de la ribosa10, dando por resultado replicativa estrés y genoma inestabilidad11. La abundancia de ribonucleótidos en DNA genómico (gDNA) y su relevancia en la replicación y mecanismos de reparación, así como las implicaciones para la estabilidad del genoma, dar motivo para investigar su exacta ocurrencia y frecuencia de una manera de todo el genoma.
Actividad Rnasa H2 no se ha encontrado en la mitocondria humana y ribonucleótidos no eficientemente por lo tanto se eliminan en la DNA mitocondrial (mtDNA). Varias vías están involucradas en el suministro de nucleótidos a las mitocondrias humanas y para investigar si los disturbios en la piscina de nucleótidos mitocondrial causan un número elevado de ribonucleótidos en mtDNA humano, hemos desarrollado un protocolo para cuantificar y mapa Estos ribonucleótidos en el ADNmt humano aislado de los fibroblastos, las células HeLa y paciente de la célula líneas12.
Mayoría en vitro enfoques (revisados en ha comentado13) para determinar la selectividad de polimerasas de la DNA contra rNTPs se basan en experimentos de ribonucleótido sola inserción o cartilla extensión donde rNTPs competidoras están incluidos en la reacción mezcla, lo que permite la identificación o cuantificación relativa de la incorporación de ribonucleótido en corto plantillas de la DNA. Enfoques cuantitativos en secuencias cortas pueden no reflejar piscinas dNTP y rNTP en concentraciones celulares y por lo tanto proporcionar información sobre selectividad de polimerasa pero son de importancia limitada sobre genomas enteros. Se ha demostrado que la cantidad relativa de ribonucleótidos incorporados durante la replicación de una plantilla de DNA más larga, como un plásmido, puede ser visualizada en un gel de secuenciación utilizando dNTPs radiactivos e hidrolización de la DNA en un medio alcalino14. Además, se ha analizado gDNA en Southern blot después de hidrólisis alcalina, permitiendo strand-sondeo y determinación de índices absolutos de ribonucleótido incorporación en vivo15. Estos enfoques permiten una comparación relativa de la frecuencia de incorporación pero no entregan ninguna penetración en la posición o la identidad de los ribonucleótidos incorporados. Enfoques más recientes para analizar el contenido de ribonucleótido gDNA en vivo, como HydEn Seq16, ribosa-Seq17, Pu Seq18o19de emRiboSeq, se aprovecha de los ribonucleótidos incrustados sensibilidad a las alcalinas o Rnasa H2, respectivamente y secuenciación de próxima generación para identificar ribonucleótidos genoma. Estos métodos no proporcionan información sobre la frecuencia absoluta de la incorporación de ribonucleótidos detectados. Añadiendo el paso de la hendidura enzimática específica de secuencia para el protocolo de HydEn-seq, el método que Describimos aquí convenientemente extiende la información obtenida desde un enfoque de secuencia, permitiendo la asignación simultánea y cuantificación de encajado ribonucleótidos12. Este método es aplicable a prácticamente cualquier organismo dado que pueden generarse extractos de ADN muy intactos y un genoma de referencia conveniente está disponible. El método podría ser adaptado para cuantificar y determinar la ubicación de cualquier lesión que puede ser digerido por una nucleasa y deja un 5´-fosfato o un fin de 5´-OH.
Para mapear y cuantificar ribonucleótidos en la DNA genomic, el método combina el escote por una endonucleasa específica de secuencia y alcalina hidrólisis generando 5´-fosfato termina en sitios donde se encuentra la secuencia de reconocimiento específico de la endonucleasa y 5´- OH termina en posiciones donde se encontraban ribonucleótidos. Puesto que los extremos libres generados se unen posteriormente con los adaptadores y secuenciaron utilizando secuenciación de próxima generación, es de importancia utilizar ADN altamente intacto y evitar la fragmentación al azar durante la preparación de biblioteca y extracción de ADN. Evaluar estas lecturas normalizadas a la lectura en los sitios de clivaje de la endonucleasa permite una cuantificación simultánea y mapeo de los ribonucleótidos detectados. 5´-extremos libres se detectan en los experimentos de control donde la hidrólisis alcalina de ADN se sustituye por el tratamiento con KCl. Los datos adquiridos proporcionan la penetración en cantidad y ubicación de ribonucleótido y permite el análisis con respecto a la frecuencia de contenido e incorporación de ribonucleótido.
Protocolo
este protocolo se describe en la figura 1 e incluye el aislamiento del gDNA, digestión con enzimas de restricción para poder cuantificar el número de ribonucleótidos, tratamiento con álcali para hidrolizar los enlaces fosfodiéster de ribonucleótidos incorporados del gDNA, fosforilación de extremos libres 5´-OH, ligadura de ssDNA de adaptadores, segunda síntesis de strand y amplificación por PCR antes de la secuencia.
1. adaptadores y cartillas del índice
- ARC49 obtener ARC140 oligonucleótidos, ARC76/77, adaptador y ARC78-ARC107 cartillas de índice (ver tabla 1).
Nota: Oligonucleótidos deben ser purificada de HPLC. ARC76/77 se ordenan como dúplex. - Preparar 100 μm las soluciones madre de cada oligonucleótido en tampón Tris-EDTA (TE) (véase la Tabla de materiales) y almacenar a -20 ° C.
- Preparar 10 soluciones de μm de ARC67/77 y 2 μm de primers ARC49 e índice diluyendo en tampón de elución (EB; vea la Tabla de materiales). Almacenar a-20 ° C.
2. Crecimiento y cosecha de las células
- las células HeLa crecen en 70 mL Dulbecco ' s medio de Eagle modificado (DMEM) suplementado con suero bovino fetal 10% en un matraz de 250 mL cono a 37 ° C.
- Cuenta el número de las células y recoger 5 x 10 6 células en un tubo de 50 mL, centrifugar durante 5 min a 200 x g y descarte el sobrenadante.
- Lavar las células con 20 mL 1 x PBS, centrifugar durante 5 min a 200 x g y descarte el sobrenadante.
- Congelar las pelotillas a-20 ° C o continuar con la purificación de DNA a.
3. Purificación de DNA y cuantificación
- purificar gDNA usando extracción de fenol-cloroformo como se describe abajo.
- Resuspender las células en 2 mL de tampón de lisis (ver la Tabla de materiales) e incubar durante 30 min a 42 ° C en un bloque calefactor.
PRECAUCIÓN: El tampón de lisis contiene componentes peligrosos. Solución de SDS es irritante, proteinasa K sensibilizantes, irritantes y tóxicas. Use ropa protectora y guantes de. - Dividir la muestra en dos tubos de 2 mL y añadir 1 volumen (V) de fenol-cloroformo-isoamyl alcohol (25:24:1).
PRECAUCIÓN: Fenol-cloroformo-isoamyl alcohol es tóxico, mutagénico, corrosivo y peligroso a ambientes acuáticos. Utilizar en una campana de humos, use ropa protectora y guantes y desechar en la basura especial fenol-cloroformo. - Mezcla por inversión para 30-60 s y centrifugar durante 5 min a 15.000 x g a temperatura ambiente.
Nota: No no un vórtice ADN para evitar la introducción de roturas del filamento al azar, que distorsionaran los resultados. - Transferir la fase superior acuosa a un nuevo tubo de 2 mL y añadir 1 V de fenol-cloroformo-isoamyl alcohol (25:24:1).
- Mezcla por inversión y centrifugar durante 5 min a 15.000 x g a 4 ° C.
- Transferir fase superior acuosa para un nuevo 2 mL del tubo y añadir 20 μl (5 M) de NaCl y 1 V de isopropanol frío.
PRECAUCIÓN: Isopropanol es inflamable, irritante y tóxico. Almacenar en un gabinete ventilado, use guantes y ropa de protección y mantenerlo lejos de las llamas. - Mezclar por inversión e incubar durante al menos 1 h a -20 ° C.
- Centrifugar 20 min a 15.000 x g, 4 º C y desechar sobrenadante.
- Pellet de ADN lavado con 200 μL frío 70% de etanol, centrifugar durante 20 min a 15.000 x g a 4 º C y descartar el sobrenadante.
PRECAUCIÓN: el etanol al 70% es inflamable e irritante. Mantener la solución de trabajo a-20 ° C, otra tienda en gabinete ventilado, llevar ropa protectora y guantes y mantenerse alejado de las llamas. - Secar el pellet de ADN a temperatura ambiente durante 20-25 min.
- ADN disolver en 100 μl TE tampón y las muestras en un tubo de la piscina.
- Resuspender las células en 2 mL de tampón de lisis (ver la Tabla de materiales) e incubar durante 30 min a 42 ° C en un bloque calefactor.
- Concentración de cuantificar el ADN utilizando un reactivo de cuantificación de dsDNA según fabricante ' Especificaciones de s (véase la Tabla de materiales).
Nota: Utilice un reactivo de cuantificación de dsDNA, porque la cuantificación espectrofotométrica de ADN puede ser afectado por fenol residual. - Tienda ADN a-20 ° C o continuar con tratamiento HincII.
4. Hidrólisis alcalina y HincII tratamiento
- Digest 1 μg de ADN en una reacción de combinación que contiene 5 μl 10 x tampón 3.1, 1 μl (10 U) HincII y libre de nucleasas H 2 O a un volumen final de 50 μl.
Nota: Para lograr las condiciones óptimas para la ligadura, segunda síntesis de strand y la amplificación de PCR, puede ser necesario aumentar la cantidad de ADN de entrada si se espera que el ADN contiene una cantidad muy baja de ribonucleótidos. Del mismo modo, puede ser necesario disminuir la entrada ADN si el número de ribonucleótidos es muy alto. - Incubar durante 30 min a 37 ° C.
- Purificar HincII tratados ADN con perlas paramagnéticas.
Nota: Mantenga el tubo de tapas abierto en los siguientes pasos para no perturbar pelotillas con la apertura de los tubos.- Añadir 1.8 V perlas paramagnéticas a cada uno de muestra y mezclar cuidadosamente mediante pipeteo, incubar a temperatura ambiente durante 10 min
- Utilizar una rejilla magnética para pellets los granos durante 5 minutos, luego retire y deseche el sobrenadante.
- Lavar el pellet con 150 μL de etanol al 70% (temperatura ambiente) para cerca de 30 s luego quite y descarte el sobrenadante.
- Lavar el precipitado con 200 μL de etanol al 70% (temperatura ambiente) durante unos 30 s, luego retire y descarte el sobrenadante.
Nota: El etanol Residual se puede quitar con una pipeta de 10 μl. Gotas se pueden girar hacia abajo brevemente antes. - Secar las muestras a temperatura ambiente durante unos 15-20 minutos
Nota: El tiempo exacto depende del volumen de los granos y la forma de la pelotilla, por lo tanto deben comprobarse visualmente los pellets. - Retirar los tubos de la rejilla magnética y eluir pellet en 45 μl EB, mezclar mediante pipeteo cuidadosamente.
- Incubar por 5 min luego los granos en la rejilla magnética de la pelotilla y uso 45 μl de DNA purificado en paso 4.4.
- Añadir 5 μl de KOH (3 M) o KCl (3 M) del ADN creando un volumen total de 50 μl.
PRECAUCIÓN: solución 3 M de KOH es corrosivo. Use ropa protectora y guantes de.
Horno - incubar por 2 h a 55 ° C en un hibridación seguida de 5 minutos en hielo.
Nota: Se recomienda realizar el tratamiento de KOH en un horno en lugar de un bloque de la calefacción para mantener un calentamiento uniforme del tubo y evitar la condensación en la tapa de. - Precipitar el ADN agregando acetato de sodio de 10 μl (3 M, pH = 5.2) y 125 μl etanol frío de 100%. Incubar en hielo por 5 min
PRECAUCIÓN: el etanol 100% es inflamable e irritante. Un gabinete ventilado, use ropa protectora y guantes y mantenerse lejos de las llamas. - Pellets gDNA por centrifugación a 21.000 x g, 4 ° C por 5 min y descarte el sobrenadante.
- Lavado ADN pellet con 250 μl frío 70% EtOH, Centrifugue a 21.000 x g, 4 ° C por 5 min y descarte el sobrenadante.
Nota: Para quitar las gotitas, el tubo se puede girar hacia abajo brevemente otra vez y puede eliminar sobrenadante con una pipeta de 10 μl. - Dejó el sedimento seco en un tubo abierto por unos 5-10 min hasta que se evapore cualquier líquido visible.
- Disuelve de pellet de ADN dejó en 20 μl EB durante 30 min a temperatura ambiente.
5. 5´ fosforilación final
- preparar la mezcla de reacción para cada muestra por adelantado contraexistente de 2.5 μl 10 x buffer de reacción Polinucleótido quinasa de T4, 1 μl (10 U) 3´-fosfatasa-menos T4 Polinucleótido quinasa y 2,5 μl ATP (10 mM).
- De transferencia 19 μl de cada ADN de la muestra en un tubo nuevo de 200 μl y desnaturalizar 3 minutos a 85 ° C en un thermo cycler.
- ADN cool las muestras en hielo y añadir 6 μl de mezcla de reacción para cada muestra.
- Mezclas de reacción de incubar a 37 ° C por 30 minutos y una parada de la reacción por incubar las muestras a 65 ° C durante 20 min.
- Purificar ADN como se describe en 4.3, con 1,8 V de perlas paramagnéticas pero eluir en 14 μl EB.
6. ssDNA ligadura
- preparar la mezcla de reacción para cada muestra compuesta de antemano 0,5 μl ATP (2 mM), 5 μl 10 x buffer de reacción RNA T4 ligasa, 5 μl CoCl 3 (NH 3) 6 mM (10), 0,5 μl ARC140 (100 μm) y 25 μl 50% PEG 8000. Mezclar por pipeteo.
PRECAUCIÓN: CoCl 3 (NH 3) 6 es cancerígeno, sensibilización y peligrosas para el medio ambiente acuático. Use ropa protectora y guantes de. - Transferencia 13 μl de ADN purificado de paso 5.5 a un tubo nuevo de 200 μl y desnaturalizar 3 minutos a 85 ° C en un thermo cycler.
- Enfriar el ADN en el hielo y añadir 36 μl de mezcla de reacción para cada muestra, mezclar mediante pipeteo y desactivación brevemente.
- Agregar 1 μl (10 U) de ARN ligasa de T4 a cada reacción, mezclar mediante pipeteo y girar hacia abajo brevemente.
- Incubar las muestras a temperatura ambiente en la oscura noche.
7. Síntesis del segundo filamento
- Purify ligarse DNA como se describe en 4.3, pero uso 0.8 V de perlas paramagnéticas, pellets los granos durante 10 minutos y eluir en 20 μl EB.
Nota: Debido a la mayor viscosidad de la mezcla de reacción de la ligadura, el primer paso de granulación se prolonga. - Transferencia de 20 μl de muestra de ADN a un tubo nuevo de PCR de 200 μl. Repita el paso de purificación con 0.8 V de granos paramagnéticos siguiendo el fabricante ' Especificaciones de s y eluir en 14 μl EB.
- Preparar la mezcla de reacción para cada muestra por adelantado que consta de 2 μl de tampón de reacción de la polimerasa de la DNA de 10 x T7, 2 μl de ARC76/77 (2 μm), 2 μl dNTPs (2 mM) y 0,8 μl BSA (1 mg/mL).
- 12,8 transferir μl purificado ADN a un tubo nuevo de 200 μL, desnaturalizar 3 minutos a 85 ° C en un thermo cycler.
- Enfriar el ADN en el hielo y añadir 6.8 μl de mezcla de reacción para cada muestra, mezclar mediante pipeteo, desactivación brevemente e incubar 5 min a temperatura ambiente.
- Añadir 0,4 μL (4 U) T7 DNA polimerasa a cada reacción e incubar 5 min a temperatura ambiente.
- Purificar ADN como se describe en 4.3, con 0.8 V de perlas paramagnéticas y eluir en EB de 11 μl.
8. Amplificación por PCR y cuantificación de biblioteca
- Prepare la mezcla de reacción para cada muestra en un nuevo 200 tubo de μl de antemano consistente en 7.5 μl ARC49 (2 μm), cartilla de índice de 7.5 μl (2 μm, único para cada muestra) y 25 μl de 2 x arranque en caliente mezcla listo.
- Añadir 10 μl de muestra de ADN en cada reacción. Ampliar la biblioteca con las siguientes condiciones: desnaturalizar a 95 ° C por 45 s, seguido por 18 ciclos de 98 ° C durante 15 s, 65 ° C durante 30 s, 72 ° C por 30 s, terminando con una elongación final a 72 ° C por 2 minutos mantenga las muestras a 4 ° C después de la amplificación de.
- Purificar las bibliotecas como se describe en 4.3, con 0,8 V de perlas paramagnéticas y procederá a la elución en 20 μl de tampón TE.
- Cuantificar bibliotecas utilizando un reactivo de cuantificación de dsDNA, según el fabricante ' Especificaciones de s (véase la Tabla de materiales).
- Almacenar las muestras a-20 ° C o continuar con el análisis de la biblioteca.
9. Análisis de la biblioteca y Pooling
- determinar la calidad de cada biblioteca y estimar el tamaño de fragmento promedio utilizando un sistema de electroforesis digital.
Nota: El tamaño del fragmento promedio se evalúa por estimar que el área bajo la curva del electroferograma es reducido a la mitad, haciendo caso omiso de picos de los marcadores. Resultados representativos de los perfiles de biblioteca adecuado tras el tratamiento KOH o KCl se dan en la figura 2A. - Calcular la concentración (nM) de las bibliotecas como:
/(p*650) (c/10 3)] * 10 9
donde c es la concentración de la biblioteca en ng/μl y p es el tamaño de fragmento promedio en bp, como estimado en 9.1. - Piscina molares cantidades iguales de hasta 24 bibliotecas amplificadas con iniciadores de diferente índice de secuenciación. Añadir buffer TE a un volumen final de 25 μl y la concentración de 10 nM.
Nota: Dependiendo del número de bibliotecas para combinarse, se ajusta la cantidad de ADN de cada biblioteca. Si se detectaron dímeros de primer paso 9.1 como un pico distinto de unos 130 bp, el volumen final de la piscina de la biblioteca puede superar los 25 μl, ya que el paso de purificación se repite como se describe en 4.3, con 0,8 V de perlas paramagnéticas, y ADN se eluyó en 25 μl TE buf Fer.- Determine la concentración de piscina biblioteca nueva utilizando un reactivo de cuantificación de dsDNA según fabricante ' Especificaciones de s y el promedio máximo tamaño como se describe anteriormente. Proceder a la secuenciación y análisis de datos (secciones 10 y 11).
10. Secuencia
- realizar secuenciación del extremo apareado 75-base en bibliotecas combinado 12.
11. Análisis de datos
- Trim todas las lecturas para eliminar secuencias de adaptador de filtro para la calidad y leer longitud.
Nota: Esto puede hacerse usando cutadapt 1.2.1 20 con el comando ' cutadapt -f fastq - partido-leer-comodines--tranquilo -m 15 - q 10 - un NNNNNNN < archivo >', donde NNNNNNN se sustituye por la secuencia del adaptador real y < archivo > es reemplazado con el nombre del fichero fastq. - Eliminar compañeros de lecturas que fueron descartadas en el paso anterior utilizando secuencias de comandos personalizadas.
- Pares de alinear Mate 1 de restantes a un índice que contiene la secuencia de los oligonucleótidos utilizados en la preparación de la biblioteca (e.g., usando corbatín 0.12.8 21 y la línea de comandos opciones - m1-v2). Deseche todos los pares con alineaciones exitosas.
- Alinear pares restantes en el genoma de referencia de organismo con corbatín de la línea de comandos opciones - v2 -X 10000--mejor.
- Mapa Lee eso palmo entre mitocondrial molécula inicial y final alineando Mate 1 de todos los pares de no alineados (con pajarita de la línea de comandos opciones - v2).
- Determinar el recuento de fin de 5´ para todos alineaciones de la final individuales y pareadas. Desplace la posición de éstos por uno base aguas arriba hasta la posición donde estaban los ribonucleótidos hidrolizados.
- Exportar datos desde el archivo de bowtie formato a un formato de archivo bedgraph usando scripts personalizados para visualización en común browsers del genoma. Normalizar las lecturas para cada filamento a Lee por millones.
- Usando la posición y el conteo desde el archivo bedgraph, referencia la secuencia del genoma del organismo para determinar la identidad de incorpelaboradas ribonucleótidos.
Nota: Para el genoma mitocondrial humano Lee de las regiones 16.200-300 y 5.747-5.847 de cada hebra debe excluirse ya que estas regiones contienen muchos 5´-extremos libres, no relacionadas con la incorporación de ribonucleótido por ADN polimerasa γ. - Dividir la Lee total, no incluye lecturas en los once sitios de HincII, con el número promedio de lecturas por sitio HincII para obtener el número de ribonucleótidos por rotura de la hebra, (es decir, el número de ribonucleótidos por molécula mitochondrial).
Resultados
Ilustrar la metodología descritos, representante de los datos se generaron análisis de ADN mitocondrial humano de HeLa células12. Figura 2B muestra lecturas resumidas en todos sitios HincII en pesados (HS) y cadena ligera (LS) del ADNmt humano después del tratamiento de KCl (paneles de la izquierda). Alrededor del 70% de todos los 5´-extremos detectados localizar a los sitios de corte, demostrando la gran eficiencia de la digestión HincII. Tratamiento de bibliotecas con KOH para hidrolizar el ADN en ribonucleótidos incrustadas disminuye el número de lecturas en sitios HincII a alrededor del 40% (figura 2B, paneles de la derecha). Esto se espera ya que se genera gran cantidad de fines de 5´ en los sitios de la incorporación de ribonucleótido y es indicativo de una suficiente calidad de biblioteca. Figura 2 ilustra la localización y la frecuencia de los 5´-extremos (verde) de después de Lee y KCl tratamiento generados por HydEn-seq (magenta) tras el tratamiento KOH, detección de extremos libres de 5´ y extremos generados en ribonucleótidos por hidrólisis alcalina. 5´-extremos libres y ribonucleótidos localizar al HS de mtDNA humano se muestran en el panel de la izquierda y los localiza al LS se muestran en el panel derecho. Los números relativos de raw lee en ribonucleótidos (Figura 2D, panel superior) o HincII sitios (panel inferior) en HS y LS de mtDNA muestran, respectivamente, una cobertura más 14-fold o 31-fold de la LS en relación con el capítulo, mientras que no se observó una tendencia similar para ADN nuclear. Este filamento se puede explicar por la clara diferencia en la composición base de los dos filamentos e ilustra la importancia de la normalización a Lee en sitios HincII.
Normalización de lectura cuenta a HincII da una medida cuantitativa del número de ribonucleótidos por genoma mitocondrial (Figura 3A). Como se ilustra en la figura 3B, la Lee después tratamiento de KOH por cada ribonucleótido normalizado a la composición de la secuencia de cada cadena muestra una proporción diferente de 1, indicando una distribución no aleatoria de Lee sugiriendo un ribonucleótido distinto patrón y una calidad alta de la biblioteca. Que la relación se ve afectada por la digestión anterior con HincII, verificar la especificidad de la enzima clivaje. Normalizar la lee en los sitios de ribonucleótidos incrustados en sitios de clivaje HincII, así como el contenido de nucleótidos del genoma, genera una medida cuantitativa de cuántos de cada ribonucleótido se incorporan por 1.000 bases complementarias ( Figura 3).
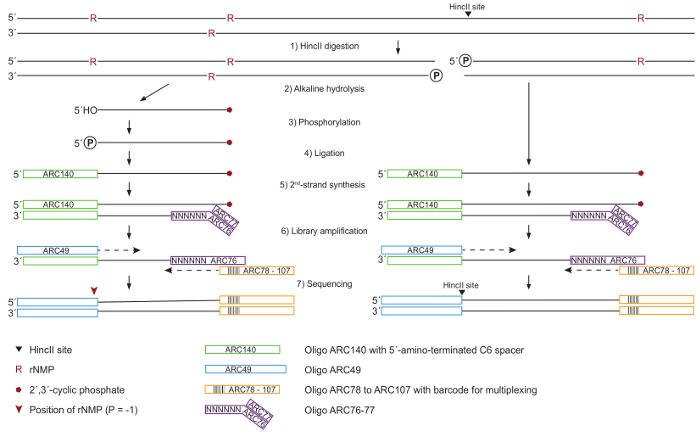
Figura 1: esquema de procesamiento de ADN y la preparación de la biblioteca. (1) extracción de ADN genómico todo es dividido por HincII para la normalización en la cuantificación posterior de ribonucleótidos, generando extremos embotados en sitios HincII (flecha negra). (2) el ADN se trata con KOH para hidrolizar en sitios de ribonucleótido, conduce a 2´, 3´-cíclico fosfato (Pentágono rojo) en los 3´-extremos y extremos gratis 5´-OH. (3) extremos 5´-OH son fosforiladas por T4 Polinucleótido kinasa 3´-fosfatasa-minus. (4) todos los 5´-extremos lleva un grupo fosfato se unen a los oligonucleótidos ARC140 por ligasa de T4 RNA. (5) la segunda cadena se sintetiza utilizando T7 polimerasa de la DNA y las ARC76-77 oligonucleótidos que contienen al azar N6 secuencias. (6) la biblioteca es amplificada por una ADN polimerasa de alta fidelidad usando ARC49 y uno de los ARC78 a ARC107 cartillas de índice que contiene un código de barras único para multiplexación. (7) 5´-extremos están ubicados por la secuencia final emparejado. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: validación de un método. Electropherograms representante (A) mediante un sistema automatizado de electroforesis para determinar la calidad de generar bibliotecas tratadas con KOH o KCl. (B) resumidos señal en sitios HincII pesados (HS) y cadena ligera (LS) mtDNA humano tras el tratamiento KOH (paneles de la derecha) o KCl (paneles de la izquierda). Circos (C) la figura de 5´-extremos libres (verde) y de HydEn-Seq (5´-extremos libres y ribonucleótidos, magentas) en HS (panel izquierdo) y mtDNA humano LS (panel derecho). Picos se normalizan a por millones Lee y el pico máximo se ajusta para el máximo número de lecturas de la biblioteca de HydEn-seq. (D) resumidos raw lee en ribonucleótidos (panel superior) y HincII sitios (panel inferior) en la pesada (H) y filamento de luz (L) en el ADNmt humano (Mito.) o a la inversa (RV) o hacia adelante en la cadena (FW) de nuclear (Nuc). DNA. Figuras B, C y D están adaptadas de referencia12. Barras de error representan el error estándar de la media. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Representante resultados. (A) el relativo número de ribonucleótidos normalizado a Lee en sitios HincII para KOH tratado bibliotecas en el pesado (H) o el filamento de la luz (L). (B) relación de identidad ribonucleótido a la composición del genoma de mtDNA para KOH tratados (KOH) y HincII troceados con KOH tratado (HincII + KOH) bibliotecas en la pesada (H) o luz (L) del filamento del ADNmt. (C) frecuencia de ribonucleótido normalizado a 1.000 bases complementarias para HincII y KOH trataron bibliotecas en el pesado (H) o el filamento de luz (L) del ADNmt. Las figuras son adaptadas de referencia12. Barras de error representan el error estándar de la media. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre | Secuencia de | |||
| ARC49 | AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCT | |||
| ARC76 | GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTNNNN * N * N | |||
| ARC77 | AGATCGGAAGAGCACACGTCTGAACTCCAGTC * A * C | |||
| ARC78 | CAAGCAGAAGACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC84 | CAAGCAGAAGACGGCATACGAGATACATCGGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC85 | CAAGCAGAAGACGGCATACGAGATGCCTAAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC86 | CAAGCAGAAGACGGCATACGAGATTGGTCAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC87 | CAAGCAGAAGACGGCATACGAGATCACTGTGTGACTGGAGTTCAGACGTGTGCTCTTCCGA | |||
Tabla 1: oligonucleótidos. Figuran los oligonucleótidos utilizados para HydEn siguientes Negrita indica indexación. * indica un vínculo fosfotioato. ARC140 contiene un grupo amino 5´ en lugar de un grupo de 5´-OH, en combinación con un enlazador de C6. Esta modificación reduce la formación de concatemers ARC140 durante la ligadura.
Discusión
Aquí presentamos una técnica para cuantificar ribonucleótidos en gDNA y mtDNA en particular, por la simple introducción de la hendidura de la DNA en sitios específicos de secuencia en el genoma como una adición al protocolo establecido de HydEn-seq y mapa simultáneamente. Aunque este estudio se centra en el mtDNA humano, el método de HydEn seq fue desarrollado originalmente en Saccharomyces cerevisiae, ilustrando la traducción del método a otros organismos12,16.
Para resultados confiables de este enfoque, cabe señalar algunos pasos críticos: (A) desde adaptadores de secuencia ligan a todos los 5´-extremos disponibles, es fundamental trabajar con ADN altamente intacto. DNA debe ser aislado y bibliotecas deben hacerse preferiblemente inmediatamente después de la extracción de ADN y el ADN puede ser almacenado a-20 ° C. No se recomienda almacenar el ADN en la nevera durante mucho tiempo o para congelar y descongelarlo repetidamente. (B) para generar bibliotecas adecuadas con este método, es fundamental para llevar a cabo el tratamiento de la KOH de la DNA en una estufa de incubación, en lugar de un bloque de calentamiento, asegurando la calefacción homogénea de toda la muestra y a la hidrólisis cuantitativa. (C) además, es fundamental para controlar la calidad de las bibliotecas antes de la puesta en común y secuenciación. El ADN debe ser cuantificado y analizado mediante un sistema de electroforesis automática garantizar cantidades adecuadas de la biblioteca de ADN, confirmar tamaños de fragmento correspondiente y busque dímeros de primer.
Para un análisis de datos significativos, también es importante tener en cuenta que el valor informativo de este método depende de los controles adecuados para evaluar fondo cuenta y sesgos de la secuencia o cadena. Habitualmente conseguimos una eficiencia de asignación en muestras de KCl de cerca de 70% cuando sólo la digestión con la endonucleasa específica de secuencia (figura 2B, paneles de la izquierda). Además, es importante confirmar que el tratamiento de la endonucleasa no afecta a la general detección de ribonucleótidos incorporados comparando HincII tratados y sin tratar las muestras (figura 3B). En estos experimentos, hemos utilizado HincII introducir cortes específicos, aunque también podrían utilizarse otras enzimas de restricción de alta fidelidad.
El protocolo podría ser adaptado para el estudio de otros tipos de lesiones del ADN que pueden ser procesadas 5´-fosfato o 5´-OH termina. La exactitud de los resultados depende de la especificidad del proceso y requiere controles adecuados (por ejemplo, el tipo salvaje o sin tratar) para verificación. Por otra parte, al adaptar este método a otras aplicaciones o para su uso con otros organismos, uno debe considerar que el método en su configuración actual requiere alrededor de 1 μg de DNA que es procesada en una biblioteca. Puesto que el número de extremos es dependiente en el número de ribonucleótidos incrustados, que varía dependiendo del organismo o mutante, muestras incluyendo un menor número de ribonucleótidos requeriría más de ADN para generar un número suficiente de extremos en la entrada del construcción de biblioteca posterior. Del mismo modo, si las muestras de ADN tienen un número mucho mayor de ribonucleótidos, también requeriría usando la DNA menos entrada para obtener las condiciones óptimas para la ligadura, la segunda síntesis de strand y amplificación por PCR. Es de destacar que la construcción de la biblioteca como se describe en este protocolo también generó datos que cubren el genoma nuclear (como se muestra en la Figura 2D) y sólo el análisis de datos se centró en mtDNA. Esto ilustra que genomas más grandes con frecuencias de ribonucleótido moderadamente inferiores también se capturan por este método.
Cuando se considera este método, ciertas limitaciones deben tenerse en cuenta: aunque este método debería, en teoría, ser aplicable a prácticamente cualquier organismo, un genoma de referencia adecuado es necesario para la adaptación de Lee. Además, los resultados obtenidos de nuestro protocolo representan las lecturas de un gran número de células. Patrones de incorporación específica ribonucleótido de un subconjunto de las células no pueden ser identificados por este enfoque. Si se asignan ribonucleótidos en genomas más grandes con un número muy bajo de ribonucleótidos, puede ser difícil discriminar ribonucleótidos de nicks al azar y por lo tanto son necesarios los controles adecuados.
El método se describe aquí, se extiende a las técnicas disponibles en vivo como HydEn Seq16, ribosa-Seq17, Pu Seq18, emRiboSeq19. Estos enfoques aprovechan de la sensibilidad de los ribonucleótidos incrustado a alcalino o Rnasa H2, respectivamente, utilizando secuenciación de próxima generación para identificar ribonucleótidos en todo el genoma, que permite su asignación y la comparación de incorporación relativa. Por unirse específicamente la secuencia del ADN, como se describe anteriormente, además de la hidrólisis alcalina en ribonucleótidos incrustados, la Lee para ribonucleótidos puede normalizada a los puntos del escote, permitiendo no sólo la identificación y mapeo de ribonucleótidos, sino también su cuantificación para cada molécula de ADN. La aplicación de nuestra técnica en el contexto de enfermedades relacionadas con la replicación del DNA, la reparación del ADN y TLS podría proporcionar una comprensión más profunda del papel de ribonucleótidos en subyacen mecanismos moleculares y la integridad del genoma en general.
Divulgaciones
Los autores declaran que no tienen intereses financieros que compiten.
Agradecimientos
Este estudio fue apoyado por el Consejo Sueco de investigación (www.vr.se) concede a arco (2014-6466 y la Fundación sueca para la investigación estratégica (www.stratresearch.se) arco (ICA14-0060). Chalmers University of Technology dio apoyo financiero a MKME durante este trabajo. Los fundadores no tenían ningún papel en el diseño del estudio, recopilación de datos y análisis, publicación o preparación del manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| 10x T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| 10x T4 RNA Ligase Reaction Buffer | New England Biolabs | B0216L | |
| 1x PBS | Medicago | 09-9400-100 | dissolve 1 tablet in H2O to a final volume of 1 L |
| 2-Propanol | Sigma-Aldrich | 33539-1L-GL-R | |
| 2100 Bioanalyzer | Agilent Technologies | G2940CA | |
| 50 mL Centrifuge Tube | VWR | 525-0610 | |
| Adenosine 5'-Triphosphate (ATP, 10 mM) | New England Biolabs | P0756S | dilute with EB to 2 mM |
| Agilent DNA 1000 Kit | Agilent Technologies | 5067-1504 | |
| BSA, Molecular Biology Grade (20 mg/mL) | New England Biolabs | B9000S | diltue with nuclease-free H2O to 1 mg/mL |
| Buffer EB | QIAGEN | 19086 | referred to as EB |
| CleanPCR paramagnetic beads | CleanNA | CPCR-0050 | |
| Deoxynucleotide (dNTP) Solution Mix (10 mM each) | New England Biolabs | N0447L | dilute with EB to 2 mM |
| DMEM, high glucose, GlutaMAX Supplement | Gibco | 61965026 | |
| DynaMag 96 Side | Thermo Fisher | 12331D | |
| Ethanol 99.5% analytical grade | Solveco | 1395 | dilute with milliQ water to 70% |
| Ethylenediaminetetraacetic acid solution (EDTA, 0.5 M) | Sigma-Aldrich | 03690-100ML | |
| Fetal bovine serum | Gibco | 10500056 | |
| HEPES buffer pH 8.0 (1 M) sterile BC | AppliChem | A6906,0125 | |
| Hexammine cobalt(III) chloride (CoCl3(NH3)6) | Sigma-Aldrich | H7891-5G | dissolve in nuclease-free H2O for 10 M solution, sterile filter. CAUTION: carcinogenic, sensitizing and hazardous to aquatic environment. |
| HincII | New England Biolabs | R0103S | supplied with NEBuffer 3.1 |
| Hybridiser HB-1D | Techne | FHB4DD | |
| KAPA HiFi HotStart ReadyMix (2X) | Kapa Biosystems | KK2602 | |
| Lysis buffer | 50 mM EDTA, 20 mM HEPES, NaCl 75 mM, Proteinase K (200 µg/mL), 1% SDS | ||
| Micro tube 1.5 mL | Sarstedt | 72.690.001 | |
| Microcentrifuge 5424R | Eppendorf | 5404000014 | |
| Microcentrifuge MiniStar silverline | VWR | 521-2844 | |
| Multiply µStripPro 0.2 mL tube | Sarstedt | 72.991.992 | |
| Nuclease-free water | Ambion | AM9937 | |
| Phenol – chloroform – isoamyl alcohol (25:24:1) | Sigma-Aldrich | 77617-500ML | |
| Potassium chloride (KCl) | VWR | 26764.232 | dissolve in nuclease-free H2O for 3 M solution, sterile filter |
| Potassium hydroxide (KOH) | VWR | 26668.296 | dissolve in nuclease-free H2O for 3 M solution, sterile filter |
| Proteinase K | Ambion | AM2546 | |
| Qubit 3.0 Fluorometer | Invitrogen | Q33216 | |
| Qubit Assay Tubes | Invitrogen | Q32856 | |
| Qubit dsDNA BR Assay Kit | Invitrogen | Q32850 | CAUTION: Contains flammable and toxic components |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | CAUTION: Contains flammable and toxic components |
| Refrigerated Centrifuge 4K15 | Sigma Laboratory Centrifuges | No. 10740 | |
| SDS Solution, 10% | Invitrogen | 15553-035 | |
| Sodium acetate buffer solution, pH 5.2, 3 M (NaAc) | Sigma-Aldrich | S7899 | |
| Sodium chloride (NaCl) | VWR | 27810.295 | dissolve in nuclease-free H2O for 5 M solution, sterile filter |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| T4 Polynucleotide Kinase (3' phosphatase minus) | New England Biolabs | M0236L | |
| T4 RNA Ligase 1 (ssRNA Ligase) | New England Biolabs | M0204L | supplied with PEG 8000 (50%) |
| T7 DNA Polymerase (unmodified) | New England Biolabs | M0274S | supplied with 10x T7 DNA Polymerase Reaction Buffer |
| TE Buffer | Invitrogen | 12090015 | |
| ThermoMixer F2.0 | Eppendorf | 5387000013 |
Referencias
- Traut, T. W. Physiological Concentrations of Purines and Pyrimidines. Mol. Cell. Biochem. 140, 1-22 (1994).
- McElhinny, S. A. N., et al. Abundant ribonucleotide incorporation into DNA by yeast replicative polymerases. Proc. Natl. Acad. Sci. USA. 107, 4949-4954 (2010).
- Williams, J. S., Lujan, S. A., Kunkel, T. A. Processing ribonucleotides incorporated during eukaryotic DNA replication. Nat. Rev. Mol. Cell Biol. 17, 350-363 (2016).
- Clausen, A. R., Zhang, S., Burgers, P. M., Lee, M. Y., Kunkel, T. A. Ribonucleotide incorporation, proofreading and bypass by human DNA polymerase delta. DNA Repair. 12, 121-127 (2013).
- Potenski, C. J., Klein, H. L. How the misincorporation of ribonucleotides into genomic DNA can be both harmful and helpful to cells. Nucleic Acids Res. 42, 10226 (2014).
- Vengrova, S., Dalgaard, J. Z. RNase-sensitive DNA modification(s) initiates S. pombe mating-type switching. Gene. Dev. 18, 794-804 (2004).
- Lujan, S. A., Williams, J. S., Clausen, A. R., Clark, A. B., Kunkel, T. A. Ribonucleotides Are Signals for Mismatch Repair of Leading-Strand Replication Errors. Mol. Cell. 50, 437-443 (2013).
- Ghodgaonkar, M. M., et al. Ribonucleotides Misincorporated into DNA Act as Strand-Discrimination Signals in Eukaryotic Mismatch Repair. Mol. Cell. 50, 323-332 (2013).
- DeRose, E. F., Perera, L., Murray, M. S., Kunkel, T. A., London, R. E. Solution Structure of the Dickerson DNA Dodecamer Containing a Single Ribonucleotide. Biochemistry. 51, 2407-2416 (2012).
- Li, Y. F., Breaker, R. R. Kinetics of RNA degradation by specific base catalysis of transesterification involving the 2 '-hydroxyl group. J. Am. Chem. Soc. 121, 5364-5372 (1999).
- McElhinny, S. A. N., et al. Genome instability due to ribonucleotide incorporation into DNA. Nat. Chem. Biol. 6, 774-781 (2010).
- Berglund, A. K., et al. Nucleotide pools dictate the identity and frequency of ribonucleotide incorporation in mitochondrial DNA. Plos Genet. 13, (2017).
- Brown, J. A., Suo, Z. C. Unlocking the Sugar "Steric Gate" of DNA Polymerases. Biochemistry. 50, 1135-1142 (2011).
- Sparks, J. L., et al. RNase H2-Initiated Ribonucleotide Excision Repair. Mol. Cell. 47, 980-986 (2012).
- Miyabe, I., Kunkel, T. A., Carr, A. M. The Major Roles of DNA Polymerases Epsilon and Delta at the Eukaryotic Replication Fork Are Evolutionarily Conserved. Plos Genet. 7, (2011).
- Clausen, A. R., et al. Tracking replication enzymology in vivo by genome-wide mapping of ribonucleotide incorporation. Nat. Struct. Mol. Biol. 22, 185-191 (2015).
- Koh, K. D., Balachander, S., Hesselberth, J. R., Storici, F. Ribose-seq: global mapping of ribonucleotides embedded in genomic DNA. Nat. Methods. 12, 251 (2015).
- Keszthelyi, A., Daigaku, Y., Ptasinska, K., Miyabe, I., Carr, A. M. Mapping ribonucleotides in genomic DNA and exploring replication dynamics by polymerase usage sequencing (Pu-seq). Nat. Protoc. 10, 1786-1801 (2015).
- Ding, J., Taylor, M. S., Jackson, A. P., Reijns, M. A. M. Genome-wide mapping of embedded ribonucleotides and other noncanonical nucleotides using emRiboSeq and EndoSeq. Nat. Protoc. 10, 1433-1444 (2015).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17, 10-12 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10, (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados