Method Article
מיפוי בו זמנית, כימות של Ribonucleotides ב- DNA מיטוכונדריאלי אנושי
In This Article
Summary
כאן נתאר שיטה לבצע בו זמנית quantitate ואת ribonucleotides מפה הגנום כולו ב- DNA מאוד שלם ברזולוציה נוקלאוטיד יחיד, המשלב המחשוף אנזימטי של דנ א גנומי עם הידרוליזה אלקליין שלה סוף-5´ הבאים . רצף.
Abstract
גישות הוקמה כדי להעריך את מספר ribonucleotides נוכח גנום מוגבלות כימות של ribonucleotides incorporated שימוש קצר שברי DNA סינטטיים או פלסמידים כתבניות והשערות ואז התוצאות לכל הגנום. לחלופין, מספר ribonucleotides נוכח גנום עשוי להיות מוערך באמצעות ג'ל אלקליין או שהכלים הדרומי. גישות ויוו עדכנית יותר מעסיקים הדור הבא רצפי ומאפשר מיפוי הגנום כולו של ribonucleotides, מספק את המיקום ואת זהותו של ribonucleotides מוטבע. עם זאת, הם אינם מאפשרים כימות של מספר ribonucleotides אשר משולבים לתוך הגנום. כאן נתאר כיצד למפות בו זמנית, quantitate מספר ribonucleotides אשר משולבים האנושי מיטוכונדריאלי DNA ויוו מאת הדור הבא רצפי. עלינו להשתמש DNA מאוד ללא פגע, להציג רצף ספציפי גדיל כפול מעברי מאת לעכל את זה עם endonuclease, לאחר מכן hydrolyzing ribonucleotides incorporated עם אלקלי. הקצוות שנוצר מאתרים עם מתאמי, הקצוות האלה הם וסודרו על מכונת רצף הדור הבא. ניתן לחשב את המספר המוחלט של ribonucleotides כמספר הקריאות באתר זיהוי לפי המספר הממוצע של קריאות באתר זיהוי עבור endonuclease ספציפית הרצף. פרוטוקול זה עשוי גם להיות מנוצל כדי למפות quantitate ניקס חינם ב- DNA ומאפשר התאקלמות למיפוי נגעים אחרים-DNA זה ניתן לעבד 5´-הו מסתיים או מסתיים 5´-פוספט. יתר על כן, שיטה זו ניתן ליישם כל אורגניזם, בהתחשב בכך גנום התייחסות מתאימה זמין. פרוטוקול זה ולכן מספק כלי חשוב ללמוד שכפול ה-DNA, עיבוד 5´-end, נזק לדנ א ו- DNA לתקן.
Introduction
בתא האיקריוטים, הריכוז של ribonucleotides (rNTPs) הוא גבוה בהרבה מאשר ריכוז deoxyribonucleotides (dNTPs)1. DNA polymerases להפלות ribonucleotides, אבל האפליה אינו מושלם, כתוצאה מכך, ribonucleotides במקום deoxyribonucleotides יכול לשלב הגנום במהלך שכפול ה-DNA. Ribonucleotides עשוי להיות נוקלאוטידים קאנונית הנפוץ ביותר שולבו את הגנום2. רוב ribonucleotides אלה יוסרו במהלך ההבשלה מקטע אוקאזקי RNase H2 ribonucleotide יזם כריתה תיקון (RER) או על ידי טופואיזומראז 1 (נבדקה הפניה3). Ribonucleotides שלא ניתן להסיר להישאר stably ועצוב ה DNA2,4 , עשוי להשפיע על זה בדרכים מזיקות וגם מועיל (נבדקה שנסקרה5). מלבד היכולת לשמש אותות חיובי, לדוגמה ב ההזדווגות סוג מחליף שמר הביקוע 6 , מסמן את הד המתהווה במהלך התאמה לתקן (MMR)7,8, ribonucleotides להשפיע מבנה9 ויציבות ב dna שמסביב עקב קבוצת 2´-הידרוקסיל שלהם ריבוז10, וכתוצאה מכך replicative מתח, הגנום יציבות11. השפע של ribonucleotides ב- DNA גנומי (gDNA) והרלוונטיות שלהם שכפול מנגנוני תיקון, כמו גם ההשלכות על יציבות הגנום, תן סיבה לחקור התרחשות מדויקת והתדירות שלהם באופן הגנום כולו.
RNase H2 פעילות לא נמצא בתוך המיטוכונדריה אנושי, ribonucleotides לכן יוסרו לא יעיל ב- DNA מיטוכונדריאלי (mtDNA). כמה מסלולים מעורבים את האספקה של נוקלאוטידים כדי המיטוכונדריה אנושי, כדי לבדוק הפרעות בבריכה נוקלאוטיד מיטוכונדריאלי לגרום מספר גבוה של ribonucleotides ב mtDNA אנושי, פיתחנו פרוטוקול כדי למפות quantitate אלה ribonucleotides ב mtDNA האנושי מבודד fibroblasts תאים הלה, התא החולה קווים12.
רוב במבחנה גישות (נבדקה שנסקרה13) כדי לקבוע סלקטיביות של ה-DNA polymerases נגד rNTPs מבוססים על ribonucleotide יחיד ההכנסה או פריימר ניסויים סיומת שבו מתחרים rNTPs הינם כלולים התגובה מערבבים, המאפשר זיהוי או כימות יחסי התאגדות ribonucleotide בתבניות הדי קצר. גישות כמותיים על רצפים קצרים עשוי לא לשקף dNTP בריכות rNTP בריכוזים הסלולר, ולכן מספקים תובנה פולימראז סלקטיביות אבל משמעות מוגבלת בנוגע הגנום כולו. הוכח, כי הכמות היחסית של ribonucleotides שולבו במהלך השכפול של תבנית ה-DNA ארוך יותר, כגון פלסמיד, ניתן לאבחן על ג'ל רצף באמצעות radiolabeled dNTPs ו hydrolyzing ה-DNA חצרו אלקליין14. יתר על כן, gDNA נותחה על שהכלים בדרום בעקבות הידרוליזה אלקליין, ומאפשר סטרנד חיטוט וספציפיים קביעת תעריפים מוחלטת של התאגדות ויווribonucleotide15. גישות אלה לאפשר השוואה יחסית של התאגדות תדירות אך לספק שום תובנות המיקום או זהות ribonucleotides incorporated. גישות עדכניות יותר כדי לנתח את התוכן ribonucleotide gDNA ויוו, כמו היידן-Seq16, ריבוז-Seq17, Pu-Seq18או emRiboSeq19, נצל את היתרון של ribonucleotides' מוטבע רגישות אלקליין או טיפול RNase H2, בהתאמה, ולהעסיק את הדור הבא רצפי לזהות ribonucleotides הגנום כולו. שיטות אלה אינם מספקים תובנה תדירות ההתאגדות המוחלט ribonucleotides שזוהו. באמצעות הוספת השלב של פצילות אנזימטי ספציפי רצף פרוטוקול היידן-seq, השיטה שנתאר כאן בנוחות מרחיב את המידע שנצבר בגישה רצף, ומאפשר מיפוי בו זמנית, כימות של מוטבע ribonucleotides12. שיטה זו ישימה לאורגניזם כמעט כל נתון כי ניתן להפיק תמציות DNA מאוד ללא פגע, גנום התייחסות מתאימה הינו זמין. השיטה יכול להיות מותאם quantitate, לקבוע את המיקום של כל הנגע יכול להיות מתעכל על ידי נוקלאז ולא יוצאת 5´-פוספט או קצה 5´-הו.
כדי למפות quantitate ribonucleotides ב- DNA גנומי, השיטה משלבת המחשוף מאת endonuclease ספציפי רצף ומסתיים הידרוליזה אלקליין יצירת 5´-פוספט אתרי שבו ממוקם רצף זיהוי ספציפיות עבור endonuclease ו 5´- הו מסתיים עמדות איפה שהו ribonucleotides. כיוון הפנויות שנוצר כתוצאה מכך מאתרים עם מתאמי וסודרו באמצעות הדור הבא רצפי, יש חשיבות להשתמש DNA ללא פגע מאוד ולמנוע פיצול אקראי במהלך הכנת מיצוי וספריית הדנ א. הערכת אלה קריאות מנורמל ל הקריאות באתרים המחשוף endonuclease מאפשר כימות סימולטני של מיפוי של ribonucleotides זיהה. 5´ חינם-קצוות זוהו בניסויים שליטה בו אלקליין הידרוליזה של ה-DNA מוחלף על ידי טיפול עם אשלגן כלורי. הנתונים שהושגו לספק תובנות ribonucleotide מיקום וכמות ומאפשר ניתוח לגבי תוכן ribonucleotide ותדירות ההתאגדות.
Protocol
פרוטוקול זה המותווה באיור 1, כולל את ניתוקה של gDNA, מערכת העיכול עם אנזימי הגבלה ביכולת quantitate מספר ribonucleotides, טיפול עם אלקליות כדי hydrolyze הקשרים phosphodiester של ribonucleotides שולבו gDNA, זרחון קצות 5´-אוו, ssDNA מצדו של מתאמי, סינתזת גדיל השני ו- PCR הגברה לפני רצף.
1. מתאמי, אינדקס תחל
- תחל אינדקס להשיג ARC49, ARC140 oligonucleotides, ARC76/77, מתאם, ARC78-ARC107 (ראה טבלה 1).
הערה: Oligonucleotides צריך להיות HPLC מטוהרים. ARC76/77 מסודרות כמו דופלקס. - להכין 100 מיקרומטר פתרונות מניות של כל oligonucleotide במאגר טריס-EDTA (TE) (ראה את הטבלה של חומרים) ולאחסן ב-20 מעלות צלזיוס
- להכין 10 מיקרומטר פתרונות של ARC67/77 ו- 2 מיקרומטר פתרונות של תחל ARC49 ואינדקס על ידי דילול במאגר • תנאי (EB; ראה הטבלה של חומרים). חנות ב-20 ° C
2. וצמיחה של הקציר של תאים
- הלה לגדל תאים ב- 70 מ ל Dulbecco ' s השתנה נשר בינוני (DMEM) בתוספת 10% סרום שור עוברית בבקבוקון טווה 250 מ ל- 37 מעלות צלזיוס.
- לספור את מספר התאים, לאסוף 5 x 10 תאים 6 בשפופרת 50-mL, צנטריפוגה למשך 5 דקות ב- 200 גרם x, ולמחוק את תגובת שיקוע.
- לרחוץ את התאים עם 20 מ ל 1 x PBS, צנטריפוגה למשך 5 דקות ב- 200 גרם x, ולמחוק את תגובת שיקוע.
- להקפיא את כדורי ב-20 ° C או להמשיך בטהרה דנ א.
3. כימות והערכה טיהור DNA
- לטהר gDNA באמצעות פנול-כלורופורם החילוץ, כפי שמתואר להלן.
- Resuspend התאים במאגר פירוק 2 מ"ל (ראה את הטבלה של חומרים) דגירה למשך 30 דקות ב 42 מעלות צלזיוס על בלוק חימום.
התראה: פירוק המאגר מכיל מרכיבים מסוכנים. פתרון מרחביות. הוא מרגיז, Proteinase K sensitizing, מעצבן, רעיל. ללבוש כפפות וביגוד מגן. - פיצול הדגימה שני צינורות 2 מ"ל ולהוסיף 1 נפח (V) של אלכוהול פנול-כלורופורם-isoamyl (25:24:1).
זהירות: אלכוהול פנול-כלורופורם-isoamyl היא רעילה, מוטגן, מאכל ו מסוכנים לסביבות הימית. להשתמש בברדס fume ללבוש כפפות וביגוד מגן, למחוק ב פסולת פנול מיוחד-כלורופורם. - מיקס על ידי היפוך עבור 30-60 s צנטריפוגה למשך 5 דקות ב- 15,000 x g בטמפרטורת החדר.
הערה: האם לא מערבולת DNA כדי למנוע הצגת מעברי סטרנד אקראיים, אשר לעוות את התוצאות. - להעביר את השלב העליון, מימית צינור 2 מ"ל ולהוסיף 1 V של אלכוהול פנול-כלורופורם-isoamyl (25:24:1).
- מיקס על ידי היפוך צנטריפוגה למשך 5 דקות ב 15,000 g x-4 מעלות צלזיוס
- להעביר את השלב העליון, מימית mL 2 חדש צינור ולהוסיף 20 µL NaCl (5 מ') ו- 1 V של אלכוהול איזופרופיל קר.
התראה: אלכוהול איזופרופיל הוא דליק, מעצבן, רעיל. אחסן אותו בארון מאוורר ללבוש כפפות וביגוד מגן, להרחיקו להבות. - מיקס על ידי היפוך דגירה במשך לפחות שעה ב-20 מעלות צלזיוס
- צנטריפוגה כעשרים דקות בכ-15,000 x g, 4 ° C ותגובת שיקוע להשליך. צנטריפוגה כעשרים דקות ב g x 15,000 ב 4 ° C
- צנפה DNA לשטוף עם 200 µL קר 70% אתנול, ולמחוק תגובת שיקוע.
התראה: 70% אתנול הוא דליק, מעצבן. לשמור על הפתרון עובד ב-20 ° C, חנות אחרת ארון מאוורר, ללבוש בגדים מגן, כפפות, ולשמור אותה הרחק להבות. - יבש בגדר DNA בטמפרטורת החדר במשך 20-25 דק...
- כדורי DNA להמיס 100 טה µL מאגר, בריכה הדגימות צינור אחד.
- Resuspend התאים במאגר פירוק 2 מ"ל (ראה את הטבלה של חומרים) דגירה למשך 30 דקות ב 42 מעלות צלזיוס על בלוק חימום.
- Quantitate DNA ריכוז באמצעות תגובה כימית כימות dsDNA על פי יצרן ' s מפרטים (ראה את הטבלה של חומרים).
הערה: השתמש ריאגנט כימות dsDNA, כי spectrophotometric כימות ה-DNA עשויה להיות מושפעת פנול שיורית. - מאגר DNA ב-20 ° C או להמשיך בטיפול HincII.
4. הידרוליזה אלקליין וטיפול HincII
- µg 1 תקציר של ה-DNA בתגובה תערובת המכילה 5 µL 10 x מאגר 3.1, µL 1 (10 U) HincII, ו- H נטולת נוקלאז 2 O לאמצעי הסופי של 50 µL-
הערה: כדי להשיג תנאים אופטימליים מצדו, סינתזת גדיל השני ו- PCR-הגברה, ייתכן צורך להגדיל את כמות ה-DNA קלט אם הוא צפוי כי ה-DNA מכיל מספר נמוך מאוד של ribonucleotides. באופן דומה, ייתכן צורך להקטין DNA קלט אם מספר ribonucleotides הוא גבוה מאוד. - תקופת דגירה של 30 דקות ב- 37 מעלות צלזיוס.
- HincII לטהר שטופלו DNA חרוזים פאראמגנטיים.
הערה: לשמור את הצינור המכסים פתח ב השלבים הבאים כדי לא להפריע כדורי על-ידי פתיחת הצינורות.- להוסיף V 1.8 של פאראמגנטיים חרוזים לכל אחד לטעום, לערבב היטב על ידי pipetting, ו דגירה בטמפרטורת החדר במשך 10 דקות
- השתמש מתלה מגנטי כדי הצניפה החרוזים במשך 5 דקות, ואז להסיר ולמחוק את תגובת שיקוע.
- לשטוף את גלולה עם 150 µL של 70% אתנול (טמפרטורת החדר) במשך כ- 30 s ולאחר מכן הסר ו להשליך תגובת שיקוע.
- שטיפת בגדר עם 200 µL של 70% אתנול (טמפרטורת החדר) כ 30 s ואז להסיר ולמחוק את תגובת שיקוע.
הערה: ניתן להסיר שאריות אתנול עם פיפטה 10 µL. טיפות יכול להיות טוו למטה בקצרה מראש. - יבש הדגימות בטמפרטורת החדר במשך כ- 15-20 דקות
הערה: הזמן המדויק תלוי בכמויות החרוזים ואת צורת בגדר, לכן כדורי יש לבדוק חזותית. - להסיר צינורות מהמדף מגנטי, elute צנפה ב 45 µL EB, מיקס על ידי pipetting בזהירות.
- דגירה במשך 5 דקות ואז הצניפה החרוזים על מתלה מגנטי ולהשתמש µL 45 של DNA מטוהרים בשלב 4.4.
- µL 5 להוסיף קו (3 מ') או אשלגן כלורי (3 מ') ל- DNA יצירת הנפח הכולל של 50 µL-
התראה: פתרון ז קו 3 הוא מאכל. ללבוש כפפות וביגוד מגן. - Incubate עבור 2 h ב 55 ° C בהכלאה תנור ואחריו 5 דקות על קרח
הערה: מומלץ לבצע את הטיפול KOH תנור במקום בלוק חימום כדי לשמור על חימום אחיד של הצינור וכדי למנוע עיבוי על המכסה. - לזרז את הדנ א על-ידי הוספת 10 µL סודיום אצטט (3 מ', pH = 5.2) ו- 125 µL קר 100% אתנול. דגירה על קרח במשך 5 דק
התראה: 100% אתנול הוא דליק, מעצבן. לאחסן בארון מאוורר, ללבוש כפפות וביגוד מגן ולשמור מן הלהבות. - גלולה gDNA על ידי צריך שתוציאו ב 21,000 x g, 4 מעלות צלזיוס למשך 5 דקות ולמחוק את תגובת שיקוע.
- DNA שטיפת גלולה עם 250 µL קר 70% צנטריפוגה ב 21,000 x g, 4 מעלות צלזיוס למשך 5 דקות, EtOH, ולמחוק את תגובת שיקוע.
הערה: כדי להסיר טיפות, הצינור יכול להיות טוו למטה בקצרה שוב ואת תגובת שיקוע ניתן להסיר עם פיפטה 10 µl. - תן בגדר יבש צינור פתוח למשך כ- 5-10 דקות עד כל נוזל גלוי התאדו.
- DNA תן התמוססות גלולה ב 20 µL EB במשך 30 דקות בטמפרטורת החדר.
5. 5´ סוף זרחון
- להכין את תערובת התגובה עבור כל דגימה מראש אסיריםisting של 2.5 µL 10 x polynucleotide קינאז התגובה מאגר T4, µL (10 U) 1 3´-פוספטאז-חיסור T4 polynucleotide קינאז, µL 2.5 ATP (10 מ מ)-
- µL 19 העברה של כל ה-DNA לטעום לתוך צינור 200 µL ו denature למשך 3 דקות ב 85 מעלות צלזיוס ב התרמו-הצנטרפוגה.
- הדי מגניב דוגמאות על קרח ולהוסיף 6 µL של התגובה לערבב כל דגימה.
- התגובה Incubate מתערבב ב 37 מעלות צלזיוס במשך 30 דקות ולהפסיק התגובה על ידי המקננת הדגימות ב 65 מעלות צלזיוס במשך 20 דק.
- לטהר דנ א כמו שמתואר 4.3, באמצעות 1.8 V של חרוזים פאראמגנטיים אבל elute ב- 14 µL EB-
6. ssDNA מצדו
- להכין את תערובת התגובה עבור כל דגימה מראש בהיקף של 0.5 µL ATP (2 מ מ), µL 5 10 x T4 RNA ליגאז התגובה מאגר, 5 µL CoCl 3 (NH 3) 6 (10 מ מ), 0.5 µL ARC140 (100 מיקרומטר), ו- 25 µL 50% 8000 פג. לערבב היטב על ידי pipetting.
התראה: CoCl 3 (NH 3) 6 הוא מסרטנים, sensitizing ומסוכנים לסביבה הימית. ללבוש כפפות וביגוד מגן. - µL 13 העברה של DNA מטוהרים צעד 5.5 צינור 200 µL ו denature למשך 3 דקות ב 85 מעלות צלזיוס ב התרמו-הצנטרפוגה.
- מגניב הדנ א על קרח, להוסיף µL 36 של תערובת התגובה אחד לטעום, לערבב על ידי pipetting ואת ספין למטה בקצרה.
- µL להוסיף 1 (10 U) של T4 רנ א ליגאז על כל תגובה, לערבב על ידי pipetting, ספין למטה בקצרה.
- דגירה בדגימות בטמפרטורת החדר ב הלילה האפל.
7. השני-strand סינתזה
- Purify מאתרים DNA כמתואר ב- 4.3, אבל להשתמש 0.8 V של חרוזים פאראמגנטיים, גלולה החרוזים 10 דקות ו elute ב- 20 µL EB-
הערה: בשל צמיגות גבוה יותר של המיקס התגובה מצדו, הצעד הראשון pelleting מוארך.
µL - 20 העברה של דנ א כדי צינור PCR 200 µL. חזור על השלב טיהור באמצעות 0.8 V של חרוזים פאראמגנטיים בעקבות היצרן ' s מפרטים ו elute ב- 14 µL EB-
- להכין את תערובת התגובה עבור כל דגימה מראש בהיקף של 2 µL 10 x T7 DNA פולימראז התגובה מאגר, 2 µL ARC76/77 (2 מיקרומטר), µL 2 dNTPs (2 מ מ), ואת µL 0.8 BSA (1 מ"ג/מ"ל).
- µL 12.8 להעביר מטוהרים דנ א כדי צינור 200 µL, denature למשך 3 דקות ב 85 מעלות צלזיוס ב התרמו-הצנטרפוגה.
- מגניב הדנ א על קרח ולהוסיף 6.8 µL של התגובה מיקס אחד לטעום, לערבב על ידי pipetting, ספין למטה לזמן קצר ולאחר תקופת דגירה של 5 דקות בטמפרטורת החדר.
- להוסיף 0.4 µL (4 U) T7 DNA פולימראז כל תגובה, תקופת דגירה של 5 דקות בטמפרטורת החדר.
- לטהר דנ א כמו שמתואר 4.3, באמצעות 0.8 V של חרוזים פאראמגנטיים ו elute ב- 11 µL EB-
8. PCR-הגברה, כימות ספריית
- להכין תערובת התגובה עבור כל דגימה ב 200 חדש µL צינור המורכב מראש 7.5 µL ARC49 (2 מיקרומטר), µL 7.5 מדד פריימר (2 מיקרומטר, ייחודי עבור כל דגימה) ולאחר 25 µL 2 x התחלה לוהטת מוכן לערבב.
- להוסיף 10 µL של דנ א כדי כל תגובה. להגביר את הספרייה באמצעות התנאים הבאים: denature ב 95 מעלות צלזיוס במשך 45 s, ואחריו 18 מחזורים של 98 ° C 15 s, 65 ° C ל 30 s, 72 ° C ל 30 s, המסתיימים התארכות הסופי ב-72 מעלות לדוגמאות להחזיק 2 דק ב 4 ° ג לאחר הגברה.
- לטהר ספריות כמתואר ב- 4.3, באמצעות 0.8 V פאראמגנטיים חרוזים, ו elute במאגר טה µL 20-
- Quantitate ספריות באמצעות ריאגנט כימות dsDNA, על פי היצרן ' s מפרטים (ראה את הטבלה של חומרים).
- לאחסן דגימות ב-20 ° C או להמשיך עם ניתוח הספרייה.
9. ספריית ניתוח ו- Pooling
- לקבוע את האיכות של כל ספריה, להעריך את הגודל הממוצע פרגמנט באמצעות מערכת אלקטרופורזה דיגיטלי.
הערה: גודל המקטע הממוצע הוא מוערך על ידי הערכת היכן האזור מתחת העקומה של electropherogram זה חצוי, התעלמות פסגות של סמנים. התוצאות נציג של ספריית מתאימים פרופילים לאחר טיפול קו או אשלגן כלורי מקבלים איור 2 א. - לחשב את הריכוז (ננומטר) של הספריות כמו:
/(p*650) (c/10 3)] * 10 9
איפה c הוא הריכוז של הספרייה ב- ng/µL ו- p מייצג גודל המקטע הממוצע ב- bp, כמו 9.1 מוערך ב- - בריכה שווה טוחנת כמויות של ספריות עד 24 מוגבר עם צבעי יסוד שונים אינדקס רצף. טה מאגר להוסיף נפח סופי של 25 µL וריכוז של 10 ננומטר.
הערה: בהתאם למספר ספריות כדי להיות איחדו, כמות ה-DNA של כל ספריה מותאמת. אם אותרו הדימרים פריימר בשלב 9.1 בתור ברורים לשיא של-130 bp, הנפח הסופי של הבריכה הספרייה יכולים לחרוג 25 µL, כי הצעד טיהור הוא חזר כמתואר ב- 4.3, באמצעות 0.8 V פאראמגנטיים חרוזים, הדנ א הוא eluted ב 25 µL TE buf fer.- לקבוע ריכוז בריכת ספריה חדשה באמצעות תגובה כימית כימות dsDNA לפי יצרן ' s מפרטים לבין הממוצע לשיא הגודל כמתואר לעיל. להמשיך רצף וניתוח נתונים (סעיפים 10 ו-11).
10. קביעת רצף
- לבצע 75-בסיס לזווג-קצה הרצף על ספריות במאגר 12.
11. ניתוח נתונים
- חיתוך כל קורא כדי להסיר את המתאם רצפים, סינון עבור איכות ולקרוא אורך.
הערה: ניתן לבצע זאת באמצעות cutadapt 1.2.1 20 באמצעות הפקודה ' cutadapt -f fastq - התאמה-קריאה-תווים כלליים - שקט -מ' 15 - q 10, אבנר כהן < קובץ >', איפה אבנר כהן מוחלף הרצף מתאם בפועל, < קובץ > הוא החליף את שם הקובץ fastq. - להסיר חברים הקריאות שנזרקו בשלב הקודם באמצעות קבצי script מותאמים אישית.
- ליישר 1 חבר של הנותרים זוגות לאינדקס המכיל את רצף כל oligonucleotides בהכנת ספריה (למשל, באמצעות עניבת הפרפר 0.12.8 21 ושורת הפקודה אפשרויות - m1-v2). למחוק את כל זוגות עם יישורים מוצלח.
- ליישר זוגות הנותרים הגנום הפניה האורגניזם באמצעות עניבת הפרפר עם שורת הפקודה אפשרויות - v2 -X הטוב 10000....
- מפה קורא את טווח בין מולקולה מיטוכונדריאלי ההתחלה והסוף על ידי יישור 1 חבר של כל זוגות unaligned (באמצעות עניבת הפרפר עם שורת הפקודה אפשרויות - v2).
- לקבוע הרוזן 5´-מסתיימת כל סוף יחיד ומשויכים במערכים. משמרת את המיקום של אלה עד לבסיס אחד במעלה הנהר עד למיקום שבו היו ribonucleotides הידרוליזה.
- יצא נתונים מהקובץ עניבת הפרפר תבנית לתבנית קובץ bedgraph באמצעות קבצי script מותאמים אישית עבור ויזואליזציה במשותף הגנום דפדפנים. לנרמל את הקריאות על גדיל אחד כדי קריאות לכל מיליון.
- באמצעות את המיקום ואת ספירות מהקובץ bedgraph, להפנות את רצף הגנום של האורגניזם כדי לקבוע את זהותו של incorporated ribonucleotides.
הערה: עבור גנום מיטוכונדריאלי האדם קורא מתוך האזורים 16,200-300 ו 5,747-5,847 על גדיל אחד לא להיות כלולים מאז אזורים אלה מכילים הרבה 5´-הפנויות שאינן קשורות ההתאגדות ribonucleotide על ידי ה-DNA פולימראז γ- - לחלק את סכום קוראת, לא כולל קריאות באתרים HincII 11, עם המספר הממוצע של קורא לכל אתר HincII כדי לקבל את מספר ribonucleotides לכל הפסקה לחוט אחד, (קרי מספר ribonucleotides לכל מולקולה מיטוכונדריאלי).
תוצאות
הממחישות את המתודולוגיה תוארו לעיל, נציג נתונים נוצרו ניתוח דנ א מיטוכונדריאלי האנושי של הלה תאים12. איור 2B מציג קריאות מסוכמים בכלל HincII אתרים כבדים (HS), אור גדיל (LS) של האדם mtDNA לאחר הטיפול אשלגן כלורי (לוחות שמאלה). בסביבות 70% של כל שזוהו 5´-מסתיים בתרגום כדי לחתוך-האתרים, הממחיש את יעילות גבוהה של עיכול HincII. בטיפול ספריות עם קו כדי hydrolyze את ה-DNA-ribonucleotides מוטבע מקטין את מספר הקריאות של אתרי HincII כ-40% (איור 2B, לוחות נכון). זה צפוי מאז מספר רב של קצוות 5´ נוצרות באתרים התאגדות ribonucleotide, מעידה על איכות הספרייה מספקת. איור 2C ממחיש את לוקליזציה ואת התדירות של 5´ שאינו נגמר (ירוק) לאחר טיפול אשלגן כלורי וקורא שנוצר מאת היידן-seq (מגנטה) לאחר טיפול קו, גילוי 5´ חינם-מסתיים והן הקצוות שנוצרו בשעה ribonucleotides על-ידי הידרוליזה אלקליין. 5´ חינם-קצוות, ribonucleotides ההתאמה לשפות אחרות כדי HS של האדם mtDNA מוצגים בחלונית השמאלית, אלה ההתאמה לשפות אחרות האם מוצגים בחלונית הימנית. המספרים היחסית הקריאות raw ribonucleotides (איור דו-ממדי, החלונית העליונה) או HincII אתרים (החלונית התחתונה) ב- HS ו- LS של mtDNA מציגים, בהתאמה, כיסוי חזק יותר 14-fold או 31-fold של הוא יחסי HS, בעוד דעה קדומה דומה נצפתה לא עבור DNA גרעיני. הנטיה סטרנד הזו עשויה להיות מוסברת על ידי ההבדל מובהק בהרכב בסיס שני הגדילים, ממחיש את החשיבות של נירמול כדי קריאות באתרים HincII.
נרמול לקריאה סופרת כדי HincII נותן מדד כמותי של מספר ribonucleotides לכל גנום מיטוכונדריאלי (איור 3 א). כמופיע ב- 3B איור, קריאות אחרי קו טיפול עבור כל ribonucleotide מנורמל להרכב רצף של גדיל אחד הראה יחס שונה מ- 1, המציין בהתפלגות שאינן אקראיות של קריאות רומז ribonucleotide נפרדים דפוס ואיכות גבוהה הספרייה. יחס זה אינו מושפע על ידי עיכול הקודם עם HincII, אימות המחשוף ירידה לפרטים של האנזים. נרמול הקורא את המקומות של ribonucleotides מוטבע לאלה באתרים פצילות HincII, כמו גם התוכן של נוקלאוטיד של הגנום, יוצרת מידה כמותית של כמה ribonucleotide כל משולבים לכל 1000 בסיסים משלימים ( איור 3C).
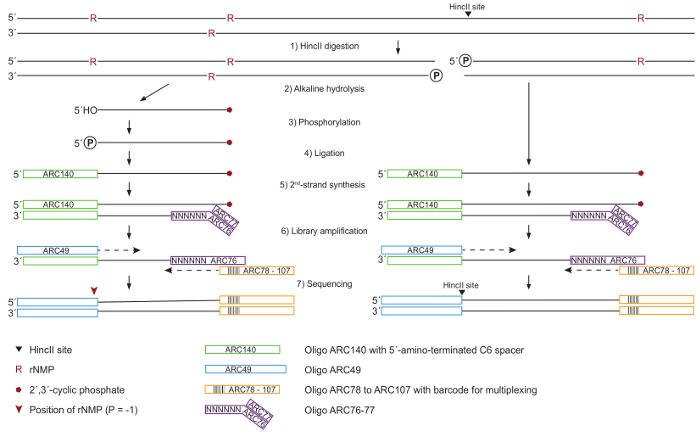
איור 1: סכימטי של ה-DNA עיבוד והכנת ספריית. (1) הדנ א כל הוא ביקע מאת HincII עבור נרמול ב כימות עוקבות של ribonucleotides, יצירת קצוות קהים באתרים HincII (ראש חץ שחור). (2) ה-DNA הוא טיפל עם קו כדי hydrolyze באתרים ribonucleotide, המוביל 2´, 3´-מחזורית פוספט (מחומש אדום)-3´-מסתיים ומסתיים 5´-אוו. (3) 5´-הו הקצוות הן phosphorylated על ידי T4 Polynucleotide קינאז 3´-פוספטאז-מינוס. (4) נגמר 5´ כל נושא קבוצת פוספט מאתרים כדי oligonucleotide ARC140 על ידי T4 RNA ליגאז. (5) יותר סטרנד השני הוא מסונתז באמצעות T7 DNA פולימראז ואת oligonucleotides ARC76-77 המכיל רצף אקראי של6 N. (6) הספרייה מוגבר על ידי אמינות גבוהה DNA פולימראז באמצעות ARC49 ו לאחד ARC78 כדי ARC107 תחל אינדקס המכיל ברקוד ייחודי עבור ריבוב. (7) 5´-הקצוות ממוקמות על ידי רצף לזווג-end. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 2: שיטת אימות. (א) electropherograms נציג שנוצר באמצעות מערכת אלקטרופורזה אוטומטית כדי לקבוע את האיכות שנוצר ספריות שטופלו אות קו או אשלגן-(B) Summarized HincII אתרים כבדים (HS), אור סטרנד (LS) mtDNA אנושי לאחר אשלגן כלורי (לוחות שמאלה) או טיפול קו (לוחות נכון). Circos (ג) איור של 5´ הפנויות (ירוק), היידן-Seq (הפנויות 5´ ו- ribonucleotides, מגנטה) HS (החלונית הימנית), mtDNA האנושי LS (לוח נכון). הפסגות הן מנורמל לכל קורא מיליון, הפסגה מרבי מותאם המספר המרבי של פעולות הקריאה של הספרייה היידן-seq. (ד) Summarized גלם קורא ribonucleotides (לוח העליון), HincII אתרים (החלונית התחתונה) כבד (H), אור (L) גדיל mtDNA אנושי (Mito.) או הפוכה (RV) או (FW) קדמי, תוך הגרעין (Nuc). ה-DNA. B, C ו- D הם ממאמרו של הפניה12. קווי שגיאה מייצגים שגיאת התקן של הממוצע. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 3: תוצאות נציג. (א) היחסית מספר ribonucleotides מנורמל כדי קריאות באתרים HincII עבור ספריות KOH שטופלו על כבד (H) או סטרנד אור (L). (B) היחס בין הזהות ribonucleotide להרכב הגנום mtDNA עבור קו (קו) ובא HincII ביקע עם קו מטופלים (HincII + קו) ספריות כבד (H) או (L) אור, לשריד mtDNA. (ג) Ribonucleotide תדירות מנורמל ל 1000 בסיסים משלימים עבור HincII ו KOH מטופלים ספריות כבד (H) או אור (L) לשריד mtDNA. דמויות מותאמים הפניה12. קווי שגיאה מייצגים שגיאת התקן של הממוצע. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
| שם | רצף | |||
| ARC49 | AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCT | |||
| ARC76 | GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTNNNN * N * N | |||
| ARC77 | AGATCGGAAGAGCACACGTCTGAACTCCAGTC * A * C | |||
| ARC78 | CAAGCAGAAGACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC84 | CAAGCAGAAGACGGCATACGAGATACATCGGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC85 | CAAGCAGAAGACGGCATACGAGATGCCTAAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC86 | CAAGCAGAAGACGGCATACGAGATTGGTCAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC87 | CAAGCAGAAGACGGCATACGAGATCACTGTGTGACTGGAGTTCAGACGTGTGCTCTTCCGA | |||
טבלה 1: Oligonucleotides. נקובים oligonucleotides המשמש היידן-תת סעיף. הפנים מודגש מציין יצירת אינדקס. * מציין כי איגרת חוב של phosphorothioate. ARC140 מכיל קבוצת אמינו 5´ במקום קבוצת 5´-הו, בשילוב עם מקשר C6. שינוי זה מפחית היווצרות ARC140 concatemers במהלך מצדו.
Discussion
כאן אנו מציגים טכניקה כדי בו-זמנית למפות לכמת ribonucleotides gDNA, ואת mtDNA בפרט, על ידי המבוא פשוטה של ביקוע DNA באתרים ספציפיים רצף הגנום תוספת פרוטוקול ה-היידן-seq הוקמה. בעוד המחקר מתמקד mtDNA אנושי, השיטה היידן-seq פותחה במקור ב שמר האפייה, הממחישות התרגום של השיטה אורגניזמים אחרים,12,16.
עבור תוצאות אמינות המתקבל גישה זו, ראוי לציין כמה צעדים קריטיים: (א) מאז רצף מתאמי מאתרים ומפסיקים את כל הקצוות 5´ זמין, חשוב לעבוד עם ה-DNA מאוד ללא פגע. DNA הנשאים ספריות צריכה להיעשות רצוי מיד לאחר בידוד ה-DNA, או ה-DNA שניתן לאחסן ב-20 ° C. לא מומלץ לאחסן DNA במקרר במשך זמן רב או כדי שוב ושוב הקפאה והפשרה של זה. (ב) כדי ליצור ספריות מתאים בשיטה זו, חשוב לבצע את הטיפול קו ה-dna תנור דגירה, במקום בלוק חימום, שמבטיח חימום הומוגנית של כל דגימה, הידרוליזה כמותית. (ג) יתר על כן, חיוני כדי לקבוע את האיכות של ספריות לפני באגירת ורצף. הדנ א צריך ניתן לכמת, נותחה באמצעות מערכת אלקטרופורזה אוטומטית כדי להבטיח כמות מספקת של ספריית ה-DNA, לאשר בגדלים המתאימים פרגמנט ובדוק הדימרים פריימר.
עבור ניתוח נתונים משמעותיים, חשוב גם לציין כי ערך אינפורמטיבי של שיטה זו הוא תלוי הפקדים המתאימים כדי להעריך ספירות הרקע והנטיות רצף או strand. אנחנו באופן שגרתי להשיג יעילות מיפוי של אשלגן כלורי דוגמאות קרוב 70% כאשר עיכול רק עם רצף ספציפי endonuclease (איור 2B, לוחות שמאלה). בנוסף, חשוב לוודא כי הטיפול endonuclease אינו משפיע הכולל זיהוי של ribonucleotides incorporated על-ידי השוואת HincII ובא לא מטופל דגימות (איור 3B). בניסויים אלה, השתמשנו HincII להציג את האתר הספציפי חתכים, למרות אנזימי הגבלה אחרים באיכות גבוהה יכול לשמש גם.
הפרוטוקול יכול להיות מותאם ללמוד סוגים אחרים של נגעים DNA זה יעובד 5´-פוספט או 5´-הו מסתיים. הדיוק של התוצאות תלוי יחודיות של עיבוד ודורש הפקדים המתאימים (למשל, פראי סוג או לא מטופל) לצורך אימות. יתר על כן, כאשר התאמת שיטה זו יישומים אחרים או לשימוש עם אורגניזמים אחרים, שצריך לשקול כי שיטת ההתקנה הנוכחית שלה דורש כ 1 µg של ה-DNA אשר מעובד בספריה. מאז מספר הקצוות של תלויה מספר ribonucleotides מוטבע, אשר משתנה בהתאם האורגניזם או מוטציה, דגימות כולל מספר נמוך יותר של ribonucleotides ידרוש יותר קלט דנ א כדי להפיק מספר מספיק של מסתיים ב ספריה הבאים בנייה. באופן דומה, אם דגימות די אן איי יש מספר גבוה יותר של ribonucleotides, זה גם ידרוש באמצעות DNA פחות קלט כדי להשיג תנאים אופטימליים מצדו השני סינתזת גדיל, הגברה PCR. ראוי לציין כי בניית ספריה כפי שמתואר פרוטוקול זה נוצר גם נתונים המכסים את הגנום הגרעיני (כפי שהוא מוצג ביישום איור דו-ממדי), רק ניתוח הנתונים היה ממוקד mtDNA. זה ממחיש כי הגנום גדול עם תדרים נמוכים למדי ribonucleotide גם בשבי על ידי שיטה זו.
כאשר בוחנים שיטה זו, מגבלות מסוימות צריכים להילקח בחשבון: אף על פי שיטה זו, תיאורטית, יהיה החלים כמעט כל אורגניזם, גנום התייחסות מתאימה יש צורך היישור של קריאות. יתר על כן, התוצאות המתקבלות מן הפרוטוקול שלנו לייצג את הקריאות בין מספר גדול של תאים. אין אפשרות לזהות דפוסי התאגדות ribonucleotide ספציפיים של קבוצת משנה של תאים על-ידי גישה זו. אם ribonucleotides ממופים ב הגנום גדולות יותר עם מספר נמוך מאוד של ribonucleotides, זה יכול להיות מאתגר להפלות ribonucleotides של ניקס אקראי, הפקדים המתאימים לכן נחוצים.
השיטה נתאר כאן, מרחיב את הטכניקות הזמינות ויוו כגון היידן-Seq16, ריבוז-Seq17, Pu-Seq18או emRiboSeq19. גישות אלה מנצלים הרגישות של ribonucleotides מוטבע אלקליין או טיפול RNase H2, בהתאמה, העסקת הדור הבא רצפי לזהות ribonucleotides הגנום כולו, אשר מאפשר מיפוי שלהם, השוואת יחסי שיתוף. על ידי ביקוע רצף הדנ א במיוחד, כפי שתוארה לעיל, בנוסף הידרוליזה אלקליין ב ribonucleotides מוטבע, הקריאות עבור ribonucleotides יכול להיות מנורמל לאתרים האלה המחשוף, ומאפשר לא רק הזיהוי ומיפוי ribonucleotides, אבל גם שלהם כימות עבור כל מולקולה DNA. היישום של הטכניקה שלנו בהקשר של מחלות הקשורות שכפול ה-DNA, תיקון ה-DNA, ו- TLS יכול לספק הבנה עמוקה יותר של התפקיד של ribonucleotides ב בבסיס המנגנונים המולקולריים ותקינות הגנום באופן כללי.
Disclosures
המחברים מצהירים כי יש להם אינטרסים כלכליים אין מתחרים.
Acknowledgements
מחקר זה נתמך על ידי מועצת המחקר השבדי (www.vr.se) מעניקה קשת (2014-6466 וקרן שבדי למחקר אסטרטגי (www.stratresearch.se) כדי ארק (ICA14-0060). האוניברסיטה הטכנולוגית של צ'אלמרס מסופקים תמיכה כספית MKME במהלך עבודה זו. התורמים שחיים היה אין תפקיד תכנון המחקר, איסוף נתונים, ניתוח, ההחלטה לפרסם או אופן ההכנה של כתב היד.
Materials
| Name | Company | Catalog Number | Comments |
| 10x T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| 10x T4 RNA Ligase Reaction Buffer | New England Biolabs | B0216L | |
| 1x PBS | Medicago | 09-9400-100 | dissolve 1 tablet in H2O to a final volume of 1 L |
| 2-Propanol | Sigma-Aldrich | 33539-1L-GL-R | |
| 2100 Bioanalyzer | Agilent Technologies | G2940CA | |
| 50 mL Centrifuge Tube | VWR | 525-0610 | |
| Adenosine 5'-Triphosphate (ATP, 10 mM) | New England Biolabs | P0756S | dilute with EB to 2 mM |
| Agilent DNA 1000 Kit | Agilent Technologies | 5067-1504 | |
| BSA, Molecular Biology Grade (20 mg/mL) | New England Biolabs | B9000S | diltue with nuclease-free H2O to 1 mg/mL |
| Buffer EB | QIAGEN | 19086 | referred to as EB |
| CleanPCR paramagnetic beads | CleanNA | CPCR-0050 | |
| Deoxynucleotide (dNTP) Solution Mix (10 mM each) | New England Biolabs | N0447L | dilute with EB to 2 mM |
| DMEM, high glucose, GlutaMAX Supplement | Gibco | 61965026 | |
| DynaMag 96 Side | Thermo Fisher | 12331D | |
| Ethanol 99.5% analytical grade | Solveco | 1395 | dilute with milliQ water to 70% |
| Ethylenediaminetetraacetic acid solution (EDTA, 0.5 M) | Sigma-Aldrich | 03690-100ML | |
| Fetal bovine serum | Gibco | 10500056 | |
| HEPES buffer pH 8.0 (1 M) sterile BC | AppliChem | A6906,0125 | |
| Hexammine cobalt(III) chloride (CoCl3(NH3)6) | Sigma-Aldrich | H7891-5G | dissolve in nuclease-free H2O for 10 M solution, sterile filter. CAUTION: carcinogenic, sensitizing and hazardous to aquatic environment. |
| HincII | New England Biolabs | R0103S | supplied with NEBuffer 3.1 |
| Hybridiser HB-1D | Techne | FHB4DD | |
| KAPA HiFi HotStart ReadyMix (2X) | Kapa Biosystems | KK2602 | |
| Lysis buffer | 50 mM EDTA, 20 mM HEPES, NaCl 75 mM, Proteinase K (200 µg/mL), 1% SDS | ||
| Micro tube 1.5 mL | Sarstedt | 72.690.001 | |
| Microcentrifuge 5424R | Eppendorf | 5404000014 | |
| Microcentrifuge MiniStar silverline | VWR | 521-2844 | |
| Multiply µStripPro 0.2 mL tube | Sarstedt | 72.991.992 | |
| Nuclease-free water | Ambion | AM9937 | |
| Phenol – chloroform – isoamyl alcohol (25:24:1) | Sigma-Aldrich | 77617-500ML | |
| Potassium chloride (KCl) | VWR | 26764.232 | dissolve in nuclease-free H2O for 3 M solution, sterile filter |
| Potassium hydroxide (KOH) | VWR | 26668.296 | dissolve in nuclease-free H2O for 3 M solution, sterile filter |
| Proteinase K | Ambion | AM2546 | |
| Qubit 3.0 Fluorometer | Invitrogen | Q33216 | |
| Qubit Assay Tubes | Invitrogen | Q32856 | |
| Qubit dsDNA BR Assay Kit | Invitrogen | Q32850 | CAUTION: Contains flammable and toxic components |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | CAUTION: Contains flammable and toxic components |
| Refrigerated Centrifuge 4K15 | Sigma Laboratory Centrifuges | No. 10740 | |
| SDS Solution, 10% | Invitrogen | 15553-035 | |
| Sodium acetate buffer solution, pH 5.2, 3 M (NaAc) | Sigma-Aldrich | S7899 | |
| Sodium chloride (NaCl) | VWR | 27810.295 | dissolve in nuclease-free H2O for 5 M solution, sterile filter |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| T4 Polynucleotide Kinase (3' phosphatase minus) | New England Biolabs | M0236L | |
| T4 RNA Ligase 1 (ssRNA Ligase) | New England Biolabs | M0204L | supplied with PEG 8000 (50%) |
| T7 DNA Polymerase (unmodified) | New England Biolabs | M0274S | supplied with 10x T7 DNA Polymerase Reaction Buffer |
| TE Buffer | Invitrogen | 12090015 | |
| ThermoMixer F2.0 | Eppendorf | 5387000013 |
References
- Traut, T. W. Physiological Concentrations of Purines and Pyrimidines. Mol. Cell. Biochem. 140, 1-22 (1994).
- McElhinny, S. A. N., et al. Abundant ribonucleotide incorporation into DNA by yeast replicative polymerases. Proc. Natl. Acad. Sci. USA. 107, 4949-4954 (2010).
- Williams, J. S., Lujan, S. A., Kunkel, T. A. Processing ribonucleotides incorporated during eukaryotic DNA replication. Nat. Rev. Mol. Cell Biol. 17, 350-363 (2016).
- Clausen, A. R., Zhang, S., Burgers, P. M., Lee, M. Y., Kunkel, T. A. Ribonucleotide incorporation, proofreading and bypass by human DNA polymerase delta. DNA Repair. 12, 121-127 (2013).
- Potenski, C. J., Klein, H. L. How the misincorporation of ribonucleotides into genomic DNA can be both harmful and helpful to cells. Nucleic Acids Res. 42, 10226 (2014).
- Vengrova, S., Dalgaard, J. Z. RNase-sensitive DNA modification(s) initiates S. pombe mating-type switching. Gene. Dev. 18, 794-804 (2004).
- Lujan, S. A., Williams, J. S., Clausen, A. R., Clark, A. B., Kunkel, T. A. Ribonucleotides Are Signals for Mismatch Repair of Leading-Strand Replication Errors. Mol. Cell. 50, 437-443 (2013).
- Ghodgaonkar, M. M., et al. Ribonucleotides Misincorporated into DNA Act as Strand-Discrimination Signals in Eukaryotic Mismatch Repair. Mol. Cell. 50, 323-332 (2013).
- DeRose, E. F., Perera, L., Murray, M. S., Kunkel, T. A., London, R. E. Solution Structure of the Dickerson DNA Dodecamer Containing a Single Ribonucleotide. Biochemistry. 51, 2407-2416 (2012).
- Li, Y. F., Breaker, R. R. Kinetics of RNA degradation by specific base catalysis of transesterification involving the 2 '-hydroxyl group. J. Am. Chem. Soc. 121, 5364-5372 (1999).
- McElhinny, S. A. N., et al. Genome instability due to ribonucleotide incorporation into DNA. Nat. Chem. Biol. 6, 774-781 (2010).
- Berglund, A. K., et al. Nucleotide pools dictate the identity and frequency of ribonucleotide incorporation in mitochondrial DNA. Plos Genet. 13, (2017).
- Brown, J. A., Suo, Z. C. Unlocking the Sugar "Steric Gate" of DNA Polymerases. Biochemistry. 50, 1135-1142 (2011).
- Sparks, J. L., et al. RNase H2-Initiated Ribonucleotide Excision Repair. Mol. Cell. 47, 980-986 (2012).
- Miyabe, I., Kunkel, T. A., Carr, A. M. The Major Roles of DNA Polymerases Epsilon and Delta at the Eukaryotic Replication Fork Are Evolutionarily Conserved. Plos Genet. 7, (2011).
- Clausen, A. R., et al. Tracking replication enzymology in vivo by genome-wide mapping of ribonucleotide incorporation. Nat. Struct. Mol. Biol. 22, 185-191 (2015).
- Koh, K. D., Balachander, S., Hesselberth, J. R., Storici, F. Ribose-seq: global mapping of ribonucleotides embedded in genomic DNA. Nat. Methods. 12, 251 (2015).
- Keszthelyi, A., Daigaku, Y., Ptasinska, K., Miyabe, I., Carr, A. M. Mapping ribonucleotides in genomic DNA and exploring replication dynamics by polymerase usage sequencing (Pu-seq). Nat. Protoc. 10, 1786-1801 (2015).
- Ding, J., Taylor, M. S., Jackson, A. P., Reijns, M. A. M. Genome-wide mapping of embedded ribonucleotides and other noncanonical nucleotides using emRiboSeq and EndoSeq. Nat. Protoc. 10, 1433-1444 (2015).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17, 10-12 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10, (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved