Method Article
Ejecutar consultas de mayor complejidad relacional (MySQL) y NoSQL (MongoDB y existen) crecen de tamaño ISO/EN 13606 EHR estandarizadas las bases de datos
En este artículo
Resumen
Este estudio compara relacionales y no relacionales (NoSQL) normalizar sistemas de información médica. La complejidad computacional de los tiempos de respuesta de consulta de dichos sistemas de gestión de base de datos (DBMS) se calcula utilizando bases de datos de tamaño doble. Estos resultados ayudan a la discusión de la idoneidad de cada enfoque de base de datos a diferentes escenarios y problemas.
Resumen
Esta investigación muestra un protocolo para evaluar la complejidad computacional de consultas relacional y no relacionales (NoSQL (no sólo lenguaje de interrogación estructurado)) estandarizados sistemas de bases de datos de información médica electrónicos de salud (EHR) de registro (DBMS). Utiliza un conjunto de tres tamaño duplicar bases de datos, es decir, bases de datos de almacenamiento de 5000, 10.000 y 20.000 realistas estandarizado extractos de HCE, en tres sistemas de gestión de base de datos (DBMS): relacional MySQL el Mapeo objeto-relacional (ORM), basado en documentos NoSQL MongoDB y lenguaje de marcado extensible nativos existen NoSQL (XML).
Se calcularon los tiempos de respuesta promedio a seis consultas de mayor complejidad, y los resultados mostraron un comportamiento lineal en los casos de NoSQL. En el campo de NoSQL MongoDB presenta una pendiente lineal mucho más plana que existen.
Sistemas NoSQL también pueden ser más apropiados mantener sistemas de información médica estandarizados debido a la especial naturaleza de las políticas de actualización de la información médica, que no debe afectar a la consistencia y la eficiencia de los datos almacenados en bases de datos NoSQL.
Una limitación de este protocolo es la falta de resultados directos de la mejora de los sistemas relacional como mapeo relacional de arquetipo (brazo) con los mismos datos. Sin embargo, la interpolación de resultados de base de datos de tamaño doblar a los presentados en la literatura y otros resultados publicados sugieren que los sistemas NoSQL sería más apropiados en muchos escenarios específicos y problemas a resolver. Por ejemplo, NoSQL puede ser apropiado para tareas basadas en documentos como extractos her utilizados en clínica, edición y visualización o situaciones donde el objetivo es no sólo a la información médica de la consulta, sino también a restaurar el EHR en exactamente su forma original.
Introducción
NoSQL (no solo SQL) DBMS han surgido recientemente como una alternativa para el DBMS relacional tradicional (RDMBS). RDBMS han dominado la forma de los datos fueron almacenados en los sistemas de base de datos durante décadas. Cálculo y álgebra relacional bien estudiado y entendido han garantizado la eficiencia y la consistencia de RDBMS1. Sistemas NoSQL no serán sustitutos de sistemas relacionales, pero podría comportarse de manera ventajosa en ciertos escenarios y en diferentes condiciones.
Algunos de estos escenarios particulares y condiciones ocurriría en el diseño de la persistencia de la base de datos de los sistemas de registro de salud electrónico (EHR) para administrar y almacenar información médica. Para ser interoperables y sostenible en la práctica, varias normas internacionales como ISO/EN 13606, openEHR, HL72,3,4,5 se han utilizado para estandarizar extractos de HCE.
Varios estándares tales como ISO/EN 13606 y openEHR han separado la información y el conocimiento en dos diferentes niveles de abstracción, representado por el modelo de referencia (RM) y estructuras de datos especiales llamadas arquetipos. Esta separación a menudo es llamada el modelo dual y permite sistemas EHR que conocimientos médicos y semánticamente interoperable para evolucionar sin reprogramar todo el sistema EHR y, en consecuencia, a ser mantenible y sostenible en la práctica6 . Sin embargo, el modelo dual implementado en sistemas EHR estandarizados requiere que la organización de la información sigue una estructura específica, y esto tiene profundas consecuencias en el nivel de persistencia de base de datos del sistema es diseñado7.
Objeto relacional Mapping (ORM)8 es una metodología para implementar un sistema de HME utilizando el paradigma de base de datos relacional. ORM mapas exhaustivamente la estructura de archivos XML (eXtensible Markup Language) extractos EHR estandarizados utilizados por el sistema de base de datos relacional. ORM construye muchas tablas relacionales exhaustivamente siguiendo la estructura de los archivos XML estandarizados de EHR extractos. Estas tablas relacionales están relacionadas a través de muchas de las claves externas y el sistema resultante puede no ser muy eficiente.
Se han propuesto varias mejoras relacionales ORM. Nodo + ruta9 de openEHR reduce el número de tablas relacionales serializar subárboles del extracto entero archivo XML en BLOBs (objetos grandes binarios). Sin embargo, esta simplificación causa lógica compleja recuperación, dañar consultas complejas. Arquetipo de mapeo relacional (brazo)10 genera un modelo de base de datos impulsado por arquetipos, construcción de un nuevo esquema relacional basado en asignaciones entre arquetipos y tablas relacionales. En consecuencia, la información no médica del extracto de HCE se pierde.
Muchas bases de NoSQL basada en documento almacenan documentos todo como BLOBs todos respetando una original XML o JSON (JavaScript Object Notation) formato. Esto significa que no se construyen tablas relacionales. Estas bases de datos NoSQL no tienen ningún esquema y no soportan que se une o (ácido) propiedades11, es decir, atomicidad, consistencia, aislamiento o durabilidad. Como resultado, pueden ser muy ineficientes si un elemento de un documento hace referencia a elementos del mismo u otros tales documentos utilizando un enlace de direccionamiento indirecto. Esto sucede porque, con el fin de mantener la coherencia, la totalidad de los documentos de referencias tienen que ser procesadas secuencialmente. Sin embargo, una base de datos no relacionales puede todavía apropiada si la principal tarea desempeñada por el SGBD es una tarea basada en el documento. Esto es porque los datos pueden permanecer en una forma más estrechamente aproximar su representación verdad usando una base de datos NoSQL basada en el documento, aunque esto también es debido a las políticas de la especial persistencia logradas por documentos médicos de EHR (véase la sección discusión).
El propósito de estos métodos es mostrar varios experimentos para comparar la aplicación de la capa de persistencia de un sistema de HCE estandarizada utilizando tres diferentes DBMS: uno relacional (MySQL) y dos NoSQL (basadas en documentos MongoDB y XML nativo). Su complejidad computacional ha sido calculado y comparado usando tres diferentes bases de tamaño creciente y seis distintas consultas de mayor complejidad. Los servidores de base de tres datos han instalado y configurado localmente en el mismo ordenador donde se ha ejecutado la consulta. Ver la Tabla de materiales por técnicas.
Experimentos de concurrencia también se han realizado para comparar el rendimiento de MySQL y de NoSQL MongoDB DBMS relacionales. Las mejoras descritas de ORM (nodo + ruta y brazo) también han sido comparadas con resultados apropiados relevantes de la literatura10.
Sistemas de gestión de bases de datos están evolucionando continuamente a un ritmo acelerado. Nadie podría pensar acerca de este desarrollo exponencial cuando el único paradigma vigente era el modelo relacional. Por poner un ejemplo, véase por ejemplo12, donde se propone un modelo para implementar el tiempo de respuesta mejor bases de datos relacionales conservando las propiedades de ácido.
Protocolo
1. construir un SGBD relacional MySQL para almacenar bases de datos de extractos de HCE estandarizada tamaño doble tres
- Importar el W3C (World Wide Web Consortium) esquema de XML correspondiente a la ISO/EN13606 RM y los tipos de datos ISO21090 en un 'IDE Java' (entorno de desarrollo integrado).
Nota: ISO significa International Standards Organization. EN los soportes para la norma europea. - Ejecutar la JAXB (Java XML Binding) Plug-in en el IDE; Esto produce clases Java correspondientes a la estructura de los elementos de los archivos XML de extractos de HCE.
- La etiqueta manualmente las clases Java con etiquetas JPA. Estas etiquetas se refieren a los cardinalities y otras relaciones entre las tablas relacionales de la base de datos MySQL.
- Importar las bibliotecas de la JPA (Java API de persistencia) en el IDE y ejecutar el método que se construye una base de datos MySQL de las clases de Java etiquetadas.
- Crear tres directorios con extractos de HCE realista de 5.000, 10.000 y 20.000 archivos XML.
- Ejecutar el método JPA para cargar un extracto XML en el DBMS MySQL en todos los extractos de la guía de 5.000 extractos.
- Repita el paso 1.6 dos veces, una vez con el directorio de 10.000 extractos y una vez con el directorio de extractos de 20.000.
2. construir un DBMS de NoSQL MongoDB para almacenar bases de datos de extractos de HCE estandarizada tamaño doble tres
- Proceso de cada uno de los tres directorios que contiene 5.000, 10.000 y 20.000 realistas EHR extractos de archivos XML con un programa estándar para convertir archivos XML a archivos JSON, como json.org.XML. Tres directorios con archivos JSON de 5.000, 10.000 y 20.000 deben ser producidos.
- Lanzar MongoDB GUI (interfaz gráfica de usuario, véase Tabla de materiales).
- Lanzamiento de MongoDB 2.6 servidor ejecutando el programa mongod desde una ventana de DOS (Disk Operating System) sistema.
- Conecte la interfaz gráfica de MongoDB al servidor localhost puerto 27017.
- Seleccione el menú "conectar".
- Escriba un nombre para la conexión (por ejemplo ' primero').
- Escriba localhost:27017 en el cuadro de texto servidor de DB.
- Pulse el botón "Connect"; un árbol con las bases de datos actuales debe aparecer en la izquierda.
- Construir una base de datos que contiene 5.000 EHR estandarizado extractos.
- Haga clic en el nombre de la conexión en la parte superior del árbol a la izquierda.
- Seleccione el menú "archivo".
- Elija "añadir base de datos".
- Introduzca el nombre de la base de datos en el cuadro de diálogo que aparece.
- Haga clic en Aceptar.
- Construir una colección que contiene 5.000 EHR estandarizado extractos.
- Haga clic en el nombre de la base de datos en el árbol de la izquierda.
- Seleccione el menú "base de datos".
- Elija "AddCollection".
- Introduzca el nombre de la colección en el cuadro de diálogo que aparece.
- Haga clic en " crear".
- Haga clic en el nombre de la colección.
- Seleccione el menú "importar".
- Seleccione el botón de radio ''JSON - shell mongo / / mongoexport ".
- Haga clic en "siguiente".
- Presione el botón "Agregar archivos de código fuente".
- Navegar en la computadora usando el cuadro de diálogo.
- Abra el directorio que contiene 5.000 JSON extraer archivos.
- Seleccionar todos los archivos en el directorio.
- Pulsa "Open". La lista de archivos JSON debe aparecer en el cuadro de diálogo de importación.
- Pulse "siguiente"; en la izquierda aparece una vista previa de la nueva colección en la base de datos.
- Pulse "siguiente".
- Presione "iniciar importación". El progreso de la importación aparece abajo a la izquierda, indicando el número de archivos importados y el tiempo transcurrido.
- Repita los pasos 5 y 6 para construir una colección de 10.000 EHR estandarizado extractos.
- Repita los pasos 5 y 6 para construir una colección de 20.000 EHR estandarizado extractos.
3. construir un NoSQL existen DBMS a tienda tres doble tamaño estandarizado EHR extractos de bases de datos
- Lanzamiento de la base de datos existen .
- Mediante el icono de la base de datos, abra al cliente de administrador de Java.
- Introduzca la contraseña de admin.
- Pulse el botón "Connect".
- Construir una colección que contiene 5.000 EHR estandarizado extractos.
- En la barra de herramientas, seleccione el menú "crear una nueva colección".
- En el cuadro de diálogo que aparece, escriba el nombre de la nueva colección.
- Haga clic en "Aceptar"; aparece la nueva colección.
- Seleccione el nombre de la colección.
- En la barra de herramientas, seleccione el menú "archivos de la tienda en la base de datos".
- Navegar en la computadora usando el cuadro de diálogo.
- Seleccione el directorio que contiene 5.000 archivos de extracto XML estandarizados.
- Haga clic en el botón "Seleccione los archivos o directorios para almacenar". Observe que aparece un cuadro de diálogo que muestra el progreso, los archivos están almacenados y el porcentaje de la base de datos creada.
- Repita el paso 5 para crear una colección que contiene 10.000 EHR estandarizado extractos.
- Repita el paso 5 para crear una colección que contiene 20.000 EHR estandarizado extractos.
4. diseñar y ejecutar en las bases de datos relacionales MySQL 3 6 consultas de mayor complejidad
- Diseño de seis consultas de mayor complejidad según los arquetipos utilizados por los extractos de HCE.
- Programa una secuencia de comandos SQL con la primera consulta en la base de datos MySQL. La SQL debe adaptarse a la estructura especial de la base de datos MySQL debido a la estandarización de extractos (arquetipos). La base de datos de mapas de toda la estructura de los extractos. Como resultado, la consulta SQL es bastante compleja.
- Identificar los atributos de las bases de datos que serían acelerar el tiempo de respuesta de las consultas si un índice se construyó sobre ellas y, a continuación construcción de tales índices, aunque la mayoría de los índices se construyen automáticamente por el DBMS.
- Si una consulta necesita un índice automáticamente no construido, construir manualmente.
- Conectarse al servidor de MySQL (suplementario Figura 1).
- Seleccione y haga clic en el nombre de la base de datos a la izquierda.
- Seleccione y haga clic en la tabla relacional donde reside el campo indizado.
- Haga clic en la pestaña "estructura".
- Seleccione y haga clic en la columna donde se construirá el índice.
- Haga clic en "Índice". Tenga en cuenta que la sentencia SQL, el índice de construcción aparece, y aparece un mensaje que indica que la oración se ha construido con éxito.
- Ejecutar la primera consulta.
- Seleccione y haga clic en el nombre de la base de datos a la izquierda.
- Haga clic en la pestaña "SQL".
- Escriba o pegue el código SQL de la primera consulta (ver figura 2 complementaria).
- Pulse "continuar". Observe que aparece la primera pantalla de la lista de resultados, junto con un mensaje con el tiempo de ejecución de la consulta.
- Repita la ejecución 5 veces y calcular el tiempo medio de respuesta.
- Repita el paso 5 con consultas de 2 a 6.
- Ver todo el proceso tres veces, con las bases de datos de extractos de 5.000, 10.000 y 20.000.
5. diseñar y ejecutar en las bases de datos NoSQL MongoDB 3 6 consultas de mayor complejidad
- Lanzar la GUI de MongoDB (véase Tabla de materiales).
- Iniciar el servidor de MongoDB 2.6 ejecutando el programa mongod desde una ventana DOS del sistema (ver figura 3 complementaria).
- Siga el paso 2.4 para conectar la interfaz gráfica de MongoDB al servidor localhost puerto 27017.
- Seleccione y expanda la base de datos MongoDB en el lado izquierdo.
- Seleccione la colección.
- Haga clic en el menú de "colección" en la barra de herramientas.
- Ejecutar la primera consulta de MongoDB.
- Haga doble clic en el botón de "Query Builder".
- Haga doble clic en el "campo de consulta" del generador de consultas en la derecha.
- Escriba el campo de la consulta de MongoDB en el campo cuadro de texto del panel de consulta (ver complementarios figura 4).
- Escriba el valor de la consulta de MongoDB en el valor cuadro de texto del panel de consulta.
Nota: Esta consulta debe ser algo como {"ns3:EHRExtract.allCompositions.content.items.parts.parts.name.ns2:originalText. valor":"Descripcion"}. El campo el valor cotizado y son separados por punto y coma. - Haga doble clic en el campo de la proyección del generador de consultas
- Escriba la primera proyección en el cuadro de texto de proyección (ver complementarios Figura 5).
- Haga doble clic en el campo de proyección para añadir un nuevo cuadro de texto de proyección.
- Escriba la segunda proyección en el cuadro de texto de proyección.
Nota: Una proyección selecciona una parte del documento obtenido por la consulta. Debe ser algo como {"ns3:EHRExtract. allCompositions.content.items.parts.parts.value.value": 1} y {" ns3: EHRExtract.all Compositions.content.items.parts.parts.value.nullFlavor ": 1} - Haga clic en el botón de play azul para ejecutar la consulta.
- Visualizar el código de consulta en la pestaña código de consulta.
- Ver los detalles del resultado en la ficha explicar: número de resultados, tiempo de ejecución en milisegundos.
- Ver, ampliar y examinar los resultados en la pestaña de resultado.
- Si se requiere más procesamiento de la consulta, escriba un programa en Java con el driver de MongoDB Java con la consulta y un método para procesar los resultados.
- Repita la ejecución 5 veces y calcular el tiempo medio de respuesta.
- Paso de 5.7 para las 2 restantes a través de 6 consultas.
- Repetir todo el proceso en los 5.000, 10.000 y 20.000 extractos de bases de datos MongoDB.
6. diseñar y ejecutar en los 3 NoSQL existen consultas de complejidad cada vez mayor de bases de datos 6
- Lanzar el existen DBMS.
- Abra al cliente de administrador de Java.
- Presione el botón "conectar a la base de datos".
- Seleccione la base de datos y haga clic en él.
- Haga clic en el menú "consultar base de datos de XPath"; aparece el cuadro de diálogo de consulta.
- Ejecutar la consulta XPath de primera (ver complementarios Figura 6).
- Escriba o pegue el código XPath de la primera consulta en la parte superior del cuadro de diálogo.
- Haga clic en el menú "Ejecutar" en la barra de herramientas del cuadro de diálogo.
- Ver resultados de XML mediante la ficha "XML" en la parte inferior del cuadro de diálogo.
- Ver el número de resultados de compilación y tiempo de ejecución en la parte inferior del cuadro de diálogo.
- Repita la ejecución 5 veces y calcular el tiempo medio de respuesta.
- Repita el paso 6 para consultas de 2 a 6.
- Hacer todo el proceso tres veces, para los extractos de 5.000, 10.000 y 20.000 existen bases de datos.
7. diseñar y ejecutar un experimento de concurrencia con el MySQL y MongoDB 5.000 extractos de bases de datos
Nota: Se ha eliminado la base de datos existen del experimento en este momento debido a la peor performance en los experimentos anteriores.
- Seleccione las consultas con las tres respuestas de tiempo más cortas en los experimentos anteriores utilizando las bases de datos de 5.000 extractos (típicamente debajo de varios segundos).
- Identificar y construir manualmente índices de atributos apropiados para las consultas, si es necesario.
- Programa dos aplicaciones multihilo de Java, uno para MySQL y el otro para MongoDB; cada aplicación tendrá tres subprocesos de prioridad diferente, uno para cada consulta seleccionada en el paso 1.
- Ejecutar y calcular las CPU (Unidad Central de procesamiento) usar la distribución para cada subproceso (consulta).
- Ejecutar cada aplicación multiproceso, haga clic en el botón ejecutar cinco veces durante cada período de 10 minutos y calcular la más ejecutada (máxima prioridad) consulta rendimiento promedio y el tiempo promedio de las tres consultas.
- Ver las consultas de ejecución, con prioridades y tiempo de ejecución.
- Calcular el rendimiento promedio y tiempo medio de respuesta de cada una de las tres consultas.
Resultados
Seis distintas consultas en realista extractos estandarizados de EHR que contiene información sobre los problemas de los pacientes, incluyendo sus nombres, las fechas iniciales y finales y la gravedad, se muestran en la tabla 1.
Tiempos de respuesta promedio de las seis consultas en las tres bases de datos de tamaño doble en cada DBMS se muestran en las tablas 2-4. Las figuras 1-6 muestran los mismos resultados gráficamente (aviso que los ejes verticales utilizan escalas muy diferentes a lo largo de estas cifras).
El fuerte comportamiento lineal de la complejidad computacional es evidente a lo largo de todas las consultas de las bases de datos NoSQL, aunque con la precaución apropiada debido al tamaño relativamente pequeño de los 3 conjuntos de datos utilizados. Sin embargo, la base de datos relacional ORM muestra un claro comportamiento lineal. La base de datos MongoDB tiene una pendiente mucho más plana que la base de datos existen.
Resultados de la mejora de los sistemas relacional discutida en la introducción Publicada en la literatura pueden encontrarse en la tabla 5. Interpolando MongoDB resultados del cuadro 3 con consultas similares y tamaños de base de datos de los resultados del brazo de tabla 5 es igual a ambos sistemas de bases de datos en la Q1, pero favorece a MongoDB en Q3.
Los resultados de los experimentos de simultaneidad se pueden encontrar en la tabla 5 y tabla6. MongoDB late MySQL tanto en tiempo de respuesta y rendimiento. De hecho, MongoDB se comporta mejor en concurrencia que en aislamiento y se erige como una impresionante base de datos en ejecución concurrente.
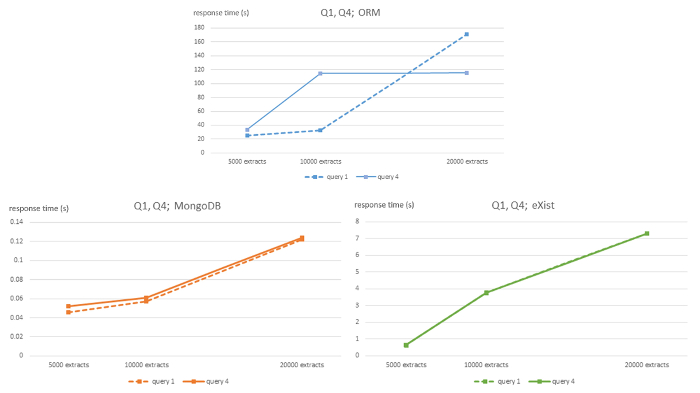
Figura 1 : Complejidad algorítmica de ORM MySQL, MongoDB, existen DBMS para consultas Q1 y Q4 y. Esta cifra se modificó de7 con licencia de Creative Commons (http://creativecommons.org/ licenses/by/4.0/) y muestra los tiempos de respuesta en segundos para 5.000, 10.000 y 20.000 tamaño EHR extractos de bases de datos para cada SGBD y consultas Q1 y Q4. Haga clic aquí para ver una versión más grande de esta figura.
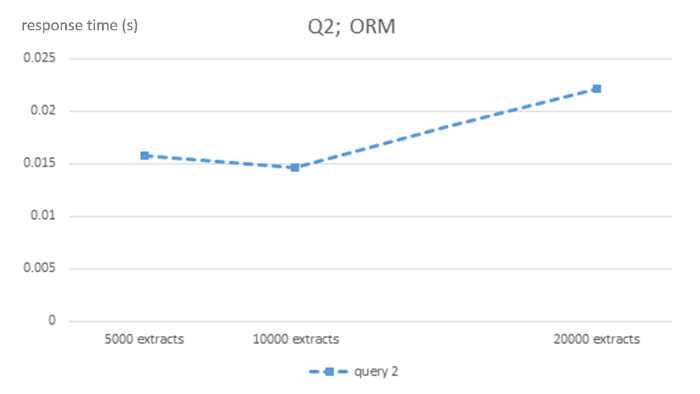
Figura 2 : Complejidad algorítmica de DBMS MySQL de ORM para la consulta Q2. Esta figura muestra los tiempos de respuesta en segundos para 5.000, 10.000 y 20.000 tamaño EHR extractos ORM MySQL base de datos para la consulta Q2. Haga clic aquí para ver una versión más grande de esta figura.
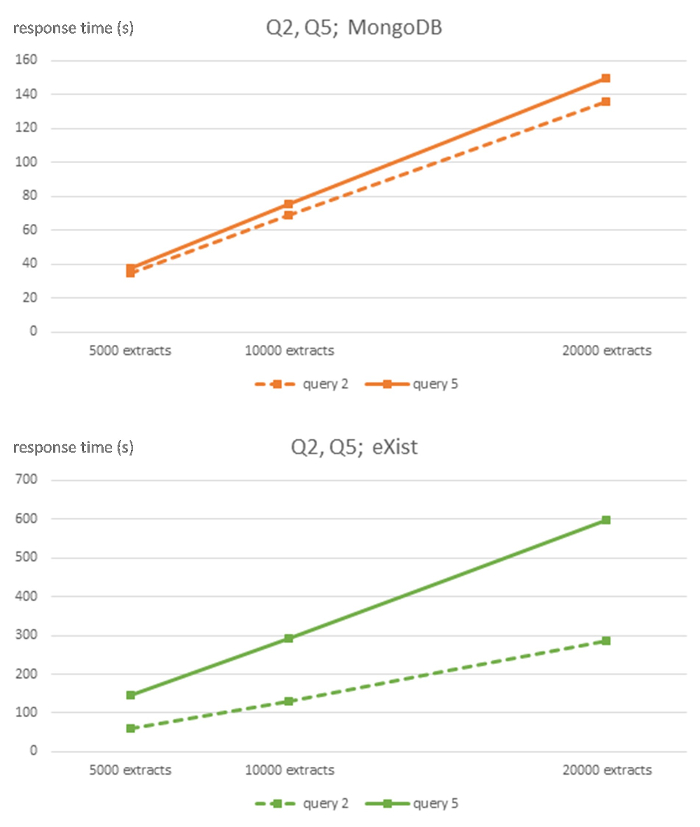
Figura 3 : Complejidad algorítmica de MongoDB y existen DBMS para consultas de Q2 y Q5. Esta cifra se modificó de7 con licencia Creative Commons (http://creativecommons.org/licenses/ por / 4,0) y tiempos de respuesta en segundos para 5.000, 10.000, 20.000 tamaño y her extractos de bases de datos para cada SGBD y consultas Q2 y Q5. Haga clic aquí para ver una versión más grande de esta figura.
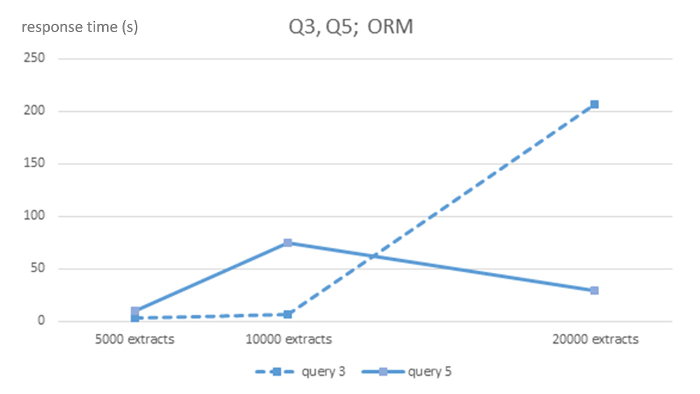
Figura 4 : Complejidad algorítmica de DBMS MySQL de ORM para consultas Q3 y Q5. Muestra los tiempos de respuesta en segundos para 5.000, 10.000 y 20.000 tamaño EHR extractos de bases de datos para cada SGBD y consultas Q3 y Q5. Haga clic aquí para ver una versión más grande de esta figura.
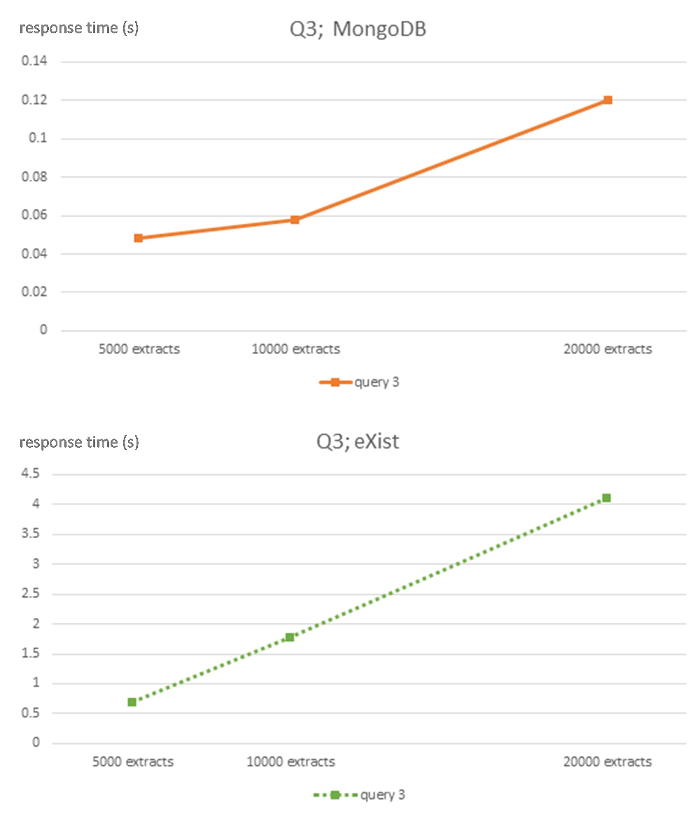
Figura 5: complejidad algorítmica de MongoDB DBMS para consulta Q3 y existen. Esta cifra se modificó de7 con licencia de Creative Commons (http://creativecommons.org/licenses/ por/4.0) y muestra los tiempos de respuesta en segundos para 5.000, 10.000 y 20.000 tamaño EHR extractos de bases de datos para cada DBMS y consulta Q3. Haga clic aquí para ver una versión más grande de esta figura.
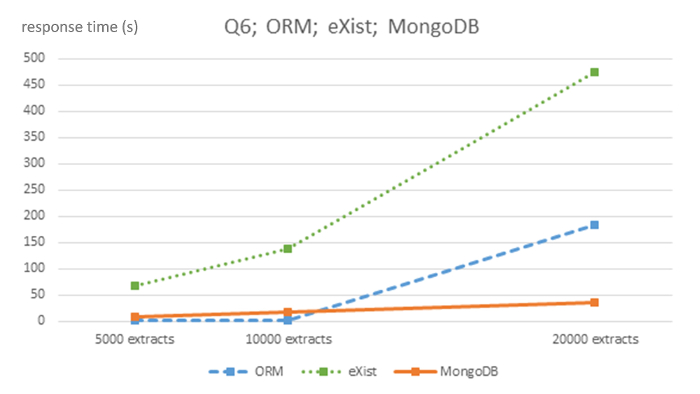
Figura 6 : Complejidad algorítmica de ORM MySQL, existen y MongoDB DBMS para consulta Q6. Esta cifra se modificó de7 con licencia de Creative Commons (http://creativecommons.org/licenses/ por/4.0) y muestra los tiempos de respuesta en segundos para 5.000, 10.000 y 20.000 tamaño EHR extractos de bases de datos para cada DBMS y consulta Q6. Haga clic aquí para ver una versión más grande de esta figura.
| Consulta | |
| Q1 | Encontrar todos los problemas de un solo paciente |
| Q2 | Encontrar todos los problemas de todos los pacientes |
| Q3 | Encontrar la fecha inicial, fecha de resolución y la gravedad |
| de un solo problema de un solo paciente | |
| Q4 | Encontrar la fecha inicial, fecha de resolución y la gravedad |
| de todo problema de problemas de un solo paciente | |
| Q5 | Encontrar la fecha inicial, fecha de resolución y la gravedad |
| de todo problema de problemas de todos los pacientes | |
| Q6 | Encontrar a todos los pacientes con 'faringitis' problema, |
| inicial de fecha > = 16 de octubre de 2007 ', fecha de resolución | |
| < = 05 de junio de 2008 ' y 'alta' de gravedad |
Tabla 1: las seis consultas realizaron en los relacionales y bases de datos NoSQL extractos de HCE estandarizada que contiene acerca de los problemas de los pacientes. Esta tabla ha sido modificada de7 con licencia de Creative Commons (http://creativecommons.org/ licenses/by/4.0/) y muestra las seis consultas creciente complejidad realizadas en las tres bases de datos crecen de tamaño para cada DBMS expresado en natural lengua.
| ORM y MySQL | 5000 docs | 10.000 documentos | 20.000 documentos |
| Q1 (s) | 25.0474 | 32.6868 | 170.7342 |
| Q2 (s) | 0.0158 | 0.0147 | 0.0222 |
| Q3 (s) | 3.3849 | 6.4225 | 207.2348 |
| Q4 (s) | 33.5457 | 114.6607 | 115.4169 |
| Q5 (s) | 9.6393 | 74.3767 | 29.0993 |
| P6 (s) | 1.4382 | 2.4844 | 183.4979 |
| Tamaño de la base de datos | 4.6 GB | 9.4 GB | 19.4 GB |
| Extractos totales | 5000 | 10.000 | 20.000 |
Tabla 2: promedio de tiempos de respuesta en segundos de las seis consultas en bases de datos de tamaño doble de lo SGBD relacional ORM MySQL. Esta tabla muestra seis tiempos de respuesta para cada consulta tres tamaño duplicar bases de datos utilizando el DBMS relacional ORM MySQL y el tamaño en memoria de las tres bases de datos.
| MongoDB | 5000 docs | 10.000 documentos | 20.000 documentos | pendiente (*10exp(-6)) |
| Q1 (s) | 0.046 | 0.057 | 0.1221 | 5,07 |
| Q2 (s) | 34.5181 | 68.6945 | 136.2329 | 6,780.99 |
| Q3 (s) | 0.048 | 0.058 | 0.1201 | 4.81 |
| Q4 (s) | 0.052 | 0.061 | o.1241 | 4.81 |
| Q5 (s) | 38.0202 | 75.4376 | 149.933 | 7460.85 |
| P6 (s) | 9.5153 | 18.5566 | 36.7805 | 1,817.68 |
| Tamaño de la base de datos | 1,95 GB | 3.95GB | 7,95 GB | |
| Extractos totales | 5000 | 10.000 | 20.000 |
Tabla 3: promedio de tiempos de respuesta en segundos de las seis consultas en bases de datos de tamaño doble de los DBMS de NoSQL MongoDB. Esta tabla ha sido modificada de7 con licencia de Creative Commons (http://creativecommons.org/ licenses/by/4.0/) y muestra los tiempos de respuesta de seis de cada consulta para las tres tamaño duplicar bases de datos utilizando el DBMS de NoSQL MongoDB y el tamaño en memoria las tres bases de datos. También se indica la pendiente lineal de cada consulta.
| Existen | 5000 docs | 10.000 documentos | 20.000 documentos | pendiente (*10exp(-6)) |
| Q1 (s) | 0.6608 | 3.7834 | 7.3022 | 442.76 |
| Q2 (s) | 60.7761 | 129.3645 | 287.362 | 15,105.73 |
| Q3 (s) | 0.6976 | 1.771 | 4.1172 | 227.96 |
| Q4 (s) | 0.6445 | 3.7604 | 7.3216 | 445.17 |
| Q5 (s) | 145.3373 | 291.2502 | 597.7216 | 30,158.93 |
| P6 (s) | 68.3798 | 138.9987 | 475.2663 | 27,125.82 |
| Tamaño de la base de datos | 1,25 GB | 2,54 GB | 5,12 GB | |
| Extractos totales | 5000 | 10.000 | 20.000 |
Tabla 4: promedio de tiempos de respuesta en segundos de las seis consultas en bases de datos de tamaño doble de existen DBMS NoSQL. Esta tabla ha sido modificada de7 con licencia de Creative Commons (http://creativecommons.org/ licenses/by/4.0/) y muestra los tiempos de respuesta de seis de cada consulta tres tamaño duplicar bases de datos utilizando el NoSQL existen DBMS y el tamaño en memoria de las tres bases de datos. También se indica la pendiente lineal de cada consulta.
| Papel de brazo | BRAZO (s) | Nodo + ruta (s) | |
| Q1 | Consulta 2.1 | 0.191 | 24.866 |
| Q3 | Consulta 3.1 | 0.27 | 294.774 |
| Tamaño de la base de datos | 2,90 GB | 43,87 GB | |
| Extractos totales | 29.743 | 29.743 |
Tabla 5: promedio de tiempos de respuesta en segundos de consultas similares a Q1 y Q3 de la mejora de los sistemas relacional presentados en 10 . Esta tabla ha sido modificada de7 con licencia de Creative Commons (http://creativecommons.org/ licenses/by/4.0/) y muestra las dos consultas más similar a Q1 y Q3 presentado en10 correspondiente a dos sistemas de bases de datos relacionales mejorada y sus tiempos de respuesta. También se muestran los tamaños de dos base de datos.
| ORM y MySQL | Rendimiento de procesamiento | Tiempo de respuesta |
| Q1 (s) | 4,711.60 | 0.0793 |
| Q3 (s) | 4,711.60 | 0.1558 |
| Q4 (s) | 4,711.60 | 0.9674 |
Tabla 6: promedio de tiempo de respuesta y rendimiento en segundos de consultas Q1, Q3 y Q4 de ORM MySQL relacional DBMS en ejecución concurrente. Esta tabla ha sido modificada de7 con licencia de Creative Commons (http://creativecommons.org/ licenses/by/4.0/) y muestra el mayor rendimiento promedio de las tres consultas del paciente y sus tiempos de respuesta promedio en el concurrente experimento de ejecución mediante el sistema relacional ORM MySQL.
| MongoDB | Rendimiento de procesamiento | Tiempo de respuesta |
| Q1 (s) | 178,672.60 | 0.003 |
| Q3 (s) | 178,672.60 | 0.0026 |
| Q4 (s) | 178,672.60 | 0.0034 |
Tabla 7: promedio de tiempo de respuesta y rendimiento en segundos de consultas Q1, Q3 y Q4 de MongoDB, NoSQL DBMS en ejecución concurrente. Esta tabla ha sido modificada de7 con licencia de Creative Commons (http://creativecommons.org/ licenses/by/4.0/) y muestra el mayor rendimiento promedio de las tres consultas del paciente y sus tiempos de respuesta promedio en el concurrente experimento de ejecución mediante el sistema MongoDB NoSQL.
Suplementario Figura 1: la captura de pantalla muestra la pantalla de software para conectarse al servidor MySQL. Haga clic aquí para descargar esta figura.
Suplementario Figura 2: la captura de pantalla muestra la interfaz SQL al servidor MySQL en la primera consulta SQL se ha escrito. Haga clic aquí para descargar esta figura.
3 figura suplementaria: el 2.6 de MongoDB localhost servidor es lanzado utilizando una ventana DOS del sistema ejecutando el servidor mongod. Haga clic aquí para descargar esta figura.
Suplementario figura 4: la captura de pantalla muestra la consulta escrita en los cuadros de texto del generador de consultas, como se muestra en pasos 5.7.1 a través 5.7.4. La captura de pantalla ilustra paso 5.7.3. Por favor haga clic aquí para descargar esta figura.
Suplementario Figura 5: la imagen muestra el paso 5.7.6. Haga clic aquí para descargar esta figura.
Suplementario Figura 6: la captura de pantalla ilustra la escritura de la consulta de XPath en theupper parte del cuadro de diálogo. Haga clic aquí para descargar esta figura.
Discusión
Este protocolo muestra que sistemas ORM relacionales pura parece prácticos para único paciente consulta (Q1, Q3 y Q4) puesto que los tiempos de respuesta son más lentos, probablemente debido a un elevado número de tablas relacionales realizar muchas operaciones de combinación costosos y debido a la sistema de almacenamiento utilizado por el tipo específico de base de datos. Bases de datos NoSQL almacenan datos de manera basado en el documento, mientras que sistemas relacionales utilizan una manera basados en tablas que se propaga cada documento a lo largo de la base de datos entera. Sistemas NoSQL muestran una pendiente lineal, con MongoDB realizar considerablemente más rápido que existen DBMS. En concurrencia, MongoDB también se comporta mucho mejor que relacional ORM MySQL7.
Este protocolo presenta un protocolo de resolución de problemas para los resultados presentados en el7 con respecto al DBMS MySQL de ORM. El servidor MySQL ha sido actualizado a la versión más reciente y los resultados han sido ligeramente modificados. Además, un punto crítico en el documento de los sistemas NoSQL MongoDB es que puede conservar consistencia cuando almacenar información médica7 porque cuando un extracto de HCE se actualiza, no se sobreescribe, pero un todo Extracto de nuevo con los nuevos datos es generados y almacenados en el sistema, y el extracto de la original se mantiene. Este es un requisito estricto de información médica, porque algunos médicos pueden haber hecho importantes decisiones médicas basadas en los datos originales.
El sistema de brazo relacional mejorado drásticamente disminuye el número de tablas relacionales y mejora relacional. Sin embargo, puesto que modifica el esquema relacional, puede consultar información médica en poder de los extractos, pero extractos no pueden ser recuperados en su forma original exacta.
Para bases de datos muy grandes de secundaria utilizan (investigación), no está claro qué sistema de base de datos es más apropiado, puesto que las consultas de todo paciente (Q2 y Q5) se comportan mejor en ORM que en sistemas NoSQL, pero estos sistemas se desempeñan mejor que el simplificado relacional sistemas en 12. Consideramos Q6 una consulta especial entre clínica y secundaria uso cuyo comportamiento no puede ser determinado por los resultados producidos por estos experimentos.
Sin embargo, una limitación del método es la indisponibilidad de experimentos directos comparando el mejor sistema relacional brazo con NoSQL MongoDB sobre consultas de práctica médica de paciente con exactamente los mismos datos utilizados en el protocolo. Mantuvimos los resultados interpolación tabla 3 y tabla 5 sobre consultas del paciente hasta que se realizó el experimento incluyendo el brazo de optimizada en el protocolo. Os dejamos estos experimentos para futuras aplicaciones. Un paso crítico dentro del protocolo es la selección de la base de datos gratis, versiones de software similares de los últimos años, para que podamos comparar estado de arte exacto de las tres tecnologías.
Este es uno de los primeros intentos para comparar directamente relacionales y sistemas NoSQL con información médica real, realista, estandarizada. Sin embargo, el sistema específico a utilizar depende mucho de la situación real y problema resuelto8.
Divulgaciones
Los autores no tienen nada que revelar. Los conjuntos de datos utilizados en estos experimentos fueron proporcionados por varios hospitales españoles bajo licencia para estos experimentos y en consecuencia no están públicamente disponibles. El esquema ISO/EN 13606 RM XML fue proporcionado por la Universidad College Londres centro de informática de la salud y la educación multiprofesional (campana).
Agradecimientos
Los autores desean dar las gracias a Dr Dipak Kalra, líder de la fuerza de tarea EHRCom que define el estándar 13606 de ISO/EN y su equipo de University College de Londres para su permiso usar la EN/ISO 13606 W3C XML Schema.
Este trabajo fue apoyado por Instituto de Salud Carlos III [números de concesión PI15/00321, PI15/00003, PI1500831, PI15CIII/00010 y RD16CIII].
Materiales
| Name | Company | Catalog Number | Comments |
| MySQL 5.7.20 | MySQL experiments | ||
| Red Hat Enterprise Linux Server release 7.4 (Maipo), 2.60GHz, RAM 16GB | |||
| MongoDB 2.6 | MongoDB experiments | ||
| Windows 7, 2.66GHz, RAM 12GB | |||
| eXist 3.0RC1 | eXist experiments | ||
| Windows 7, 2.66GHz, RAM 12GB | |||
| Studio 3T 5.5.1 | 3T Software Labs Gmbh | MongoDB GUI |
Referencias
- Codd, E. F. A relational model for large shared data banks. Comm ACM. 13 (6), 377-387 (1970).
- Kalra, D., Lloyd, D. . ISO 13606 electronic health record communication part 1: reference model. , (2008).
- Kalra, D., et al. . Electronic health record communication part 2: archetype interchange specification. , (2008).
- Kalra, D., Beale, T., Heard, S. The openEHR foundation. Stud. Health Technol. Inform. 115, 153-173 (2005).
- Beale, T. Archetypes: constraint-based domain models for future proof information systems. OOPSLA, Workshop Behav Semant. , (2002).
- Sánchez-de-Madariaga, R., et al. Examining database persistence of ISO/EN 13606 standardized electronic health record extracts: relational vs. NoSQL approaches. BMC Medical Informatics and Decision Making. 32 (2), 493-503 (2017).
- Ireland, C., Bowers, D., Newton, M., Waugh, K. Understanding object-relational mapping: a framework based approach. Int. J. Adv. Softw. 2, 202-216 (2009).
- . Node+Path Persistence Available from: https://openehr.atlassian.net/wiki/spaces/dev/pages/6553626/Node+Path+Persistence (2017)
- Wang, L., Min, L., Wang, R., et al. Archetype relational mapping - a practical openEHR persistence solution. BMC Medical Informatics and Decision Making. 15, 88 (2015).
- Kaur, K., Rani, R. Managing data in healthcare information systems: many models, one solution. Computer. March. , 52-59 (2015).
- Sabo, C., Pop, P. C., Valean, H., Danciulescu, D. An innovative approach to manage heterogeneous information using relational database systems. Advances in Intelligent Systems and Computing. 557, (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados
Utilizamos cookies para mejorar su experiencia en nuestra página web.
Al continuar usando nuestro sitio web o al hacer clic en 'Continuar', está aceptando nuestras cookies.