Method Article
Executando consultas complexidade crescente em relacional (MySQL) e NoSQL (MongoDB e existir) crescimento tamanho ISO/EN 13606 EHR padronizada bancos de dados
Neste Artigo
Resumo
Este estudo compara relacionais e não relacionais (NoSQL) padronizado de sistemas de informação médica. A complexidade computacional dos tempos de resposta da consulta de tais sistemas de gerenciamento de banco de dados (DBMS) é calculada usando bancos de dados de dobrar de tamanho. Estes resultados ajudam a discussão sobre a adequação de cada abordagem de banco de dados para diferentes cenários e problemas.
Resumo
Esta pesquisa mostra um protocolo para avaliar a complexidade computacional de uma consulta relacional e não-relacionais (NoSQL (não só de Structured Query Language)) padronizado de sistemas de banco de dados eletrônico de saúde record (EHR) informações médicas (DBMS). Ele usa um conjunto de três dobrando de tamanho bancos de dados, ou seja, bancos de dados armazenar 5000, 10.000 e 20.000 realista padronizado extractos de RSE, em sistemas de gestão de três diferentes banco de dados (DBMS): relacionais MySQL o mapeamento objeto-relacional (ORM), baseado em documento NoSQL MongoDB e nativo extensible markup language (XML) NoSQL existe.
Foram calculados os tempos de resposta média de seis consultas de complexidade crescente, e os resultados mostraram um comportamento linear nos casos NoSQL. No campo de NoSQL, MongoDB apresenta um declive linear muito mais plano do que existe.
NoSQL sistemas também podem ser mais apropriados manter os sistemas de informação médica padronizada devido à natureza especial das políticas atualização de informações médicas, que não deverá afectar a consistência e a eficiência dos dados armazenados em bancos de dados NoSQL.
Uma limitação do presente protocolo é a falta de resultados diretos da melhoria dos sistemas relacional, tais como mapeamento relacional do arquétipo (braço) com os mesmos dados. No entanto, a interpolação dos resultados de duplicação-tamanho de banco de dados com aqueles apresentados na literatura e outros resultados publicados sugerem que sistemas NoSQL podem ser mais apropriados em muitos cenários específicos e problemas a serem resolvidos. Por exemplo, NoSQL pode ser apropriado para tarefas baseadas em documentos como EHR extratos utilizados na prática clínica, ou edição e visualização ou situações onde o objectivo é não só a informação de consulta médica, mas também para restaurar a RSE em sua forma original, exatamente.
Introdução
NoSQL (não apenas SQL) DBMS recentemente surgiram como uma alternativa ao SGBD relacional tradicional (RDMBS). RDBMS têm dominado o caminho de dados foram armazenados em sistemas de banco de dados por décadas. Cálculo e álgebra relacional bem estudada e compreendida tem garantido a eficácia e a coerência do RDBMS1. Sistemas NoSQL não tornará substitutos para sistemas relacionais, mas eles poderiam comportar-se vantajosamente em determinados cenários e sob várias condições.
Alguns destes cenários específicos e condições ocorreria ao projetar a persistência de banco de dados dos sistemas de registo de saúde electrónico (RSE), usado para gerenciar e armazenar informações médicas. A fim de ser interoperável e sustentável na prática, várias normas internacionais como ISO/EN 13606, openEHR e HL72,3,4,5 têm sido utilizados para padronizar os extratos de RSE.
Diversos padrões como ISO/EN 13606 e openEHR tem separado a informação e conhecimento em dois níveis diferentes de abstração, representado pelo modelo de referência (RM) e estruturas de dados especial chamadas arquétipos. Esta separação é muitas vezes chamada o modelo dual e permite sistemas RSE ser semanticamente interoperável e médico conhecimento evoluir sem reprogramar todo o sistema de RSE e, consequentemente, para ser de fácil manutenção e sustentável na prática6 . No entanto, o dupla modelo implementado em sistemas padronizados de RSE requer que a organização das informações segue uma estrutura específica, e isso tem consequências profundas na maneira que o nível de persistência do banco de dados do sistema é projetado7.
Objeto de mapeamento relacional (ORM)8 é uma metodologia para implementar um sistema de RSE, utilizando o paradigma de banco de dados relacional. ORM exaustivamente mapeia a estrutura do padronizado EHR extrai arquivos XML (eXtensible Markup Language) usado pelo sistema para um banco de dados relacional. ORM constrói muitas tabelas relacionais exaustivamente seguindo a estrutura dos arquivos XML extratos EHR padronizadas. Estas tabelas relacionais relacionam-se através de muitas chaves estrangeiras e o sistema resultante pode não ser muito eficiente.
Foram propostas várias melhorias relacionais de ORM. Caminho do nó +9 do openEHR reduz o número de tabelas relacionais por serialização subárvores do extrato todo arquivo XML em BLOBs (objetos binários grandes). No entanto, essa simplificação faz com que lógica de recuperação complexa, danificando consultas complexas. Arquétipo de mapeamento relacional (braço)10 gera um modelo de banco de dados conduzido por arquétipos, construindo uma nova esquema relacional com base em mapeamentos entre arquétipos e tabelas relacionais. Consequentemente, algumas das informações não-médicos do extracto de RSE é perdida.
Muitos documentos com base em bancos de dados NoSQL armazenam documentos todo como BLOBs inteiras respeitando um original de XML ou JSON (JavaScript Object Notation) formato. Isto significa que não há tabelas relacionais são construídas. Esses bancos de dados NoSQL não tem nenhum esquema e eles não suportam junções ou de Propriedades (ácido)11, ou seja, atomicidade, consistência, isolamento ou durabilidade. Como resultado, eles podem ser muito ineficientes se um elemento de um documento faz referência a elementos do mesmo ou outros tais documentos utilizando um link de indireção. Isso acontece porque, a fim de manter a consistência, a totalidade dos documentos referenciados têm de ser processadas sequencialmente. No entanto, um banco de dados não-relacionais pode ser continua a justificar-se a principal tarefa realizada pelo SGBD é uma tarefa com base em documento. Isso ocorre porque os dados podem permanecer em um formulário mais pròxima aproximando sua verdadeira representação usando um documento-base NoSQL de dados, embora isto é também devido as condições de persistência especial realizadas pelos documentos médicos de RSE (consulte a seção de discussão).
A finalidade destes métodos é apresentar vários experimentos para comparar a implementação da camada de persistência de um sistema padronizado de RSE usando três diferentes SGBDs: uma relacional (MySQL) e dois NoSQL (baseado no documento MongoDB e XML nativo existem). Sua complexidade computacional tem sido calculado e comparados usando três crescente tamanho bancos de dados diferentes e seis consultas diferentes de complexidade crescente. Os servidores de banco de três dados foram instalados e configurados localmente no mesmo computador onde foram executadas as consultas. Consulte a Tabela de materiais para detalhes técnicos.
Também foram realizados experimentos de simultaneidade para comparar o desempenho de MySQL e de NoSQL MongoDB DBMSs relacional. As melhorias de ORM descritas (caminho do nó + e braço) também foram comparadas usando relevantes resultados adequados do literatura10.
Sistemas de gerenciamento de banco de dados estão evoluindo continuamente a um ritmo acelerado. Ninguém pensaria sobre este desenvolvimento exponencial quando o único paradigma existente foi o modelo relacional. Para dar um exemplo, ver por exemplo12, onde um modelo foi proposto para implementar o tempo de resposta melhorados bancos de dados relacionais mantendo as propriedades ACID.
Protocolo
1. construir um SGBD relacional do MySQL para armazenar três duplo tamanho padronizado EHR extratos de bancos de dados
- Importar o W3C (World Wide Web Consortium) XML esquema correspondente para o ISO/EN13606 RM e os tipos de dados de ISO21090 em um 'IDE Java' (Integrated Development Environment).
Nota: ISO significa International Standards Organization. PT defende norma europeia. - Executar o JAXB (Java XML Binding) plug-in no IDE; Isto produz classes Java correspondente à estrutura dos elementos dos arquivos XML EHR extratos.
- Marca manualmente as classes Java produzidas com rótulos JPA. Esses rótulos referem as cardinalidades e outras relações entre as tabelas relacionais de banco de dados MySQL.
- Importar as bibliotecas da JPA (API de persistência Java) para o IDE e executa o método que cria um banco de dados MySQL fora as classes de Java marcados.
- Crie três diretórios com 5.000, 10.000 e 20.000 EHR realista extrai arquivos XML.
- Execute o método JPA para carregar um extracto XML para o MySQL DBMS em todos os extratos do diretório 5.000 extratos.
- Repita a etapa 1.6 duas vezes, uma vez com o diretório de 10.000 extratos e uma vez com o diretório de 20.000 extratos.
2. Construa um DBMS NoSQL MongoDB para armazenar três duplo tamanho padronizado EHR extratos de bancos de dados
- Processo de cada dos três diretórios contendo 5.000, 10.000 e 20.000 realista EHR extrai arquivos XML com um programa padrão para converter arquivos XML para arquivos JSON, como json.org.XML. Três diretórios com 5.000, 10.000 e 20.000 arquivos JSON devem ser produzidos.
- Lançar um MongoDB GUI (Graphic User Interface, consulte a Tabela de materiais).
- Lançar o MongoDB 2.6 servidor executando o programa mongod de uma janela do sistema DOS (Disk Operating System).
- Conecte o MongoDB GUI ao servidor localhost usando a porta 27017.
- Selecione o menu "Connect".
- Escreva um nome para a conexão (por exemplo ' primeiro').
- Escreva localhost:27017 na caixa de texto servidor DB.
- Pressione o botão "Connect"; uma árvore com os bancos de dados atuais deve aparecer à esquerda.
- Construa um banco de dados contendo 5.000 EHR padronizado extractos.
- Clique sobre o nome da conexão no topo da árvore à esquerda.
- Selecione o menu "arquivo".
- Escolha "Adicionar Banco de dados".
- Digite o nome do banco de dados na caixa de diálogo que aparece.
- Clique Okey.
- Construa uma coleção contendo 5.000 EHR padronizado extractos.
- Clique sobre o nome do banco de dados na árvore à esquerda.
- Selecione o menu "banco de dados".
- Escolha "AddCollection".
- Digite o nome da coleção na caixa de diálogo que aparece.
- Clique em " criar".
- Clique sobre o nome da coleção.
- Selecione o menu "importação".
- Escolha o botão de opção 'JSON - escudo mongo / / mongoexport ".
- Clique em "próximo".
- Pressione o botão "Adicionar arquivos de fonte".
- Navegar no computador usando a caixa de diálogo.
- Abra o diretório que contém 5.000 JSON extrair arquivos.
- Selecione todos os arquivos no diretório.
- Pressione "aberto". A lista de arquivos JSON deve aparecer na caixa de diálogo de importação.
- Prima "seguinte"; um preview da nova coleção no banco de dados aparece no lado esquerdo.
- Pressione "próximo".
- Prima "Iniciar importação". O progresso da importação aparece em baixo à esquerda, indicando o número de arquivos importados e o tempo decorrido.
- Repita as etapas 5 e 6 para construir uma coleção de 10.000 EHR padronizado extractos.
- Repita as etapas 5 e 6 para construir uma coleção de 20.000 EHR padronizado extractos.
3. construir um NoSQL existe DBMS para loja três duplo tamanho padronizado EHR extrai os bancos de dados
- Inicie o banco de dados existe .
- Usando o ícone do banco de dados, abra o cliente de Admin de Java.
- Digite a senha do admin.
- Prima o botão "Connect".
- Construa uma coleção contendo 5.000 EHR padronizado extractos.
- Na barra de ferramentas, selecione o menu "criar uma nova coleção".
- Na caixa de diálogo que aparece, digite o nome da nova coleção.
- Clique em "aceitar"; a nova coleção irá aparecer.
- Selecione o nome da coleção.
- Na barra de ferramentas, selecione o menu "armazenar arquivos no banco de dados".
- Navegar no computador usando a caixa de diálogo.
- Selecione o diretório contendo 5.000 arquivos XML padronizados de extrato.
- Clique no botão "Selecione os arquivos ou diretórios para armazenar". Observe que uma caixa de diálogo aparece mostrando o progresso, os arquivos a serem armazenados e a porcentagem de banco de dados criado.
- Repita a etapa 5 para construir uma coleção contendo 10.000 EHR padronizado extractos.
- Repita a etapa 5 para construir uma coleção contendo 20.000 EHR padronizado extractos.
4. projetar e executar em 3 bancos de dados relacionais MySQL 6 consultas de complexidade crescente
- O projeto seis consultas de complexidade crescente, de acordo com os arquétipos usados por extratos de RSE.
- Programe um script SQL com a primeira consulta no banco de dados MySQL. O SQL deve adaptar-se à estrutura especial do banco de dados MySQL devido a padronização de extratos (arquétipos). O banco de dados mapeia toda a estrutura dos extratos. Como resultado, a consulta SQL é bastante complexa.
- Identifica os atributos de bancos de dados que iria acelerar o tempo de resposta das consultas se um índice foi construído sobre eles, então, construir tais índices, embora a maioria dos índices são criados automaticamente pelo DBMS.
- Se uma consulta precisa de um índice não-automaticamente feito, construí-lo manualmente.
- Conecte ao servidor MySQL (complementar a Figura 1).
- Selecione e clique no nome do banco de dados do lado esquerdo.
- Selecione e clique na tabela relacional onde reside o campo indexado.
- Clique na guia "estrutura".
- Selecione e clique na coluna onde o índice será construído.
- Clique em "índice". Observe que a sentença SQL construindo o índice aparece, e é exibida uma mensagem informando que a frase foi construída com sucesso.
- Execute a primeira consulta.
- Selecione e clique no nome do banco de dados do lado esquerdo.
- Clique na guia "SQL".
- Escreva ou cole o código SQL da primeira consulta (ver Figura complementar 2).
- Pressione "continuar". Observe que aparece a primeira tela da lista de resultados, juntamente com uma mensagem com o tempo de execução da consulta.
- Repita a execução 5 vezes e calcular o tempo médio de resposta.
- Repita o passo 5 com consultas de 2 a 6.
- Faz todo o processo três vezes, com 5.000, 10.000 e 20.000 extratos de bancos de dados.
5. projetar e executar em 3 bancos de dados NoSQL MongoDB 6 consultas de complexidade crescente
- Inicie a GUI do MongoDB (ver Tabela de materiais).
- Iniciar o servidor MongoDB 2.6 executando o programa mongod de uma janela do sistema DOS (ver complementar a Figura 3).
- Siga o passo 2.4 para conectar o MongoDB GUI ao servidor localhost usando a porta 27017.
- Selecione e expanda o banco de dados MongoDB no lado esquerdo.
- Selecione a coleção.
- Clique no menu "coleção" na barra de ferramentas.
- Execute a primeira consulta de MongoDB.
- Clique duas vezes no botão de "Query Builder".
- Clique duas vezes no "campo de consulta" do construtor de consultas à direita.
- Escreva o campo da consulta MongoDB no campo textbox do painel de consulta (ver complementar a Figura 4).
- Escreva o valor da consulta MongoDB na caixa de texto valor do painel de consulta.
Nota: Esta consulta deve ser algo como {ns3:EHRExtract.allCompositions.content.items.parts.parts.name.ns2:originalText". valor":"Descripcion"}. O campo e o valor são citados e separados por ponto e vírgula. - Clique duas vezes no campo de projeção do construtor de consultas
- Escrever a primeira projecção na projeção de texto (consulte complementar a Figura 5).
- Clique duas vezes no campo de projeção para adicionar um novo textbox de projeção.
- Escreva a segunda projeção na projeção de texto.
Nota: Uma projeção seleciona uma parte do documento obtida pela consulta. Isto devem ser algo como {ns3:EHRExtract". allCompositions.content.items.parts.parts.value.value": 1} e {" ns3: EHRExtract.all Compositions.content.items.parts.parts.value.nullFlavor ": 1} - Clique sobre o botão azul play para executar a consulta.
- Visualize o código de consulta na guia código de consulta.
- Visualizar os detalhes do resultado na guia explicar: número de resultados, o tempo de execução em milissegundos.
- Visualizar, ampliar e examinar os resultados na guia do resultado.
- Se posterior processamento da consulta é necessária, escreva um programa em Java com o driver MongoDB Java com a consulta e um método para processar os resultados.
- Repita a execução 5 vezes e calcular o tempo médio de resposta.
- Passo 5.7 para os restantes 2 através de 6 consultas.
- Repita todo o processo na 5.000, 10.000 e 20.000 extrai os bancos de dados MongoDB.
6. projetar e executar no 3 NoSQL existem consultas de crescente complexidade de 6 bancos de dados
- Lançar a existir DBMS.
- Abra o cliente de Admin de Java.
- Pressione o botão "conectar ao banco de dados".
- Selecione o banco de dados e clique sobre ele.
- Clique no menu "consultar o banco de dados usando o XPath"; aparece a caixa de diálogo de consulta.
- Executar a primeira consulta XPath (ver complementar a Figura 6).
- Escreva ou cole o código a XPath da primeira consulta na parte superior da caixa de diálogo.
- Clique no menu "executar" na barra de ferramentas da caixa de diálogo.
- Ver os resultados XML usando a guia "XML" na parte inferior da caixa de diálogo.
- Ver o número de resultados e compilação e tempo de execução na parte inferior da caixa de diálogo.
- Repita a execução 5 vezes e calcular o tempo médio de resposta.
- Repita a etapa 6 para consultas de 2 a 6.
- Fazer todo o processo três vezes, para os extratos de 5.000, 10.000 e 20.000 existem bancos de dados.
7. projetar e executar um experimento de simultaneidade usando o MySQL e MongoDB 5.000 extrai os bancos de dados
Nota: O banco de dados existe foi removido o experimento neste momento devido ao pior desempenho nas experiências anteriores.
- Selecione as consultas com as três respostas mais curtas de tempo nas experiências anteriores usando os bancos de dados de 5.000 extratos (tipicamente sob vários segundos).
- Identificar e construir manualmente índices de atributo apropriado para essas consultas, se necessário.
- Programa dois aplicativos multithread do Java, uma para o MySQL e outra para MongoDB; cada aplicativo terá três threads de prioridade diferentes, um para cada consulta selecionada na etapa 1.
- Executar e calcular a CPU (Unidade Central de processamento) use a distribuição para cada thread (consulta).
- Executar cada aplicativo multithread, clicando no botão executar cinco vezes durante cada intervalo de 10 min e calcular a executada mais (maior prioridade) consultar a taxa de transferência média e a resposta do tempo médio das três consultas.
- Visualize as consultas em execução, com prioridades e tempo de execução.
- Calcule a taxa de transferência média e tempo médio de resposta de cada uma das três consultas.
Resultados
Seis diferentes consultas executadas em realistas extratos padronizados de RSE que contém informações sobre os problemas dos pacientes, incluindo seus nomes, datas inicial e final e severidade, são mostradas na tabela 1.
Tempos de resposta média das seis consultas das três bases de dados da duplicação-tamanho em cada SGBD são mostrados nas tabelas 2-4. Figuras 1-6 , mostrar os mesmos resultados graficamente (Observe que os eixos verticais usam muito diferentes escalas ao longo destas figuras).
O comportamento linear forte da complexidade computacional é evidente durante todo todas as consultas de bancos de dados NoSQL, embora com cuidado apropriado devido ao tamanho relativamente pequeno de 3 conjuntos de dados utilizados. No entanto, o banco de dados relacional de ORM mostra um comportamento linear pouco claro. Banco de dados MongoDB tem uma inclinação muito mais plana do que o banco de dados existe.
Resultados pela melhoria dos sistemas relacional, discutido na introdução publicada na literatura podem ser encontrados na tabela 5. MongoDB resultados da tabela 3 com consultas semelhantes e tamanhos de banco de dados dos resultados de braço de tabela 5 de interpolação é igual a ambos os sistemas de banco de dados no Q1, mas favorece o MongoDB no 3º trimestre.
Os resultados dos experimentos simultaneidade podem ser encontrados na tabela 5 e tabela6. MongoDB bate MySQL tanto na taxa de transferência e tempo de resposta. Na verdade, o MongoDB se comporta melhor em simultaneidade do que isoladamente e se destaca como um impressionante banco de dados em execução simultânea.
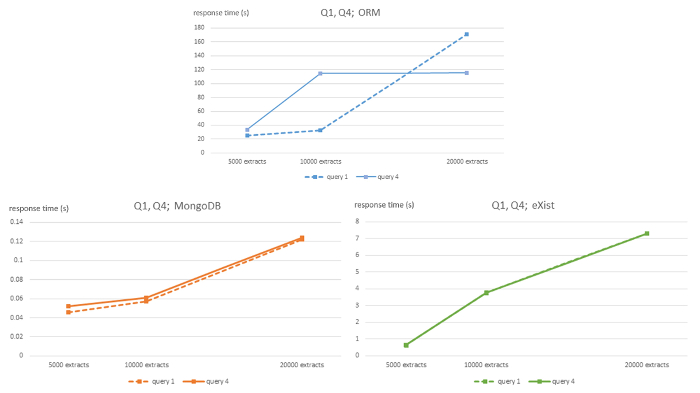
Figura 1 : Complexidade algorítmica de ORM MySQL, MongoDB, e existem DBMS para consultas Q1 e Q4. Esta figura foi modificada em7 usando a licença Creative Commons (http://creativecommons.org/ licenses/by/4.0/) e mostra os tempos de resposta em segundos para 5.000, 10.000 e 20.000 porte EHR extrai os bancos de dados para cada SGBD e consultas Q1 e Q4. Clique aqui para ver uma versão maior desta figura.
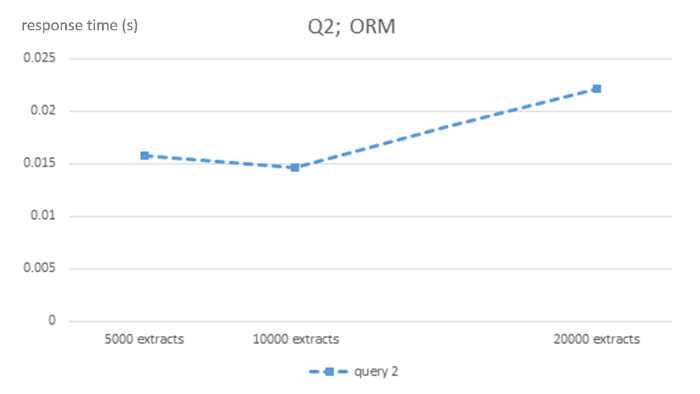
Figura 2 : Complexidade algorítmica de ORM SGBD de MySQL para consulta Q2. Esta figura mostra tempos de resposta em segundos para 5.000, 10.000 e 20.000 porte EHR extrai ORM MySQL banco de dados para consulta Q2. Clique aqui para ver uma versão maior desta figura.
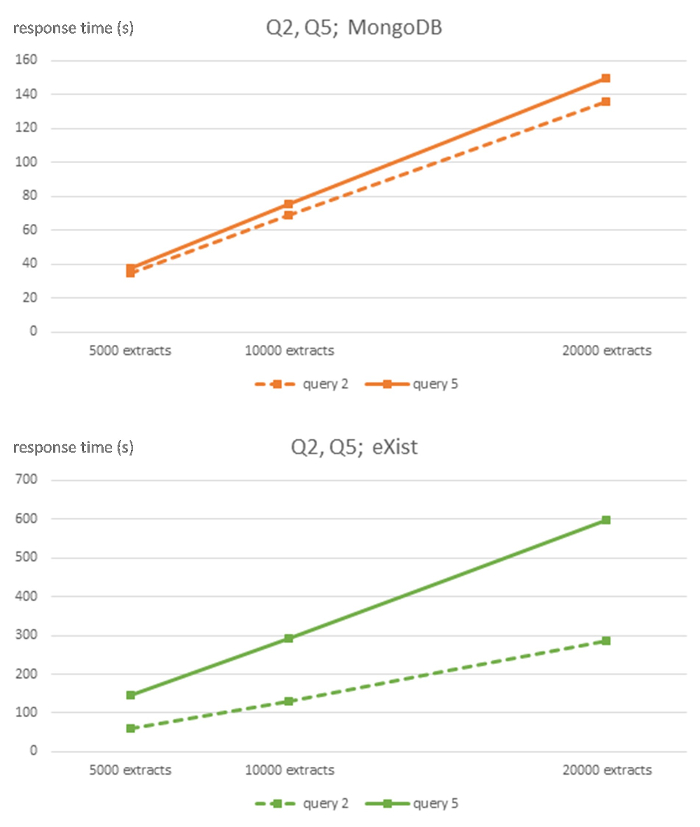
Figura 3 : Complexidade algorítmica do MongoDB e existem DBMS para consultas Q2 e Q5. Esta figura foi modificada em7 usando a licença Creative Commons (http://creativecommons.org/licenses/ por / 4.0) e mostra os tempos de resposta em segundos para 5.000, 10.000 e 20.000 tamanho EHR extrai os bancos de dados para cada SGBD e consultas Q2 e Q5. Clique aqui para ver uma versão maior desta figura.
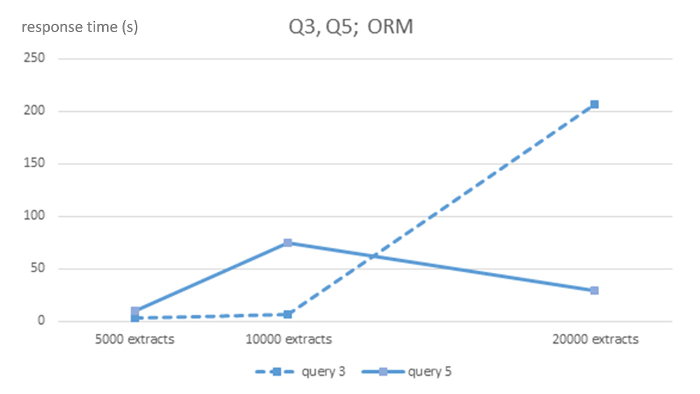
Figura 4 : Complexidade algorítmica de ORM MySQL SGBD para consultas Q3 e Q5. Mostra tempos de resposta em segundos para 5.000, 10.000 e 20.000 porte EHR extrai os bancos de dados para cada SGBD e consultas Q3 e Q5. Clique aqui para ver uma versão maior desta figura.
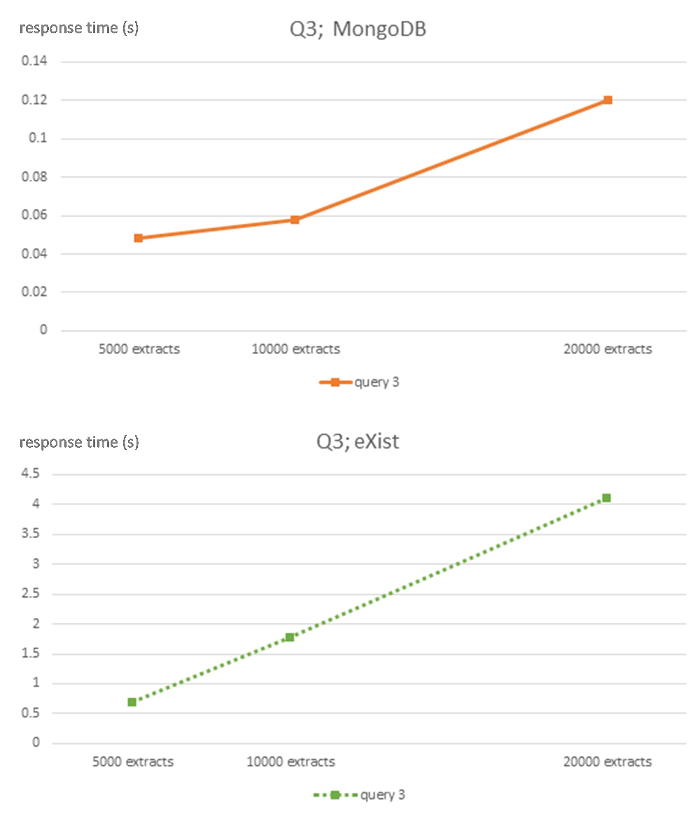
Figura 5: complexidade algorítmica de eXist e MongoDB DBMS para consulta Q3. Esta figura foi modificada em7 usando a licença Creative Commons (http://creativecommons.org/licenses/ por/4.0 /) e mostra os tempos de resposta em segundos para 5.000, 10.000 e 20.000 porte EHR extrai os bancos de dados para cada SGBD e consulta Q3. Clique aqui para ver uma versão maior desta figura.
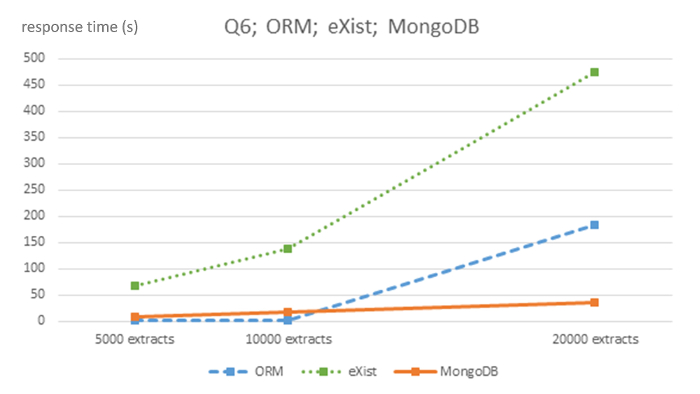
Figura 6 : Complexidade algorítmica de ORM MySQL, existir e MongoDB DBMS para consultar Q6. Esta figura foi modificada em7 usando a licença Creative Commons (http://creativecommons.org/licenses/ por/4.0 /) e mostra os tempos de resposta em segundos para 5.000, 10.000 e 20.000 porte EHR extrai os bancos de dados para cada SGBD e consulta Q6. Clique aqui para ver uma versão maior desta figura.
| Consulta | |
| Q1 | Encontrar todos os problemas de um único paciente. |
| Q2 | Encontrar todos os problemas de todos os pacientes |
| Q3 | Encontrar a data inicial, a data de resolução e a gravidade |
| de um único problema de um único paciente. | |
| Q4 | Encontrar a data inicial, a data de resolução e a gravidade |
| algum problema problemas de um único paciente. | |
| Q5 | Encontrar a data inicial, a data de resolução e a gravidade |
| algum problema problemas de todos os pacientes | |
| Q6 | Encontrar todos os pacientes com faringite problema' ', |
| inicial data > = 16 de outubro de 2007 ', data de resolução | |
| < = 5 de junho de 2008 ' e severidade 'alta' |
Tabela 1: as seis consultas realizadas sobre o relacional e bancos de dados NoSQL EHR padronizada contendo extratos sobre os problemas dos pacientes. Esta tabela foi modificada em7 usando a licença Creative Commons (http://creativecommons.org/ licenses/by/4.0/) e mostra as seis consultas complexidade crescente realizadas três tamanho crescimento bancos de dados para cada SGBD expressado em natural linguagem.
| ORM/MySQL | 5000 docs | docs de 10.000 | 20.000 docs |
| Q1 (s) | 25.0474 | 32.6868 | 170.7342 |
| Q2 (s) | 0.0158 | 0.0147 | 0.0222 |
| Q3 (s) | 3.3849 | 6.4225 | 207.2348 |
| Q4 (s) | 33.5457 | 114.6607 | 115.4169 |
| Q5 (s) | 9.6393 | 74.3767 | 29.0993 |
| Q6 (s) | 1.4382 | 2.4844 | 183.4979 |
| Tamanho de banco de dados | 4.6 GB | 9.4 GB | 19.4 GB |
| Total de extratos | 5000 | 10.000 | 20.000 |
Tabela 2: média de tempos de resposta em segundos das seis consultas em bancos de dados de duplicação-tamanho do DBMS relacionais MySQL ORM. Esta tabela mostra seis tempos de resposta para cada consulta para os três dobrando de tamanho bancos de dados usando o SGBD relacional MySQL ORM e o tamanho de memória de três bancos de dados.
| MongoDB | 5000 docs | docs de 10.000 | 20.000 docs | inclinação (*10exp(-6)) |
| Q1 (s) | 0,046 | 0,057 | 0.1221 | 5.07 |
| Q2 (s) | 34.5181 | 68.6945 | 136.2329 | 6,780.99 |
| Q3 (s) | 0,048 | 0.058 | 0.1201 | 4.81 |
| Q4 (s) | 0.052 | 0.061 | o.1241 | 4.81 |
| Q5 (s) | 38.0202 | 75.4376 | 149.933 | 7460.85 |
| Q6 (s) | 9.5153 | 18.5566 | 36.7805 | 1,817.68 |
| Tamanho de banco de dados | 1,95 GB | 3,95 GB | 7,95 GB | |
| Total de extratos | 5000 | 10.000 | 20.000 |
Tabela 3: média de tempos de resposta em segundos das seis consultas em bancos de dados de duplicação-tamanho do DBMS de NoSQL MongoDB. Esta tabela foi modificada em7 usando a licença Creative Commons (http://creativecommons.org/ licenses/by/4.0/) e mostra os tempos de resposta de seis de cada consulta para os três dobrando de tamanho bancos de dados usando o NoSQL MongoDB DBMS e o tamanho de memória os três bancos de dados. Também é mostrado o declive linear de cada consulta.
| Existem | 5000 docs | docs de 10.000 | 20.000 docs | inclinação (*10exp(-6)) |
| Q1 (s) | 0.6608 | 3.7834 | 7.3022 | 442.76 |
| Q2 (s) | 60.7761 | 129.3645 | 287.362 | 15,105.73 |
| Q3 (s) | 0.6976 | 1.771 | 4.1172 | 227.96 |
| Q4 (s) | 0.6445 | 3.7604 | 7.3216 | 445.17 |
| Q5 (s) | 145.3373 | 291.2502 | 597.7216 | 30,158.93 |
| Q6 (s) | 68.3798 | 138.9987 | 475.2663 | 27,125.82 |
| Tamanho de banco de dados | 1,25 GB | 2,54 GB | 5,12 GB | |
| Total de extratos | 5000 | 10.000 | 20.000 |
Tabela 4: média de tempos de resposta em segundos das seis consultas em bancos de dados de duplicação-tamanho do existe NoSQL DBMS. Esta tabela foi modificada em7 usando a licença Creative Commons (http://creativecommons.org/ licenses/by/4.0/) e mostra os tempos de resposta de seis de cada consulta para os três dobrando de tamanho bancos de dados usando o NoSQL existem DBMS e o tamanho de memória de os três bancos de dados. Também é mostrado o declive linear de cada consulta.
| Papel de braço | BRAÇO (s) | + Caminho do nó (s) | |
| Q1 | Consulta 2.1 | 0.191 | 24.866 |
| Q3 | Consulta 3.1 | 0,27 | 294.774 |
| Tamanho de banco de dados | 2,90 GB | 43,87 GB | |
| Total de extratos | 29.743 | 29.743 |
Tabela 5: média de tempos de resposta em segundos de consultas semelhantes a Q1 e Q3 dos sistemas relacionais melhorados apresentado em 10 . Esta tabela foi modificada em7 usando a licença Creative Commons (http://creativecommons.org/ licenses/by/4.0/) e mostra as duas consultas a maioria-semelhante a Q1 e Q3, apresentado em10 correspondente a dois sistemas de banco de dados relacional melhorada e seus tempos de resposta. Os tamanhos de dois banco de dados também são mostrados.
| ORM/MySQL | Taxa de transferência | Tempo de resposta |
| Q1 (s) | 4,711.60 | 0.0793 |
| Q3 (s) | 4,711.60 | 0.1558 |
| Q4 (s) | 4,711.60 | 0.9674 |
Tabela 6: média de tempo de resposta e throughput em segundos de consultas Q1, Q3 e Q4 de ORM MySQL SGBD relacional em execução simultânea. Esta tabela foi modificada em7 usando a licença Creative Commons (http://creativecommons.org/ licenses/by/4.0/) e mostra a maior taxa de transferência média de três consultas único paciente e seus tempos de resposta média em simultâneo o experiência de execução usando o sistema relacional MySQL ORM.
| MongoDB | Taxa de transferência | Tempo de resposta |
| Q1 (s) | 178,672.60 | 0,003 |
| Q3 (s) | 178,672.60 | 0.0026 |
| Q4 (s) | 178,672.60 | 0.0034 |
Tabela 7: média de tempo de resposta e throughput em segundos de consultas Q1, Q3 e Q4 de DBMS de NoSQL MongoDB em execução simultânea. Esta tabela foi modificada em7 usando a licença Creative Commons (http://creativecommons.org/ licenses/by/4.0/) e mostra a maior taxa de transferência média de três consultas único paciente e seus tempos de resposta média em simultâneo o experiência de execução usando o sistema de MongoDB NoSQL.
Complementar Figura 1: A imagem mostra a tela do software para se conectar ao servidor MySQL. Clique aqui para baixar esta figura.
Complementar Figura 2: A captura de tela mostra a interface do SQL para o servidor MySQL onde foi escrita a primeira consulta SQL. Clique aqui para baixar esta figura.
Complementar Figura 3: O MongoDB 2.6 localhost servidor é iniciado usando uma janela de sistema do DOS, executando o servidor mongod. Clique aqui para baixar esta figura.
Complementar Figura 4: A imagem mostra a consulta escrita nas caixas de texto do construtor de consultas, conforme mostrado nas etapas 5.7.1 através de 5.7.4. A imagem ilustra passo 5.7.3. , Por favor clique aqui para baixar esta figura.
Complementar Figura 5: A imagem mostra o passo 5.7.6. Clique aqui para baixar esta figura.
Complementar Figura 6: A imagem ilustra a escrita da consulta XPath no Upper parte da caixa de diálogo. Clique aqui para baixar esta figura.
Discussão
Este protocolo mostra que sistemas ORM relacionais puros não parece práticos para consultas único paciente (Q1, Q3 e Q4) desde os tempos de resposta são mais lentos, provavelmente devido a um elevado número de tabelas relacionais realizando muitas operações de junção caro e devido à sistema de armazenamento usado pelo tipo específico de banco de dados. Bancos de dados NoSQL armazenam dados em uma forma com base em documento, enquanto os sistemas relacionais usam uma forma baseada na tabela que se espalha cada documento em todo o banco de dados inteiro. NoSQL sistemas mostram uma inclinação linear, com MongoDB realizando consideravelmente mais rápido do que existe de DBMS. Simultaneidade, MongoDB também se comporta muito melhor do que relacionais MySQL ORM7.
Este protocolo apresenta um protocolo de resolução de problemas para os resultados apresentados em7 sobre o SGBD MySQL de ORM. O sistema MySQL foi atualizado para a versão mais recente e os resultados foram ligeiramente modificados. Além disso, um ponto crítico em sistemas baseados em documentos de NoSQL como MongoDB é que eles podem preservar a consistência quando armazenar informações médicas7 , porque quando um extrato de RSE é atualizado, ele não é substituído, mas um todo novo extrato com os novos dados é gerado e armazenado no sistema, e o extrato original é mantido. Isto é uma exigência rigorosa de informação médica, porque alguns médicos podem ter feito importantes decisões médicas baseadas nos dados originais.
O sistema de braço relacional melhorado drasticamente diminui o número de tabelas relacionais e melhora o desempenho relacional. Desde que ele modifica o esquema relacional, informação médica, realizada pelos extractos pode ser consultada, porém, extractos não possam ser recuperados em suas formas exatas do originais.
Para bancos de dados muito grandes no secundário usam (pesquisa), não está claro qual sistema de banco de dados é mais apropriado, já que as consultas de todos-paciente (Q2 e Q5) comportar-se melhor em ORM do que em sistemas NoSQL, mas esses sistemas executam melhor do que o simplificado relacional sistemas em 12. Consideramos o Q6 uma consulta especial entre prática clínica e secundário usar cujo comportamento não pode ser determinado pelos resultados gerados por estas experiências.
No entanto, uma limitação do método é o inavailability de experiências diretas comparando o relacional braço sistema melhorado com NoSQL MongoDB sobre prática único paciente, médico consultas com exatamente os mesmos dados usados no protocolo. Mantivemos os resultados de interpolação tabela 3 e tabela 5 sobre consultas único paciente até que a experiência incluindo braço otimizado no protocolo foi realizada. Deixamos estas experiências para aplicações futuras. Um passo crítico no âmbito do protocolo é a seleção de base de dados gratuita, versões de software semelhante de anos recentes, para que nós pode comparar o exato estado-de-the-art das três tecnologias.
Esta é uma das primeiras tentativas de comparar diretamente relacional e sistemas NoSQL usando informação médica real, realista, padronizada. No entanto, o sistema específico a ser usado depende muito o cenário real e o problema ser resolvido8.
Divulgações
Os autores não têm nada para divulgar. Os conjuntos de dados utilizados nesses experimentos foram fornecidos por vários hospitais espanhóis sob licença para estas experiências e, consequentemente, não são acessíveis ao público. O esquema XML de RM ISO/EN 13606 foi fornecido pelo centro da University College London para informática em saúde & multiprofissional educação (sinal sonoro).
Agradecimentos
Os autores gostaria de agradecer o Dr. Dipak Kalra, líder da força-tarefa EHRCom que definiu o ISO/EN 13606 padrão e sua equipe do University College de Londres para sua permissão usar o ISO/EN 13606 W3C XML Schema.
Este trabalho foi financiado pelo Instituto de Salud Carlos III [concessão números PI15/00321, PI15/00003, PI1500831, PI15CIII/00010 e RD16CIII].
Materiais
| Name | Company | Catalog Number | Comments |
| MySQL 5.7.20 | MySQL experiments | ||
| Red Hat Enterprise Linux Server release 7.4 (Maipo), 2.60GHz, RAM 16GB | |||
| MongoDB 2.6 | MongoDB experiments | ||
| Windows 7, 2.66GHz, RAM 12GB | |||
| eXist 3.0RC1 | eXist experiments | ||
| Windows 7, 2.66GHz, RAM 12GB | |||
| Studio 3T 5.5.1 | 3T Software Labs Gmbh | MongoDB GUI |
Referências
- Codd, E. F. A relational model for large shared data banks. Comm ACM. 13 (6), 377-387 (1970).
- Kalra, D., Lloyd, D. . ISO 13606 electronic health record communication part 1: reference model. , (2008).
- Kalra, D., et al. . Electronic health record communication part 2: archetype interchange specification. , (2008).
- Kalra, D., Beale, T., Heard, S. The openEHR foundation. Stud. Health Technol. Inform. 115, 153-173 (2005).
- Beale, T. Archetypes: constraint-based domain models for future proof information systems. OOPSLA, Workshop Behav Semant. , (2002).
- Sánchez-de-Madariaga, R., et al. Examining database persistence of ISO/EN 13606 standardized electronic health record extracts: relational vs. NoSQL approaches. BMC Medical Informatics and Decision Making. 32 (2), 493-503 (2017).
- Ireland, C., Bowers, D., Newton, M., Waugh, K. Understanding object-relational mapping: a framework based approach. Int. J. Adv. Softw. 2, 202-216 (2009).
- . Node+Path Persistence Available from: https://openehr.atlassian.net/wiki/spaces/dev/pages/6553626/Node+Path+Persistence (2017)
- Wang, L., Min, L., Wang, R., et al. Archetype relational mapping - a practical openEHR persistence solution. BMC Medical Informatics and Decision Making. 15, 88 (2015).
- Kaur, K., Rani, R. Managing data in healthcare information systems: many models, one solution. Computer. March. , 52-59 (2015).
- Sabo, C., Pop, P. C., Valean, H., Danciulescu, D. An innovative approach to manage heterogeneous information using relational database systems. Advances in Intelligent Systems and Computing. 557, (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados