Method Article
Detección de copia baja número DNA Viral integradora formada por In Vitro la infección Hepatitis B
En este artículo
Resumen
Aquí describimos la generación en vitro de ADN del VHB a través de un sistema de infección de virus de Hepatitis B y la detección altamente sensible de su integración de (1 – 2 copias) con inversa nested PCR.
Resumen
Hepatitis B Virus (HBV) es un patógeno común transmitidas por la sangre que causa cáncer de hígado y cirrosis hepática resultante de la infección crónica. Generalmente, el virus se replica por una ADN episomal intermedio; sin embargo, se ha observado integración de fragmentos de ADN del VHB en el genoma del anfitrión, aunque esta forma no es necesaria para la replicación viral. El propósito exacto, momento y mecanismos que ADN de VHB se produce integración es todavía no claro, pero recientes datos muestran que se produce muy temprano después de la infección. Aquí, la generación en vitro y detección de integraciones de HBV DNA se describen en detalle. Nuestro protocolo específicamente amplifica solo copias de integraciones de ADN de virus de la célula y permite la cuantificación absoluta y resolución solo-pares de la secuencia de salida. Esta técnica se ha aplicado a varios tipos de células susceptibles de HBV (incluyendo hepatocitos humanos primarios), a varios mutantes del VHB y en conjunto con varias exposiciones de la droga. Prevemos esta técnica convirtiéndose en una prueba clave para determinar los mecanismos moleculares subyacentes de este fenómeno clínico relevante.
Introducción
VHB es un virus de ADN bicatenario que pueden causar la infección crónica de toda la vida, cirrosis del higado y hepatocelular carcinoma (HCC)1,2,3. Mientras que hay múltiples mecanismos moleculares conduce HBV persistencia4 (p. ej., alta estabilidad de la plantilla transcripcional viral epigenética), evasión de la vigilancia inmune y baja rotación de hepatocitos en el hígado y sus asociados riesgo de HCC iniciación5,6 (p. ej., inflamación crónica y activación de celulares estrés vías), integración del ADN del VHB en el genoma de la célula huésped (un mecanismo reportado para ambos de estos fenómenos) ha sido poco estudiada . Una de las principales razones es la falta de adecuado en vitro infección sistemas de HBV que permite la detección confiable de eventos de integración. Aquí, describimos un protocolo desarrollado recientemente en vitro generación y detección de integraciones de HBV DNA que puede ser utilizado para dilucidar los mecanismos moleculares subyacentes y sus consecuencias.
Replicación del VHB y la formación de la integración de ADN del VHB ha sido revisada previamente en detalle7. Brevemente, VHB entra en hepatocitos con el péptido de Co-transporting Taurocholate del sodio (NTCP) como el principal receptor celular para infección8,9. Los nucleocapsids HBV que contiene el ADN circular relajado de HBV (rcDNA) genoma entra en el núcleo, donde la rcDNA se convierte en ADN circular covalentemente cerrado episomal (cccDNA). El cccDNA nuclear actúa como plantilla transcripcional viral mRNAs y pre-genomic RNA (pgRNA)10. PgRNA y HBV polimerasa entonces se empaquetan en nucleocapsids recién formado (formados por dímeros de la proteína core VHB). PgRNA VHB es transcripción inversa dentro de la nucleocápside, dando por resultado un genoma rcDNA o una doble cadena linear DNA (dslDNA) genoma11,12. Estos maduros nucleocápside que contiene genomas de ADN de VHB es finalmente envuelto y exportados como viriones.
Infección de hepatocitos por sobres partículas que contienen moléculas de dslDNA puede resultar en la integración viral en el anfitrión célula genoma13, conduce a formas de replicación incompetente de HBV DNA7,14,15. Integración de la DNA de HBV ocurre en el sitio de cromosómico doble cadena DNA roturas15. La acumulación de pruebas sugiere que cada evento de integración se produce en una posición esencialmente al azar en el anfitrión genoma celular16,17. Además, la integración de DNA de HBV ocurre algo raramente, a un ritmo de 1 por 104 células13,18,19,20. Preguntas importantes sobre la integración de ADN del VHB siguen sin respuesta, particularmente con respecto a la exactas vías moleculares involucradas, la dependencia de virales y factores del anfitrión, los antígenos virales expresados de forma integrada y su posible contribución persistencia viral7. Hemos establecido un modelo en vitro para arrojar luz sobre algunas de estas preguntas.
La rareza de HBV DNA eventos de integración (tanto con respecto a la tasa de integración por la célula y el número de copias de cada integración única) en la infección por HBV en vitro modelos hacen ellos difíciles de detectar. Mitosis de la célula es limitado en nuestro sistema in vitro , como divisoria de las células no son compatibles con infección eficiente. Así, a diferencia de en paciente tejidos del hígado donde produce la gran expansión clonal de hepatocitos18,19,20, muy que pocas copias (1-2) de cada integración están presentes en un determinado grupo de células infectadas en vitro . También hemos encontrado que la integración ocurre principalmente durante la infección inicial de hepatocitos (y no continuamente en los hepatocitos infectados crónicamente)13. Por consiguiente, integración de ADN del VHB no puede aumentarse simplemente cultivo de células para un período más largo.
En general, los métodos utilizados previamente para detectar ADN de VHB integrado, incluyendo meridional blot hibridación21,22,23,24,25, Alu-PCR26,27 , ligadura mediada por cassette PCR28y genoma secuencia29,30,31,32,33, no tienen la sensibilidad para detectar solo copias de integraciones. Nosotros y otros investigadores hemos utilizado inversa nested PCR (invPCR) para detectar la integración de ADN hepadnaviral en pato, marmota y hígados infectados humanos13,14,18,19, 34,35,36. Otros han introducido modificaciones procesales para la técnica de invPCR que pueden alterar la generación de señales de falsos positivos y restringir la capacidad de cuantificación37. Si el ensayo se realiza como se describe en este protocolo, invPCR representa un análisis sensible y específico que identifica y cuantifica (en números absolutos copia) múltiples integraciones de HBV DNA. El cruce de ADN VHB-célula se ordena en una resolución de la solo-base par, permitiendo estudios bioinformatical de viral y anfitrión DNA secuencias en los sitios de integración13.
Anteriormente hemos descrito38 resultados de invPCR en el tejido infectado por el ADN de VHB extraído de una amplia gama de fuentes, incluyendo los tejidos del hígado congelado rápido, secciones parafina-encajadas de hígado y muestras de tejido muy pequeña aisladas por Microdisección láser19. Este protocolo describe una actualización de un análisis de invPCR utilizando el ADN extraído de tejidos derivados de cultura de la célula después de la infección en vitro , en el cual se generan números de copia baja de integraciones (1 – 2 copias por integración). La HBV DNA integraciones formados en vitro se asemejan a las que se encuentran en los tejidos del paciente con respecto a su distribución en el genoma celular y conexiones dentro de la secuencia viral que es integrado13,16, sugiriendo un vía comparable a dentro un hígado infectado.
Protocolo
1. infección y extracción de ADN de la célula
- Mantener cultivadas Huh7 NTCP células8,9 en medio modificado Eagle de Dulbecco (DMEM) suplementado con 10% v/v suero fetal bovino, 1 x y 2 mM L-glutamina, penicilina/estreptomicina.
Nota: Esto se ha divulgado previamente en detalle40. - Semilla 2 x 105 células/mL Huh7 NTCP células en una placa de 12 pozos con 1 mL de DMEM.
- Si probando tratamientos con células (p. ej., inhibidores de la VHB), aplicarlos a la cultura sobrenadante 4 h después de la siembra (1 día antes de la infección por VHB). Para un control negativo, aplicar 200 nM Myrcludex B (un potente HBV entrada inhibidor41).
- Usar sobrenadante de42 columnas heparina purificada de HepAD3843 como un inóculo para infectar células en 1.000 VGE/celular en 500 μl de medio de cultivo (DMEM suplementado con suero bovino fetal 10% v/v, 1 x 2 mM L-glutamina y penicilina/estreptomicina y 2.5% v/v DMSO), que contiene 4% p/v polietilenglicol 8000 [disuelto en 1 x solución salina tamponada con fosfato (PBS)].
- Las células en una incubadora de 37 ° C (fijado en 5% CO2 y el 90% de humedad) de la cultura.
- Lavan las células dos veces con 1 mL de PBS estéril de 1 x a post-infección 16-24 h.
- Sustituir los medios de cultivo cada 2 días después de la infección hasta la cosecha.
- En infecciones posterior al día 3, tratar las células con 5 μm Tenofovir disoproxil y 10 μm lamivudina para limitar la producción de productos intermedios replicativos HBV que amplificable por invPCR.
- Infección posterior al día 5, trypsinize las células con 200 μL de tripsina-EDTA y resuspender en 2 mL de DMEM (suplementado con suero bovino fetal 10% v/v, 1 x penicilina/estreptomicina y 2 mM L-glutamina), que también contiene 5 μm Tenofovir disoproxil y 10 μm Lamivudina.
- Transferencia de las suspensiones celulares a una placa de 6 pozos para inducir a una ronda de la mitosis (que se ha divulgado para inducir pérdida de HBV cccDNA44 y otros intermedios de HBV DNA que son amplificable por invPCR).
- Post-infección día 7, trypsinize las células ampliadas en 400 μL de tripsina-EDTA y agite la mezcla en 1 mL de DMEM. Coloque la suspensión en un tubo de 1,5 mL, sedimenten las células por centrifugación a 500 x g durante 5 minutos y retirar el sobrenadante por aspiración.
- (Opcional) Almacenar los pellets de células a-20 ° C hasta que estén listos para la extracción de ADN.
- Extraer el ADN del precipitado de células usando un kit de extracción de ADN, según las instrucciones del fabricante. Calcular la concentración final de ADN mediante densitometría óptica.
Nota: El rendimiento de ADN en un volumen de elución de 100 μL es generalmente ~ 250-400 ng/μL.
2. inversión de la DNA
- ~1.5–2.5 alícuota μg del ADN total extraído de paso 1 en un tubo PCR 200 μL. Añadir la cantidad apropiada de mezcla de maestra de enzima de la restricción para dar lugar a un volumen de reacción 40 μL que contiene 1 x buffer de reacción de síntesis (e.g., buffer de CutSmart) y 10 U NcoI-HF.
- Homogeneizar las reacciones y girar hacia abajo en una centrifugadora de tubo pequeño. Incubar la reacción de la enzima de la restricción en una máquina PCR a 37 ° C por 1 h para la eficacia óptima digestión.
- Inactivar la enzima de la restricción por la incubación a 80 ° C por 20 min.
- La transferencia de la reacción de la enzima de la restricción entera a un tubo de 1,5 mL. Añadir 400 μL de 1 x de buffer de T4 DNA ligasa y 500 U de T4 ADN ligasa y mezclar bien. El volumen de reacción grande anima intra molecular (a diferencia de la molecular) ligadura de los fragmentos de ADN digeridos.
- Incubar la reacción de la ligadura a temperatura ambiente durante 2 h para ligadura completa.
- Inactivar el ligase de la DNA de T4 a 70 ° C por 20 min.
- Añada 10 μL de 10% w/v sodio dodecil sulfato para asegurar la completa inactivación de la ligasa de T4.
- Mezcle los tubos Vortex de pulso y girar brevemente por la mezcla de reacción. Añadir NaCl a una concentración final de 100 m m y dextrano (35-45 kDa) a una concentración final de 90 μg/mL. Mezcle los tubos Vortex de pulso y girar brevemente por la mezcla de reacción.
- Agregar 900 μL de etanol al 100% y mezclar por inversión. Precipitar el ADN a-20 ° C durante la noche.
- Pelotilla de la DNA precipitada por centrifugación a 14.000 x g durante 15 minutos quite el sobrenadante por aspiración con una pipeta de P200. Lavar el pellet con 500 μL de etanol al 70% v/v y centrifugación a 14.000 x g durante 15 minutos.
- Eliminar el etanol por aspiración con una pipeta de P200. Secar el pellet de ADN a temperatura ambiente durante 20 minutos.
- Disolver los pellets en 20 μL de H2O. agregar 20 μL de una mezcla maestra de enzima de la restricción para dar lugar a un volumen de reacción 40 μL que contiene 1 x buffer de reacción digest, 5 U de BsiHKAI y 5 U de Sph-HF incubar la reacción de la enzima de la restricción en una bloque de calor a 37 º C (la temperatura óptima para el Sph-HF) durante 1 hora.
- Girar brevemente por la mezcla de reacción. Incubar la reacción de la enzima de la restricción en un bloque de calor a 65 ° C (la temperatura óptima para BsiHKAI) durante 1 hora.
- Girar brevemente por la mezcla de reacción.
- Almacenar el ADN invertido a-20 ° C hasta el siguiente paso.
3. anidado PCR
- Quitar amplicones de potencial de un sello de silicona reutilizable mat usando una solución de degradación de ADN, enjuagar la alfombra con agua libre de ADN y secado con aire a temperatura ambiente mientras se prepara la mezcla PCR.
- Preparar 1 mL de una mezcla de x PCR 1 que contiene el delantero externo (5'-TTC GCT TCA CCT CTG CAC G-3') y reversa (5'-AAA GGA CGT CCC GCG CAG-3') cebadores en una concentración de 0,5 μm.
- Añadir 170 μL de la mezcla de x PCR 1 a los pocillos A1 y E1 en una placa de PCR de 96 pocillos.
- Añadir 120 μL de la mezcla de x PCR 1 a pozos B1 a H1.
- Añadir 10 μL del ADN invertido del paso 2 en los pocillos A1 y E1 (2 diferentes muestras invertidas pueden analizarse en la misma placa PCR). Mezcla de la reacción en cada pozo transfiriendo suavemente unas 10 veces usando una P1000 a 100 μL.
- Muestras diluidas en serie de pozos A1 a D1 en una proporción de 1:3 por la transferencia de 60 μL a cada paso. Mezcla de los pozos en cada paso transfiriendo suavemente les sobre 10 veces usando una P1000 a 100 μL. Evitar la formación de burbujas. Repita el paso 3.6 para E1 bien, diluir hasta H1 bien.
- Alícuota de 10 μL de la mezcla de reacción mediante una pipeta multicanal de pozos A1-H1 en pocillos A2-H2, A3-H3, y así sucesivamente hasta llegar a A12-H12 los pocillos de la placa de 96 pocillos. Cubra la placa de la polimerización en cadena con la placa de silicio seco del paso 3.1, presionando firmemente.
- Colocar la placa en una máquina PCR y ejecutar el siguiente programa: 10 min a 95 ° C; 35 ciclos de 15 s a 95 ° C, 15 s a 54 ° C y a 3 min a 72 ° C; 7 minutos a 72 ° C; Luego, sostenga la placa a temperatura ambiente.
- Los pernos de un replicador de 96 pines para partir con un mechero de Bunsen de calor y luego enfriar durante al menos 5 minutos a temperatura ambiente.
- Rellenar los pocillos de una placa PCR de segunda con 10 μL de mezcla de x PCR 1 (que contiene un tampón de carga-lista de gel) y el delantero interno (5'-CGC ATG GAG ACC ACC GTG A-3') y reversa (5'-CAC AGC CTA GCA ATG de GCC G-3') a 0,5 μm.
- Cuidadosamente retire la placa de silicio de la placa de la polimerización en cadena de la placa de 96 pozos de la primera ronda PCR y evitar la contaminación cruzada entre pozos.
- Utilizar el replicador de enfriado para transferir los productos PCR de la placa de 96 pocillos de PCR de la primera ronda a la placa de 96 pocillos nueva alícuotas. Llevar a cabo la PCR anidada utilizando las mismas condiciones que en el paso 3.8, excepto cambiando el paso de desnaturalización inicial a 95 ° C de 10 min a 2 min.
4. PCR producto aislamiento y extracción de Gel
- Analizar los productos PCR por electroforesis del gel mediante un pozo de 96 con gel de agarosa de w/v de 1.3%. Un gel de agarosa de 100 mL, ejecutar a 200 V durante 10 – 15 minutos.
- Suprimir las bandas de DNA de los geles de agarosa utilizando pajitas de beber desechables. Para cada producto PCR, coloque la paja y gel de agarosa Conecte un tubo de 1,5 mL y recorte la paja a medida con unas tijeras.
Nota: Es suficiente para aislar grupos solamente de las diluciones en las que se pueden resolver solo productos de la PCR. - (Opcional) Almacenar los tubos a-20 ° C para posterior extracción.
- Expulsar los tacos de agarosa en cada tubo, apretando en el extremo del fragmento paja. Añadir 300 μL de tampón de extracción de gel y 5 μL de la mezcla de gel de extracción de vidrio grano a cada tubo.
- Extraer los productos PCR según las instrucciones del fabricante para el kit de extracción de gel (excepto usando volúmenes de medio para el lavado de pasos) y eluir el ADN de los granos con 30 μL de agua.
- Presentar el ADN purificado de Sanger secuenciación (según las instrucciones del proveedor) con el primer avance en la segunda ronda de polimerización en cadena jerarquizada.
5. Análisis de la secuencia
- Confirmar a las ensambladuras de ADN de virus-célula realizando análisis de explosión de nucleótido (usando alineando a la colección de nucleótido entero por defecto).
Nota: Resultados representativos se detallan en la figura 3 .- Si sólo se observa un alineamiento parcial de la secuencia de corte 5' secuencia de la DNA de HBV antes de volver a ejecutar un análisis de la explosión.
- Aplicar criterios de selección rigurosos para determinar si una secuencia dada representa una Unión de verdadera integración como sigue:
- Incluyen solamente las secuencias que contienen > 20 bp de una secuencia celular seguro asignación al genoma celular.
- No cualquier secuencia que contienen sitios de restricción de cualquier enzimas usadas en 10 bp de la ensambladura de la integración de supuestos virus-célula, que probablemente representan vitro ligadura eventos en lugar de uniones de verdadera integración.
- Caso omiso de cualquier secuencia con los niveles de fluorescencia evidente pico diferentes a cada lado de la ensambladura de supuesta integración en Cromatógrafos de secuenciación, ya que son artefactos probablemente generados por la contaminación cruzada durante la reacción de secuenciación o cromatografía capilar.
- Definir eventos de integración único que con el VHB exacta y secuencias celulares en el cruce de virus-célula.
Nota: Repetición de eventos de integración único puede resultar de la expansión clonal de las células que contienen esas integraciones o contaminación cruzada durante la PCR (que puede ser probada usando reacciones de control negativo, más detalladas en los resultados representativos a continuación). Integraciones repetidas en secuencias celulares específicas se esperan para mostrar diferentes sitios terminales de HBV para cada evento de integración, como vías de unirse a final no homóloga son usados15.
- Calcular la frecuencia de integración multiplicando el factor de dilución de las plantillas de ADN invertidos por el número de uniones célula virus detectado en esa dilución, seguido de la normalización a la cantidad de entrada total de ADN en la reacción de inversión. En general, la frecuencia de integración es del orden de 1:104 células.
Resultados
Una representación esquemática de la técnica de invPCR se muestra en la figura 1. Una electroforesis en agarosa al ejemplo de una invPCR exitosa se muestran en la figura 2 antes y después del aislamiento del producto PCR. Figura 3 muestra el resultado de análisis de la explosión del paso 5.1 en los casos de (i) amplificación de unión de ADN de virus-célula y (ii) la amplificación de un intermediario replicativo de HBV DNA.
Resultados de controles positivos esperados: células Huh7 NTCP infectadas con cepas HBV heparina purificada como se describió anteriormente (figura 1) pueden servir como un control positivo. En este caso, cada producto PCR se debe alcanzar por la dilución de 1:3 de segunda o tercera de cada muestra (correspondiente a ~ 104 equivalentes de la célula por dilución, figura 2). En estas diluciones, aproximadamente el 50% de los productos representará a cruces de ADN verdadero virus-célula, mientras que la otra mitad representa la amplificación de productos intermedios de la DNA de HBV (figura 3).
En anteriores estudios13, hemos encontrado pocos casos de uniones repetidas virus-célula, no sugiriendo celulares sitios genómicos de integración preferente de HBV DNA. También encontramos que >80% cruces de virus-célula ocurren entre los nucleótidos 1732 y 1832 (según el nucleótido numeración del genotipo del VHB D, GenBank accesión #U95551.1), donde este último representa la terminal derecha de la forma de dslDNA HBV. Secuencias de ensambladuras del verdadero virus-célula encontradas a través de nuestro método en experimentos en vitro están públicamente disponibles en GenBank (números MH057851 a MH058006).
Espera que los resultados de los controles negativos: no infectadas las células Huh7 NTCP o inoculados Huh7 NTCP células previamente tratadas con Myrcludex B pueden actuar como controles negativos. InvPCR análisis de la DNA extraída de estas células no debe producir ningún producto PCR. Corriendo estas muestras control negativo en la misma placa PCR como muestras positivas permite probar la contaminación cruzada durante el proceso de la PCR-anidada. La prueba más rigurosa para la contaminación cruzada es el funcionamiento de una muestra control negativo en los pozos A-D y la muestra positiva en pozos E-H sobre una placa de 96 pocillos. En este caso, la dilución más concentrada de la muestra positiva se ejecuta en una fila junto a la menos concentrada dilución de la muestra negativa. Si los productos de amplificación se observan en las muestras control negativo, Instituto de las medidas sugeridas en la discusión para reducir al mínimo la contaminación cruzada de eventos.
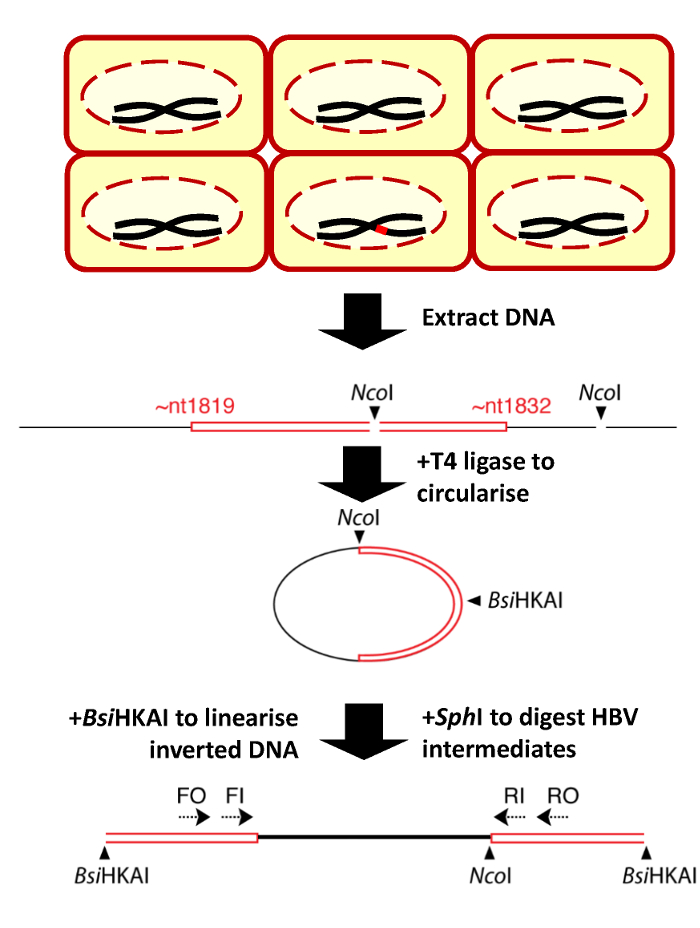
Figura 1: Figura esquemática del proceso de invPCR para detectar integraciones de HBV DNA. Después de la infección de HBV, sólo una minoría de células Huh7 NTCP contiene VHB dslDNA (rojo) integra en el genoma de la célula huésped (rojo), representado en la parte superior. ADN total es entonces extraído de las células (paso 1), invertida (paso 2) y amplificado por PCR anidada (paso 3). Se muestran las posiciones relativas de los sitios de restricción enzimática (Ncoy BsiHKAI) en el cruce de ADN de células de virus suprimido. La figura es una adaptación de una anterior publicación13. Haga clic aquí para ver una versión más grande de esta figura.
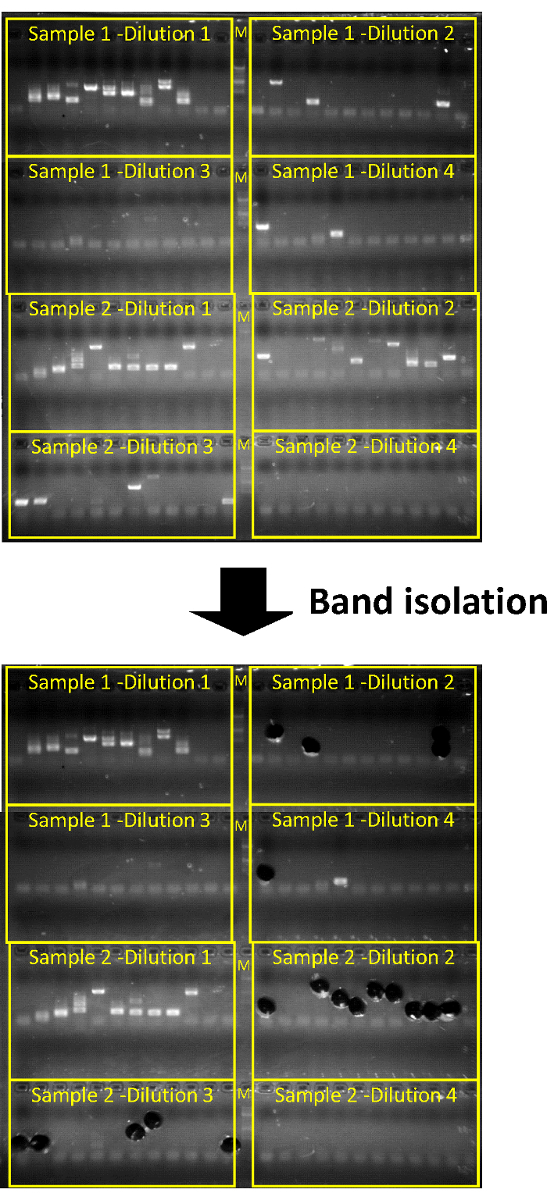
Figura 2: gel de agarosa electroforesis y extracción de gel posterior de un exitoso invPCR. "M" representa la escala de marcadores de ADN. Cada fila de 12 representa repeticiones técnicas en una sola dilución en la placa de la polimerización en cadena. Dos muestras de ADN invertidas se pueden ejecutar por gel de agarosa/placa PCR. Los productos PCR para cada dilución deben valorar hacia fuera en a ~ 1:3 cociente. Extracción de los productos de las dos diluciones menos concentradas donde bandas solo pueden ser fácilmente aislados es suficiente, como tasa de integración de DNA de HBV se determina por titulación punto final. Haga clic aquí para ver una versión más grande de esta figura.
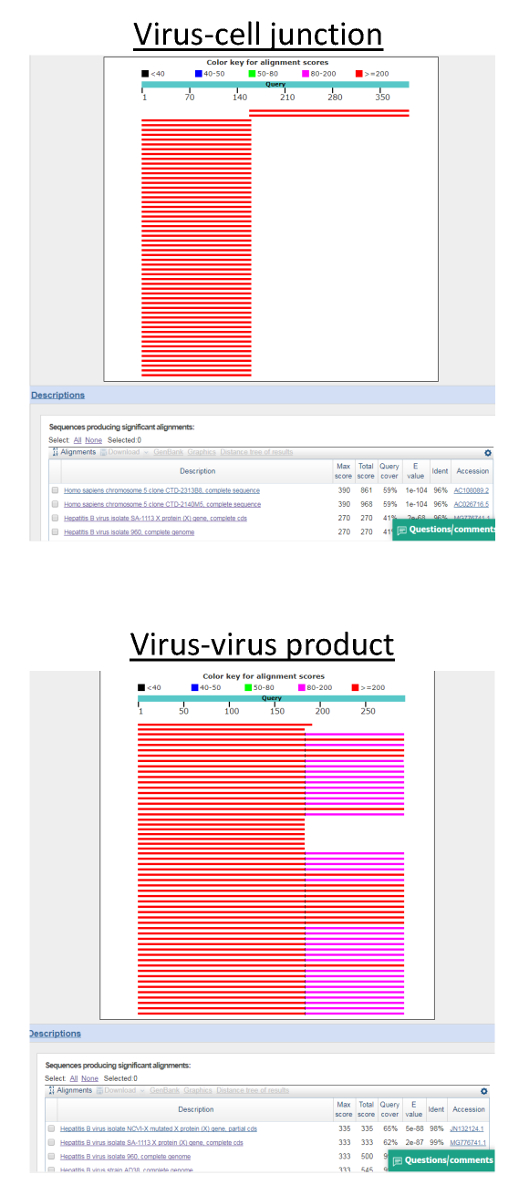
Figura 3: Análisis de la explosión de productos invPCR. Como largas extensiones de secuencia se generan de Sanger secuenciación, la fuente de secuencias de la DNA es generalmente clara. Fuerte alineación para VHB y genomas celulares se observa en distintas áreas del archivo de secuencia FASTA, generados a partir de Sanger secuenciación en el panel superior, lo que sugiere un cruce de ADN verdadero virus-célula. En el panel inferior, la secuencia se alinea solamente con fragmentos del genoma del VHB, lo que sugiere un producto generado por la amplificación de un genoma de HBV DNA cambiado. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Antes de realizar el protocolo, es importante tener en cuenta que este ensayo invPCR es una técnica altamente sensible, capaz de amplificar solo copias de la plantilla de la DNA. Por lo tanto, limitar la contaminación de productos de la PCR es de suma importancia. Estrategias generales para limitar la contaminación del producto PCR los siguientes. (i) establecer áreas separadas físicamente para los diferentes pasos del método. Cada área debe tener batas de laboratorio independiente, y guantes deben cambiarse cuando se mueve entre estas áreas. Hemos enumerado estas áreas a continuación en orden de más a menos probable ser contaminado: PCR producto extracción y secuenciación de montaje zona de reacción (post-PCR); Además de la plantilla PCR y pin flameado de transferencia de zona (hemos utilizado campanas de PCR con una lámpara UV descontaminante con buenos resultados); Área de extracción e inversión de DNA (pre-PCR); y una zona de "ADN plantilla libre" utilizado exclusivamente para la preparación de acciones y soluciones de la polimerización en cadena. (ii) ser conscientes del flujo de aire en el laboratorio como un conductor de potencial de contaminación cruzada. En particular, la contaminación cruzada de las reacciones de PCR durante la etapa de transferencia pin flameado es más probable que se producen y se conducen a la cuantificación incorrecta de la frecuencia de la integración. Capillas de la PCR pueden utilizarse para limitar estas corrientes cruzadas. Pozos de control negativo (p. ej., tratados con Myrcludex B muestras o no controles de plantilla) en la PCR también pueden utilizarse para probar la contaminación cruzada. (iii) limitar los contaminantes potenciales de la PCR en pipetas y las superficies de trabajo limpiándolos regularmente hacia abajo con una solución de degradación del ADN.
El protocolo especificado aquí se arregla para detectar la integración de un conocido clon de VHB infeccioso generado a partir de la HepAD38 de la célula línea42. Si el inóculo utilizado es de una secuencia diferente de ADN VHB (por ej., de suero del paciente), entonces el genoma HBV debe secuenciado primero para confirmar la compatibilidad de primers PCR y el diseño de inversión. En los estudios previamente publicados19, hemos utilizado 3 juegos de cebadores que se unen a secuencias de HBV DNA conservadas flanqueando el lugar esperado de uniones de integración de ADN del VHB. Otras secuencias de la cartilla y los protocolos se han descrito para determinar la secuencia genomic de HBV44,45,46,47,48 y pueden funcionar con éxito.
Además, pueden utilizarse diferentes células susceptibles de HBV (por ejemplo, las células HepG2 NTCP primaria hepatocitos humanos, distinguido HepaRG,); sin embargo, hemos encontrado que las células Huh7 NTCP proporcionan la mayor relación de señal a ruido, al considerar el número de virus-célula ensambladuras de la DNA que son amplificadas en comparación con el DNA del virus intermedio que es amplificado13. En particular, células terminalmente diferenciadas como hepatocitos humanos primarios o HepaRG diferenciado no eficientemente sufren mitosis, dando lugar a un alto nivel de amplificable intermedios replicativos de HBV DNA restante dentro de las células. Hemos encontrado que el ~ 90% de secuencias amplificadas representan reordenamientos de ADN VHB (y no eventos de integración) en células diferenciadas terminalmente, en comparación con 70 – 50% de las líneas de células de hepatoma13. Estos productos son generalmente genomas de ADN de VHB que contienen canceladuras grandes en la superficie y la base de marcos de lectura abierta, o pueden representar cuasi-especies HBV con un adicional Ncodel sitio a antes de Sphsitio. La amplificación de estas especies HBV (incluyendo un diagrama esquemático) se han descrito en detalle previamente13.
Hay varias desventajas a este método. Debido a la naturaleza laboriosa y varios día de este protocolo, invPCR no es adecuado para análisis de alto rendimiento de un gran número de muestras. Además, como se basa en limitar la valoración de la dilución, nuestro método no es muy precisa en la cuantificación; Aunque, deben medir fácilmente cambios de nivel de registro en la frecuencia de integración.
Además, nuestro protocolo de inversión sólo es adecuado para detectar integraciones que ocurren entre la región DR2 y DR1 del genoma del VHB, como la mayoría de integraciones de HBV ocurren dentro de esta región49. NGS el análisis de tejidos del paciente VHB ha demostrado que una gran minoría (hasta ~ 50%) puede también ocurrir fuera de esta región49. Nuevos diseños de invPCR son teóricamente capaces de detectar estos otros sitios de la integración, aunque ellos no (a nuestro conocimiento) han llevado a cabo todavía. Correlativamente, debido a los sitios de la enzima de restricción necesaria requeridos aguas abajo de la secuencia de cruce de virus-célula de la reacción de inversión, invPCR no detecta todas las integraciones que ocurren dentro de las regiones del genoma HBV DR2 y DR1 (es decir, han estimado que el ~ 10% de integraciones todos son detectable usando en silico simulaciones13). Sin embargo, cuando se aplica a un lote centrado de muestras con una pequeña cantidad de diferentes tratamientos, invPCR es uno de los métodos sólo prácticos para la detección de ADN de VHB integrado en una sola resolución de base par.
Visualizamos, por tanto, aplicaciones de este método que sirve un papel clave en la búsqueda de virus (a través de mutaciones del inóculo HBV), celular (golpe de gracia o la sobreexpresión de genes celulares específicos o a través de aplicación de varias drogas) y ambientales ( por ejemplo, la exposición a estrés oxidativo) factores que inducen la integración del DNA del VHB. Con este método y sistemas de infección de HBV recién desarrollados, nos permiten un control sin precedentes de estos factores para que sean bien aisladas y mejor caracterizado. También esperamos que esto conduzca a una comprensión clave de las consecuencias celulares de la integración de ADN HBV, incluyendo a qué antígenos víricos de la medida (por ejemplo, HBx o HBsAg50) se expresan de forma integrada, lo que controla esta expresión, y si cambia o no integración de HBV significativamente el fenotipo celular hacia un estado más pro-oncogénico. Los resultados de estos estudios futuros tendrán un profundo impacto en las estrategias terapéuticas que se usa para tratar la hepatitis crónica B y en la comprensión básica del virus sí mismo.
Divulgaciones
S.U. es un co-solicitante y co-inventor de patentes protege B Myrcludex como un inhibidor de HBV/HDV. T.T. no declara conflictos de interés.
Agradecimientos
Este trabajo recibida financiación del centro alemán para investigación de infección (DZIF), TTU Hepatitis proyectos 5.807 5.704, la TRR179 de la Deutsche Forschungsgemeinschaft (DFG) (TP 15) y el centro australiano para la investigación de virología Hepatitis y VIH.
Estamos agradecidos al profesor William Mason para su parte en el desarrollo del método original de invPCR y demostrando a nosotros. Nos gustaría agradecer a los Drs. Yi Ni y Florian A. Lempp reactivos (líneas celulares e inóculo HBV). Reconocemos Anja Rippert, Franziska Schlund, Sarah Engelhardt y Dr. Katrin Schöneweis para asistencia técnica. Agradecemos a Miriam Kleinig para corrección y a profesores Nicholas Shackel y Ralf Bartenschlager para apoyo continuo.
Materiales
| Name | Company | Catalog Number | Comments |
| Dulbecco’s PBS | PAA | H15-002 | |
| DMEM medium | Thermo Fisher Scientific | 41965 | |
| Fetal bovine serum | PAA | A15-151 | Heat-inactivate before use |
| Penicillin, 10,000 U/mL; Streptomycin 10mg/ml, 100× | PAA | P11-010 | |
| L-glutamine, 200 mM | PAA | M11-004 | |
| Trypsin, 0.5 mg/mL; EDTA, 0.22 mg/mL, 1× | PAA | L11-004 | |
| PEG, MW 8000 | Sigma-Aldrich | 89510 | Stock at 40% w/v in 1xPBS, autoclave before use |
| DMSO for spectroscopy | Merck | 102950 | |
| Tenofovir disoproxil | Sigma-Aldrich | CDS021622 | Dissolve in DMSO |
| Lamivudine | Sigma-Aldrich | L1295 | Dissolve in DMSO |
| NucleoSpin Tissue kit | Macherey Nagel | 740952.250 | |
| NanoDrop 2000/2000c Spectrolphotometer | Thermo Fisher Scientific | ND-2000 | |
| NcoI-HF | NEB | R3193L | |
| T4 DNA ligase | NEB | M0202T | |
| Sodium dodecyl sulfate | Sigma-Aldrich | L3771 | |
| Sodium Chloride | Sigma-Aldrich | 433209 | |
| Dextran (35-45 kDa) | Sigma-Aldrich | D1662-10G | |
| Absolute Ethanol | Sigma-Aldrich | 32205-1L-D | |
| BsiHKAI | NEB | R0570L | |
| SphI-HF | NEB | R3182L | |
| Amplitaq Gold Taq kit | Thermo Fisher Scientific | 4331816 | Use for first nest PCR |
| Silicon sealing Mats for 96-Well PCR Plates | Biorad | 2239442 | |
| DNAZap PCR DNA Degradation Solution | Thermo Fisher Scientific | AM9890 | |
| 96-well PCR plates | Sigma Aldrich | CLS6509 SIGMA | |
| 96-pin replicator | Thermo Fisher Scientific | 250520 | |
| GoTaq Flexi PCR kit | Promega | M8295 | Use for second nest PCR, use green buffer for easy loading of agarose gel |
| Biozym LE Agarose | Biozym | 840004 | |
| QIAEX II Gel extraction kit | QIAGEN | 20051 |
Referencias
- Lin, X., et al. Chronic hepatitis B virus infection in the Asia-Pacific region and Africa: review of disease progression. Journal of Gastroenterology and Hepatology. 20 (6), 833-843 (2005).
- Iloeje, U. H., et al. Predicting cirrhosis risk based on the level of circulating hepatitis B viral load. Gastroenterology. 130 (3), 678-686 (2006).
- Huang, Y. T., et al. Lifetime risk and sex difference of hepatocellular carcinoma among patients with chronic hepatitis B and C. Journal of Clinical Oncology. 29 (27), 3643-3650 (2011).
- Nassal, M. HBV cccDNA: viral persistence reservoir and key obstacle for a cure of chronic hepatitis B. Gut. 64 (12), 1972-1984 (2015).
- Tu, T., Budzinska, M. A., Shackel, N. A., Jilbert, A. R. Conceptual models for the initiation of hepatitis B virus-associated hepatocellular carcinoma. Liver International. 35 (7), 1786-1800 (2015).
- Tu, T., Buhler, S., Bartenschlager, R. Chronic viral hepatitis and its association with liver cancer. Biological Chemistry. 398 (8), 817-837 (2017).
- Tu, T., Budzinska, M. A., Shackel, N. A., Urban, S. HBV DNA Integration: Molecular Mechanisms and Clinical Implications. Viruses. 9 (4), (2017).
- Yan, H., et al. Sodium taurocholate cotransporting polypeptide is a functional receptor for human hepatitis B and D virus. eLife. 1, e00049 (2012).
- Ni, Y., et al. Hepatitis B and D viruses exploit sodium taurocholate co-transporting polypeptide for species-specific entry into hepatocytes. Gastroenterology. 146 (4), 1070-1083 (2014).
- Tuttleman, J. S., Pourcel, C., Summers, J. Formation of the pool of covalently closed circular viral DNA in hepadnavirus-infected cells. Cell. 47 (3), 451-460 (1986).
- Staprans, S., Loeb, D. D., Ganem, D. Mutations affecting hepadnavirus plus-strand DNA synthesis dissociate primer cleavage from translocation and reveal the origin of linear viral DNA. Journal of Virology. 65 (3), 1255-1262 (1991).
- Wang, G. H., Seeger, C. The reverse transcriptase of hepatitis B virus acts as a protein primer for viral DNA synthesis. Cell. 71 (4), 663-670 (1992).
- Tu, T., Budzinska, M. A., Vondran, F. W. R., Shackel, N. A., Urban, S. Hepatitis B virus DNA integration occurs early in the viral life cycle in an in vitro infection model via NTCP-dependent uptake of enveloped virus particles. Journal of Virology. , (2018).
- Yang, W., Summers, J. Integration of hepadnavirus DNA in infected liver: evidence for a linear precursor. Journal of Virology. 73 (12), 9710-9717 (1999).
- Bill, C. A., Summers, J. Genomic DNA double-strand breaks are targets for hepadnaviral DNA integration. Proceedings of the National Academy of Sciences of the United States of America. 101 (30), 11135-11140 (2004).
- Budzinska, M., Shackel, N. A., Urban, S., Tu, T. Sequence Analysis of Integrated Hepatitis B Virus DNA during HBeAg-Seroconversion. Emerging Microbes and Infections. , (2018).
- Budzinska, M., Shackel, N. A., Urban, S., Tu, T. Cellular genomic sites of HBV DNA integration. Genes. , (2018).
- Mason, W. S., Liu, C., Aldrich, C. E., Litwin, S., Yeh, M. M. Clonal expansion of normal-appearing human hepatocytes during chronic hepatitis B virus infection. Journal of Virology. 84 (16), 8308-8315 (2010).
- Tu, T., et al. Clonal expansion of hepatocytes with a selective advantage occurs during all stages of chronic hepatitis B virus infection. Journal of Viral Hepatitis. 22 (9), 737-753 (2015).
- Mason, W. S., et al. HBV DNA Integration and Clonal Hepatocyte Expansion in Chronic Hepatitis B Patients Considered Immune Tolerant. Gastroenterology. 151 (5), 986-998 (2016).
- Shafritz, D. A., Shouval, D., Sherman, H. I., Hadziyannis, S. J., Kew, M. C. Integration of hepatitis B virus DNA into the genome of liver cells in chronic liver disease and hepatocellular carcinoma. Studies in percutaneous liver biopsies and post-mortem tissue specimens. New England Journal of Medicine. 305 (18), 1067-1073 (1981).
- Hino, O., et al. Detection of hepatitis B virus DNA in hepatocellular carcinomas in Japan. Hepatology. 4 (1), 90-95 (1984).
- Fowler, M. J., et al. Integration of HBV-DNA may not be a prerequisite for the maintenance of the state of malignant transformation. An analysis of 110 liver biopsies. Journal of Hepatology. 2 (2), 218-229 (1986).
- Chen, J. Y., Harrison, T. J., Lee, C. S., Chen, D. S., Zuckerman, A. J. Detection of hepatitis B virus DNA in hepatocellular carcinoma: analysis by hybridization with subgenomic DNA fragments. Hepatology. 8 (3), 518-523 (1988).
- Esumi, M., Tanaka, Y., Tozuka, S., Shikata, T. Clonal state of human hepatocellular carcinoma and non-tumorous hepatocytes. Cancer Chemotherapy and Pharmacology. 23 Suppl, S1-S3 (1989).
- Murakami, Y., et al. Large scaled analysis of hepatitis B virus (HBV) DNA integration in HBV related hepatocellular carcinomas. Gut. 54 (8), 1162-1168 (2005).
- Kimbi, G. C., Kramvis, A., Kew, M. C. Integration of hepatitis B virus DNA into chromosomal DNA during acute hepatitis B. World Journal of Gastroenterology. 11 (41), 6416-6421 (2005).
- Tamori, A., et al. Alteration of gene expression in human hepatocellular carcinoma with integrated hepatitis B virus DNA. Clinical Cancer Research. 11 (16), 5821-5826 (2005).
- Sung, W. K., et al. Genome-wide survey of recurrent HBV integration in hepatocellular carcinoma. Nature Genetics. 44 (7), 765-769 (2012).
- Fujimoto, A., et al. Whole-genome sequencing of liver cancers identifies etiological influences on mutation patterns and recurrent mutations in chromatin regulators. Nature Genetics. 44 (7), 760-764 (2012).
- Jiang, S., et al. Re-evaluation of the Carcinogenic Significance of Hepatitis B Virus Integration in Hepatocarcinogenesis. Proceedings of the National Academy of Sciences of the United States of America. 7 (9), e40363 (2012).
- Jiang, Z., et al. The effects of hepatitis B virus integration into the genomes of hepatocellular carcinoma patients. Genome Research. 22 (4), 593-601 (2012).
- Kan, Z., et al. Whole-genome sequencing identifies recurrent mutations in hepatocellular carcinoma. Genome Research. 23 (9), 1422-1433 (2013).
- Summers, J., et al. Hepatocyte turnover during resolution of a transient hepadnaviral infection. Proceedings of the National Academy of Sciences of the United States of America. 100 (20), 11652-11659 (2003).
- Mason, W. S., Jilbert, A. R., Summers, J. Clonal expansion of hepatocytes during chronic woodchuck hepatitis virus infection. Proceedings of the National Academy of Sciences of the United States of America. 102 (4), 1139-1144 (2005).
- Mason, W. S., et al. Detection of clonally expanded hepatocytes in chimpanzees with chronic hepatitis B virus infection. Journal of Virology. 83 (17), 8396-8408 (2009).
- Chauhan, R., Churchill, N. D., Mulrooney-Cousins, P. M., Michalak, T. I. Initial sites of hepadnavirus integration into host genome in human hepatocytes and in the woodchuck model of hepatitis B-associated hepatocellular carcinoma. Oncogenesis. 6 (4), e317 (2017).
- Tu, T., Jilbert, A. R. Detection of Hepatocyte Clones Containing Integrated Hepatitis B Virus DNA Using Inverse Nested PCR. Methods in Molecular Biology. 1540, 97-118 (2017).
- Ni, Y., Urban, S. Hepatitis B Virus Infection of HepaRG Cells, HepaRG-hNTCP Cells, and Primary Human Hepatocytes. Methods in Molecular Biology. 1540, 15-25 (2017).
- Schulze, A., Schieck, A., Ni, Y., Mier, W., Urban, S. Fine mapping of pre-S sequence requirements for hepatitis B virus large envelope protein-mediated receptor interaction. Journal of Virology. 84 (4), 1989-2000 (2010).
- Lempp, F. A., et al. Evidence that hepatitis B virus replication in mouse cells is limited by the lack of a host cell dependency factor. Journal of Hepatology. 64 (3), 556-564 (2016).
- Ladner, S. K., et al. Inducible expression of human hepatitis B virus (HBV) in stably transfected hepatoblastoma cells: a novel system for screening potential inhibitors of HBV replication. Antimicrobial Agents and Chemotherapy. 41 (8), 1715-1720 (1997).
- Allweiss, L., et al. Proliferation of primary human hepatocytes and prevention of hepatitis B virus reinfection efficiently deplete nuclear cccDNA in vivo. Gut. , (2017).
- Yang, Z. T., et al. Characterization of Full-Length Genomes of Hepatitis B Virus Quasispecies in Sera of Patients at Different Phases of Infection. Journal of Clinical Microbiology. 53 (7), 2203-2214 (2015).
- Chook, J. B., et al. Universal Primers for Detection and Sequencing of Hepatitis B Virus Genomes across Genotypes A to G. Journal of Clinical Microbiology. 53 (6), 1831-1835 (2015).
- Li, F., et al. Whole genome characterization of hepatitis B virus quasispecies with massively parallel pyrosequencing. Clinical Microbiology and Infection. 21 (3), 280-287 (2015).
- Zhou, T. C., et al. Evolution of full-length genomes of HBV quasispecies in sera of patients with a coexistence of HBsAg and anti-HBs antibodies. Scientific Reports. 7 (1), 661 (2017).
- Long, Q. X., Hu, J. L., Huang, A. L. Deep Sequencing of the Hepatitis B Virus Genome: Analysis of Multiple Samples by Implementation of the Illumina Platform. Methods in Molecular Biology. 1540, 211-218 (2017).
- Li, X., et al. The function of targeted host genes determines the oncogenicity of HBV integration in hepatocellular carcinoma. Journal of Hepatology. 60 (5), 975-984 (2014).
- Wooddell, C. I., et al. RNAi-based treatment of chronically infected patients and chimpanzees reveals that integrated hepatitis B virus DNA is a source of HBsAg. Science Translational Medicine. 9 (409), (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados