Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Confección de ensayos de citotoxicidad in vivo para estudiar inmunodominancia en respuestas de células T CD8+ específicas de tumores
En este artículo
Resumen
Describimos aquí un ensayo de matanza in vivo basado en citometría de flujo que permite el examen de inmunodominancia en las respuestas de linfocitos T citotóxicos (CTL) a un antígeno tumoral modelo. Proporcionamos ejemplos de cómo este ensayo elegante se puede emplear para estudios mecaniísticos y para pruebas de eficacia de fármacos.
Resumen
Los ensayos de citotoxicidad con base en el éster de carboxyfluorescein succinimidil (CFSE) permiten la cuantificación sensible y precisa de las respuestas de linfocitos T de CD8+ citolíticos (CTL) obtenidas contra péptidos derivados de tumores y patógenos. Ofrecen varias ventajas sobre los ensayos de matanza tradicionales. En primer lugar, permiten el seguimiento de la citotoxicidad por CTL mediada dentro de los órganos linfoides secundarios arquitectónicamente intactos, típicamente en el bazo. En segundo lugar, permiten estudios mecaniísticos durante las fases de cebado, efector y recuperación de las respuestas CTL. En tercer lugar, proporcionan plataformas útiles para las pruebas de eficacia de vacunas/fármacos en un entorno verdaderamente in vivo. Aquí, proporcionamos un protocolo optimizado para el examen de las respuestas CTL concomitantes contra más de un epitopo péptido de un antígeno tumoral modelo (AG), a saber, el virus Simia 40 (SV40)-codificado grande t AG (t AG). Al igual que la mayoría de las otras proteínas del tumor clínicamente relevantes, T AG alberga muchos péptidos potencialmente inmunogénicos. Sin embargo, sólo cuatro de estos péptidos inducen respuestas CTL detectables en ratones C57BL/6. Estas respuestas se organizan sistemáticamente en un orden jerárquico basado en su magnitud, que forma la base para TCD8 "inmunodominancia" en este potente sistema. En consecuencia, la mayor parte de la respuesta TCD8 específica de t AG se centra en un único epítopo inmunodominante, mientras que los otros tres epítopos se reconocen y responden sólo débilmente. La inmunodominación compromete la amplitud de las respuestas antitumorales TCD8 y es, como tal, considerado por muchos como un impedimento para la vacunación exitosa contra el cáncer. Por lo tanto, es importante entender los factores celulares y moleculares y los mecanismos que dictan o dan forma a la inmunodominación TCD8 . El protocolo que Describimos aquí está adaptado a la investigación de este fenómeno en el modelo de inmunización de T AG, pero se puede modificar y extender fácilmente a estudios similares en otros modelos de tumores. Proporcionamos ejemplos de cómo el impacto de las intervenciones inmunoterapéuticas experimentales se puede medir utilizando ensayos de citotoxicidad in vivo.
Introducción
Las células t CD8+ convencionales (tCD8) juegan partes importantes en la vigilancia inmune contra el cáncer. Funcionan principalmente en la capacidad de los linfocitos T citolíticos (CTLs) que reconocen antígenos péptidos específicos o asociados al tumor (AGS) que se muestran dentro de la hendidura cerrada de las moléculas de clase I del complejo de histocompatibilidad principal (MHC). Los CTLs completamente armados utilizan su arsenal citotóxico para destruir las células malignas. El anticancerígeno TCD8 puede detectarse en la circulación o incluso en las masas primarias y metastásicas de muchos pacientes con cáncer y animales portadores de tumores. Sin embargo, a menudo son anérgicas o exhaustas y no logran erradicar el cáncer. Por lo tanto, muchas modalidades de inmunoterapia están diseñadas para aumentar las frecuencias TCD8 anticancerígenas y para restaurar y potenciar sus funciones.
Las proteínas tumorales albergan muchos péptidos, algunos de los cuales pueden ser inmunogénicos y potencialmente inmunoprotectores. Sin embargo, las respuestas TCD8 cuantificables se provocan con magnitudes variables contra pocos péptidos solamente. Esto crea una "jerarquía de inmunodominancia" entre TCD8 clones1. En consecuencia, inmunodominante (ID) TCD8 ocupan rangos jerárquicos prominentes, que es comúnmente juzgado por su abundancia. Por el contrario, las células TCD8 cuyo receptor de linfocitos t (TCR) es específico para epitopos SUBDOMINANTES (SD) ocurren en frecuencias más bajas. Nosotros y otros hemos identificado algunos de los factores que dictan o dan forma a la inmunodominación en las respuestas TCD8 . Estos incluyen, entre otros, el modo de presentación de AG a TCD8 ingenuos (es decir, presentación directa, presentación cruzada, travestismo)2,3,4, el tipo de células presentadoras de AG (APCs) participandoen la activación5TCD8 , la abundancia y estabilidad de la proteína AGS6,7 y la eficiencia y cinética de su degradación por proteasomas7,8, el selectividad relativa del transportador asociado con el procesamiento de AG (TAP) para los péptidos9, la afinidad de los péptidos liberados para las moléculas de MHC I9,10, la presencia, las frecuencias precursoras y la diversidad de TCR de cognar tCD8 en las agrupaciones de células t11,12,13, competencia cruzada entre las células t para el acceso a los APCs14,15, y la capacidad fratricida de tCD8 clones16. Además, la inmunodominación TCD8 está sometida a mecanismos inmunoreguladores mediadas por varios tipos de células supresoras, como las células t (nTreg) reguladorasque ocurren de forma natural, la molécula Co-inhibitoria de la superficie celular programada muerte-1 (PD-1)16, y ciertas enzimas intracelulares como indoleamina 2, 3-dioxigenasa (ido)18 y el objetivo de mamífero de rapamicina (mTOR)19. Es importante tener en cuenta, sin embargo, que los factores anteriores no siempre tienen en cuenta completamente la inmunodominación.
Aparte de la biología básica de la inmunodominación TCD8 , el examen de este fenómeno intrigante tiene importantes implicaciones en la inmunología del cáncer y la inmunoterapia. En primer lugar, un estado de identificación no confiere necesariamente a un clon dado de TCD8 la capacidad de prevenir el inicio del tumor o la progresión20. Si y cómo los ID y SD TCD8 contribuyen a la inmunidad antitumoral pueden depender del tipo y el grado de malignidad y el sistema experimental empleado. En segundo lugar, se cree que ID TCD8 clones pueden ser "demasiado visibles" para el sistema inmunológico y por lo tanto más propensos a los mecanismos de tolerancia central y/o periférica16,21. En tercer lugar, los tumores heterogénicos pueden contener células neoplásicas que evitan la detección por muchos, si no la mayoría, CTLs mostrando sólo un espectro estrecho de péptidos: MHC complejos. Bajo estas circunstancias, TCD8 respuestas de amplitud insuficiente son propensos a permitirse tales células tumorales una ventaja de supervivencia, potenciando así su crecimiento de la salida22. Es por las razones anteriores que muchos ver inmunodominación como un obstáculo a la vacunación exitosa a base de TCD8y terapias contra el cáncer.
La inoculación de ratones C57BL/6 con virus simio 40 (SV40)-células transformadas que expresan gran tumor AG (T AG) proporciona un potente sistema preclínico para estudiar el inmunopredominio de TCD8 . Este modelo ofrece varios beneficios. En primer lugar, los epítopos peptídicos de esta oncoproteína clínicamente relevante están bien caracterizados en esta cepa de ratón23 (tabla 1). En segundo lugar, los epítopos de T AG, que se denominan sitios I, II/III, IV y V, desencadenan respuestas TCD8 que se organizan sistemáticamente en el siguiente orden jerárquico: sitio iv > > sitio I ≥ sitio II/III > ≫ sitio V. en consecuencia, el sitio IV-específico TCD8 Monte la respuesta más robusta a T AG. Por el contrario, los sitios I y II/III son subdominantes, y los TCD8 específicos del sitio V son menos abundantes y por lo general sólo son detectables en ausencia de capacidad de respuesta a otros epítopos23,24. En tercer lugar, la línea celular del tumor T AG+ utilizada en el protocolo aquí descrito, es decir, C57SV células de fibrosarcoma, y las utilizadas en nuestras investigaciones anteriores16,17,18,19 ,25,26, se transforman con fragmentos subgenómicos SV4025. Por lo tanto, son incapaces de ensamblar y liberar SV40 viriones que potencialmente podrían infectar APCs del host. Además, las células C57SV carecen de moléculas coestimuladores clásicas como CD80 (B7-1), CD86 (B7-2) y ligando del CD137 (4-1BBL)16. Los atributos anteriores hacen que estas líneas sean ideales para el examen de la activación in vivo de TCD8 mediante cebado cruzado. La cebado cruzado es una vía importante para inducir respuestas TCD8 , especialmente aquellas lanzadas contra células tumorales de origen no hematopoyético que fallan directamente en las células T ingenuas25.
Las frecuencias y/o funciones antitumorales TCD8 pueden controlarse mediante la tinción de MHC I tetrámero, la tinción intracelular para citoquinas efectoras (p. ej., el interferón [IFN]-γ) o las moléculas líticas (p. ej., perforación), los ensayos de inmunosolla ligada a enzimas (ELISPOT) y ex ensayos de citotoxicidad vivo. Desde su creación en los 199027,28, carboxyfluorescein succinimidil Ester (CFSE)-basado en ensayos de matanza in vivo han permitido la evaluación de las respuestas citotóxicas mediadas por antiviral CTLs29,30 , 31, antitumorales CTLs16,32, natural killer (NK) células33, glicolipid reactivo natural invariante T (iNKT) células34, y pre existente y de Novo específico del donante aloanticuerpos26. Por lo tanto, sus aplicaciones pueden ser de interés para un amplio número de lectores, incluyendo pero no limitado a los investigadores que trabajan en las áreas de Inmunología tumoral e inmunoterapia, inmunidad antipatógeno, y el diseño de la vacuna preventiva y terapéutica.
Para evaluar la citotoxicidad mediada por células en escenarios típicos, dos poblaciones de splenocitos ingenuos que exhiben un AG irrelevante o un AG (s) cognado están etiquetados con dos dosis diferentes de CFSE, mezcladas en igual número e inyectadas en ingenua (control) o asesino ratones que albergan células. A continuación, la citometría de flujo examina la presencia/ausencia de cada población objetivo.
Hemos optimizado y empleado ensayos de matanza in vivo en nuestros estudios sobre inmunodominancia en las respuestas antiviral y antitumoral T CD812,16,17. Aquí proporcionamos un protocolo detallado para la evaluación simultánea de las respuestas de ID y SD TCD8 a los epítopos de t AG, que pueden adoptarse fácilmente para investigaciones similares en otros sistemas experimentales. También proporcionamos resultados representativos que demuestran que el agotamiento de células nTreg y el bloqueo PD-1 pueden mejorar selectivamente la citotoxicidad inducida por ID TCD8-y SD tCD8, respectivamente. Al final, discutiremos múltiples ventajas de los ensayos de matanza in vivo, así como algunas de sus limitaciones inherentes.
Protocolo
Los experimentos descritos aquí siguen protocolos de uso de animales aprobados por entidades institucionales y se adhieren a las directrices nacionales establecidas.
1. inoculación de ratones C57BL/6 con T AG-expresando células tumorales
- Cultivar la línea celular de fibrosarcoma SV40 transformada C57SV (o una línea celular de T AG+ adherente similar) en el medio de águila modificada de Dulbecco (DMEM) con 4,5 g/l D-glucosa y l-glutamina (1x) y complementado con piruvato sódico de 1 mm y 10% inactivados por calor suero bovino fetal (FBS) en matraces tratados con cultivo de tejidos a 37 ° c en atmósfera humidificada que contengan 10% de CO2.
- Una vez que las células se vuelven completamente confluentes o ligeramente sobreconfluidas, retire suavemente y deseche el medio y enjuague la monocapa con suero estéril amortiguado por fosfato (PBS) precalentado.
Nota: la expresión T AG máxima se logra cuando las células T AG+ alcanzan el 100% de confluencia. - Dentro de un gabinete biológico de seguridad, añadir tripsin-EDTA pre-calentado (0,25%) para cubrir la monocapa a temperatura ambiente hasta que las células se desalojan en parches. Toque los lados del matraz (s) de cultivo varias veces para liberar las células adherentes restantes.
Nota: si es necesario y para agilizar el proceso de tripsinización, transfiera el (los) matraz (s) a una incubadora de 37 ° c. Las células desalojadas adoptarán rápidamente una forma redondeada bajo un microscopio de luz. Este paso debe durar aproximadamente 5 minutos. - Añadir 5 mL de grumos medio y disociado DMEM para preparar una suspensión de una sola célula pipeteando el contenido de cada matraz hacia arriba y hacia abajo.
- Transfiera la suspensión de la célula a través de un colador de células con poros de 70 μm en un tubo.
- Gire hacia abajo el tubo a 400 x g durante 5 min a 4 ° c.
- Deseche el sobrenadante. Resuspend células granuladas en 10 mL de PBS estéril frío.
- Repita los pasos 1,6 y 1,7 dos veces.
- Cuente las células con un hemociómetro. Prepare una suspensión uniforme que contenga 4 x 107 células/ml PBS estéril.
- Inyectar 500 μL de la suspensión anterior intraperitonealmente (i.p.) en cada adulto (6-12-semana-edad) macho o hembra C57BL/6 ratón.
2. regímenes de tratamiento
-
Régimen de tratamiento para examinar la contribución de las células nTreg a TCD8 inmunodominancia
- Cuatro días antes de la preparación in vivo de ratones C57BL/6 con células C57SV (paso 1,10), inyectar cada animal una vez i.p. con 0,5 mg de una baja endotoxina, anticuerpo monoclonal anti-CD25 sin azida (mAb) (Clone PC-61.5.3), que agota las células nTreg, o con una rata IgG1 isotipo control (p. ej., clonar KLH/G1-2-2, clonar HRPN o clonar TNP6A7).
-
Régimen de tratamiento para probar la significancia in vivo de PD-1-PD-L1 (2) interacciones en el modelado de inmunodominancia TCD8
Nota: la participación de PD-1 por PD-L1 a menudo, pero no siempre, medita la co-inhibición y/o agotamiento de TCD8específico de AG. Por lo tanto, el tratamiento con anti-PD-1 se puede realizar en paralelo con la administración de mAbs anti-PD-L1 y anti-PD-L2 para revelar la interacción intercelular exacta involucrada en un fenómeno biológico.
3. preparación de los Splenocitos objetivo
- Eutanasia ratones ingenuos C57BL/6 de sexo igualado (6-12 semanas de edad) que servirán como donantes de esplocito por dislocación cervical.
- Coloque cada ratón con el abdomen hacia arriba dentro de un gabinete biológico de seguridad. Pulverizar la piel con un 70% (v/v) EtOH. Usando fórceps y tijeras estériles, levante la piel y haga una pequeña incisión ventral en la línea media. Luego, corta la piel de forma cruzada para exponer el peritoneo.
- Usando fórceps, levanta el peritoneo de forma similar a una carpa sin arrebatar ninguno de los órganos internos. Corta el peritoneo abierto para exponer la cavidad peritoneal y retira suavemente el bazo.
- Colocar el bazo (s) dentro de una amoladora de tejido Dounce de 15 mL que contiene 5 mL de PBS estéril. Aplicar presión manual usando el émbolo de vidrio de la amoladora hasta que el tejido esplémico se disipa en una suspensión de células rojas homogéneas.
Nota: dependiendo del número de animales receptores por grupo experimental, pueden necesitarse varios bazos de ratón de donante para la preparación de células diana. Hasta 3 bazos se pueden homogeneizar juntos dentro de una amoladora de 15 ml. - Transfiera el homogeneiado a un tubo de 15 mL. Gire hacia abajo el tubo a 400 x g durante 5 min a 4 ° c.
- Deseche el sobrenadante. Resuspend células granuladas en 4 mL de tampón de lising de cloruro de amonio-potasio (ACK) durante 4 min para eliminar los eritrocitos.
Nota: este es un paso sensible al tiempo. La sobreexposición de los esplocitos al tampón de lising ACK aumentará su fragilidad y los hará susceptibles a la muerte celular no específica. - Para cada tubo, añadir 8 mL de Medio RPMI 1640 conteniendo 10% de FBS inactivados por calor, L-alanyl-L-glutamina, 0,1 mM medios esenciales mínimos (MEM) aminoácidos no esenciales, 1 mM piruvato sódico, 10 mM HEPES, y 1x penicilina/estreptomicina, que en adelante será conoce como medio RPMI completo (tabla de materiales).
- Transfiera el contenido a través de poros de 70 μm de un colador celular a un nuevo tubo de 15 mL.
- Gire hacia abajo el tubo a 400 x g durante 5 min a 4 ° c.
- Deseche el sobrenadante. Resuspend células granuladas en 12 mL de RPMI completo.
- Divida la suspensión de esplococitos en 3 porciones iguales (4 mL cada una) en 3 tubos separados.
4. recubrimiento de los Splenocitos objetivo con péptidos irrelevantes y cognados
- Etiquetar los tubos de acuerdo con los péptidos que se utilizarán para el pulso de los esplocitos Diana. Los esplocitos de control se pulsarán con un péptido irrelevante, y cada población de esplocitos objetivo cognados se pulsará con un péptido sintético correspondiente al epítopo inmunodominante derivado de T AG (sitio IV) o un epítopo Subdominante de T AG (sitio I o sitio II/III) (cuadro 1).
Nota: la elección de péptidos irrelevantes depende de la conexión experimental y de la cepa del ratón utilizada en cada investigación. Los autores a menudo utilizan gB498-505 (un epitopo de péptido inmunodominante de h-2Kb-restringido de virus del herpes simple [VHS]-1) y/o GP33-41 (un epitopo de péptido inmunodominante de h-2Dbrestringido de linfocíticos virus de la coriomeningitis ([LCMV]) en ratones C57BL/6 (tabla 1). Estos péptidos son opciones óptimas porque: (i) se derivan de patógenos no encontrados anteriormente en el modelo de ratón descrito aquí; (II) similares a los péptidos derivados de T AG, gB498-505 y GP33-41 están restringidos y se unen a las moléculas H-2b . En los ensayos de muerte in vivo de tres picos, cada uno de los dos picos que corresponden a las células diana pueden representar esplocitos pulsados con un péptido inmunodominante o Subdominante. La elección de cada conjunto de péptidos varía de acuerdo con los objetivos de cada experimento. Vea la figura 1 y la figura 2 como ejemplos de dicha variación. Para el resto de este protocolo, los sitios I y IV derivados de T AG representarán péptidos subdominantes e inmunodominantes, respectivamente. - Pulse el contenido de cada tubo etiquetado con 1 μM del péptido respectivo durante 1 h a 37 ° c y 5% CO2.
- Utilice un colador de celda separado (con poros de 70 μm) para cada tubo para eliminar grumos y desechos Si es necesario.
- Gire hacia abajo el tubo a 400 x g durante 5 min a 4 ° c. Deseche el sobrenadante.
- Resuspend las células granuladas en 12 mL de PBS frío estéril y repita el paso 4,4 una vez más.
Nota: es importante eliminar tanto FBS como sea posible porque FBS puede enlazar CFSE en el siguiente paso.
5. etiquetado de los esplocitos objetivo con CFSE
- Resuspendio los esplocitos pulsados con péptidos en 4 mL de PBS estéril.
- Añadir CFSE a 0,025 μM, 0,25 μM y 2 μM en los tubos que contengan esplocitos de péptidos, sitios I y del sitio IV, respectivamente.
Nota: para lograr un etiquetado CFSE uniforme, sostenga cada tubo en un ángulo de 45 ° antes de añadir el CFSE al lado ligeramente por encima de la suspensión de la célula seguida inmediatamente por un suave vórtex. Esto asegurará la aparición de histogramas lisos al final. Las variaciones de las intensidades de CFSE no son infrecuentes en lotes y dependientes de la edad. Por lo tanto, uno puede necesitar experimentar con dosis diferenciales de CFSE antes de decidir sobre las concentraciones óptimas que se utilizarán.
PRECAUCIÓN: el CFSE es tóxico a concentraciones superiores a 5 μM. - Colocar los tubos dentro de una incubadora de 37 ° c durante 15 min e invertirlos una vez cada 5 min.
- Agregue 3 mL de FBS inactivados por calor a cada tubo para detener la reacción de CFSE. Recarga el contenido con PBS estéril.
- Gire hacia abajo el tubo a 400 x g durante 5 min a 4 ° c. Deseche el sobrenadante.
- Resuspend células granuladas en 12 mL de PBS estéril y repita el paso 5,5.
6. examen del etiquetado CFSE adecuado/igual de las poblaciones de Esesplocitos objetivo
- Resuspend células granuladas en 3 mL de PBS.
- Vórtice los tubos suavemente. Transferir 10 μL, cada uno, debajaCFSE, CFSEintermedio (int), y CFSE suspensiones de célulasaltas en un 5 ml de fondo redondo de poliestireno activado por fluorescencia (facs) tubo que contiene 200 μL de PBS.
- Interrogue las células utilizando un CITOMETRO de flujo equipado con un láser de 488 nm. Dibuje una compuerta de linfocitos basada en las propiedades de dispersión hacia adelante (FSC) y dispersión lateral (SSC) de las células antes de adquirir 5000 eventos que caen dentro de la puerta de linfocitos en el canal FL-1.
- Dentro de la población "padre" CFSE+ , dibuje puertas de histograma adicionales para identificar las subpoblaciones CFSELow, CFSEinty CFSEHigh .
- Confirme los números de evento iguales o casi iguales dentro de las tres puertas. Si es necesario, ajuste los números de celda en los tubos de "fuente" (paso 6,1) antes de mezclar e inyectar los esplocitos objetivo en ratones ingenuos y imprimados en la sección 7.
7. inyección de células diana etiquetadas por CFSE en los destinatarios ingenuos y con cebado de T-AG
- Vórtice suavemente los tubos de origen. Transfiera las tres suspensiones de células con etiqueta CFSE en proporciones iguales en un tubo nuevo.
- Recarga el contenido con PBS estéril.
- Gire hacia abajo el tubo a 400 x g durante 5 min a 4 ° c. Resuspend las células granuladas con PBS estéril.
- Cuente las células en azul tripan por un hemociómetro para asegurar la viabilidad celular de al menos el 95%.
- Ajustar el volumen para inyectar 1 x 107 células de objetivo mixto/200 ΜL de PBS por vía intravenosa (i.v.), a través de la vena de cola, en cada ratón C57BL/6 receptor.
Nota: Almacene las células en hielo entre las inyecciones. Mezcle suavemente las células diana antes de cada inyección. Anote el tiempo exacto de inyección para cada ratón, que determinará cuándo el animal tendrá que ser eutanzado. Es importante mantener la duración de la citotoxicidad in vivo consistente entre todos los animales en el mismo experimento.
8. adquisición de datos
- Dos o cuatro horas después de la inyección de células diana etiquetadas con CFSE, eutanasia a los ratones receptores por dislocación cervical.
Nota: la duración de la citotoxicidad in vivo puede variar dependiendo del sistema experimental empleado, la inmunogenicidad de los Ags objetivo, la abundancia anticipada de TCD8 específico del antígeno peptídico en el bazo, y la robustez de su función lítica entre otros factores. - Retire y procese cada bazo por separado como en los pasos 3.2 − 3.9.
- Desechar el sobrenadante y resuspender las células peladas en 3 mL de PBS.
Nota: tenga cuidado adicional para manejar el tejido esplémico y preparaciones celulares a 4 ° c o en hielo antes de análisis citofluométricos. Esto es para prevenir la continuación de la citotoxicidad ex vivo. - Transfiera aproximadamente 1 x 107 células de cada bazo procesado a un tubo FACS limpio.
- Interrogue las células inmediatamente utilizando un CITOMETRO de flujo equipado con un láser de 488 nm. Dibuje una puerta de linfocito basada en las propiedades FSC y SSC de las células.
- Identifique los esplocitos del receptor CFSE y las celdas de destino transferidas. Dibuje puertas adicionales que acomode distintas poblaciones de células diana de CFSELow, CFSEinty CFSE.
- Adquiera un total de 2000 eventosbajos CFSE en el canal FL-1.
9. Análisis de datos
- Calcule la lisis específica de cada población de células objetivo cognado utilizando la siguiente fórmula:
% Citotoxicidad específica =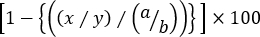
donde x = CFSEint/ número de evento alto en t AG-el ratón Primed, y = CFSE número de eventobajo en t AG-el ratón Primed, a = CFSEint/ número de evento alto en el ratón ingenuo, y b = CFSEbajo el evento número en el ratón ingenuo.
Nota: en los ensayos de citotoxicidad de tres picos en los que se evalúa la lisis específica de más de una población objetivo cognada, no es apropiado utilizar frecuencias celulares Diana. Esto es simplemente porque la frecuencia de una población de células diana cognada está influenciada no sólo por el porcentaje de los controles irrelevantes, sino también por el de los otros esplocitos objetivo cognado. Por lo tanto, los números de evento dentro de cada puerta deben usarse en la fórmula anterior para calcular con precisión la lisis de cada población de células diana cognada (ya sea CFSEint o CFSEHigh cells) contra controles debajo CFSE.
Resultados
El objetivo del experimento cuyos resultados se muestran en la figura 1 fue determinar si la presencia y las funciones de las células nTreg forman o alteran la jerarquía de inmunodominancia de tCD8específico de t AG. C57BL/6 ratones fueron inyectados i.p. con PBS o con 0,5 mg de un anti-CD25 mAb (Clone PC-61.5.3 [PC61]) cuatro días antes de que recibieran 2 x 107 C57SV células tumorales i.p. En experimentos separados, se usó un control de isotipo IgG1 de rata en l...
Discusión
Los ensayos de citotoxicidad in vivo basados en CFSE ofrecen varias ventajas sobre los ensayos de matanza tradicionales, como el cromo radioactivo (51CR) y los ensayos de liberación colorimétrico de lactato deshidrogenasa (LDH). Primero, permiten el monitoreo de la función CTL dentro de un órgano linfoide secundario arquitectónicamente intacto.
En segundo lugar, el asesinato específico de células Diana en ensayos de citotoxicidad in vivo refleja el número absoluto de TC...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por los institutos canadienses de investigación sanitaria (CIHR), las subvenciones MOP-130465 y PJT-156295 a SMMH. JC está parcialmente respaldada por una beca de posgrado de la Reina Isabel II en ciencia y tecnología del Ministerio de formación, colegios y universidades de Ontario. CEM fue un beneficiario de una beca de posgrado de Alexander Graham Bell Canada (doctorado) de Ciencias naturales y el Consejo de investigación de ingeniería de Canadá (NSERC).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA (1X) | Thermo Fisher Scientific | 25200-056 | |
| ACK Lysing Buffer | Thermo Fisher Scientific | A1049201 | |
| Anti-mouse CD25 (clone PC-61.5.3) | Bio X Cell | BE0012 | |
| Anti-mouse PD-1 (clone RMP1-14) | Bio X Cell | BE0146 | |
| CFSE | Thermo Fisher Scientific | C34554 | |
| DMEM (1X) | Thermo Fisher Scientific | 11965-092 | |
| Fetal bovine serum (FBS) | Wisent Bioproducts | 080-150 | Heat-inactivate prior to use |
| GlutaMAX (100X) | Thermo Fisher Scientific | 35050-061 | |
| HEPES (1M) | Thermo Fisher Scientific | 15630080 | 10 mM final concentration |
| MEM Non-Essential Amino Acids Solution (100X) | Thermo Fisher Scientific | 11140-050 | |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781 | Stock is 100X |
| Rat IgG1 (clone KLH/G1-2-2) | SouthernBiotech | 0116-01 | Isotype control |
| Rat IgG1 (clone HRPN) | Bio X Cell | BE0088 | Isotype control |
| Rat IgG1 (clone TNP6A7) | Bio X Cell | BP0290 | Isotype control |
| Rat IgG2a (clone 2A3) | Bio X Cell | BP0089 | Isotype control |
| RPMI 1640 (1X) | Thermo Fisher Scientific | 11875-093 | |
| Sodium Pyruvate (100 mM) | Thermo Fisher Scientific | 11360-070 | 1 mM final concentration |
Referencias
- Yewdell, J. W., Bennink, J. R. Immunodominance in major histocompatibility complex class I-restricted T lymphocyte responses. Annual Review of Immunology. 17, 51-88 (1999).
- Chen, W., et al. Reversal in the immunodominance hierarchy in secondary CD8+ T cell responses to influenza A virus: roles for cross-presentation and lysis-independent immunodomination. The Journal of Immunology. 173 (8), 5021-5027 (2004).
- Otahal, P., et al. Inefficient cross-presentation limits the CD8+ T cell response to a subdominant tumor antigen epitope. The Journal of Immunology. 175 (2), 700-712 (2005).
- Lauron, E. J., et al. Cross-priming induces immunodomination in the presence of viral MHC class I inhibition. PLoS Pathogens. 14 (2), e1006883 (2018).
- Crowe, S. R., et al. Differential antigen presentation regulates the changing patterns of CD8+ T cell immunodominance in primary and secondary influenza virus infections. The Journal of Experimental Medicine. 198 (3), 399-410 (2003).
- Probst, H. C., et al. Immunodominance of an antiviral cytotoxic T cell response is shaped by the kinetics of viral protein expression. The Journal of Immunology. 171 (10), 5415-5422 (2003).
- Gileadi, U., et al. Generation of an immunodominant CTL epitope is affected by proteasome subunit composition and stability of the antigenic protein. The Journal of Immunology. 163 (11), 6045-6052 (1999).
- Zanker, D., Waithman, J., Yewdell, J. W., Chen, W. Mixed proteasomes function to increase viral peptide diversity and broaden antiviral CD8+ T cell responses. The Journal of Immunology. 191 (1), 52-59 (2013).
- Deng, Y., Yewdell, J. W., Eisenlohr, L. C., Bennink, J. R. MHC affinity, peptide liberation, T cell repertoire, and immunodominance all contribute to the paucity of MHC class I-restricted peptides recognized by antiviral CTL. The Journal of Immunology. 158 (4), 1507-1515 (1997).
- Chen, W., Khilko, S., Fecondo, J., Margulies, D. H., McCluskey, J. Determinant selection of major histocompatibility complex class I-restricted antigenic peptides is explained by class I-peptide affinity and is strongly influenced by nondominant anchor residues. The Journal of Experimental Medicine. 180 (4), 1471-1483 (1994).
- Kotturi, M. F., et al. Naive precursor frequencies and MHC binding rather than the degree of epitope diversity shape CD8+ T cell immunodominance. The Journal of Immunology. 181 (3), 2124-2133 (2008).
- Haeryfar, S. M., et al. Terminal deoxynucleotidyl transferase establishes and broadens antiviral CD8+ T cell immunodominance hierarchies. The Journal of Immunology. 181 (1), 649-659 (2008).
- Leon-Ponte, M., Kasprzyski, T., Mannik, L. A., Haeryfar, S. M. Altered immunodominance hierarchies of influenza A virus-specific H-2(b)-restricted CD8+ T cells in the absence of terminal deoxynucleotidyl transferase. Immunological Investigations. 37 (7), 714-725 (2008).
- Kedl, R. M., et al. T cells compete for access to antigen-bearing antigen-presenting cells. The Journal of Experimental Medicine. 192 (8), 1105-1113 (2000).
- Kastenmuller, W., et al. Cross-competition of CD8+ T cells shapes the immunodominance hierarchy during boost vaccination. The Journal of Experimental Medicine. 204 (9), 2187-2198 (2007).
- Memarnejadian, A., et al. PD-1 Blockade Promotes Epitope Spreading in Anticancer CD8(+) T Cell Responses by Preventing Fratricidal Death of Subdominant Clones To Relieve Immunodomination. The Journal of Immunology. 199 (9), 3348-3359 (2017).
- Haeryfar, S. M., DiPaolo, R. J., Tscharke, D. C., Bennink, J. R., Yewdell, J. W. Regulatory T cells suppress CD8+ T cell responses induced by direct priming and cross-priming and moderate immunodominance disparities. The Journal of Immunology. 174 (6), 3344-3351 (2005).
- Rytelewski, M., et al. Suppression of immunodominant antitumor and antiviral CD8+ T cell responses by indoleamine 2,3-dioxygenase. PLoS One. 9 (2), e90439 (2014).
- Maleki Vareki, S., et al. Differential regulation of simultaneous antitumor and alloreactive CD8(+) T-cell responses in the same host by rapamycin. American Journal of Transplantation. 12 (1), 233-239 (2012).
- Irvine, K., Bennink, J. Factors influencing immunodominance hierarchies in TCD8+ -mediated antiviral responses. Expert Review of Clinical Immunology. 2 (1), 135-147 (2006).
- Grossmann, M. E., Davila, T., Celis, T. Avoiding tolerance against prostatic antigens with subdominant peptide epitopes. Journal of Immunotherapy. 24 (3), 237-241 (2001).
- Schreiber, H., Wu, T. H., Nachman, J., Kast, W. M. Immunodominance and tumor escape. Seminars in Cancer Biology. 12 (1), 25-31 (2002).
- Mylin, L. M., et al. Quantitation of CD8(+) T-lymphocyte responses to multiple epitopes from simian virus 40 (SV40) large T antigen in C57BL/6 mice immunized with SV40, SV40 T-antigen-transformed cells, or vaccinia virus recombinants expressing full-length T antigen or epitope minigenes. Journal of Virology. 74 (15), 6922-6934 (2000).
- Fu, T. M., et al. An endoplasmic reticulum-targeting signal sequence enhances the immunogenicity of an immunorecessive simian virus 40 large T antigen cytotoxic T-lymphocyte epitope. Journal of Virology. 72 (2), 1469-1481 (1998).
- Chen, W., et al. Cross-priming of CD8+ T cells by viral and tumor antigens is a robust phenomenon. European Journal of Immunology. 34 (1), 194-199 (2004).
- Memarnejadian, A., Meilleur, C. E., Mazzuca, D. M., Welch, I. D., Haeryfar, S. M. Quantification of Alloantibody-Mediated Cytotoxicity In Vivo. Transplantation. 100 (5), 1041-1051 (2016).
- Aichele, P., et al. Peptide antigen treatment of naive and virus-immune mice: antigen-specific tolerance versus immunopathology. Immunity. 6 (5), 519-529 (1997).
- Oehen, S., Brduscha-Riem, K. Differentiation of naive CTL to effector and memory CTL: correlation of effector function with phenotype and cell division. The Journal of Immunology. 161 (10), 5338-5346 (1998).
- Coles, R. M., Mueller, S. N., Heath, W. R., Carbone, F. R., Brooks, A. G. Progression of armed CTL from draining lymph node to spleen shortly after localized infection with herpes simplex virus 1. The Journal of Immunology. 168 (2), 834-838 (2002).
- Barber, D. L., Wherry, E. J., Ahmed, R. Cutting edge: rapid in vivo killing by memory CD8 T cells. The Journal of Immunology. 171 (1), 27-31 (2003).
- Meilleur, C. E., et al. Bacterial superantigens expand and activate, rather than delete or incapacitate, preexisting antigen-specific memory CD8+ T cells. The Journal of Infectious Diseases. , (2018).
- Goldszmid, R. S., et al. Dendritic cells charged with apoptotic tumor cells induce long-lived protective CD4+ and CD8+ T cell immunity against B16 melanoma. The Journal of Immunology. 171 (11), 5940-5947 (2003).
- Oberg, L., et al. Loss or mismatch of MHC class I is sufficient to trigger NK cell-mediated rejection of resting lymphocytes in vivo - role of KARAP/DAP12-dependent and -independent pathways. European Journal of Immunology. 34 (6), 1646-1653 (2004).
- Wingender, G., Krebs, P., Beutler, B., Kronenberg, M. Antigen-specific cytotoxicity by invariant NKT cells in vivo is CD95/CD178-dependent and is correlated with antigenic potency. The Journal of Immunology. 185 (5), 2721-2729 (2010).
- Brinster, R. L., et al. Transgenic mice harboring SV40 T-antigen genes develop characteristic brain tumors. Cell. 37 (2), 367-379 (1984).
- Tatum, A. M., et al. CD8+ T cells targeting a single immunodominant epitope are sufficient for elimination of established SV40 T antigen-induced brain tumors. The Journal of Immunology. 181 (6), 4406-4417 (2008).
- Schell, T. D., Tevethia, S. S. Control of advanced choroid plexus tumors in SV40 T antigen transgenic mice following priming of donor CD8(+) T lymphocytes by the endogenous tumor antigen. The Journal of Immunology. 167 (12), 6947-6956 (2001).
- Greenberg, N. M., et al. Prostate cancer in a transgenic mouse. Proceedings of the National Academy of Sciences of the United States of America. 92 (8), 3439-3443 (1995).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados