È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sartoria in vivo saggi di citotossicità per studiare l'Immunodominanza nelle risposte delle cellule CD8+ T specifiche del tumore
In questo articolo
Riepilogo
Descriviamo qui un saggio di uccisione a base di citometria in vivo che consente di esaminare l'immunodominanza nelle risposte citotossiche dei linfociti T (CTL) a un antigene tumorale modello. Forniamo esempi di come questo saggio elegante può essere impiegato per gli studi meccanicistici e per i test di efficacia dei farmaci.
Abstract
I saggi di citotossicità in vivo (CFSE) a base di carbossimetilfluoruro di succinimidile (CFC) consentono una quantificazione sensibile e accurata delle risposte dei linfociti T (CTL) di CD8 e citolitici provocate contro i peptidi derivati dal tumore e dal patogeno. Offrono diversi vantaggi rispetto ai saggi di uccisione tradizionali. In primo luogo, consentono il monitoraggio della citotossicità CTL-mediata all'interno di organi linfoidi secondari architettonicamente intatti, tipicamente nella milza. In secondo luogo, consentono studi meccanicistici durante le fasi di adescamento, effettore e richiamo delle risposte CTL. In terzo luogo, forniscono piattaforme utili per i test di efficacia dei vaccini/farmaci in un ambiente veramente in vivo. Qui, forniamo un protocollo ottimizzato per l'esame delle risposte CTL concomitanti contro più di un epitopo peptidico di un antigene tumorale modello (AG), vale a dire, Simian virus 40 (SV40)-codificato large T AG (T AG). Come la maggior parte delle altre proteine tumorali clinicamente rilevanti, T AG ospita molti peptidi potenzialmente immunogenici. Tuttavia, solo quattro di questi peptidi inducono risposte CTL rilevabili in topi topi C57BL/6. Queste risposte sono disposte in modo coerente in un ordine gerarchico in base alla loro grandezza, che costituisce la base per TCD8 "immunodominio" in questo potente sistema. Di conseguenza, la maggior parte della risposta tCD8 specifica t AG è focalizzata contro un singolo epitopo immunodominante, mentre gli altri tre epitopi sono riconosciuti e hanno risposto solo debolmente. L'immunodominio compromette l'ampiezza delle risposte antitumorali TCD8 ed è, come tale, considerata da molti come un impedimento alla vaccinazione di successo contro il cancro. Pertanto, è importante comprendere i fattori cellulari e molecolari e i meccanismi che dettano o plasmano l'immunodominanza TCD8 . Il protocollo che descriviamo qui è adattato all'indagine di questo fenomeno nel modello di immunizzazione T AG, ma può essere prontamente modificato ed esteso a studi simili in altri modelli tumorali. Forniamo esempi di come l'impatto degli interventi immunoterapeutici sperimentali possa essere misurato utilizzando saggi di citotossicità in vivo.
Introduzione
Le cellule t CD8+ convenzionali (tCD8) giocano parti importanti nella sorveglianza immunitaria anticancro. Essi funzionano principalmente nella capacità dei linfociti T citolitici (CTLs) che riconoscono i peptidi antigeni specifici del tumore o associati (AGS) visualizzati all'interno della fessura chiusa delle principali molecole di classe I di istocompatibilità (MHC). I CTL completamente armati utilizzano il loro arsenale citotossico per distruggere le cellule maligne. Anticancro TCD8 può essere rilevato in circolazione o anche all'interno di masse primarie e metastatiche di molti pazienti oncologici e animali tumorali. Tuttavia, sono spesso anergici o esausti e non riescono a sradicare il cancro. Pertanto, molte modalità immunoterapeutiche sono progettate per aumentare le frequenze TCD8 anticancro e per ripristinare e potenziare le loro funzioni.
Le proteine tumorali ospitano molti peptidi, alcuni dei quali possono essere immunogenici e potenzialmente immunoprotettivi. Tuttavia, le risposte TCD8 quantificabili sono suscitate con magnitudini variabili contro pochi peptidi solo. Questo crea una "gerarchia di immunodominanza" tra i cloni TCD8 1. Di conseguenza, immunodominante (ID) TCD8 occupano prominenti ranghi gerarchici, che è comunemente giudicato dalla loro abbondanza. Al contrario, le cellule TCD8 il cui recettore delle cellule t (TCR) è specifico per gli epitopi SUBDOMINANTI (SD) si verificano nelle frequenze più basse. Noi e altri abbiamo identificato alcuni dei fattori che dettano o plasmano l'immunodominanza nelle risposte TCD8 . Questi includono, tra gli altri, la modalità di presentazione AG a naïve TCD8 (cioè, presentazione diretta, cross-presentazione, cross-dressing)2,3,4, il tipo di cellule presentanti AG (APCs) partecipando all'attivazione T CD85, l'abbondanza e la stabilità delle proteine AGS6,7 e l'efficienza e la cinetica del loro degrado da parte dei proteasomi7,8, il selettività relativa del trasportatore associato alla lavorazione AG (TAP) per i peptidi9, l'affinità dei peptidi liberati per le molecole MHC I9,10, la presenza, le frequenze dei precursori e la diversità di TCR di imparentata tCD8 in pool di cellule t11,12,13, Cross-Competition tra le cellule t per l'accesso a APCs14,15, e la capacità fratricida di tCD8 cloni16. Inoltre, l'immunodominanza TCD8 è sottoposto a meccanismi immunoregolanti mediati da diversi tipi di cellule soppressori, come le cellule t (nTreg) regolatorie naturali (n)17, la molecola co-inibitoria della superficie cellulare programmata morte-1 (PD-1)16, e alcuni enzimi intracellulari come indoleamine 2, 3-diossigenasi (ido)18 e l'obiettivo dei mammiferi della rapamicina (mTOR)19. È importante notare, tuttavia, che i fattori di cui sopra non sempre completamente conto per l'immunodominanza.
Oltre alla biologia di base dell'immunodominanza TCD8 , l'esame di questo intrigante fenomeno ha importanti implicazioni nell'immunologia e immunoterapia del cancro. In primo luogo, uno stato ID non conferisce necessariamente su un determinato clone TCD8 la capacità di prevenire l'iniziazione tumorale o progressione20. Se e come ID e SD TCD8 contribuiscono all'immunità antitumorale può dipendere dal tipo e l'entità di malignità e il sistema sperimentale impiegato. In secondo luogo, si ritiene che i cloni ID TCD8 possano essere "troppo visibili" al sistema immunitario e, di conseguenza, più inclini ai meccanismi di tolleranza centrale e/o periferica16,21. Terzo, i tumori eterogenei possono contenere cellule neoplastiche che evitano il rilevamento da molti, se non la maggior parte, CTLs visualizzando solo uno spettro ristretto di peptidi: MHC complessi. In queste circostanze, TCD8 risposte di ampiezza insufficiente sono suscettibili di permettersi tali cellule tumorali un vantaggio di sopravvivenza, potenziando così la loro conseguenza22. È per le ragioni di cui sopra che molti vista immunodominanza come un ostacolo per la vaccinazione di successo TCD8-based e terapie contro il cancro.
L'inoculo di topi C57BL/6 topi con il virus simian 40 (SV40)-cellule trasformate che esprimono grande tumore AG (T AG) fornisce un potente sistema preclinico per studiare l'immunodominanza TCD8 . Questo modello offre diversi vantaggi. In primo luogo, gli epitopi peptidici di questa oncoproteina clinicamente rilevante sono ben caratterizzati in questo ceppo di topo23 (tabella 1). In secondo luogo, T AG epitopi, che sono chiamati siti I, II/III, IV, e V, trigger TCD8 risposte che sono sistematicamente disposti nel seguente ordine gerarchico: sito IV > > sito I ≥ sito II/iii > ≫ sito V. di conseguenza, il sito IV specifico TCD8 montare la risposta più robusta a T AG. Al contrario, i siti I e II/III sono subdominanti e i TCD8 specifici del sito V sono meno abbondanti e di solito rilevabili solo in assenza di reattività ad altri epitopi23,24. In terzo luogo, la linea di cellule tumorali T AG+ utilizzata nel protocollo qui descritto, vale a dire C57SV cellule di fibrosarcoma, e quelle utilizzate nelle nostre indagini precedenti16,17,18,19 ,25,26, sono trasformati con frammenti SV40 subgenomici25. Pertanto, essi non sono in grado di assemblare e rilasciare SV40 virioni che potrebbero potenzialmente infettare host APCs. Inoltre, le cellule C57SV sono prive di molecole classiche costimulatorie come CD80 (B7-1), CD86 (B7-2) e CD137 ligando (4-1BBL)16. Gli attributi di cui sopra rendono queste linee ideali per l'esame dell'attivazione in vivo TCD8 tramite cross-adescamento. L'adescamento incrociato è una via importante nell'indurre le risposte TCD8 , in particolare quelle lanciate contro le cellule tumorali di origine non ematopoietica che non riescono a prime cellule T naïve direttamente25.
Le frequenze e/o le funzioni antitumor TCD8 possono essere monitorate dalla colorazione del TETRAMERO MHC I, dalla colorazione intracellulare per le citochine effettrici (ad es. interferone [IFN]-γ) o dalle molecole lisiche (ad es. perforina), dai dosaggi di immunospot (ELISPOT) collegati agli enzimi ed ex saggi di citotossicità in vivo. Sin dalla loro nascita negli anni novanta27,28, i saggi di uccisione in vivo di carbossilfluorite succinimidilestere (CFSE) hanno consentito la valutazione delle risposte citotossiche mediate da CTLs antivirali29,30 , 31, antitumorale CTLs16,32, cellule Natural Killer (NK)33, cellule di Natural Killer T (iNKT) invarianti di glycolipid-reattivo34, e pre-esistenti e de novo donatori specifici alloanticorpi26. Pertanto, le loro applicazioni possono essere di interesse per un ampio pubblico, compresi ma non limitati a investigatori che lavorano nelle aree di immunologia e immunoterapia tumorale, immunità anti-patogeni, e la progettazione preventiva e terapeutica del vaccino.
Per valutare la citotossicità cellulare-mediata in scenari tipici, due popolazioni di splenociti ingenui che mostrano una AG irrilevante o una AG (s) cognata sono etichettate con due diverse dosi di CFSE, mescolate in numero uguale e iniettato in naïve (controllo) o killer topi che ospitano cellule. La presenza/assenza di ogni popolazione bersaglio viene quindi esaminata dalla citometria a flusso.
Abbiamo ottimizzato e impiegato i saggi di uccisione in vivo nei nostri studi sull'immunodominanza nelle risposte antivirali e antitumore T CD812,16,17. Qui, forniamo un protocollo dettagliato per la valutazione simultanea di ID e SD TCD8 risposte agli epitopi di t AG, che possono essere prontamente adottati per indagini simili in altri sistemi sperimentali. Forniamo anche risultati rappresentativi che dimostrano che la deplezione delle cellule nTreg e il blocco PD-1 possono migliorare selettivamente l'IDtCD8-e la citotossicità indotta da SD t CD8, rispettivamente. Alla fine, discuteremo i molteplici vantaggi dei saggi di uccisione in vivo e di alcuni dei loro limiti intrinseci.
Protocollo
Gli esperimenti qui descritti seguono i protocolli di uso degli animali approvati da enti istituzionali e aderiscono a linee guida nazionali consolidate.
1. inoculo di topi C57BL/6 topi con cellule tumorali che esprimono T AG
- Far crescere la linea di cellule fibrosarcoma SV40-trasformata C57SV (o una linea cellulare T AG+ aderente simile) nel medium dell'Aquila modificato di Dulbecco (DMEM) con 4,5 g/l D-glucosio e l-glutammina (1x) e completato con 1 mm di piruvato di sodio e 10% di calore inattivato siero bovino fetale (FBS) nei palloni trattati con colture tissutali a 37 ° c in atmosfera umidificata contenente 10% di CO2.
- Una volta che le cellule diventano completamente confluenti o leggermente sovconfluenti, rimuovere delicatamente e scartare il mezzo e sciacquare il monostrato con soluzione salina sterile tampone fosfato pre-riscaldato (PBS).
Nota: la massima espressione T AG si ottiene quando le cellule T AG+ raggiungono il 100% di confluenza. - All'interno di un armadietto di sicurezza biologico, aggiungere tripsin-EDTA pre-riscaldato (0,25%) per coprire il monostrato a temperatura ambiente fino a quando le cellule vengono disalloggiate in patch. Toccare i lati del pallone (i) di coltura più volte per liberare le restanti cellule aderenti.
Nota: se necessario e per accelerare il processo di tripsinizzazione, trasferire il pallone (i) in un incubatore 37 ° c. Le cellule disalloggiate adotteranno rapidamente una forma arrotondata sotto un microscopio leggero. Questo passo dovrebbe durare circa 5 min. - Aggiungere 5 mL di mezzo DMEM e dissociare i gruppi per preparare una sospensione a singola cellula pipettando il contenuto di ciascun pallone su e giù.
- Trasferire la sospensione cellulare attraverso un filtro a cellule con pori di 70 μm in un tubo.
- Girare verso il basso il tubo a 400 x g per 5 min a 4 ° c.
- Scartare il supernatante. Risospendere le cellule pellettate in 10 mL di PBS sterile freddo.
- Ripetere i passaggi 1,6 e 1,7 due volte.
- Contare le cellule usando un hemocytometer. Preparare una sospensione uniforme contenente 4 x 107 cellule/ml sterile PBS.
- Iniettare 500 μl della sospensione di cui sopra intraperitoneale (IP) in ogni adulto (6-12-settimana-vecchio) maschio o femmina topi C57BL/6 mouse.
2. regimi terapeutici
-
Regime di trattamento per esaminare il contributo delle cellule nTreg all'immunodominanza TCD8
- Quattro giorni prima dell'adescamento in vivo di topi C57BL/6 topi con cellule C57SV (fase 1,10), iniettare ogni animale una volta con 0,5 mg di un anticorpo monoclonale anti-CD25 (mAb) a bassa endotossina, privo di azide (clone PC-61.5.3), che esaurisce le cellule di nTreg, o con un isotipo IgG1 del ratto controllo (ad esempio, clone KLH/G1-2-2, clone HRPN o clone TNP6A7).
-
Regime di trattamento per testare l'importanza in vivo delle interazioni PD-1-PD-L1 (2) nel plasmare l'immunodominanza TCD8
Nota: l'impegno di PD-1 da PD-L1 spesso, ma non sempre, media la co-inibizione e/o l'esaurimento di AG-specifico TCD8. Pertanto, il trattamento con anti-PD-1 può essere eseguito parallelamente alla somministrazione di mAbs anti-PD-L1 e anti-PD-L2 per rivelare l'esatta interazione intercellulare coinvolta in un fenomeno biologico.
3. preparazione dei splenociti bersaglio
- Euthanize del sesso-abbinato naïve topi C57BL/6 topi (6-12 settimane di età) che servirà come donatori splenociti per lussamento cervicale.
- Posizionare ogni mouse con l'addome rivolto verso l'alto all'interno di un armadio di sicurezza biologico. Spruzzare la pelle con 70% (v/v) EtOH. Utilizzando pinze e forbici sterili, sollevare la pelle e fare una piccola incisione della linea mediana ventrale. Quindi, tagliare la pelle in un modo simile a croce per esporre il peritoneo.
- Utilizzando pinze, tirare il peritoneo in una tenda-come la moda senza strappare uno degli organi interni. Tagliare il peritoneo aperto per esporre la cavità peritoneale e rimuovere delicatamente la milza.
- Posizionare la milza (s) all'interno di una smerigliatrice di tessuto Dounce da 15 mL contenente 5 mL di PBS sterile. Applicare la pressione manuale utilizzando lo stantuffo di vetro della smerigliatrice fino a quando il tessuto splenico si disseppi in una sospensione di cellule rosse omogenee.
Nota: a seconda del numero di animali riceventi per gruppo sperimentale, possono essere necessari diversi milze del topo donatore per la preparazione delle cellule bersaglio. Fino a 3 milze possono essere omogeneizzati insieme all'interno di una smerigliatrice da 15 ml. - Trasferire l'omogeneato in un tubo da 15 mL. Girare verso il basso il tubo a 400 x g per 5 min a 4 ° c.
- Scartare il supernatante. Risospendere le cellule pellettate in 4 mL di tampone di lising di ammonio-cloruro-potassio (ACK) per 4 min per eliminare gli eritrociti.
Nota: questo è un passaggio sensibile al tempo. Sovraesporre i splenociti al tampone di lising ACK aumenterà la loro fragilità e li renderà suscettibili alla morte cellulare non specifica. - Per ogni tubo, aggiungere 8 mL di RPMI 1640 medium contenente 10% FBS inattivati dal calore, L-alanyl-L-Glutammina, 0,1 mM minimi essenziali (MEM) aminoacidi non essenziali, 1 mM di piruvato di sodio, 10 mM di HEPES, e 1x penicillina/streptomicina, che sarà in seguito indicato come mezzo RPMI completo (tabella dei materiali).
- Trasferire il contenuto attraverso i pori di 70 μm di un filtro cellulare in un nuovo tubo da 15 mL.
- Girare verso il basso il tubo a 400 x g per 5 min a 4 ° c.
- Scartare il supernatante. Risospendere le cellule pellettate in 12 mL di RPMI completa.
- Dividere la sospensione splenocitaria in 3 porzioni uguali (4 mL ciascuna) in 3 tubi separati.
4. rivestimento target splenociti con peptidi irrilevanti e cognati
- Etichettare i tubi secondo i peptidi che verranno utilizzati per pulsare splenociti bersaglio. I splenociti di controllo saranno pulsati con un peptide irrilevante, e ogni popolazione di splenociti bersaglio cognato sarà pulsata con un peptide sintetico corrispondente all'epitopo immunodominante derivato da T AG (sito IV) o un epitopo di T AG Subdominante (sito I o sito II/III) (tabella 1).
Nota: la scelta di peptidi irrilevanti dipende dalla configurazione sperimentale e dal ceppo di topo utilizzato in ogni indagine. Gli autori usano spesso gB498-505 (un epitopo di peptide immunodominante con restrizionebh-2k di virus dell'herpes simplex [HSV]-1) e/o GP33-41 (un epitopo del peptide immunodominante h-2Db-Restricted di linfocitico virus della choriomeningite ([LCMV]) in topi C57BL/6 topi (tabella 1). Questi peptidi sono scelte ottimali perché: (i) sono derivati da patogeni non precedentemente incontrati nel modello di topo descritto qui; (II) simili ai peptidi derivati da T AG, i gB498-505 e i GP33-41 sono limitati da e si legano alle molecole di H-2b . Nei saggi di uccisione in vivo a tre picchi, ciascuno dei due picchi che corrispondono alle cellule bersaglio imparentata può rappresentare splenociti pulsati con un peptide immunodominante o Subdominante. La scelta di ogni set di peptidi varia in base agli obiettivi di ogni esperimento. Vedere Figura 1 e figura 2 come esempi di tale variazione. Per il resto di questo protocollo, I siti di derivazione I e IV di T AG rappresenteranno rispettivamente peptidi subdominanti e immunodominanti. - Pulsare il contenuto di ogni tubo etichettato con 1 μM del rispettivo peptide per 1 h a 37 ° c e 5% CO2.
- Utilizzare un filtro a cella separato (con pori di 70 μm) per ogni tubo per rimuovere i residui e i detriti, se necessario.
- Girare verso il basso il tubo a 400 x g per 5 min a 4 ° c. Scartare il supernatante.
- Risospendere le cellule pellettate in 12 mL di PBS sterile freddo e ripetere il passo 4,4 ancora una volta.
Nota: è importante rimuovere quanto più FBS possibile perché FBS può associare CFSE nel passaggio successivo.
5. etichettatura splenociti bersaglio con CFSE
- Risospendere la splenociti pulsata peptidica in 4 mL di PBS sterile.
- Aggiungere CFSE a 0,025 μM, 0,25 μM e 2 μM nei tubi contenenti rispettivamente splenociti di peptide, sito I-e sito IV-pulsato.
Nota: per ottenere l'etichettatura CFSE uniforme, tenere ogni tubo ad un angolo di 45 ° prima di aggiungere CFSE al lato leggermente sopra la sospensione cellulare seguita immediatamente da un delicato vortexing. Questo garantirà la comparsa di istogrammi lisci alla fine. Le variazioni batch-to-batch e dipendenti dall'età nelle intensità CFSE non sono rare. Pertanto, si può avere bisogno di sperimentare con le dosi differenziali di CFSE prima di decidere sulle concentrazioni ottimali da utilizzare.
Attenzione: il CFSE è tossico a concentrazioni superiori a 5 μM. - Posizionare i tubi all'interno di un incubatore di 37 ° c per 15 min e invertirli una volta ogni 5 min.
- Aggiungere 3 mL di FBS inattivati dal calore a ciascun tubo per fermare la reazione di CFSE. Rifinire il contenuto con PBS sterile.
- Girare verso il basso il tubo a 400 x g per 5 min a 4 ° c. Scartare il supernatante.
- Risospendere le cellule pellettate in 12 mL di PBS sterile e ripetere il passo 5,5.
6. esame dell'etichettatura CFSE adeguata/equa delle popolazioni di splenociti bersaglio
- Risospendere le cellule pellettate in 3 mL di PBS.
- Agitare delicatamente i tubi. Trasferire 10 μL, ciascuno, di CFSEbasso, CFSEintermedio (int), e le sospensioniad alta cella CFSE in un 5 ml di polistirolo di base rotonda a fluorescenza (FACS), contenente 200 μl di PBS.
- Interrogare le cellule utilizzando un citometro a flusso dotato di un laser 488 nm. Disegna un cancello linfocitario basato sulle proprietà di dispersione in avanti (FSC) e dispersione laterale (SSC) delle cellule prima di acquisire 5000 eventi che rientrano nel cancello dei linfociti nel canale FL-1.
- All'interno della popolazione CFSE+ "Parent", disegna ulteriori porte di istogramma per identificare le sottopopolazioni CFSElow, CFSEinte CFSEHigh .
- Confermare i numeri di evento uguali o quasi uguali all'interno dei tre cancelli. Se necessario, regolare i numeri di cella nei tubi "sorgente" (passaggio 6,1) prima di miscelare e iniettare splenociti bersaglio in topi ingenui e innescati nella sezione 7.
7. iniezione di cellule bersaglio etichettate con CFSE in destinatari naïve e con primed T-AG
- Agitare delicatamente i tubi sorgente. Trasferire le tre sospensioni di celle con etichetta CFSE in rapporti uguali in un nuovo tubo.
- Rifinire il contenuto con PBS sterile.
- Girare verso il basso il tubo a 400 x g per 5 min a 4 ° c. Risospendere le cellule pellettate con PBS sterile.
- Contare le cellule in Trypan blu da un emocytometro per garantire la vitalità cellulare di almeno 95%.
- Regolare il volume al fine di iniettare 1 x 107 cellule bersaglio miste/200 μl PBS per via endovenosa (i.v.), tramite vena di coda, in ogni destinatario topi C57BL/6 mouse.
Nota: conservare le cellule sul ghiaccio tra le iniezioni. Mescolare delicatamente le cellule bersaglio prima di ogni iniezione. Registrare l'ora esatta di iniezione per ogni topo, che determinerà quando l'animale avrà bisogno di essere eutanizzata. È importante mantenere la durata della citotossicità in vivo consistente tra tutti gli animali nello stesso esperimento.
8. acquisizione dati
- Due o quattro ore dopo l'iniezione di cellule bersaglio con etichetta CFSE, eutanizzare i topi destinatari per lussazione cervicale.
Nota: la durata della citotossicità in vivo può variare a seconda del sistema sperimentale impiegato, dell'immunogenicità dell'AGS bersaglio, dell'abbondanza prevista di TCD8 specifico per l'antigene peptidico nella milza e della robustezza della loro funzione Lisica tra gli altri fattori. - Rimuovere ed elaborare ogni milza separatamente come nei passaggi 3.2 − 3.9.
- Eliminare il supernatante e risospendere le cellule pellettate in 3 mL di PBS.
Nota: prestare attenzione a maneggiare il tessuto splenico e le preparazioni cellulari a 4 ° c o su ghiaccio prima delle analisi cytofluorimetriche. Questo per prevenire la continua citotossicità ex vivo. - Trasferire circa 1 x 107 cellule da ogni milza elaborata in un tubo FACS pulito.
- Interrogare immediatamente le cellule utilizzando un citometro a flusso dotato di un laser a 488 nm. Disegna una porta linfocitaria basata sulle proprietà FSC e SSC delle cellule.
- Identifica i splenociti di CFSE- Recipient e le cellule bersaglio CFSE+ trasferite. Disegna altri cancelli che ospitano distinte CFSElow, CFSEinte CFSEHigh target popolazioni di cellule.
- Acquisisci un totale di 2000 eventiBassi CFSE nel canale FL-1.
9. analisi dei dati
- Calcolare la lisi specifica di ogni popolazione di cellule bersaglio imparentata utilizzando la seguente formula:
% Citotossicità specifica =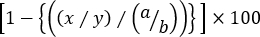
dove x = CFSEint/High numero di evento in t AG-mouse innescato, y = CFSEbasso numero di evento in t AG-mouse innescato, a = CFSEint/alto numero di evento in mouse ingenuo, e b = CFSEbasso evento numero in un mouse ingenuo.
Nota: nei saggi di citotossicità a tre picchi in cui viene valutata la lisi specifica di più di una popolazione bersaglio cognata, non è opportuno utilizzare le frequenze delle cellule bersaglio. Questo è semplicemente perché la frequenza di una popolazione di cellule bersaglio imparentata è influenzata non solo dalla percentuale dei controlli irrilevanti, ma anche da quella degli altri splenociti bersaglio affini. Pertanto, i numeri di evento all'interno di ogni cancello devono essere utilizzati nella formula sopra per calcolare accuratamente la lisi di ogni popolazione di cellule bersaglio imparentata (sia CFSEint o CFSEalte celle) contro i controlli dibassa CFSE.
Risultati
L'obiettivo dell'esperimento i cui risultati sono rappresentati nella Figura 1 è stato quello di determinare se la presenza e le funzioni delle cellule nTreg plasmano o alterano la gerarchia di immunodominanza del tCD8specifico di t AG. TOPI C57BL/6 topi sono stati iniettati IP con PBS o con 0,5 mg di un anti-CD25 mAb (clone PC-61.5.3 [PC61]) quattro giorni prima hanno ricevuto 2 x 107 C57SV cellule tumorali IP In esperimenti separati, un controllo isotipo IgG1 ratto ?...
Discussione
I saggi di citotossicità in vivo basati su CFSE offrono diversi vantaggi rispetto ai saggi tradizionali di uccisione come il cromo radioattivo (51CR) Release e i saggi di rilascio di lattato deidrogenasi colorimetrico (LDH). In primo luogo, consentono il monitoraggio della funzione CTL all'interno di un organo linfoide secondario architettonicamente intatto.
In secondo luogo, l'uccisione specifica di cellule bersaglio in saggi di citotossicità in vivo riflette il numero assoluto d...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da Canadian Institutes of Health Research (CIHR) concede MOP-130465 e PJT-156295 a SMMH. JC è parzialmente supportato da una borsa di studio di laurea Queen Elizabeth II in scienza e tecnologia presso il Ministero della formazione, College e Università dell'Ontario. CEM è stato un destinatario di una borsa di studio laureato Alexander Graham Bell Canada (dottorato) da scienze naturali e ingegneria ricerca del Canada (NSERC).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA (1X) | Thermo Fisher Scientific | 25200-056 | |
| ACK Lysing Buffer | Thermo Fisher Scientific | A1049201 | |
| Anti-mouse CD25 (clone PC-61.5.3) | Bio X Cell | BE0012 | |
| Anti-mouse PD-1 (clone RMP1-14) | Bio X Cell | BE0146 | |
| CFSE | Thermo Fisher Scientific | C34554 | |
| DMEM (1X) | Thermo Fisher Scientific | 11965-092 | |
| Fetal bovine serum (FBS) | Wisent Bioproducts | 080-150 | Heat-inactivate prior to use |
| GlutaMAX (100X) | Thermo Fisher Scientific | 35050-061 | |
| HEPES (1M) | Thermo Fisher Scientific | 15630080 | 10 mM final concentration |
| MEM Non-Essential Amino Acids Solution (100X) | Thermo Fisher Scientific | 11140-050 | |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781 | Stock is 100X |
| Rat IgG1 (clone KLH/G1-2-2) | SouthernBiotech | 0116-01 | Isotype control |
| Rat IgG1 (clone HRPN) | Bio X Cell | BE0088 | Isotype control |
| Rat IgG1 (clone TNP6A7) | Bio X Cell | BP0290 | Isotype control |
| Rat IgG2a (clone 2A3) | Bio X Cell | BP0089 | Isotype control |
| RPMI 1640 (1X) | Thermo Fisher Scientific | 11875-093 | |
| Sodium Pyruvate (100 mM) | Thermo Fisher Scientific | 11360-070 | 1 mM final concentration |
Riferimenti
- Yewdell, J. W., Bennink, J. R. Immunodominance in major histocompatibility complex class I-restricted T lymphocyte responses. Annual Review of Immunology. 17, 51-88 (1999).
- Chen, W., et al. Reversal in the immunodominance hierarchy in secondary CD8+ T cell responses to influenza A virus: roles for cross-presentation and lysis-independent immunodomination. The Journal of Immunology. 173 (8), 5021-5027 (2004).
- Otahal, P., et al. Inefficient cross-presentation limits the CD8+ T cell response to a subdominant tumor antigen epitope. The Journal of Immunology. 175 (2), 700-712 (2005).
- Lauron, E. J., et al. Cross-priming induces immunodomination in the presence of viral MHC class I inhibition. PLoS Pathogens. 14 (2), e1006883 (2018).
- Crowe, S. R., et al. Differential antigen presentation regulates the changing patterns of CD8+ T cell immunodominance in primary and secondary influenza virus infections. The Journal of Experimental Medicine. 198 (3), 399-410 (2003).
- Probst, H. C., et al. Immunodominance of an antiviral cytotoxic T cell response is shaped by the kinetics of viral protein expression. The Journal of Immunology. 171 (10), 5415-5422 (2003).
- Gileadi, U., et al. Generation of an immunodominant CTL epitope is affected by proteasome subunit composition and stability of the antigenic protein. The Journal of Immunology. 163 (11), 6045-6052 (1999).
- Zanker, D., Waithman, J., Yewdell, J. W., Chen, W. Mixed proteasomes function to increase viral peptide diversity and broaden antiviral CD8+ T cell responses. The Journal of Immunology. 191 (1), 52-59 (2013).
- Deng, Y., Yewdell, J. W., Eisenlohr, L. C., Bennink, J. R. MHC affinity, peptide liberation, T cell repertoire, and immunodominance all contribute to the paucity of MHC class I-restricted peptides recognized by antiviral CTL. The Journal of Immunology. 158 (4), 1507-1515 (1997).
- Chen, W., Khilko, S., Fecondo, J., Margulies, D. H., McCluskey, J. Determinant selection of major histocompatibility complex class I-restricted antigenic peptides is explained by class I-peptide affinity and is strongly influenced by nondominant anchor residues. The Journal of Experimental Medicine. 180 (4), 1471-1483 (1994).
- Kotturi, M. F., et al. Naive precursor frequencies and MHC binding rather than the degree of epitope diversity shape CD8+ T cell immunodominance. The Journal of Immunology. 181 (3), 2124-2133 (2008).
- Haeryfar, S. M., et al. Terminal deoxynucleotidyl transferase establishes and broadens antiviral CD8+ T cell immunodominance hierarchies. The Journal of Immunology. 181 (1), 649-659 (2008).
- Leon-Ponte, M., Kasprzyski, T., Mannik, L. A., Haeryfar, S. M. Altered immunodominance hierarchies of influenza A virus-specific H-2(b)-restricted CD8+ T cells in the absence of terminal deoxynucleotidyl transferase. Immunological Investigations. 37 (7), 714-725 (2008).
- Kedl, R. M., et al. T cells compete for access to antigen-bearing antigen-presenting cells. The Journal of Experimental Medicine. 192 (8), 1105-1113 (2000).
- Kastenmuller, W., et al. Cross-competition of CD8+ T cells shapes the immunodominance hierarchy during boost vaccination. The Journal of Experimental Medicine. 204 (9), 2187-2198 (2007).
- Memarnejadian, A., et al. PD-1 Blockade Promotes Epitope Spreading in Anticancer CD8(+) T Cell Responses by Preventing Fratricidal Death of Subdominant Clones To Relieve Immunodomination. The Journal of Immunology. 199 (9), 3348-3359 (2017).
- Haeryfar, S. M., DiPaolo, R. J., Tscharke, D. C., Bennink, J. R., Yewdell, J. W. Regulatory T cells suppress CD8+ T cell responses induced by direct priming and cross-priming and moderate immunodominance disparities. The Journal of Immunology. 174 (6), 3344-3351 (2005).
- Rytelewski, M., et al. Suppression of immunodominant antitumor and antiviral CD8+ T cell responses by indoleamine 2,3-dioxygenase. PLoS One. 9 (2), e90439 (2014).
- Maleki Vareki, S., et al. Differential regulation of simultaneous antitumor and alloreactive CD8(+) T-cell responses in the same host by rapamycin. American Journal of Transplantation. 12 (1), 233-239 (2012).
- Irvine, K., Bennink, J. Factors influencing immunodominance hierarchies in TCD8+ -mediated antiviral responses. Expert Review of Clinical Immunology. 2 (1), 135-147 (2006).
- Grossmann, M. E., Davila, T., Celis, T. Avoiding tolerance against prostatic antigens with subdominant peptide epitopes. Journal of Immunotherapy. 24 (3), 237-241 (2001).
- Schreiber, H., Wu, T. H., Nachman, J., Kast, W. M. Immunodominance and tumor escape. Seminars in Cancer Biology. 12 (1), 25-31 (2002).
- Mylin, L. M., et al. Quantitation of CD8(+) T-lymphocyte responses to multiple epitopes from simian virus 40 (SV40) large T antigen in C57BL/6 mice immunized with SV40, SV40 T-antigen-transformed cells, or vaccinia virus recombinants expressing full-length T antigen or epitope minigenes. Journal of Virology. 74 (15), 6922-6934 (2000).
- Fu, T. M., et al. An endoplasmic reticulum-targeting signal sequence enhances the immunogenicity of an immunorecessive simian virus 40 large T antigen cytotoxic T-lymphocyte epitope. Journal of Virology. 72 (2), 1469-1481 (1998).
- Chen, W., et al. Cross-priming of CD8+ T cells by viral and tumor antigens is a robust phenomenon. European Journal of Immunology. 34 (1), 194-199 (2004).
- Memarnejadian, A., Meilleur, C. E., Mazzuca, D. M., Welch, I. D., Haeryfar, S. M. Quantification of Alloantibody-Mediated Cytotoxicity In Vivo. Transplantation. 100 (5), 1041-1051 (2016).
- Aichele, P., et al. Peptide antigen treatment of naive and virus-immune mice: antigen-specific tolerance versus immunopathology. Immunity. 6 (5), 519-529 (1997).
- Oehen, S., Brduscha-Riem, K. Differentiation of naive CTL to effector and memory CTL: correlation of effector function with phenotype and cell division. The Journal of Immunology. 161 (10), 5338-5346 (1998).
- Coles, R. M., Mueller, S. N., Heath, W. R., Carbone, F. R., Brooks, A. G. Progression of armed CTL from draining lymph node to spleen shortly after localized infection with herpes simplex virus 1. The Journal of Immunology. 168 (2), 834-838 (2002).
- Barber, D. L., Wherry, E. J., Ahmed, R. Cutting edge: rapid in vivo killing by memory CD8 T cells. The Journal of Immunology. 171 (1), 27-31 (2003).
- Meilleur, C. E., et al. Bacterial superantigens expand and activate, rather than delete or incapacitate, preexisting antigen-specific memory CD8+ T cells. The Journal of Infectious Diseases. , (2018).
- Goldszmid, R. S., et al. Dendritic cells charged with apoptotic tumor cells induce long-lived protective CD4+ and CD8+ T cell immunity against B16 melanoma. The Journal of Immunology. 171 (11), 5940-5947 (2003).
- Oberg, L., et al. Loss or mismatch of MHC class I is sufficient to trigger NK cell-mediated rejection of resting lymphocytes in vivo - role of KARAP/DAP12-dependent and -independent pathways. European Journal of Immunology. 34 (6), 1646-1653 (2004).
- Wingender, G., Krebs, P., Beutler, B., Kronenberg, M. Antigen-specific cytotoxicity by invariant NKT cells in vivo is CD95/CD178-dependent and is correlated with antigenic potency. The Journal of Immunology. 185 (5), 2721-2729 (2010).
- Brinster, R. L., et al. Transgenic mice harboring SV40 T-antigen genes develop characteristic brain tumors. Cell. 37 (2), 367-379 (1984).
- Tatum, A. M., et al. CD8+ T cells targeting a single immunodominant epitope are sufficient for elimination of established SV40 T antigen-induced brain tumors. The Journal of Immunology. 181 (6), 4406-4417 (2008).
- Schell, T. D., Tevethia, S. S. Control of advanced choroid plexus tumors in SV40 T antigen transgenic mice following priming of donor CD8(+) T lymphocytes by the endogenous tumor antigen. The Journal of Immunology. 167 (12), 6947-6956 (2001).
- Greenberg, N. M., et al. Prostate cancer in a transgenic mouse. Proceedings of the National Academy of Sciences of the United States of America. 92 (8), 3439-3443 (1995).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon