Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Efecto isotópico en el proceso de transferencia de doble protones de porficeno investigado por el método QM/MM mejorado
En este artículo
Resumen
Aquí se presenta un protocolo que utiliza el método QM/MM mejorado para investigar el efecto isotópico en el proceso de transferencia de doble protones en porficeno.
Resumen
La sustitución de deuterio único en porficeno conduce a una geometría molecular asimétrica, que puede afectar al proceso de transferencia de doble protones en la molécula de porficeno. En este estudio, aplicamos un método mejorado QM/MM llamado SITS-QM/MM para investigar los efectos del isótopo de hidrógeno/deuterio (H/D) en la transferencia de doble protones en porficéo. Los cambios de distancia en las simulaciones de dinámica molecular SITS-QM/MM sugirieron que el deuterio sustituyó al porficeno adoptó el mecanismo de transferencia de doble protones escalonado. El análisis estructural y los cambios de energía libre del proceso de transferencia de doble protones indicaron que la sustitución isotópica asimétrica comprimió sutilmente los enlaces de hidrógeno covalentes y puede alterar la ubicación original del estado de transición.
Introducción
El proceso de transferencia de protones en porficéos tiene aplicaciones potenciales en el desarrollo de interruptores moleculares, transistores y dispositivos de almacenamiento de información1,2. En particular, la tautomerización en porficéos a través del proceso de transferenciade doble protones ha atraído un amplio interés en los campos de la espectroscopia y la fotofísica 2. Los átomos de hidrógeno interno de porficeno pueden migrar de un isómero trans al otro isómero trans equivalente a través del proceso de transferencia de doble protones como se muestra en la Figura 1. Se han propuesto dos mecanismos para el proceso de transferencia de doble protones: el mecanismo concertado y el mecanismo escalonado3,4. En el proceso concertado de transferencia de doble protones, ambos átomos de protones se mueven al estado de transición sincrónicamente de una manera simétrica, mientras que un protón completa la transferencia antes que el otro protón en un proceso escalonado. Dos átomos de hidrógeno pueden transferirse simultáneamente o escalonadamente dependiendo de la fuerza de correlación entre dos átomos de hidrógeno5.
La sustitución isotópica se ha utilizado para detectar las propiedades estructurales de las moléculas y velocidad constantes de la cinética de reacción6. La sustitución de deuterio único en el hidrógeno interno del porficeno conduce a una forma asimétrica de la molécula. El enlace de hidrógeno puede expandirse o contraerse debido a la diferencia de masa entre los átomos de hidrógeno y deuterio. La sustitución isotópica introduce una perturbación en el andamio del porficeno. Se plantea la cuestión de si la estructura asimétrica afectaría el proceso de transferencia de protones. Limbach y compañeros de trabajo informaron que la sustitución del hidrógeno por deuterio comprimirá tanto los enlaces de hidrógeno, como el acoplamiento cooperativo de dos enlaces de hidrógeno en porficeno puede favorecer el mecanismo concertado7,mientras que Yoshikawa declaró que el deuteración haría que el mecanismo escalonado contribuyera más que el mecanismo concertado8. Se han desarrollado técnicas experimentales, como la espectroscopia de fuerza, paracapturar detalles de tautomerización en un solo porficeno 9. Sin embargo, sigue siendo difícil determinar los detalles atómicos de la transferencia de protones experimentalmente debido a su naturaleza transitoria.
Los cálculos teóricos y las simulaciones pueden actuar como herramientas complementarias para esclarecer los mecanismos de reacción de la transferencia de protones. Entre los diferentes métodos teóricos, las simulaciones de dinámica molecular (MD) pueden monitorear los movimientos dinámicos de cada átomo, y se ha utilizado ampliamente para revelar mecanismos complejos en reacciones químicas y enzimáticas. Sin embargo, las simulaciones MD regulares tienden a sufrir problemas de muestreo insuficientes, especialmente cuando existe una barrera de energía alta en el proceso de interés. Por lo tanto, se han desarrollado métodos de muestreo mejorados, que incluyen el muestreo de ruta de transición10,11, muestreo paraguas (EE.UU.)12,13, y muestreo de templado integrado (ITS)14, 15. La combinación de diferentes métodos de muestreo mejorados puede aumentar aún más la eficiencia de muestreo16,17,18. Para aprovechar los algoritmos de muestreo mejorados en la simulación de reacciones químicas, hemos implementado el método de muestreo de templado integrado selectivo (SITS) con potencial mecánico cuántico y mecánico molecular (QM/MM) recientemente19. El método SITS-QM/MM propuesto combina las ventajas de ambos métodos: el método SITS acelera el muestreo y puede explorar todos los canales de reacción posibles sin conocimiento previo del mecanismo de reacción, y QM/MM proporciona una descripción más precisa de el proceso de formación de unión y ruptura de unión, que no puede ser simulado por los métodos MM únicamente. El enfoque SITS-QM/MM implementado ha descubierto con éxito la transferencia concertada de doble protones, mecanismo de transferencia de doble protones no correlacionado y correlacionado en diferentes sistemas, sin predefinir las coordenadas de reacción19. En el caso del porficeno, se ha notificado el carácter de transferencia de protones escalonapero pero correlacionado19. El método híbrido SITS-QM/MM se utilizó para investigar el efecto isotópico en el porficeno en nuestro estudio, y a continuación se muestran las descripciones detalladas del algoritmo y el protocolo de nuestro método.
Hemos implementado el método SITS con potencial sin QM/MM híbrido. Se definió el potencial efectivo de SITS para incluir la energía potencial a diferentes temperaturas con los factores de ponderación nk para cubrir rangos de temperatura más amplios,
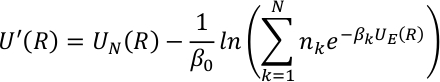
donde, N es el número de términos canónicos, la temperatura inversa es la temperatura inversa, y nk es el factor de ponderación correspondiente para cada componente canónico. UE (R) y UN(R) representan los términos mejorados y no mejorados en SITS y se definen como,
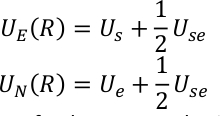
U s, Use y Ue son la energía potencial del subsistema, la interacción entre el subsistema y el medio ambiente, y la energía potencial del medio ambiente. El potencial QM/MM se expresa como una suma híbrida de tres componentes,
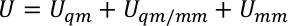
donde Uqm, Uqm/mmy Umm son el término de energía interna del subsistema QM, la energía de interacción entre las regiones QM y MM y la energía de interacción dentro del subsistema MM, respectivamente. El término Uqm/mm se puede dividir en tres componentes, que incluyen los términos electrostáticos, van der Waals y energía de interacción covalente entre los átomos QM y MM,

Asignamos, 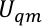
 y en un término de Us en SITS,
y en un término de Us en SITS,
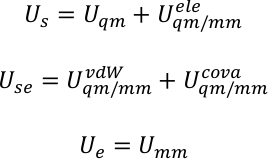
El potencial total del sistema se descompuso entonces en la energía del subsistema U s, la energía de interacción entre el subsistema y el medio ambiente Use, y la energía del medio ambiente Ue. Por ejemplo, en el sistema de la obra actual, el subsistema es la porficencia, y el medio ambiente el agua.
El perfil PMF a lo largo de una variable colectiva (R) se deriva como,
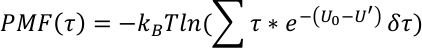
Las coordenadas de reacción generalmente utilizadas para cada transferencia de hidrógeno de N1-H1- N2 son q1 o (r1ar2)/2 y q2 a r1 + r2, donde se encuentran1 es la distancia de N1-H1 y r2 es la distancia de H1-N2.
El método se ha implementado en el paquete de simulación QM/MM MD QM4D20. El código fuente completo y la documentación se pueden encontrar aquí: http://www.qm4d.info/.
Generalmente, las simulaciones SITS-QM/MM MD implican cuatro pasos: pre-equilibrio (pre-sits); optimización nk (opt-sits); simulación de producción y análisis de datos.
Protocolo
1. Modelo de construcción
- Construir la estructura del porficeno: Abra el software GaussView haciendo doble clic en el ratón. A continuación, haga clic en el botón Fragmento de elemento en el menú de GaussView para elegir los elementos necesarios. Construir pórfido. A continuación, haga clic en el botón Archivo para guardar como el archivo pdb.
- Solvate el modelo: Solvate porficene en una caja de agua cúbica TIP3P21 con una longitud de borde de 38o mediante la emisión del comando en el sistema operativo linux: genbox_d -cp prp-vac.pdb -cs spc216.gro -o solv.pdb -maxsol 1484 -box 3.8.
- Construir deuterio porficeno: Ejecute el siguiente comando para generar el archivo de topología: cns < ppi_solv.inp. A continuación, abra prp-wat.psf con el comando vi y cambie la masa de H1 de 1.00800 a 2.01600 para reemplazar un átomo de hidrógeno intramolecular de porficeno con deuterio para construir un solo deuterio sustituido por el porficeno.
-
Establezca los parámetros normales de simulación de MD: Método de entrada scctb, integral 0.5 fsy cutoff 12 en el archivo de entrada MD abriéndolo con el comando vi.
NOTA: Adoptar una distancia de corte de 12o para calcular las interacciones tanto vdW como electrostáticas. Simular la molécula de porficeno con el método DFTB/MIO22. Establezca el paso de tiempo de integración como 0,5 fs para simulaciones MD. Mantenga la temperatura del sistema de simulación a 300 K con el termostato Langevin. A continuación, realice las simulaciones con el software QM4D siguiendo los pasos a continuación.
2. Pre-sits
-
Configurar parámetros de temperatura: Entrada templow 260, temphigh 1100 y ntemp 160 en el archivo de entrada.
NOTA: El rango de temperatura de 260K a 1100K fue distribuido por el software QM4D a 160 puntos de temperatura durante las simulaciones MD. Los archivos de entrada de plantilla se incluyen en Archivos suplementarios. -
Inicie las pre-sits: Establezca runtype 100 y step 120,000 en el archivo de entrada. A continuación, ejecute el siguiente comando: $PATH/qm4d $INPUTFILE > $OUTPUTFILE.
NOTA: El paso total es de 120.000, pero se puede ajustar en función de la necesidad específica. Los parámetros sugeridos de las simulaciones MD se guardan en el $INPUTFILE. El mismo comando también se utiliza en los siguientes pasos de opt-sits y simulación de producción, con el archivo de entrada modificado en consecuencia. -
Cálculo de las energías descompuestas
- Extraer cambios de energía: Durante la etapa de pre-sits, monitoree la energía de cada término para calcular los valores medios, como se muestra en la Figura1. Utilice el comando grep Linux para extraer la energía de la siguiente manera:
grep 'SITS-ener0' $INPUTFILE á awk ''a+'$3;b+'$4;c+'$5'END'print a/NR,b/NR,c/NR'. - Modificar energías medias en el archivo de entrada MD: Calcule las energías medias basadas en la salida de la línea de comandos anterior, y modifique la línea vshift0 -30801.95; vshift1 -26.88; vshift2 -13888.28 en el archivo de entrada con los promedios recién generados.
NOTA: Los números -30801.85, -26.88 y -13888.28 son las energías promedio en el sistema modelo actual. Modifique los valores en función de los sistemas específicos.
- Extraer cambios de energía: Durante la etapa de pre-sits, monitoree la energía de cada término para calcular los valores medios, como se muestra en la Figura1. Utilice el comando grep Linux para extraer la energía de la siguiente manera:
3. Opt-sits
- Inicie opt-sits: Establezca runtype 0 en el archivo de entrada. A continuación, inicie el programa QM4D escribiendo el comando como se muestra en el paso 2.2 para iniciar el paso de optimización.
-
Monitoreo de los cambios energéticos y nk valores.
- Trazar la propagación de energía con el programa de "gracia" y asegurarse de que la fluctuación de energía puede cubrir los extremos más bajos y más altos del rango de temperatura.
- Después de la optimización, guarde los valores nk finales del paso opt-sits en un nuevo archivo, que se denomina nk.dat en este protocolo.
4. Ejecución de simulaciones de producción
- Preparar el archivo de entrada MD: Establezca runtype 1 en el nuevo archivo de entrada para iniciar el paso de simulación de producción. Especifique el nombre de archivo del archivo nk almacenado como nkfile nk.dat en el archivo de entrada. El número de pasos de tiempo se estableció como 6.400.000 en los sistemas actuales.
-
Iniciar simulación MD de producción: Ejecute el siguiente comando para iniciar simulaciones MD: $INPUTFILE $PATH/qm4d > $OUTPUTFILE.
NOTA: Asegúrese de que el software QM4D puede leer los valores nk. El tiempo de simulación depende del sistema, así que cambie el paso de simulación en función de demandas específicas. Seleccione un número adecuado de pasos de tiempo para garantizar suficiente tiempo de simulación para su propio sistema. Este paso probablemente consume mucho tiempo, así que guarde los archivos de reinicio para evitar reiniciar la producción desde el principio una vez que se interrumpa.
5 Análisis de datos
-
Seguimiento de los cambios de distancia
- Supervise el proceso de formación y rotura de enlaces durante la fase de producción, utilice el comando grep para comprobar los cambios de distancia de H1-N1 y H1-N2 a lo largo del tiempo de simulación. La misma operación se puede llevar a cabo para H2-N3 y H2-N4. A continuación, trace la propagación de distancia utilizando el valor de distancia acumulado durante las simulaciones de producción.
-
Extraer coordenadas de reacción
- Extraiga las coordenadas de reacción y los términos de energía del archivo de salida de producción generado por el comando QM4D por grep:
grep 'dist 1' $OUTPUTFILE awk ''imprimir $5'' > distancia1;
grep 'ener0' $OUTPUTFILE > ener0. - Organice los datos en cuatro columnas: q1, q2, U0 y U' (U0 y U' son la energía normal de salida y la energía ponderada), y escríbalos en el archivo de datos en cada período de tiempo.
- Extraiga las coordenadas de reacción y los términos de energía del archivo de salida de producción generado por el comando QM4D por grep:
-
Cálculo de la energía libre
- Calcule la energía libre emitiendo el siguiente comando:
sits-pmf 300 $INPUTFILE PMF2 [hist_minx hist_maxx num_binsx] [hist_miny hist_maxy num_binsy] > $OUTPUTFILE.
NOTA: sits-pmf es el método de análisis basado en histograma. [hist_minx hist_maxx num_binsx] define el intervalo y el número de ubicaciones para la primera coordenada de reacción. Las coordenadas de reacción de la segunda se pueden establecer mediante [hist_miny hist_maxy num_binsy]. - Para proyectar la energía libre en el paisaje bidimensional, escriba el siguiente comando:
sits-pmf 300 h1-2d.dat PMF2 -0.6 0.6 24 2.45 4.25 36 > sits-pmf.out.
NOTA: Utilice un total de 24 bins y 36 bins para cubrir los cambios de distancia en dos coordenadas de reacción seleccionadas, q1 y q2, respectivamente. Guarde los datos PMF 2D para cada hidrógeno/deuterio en el archivo sits-pmf.out.
- Calcule la energía libre emitiendo el siguiente comando:
Resultados
El efecto de sustitución de deuterio único en el proceso de transferencia de doble protones en porficeno se examinó en el protocolo actual (Figura1). Se comprobó la energía potencial del subsistema QM y el agua durante el paso de preequilibrio y optimización para asegurarse de que la energía se ha ampliado a un rango de energía más amplio (Figura2). Los cambios representativos de distancia y ángulo (Figura3 y
Discusión
La estructura del porficeno se mostró en la Figura1. El potencial híbrido QM/MM de incrustación electrostática con el método SITS se utilizó para describir las reacciones químicas en el agua23,24. La transferencia de protones se produce dentro del porficeno3 y por lo tanto el porficeno se establece como región QM y el agua de recordatorio se establece como región MM. Aquí adoptamos DFTB/MIO como nues...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Esta investigación cuenta con el apoyo del Programa Nacional de Investigación y Desarrollo Clave de China (2017YFA0206801, 2018YFA0208600), la Fundación nacional de ciencias naturales de la provincia de Jiangsu y la Fundación Nacional de Ciencias Naturales de China (91645116). L.X es el Profesor Nombrado Por Zhong-Wu de la Universidad Tecnológica de Jiangsu. Los autores reconocen las sugerencias del Dr. Hao Hu y el Dr. Mingjun Yang.
Materiales
| Name | Company | Catalog Number | Comments |
| operating system | CentOS Linux release 6.0 | ||
| QM4D software | http://www.qm4d.info/ | in-house program | |
| Computer desktop | HP |
Referencias
- Waluk, J. Ground- and excited-state tautomerism in porphycenes. Accounts of Chemical Research. 39 (12), 945-952 (2006).
- Waluk, J. Spectroscopy and tautomerization studies of porphycenes. Chemical Reviews. 117 (4), 2447-2480 (2017).
- Kozlowski, P. M., Zgierski, M. Z., Baker, J. The inner-hydrogen migration and ground-state structure of porphycene. The Journal of Chemical Physics. 109 (14), 5905-5913 (1998).
- Yoshikawa, T., Sugawara, S., Takayanagi, T., Shiga, M., Tachikawa, M. Theoretical study on the mechanism of double proton transfer in porphycene by path-integral molecular dynamics simulations. Chemical Physics Letters. 496 (1-3), 14-19 (2010).
- Smedarchina, Z., Shibl, M. F., Kühn, O., Fernández-Ramos, A. The tautomerization dynamics of porphycene and its isotopomers - concerted versus stepwise mechanisms. Chemical Physics Letters. 436 (4-6), 314-321 (2007).
- Wolfsberg, M., Hook, W. A., Paneth, P., Rebelo, L. P. N. Isotope effects. The Chemical, Geological, and Bio Sciences. , (2009).
- Pietrzak, M., Shibl, M. F., Bröring, M., Kühn, O., Limbach, H. H. 1H/2H NMR studies of geometric H/D isotope effects on the coupled hydrogen bonds in porphycene derivatives. Journal of the American Chemical Society. 129 (2), 296-304 (2007).
- Yoshikawa, T., Sugawara, S., Takayanagi, T., Shiga, M., Tachikawa, M. Quantum tautomerization in porphycene and its isotopomers: Path-integral molecular dynamics simulations. Chemical Physics. 394 (1), 46-51 (2012).
- Ladenthin, J. N., et al. Force-induced tautomerization in a single molecule. Nature Chemistry. 8 (10), 935-940 (2016).
- Dellago, C., Bolhuis, P. G., Csajka, F. S., Chandler, D. Transition path sampling and the calculation of rate constants. The Journal of Chemical Physics. 108 (5), 1964-1977 (1998).
- Bolhuis, P. G., Chandler, D., Dellago, C., Geissler, P. L. Transition path sampling: Throwing ropes over rough mountain passes, in the dark. Annual Review of Physical Chemistry. 53 (1), 291-318 (2002).
- Torrie, G. M., Valleau, J. P. Nonphysical sampling distributions in monte carlo free-energy estimation: Umbrella sampling. Journal of Computational Physics. 23 (2), 187-199 (1977).
- Kästner, J. Umbrella sampling. Wiley Interdisciplinary Reviews: Computational Molecular Science. 1 (6), 932-942 (2011).
- Gao, Y. Q. An integrate-over-temperature approach for enhanced sampling. Journal of Chemical Physics. 128 (6), 064105 (2008).
- Yang, L., Gao, Y. Q. A selective integrated tempering method. Journal of Chemical Physics. 131 (21), 214109 (2009).
- Yang, M., Yang, L., Gao, Y., Hu, H. Combine umbrella sampling with integrated tempering method for efficient and accurate calculation of free energy changes of complex energy surface. The Journal of Chemical Physics. 141 (4), 044108 (2014).
- Yang, Y. I., Zhang, J., Che, X., Yang, L., Gao, Y. Q. Efficient sampling over rough energy landscapes with high barriers: A combination of metadynamics with integrated tempering sampling. The Journal of Chemical Physics. 144 (9), 094105 (2016).
- Xie, L., Shen, L., Chen, Z. N., Yang, M. Efficient free energy calculations by combining two complementary tempering sampling methods. The Journal of Chemical Physics. 146 (2), 024103 (2017).
- Xie, L., Cheng, H., Fang, D., Chen, Z. N., Yang, M. Enhanced QM/MM sampling for free energy calculation of chemical reactions: A case study of double proton transfer. The Journal of Chemical Physics. 150 (4), 044111 (2019).
- Hu, X., Hu, H., Yang, W. . QM4D: an integrated and versatile quantum mechanical/molecular mechanical simulation package (http://www.qm4d.info/). , (2016).
- Jorgensen, W. L., Chandrasekhar, J., Madura, J. D., Impey, R. W., Klein, M. L. Comparison of simple potential functions for simulating liquid water. The Journal of Chemical Physics. 79 (2), 926-935 (1983).
- Walewski, L., et al. Scc-dftb energy barriers for single and double proton transfer processes in the model molecular systems malonaldehyde and porphycene. International Journal of Quantum Chemistry. 106 (3), 636-640 (2006).
- Bakowies, D., Thiel, W. Hybrid models for combined quantum mechanical and molecular mechanical approaches. The Journal of Physical Chemistry. 100 (25), 10580-10594 (1996).
- Hu, H., Yang, W. Development and application of ab initio QM/MM methods for mechanistic simulation of reactions in solution and in enzymes. Journal of Molecular Structure: THEOCHEM. 898 (1-3), 17-30 (2009).
- Xie, L., Yang, M., Chen, Z. N. Understanding the entropic effect in chorismate mutase reaction catalyzed by isochorismate-pyruvate lyase from pseudomonas aeruginosa (PchB). Catalysis Science, Technology. 9 (4), 957-965 (2019).
- Shibl, M. F., Pietrzak, M., Limbach, H. H., Kühn, O. Geometric H/D isotope effects and cooperativity of the hydrogen bonds in porphycene. ChemPhysChem. 8 (2), 315-321 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados