Method Article
Identificación de campo de Matricaria chamomilla utilizando un sistema qPCR portátil
En este artículo
Resumen
Aquí se presenta un protocolo para la identificación de campo de Matricaria chamomilla utilizando un sistema portátil qPCR. Este protocolo fácil de realizar es ideal como método para confirmar la identidad de una especie botánica en lugares donde el acceso a equipos de laboratorio y experiencia es limitado, como granjas y almacenes.
Resumen
El control de calidad en los productos botánicos comienza con el suministro de materia prima. Tradicionalmente, la identificación botánica se realiza a través de la evaluación morfológica y métodos analíticos químicos. Sin embargo, la falta de disponibilidad de botánicos, especialmente en los últimos años, junto con la necesidad de mejorar el control de calidad para combatir las tensiones en la cadena de suministro provocadas por el aumento de la demanda de los consumidores y el cambio climático, requiere enfoques alternativos. El objetivo de este protocolo es facilitar la identificación de especies botánicas utilizando un sistema portátil qPCR sobre el terreno o en cualquier entorno, donde el acceso a equipos de laboratorio y experiencia es limitado. El ADN objetivo se amplifica utilizando qPCR a base de tinte, con ADN extraído de materiales de referencia botánicos que sirven como un control positivo. El ADN objetivo se identifica por su amplificación específica y la coincidencia de su pico de fusión con el control positivo. Se ha incluido una descripción detallada de los pasos y parámetros, desde la recopilación práctica de muestras de campo hasta la extracción de ADN, la amplificación de PCR, seguida de la interpretación de datos, para garantizar que los lectores puedan replicar este protocolo. Los resultados producidos se alinean con los métodos tradicionales de identificación botánica de laboratorio. El protocolo es fácil de realizar y rentable, lo que permite realizar pruebas de calidad en materias primas lo más cerca posible del punto de origen de la cadena de suministro.
Introducción
La práctica de usar productos botánicos para mantener y mejorar la salud se remonta a miles de años. Debido a las tensiones en la cadena de suministro provocadas por el aumento de la demanda de los consumidores1, prácticas de cosecha insostenibles y cambio climático2, la adulteración botánica se está convirtiendo en una preocupación creciente en la industria de alimentos y suplementos dietéticos3. La presencia de especies botánicas no declaradas o mal identificadas puede conducir a una eficacia reducida, o incluso problemas de seguridad. Por ejemplo, el cohosh negro (Actaea racemosa), utilizado para el tratamiento de las molestias premenstruales, puede sustituirse por una especie asiática de bajo precio con un apoyo de datos clínicos limitados para su eficacia4. En un caso más grave, la sustitución de Aristolochia fangchi por Stephania terandra en un estudio clínico para bajar de peso utilizando hierbas chinas condujo a nefrotoxicidad grave e insuficiencia renal en algunos participantes5,6. Las dos especies diferentes compartían un nombre común chino "Fang Ji". Estos casos ponen de relieve la necesidad de un control de calidad más estricto, empezando por la identificación de las materias primas7,preferiblemente lo más cerca posible del punto de origen de la cadena de suministro, para que los recursos puedan asignarse eficientemente al material de identidad correcta.
Se pueden utilizar una serie de enfoques ortogonales para la identificación botánica. Tradicionalmente, la identificación botánica se realiza a través de la evaluación morfológica8,9 y los métodos analíticos químicos10,11,12,13. La identificación morfológica se basa en diferencias en las características macroscópicas y microscópicas de los materiales vegetales si existen diferencias (Figura 1). Sin embargo, la falta de programas de formación sobre botánica clásica en los últimos años ha dado lugar a una escasez de expertos14,lo que hace que este enfoque sea poco práctico para el control de calidad rutinario. Su aplicación en materiales botánicos en polvo también es limitada. Los métodos analíticos químicos se utilizan ampliamente en farmacopeas y laboratorios, pero no son ideales para pruebas de campo debido al tamaño de instrumentos como la cromatografía de capa delgada de alto rendimiento (HPTLC), la cromatografía líquida de alto rendimiento (HPLC) y la espectroscopia de resonancia magnética nuclear (RMR)(Figura 2)y los requisitos ambientales. Recientemente, los métodos genómicos han surgido como una técnica alternativa para la autenticación y sustitución de especies botánicas y han demostrado ser eficientes y precisos. Los métodos genómicos explotan la alta fidelidad y especificidad de la información genética en materiales vegetales15,16,17,18,19. Las herramientas de diagnóstico molecular están disponibles en forma de dispositivos portátiles, y a menudo incluyen herramientas automatizadas de interpretación de datos que reducen la barrera al uso de la tecnología, por lo que este enfoque es ideal para la identificación de campo20,,21,,22,,23,,24. Una vez diseñado y validado el método de análisis molecular25,,26,,27,puede ser realizado por cualquier personal con formación básica en biología molecular. Entre las diferentes herramientas portátiles disponibles, la PCR en tiempo real en secuencias de ADN es una de las opciones rentables28. La combinación de un dispositivo portátil, junto con el análisis molecular personalizado y validado, permite la verificación de especies botánicas e ingredientes fuera del laboratorio, como en granjas y almacenes de materiales botánicos, reduciendo el tiempo y los costos asociados con los métodos tradicionales.
El objetivo de este protocolo es introducir un método de identificación botánica en situaciones en las que el acceso a los equipos de laboratorio y la experiencia es limitado o no está disponible, utilizando un sistema portátil qPCR. El método se demuestra en un campo de Matricaria chamomilla (Figura 3A), comúnmente conocida como manzanilla alemana, ampliamente utilizado por sus propiedades antiinflamatorias y antioxidantes29. Se puede confundir con especies relacionadasde apariencia u olor similar, especialmente de los géneros Chamaemelum, Tanacetumy Crisantemo30,,31,,32. Entre las especies relacionadas, Chamaemelum nobile, también conocida como manzanilla romana, se encuentra una notable con niveles de producción comparables en el comercio(Figura 3B). El método demostrado fue diseñado no sólo para identificar la especie botánica objetivo M. chamomilla,sino también detectar su pariente cercano, C. nobile, basado en la amplificación específica de secuencias de ADN.
Este artículo explica, en detalle, cómo realizar la identificación botánica de campo de M. chamomilla utilizando el análisis intercalado de qPCR basado en tinte y curva de fusión en un dispositivo portátil. El protocolo incluye la recolección de muestras botánicas del campo, la extracción de ADN in situ y la configuración de la reacción PCR en tiempo real. Para garantizar una conclusión válida, se utilizan como control positivo el ADN genómico botánico de destino M. chamomilla y el ADN genómico botánico no objetivo C. nobile, preextraído de materiales de referencia botánicos certificados. La especificidad de este método se demuestra mediante la realización de pruebas de identificación de M. chamomilla y C. nobile individualmente en muestras y controles. El control negativo no plantilla se utiliza para excluir los resultados falsos positivos causados por la contaminación por PCR.
Protocolo
1. Recolección de muestras
- Configure un área de prueba en el campo con una superficie plana y horizontal.
- Identificar una planta representativa que refleje las características de la mayoría de las plantas en el campo de la flor de manzanilla (Figura 4).
- Escoja una cabeza de flor de la planta representativa con guantes estériles.
- Coloque la muestra en un tubo de recogida de 2,0 ml.
- Repita los pasos 1.3 a 1.4 y recoja un prospecto (aproximadamente 0,5–0,7 cm de largo) de la misma planta.
NOTA: La flor y la hoja de M. chamomilla son lo suficientemente pequeñas como para sentarse en la parte inferior de un tubo de recolección de 2.0 ml. Para otros botánicos con mayor superficie, se puede utilizar un punzón de papel o tijeras (enjuagados en etanol 70% antes de su uso) para aislar muestras de tejido para análisis. Cuando se requiera un muestreo múltiple, enjuague el punzón o las tijeras de papel entre la manipulación de diferentes muestras.
2. Extracción de ADN
- Precalentar la incubadora de baño seco a 95oC.
- A cada tubo de recogida, añada 100 l de la solución de extracción del kit de extracción de ADN vegetal (incluido en Tabla de materiales). Para una mejor eficiencia de extracción de ADN, muele la muestra de tejido en la solución de extracción utilizando un tejido de pestle.
- Cierre el tubo. Asegúrese de que el tejido botánico esté cubierto con la solución de extracción durante todo el proceso de extracción.
- Colocar los tubos de recogida en una incubadora de baño seco precalentada e incubar los tubos de recogida a 95 oC durante 10 min.
- Después de 10 minutos, saque los tubos de la incubadora de baño seco.
- Añadir 100 l de la solución de dilución del mismo kit de extracción de ADN y mezclar la solución pipeteando hacia arriba y hacia abajo varias veces.
- Repita el mismo paso para la extracción de prospectos.
- Agitar para mezclar la solución aún más. El tejido vegetal generalmente no parece estar degradado después de este tratamiento. El color líquido puede cambiar y volverse turbio.
NOTA: La solución diluida se puede almacenar a temperatura ambiente durante la noche si no se procede inmediatamente. No es necesario eliminar los desechos celulares del tejido vegetal antes de almacenarlos. El líquido en los tubos contiene las plantillas de ADN para la amplificación de PCR aguas abajo.
3. Configuración de la reacción PCR
- Configure las condiciones de termociclismo del instrumento qPCR de acuerdo con las especificaciones del fabricante. Aplicar el perfil de termociclo PCR enumerado en (Tabla 1), que comienza con un paso de temperatura constante para la desnaturalización inicial, seguido de 25 ciclos de amplificación, y termina con rampa de temperatura para obtener una curva de fusión de alta resolución.
- Descongelar la mezcla maestra qPCR y las imprimaciones (Tabla 2) a temperatura ambiente antes de su uso.
- Planificar la reacción que se cargará en cada pozo: pozos que contengan control positivo con especies objetivo, control positivo con especies no objetivo, muestras y control negativo (Figura 5).
- En este ejemplo, se utilizan diez pozos: cinco para la prueba de identificación de la manzanilla alemana y los cinco restantes para la prueba de identificación de la manzanilla romana. Para cada tipo de prueba de identificación de especies, un pozo contiene un control positivo con ADN extraído del material de referencia de especies objetivo, un pozo contiene un control positivo con ADN extraído del material de referencia de especies no dirigidas, dos pozos están llenos de muestras de ADN de flores y hojas extraídas del campo, y un pozo se asigna para un control negativo. La Tabla 3 describe cada tipo de pozo.
- Preparar una mezcla maestra de reacción de acuerdo con el manual para cada prueba de identificación de especies botánicas. Una mezcla maestra de reacción típica contiene qPCR Master Mix universal (2x), imprimaciones específicas para especies directas y inversas y agua libre de nucleasas. La Tabla 4 enumera la composición del sistema de reacción.
NOTA: Si no se utiliza inmediatamente, almacene la mezcla maestra de reacción qPCR a +2 oC a +8 oC en un refrigerador o mini-nevera. - Mezclar a fondo la mezcla maestra de reacción por pipeteo antes de su uso.
- Coloque el cartucho qPCR boca arriba sobre una superficie plana y estable.
- Cargue 18 l de la mezcla maestra de reacción configurada en el paso 3.4 en los pomos del cartucho de acuerdo con los pozos definidos en el paso 3.3. Para esta demostración, agregue la mezcla maestra de reacción de prueba de identificación de manzanilla alemana en pozos etiquetados para la prueba GC (GCT en pozos 1, 3, 5, 7, 9) y la prueba de identificación de manzanilla romana master-mix en pozos etiquetados para la prueba RC (RCT en pozos 2, 4, 6, 8, 10) (ver Figura 5).
- Transfiera 2 l de ADN de muestra del sobrenadante de tubos de extracción de ADN y controles positivos de ADN preextraídos en pozos de cartucho precargados con mezcla maestra qPCR. Después de agregar cada plantilla de ADN a la mezcla maestra qPCR, mezcle suavemente la solución pipeteando.
NOTA: Evite los desechos celulares flotantes al transferir ADN desde el tubo de extracción de ADN. Utilice minicentrífuga para separar el sobrenadante y los desechos celulares, si es necesario. - Selle cuidadosamente el cartucho con película adhesiva. Cargue el cartucho en la cámara de termociclismo y ciérrelo.
- Ajuste el instrumento qPCR para que se ejecute.
Resultados
Siguiendo el protocolo descrito en la sección 1, el ADN botánico de la cabeza y la hoja de la flor se extrajeron en el sobrenadante después de la incubación térmica del tubo de recolección a 95 oC durante 10 min. En el estudio actual, el sobrenadante mostró un color amarillo y verdoso tanto para la flor como para la hoja, lo que indica que una variedad de compuestos naturales fueron liberados en el sobrenadante con ADN botánico(Figura 6). Aunque más tarde se logró una amplificación fiable de la PCR en triplicado para toda la plantilla de ADN extraído de campo, la evaluación de la calidad del ADN se realizó en el laboratorio como referencia. La concentración del extracto de ADN de la cabeza de la flor, determinada por la fluorometría, osciló entre 3,69–5,36 ng/l, mientras que la concentración de extracto de ADN de la hoja osciló entre 6,42–9,29 ng/l. Las proporciones de absorción A260/A280 y A260/A230 de extractos de ADN de flores y hojas se midieron mediante espectrofotometría. Sin embargo, debido a la superposición entre el ADN y el espectro de absorción UV fitoquímico, estas relaciones no se pudieron medir fiabilidad (datos no mostrados).
Se utilizó un tinte fluorescente intercalante para monitorear la amplificación de los fragmentos de destino en tiempo real. Dado que tanto las imprimaciones específicas M. chamomilla como C. nobile se dirigen a la región del espaciador interno transcrito 2 (ITS2), que tiene decenas a cientos de copias en el genoma de la planta, 25 ciclos de amplificación de PCR son suficientes para generar suficientes amplicons para la identificación de especies de manzanilla. En la Figura 7, el valor de Ct para el control positivo de M. chamomilla en la prueba de identificación de M. chamomilla fue inferior a 25 (GCP_GCT), mientras que después de 25 ciclos de amplificación, la fluorescencia del mismo control en la prueba de identificación C. nobile estaba por debajo del umbral de detección (GCP_RCT). Por otro lado, después de 25 ciclos, la fluorescencia para el control positivo de C. nobile en la prueba de identificación de M. chamomilla estaba por debajo del umbral de detección (RCP_GCT), mientras que el valor de Ct para el mismo control en la prueba de identificación C. nobile fue inferior a 25 (RCP_RCT). La amplificación de los controles positivos objetivo y no objetivo en sus respectivos ensayos demuestran la especificidad del ensayo de identificación M. chamomilla. Para el ADN de muestra, el extracto de ADN de la cabeza de la flor de campo y de la hoja produjo valores de Ct de 15,18 y 19,41 en la prueba de identificación de M. chamomilla, respectivamente (Sample1(FLOWER)_GCT y Sample2(LEAF)_GCT). Ambas muestras no se amplificaron en la prueba de identificación C. nobile (Sample1(FLOWER)_RCT y Sample2(LEAF)_RCT). Los patrones de amplificación de ambas muestras coincidieron con el patrón de amplificación del control positivo de M. chamomilla. Los controles negativos no se amplificaron tanto en las pruebas de identificación de M. chamomilla como en las de C. nobile (NC_GCT y NC_RCT), excluyendo la posibilidad de falsos positivos causados por la contaminación por PCR. Para confirmar aún más la amplificación específica en controles y muestras positivas, las fracciones del producto final de PCR de cada pozo se ejecutaron con gel de agarosa al 2% en el laboratorio(Figura 8). Para la prueba de identificación de M. chamomilla, ambas muestras de campo arrojaron amplicons corriendo en la misma posición que el control positivo M. chamomilla con un tamaño estimado ligeramente superior a 100 bp (tamaño teórico 102 bp). Para la prueba de identificación C. nobile, las especies no objetivo C. nobile positive control dieron lugar a una banda entre 50 y 100 bp, ajustando el tamaño teórico de 65 bp. El resto de los carriles no mostró ningún producto de amplificación específico, que estaba de acuerdo con la ausencia de señal fluorescente para estos pozos, como se observó en las pruebas de campo.
Después de la amplificación de PCR, se realizó un análisis de la curva de fusión para evaluar las características de disociación del ADN de doble cadena (dsDNA) durante el calentamiento. A medida que la temperatura aumentaba durante el ciclo final para el análisis de la curva de fusión, los aumentos de temperatura hicieron que los amplicons de doble hebra se disociaran. El tinte fluorescente intercalante se liberó gradualmente en la solución, disminuyendo la intensidad de la fluorescencia (Figura 9A). El punto de inflexión de la primera curva derivada se utilizó para determinar la temperatura de fusión (Tm) (Figura 9B), que depende principalmente de la longitud del fragmento de ADN y el contenido de GC. La combinación del valor ct con la temperatura de fusión puede aumentar la especificidad del análisis qPCR. En el estudio actual, el pico de temperatura de fusión de M. chamomilla positive control PCR amplicon se produjo a 85,6 oC (GCP_GCT) y era distinto del pico de temperatura de fusión de C. nobile positive control PCR amplicon a 79,1 oC (RCP_RCT). El amplícono PCR de la cabeza y la hoja de la flor de campo produjo picos de temperatura de fusión a 85,2 oC y 84,8 oC, respectivamente (Sample1(FLOWER)_GCT y Sample2(LEAF)_GCT). Para evaluar las variaciones de temperatura de fusión medidas por el sistema portátil qPCR, se recogieron puntos de datos adicionales para confirmar que las temperaturas de fusión de la muestra siempre estuvieron cerca de la temperatura de fusión obtenida del control positivo de M. chamomilla (dentro de 2 oC) y estaban lejos de la temperatura de fusión de C. no positivebile control amplicon (Figura 10). A veces se reportaron picos de temperatura de fusión en otros pozos. Sin embargo, sus valores de Ct no eran inferiores a 25 y los picos de temperatura de fusión no estaban cerca del control positivo de M. chamomilla o C. nobile (más de 2 oC de separación).
En resumen, la prueba de identificación del campo M. chamomilla puede interpretarse sobre la base de las reglas de decisión resumidas en el Cuadro 5. Con todos los controles positivos positivos positivos para las especies botánicas putativas, negativos para las otras especies, y controles negativos que dieron negativo, ambas muestras de campo se determinó que contienen M. chamomilla pero no C. nobile. Además, para alinear los resultados de las pruebas de campo con otras técnicas analíticas, las conclusiones sobre el terreno se confirmaron aún más mediante un método de codificación de barras de ADN25 previamente validado (datos no mostrados).

Figura 1: Identificación morfológica de materiales botánicos. (A) Flores Hibiscus rosa-sinensis, raíces de Longa de Curcuma, hojas de Malva Sylvestris, hojas de Rosmarinus officinalis, semillas de coriandrum sativum, raíces de cocina Zingiber. (B) Las escamas de Petroselinum crispum y Apium graveolens son difíciles de diferenciar. Haga clic aquí para ver una versión más grande de esta figura.
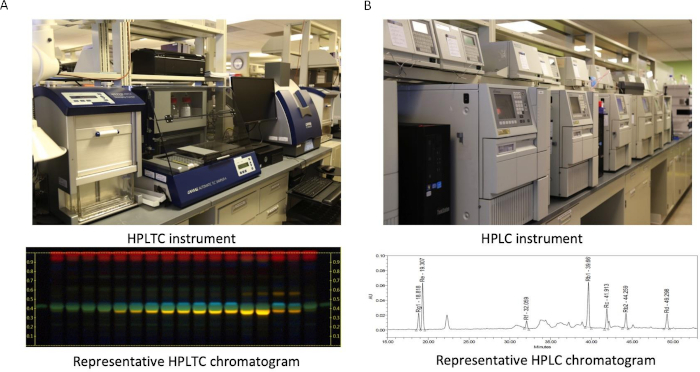
Figura 2: Identificación química de materiales botánicos. (A) HPTLC y un cromatograma HPTLC representativo. (B) HPLC y un cromatograma HPLC representativo. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Matricaria chamomilla y Chamaemelum nobile en el campo. (A) Matricaria chamomilla, adaptada de Wikipedia bajo CC BY-SA 3.0, https://en.wikipedia.org/wiki/Matricaria_chamomilla#/media/File:Matricaria_February_2008-1.jpg. (B) Chamaemelum nobile, adaptado de Wikipedia bajo CC BY-SA 3.0, https://en.wikipedia.org/wiki/Chamomile#/media/File:Chamaemelum_nobile_001.JPG. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Recolección de piezas vegetales de M. chamomilla del campo. Haga clic aquí para ver una versión más grande de esta figura.
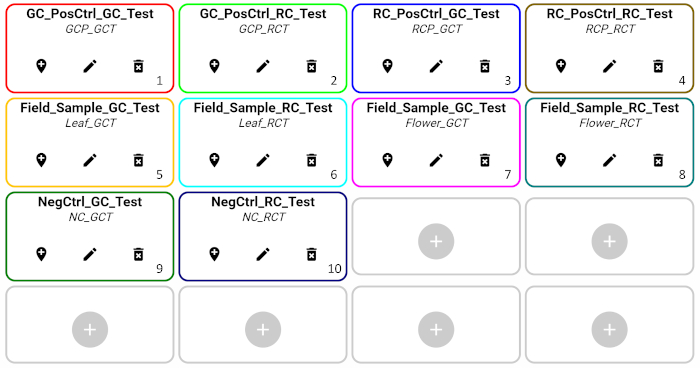
Figura 5: Diseño de pozos de prueba en la demostración. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Extracto de ADN de campo en tubos de recogida. El tejido botánico permanece en el tubo original y está cubierto por un extracto de ADN amarillento. Haga clic aquí para ver una versión más grande de esta figura.
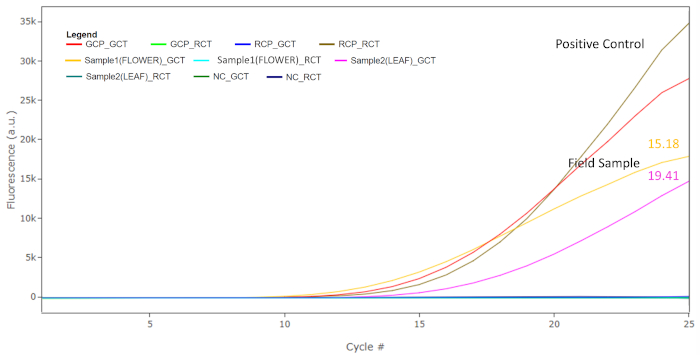
Figura 7: Gráfica de fluorescencia que muestra la acumulación de productos PCR en 25 ciclos de termociclismo. El control positivo de M. chamomilla y el control positivo C. nobile muestran valores de Ct inferiores a 25 en las pruebas de identificación M. chamomilla y C. nobile, respectivamente. Las muestras de flores y hojas de campo fueron amplificadas por la prueba de identificación de M. chamomilla con valores de Ct de 15,18 y 19,41. El resto de los pozos no se amplificaron. Haga clic aquí para ver una versión más grande de esta figura.
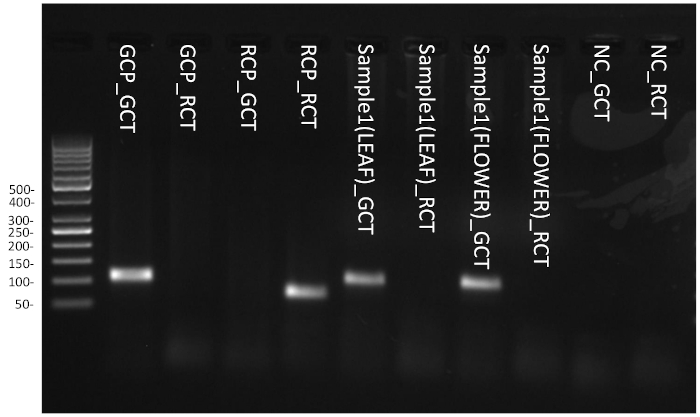
Figura 8: Electroforesis de gel de productos de amplificación de PCR de campo. Haga clic aquí para ver una versión más grande de esta figura.
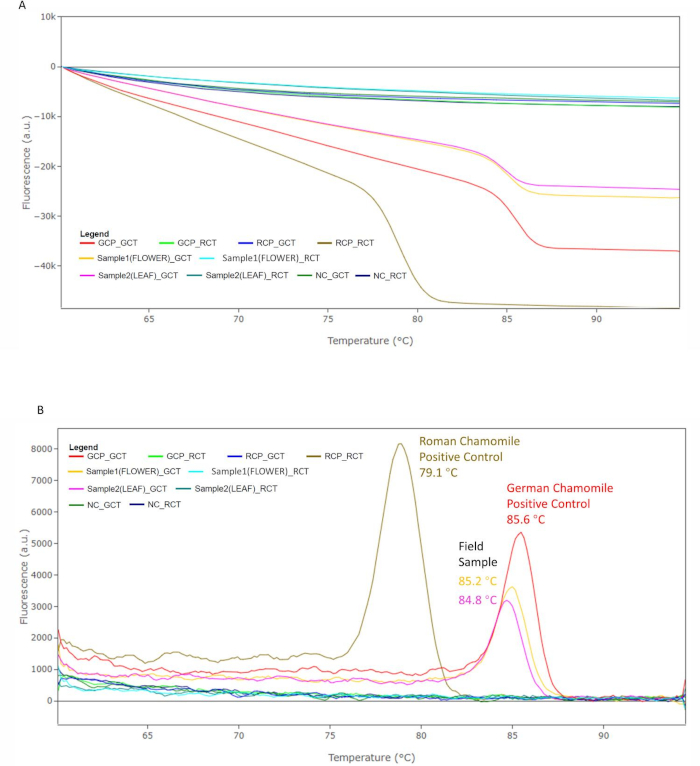
Figura 9: Análisis de temperatura de fusión. (A) La señal de fluorescencia en cada pozo disminuye con el aumento de la temperatura. (B) La identidad de los productos PCR se confirmó mediante el pico de temperatura de fusión en el análisis de la curva de fusión. Las muestras de flores y hojas de campo muestran picos a 85,2 oC y 84,8 oC. Estos están cerca del pico producido por M. chamomilla control positivo. El control positivo de C. nobile produjo un pico a 79,1 oC, que es diferente de las otras tres muestras. Haga clic aquí para ver una versión más grande de esta figura.
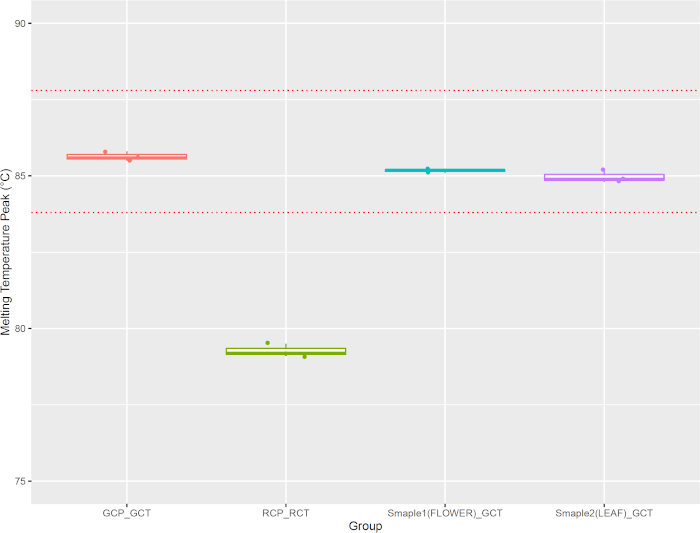
Figura 10: Variación máxima de temperatura de fusión entre el control positivo y las muestras de campo. Haga clic aquí para ver una versión más grande de esta figura.
| Etapa | Ciclo | Temperatura | Hora |
| Temperatura constante | 1 | 95 oC | 60s |
| Amplificación | 25 | 95 oC | 30s |
| 60 oC | 30s | ||
| Curva de fusión | 1 | 60 oC | Rampa 0,05 oC/s |
| 95 oC |
Tabla 1: condiciones de termociclemiento qPCR para las pruebas de identificación M. chamomilla y C. nobile.
| Ensayo | Primer nombre | Secuencia 5'-3' | Posición | Región | Tamaño del Amplicon | |
| Matricaria recutita | ZL3 | TCGTCGGTCGCAAGGATAAG | Adelante | ITS2 | 102 bp | |
| ZL4 | TAAACTCAGCGGGTAGTCCC | Marcha atrás | ||||
| Chamaemelum nobile | ZL11 | TGTCGCACGTTGCTAGGAAGCA | Adelante | ITS2 | 65 bp | |
| ZL12 | TCGAAGCGTCATCCTAAGACAAC | Marcha atrás | ||||
Tabla 2: Pares de imprimación para pruebas de identificación M. chamomilla y C. nobile.
| Bien posicionamiento | Bien nombre | Descripción | |
| 1 | GC_PosCtrl_GC_Test | Control positivo de la manzanilla alemana bajo prueba GC | |
| 2 | GC_PosCtrl_RC_Test | Control positivo de la manzanilla alemana bajo prueba RC | |
| 3 | RC_PosCtrl_GC_Test | Control positivo de la manzanilla romana bajo prueba DE GC | |
| 4 | RC_PosCtrl_RC_Test | Manzanilla romana control positivo bajo RC Test | |
| 5 | Field_Sample_GC_Test | Muestra de tejido de hoja bajo prueba de GC | |
| 6 | Field_Sample_RC_Test | Muestra de tejido de hoja bajo prueba RC | |
| 7 | Field_Sample_GC_Test | Muestra de tejido floral bajo prueba de GC | |
| 8 | Field_Sample_RC_Test | Muestra de tejido floral bajo prueba RC | |
| 9 | NegCtrl_GC_Test | Muestra de control negativo bajo prueba DE GC | |
| 10 | NegCtrl_RC_Test | Muestra de control negativo bajo rc Test | |
Tabla 3: Tipos de pozos y descripciones para las pruebas de identificación M. chamomilla y C. nobile.
| Reactivo | GC_Test | RC_Test |
| Mezcla universal qPCR* | 10 l | 10 l |
| Imprimación ZL3 (10 m) | 0,4 l | Na |
| Imprimación ZL4 (10 m) | 0,4 l | Na |
| Imprimación ZL11 (10 m) | Na | 0,4 l |
| Imprimación ZL12 (10 m) | Na | 0,4 l |
| H2O (sin nucleasas) | 7.2 L | 7.2 L |
| * contiene Hot Start Taq DNA Polymerase | ||
Tabla 4: Composición de mezcla maestra para M. chamomilla y C. nobile pruebas de identificación.
| Nombre del pozo | Resultado esperado | Criterios de Resultados Positivos | Criterios de Resultados Negativos |
| Detectado / Presente | No Detectado / Ausente | ||
| GC_PosCtrl_GC_Test | Detectado | Ct < 25 y 84 | - |
| GC_PosCtrl_RC_Test | No detectado | - | Sin valor de Ct dentro de 25 ciclos |
| RC_PosCtrl_GC_Test | No detectado | - | Sin valor de Ct dentro de 25 ciclos |
| RC_PosCtrl_RC_Test | Detectado | Ct < 25 y 79 | - |
| Field_Sample_Leaf_GC_Test | Presente | Ct < 25 y 84 | Sin valor de Ct dentro de 25 ciclos |
| Field_Sample_Leaf_RC_Test | Ausente | - | Sin valor de Ct dentro de 25 ciclos |
| Field_Sample_Flower_GC_Test | Presente | Ct < 25 y 84 | Sin valor de Ct dentro de 25 ciclos |
| Field_Sample_Flower_RC_Test | Ausente | - | Sin valor de Ct dentro de 25 ciclos |
| NegCtrl_GC_Test | No detectado | - | Sin valor de Ct dentro de 25 ciclos |
| NegCtrl_RC_Test | No detectado | - | Sin valor de Ct dentro de 25 ciclos |
Tabla 5: Reglas para la interpretación de los resultados de qPCR.
Discusión
El diseño de imprimaciones y la selección de plantillas son los pasos cruciales para obtener una amplificación qPCR eficiente y específica. Después de identificar una plantilla adecuada, el software de diseño de imprimación se utiliza normalmente para ayudar a la selección de imprimaciones basadas en variables de diseño como la longitud de imprimación, la temperatura de fusión y el contenido de GC33,,34. La optimización y validación se puede realizar en las condiciones experimentales esperadas del ensayo para garantizar la especificidad, sensibilidad y robustez de una reacción PCR35. El diseño de imprimación subóptimo puede dar lugar a la formación de imprimación-dimer, en la que las interacciones de imprimación producen productos no específicos36.
Los controles sin plantilla (NTC) utilizados en este estudio comprueban tanto la contaminación del ADN como la presencia de imprimadores que podrían afectar el ensayo. Los resultados no mostraron amplificación, una buena indicación de que tanto la contaminación del ADN como los dimers de imprimación no son una preocupación. La contaminación del ADN y los dimers de imprimación se manifiestan en curvas de fusión sin controles de plantilla, y como picos adicionales en curvas de fusión de controles positivos. Normalmente, se espera que la curva de fusión de un control positivo contenga un único pico, a menos que los subdominios ricos en AT de la plantilla causen un derretimiento desigual. Se podrían predecir picos dobles simulando ensayos de fusión utilizando el software uMELT37. En este estudio, el estándar de oro de la ejecución del producto PCR en gel de agarosa se utilizó para confirmar la presencia del producto de PCR objetivo y la ausencia de contaminación y imprimación-dimers.
Un desafío considerable en el análisis molecular de material botánico es obtener ADN de buena calidad tras el proceso de extracción de ADN botánico. Los materiales botánicos se comercializan y consumen por los compuestos químicos activos que se asocian con beneficios para la salud. En el proceso de extracción de ADN, estos compuestos químicos también se liberarán en la solución de extracción de ADN, lo que potencialmente causará inhibición de la PCR, lo que resulta en fallas en la amplificación de PCR. Se han desarrollado varios kits de purificación de ADN vegetal utilizando disolventes orgánicos y columnas para eliminar compuestos químicos derivados de los botánicos38. Sin embargo, la campana de humo y la centrífuga de alta velocidad necesaria para ayudar a estos kits no están disponibles en el campo.
En el protocolo actual, el método simplificado de extracción de ADN utiliza un kit de extracción de ADN de planta comercial (ver Tabla de materiales para más detalles). Tuvo la capacidad de neutralizar sustancias inhibidoras comunes para obtener resultados reproducibles y produjo resultados consistentes para M. chamomilla y C. nobile. Tanto M. chamomilla como C. nobile florecen cabezas y hojas dieron como productor de ampnados PCR con picos de fusión específicos, lo que indica que la presencia de inhibidores de PCR no era una preocupación. Para otras plantas con niveles más altos de inhibidores de PCR, amplificar el ADN en su extracción original puede ser menos eficiente. Para reducir la inhibición y mejorar la eficiencia de amplificación, con acceso a toda la planta, otras partes de la planta con menor contenido de polisacárido y polifenoles también se pueden utilizar con fines de identificación. Si hay un acceso limitado a diferentes partes de plantas, hojas más jóvenes o pétalos diseccionados de las cabezas de las flores, que típicamente tienen un menor contenido fenólico39,puede ofrecer una mejor oportunidad de éxito. Dado que las secuencias de ADN son consistentes en toda la planta, cualquier parte de la planta se puede utilizar para confirmar la identidad de la especie. Si la amplificación de PCR sigue siendo subóptima, el extracto de ADN original se puede diluir aún más antes de que se pueda utilizar pcR, o se pueden utilizar protocolos de purificación de laboratorio más sofisticados.
Otro desafío para el análisis de ITP son los resultados falsos positivos causados por la contaminación del ADN, que pueden afectar negativamente a la interpretación de los datos. Por lo general, se controla mediante limpieza activa, uso de equipos dedicados y restricción del trabajo a áreas designadas. Usando qPCR, todo el análisis de PCR se puede realizar en un sistema cerrado, lo que reduce en gran medida la posibilidad de contaminación por amperón pcR en un entorno que no está bien controlado. Además, el ADN ambiental tampoco debe mostrar un falso positivo debido a la especificidad del ensayo, según un estudio de validación anterior40.
Hay margen de mejora. En el protocolo presentado aquí, el tinte intercalante se utilizó para mostrar la amplificación del fragmento objetivo en tiempo real. La especificidad del método se confirma aún más por la temperatura de fusión característica, que es distinta entre M. chamomilla y C. nobile amplicons. Por lo tanto, la intercalación de PCR a base de tinte puede responder eficientemente a la pregunta "¿Qué es esta especie de planta?" en el campo. Sin embargo, además de la necesidad de realizar una identificación botánica en una sola planta aislada del campo, en muchas circunstancias, los polvos botánicos o extractos en el almacén también se beneficiarán de una evaluación rápida de la identidad in situ. Para estos tipos de materiales, es posible que sea necesario abordar preguntas adicionales, como "¿Qué hay en este material?", "¿Contiene la especie botánica que estoy buscando?", "¿Contiene adulterantes que quiero evitar?", y "¿Es sustituido total o parcialmente por otras especies botánicas que son dañinas?". En lugar de utilizar tinte intercalante, se pueden diseñar diferentes sondas qPCR para atacar amplicons de diferentes botánicos en un sistema de reacción, manteniendo al mismo tiempo una alta especificidad y eficiencia del ensayo. El desarrollo de qPCR basado en sondas y la utilización de un sistema portátil qPCR que ofrece detección de múltiples canales pueden ampliar aún más la aplicación de pruebas de campo como un ensayo apto para el propósito a un entorno más amplio, como almacenes de materiales botánicos y centros de distribución para responder preguntas más complicadas. Además, el uso de múltiples sondas también permite al usuario incluir amplificación interna en cada sistema de reacción, por lo que más información estará disponible cuando se sospeche la inhibición de la PCR.
El protocolo presentado aquí tiene las siguientes ventajas en comparación con las tecnologías existentes utilizadas para el mismo propósito. En primer lugar, para los métodos tradicionales de identificación morfológica y química, el procedimiento y sus resultados deben ser llevados a cabo e interpretados por expertos. las pruebas de identificación basadas en qPCR pueden ser realizadas por personas con formación básica en biología molecular e interpretadas de una manera más estandarizada. En segundo lugar, en comparación con la identificación y diferenciación de especies basadas en qPCR que normalmente se realizan en el laboratorio, el protocolo de identificación de campo que utiliza un instrumento portátil con una gran huella, como una centrífuga de alta velocidad, un equipo de evaluación de la calidad del ADN, un ciclo térmico con detector de fluorescencia y un ordenador que ejecute un software especial. Por lo tanto, la identificación de especies basadas en ADN se puede realizar en cualquier entorno sin demora. En tercer lugar, la búsqueda de materiales botánicos es una tarea que requiere una operación global. Con los avances en los servicios en la nube y la inteligencia artificial, un dispositivo portátil puede recibir potencialmente métodos desarrollados y validados por expertos en el laboratorio, ser operado por no expertos en áreas remotas y producir certificaciones objetivas de terceros. Por lo tanto, esta opción es más convincente que nunca con el trabajo remoto convirtiéndose en la tendencia.
En resumen, el protocolo aquí demostró la identificación de campo de M. chamomilla utilizando un sistema portátil qPCR. La aplicación exitosa de esta técnica generará resultados muy precisos en la identificación botánica y ayudará a los fabricantes y proveedores botánicos a calificar los materiales botánicos de manera oportuna y rentable.
Divulgaciones
Certificamos que Zhengxiu Yang, Zheng Quan, Tiffany Chua, Leo Li, Yanjun Zhang, Silva Babajanian, Tricia Chua, Peter Chang, Gary Swanson, Zhengfei Lu son empleados de Herbalife International of America, Inc. Certificamos que Francesco Buongiorno, Isabella Della Noce y Lorenzo Colombo son empleados de Hyris Ltd. que produce el instrumento portátil qPCR utilizado en este artículo.
Agradecimientos
Agradecemos a James Shan por sus esfuerzos en la grabación de video de campo. Agradecemos a Jon Byron y Matthew Semerau por su trabajo en la edición de vídeo. Agradecemos a Ansen Luo, Harry Du y Frank Deng por su apoyo en la localización del campo de prueba. Agradecemos a Maria Rubinsky por sus valiosos comentarios sobre todo el manuscrito. Todas las personas reconocidas son empleados de Herbalife International of America, Inc.
Materiales
| Name | Company | Catalog Number | Comments |
| Battery | TNE | 78000mAh | Provide field power supply |
| bCUBE | HYRIS | bCUBE 2.0 | Portable qPCR instrument |
| Cartridges(16 Well) | HYRIS | NA | Consumables for bCUBE |
| Electric pipette | Eppendorf | NA | Handling liquid |
| Extract-N-AmpTM plant PCR kit | SIGMA | XNAP2-1KT | Plant DNA extraction kit |
| German chamomile (Matricaria chamomilla L) flower botanical reference materials | AHP | 2264 | Used as positive control |
| Mini dry bath | Yooning | MiniH-100L | For DNA extraction |
| Nuclease-free water | AMBION | AM9937 | qPCR reaction |
| Primer | Thermo Fisher Scientific | NA | qPCR reaction |
| Roman chamomile (Chamaemelum nobile) flower botancial reference materials | ChromaDex | ASB-00030806-005 | Used as positive control |
| Luna universal qPCR master mix | NEB | M3003L | qPCR reaction |
Referencias
- Smith, T., Gillespie, M., Eckl, V., Knepper, J., Reynolds, C. Herbal supplement sales in US increase by 9.4% in 2018. HerbalGram. 123, 62-73 (2019).
- Israelsen, L. D. The challenge of regulation, globalization and climate change on botanicals and traditional medicines: respecting tradition while embracing change. Planta Medica. 74 (3), 36 (2008).
- Cardellina, J. H. Challenges and opportunities confronting the botanical dietary supplement industry. Journal of Natural Products. 65 (7), 1073-1084 (2002).
- Foster, S. Exploring the peripatetic maze of black cohosh adulteration: a review of the nomenclature, distribution, chemistry, market status, analytical methods and safety. HerbalGram. 98, 32-51 (2013).
- Vanherweghem, J. L. Misuse of herbal remedies: The case of an outbreak of terminal renal failure in Belgium (Chinese herbs nephropathy). The Journal of Alternative and Complementary Medicine. 4 (1), 9-13 (1998).
- Vanherweghem, J. L., et al. Rapidly progressive interstitial renal fibrosis in young women: association with slimming regimen including Chinese herbs. The Lancet. 341 (8842), 387-391 (1993).
- Khan, I. A., Smillie, T. Implementing a "quality by design" approach to assure the safety and integrity of botanical dietary supplements. Journal of Natural Products. 75 (9), 1665-1673 (2012).
- Applequist, W. . The Identification of Medicinal Plants: A Handbook of the Morphology of Botanicals in Commerce. , (2006).
- Upton, R., Graff, A., Jolliffe, G., Länger, R., Williamson, E. . American Herbal Pharmacopoeia: Botanical Pharmacognosy - Microscopic Characterization of Botanical Medicines. , (2016).
- Frommenwiler, D. A., et al. Comprehensive HPTLC fingerprinting for quality control of an herbal drug - the case of angelica gigas root. Planta Medica. 84 (6-7), 465-474 (2018).
- Heyman, H. M., Meyer, J. J. M. NMR-based metabolomics as a quality control tool for herbal products. South African Journal of Botany. 82, 21-32 (2012).
- Lazarowych, N. J., Pekos, P. Use of fingerprinting and marker compounds for identification and standardization of botanical drugs: strategies for applying pharmaceutical HPLC analysis to herbal products. Drug Information Journal. 32 (2), 497-512 (1998).
- Tankeu, S., et al. Hyperspectral imaging and support vector machine: a powerful combination to differentiate black cohosh (actaea racemosa) from other cohosh species. Planta Medica. 84 (6-7), 407-419 (2018).
- Rodman, J. E., Cody, J. H. The taxonomic impediment overcome: NSF's Partnerships for Enhancing Expertise in Taxonomy (PEET) as a model. Systematic Biology. 52 (3), 428-435 (2003).
- Chen, S., et al. A renaissance in herbal medicine identification: from morphology to DNA. Biotechnology Advances. 32 (7), 1237-1244 (2014).
- Li, X., et al. Plant DNA barcoding: from gene to genome. Biological Reviews. 90 (1), 157-166 (2015).
- Madesis, P., Ganopoulos, I., Sakaridis, I., Argiriou, A., Tsaftaris, A. Advances of DNA-based methods for tracing the botanical origin of food products. Food Research International. 60, 163-172 (2014).
- Newmaster, S. G., Ragupathy, S., Janovec, J. A botanical renaissance: state-of-the-art DNA bar coding facilitates an automated identification technology system for plants. International Journal of Computer Applications in Technology. 35 (1), 50-60 (2009).
- Parveen, I., Gafner, S., Techen, N., Murch, S. J., Khan, I. A. DNA barcoding for the identification of botanicals in herbal medicine and dietary supplements: strengths and limitations. Planta Medica. 82 (14), 1225-1235 (2016).
- Agrawal, N., Hassan, Y. A., Ugaz, V. M. A pocket-sized convective PCR thermocycler. Angewandte Chemie International Edition. 46 (23), 4316-4319 (2007).
- Almassian, D. R., Cockrell, L. M., Nelson, W. M. Portable nucleic acid thermocyclers. Chemical Society Reviews. 42 (22), 8769-8798 (2013).
- Benítez-Páez, A., Portune, K. J., Sanz, Y. Species-level resolution of 16S rRNA gene amplicons sequenced through the MinION portable nanopore sequencer. Gigascience. 5 (1), 4 (2016).
- Emanuel, P. A., et al. Detection of Francisella tularensis within infected mouse tissues by using a hand-held PCR thermocycler. Journal of Clinical Microbiology. 41 (2), 689-693 (2003).
- Quick, J., et al. Real-time, portable genome sequencing for Ebola surveillance. Nature. 530 (7589), 228 (2016).
- Lu, Z., et al. Single-laboratory validation of a two-tiered DNA barcoding method for raw botanical identification. Journal of AOAC International. 102 (5), 1435-1447 (2019).
- Sgamma, T., et al. DNA barcoding for industrial quality assurance. Planta Medica. 83 (14-15), 1117-1129 (2017).
- Wallinger, C., et al. Rapid plant identification using species-and group-specific primers targeting chloroplast DNA. PLoS One. 7 (1), 29473 (2012).
- Newmaster, S. G., et al. Recommendations for validation of real-time PCR methods for molecular diagnostic identification of botanicals. Journal of AOAC International. , (2019).
- Singh, O., Khanam, Z., Misra, N., Srivastava, M. K. Chamomile (Matricaria chamomilla L.): an overview. Pharmacognosy Reviews. 5 (9), 82 (2011).
- Mills, S. Y., Bone, K. The Essential Guide to Herbal Safety. Elsevier Health Sciences. , 325 (2004).
- Avula, B., et al. Quantitative determination of phenolic compounds by UHPLC-UV-MS and use of partial least-square discriminant analysis to differentiate chemo-types of Chamomile/Chrysanthemum flower heads. Journal of Pharmaceutical and Biomedical Analysis. 88, 278-288 (2014).
- Guzelmeric, E., Vovk, I., Yesilada, E. Development and validation of an HPTLC method for apigenin 7-O-glucoside in chamomile flowers and its application for fingerprint discrimination of chamomile-like materials. Journal of Pharmaceutical and Biomedical Analysis. 107, 108-118 (2015).
- Dieffenbach, C. W., Lowe, T. M., Dveksler, G. S. General concepts for PCR primer design. Genome Research. 3 (3), 30-37 (1993).
- Untergasser, A., et al. Primer3Plus, an enhanced web interface to Primer3. Nucleic Acids Research. 35, 71-74 (2007).
- Bustin, S., Huggett, J. qPCR primer design revisited. Biomolecular Detection and Quantification. 14, 19-28 (2017).
- Brownie, J., et al. The elimination of primer-dimer accumulation in PCR. Nucleic Acids Research. 25 (16), 3235-3241 (1997).
- Dwight, Z., Palais, R., Wittwer, C. T. uMELT: prediction of high-resolution melting curves and dynamic melting profiles of PCR products in a rich web application. Bioinformatics. 27 (7), 1019-1020 (2011).
- Heikrujam, J., Kishor, R., Mazumder, P. B. The chemistry behind plant DNA isolation protocols. Biochemical Analysis Tools - Methods for Bio-Molecules Studies. , (2020).
- Blum-Silva, C. H., Chaves, V. C., Schenkel, E. P., Coelho, G. C., Reginatto, F. H. The influence of leaf age on methylxanthines, total phenolic content, and free radical scavenging capacity of Ilex paraguariensis aqueous extracts. Revista Brasileira de Farmacognosia. 25 (1), 1-6 (2015).
- Lu, Z., et al. Validation of a targeted PCR method for raw and processed botanical material identification: an example using matricaria chamomilla (chamomile). Journal of AOAC International. 102 (6), 1787-1797 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados