Method Article
Visualisation et d'analyse de molécules d'ARNm par fluorescence
Dans cet article
Résumé
Ce protocole décrit une méthode expérimentale pour effectuer Fluorescence In situ Hybridation (FISH) pour compter ARNm dans des cellules individuelles à une résolution de molécule unique.
Résumé
La méthode hybridation fluorescente in situ (FISH) permet de détecter des acides nucléiques dans l'environnement cellulaire native. Ici nous fournissons un protocole pour l'utilisation de FISH pour quantifier le nombre d'ARNm dans les cellules de levure simples. Les cellules peuvent être cultivées dans n'importe quelle condition d'intérêt et ensuite fixées et rendues perméables. Par la suite, plusieurs désoxyoligonucléotides simple brin conjugués à des colorants fluorescents sont utilisés pour étiqueter et de visualiser ARNm. Fluorescence limité par diffraction à partir de molécules d'ARNm individuelles est quantifié en utilisant un algorithme de détection de point pour identifier et compter le nombre d'ARNm par cellule. Bien que les méthodes de quantification plus standard de taches Nord, RT-PCR et des microréseaux d'expression génique fournissent des informations sur les ARNm moyenne dans la population en vrac, FISH facilite à la fois le comptage et la localisation de ces ARNm dans des cellules individuelles à une résolution de molécule unique.
Introduction
En utilisant des techniques de mesure en vrac, il n'est pas possible de doser le nombre de transcrits ou de l'activité transcriptionnelle des cellules individuelles dans une. En utilisant des protéines fluorescentes commandés par des promoteurs d'intérêt que les journalistes de l'expression des gènes peut résoudre ce problème dans une certaine mesure, mais le temps nécessaire pour les protéines fluorescentes pour plier obscurcit début dynamique. Protéines fluorescentes à long terme peuvent aussi ne pas signaler durée de vie des ARNm. La méthode de FISH peut être utilisé pour l'ARNm d'essai durant son cycle de vie complet, de l'initiation de la transcription dans le noyau à la suite maturation et la dégradation dans des cellules individuelles, avec une résolution de molécule unique.
L'original expériences in situ pour la visualisation des acides nucléiques utilisés sondes d'ARN radiomarqués pour sonder des éléments d'ADN. Il s'agit notamment visualiser l'ADN ribosomique dans les ovaires de la grenouille Xenopus laevis 2 et l'ADN satellite dans les tissus de la souris 3. La première fluorescent dans exper situréalisation utilise une molécule d'ARN marqué avec un fluorophore pour sonder des séquences d'ADN particulières 4. La première application de sondes fluorescentes pour la visualisation de l'ARN in situ est la visualisation de l'expression du gène d'actine de poulet dans la culture de tissus du muscle 5. Plus récemment, dans la levure bourgeonnante, le poisson a été utilisé pour étudier les oscillations dans la transcription au cours du cycle métabolique de la levure 6, la désintégration des ARNm au cours de la progression du cycle cellulaire 7, et la localisation spatiale des transcrits d'ARNm lors de la mitose 8. FISH a été utilisé dans la levure de montrer que les fluctuations non corrélées dans les gènes transcrits de manière constitutive, qui constituent plus de la moitié des gènes de la levure, proviennent de l'initiation de la transcription non corrélé 9. Chez les espèces non-levure, le poisson a été utilisé pour identifier les marqueurs de cellules souches dans l'intestin de souris 10 et pour déterminer qui pénétrance incomplète du destin des cellules peut entraîner des fluctuations de l'expression des gènes stochastiques C.elegans embryons 11.
La méthode FISH décrit ici fonctionne par hybridation marquées par un colorant, des sondes d'ADN simple brin de l'ARNm messages. Les cellules sont imagés et ARNm sont comptées en utilisant un algorithme de détection de place. Sondes simple brin peuvent être générés avec un synthétiseur d'ADN et ensuite étiquetés (appelée ici comme sondes Singer) ou commandés dans le commerce en tant que sondes pré-marqués (sondes Stellaris) 12,13. Une différence majeure entre le chanteur et Stellaris sondes est que les sondes Singer sont plus longues (~ 50 pb) et sont multi-étiquetés, tandis que les sondes Stellaris sont courts (~ 20 bp) avec une seule étiquette par sonde, tel que décrit par Raj et al 14. En outre, l'approche Stellaris utilise beaucoup plus de sondes par gène que celle de Singer (~ 30 contre 5 sondes par gène, respectivement). Ci-dessous nous vous fournissons un protocole qui décrit l'utilisation de chaque type de sonde. Dans la section 2, nous présentons un protocole pour l'étiquetage des amino-allyle sondes thymidine contenant wvec un colorant Cy choisi. Un aperçu des étapes de calcul nécessaires pour identifier les points d'ARNm unique est prévu à la section 7.
Protocole
Les figures 1 et 2 sont des schémas des procédures expérimentales FISH et la canalisation de l'analyse d'images utilisés pour quantifier images de poissons.
1. Solutions à Préparer
* Les solutions ci-dessous sont pour une utilisation avec des sondes de chanteur. Si l'aide de sondes Stellaris, remplacer "40% de formamide» par «10% de formamide" en tant tampon d'hybridation et le tampon de lavage. D'autres modifications à la mémoire tampon d'hybridation lorsque l'aide de sondes Stellaris sont (1) ajouter 1 g Sulfate de Dextran et (2) ne comprend pas les 10 mg ssDNA.
Tampon B
8 ml 1 M KH 2 PO 4
41,5 ml 1M K 2 HPO 4
109,3 g Sorbitol
Tampon sphéroplastes
890 pi de tampon B
100 VRC ul
10 pl 25.000 U / ml lyticase
2 pl β-mercaptoéthanol
tampon d'hybridation (10 ml, le volume final)
10 mg E. coli ARNt
10 mg ssDNA *
100 pi mm stock 200 VRC
40 ul de 50 mg / ml de BSA
1 ml 20X SSC
4 ml 40% de formamide *
Nucléase libre l'eau (à 10 volume final ml)
1 g Sulfate de Dextran *
* Tampon d'hybridation peut être maintenue en portions de 0,5 ml à -20 ° C pour plus de commodité.
Tampon de lavage (ml, volume final 50)
5 ml 20X SSC
20 ml 40% de formamide *
Nucléase libre l'eau (à 50 volume final ml)
Étiquetage Buffer
1,06 g de carbonate de sodium
100 ml d'eau DEPC
pH 9
2. Probe-étiquetage (sondes Singer uniquement)
Nous obtenons ces sondes par synthèse en interne en utilisant un appareil de synthèse d'oligonucléotides ABI. Typicallié, 4-5 ~ 50 pb oligonucléotides sont synthétisés qui sont homologues du gène d'intérêt, en remplaçant la thymidine amino-allyl pour plusieurs thymidines espacées d'au moins 8, de préférence de 10 pb à part +. En raison de leur sensibilité à l'ozone, nous travaillons dans un établissement exempt d'ozone lors de l'utilisation de colorants CY.
- Obtenir ~ 5 sondes et remettre en suspension dans 100 pi d'eau - les concentrations de cocher sur Nanodrop.
- Selon le nombre de sondes / gène, combiner total de 10 ug / oligonucléotides gènes (par exemple si ont 5 sondes / gène, puis vouloir 2 pg / sonde).
- Utilisez les colonnes de Qiaquick pour purifier sondes selon QlAquick Nucleotide Protocole de lot d'enlèvement.
- Ajouter 10 volumes tampon PN pour le volume total de sondes et combiné mix.
- Appliquer l'échantillon à la colonne QIAquick - si le volume total est supérieur à 750 pi, ralentit deux fois en utilisant la moitié du volume dans chaque tour
- Laisser reposer pendant 1 min.
- Centrifuger 1 min à 6000 rpm.
- Laver avec 750 ul de tampon PE.
- Centrifugeuse1 min à 6000 rpm.
- Disposer d'écoulement à travers la colonne et re-spin pendant 1 min à 13000 rpm à sécher.
- colonne QIAquick de place dans le nouveau microtube et l'ADN élue avec 50 pl H 2 O - Assurez-vous que H 2 O pH est à 7,0 et 8,5 et est placé directement sur la membrane.
- Laisser reposer pendant 1 min.
- Centrifuger pendant 1 min à 13000 rpm pour éluer l'ADN.
- Lyophiliser l'ADN à 45 ° C.
- Remettre le culot dans un tampon de marquage 10 pi et d'ajouter au tube de colorant sans toucher le colorant.
- Répéter l'opération avec un autre tampon de marquage 10 pl.
- Vortex et centrifuger le tube de colorant et de l'ADN.
- Couvrir les tubes avec du papier d'aluminium et garder dans l'obscurité à température ambiante O / N à étiqueter.
- Répétez QlAquick Nucleotide Protocole de lot d'enlèvement. Effectuer avec deux différences:
- Ajouter tampon PN 200 pi de sondes et de mettre des sondes marquées à travers des colonnes 2x.
- Effectuer 3 lavages avec le tampon PE afin de se laver de tout colorant non fixé avant l'élution. - After élution obtenir concentration via NanoDrop. L'efficacité de l'étiquetage est typiquement ~ 0,25 pmol / ng d'ADN simple brin.
3. Lamelle Préparation
- Placer les lamelles sur des lames de plasma preen chambre à vide ( http://www.plasmapreen.com/ ) (le plus proche du centre, mieux c'est).
- Mettez chambre à vide au micro-ondes et assurez-vous qu'il est scellé.
- Mettre la pompe en premier, puis tourner à vide sur une seule fois la pompe est démarrée.
- Allumez le four micro-ondes, et d'arrêter 5 sec après plasma est visible.
- Éteignez vide puis pomper.
- Tirez chambre à vide et retirer des lamelles avec une pince (ceux qui sont tombés ont besoin d'être à nouveau nettoyé).
- La place des lamelles couvre-côté nettoyé dans des plaques à 12 puits.
4. Procédure de fixation
- Croître la levure à une DO600 d'environ 0,1-0,2 dans un milieu minimal. 10 ml de cellules donne assez for ~ 10 hybridations distinctes.
- Ajouter 1/10 du volume 37% de formaldéhyde directement sur le support de croissance (10 ml de culture + 1 ml de formaldéhyde 37%) et laisser reposer pendant 45 min.
- Laver 2x avec 1 ml de glace tampon froid B dans un tube à centrifuger (peut tourner à 13000 rpm pendant 1 min).
- Ajouter 1 ml de tampon sphéroplastes.
- Incuber à 37 ° C pendant 15 min. Vérifiez cellules toutes les quelques minutes sous le microscope jusqu'à ce que la plupart des cellules sont en noir (c'est à dire pas de phase lumineuse).
- Laver 2x avec de la glace tampon froid B, tournant à faible vitesse (~ 3500 rpm).
- Ajouter 1 ml de EtOH à 70%, remettre en suspension doucement et laisser une nuit à 4 ° C (peut stocker indéfiniment à -20 ° C).
5. Procédure d'hybridation
- Préparer la solution d'hybridation: à 100 pi de tampon d'hybridation, ajouter 1-3 pi de sonde, puis vortex et centrifuger. sondes Singer utilisent 8-10 totale ng par set sonde (ie par gène). Nous avons imagé jusqu'à 3 gènes simultanément avec 3 difsonde marquée rents fixe.
Assurez-vous de réchauffer la solution d'hybridation à la température ambiante avant de l'ouvrir.
Pour les sondes de Stellaris, il est recommandé de commencer 4 réactions d'hybridation séparées en ajoutant 1 pi de chacun de 1:10, 1:20, 1:50 et 1:100 dilutions de travail des sondes pour voir celui qui est optimal. Dilutions de travail des sondes Stellaris sont préparés dans un tampon d'hybridation.
- Centrifugeuse (pour toutes les étapes ultérieures; 3500 rpm, 5 min), les cellules fixes (par exemple 200 pi) et aspirer loin de l'éthanol.
- Doucement remettre en suspension dans un tampon de lavage 1 ml qui contient le même pourcentage de formamide dans le tampon d'hybridation. Laisser reposer pendant 2-5 min.
- Centrifuger l'échantillon et le tampon de lavage aspiration, puis ajouter la solution d'hybridation. Incuber dans l'obscurité sous agitation douce, O / N à 37 ° C.
Remarque: la procédure suivante est d'application cellules / d'imagerie sur des lamelles. Pour wincinération / imagerie de cellules dans des plaques à 96 puits, y compris la solution de rechange réactive de l'oxygène de l'espèce au trésor voir http://www.biosearchtech.com/stellarisprotocols.
- Le lendemain, ou à l'avance, propre (voir procédure) et de traiter les lamelles avec 150 pi 0,01% de poly-L-lysine pendant 5 min. Aspirer, laisser sécher, laver 3x avec dH 2 O et laisser sécher.
- Dans la matinée, ajouter 1 ml de tampon de lavage de l'échantillon, remettre en suspension doucement, centrifugeuse et aspirer, puis remettre en suspension dans un autre 1 ml de tampon de lavage et incuber à 37 ° C pendant 30 min.
- Laver avec 2X SSC + 0,1% de Triton X-100 à TA 15 min sur agitateur.
- Prenez milieu de montage du congélateur pour lui permettre de venir à la TA avant le montage.
- Laver avec 1X SSC à température ambiante 15 min sur agitateur.
- Diluer DAPI dans PBS (0,1 pg / ml final) et remettre les cellules dans 150 pi.
- Lieu solution sur nettoyé / poly-Lys-traitéslamelles TED dans la plaque de 12 puits; au moins 30 min intacte.
- Retirez la solution (vous pouvez la placer sur une lamelle de rechange comme une sauvegarde) et laver 3x avec 1 ml de PBS 1X.
- Placez 3 pi de prolonger milieu de montage or sur une lame (Invitrogen P36934). Pour ce faire, un à la fois si vous avez plusieurs lamelles pour éviter le dessèchement du milieu de montage.
- Ajouter ~ 0,5 ml d'éthanol à lamelle en plaque 12 puits, enlever la lamelle et l'air sec tout en maintenant avec des pincettes.
- Placez les lamelles à côté cellule vers le bas sur un milieu de montage et laisser durcir plusieurs heures ou O / N dans l'obscurité.
- Sceller les bords avec le vernis à ongles et de procéder à l'imagerie.
6. Imagerie des cellules avec Olympus IX-81 inversé Présentation du microscope
- Pour l'acquisition d'image, nous utilisons le logiciel Slidebook (intelligente imaging.com) et un, 1,45 NA, TIRFM objectif d'huile 100X. Pour GFP série, DAPI, Cy3, Cy5 Cy3.5 et imagerie: jeux de filtres Chroma (voir réactifs).
- Utiliser un filtre DAPIde trouver et de concentrer les cellules.
- Définissez ce que le point de référence.
- Jetez un z-pile d'images autour du point de référence lorsque la distance totale est de 5 um avec 0,2 Taille de l'iM (25 avions). Répétez l'opération pour chaque canal à colorant (ie fixe chaque sonde).
- Exporter au format TIFF 16 bits pour l'analyse d'image.
7. Analyse Aperçu de l'image (sondes Singer)
Ci-dessous nous donnent un aperçu des méthodes de calcul que nous utilisons pour l'analyse des images de poissons dans MATLAB. Les fonctions MATLAB pertinentes utilisées sont regroupées sur la droite. Les algorithmes et les seuils sont actuellement accordées pour les données des sondes de style de chanteur. En utilisant des sondes de style Stellaris nécessitera une certaine adaptation, en particulier à l'étape de filtrage final (7.8).
Identification des cellules 15
- Des cellules séparées de fond en utilisant un seuil global sur DAPI images 16 [graythresh].
- Identifier les noyaux en utilisant la fonction maxima prolongée [imextendedmax].
- cellules segment en utilisant des noyaux sous forme de graines pour un algorithme bassin versant [bassin].
Trouver des sites dans chaque canal de fluorescence
- Effectuer transformation de tophat de normaliser fond et améliorer le rapport signal sur bruit [bwmorph].
- Trouver maxima pour identifier la couche mise au point correcte (dans le plan z) pour chaque spot [imregionalmax].
- Filtrer spots potentiels en utilisant un modèle de régression linéaire pour un dégradé radial.
Mesure intensité de la tache et filtrer les signaux simples vs multiple sonde
- Monter un profil gaussien 2D à la place de la couche en-focus préalablement identifiés et l'intensité estimation 17.
- Filtrer les points faibles à l'aide d'un seuil (basé sur l'histogramme). Pour les sondes de type Singer cela est important, mais l'est moins pour les sondes de Stellaris.
- Comptez points dans chaque cellule.
Résultats
La figure 3 montre les histogrammes typiques calculés à partir des images de poissons et utilisée pour déterminer le nombre d'ARNm présents dans des cellules individuelles. Un avantage important de microscopie basée sur la quantification d'ARN est que l'on peut obtenir des informations sur la localisation des transcriptions. Par exemple, nous avons utilisé FISH pour identifier les ARNm dans des cellules individuelles avec une inductible CBF1 allèle (figure 4). Parce que de nombreuses molécules d'ARNm sont présents sur le site de transcription, nous sommes en mesure d'identifier la présence et la localisation des sites de transcription dans le noyau.
En utilisant différents colorants d'ARNm d'étiquettes de gènes différents, on peut quantifier plusieurs espèces d'ARNm dans les mêmes cellules. Pour démontrer cela, les cellules de levure ont été incubées en présence de α-facteur et le sorbitol. FUS1 transcription (colorant Quasar670, rouge) est induite par α-facteur. Transcription STL1 (Quasar 570 colorant, vert) est induite par l'augmentation de l'osmolarité extracellulaire ( Figure 5). Figure 4 est un exemple de FISH avec des sondes de chanteur. Figure 5 est un exemple de FISH avec des sondes Stellaris.
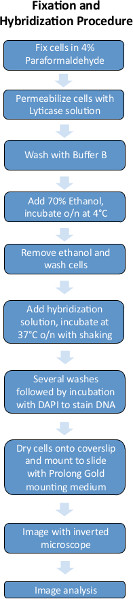
Figure 1. Schéma de la procédure expérimentale de poissons. Cliquez ici pour agrandir la figure .

Figure 2. Schéma de l'image pipeline d'analyse. Les dernières places sont déterminées dans la figure à droite.
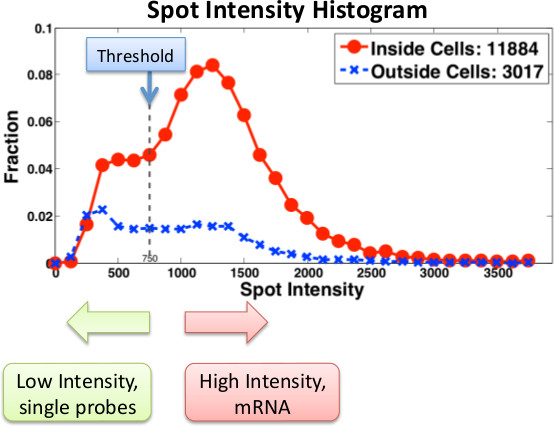
Figure 3. Histogramme des intensités de place pour un gène particulier utilisant des sondes Singer . intensités de sonde sont calculées à l'intérieur (rouge) et l'extérieur des cellules (en bleu). Taches de faible intensité sont soit le bruit ou les sondes simples. Messages d'ARNm réels sont étiquetés avec des sondes multiples. En utilisant les sondes de Stellaris, sondes simples sont moins détectables et donc les seuils et le filtrage doivent être ajustées en conséquence.
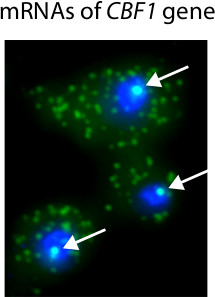
Figure 4. Les résultats représentatifs de la procédure FISH Singer. Dans cette expérience, CBF1 transcription est activée par un promoteur inductible 18. Les noyaux sont colorés en bleu avec DAPI. ARNm CBF1 sont marquées avec des sondes Cy3-étiquetés. Les flèches blanches mettent en évidence la présence de CBF1 sites de transcription dans le noyau. Transcrits d'ARNm simples sont visibles dans le cytoplasme.
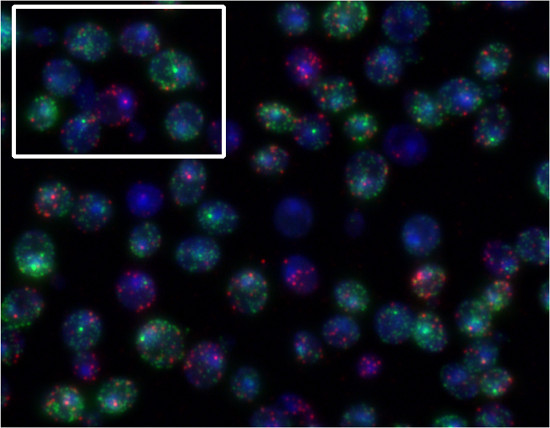
Figure 5. Les résultats représentatifs de la procédure FISH Stellaris. Cellules de levure MATa ont été simultanément exposés à 30 ng / ml α-facteur et 0,75 M sorbitol pendant 10 minutes et ont été simultanément sondé pour FUS1 (Quasar 670, rouge) et STL1 (Quasar 570, vert) transcriptions. Dans la zone en surbrillance, nous pouvons voir une cellule de répondre seulement à la phéromone (FUS1 site de départ, rouge) et un autre répondant principalement au sorbitol (site de départ STL1, vert).
Discussion
À ce jour, le poisson est principalement été une méthode à faible débit. L'utilisation de Cy3, Cy3.5 et Cy5 limite le nombre de gènes, on peut étudier dans des cellules individuelles à trois à la fois. Certaines sondes supplémentaires ont été développés (Stellaris), mais le nombre de sondes distinctes est toujours au plus sept ans. Pour contourner cette limitation, les stratégies de marquage combinatoire utilisant plusieurs fluorophores ont été utilisées pour créer des codes barres pour les différentes espèces d'ARNm 19,20. Plus récemment, Lubeck et Cai utilisés codes-barres optique et spectrale à quantifier 32 espèces différentes simultanément avec des poissons dans des cellules de levure unique 19. Une des limites de cette approche combinatoire récent est qu'elle nécessite l'utilisation de la microscopie de super-résolution. L'analyse nécessaire pour distinguer les sondes code à barres est également très complexe.
Nous avons constaté que Cy3 et Cy3.5 sont préférables à Cy5 pour les expériences de poisson. Une des limitations du colorant Cy5 est sa sensibilitéau photoblanchiment. Cependant, Stellaris a récemment développé Cy5 variantes qui sont annoncés comme plus résistant au photoblanchiment, et peut atténuer ce problème technique. C'est également intéressant de noter que le poisson est une méthode coûteuse à mettre en œuvre et que les deux chanteur et Stellaris sondes coûtent 700 $ - 1000 $ par ensemble de sondes, bien que les prix pour les sondes disponibles sur le marché devraient diminuer à l'avenir. Sobre de réactifs et d'étiquetage efficace apporte des sondes de chanteur jusque dans la gamme inférieure dans le prix.
L'un des principaux défis techniques est la séparation des spots de sondes simples ou multiples, ce qui nécessite la mise en œuvre des algorithmes sophistiqués place déterminants. Cela peut prendre un examen approfondi du manuel pour régler les paramètres d'analyse d'images pour des montages expérimentaux spécifiques. Un aperçu de notre pipeline de calcul avec des fonctions MATLAB concernés est prévue à l'article 7 du protocole. Cette question est quelque peu atténué par les sondes Stellaris qui ont seulement une étiquette par sonde. Elle nécessite donc la colocalisation de plusieurs sondes pour voir un signal.
Parce que le poisson nécessite cellules de fixation, il ne facilite pas le suivi des cellules individuelles au fil du temps. Auparavant, nous avons utilisé les données d'instantané de poisson pour reconstruire la dynamique de l'expression des gènes dans les populations de levure métaboliquement cyclistes individuels 6. Cyclisme métabolique est observée en pré-affamés, cultures continues, et se caractérise par des oscillations collectives ensemble de la population dans la consommation d'oxygène. Ces oscillations sont associés à l'échelle du génome oscillations de transcriptions qui se produisent pour la moitié des gènes de la levure à différentes phases de la consommation d'oxygène. Nous avons cherché à déterminer si le cyclisme métabolique était présente dans les cultures de levure en continu non synchronisées. Le cas échéant, relevés de notes qui sont anti-corrélés au sein des populations synchrones devraient également être anti-corrélés dans des cellules individuelles non synchronisés, et vice versa pour les transcriptions corrélés.
ent "> Pour reconstruire la dynamique de la production d'ARNm dans le temps, les données de l'instantané observées doit être comparée à ce qui est attendu d'un modèle de comportement sous-jacent. Il ya des limites théoriques lorsque ces« instantanés »des données d'expression de gènes peuvent être utilisés pour déterminer la dynamique de l'expression des gènes sous-jacents et quels types de modèles peuvent être distingués 21. Pour les données sur le cycle du métabolisme, plutôt que de montrer directement la présence d'oscillations temporelles, des mesures statistiques ont été mises en œuvre à l'appui qu'il ya effectivement un programme oscillatoire autonome cellulaire compatible avec puce vrac mesures.Déclarations de divulgation
Les auteurs déclarent qu'ils n'ont aucun intérêt financier concurrents.
Remerciements
Cette recherche a été financée par des subventions GM046406 (à DB) et par l'Institut national des sciences médicales générales Centre pour Quantitative Biology (GM071508). RSM reconnaît financement de la NSF Graduate Research Fellowship. MNM est pris en charge par un Lewis-Sigler Fellowship. Nous tenons à remercier les membres du laboratoire Botstein pour des discussions utiles et les anciens membres Allegra Petti et Nikolai Slavov pour leurs contributions au projet de cycle métabolique. Nous remercions Daniel Zenklusen et Robert Singer pour obtenir nous avons commencé avec la méthode FISH.
matériels
| Name | Company | Catalog Number | Comments |
| Vanadyl Ribonucleoside Complex | NEB | S1402S | |
| Lyticase | Sigma | L5263 | |
| E. coli tRNA | Roche | 1010954001 | |
| BSA (RNase free) | Ambion | ||
| Beta-mercapt–thanol | Fisher | 03446l | |
| DAPI, dilactate | Sigma | D9564 | |
| PBS 10X (RNase free) | Ambion | AM9624 | |
| Triton X-100 | Shelton Scientific | ||
| Dextran sulfate | Sigma | D6001 | Or equivalent |
| Saline-sodium citrate (SSC) 20X | VWR | 82021-484 | |
| Formamide (deionized) | Ambion | AM9342 | |
| Nuclease-free water | Ambion | AM9932 | |
| Alpha-D-glucose | Sigma | 158968 | For GLOX solution |
| 1 M Tris-HCl, pH 8.0 | Ambion | AM9855G | |
| 100% Ethanol | |||
| Glucose oxidase | Sigma | G0543 | For GLOX solution |
| Catalase | Sigma | C3155 | For GLOX solution |
| Concanavalin A | MP Biomedicals | 150710 | |
| Polylysine (0.01%) | Sigma | P8920 | |
| Coverslips | Warner Instruments | Cs-18R15 | |
| Prolong Gold Mounting Medium | Invitrogen | P36934 | |
| QIAquick Nucleotide Removal Kit | QIAGEN | 28304 | |
| FISH Probes | Biosearch Technologies | Custom order for your desired mRNA sequence | |
| Glass bottom 96-well plates | Nunc | 265300 | Alternative to coverslips |
| 12-well plates | BD Falcon | 351143 | |
| Cy3, Cy3.5, Cy5 dyes | GE Healthcare | monofunctional NHS-ester | |
| EQUIPMENT | |||
| Plasma-Preen I Cleaner | Terra Universal | 9505-00 | Controller (Cat #9505-17 optional) |
| Vacuum Pump | Alcatel | 205SDMLAM | For operating Plasma-Preen |
| Widefield Fluorescence Microscope | Olympus | IX81 | Or equivalent |
| 100X objective | Olympus | 1-UB617R | |
| Light Source | X-Cite | XCT 10-A | Or equivalent |
| Filter Sets | Chroma | U-NSP100V2-SPR, U-NSP101V2-SPR, U-NSP102V2-SPR, U-NSP103V2-SPR,U-NSP104V2-SPR. | |
| Cooled CCD or EMCCD Camera | Hamamatsu | C4742-98-24ER | |
Références
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of single RNA transcripts in situ. Science. 280, 585-590 (1998).
- Gall, J. G., Pardue, M. L. Formation and Detection of Rna-DNA Hybrid Molecules in Cytological Preparations. Proceedings of the National Academy of Sciences of the United States of America. 63, 378 (1969).
- Jones, K. W. Chromosomal and Nuclear Location of Mouse Satellite DNA in Individual Cells. Nature. 225, 912 (1970).
- Bauman, J. G., Wiegant, J., Borst, P., van Duijn, P. A new method for fluorescence microscopical localization of specific DNA sequences by in situ hybridization of fluorochromelabelled RNA. Exp Cell Res. 128, 485-490 (1980).
- Singer, R. H., Ward, D. C. Actin gene expression visualized in chicken muscle tissue culture by using in situ hybridization with a biotinated nucleotide analog. Proc. Natl. Acad. Sci. U.S.A. 79, 7331-7335 (1982).
- Silverman, S. J., et al. Metabolic cycling in single yeast cells from unsynchronized steady-state populations limited on glucose or phosphate. Proc. Natl. Acad. Sci. U.S.A. 107, 6946-6951 (2010).
- Trcek, T., Larson, D. R., Moldon, A., Query, C. C., Singer, R. H. Single-molecule mRNA decay measurements reveal promoter- regulated mRNA stability in yeast. Cell. 147, 1484-1497 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Mol Cell. 2, 437-445 (1998).
- Gandhi, S. J., Zenklusen, D., Lionnet, T., Singer, R. H. Transcription of functionally related constitutive genes is not coordinated. Nat. Struct. Mol. Biol. 18, 27-34 (2011).
- Itzkovitz, S., et al. Single-molecule transcript counting of stem-cell markers in the mouse intestine. Nature Cell Biology. 14, 106-U193 (2012).
- Raj, A., Rifkin, S. A., Andersen, E., van Oudenaarden, A. Variability in gene expression underlies incomplete penetrance. Nature. 463, 913-U984 (2010).
- Raj, A., Tyagi, S. Detection of individual endogenous RNA transcripts in situ using multiple singly labeled probes. Methods Enzymol. 472 (10), 365-386 (2010).
- Trcek, T., et al. Single-mRNA counting using fluorescent in situ hybridization in budding yeast. Nature Protocols. 7, 408-419 (2012).
- Raj, A., vanden Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5, 877-879 (2008).
- Otsu, N. A Tlreshold Selection Method from Gray-Level Histograms. IEEE Transactions on Systems, Man and Cybernetics. 9, 62-66 (1979).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophys J. 82, 2775-2783 (2002).
- McIsaac, R. S., et al. Fast-acting and nearly gratuitous induction of gene expression and protein depletion in Saccharomyces cerevisiae. Molecular biology of the cell. 22, 4447-4459 (2011).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nature Methods. 9, 743-U159 (2012).
- Levsky, J. M., Shenoy, S. M., Pezo, R. C., Singer, R. H. Single-cell gene expression profiling. Science. 297, 836-840 (2002).
- Wyart, M., Botstein, D., Wingreen, N. S. Evaluating Gene Expression Dynamics Using Pairwise RNA FISH Data. Plos Computational Biology. 6, (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon