Method Article
Système: Drug Repurposing Hypothesis Generation Utilisation de la "Drogues fines RE"
Dans cet article
Résumé
Here we describe a protocol using the web-based drug repurposing hypothesis generation tool: "RE:fine Drugs." This protocol can be modified to a user's preferences at the level of the query type (gene, drug or disease) and/or the range of available advanced options.
Résumé
The promise of drug repurposing is that existing drugs may be used for new disease indications in order to curb the high costs and time for approval. The goal of computational methods for drug repurposing is to enable solutions for safer, cheaper and faster drug discovery. Towards this end, we developed a novel method that integrates genetic and clinical phenotype data from large-scale GWAS and PheWAS studies with detailed drug information on the concept of transitive Drug-Gene-Disease triads. We created "RE:fine Drugs," a freely available, interactive dashboard that automates gene, disease and drug-based searches to identify drug repurposing candidates. This web-based tool supports a user-friendly interface that includes an array of advanced search and export options. Results can be prioritized in a variety of ways, including but not limited to, biomedical literature support, strength and statistical significance of GWAS and/or PheWAS associations, disease indications and molecular drug targets. Here we provide a protocol that illustrates the functionalities available in the "RE:fine Drugs" system and explores the different advanced options through a case study.
Introduction
Les processus coûteux et inefficaces associés aux approches de découverte de médicaments traditionnels, y compris les médicaments à haut débit et de criblage de composés de plomb, contribuent à des retards dans la traduction des découvertes de la recherche en thérapies pour les patients 1,2. Une moyenne de 1 milliard de dollars américains et 15-20 ans est nécessaire pour mettre un nouveau médicament sur le banc au chevet 3. En outre, 52% des médicaments échouent au cours du développement dans 1 des essais cliniques de phase, et seulement 25% des composés qui entrent dans la phase 2 de procéder en phase complète 3 études cliniques 4. L'objectif de réorientation de la drogue ou de repositionnement de médicaments est de renouveler drogue a échoué et / ou trouver de nouvelles indications pour des médicaments approuvés afin d'offrir de nouvelles thérapies pour les patients plus rapidement et avec un taux de réussite plus élevé. Reformatage de drogue peut diminuer le délai pour rendre les médicaments disponibles pour une utilisation chez les patients à 3-12 ans 5. applications médicales importantes pour la drogue repurposing comprennent: les maladies avec prognos pauvresest et le faible taux de survie, les maladies résistantes aux médicaments, les zones de recherche sur les maladies sous-financés et les populations de patients pauvres et mal desservies.
Computational reformatage de médicament est définie comme le processus de conception et de validation des flux de travail automatisés qui peuvent générer des hypothèses pour de nouvelles indications pour un médicament candidat 6. méthodes médicamenteuses repurposing informatiques existantes ont été classées, basée sur un réseau basé sur les signatures de la connaissance fondée sur des objectifs, et sur la base ciblée mécanisme, et peut être orienté de gènes, la maladie ou la drogue perspectives. En outre, les approches de calcul peuvent accélérer encore des expériences de validation de concept de validation et des études cliniques à petite échelle pour les médicaments candidats réutilisés 7. Nous avons précédemment rapporté sur "RE: Médicaments fins", un outil disponible gratuitement, interactif basé sur le Web pour la drogue repurposing génération d'hypothèse basée sur la théorie transitive des relations Drug-Gene-maladie 8. Le g globalharbon de cette méthode consiste à intégrer systématiquement divers types de données sur les médicaments, génétiques et cliniques pour permettre la drogue repurposing pour les utilisateurs de diverses communautés, y compris les cliniques, l'industrie et des organismes de réglementation. Les méthodes fondamentales de ce système ont été signalés précédemment pour l'utilisation de l' étude du génome association (GWAS) et étude d'association phenome-large (PheWAS) données dans la drogue repurposing recherche 9,10. La nouvelle combinaison de ces types de données distingue notre webtool d'autres méthodes d'objectifs 6,11.
Le RE: fine système drogues contient actuellement 60,911 hypothèses de médicaments repurposing couvrant 916 médicaments, 567 gènes et 1.770 maladies. Le webtool fournit une interface conviviale pour les chercheurs à la recherche interactive des hypothèses de repurposing de drogues et de hiérarchiser les utiliser divers critères. Par exemple, les utilisateurs peuvent filtrer les hypothèses de médicaments repurposing avec le soutien dans la littérature biomédicale et les essais cliniques Databasoi, p-valeurs significatives, les rapports de cotes d'association ou par des indications spécifiques. La seule exigence pour ce système est l'accès à Internet.
Protocole
1. Conditions d'initiation des requêtes de Gene, les médicaments ou les maladies
- Accédez à la page d'accueil pour "RE: Médicaments fines" à l'adresse suivante: http://drug-repurposing.nationwidechildrens.org. Commencez par entrer un terme de recherche dans la barre de recherche à partir de l'une des trois catégories suivantes: médicaments (générique de nom du médicament), la maladie (nouvelle indication de la maladie) ou d'un gène (symbole officiel du gène HGNC).
- Filtrer la fonction barre de recherche pour inclure uniquement "Nom du médicament", "Indication nouvelle maladie", "Symbole Gene" ou "Tous les" catégories. La barre de recherche comprend une fonction d'auto-remplissage pour les entrées de la requête.
- Mettre dans un mot-clé, et cliquez sur le bouton "Rechercher". Trier le tableau des résultats par l'une des colonnes suivantes: "Drug", "Indication enregistré", "P-Value", "P-valeur ajustée», «Rapport de cotes", "Etude", "Nouvelle indication", "Drug Indication de la Banque "," # de Medline Abstracts "," # de Clinique Registre de première instance "," potentiel "," SNP "," Gene "ou" action ".
- Accédez à l'option de recherche avancée afin d'activer la fonction d'information pharmaceutique. Cliquez sur l'icône dans la colonne "Info" pour un médicament particulier. Observer une page qui répertorie l'ensemble des informations correspondantes, y compris p-valeur pour l'association, le nom de la maladie, le nom de la drogue, les détails de gènes (NCBI lien Gene) et les détails des médicaments (lien DrugBank).
2. Exploration des options avancées
- Cliquez sur le bouton "avancé" situé sur le côté droit de la page, et plusieurs options pour affiner les résultats sont fournis. Les options de recherche avancées comprennent des modifications à ce qui suit: la drogue, l'association, la maladie, le potentiel, le gène et l'action.
- Exporter les tableaux des résultats en cliquant sur le bouton "Exporter" sur le côté droit de la page. Cliquez sur le bouton "Simple", afin de replier la fenêtre de recherche avancée.
- Sous l'onglet Avancé d'option "de drogue", spécifiez une indication de médicament particulier ou un nom de médicament supplémentaire pour filtrer les résultats.
- Sous l'onglet "association", filtrer les résultats par le niveau de signification P-Value, ajusté P-Value avec FDR, la taille de l'effet (rapport de cotes), et / ou Type d'étude (GWAS, PheWAS ou les deux).
- Sous l'onglet «maladie», spécifiez une certaine Description de la maladie pour la nouvelle utilisation prévue.
- Sous l'onglet «potentiel», les résultats de filtre selon l'une quelconque des critères suivants: (i) si l'indication de la drogue est contenue dans la base de données DrugBank, (ii) le nombre de résumés Medline avec co-occurrence de la drogue et de la maladie, ( iii) nombre d'entrées de la base de données ClinicalTrials.gov avec co-occurrence de la drogue et de la maladie et (iv) Recibler potentiel.
REMARQUE: L'option potentiel Recibler décrit la nouveauté de la découverte: (i) Connus: relation existe déjà dans la base de données DrugBank, (ii) Fortement soutenu: Un certain soutien dans les deux registres d'essais cliniques et les résumés Medline, (iii) probable: un certain soutien dans les deux registres d'essais cliniques ou les résumés Medline et (iv) Novel: aucune preuve dans le registre de l'essai clinique, ni dans les résumés Medline. - Sous l'onglet "gène", entrez un identifiant de SNP ou Gene Symbole pour filtrer les résultats par les gènes cibles de médicaments spécifiques.
- Sous l'onglet "action", spécifier le type d'action drogue contre la cible (s) de la drogue, y compris agonistes, antagonistes, autre, inconnu ou toutes (source: base de données DrugBank).
Résultats
Dans cet exemple, le gène "IL2RB" a été saisi comme une requête fondée sur les gènes, et a été automatiquement reconnu comme tel par la fonction d' auto-remplissage (Figure 1). Les hypothèses repurposing douze médicaments pour le gène IL2RB sont retournés, comme le montre la figure 2. Détaillée page d'information pour une hypothèse médicament repurposing particulier, "daclizumab" dans ce cas, est fourni à partir de la colonne "Info" (Figure 3). Les résultats ont été filtrés sur l'onglet médicament a été par tous les résultats correspondant à la «Daclizumab» médicaments, comme le montre la figure 4. La figure 5 montre que les médicaments avec une indication connue pour le terme «transplantation» de la maladie (source: base de données DrugBank). L'onglet association permet à l'utilisateur de filtrer SNP-phénotypiques relations de signification statistique (P-valeur) et la taille de l'effet génétique (odds ratio), défini comme le rapport des chances de prEsence de la maladie chez les personnes ayant un génotype spécifique (SNP allèle) sur les chances de présence de la maladie chez les individus sans l'allèle SNP. La figure 6 montre les résultats filtrés sous l'onglet d'association au sein de la gamme P-Value de 0,000001 à 0,05. La figure 7 montre des hypothèses de médicaments repurposing spécifiques pour "asthme" sur la base des nouvelles indications que nous avons trouvé dans l'étude. La figure 8 montre les résultats sous l'onglet potentiel pour filtrer par un nombre minimum de 5 résumés Medline contenant une co-occurrence des termes de la drogue et les maladies. Dans cet exemple, tous les médicaments résultats sous l'onglet du gène sont ciblées pour le gène "IL2RB", correspondant à la durée de la requête d' origine (Figure 9). Enfin, les résultats Figure 10 spectacles filtrés sous l'onglet "action" pour revenir tous les médicaments qui agissent comme agonistes sur le gène IL2RB.

Figure 1: Le RE: Médicaments fins tableau de bord interactif page d' accueil. Les utilisateurs peuvent commencer une requête en entrant un nom de médicament, nouvelle indication de la maladie ou le symbole du gène. Des liens sont également prévus pour les documents de référence GWAS et PheWAS décrivant les méthodes pour générer des hypothèses de médicaments repurposing. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
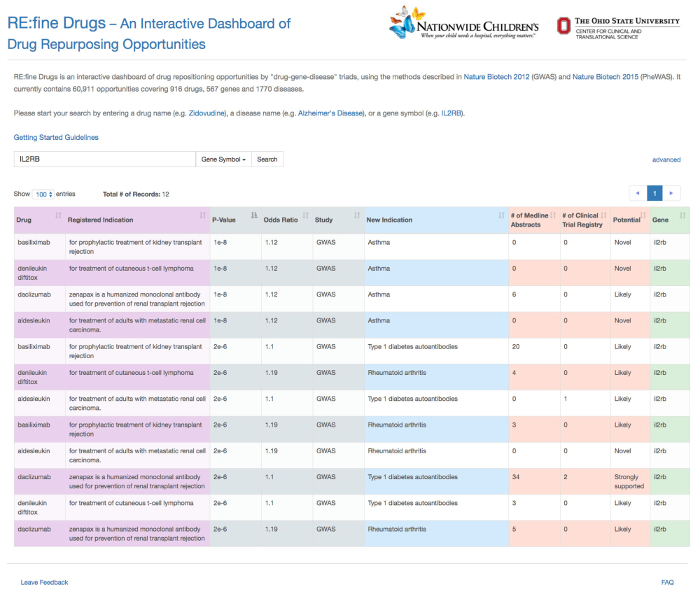
Figure 2: la fonction Auto-remplissage pour les entrées de la requête. A titre d'exemple, le terme de requête de gène "IL2RB" a été automatiquement reconnu comme un terme de gène. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
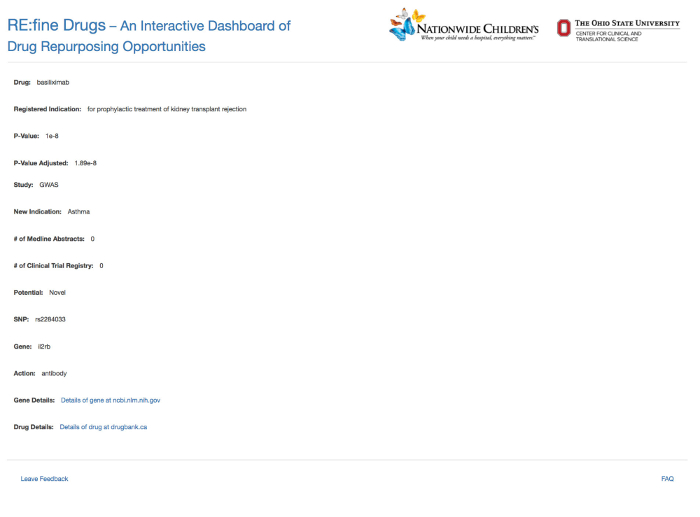
Figure 3: Drug repurposing résultats table produite à partir d' une requête fondée sur les gènes (par exemple, IL2RB). Douze médicaments repurposing hypothèses pour le gène IL2RB sont produites. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
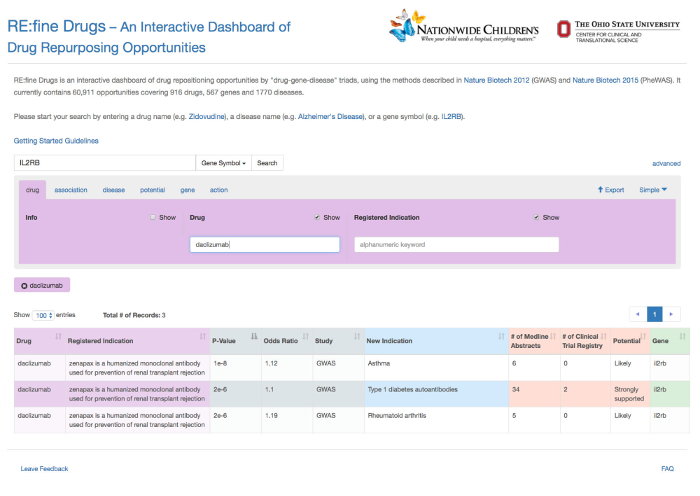
Figure 4: Page d'information pour les médicaments individuels de la page de résultats. En cliquant sur l'icône de la colonne "Info" affiche des informations détaillées pour le daclizumab de drogue. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
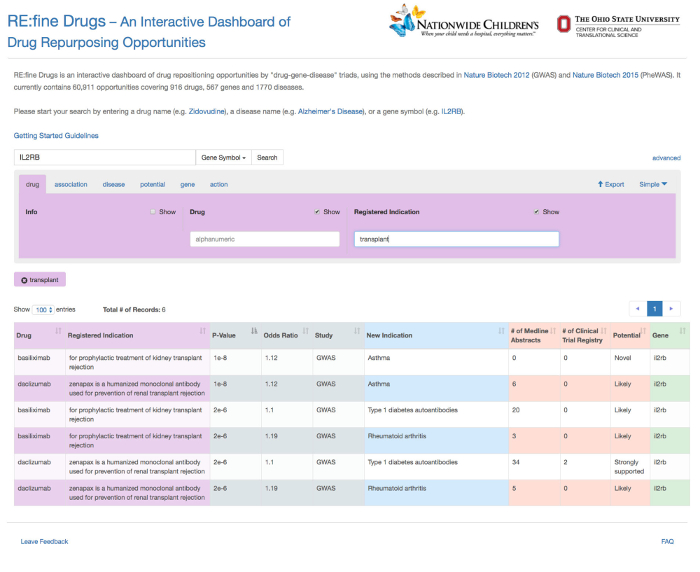
Figure 5: Recherche avancée option sous l' onglet de la drogue pour filtrer par un médicament spécifique. Dans cet exemple, trois résultats sont présentés pour le daclizumab de drogue. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
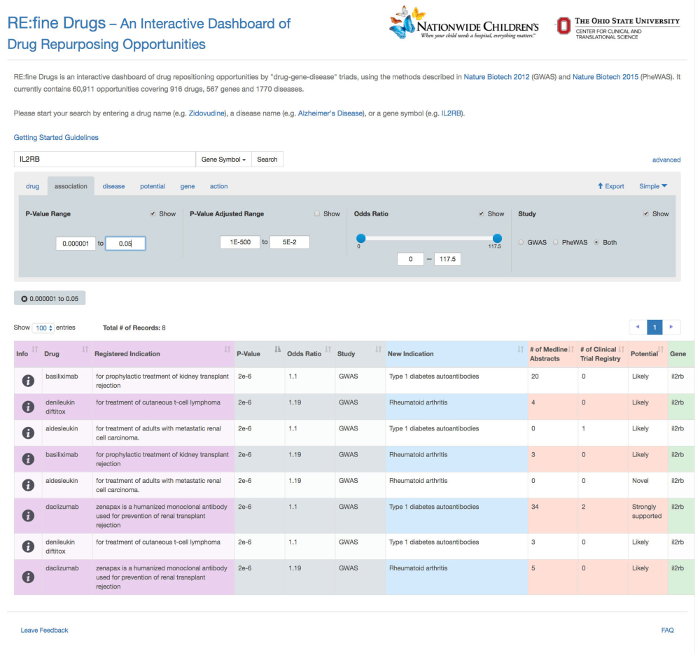
Figure 6: Recherche avancée option sous l' onglet de la drogue pour filtrer par une indication spécifique de la maladie à partir de la base de données DrugBank. Tous les médicaments ayant une indication connue pour la maladie terme «transplantation» sont présentés. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
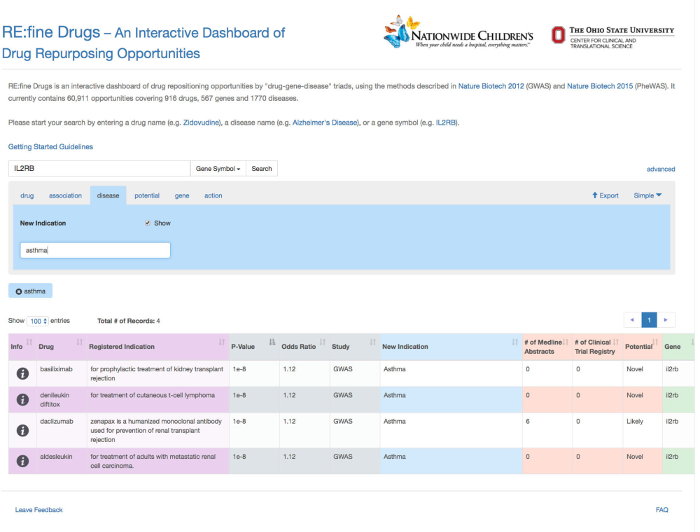
Figure 7: Recherche avancée option sous l' onglet d'association pour filtrer par niveau de signification. Dans ce cas, huit résultats sont fournis dont le niveau de signification association relève de la gamme P-Value de ,000001 à 0,05. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
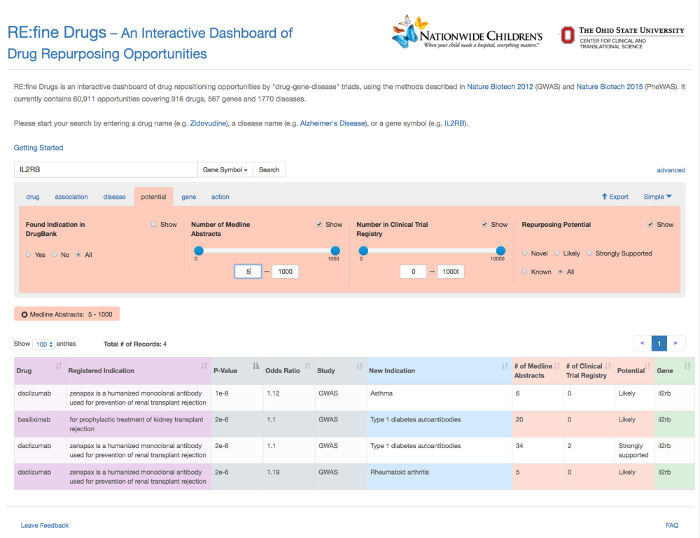
Figure 8: Recherche avancée option sous l' onglet de la maladie pour filtrer par une indication spécifique de la maladie , nous avons extrait dans cette étude. Dans cet exemple, les quatre résultats sont montrés pour l' asthme comme nouvelle utilisation de la maladie. S'il vous plaît cliquer ici pour voir un plus grand versisur ce chiffre.
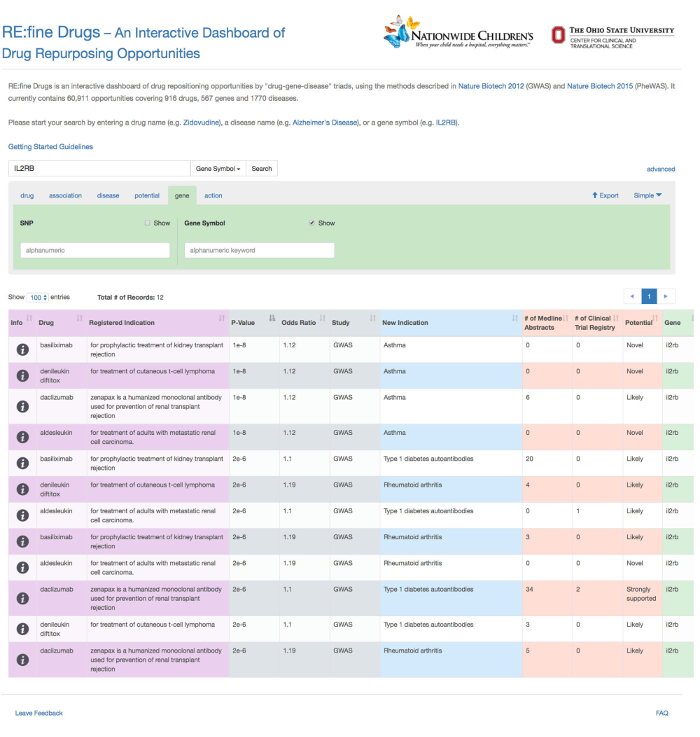
Figure 9: Recherche avancée option sous l' onglet potentiel pour filtrer par un co-occurrence des termes de la drogue et les maladies dans les résumés Medline. Dans cet exemple, quatre résultats sont présentés qui sont pris en charge par un nombre minimum de 5 résumés Medline contenant une co-occurrence des termes de la drogue et les maladies. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
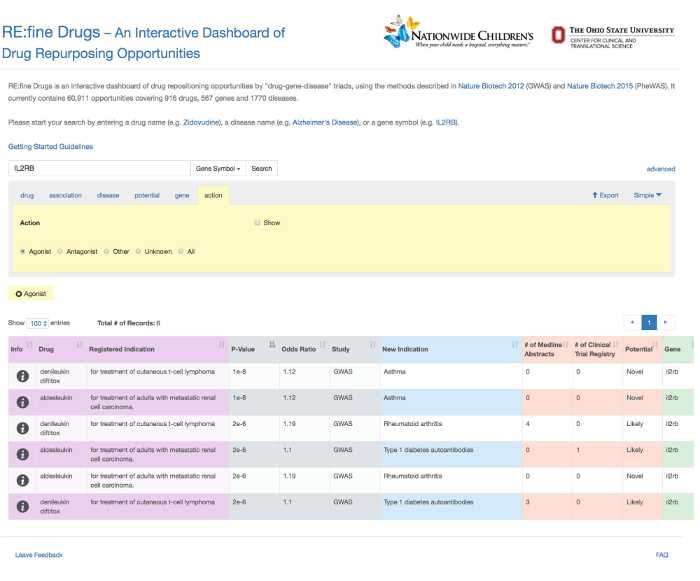
Figure 10: Recherche avancée option sous l' onglet du gène pour filtrer par un symbole de gène spécifique, où tous les résultats correspondent au gène IL2RB utilisé pour la requête initiale. Dans cet exemple, tous les médicaments résultatssous l'onglet du gène sont ciblés pour le gène "IL2RB", correspondant à la durée de la requête originale. Recherche avancée option sous l'onglet d'action pour filtrer par des médicaments agonistes seulement. Dans cet exemple, les six résultats sont renvoyés pour tous les médicaments qui agissent comme des agonistes du gène IL2RB. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Discussion
The protocol described here for the RE:fine Drugs interactive dashboard can be modified in different ways according to the user's preferences. This method uniquely integrates GWAS and PheWAS data as a novel paradigm underlying drug repurposing hypothesis generation. Specifically, this system provides access to both 52,966 PheWAS associations and 7,945 GWAS associations with advanced options to filter the results by the study type, effect size and/or significance level. Another advantage of this method over existing computational drug repurposing tools is that queries may be made from drug, gene or disease perspectives.
There are several limitations to this method. Currently, the PheWAS data is limited to primarily adult patient population from five institutions contained in the Electronic Medical Records and Genomics (eMERGE) network with a mean age of 69.5 years 12. Additionally, the "repurposing potential" feature uses co-occurrence of search terms in Medline abstracts as one of its criteria. It is well known that text mining methods using co-occurrence have limitations with respect to syntactical structure and literature bias. Thus, we recommend this feature be used as a starting point to explore the potential novelty and/or evidence supporting specific drug repurposing hypotheses and recommend additional investigation into the biomedical literature and clinical trial databases.
Future directions for this work not described here would be to extend this database to additional sources of GWAS and PheWAS data as they become available. Similar efforts to systematically translate results from large-scale GWAS studies into drug repurposing hypotheses have been previously published 9,13-14. It may be useful to compare these different workflows to predict drug candidates from GWAS data in future studies. Additionally, several other methods exist to computationally generate drug repurposing hypotheses from different data sources, including: genomics, transcriptomics, chemical structures, drug side effect profiles, as previously summarized 6,11. Future methodological advancements could also include automating drug combination predictions and providing information on drug toxicity to guide follow up studies for drug candidates.
Furthermore, the hypotheses generated from RE:fine Drugs may be further validated using electronic health records, before initiating clinical trials 15. Finally, future studies will be needed to compare this system to other target-based drug repurposing methods.
Déclarations de divulgation
The authors declare that they have no competing financial interests.
Remerciements
This work was partially supported by the National Institutes of Health (NIH) Clinical and Translational Science Awards (CTSA) Grant (UL1TR001070) to the Ohio State University's Center for Clinical and Translational Science (CCTS) and the National Library Of Medicine under Award Number T15LM011270.
matériels
| Name | Company | Catalog Number | Comments |
| Access the homepage for “RE:fine Drugs” at the following link: http://drug-repurposing.nationwidechildrens.org. | n/a | n/a | The only requirement for this system is Internet access |
Références
- Borisy, A. A., et al. Systematic discovery of multicomponent therapeutics. Proc Natl Acad Sci U S A. 100 (13), 7977-7982 (2003).
- Zhang, L., et al. High-throughput synergy screening identifies microbial metabolites as combination agents for the treatment of fungal infections. Proc Natl Acad Sci U S A. 104 (11), 4606-4611 (2007).
- Adams, C. P., Brantner, V. V. Estimating the cost of new drug development: is it really 802 million dollars? Health Aff (Millwood). 25 (2), 420-428 (2006).
- Bunnage, M. E. Getting pharmaceutical R&D back on target. Nat Chem Biol. 7 (6), 335-339 (2011).
- Ashburn, T. T., Thor, K. B. Drug repositioning: identifying and developing new uses for existing drugs. Nat Rev Drug Discov. 3 (8), 673-683 (2004).
- Hurle, M. R., et al. Computational drug repositioning: from data to therapeutics. Clin Pharmacol Ther. 93 (4), 335-341 (2013).
- Jin, G., Wong, S. T. Toward better drug repositioning: prioritizing and integrating existing methods into efficient pipelines. Drug Discov Today. 19 (5), 637-644 (2014).
- Moosavinasab, S., et al. 34;RE:fine Drugs": An Interactive Dashboard to Access Drug Repurposing Opportunities. Database (Oxford). , (2016).
- Zhang, J., et al. Use of genome-wide association studies for cancer research and drug repositioning. PLoS One. 10 (3), e0116477(2015).
- Rastegar-Mojarad, M., Ye, Z., Kolesar, J. M., Hebbring, S. J., Lin, S. M. Opportunities for drug repositioning from phenome-wide association studies. Nat Biotechnol. 33 (4), 342-345 (2015).
- Li, J., et al. A survey of current trends in computational drug repositioning. Brief Bioinform. 17 (1), 2-12 (2016).
- Denny, J. C., et al. Systematic comparison of phenome-wide association study of electronic medical record data and genome-wide association study data. Nat Biotechnol. 31 (12), 1102-1110 (2013).
- Wang, H., et al. Mining drug-disease relationships as a complement to medical genetics-based drug repositioning: Where a recommendation system meets genome-wide association studies. Clin Pharmacol Ther. 97 (5), 451-454 (2015).
- Sanseau, P., et al. Use of genome-wide association studies for drug repositioning. Nat Biotechnol. 30 (4), 317-320 (2012).
- Xu, H., et al. Validating drug repurposing signals using electronic health records: a case study of metformin associated with reduced cancer mortality. J Am Med Inform Assoc. 22 (1), 179-191 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon