Method Article
Un haut-débit Plate-forme Cell biopuces pour corrélative Analyse de la différenciation des cellules et des forces de traction
Dans cet article
Résumé
La différenciation cellulaire est régulée par une foule de facteurs microenvironnement, y compris les propriétés de la composition de la matrice et de matériaux de substrat. Nous décrivons ici une technique utilisant des puces à ADN de cellules en conjonction avec une force de traction au microscope pour évaluer la différenciation des cellules et les interactions cellule-substrat biomécaniques en fonction du contexte microenvironnement.
Résumé
microréseaux cellulaires microfabriqués, qui consistent en des combinaisons de biomolécules coordonnées imprimées sur une surface d'hydrogel élastique, fournissent un système d'ingénierie à haut débit étroitement contrôlé pour mesurer l'impact des signaux biochimiques disposés sur la différenciation cellulaire. Les efforts récents utilisant des microréseaux cellulaires ont démontré leur utilité pour les études combinatoires dans lesquelles de nombreux facteurs microenvironnement sont présentés en parallèle. Cependant, ces efforts ont porté principalement sur l'étude des effets des signaux biochimiques sur les réponses cellulaires. Ici, nous présentons une plate-forme de puces à ADN cellulaire avec les propriétés des matériaux accordables pour évaluer à la fois la différenciation des cellules par des interactions d'immunofluorescence et de la cellule-substrat biomécanique par la force de traction microscopie. Pour ce faire, nous avons développé deux formats différents en utilisant des hydrogels de polyacrylamide de variation du module de Young fabriqué soit sur des lames de microscope ou des plats à fond de verre de Pétri. Nous fournissons bespratiques de t et le dépannage pour la fabrication de biopuces sur ces substrats d'hydrogel, la culture cellulaire ultérieure sur microarrays, et l'acquisition de données. Cette plate - forme est bien adaptée à une utilisation dans les enquêtes sur les processus biologiques pour lesquels les deux biochimique (par exemple, la composition de la matrice extracellulaire) et biophysique (par exemple, la rigidité du substrat) indices peuvent jouer significative, coupant les rôles.
Introduction
Les interactions entre les cellules et les facteurs environnants microenvironnementales médient une grande variété de processus biologiques au cours du développement, l' homéostasie et la maladie pathogenèse 1, 2, 3, 4. Ces interactions comprennent l'administration microenvironnement de facteurs solubles à des cellules, la liaison cellule-matrice et interactions cellule-cellule par l'intermédiaire de la liaison ligand-récepteur. En plus des considérations biochimiques ci - dessus, les paramètres biophysiques, comme les propriétés mécaniques du substrat (par exemple, le module de Young, de la porosité) et la forme des cellules, et mécanotransduction aval associé ont de plus en plus gagné la reconnaissance en tant que médiateurs clés de la différenciation des cellules 5, 6, 7, 8, 9 , 10. Les signaux résultant de ces interactions microenvironnementales servent comme entrées aux réseaux de gènes et les voies de signalisation. En outre, ces composants cellulaires intrinsèques fournissent également une rétroaction au microenvironnement par des facteurs sécrétés et des enzymes dégradant la matrice, complétant une boucle de corégulation complexe entre les programmes génétiques cellulaires intrinsèques et des facteurs cellulaires extrinsèque microenvironnementales 5, 11, 12.
L'utilisation de systèmes d' ingénierie pour la présentation contrôlée des facteurs microenvironnement est avéré utile dans une gamme de contextes différents 13, 14, 15. Systèmes microfabriqués en particulier ont facilité la structuration spatiale précise des protéines et des cellules ainsi que des analyses hautement parallélisée via la miniaturisation 13, 16, 17, 18, 19, 20, 21, 22. Les puces à cellules représentent un tel système micro - usiné , dans lequel les combinaisons de biomolécules sont des contacts imprimés sur un substrat en hydrogel de polyacrylamide élastique 23, 24, 25. L'inclusion de composants cellulaires-adhésif (à savoir les protéines de la matrice) permet une adhérence soutenue de culture cellulaire et sur les microréseaux, qui est souvent suivie d'une analyse en aval par l'intermédiaire d'immunocytochimie et reporters fluorescents. Microréseaux cellulaires ont été productifs orientés vers la réalisation d' une meilleure compréhension du phénotype des cellules du foie 23, 26, la différenciation des précurseurs de neurones 27, Mammardécisions y progéniteur du devenir 28, embryonnaires cellules souches d' entretien / différenciation 23, 29, 30, poumon métastases du cancer 31, et réponse thérapeutique dans le mélanome 32. Récemment , nous avons démontré que l'utilisation de puces à ADN de la cellule pour définir le rôle de la matrice extracellulaire (ECM) Composition de la protéine dans la description de l' endoderme 33, la différenciation des progéniteurs du foie 34, 35, et une tumeur du poumon réponse aux médicaments des cellules 36. Dans ces travaux, nous avons mis l'accent sur l'extension des capacités combinatoires de la plate-forme de réseau et d'explorer les intersections de la signalisation cellulaire intrinsèque avec la composition et la biomécanique de la matrice extracellulaire. En outre, nous avons mis en œuvre des lectures biophysiques cette plate-forme de réseau pour fournir la capacité de quantitativement caracponctuent la le rôle de la contractilité des cellules dans la différenciation des processus 35. Pour ce faire, nous avons intégré la microscopie à force de traction (TFM) avec des microréseaux cellulaires pour permettre une évaluation à haut débit de traction générée cellulaire. TFM est une méthode largement utilisée pour mesurer des forces de traction générées cellules et a fourni des informations importantes concernant la coordination de la cellule unique et une fonction de niveau de tissu avec la composition et biomécanique du microenvironnement local 37, 38, 39, 40. Ainsi, en combinant TFM avec des microréseaux cellulaires fournit un système à haut débit pour mesurer les principaux paramètres biophysiques, physiologiquement pertinents.
La plate-forme de puces à ADN cellulaire décrit ici se compose de quatre sections: la fabrication de substrats de polyacrylamide, la fabrication de tableaux, de la culture cellulaire et test de lecture et l'analyse des données. VoirLa figure 1 pour un résumé schématique des trois premières sections expérimentales; voir la figure 2 pour un résumé schématique de la section finale avec un accent sur l' analyse des données d'immunofluorescence. Afin d'adapter la plate - forme de puces à ADN de la cellule à l' étude des interactions cellule-substrat biomécaniques, on a utilisé des substrats de Polyacrylamide du module d'accordable jeune , mais une porosité similaire, par Wen et al. 41. Pour permettre des mesures TFM des forces exercées par les cellules sur leur substrat, nous avons implémenté un format de Petri plat à fond de verre en plus du microscope en verre épais coulisse fréquemment utilisé par d'autres groupes. Ainsi, cette plate - forme de puces à ADN cellulaire est capable de mesures parallèles de la différenciation cellulaire par immunofluorescence sur des lames de microscope et les forces générées cellules par TFM sur la vaisselle à fond de verre séparés. Nous avons également appliqué plusieurs améliorations à l'approche analytique utilisée avec des microréseaux cellulaires. Spécifiquesly, au lieu de paramétrique Z-score de l'intensité globale de l'île, nous mesurons l'intensité à cellule unique et appliquer la normalisation quantile afin de tenir compte des distributions non-normales et décrire plus précisément le comportement cellulaire. Nous croyons que ces améliorations fournissent une utilité particulière vers les enquêtes sur les processus biologiques dans lesquels les signaux à la fois biochimiques et biophysiques jouent des rôles importants qui se croisent. En outre, nos améliorations analytiques permettent l'application des microréseaux cellulaires à des études d'une gamme de fonctions cellulaires pour lesquelles une seule cellule et de comportement au niveau de la population divergent.
Protocole
1. Fabrication de polyacrylamide Substrats
- des substrats en verre propres - soit des lames de microscope standard pour immunofluorescence d'extrémité ou à fond de verre de 35 mm des boîtes de Pétri pour TFM - afin d'assurer polyacrylamide optimal hydrogel fabrication et l'intégrité lors de la culture cellulaire. Alternativement, l'utilisation pré-nettoyé des substrats en verre.
- Imprégner des substrats en verre à 0,25% v / v de Triton X-100 dans de l' eau distillée (dH 2 O). Placer les substrats sur un agitateur orbital pendant 30 minutes.
- Retirer Triton X-100 solution et rincer les substrats 5 fois avec des substrats dH 2 O. Laisser immergés dans le rinçage final et le placer sur un agitateur orbital pendant 30 min.
- Retirer dH 2 O et immergez substrats dans de l' acétone. Placer les substrats sur un agitateur orbital pendant 30 minutes.
- Retirer l'acétone et immergez substrats dans le méthanol. Placer les substrats sur un agitateur orbital pendant 30 minutes.
- Retirer le méthanol et rincer les substrats 5 fois avec dH 2 substrats O. Immerger dans NaOH 0,05 N et les placer sur un agitateur orbital pendant 1 h.
ATTENTION: NaOH est très caustique et peut causer de graves brûlures de la peau et des lésions oculaires. Porter des gants, des vêtements et des lunettes de protection. - Retirer la solution de NaOH et rincer les substrats 5 fois avec dH 2 O. Utiliser de l' air comprimé filtré pour sécher des substrats et cuire au four à 110 ° C sur une plaque chaude jusqu'à ce que sec (5 - 15 min). substrats nettoyés peuvent être stockés à température ambiante indéfiniment.
- Silaniser substrats en verre propre, afin d'assurer la fixation de l'hydrogel de polyacrylamide.
- Imprégner des substrats en verre propre dans fraîchement préparée de 2% v / v 3- (triméthoxysilyl) propyle, le méthacrylate de (3-TPM) dans de l'éthanol. Placer les substrats sur un agitateur orbital pendant 30 minutes.
ATTENTION: 3-TPM est un liquide combustible. Tenir à l'écart de la chaleur, des étincelles, des flammes nues et des surfaces chaudes et utiliser uniquement dans une hotte chimique. - Retirer la solution 3-TPM et immergez substrats dans ethanol. Placer les substrats sur un agitateur orbital pendant 5 min.
- Utiliser de l'air comprimé filtré pour sécher les substrats et cuire au four à 110 ° C sur une plaque chaude jusqu'à ce que sec (5 - 15 min). substrats Silanized peuvent être stockés à température ambiante pendant 1 mois.
- Imprégner des substrats en verre propre dans fraîchement préparée de 2% v / v 3- (triméthoxysilyl) propyle, le méthacrylate de (3-TPM) dans de l'éthanol. Placer les substrats sur un agitateur orbital pendant 30 minutes.
- Option 1: Fabriquez hydrogels de polyacrylamide sur des lames de microscope silanisées pour point final immunofluorescence.
- Préparer une solution pré-polymère dans dH2Û avec l'acrylamide / pourcentage bisacrylamide souhaité (p / v) rapport pour fabriquer des substrats avec des modules de Young de 4 kPa (4% d' acrylamide, 0,4% de bisacrylamide), 13 kPa (6% d' acrylamide, 0,45 % bisacrylamide), ou 30 kPa (8% d' acrylamide, 0,55% bisacrylamide) et la porosité similaire, par Wen et al. 41. Vortex solution jusqu'à claire et filtre avec une seringue de 0,2 um. les solutions de pré-polymères peuvent être stockés à 4 ° C pendant 3 mois.
ATTENTION: L'exposition à l'acrylamide ou bisacrylamide peut entraîner une toxicité aiguë, Neurotoxicity, et l'irritation. Porter des gants, des vêtements et des lunettes de protection. - Préparer une solution de photoinitiateur de 20% en poids / volume d'Irgacure 2959 dans le methanol. Cette solution de photoinitiateur ne peut pas être stocké et doit être préparé frais chaque fois.
- Mélanger le pré-polymère et des solutions de photo-initiateurs dans un rapport de 9: 1 (photoinitiateur pré-polymère). dégazer le cas échéant avec une chambre à vide pendant 15 min pour éliminer les bulles.
- Placer les lames silanisées dans un plateau de séchage en verre et pipeter 100 pi de 9: 1 de pré-polymère: Solution de photo-initiateur sur chaque lame. couvrir délicatement chaque lame avec une lamelle mm 22 × 60, tout en évitant la création de bulles. A noter que la lamelle empêche l'inhibition de la réaction de polymérisation par l'oxygène.
- Placer le plateau de séchage dans un agent de réticulation aux UV et exposer les diapositives à 365 nm UV A pendant 10 min (4 W / m 2). Optimiser le temps de polymérisation, au besoin. Des expositions plus longues difficultés de risque de retirer la lamelle en raison de surpolymérisation. expositions plus courtes rISK underpolymerization et une faible stabilité d'hydrogel.
- Immergez hydrogels dans dH 2 O pendant 5 min. Retirer les lamelles avec un rasoir, en prenant soin de ne pas endommager les hydrogels polymérisés.
- Laisser hydrogels dH 2 O à température ambiante pendant 1 à 3 j, la modification dH 2 O par jour. Déshydrater hydrogels à 50 ° C sur une plaque chaude jusqu'à ce que sec (15 - 30 min) et conserver à la température ambiante pendant jusqu'à 3 mois.
- Préparer une solution pré-polymère dans dH2Û avec l'acrylamide / pourcentage bisacrylamide souhaité (p / v) rapport pour fabriquer des substrats avec des modules de Young de 4 kPa (4% d' acrylamide, 0,4% de bisacrylamide), 13 kPa (6% d' acrylamide, 0,45 % bisacrylamide), ou 30 kPa (8% d' acrylamide, 0,55% bisacrylamide) et la porosité similaire, par Wen et al. 41. Vortex solution jusqu'à claire et filtre avec une seringue de 0,2 um. les solutions de pré-polymères peuvent être stockés à 4 ° C pendant 3 mois.
- Option 2: Fabriquez fluorescentes polyacrylamide hydrogels contenant des perles-sur silanisées 35 mm plats à fond de verre de Petri pour l'évaluation en direct des interactions cellule-substrat à l'aide de TFM.
- Soniquer une solution mère de 1 um perles fluorescentes pendant 15 min pour disperser les agrégats.
- Préparer une solution pré-polymère dans dH2Û avec l'acrylamide / pourcentage bisacrylamide souhaité (p / v) rapport pour fabriquer des substrats avec des modules de Young de 4 kPa (4% d' acrylamide, 0,4% de bisacrylamide), 13 kPa (6% d' acrylamide, 0.45% Bisacrylamide), ou 30 kPa (8% d' acrylamide, 0,55% bisacrylamide) et la porosité similaire, par Wen et al. 41. Vortex solution jusqu'à claire et filtre avec une seringue de 0,2 um. les solutions de pré-polymères peuvent être stockés à 4 ° C pendant 3 mois.
ATTENTION: L'exposition à l'acrylamide ou bisacrylamide peut entraîner une toxicité aiguë, la neurotoxicité, et l'irritation. Porter des gants, des vêtements et des lunettes de protection. - Ajouter des perles fluorescentes à la solution de pré-polymère à une concentration finale de 0,2% v / v et le vortex pour mélanger.
- Préparer une solution de photoinitiateur de 20% en poids / volume d'Irgacure 2959 dans le methanol. Cette solution de photoinitiateur ne peut pas être stocké et doit être préparé frais chaque fois.
- Mélanger les solutions pré-polymère / perles et photoamorceurs dans un 9: 1 (pré-polymère / perle: photoinitiateur) ratio. dégazer le cas échéant avec une chambre à vide pendant 15 min pour éliminer les bulles.
- Placez silanisées 35 mm plats à fond de verre de Petri dans un plateau de séchage en verre et pipet 20 pi de 9: Solution de photo-initiateur sur le centre de chaque boîte: 1 de pré-polymère / perle. couvrir délicatement chaque lame avec une lamelle circulaire de 12 mm, tout en évitant la création de bulles. A noter que la lamelle empêche l'inhibition de la réaction de polymérisation par l'oxygène.
- Afin de répartir les billes fluorescentes sur la surface de l'hydrogel, inverser les plats et le laisser à la température ambiante pendant 20 min, par Knoll et al. 42.
- Alors qu'il était encore inversé, exposer des plats à 365 nm UV A pendant 10 min (4 W / m 2). Optimiser le temps de polymérisation, au besoin. Des expositions plus longues difficultés de risque de retirer la lamelle en raison de surpolymérisation. Shorter underpolymerization expositions au risque et une faible stabilité d'hydrogel.
- Immergez hydrogels à 0,1 M 4- (2-hydroxyéthyl) -1-piperazineethanesulfonic (HEPES) tampon et laisser à température ambiante dans l'obscurité pendant une nuit. Retirez soigneusement les lamelles avec un rasoir, en prenant soin de ne pas endommager le polymehydrogels risé.
- Déshydrater hydrogels à 50 ° C sur une plaque chaude jusqu'à ce que sec (15 - 30 min). Les hydrogels peuvent être stockés à température ambiante dans l'obscurité pendant 3 mois.
2. Fabrication de tableaux
- Préparation des tampons pour imprimer des biomolécules. Utilisez le tampon d'impression approprié pour les biomolécules d'intérêt. Facteur de croissance (GF), le tampon d'impression est largement adapté à d'autres classes de molécules, telles que des ligands de cellule à cellule.
- Pour préparer 2 x tampon d'impression en protéines ECM, ajouter 164 mg d'acétate de sodium et 37,2 mg d'acide éthylènediaminetétraacétique (EDTA) à 6 ml dH 2 O vortex et laisser incuber à 37 ° C pour solubiliser complètement. Après solubilisation, ajouter 50 pi de préchauffée de Triton X-100 et 4 mL de glycérol. Vortex et incuber à 37 ° C à nouveau pour solubiliser. Ajouter 40 - 80 ul d'acide acétique glacial, titrer pour ajuster le pH à 4,8. 2 x tampon d'impression en protéines ECM peut être stocké à 4° C pendant 1 mois.
ATTENTION: L'acide acétique est inflammable et corrosif. Porter des gants, des vêtements et des lunettes de protection. - Pour préparer un tampon de 2 x d'impression GF, ajouter de l'acétate de sodium et 105,5 mg 37,2 mg d'EDTA à 6 ml de solution saline tamponnée au phosphate (PBS). Vortexer et incuber à 37 ° C pour solubiliser complètement. Après solubilisation, ajouter 100 mg de 3 - [(3-cholamidopropyl) diméthylammonio] -1-propanesulfonate (CHAPS) et 4 ml de glycérol. un tampon d'impression de la protéine GF 2 × peut être conservé à 4 ° C pendant 1 mois.
- Pour préparer 2 x tampon d'impression en protéines ECM, ajouter 164 mg d'acétate de sodium et 37,2 mg d'acide éthylènediaminetétraacétique (EDTA) à 6 ml dH 2 O vortex et laisser incuber à 37 ° C pour solubiliser complètement. Après solubilisation, ajouter 50 pi de préchauffée de Triton X-100 et 4 mL de glycérol. Vortex et incuber à 37 ° C à nouveau pour solubiliser. Ajouter 40 - 80 ul d'acide acétique glacial, titrer pour ajuster le pH à 4,8. 2 x tampon d'impression en protéines ECM peut être stocké à 4° C pendant 1 mois.
- Préparer la plaque source.
- Dans un V-bottom microplaques 384 puits, mélanger des volumes égaux de 2 x tampon d'impression avec chaque solution de biomolécules au double de la concentration cible.
REMARQUE: une concentration cible appropriée pour des protéines d'ECM les plus courants est de 250 pg / ml alors que les concentrations cibles pour d'autres types de facteurs disposés varient en fonction de la rétention dans l'hydrogel et la fonction biologique. Le volume total dans chaque puits peut êtreaussi bas que 5 pi et ne doivent pas être plus de 15 pi. En plus des combinaisons de biomolécules d'intérêt, inclure un marqueur fluorescent rangés dans le but de faciliter l'analyse d'image en aval. Utiliser la rhodamine conjugué dextrane (2,5 mg / ml). - Mélanger soigneusement chaque puits par pipetage, en prenant soin de ne pas générer des bulles. Centrifuger la microplaque source pendant 1 min à 100 x g. Fabriquez microréseaux utilisant des plaques de source préparés le même jour et stockées à 4 ° C jusqu'à microarray fabrication.
- Dans un V-bottom microplaques 384 puits, mélanger des volumes égaux de 2 x tampon d'impression avec chaque solution de biomolécules au double de la concentration cible.
- broches Nettoyer selon les instructions du fabricant avant chaque essai microarray de fabrication. Chargez broches propres directement dans la tête d'impression de l'microarrayer.
- Préparer microarrayer et programme en utilisant le logiciel du fabricant. Bien que les étapes ci-dessous sont en partie spécifiques au microarrayer particulier utilisé ici, le fonctionnement de la plupart microarrayers est similaire.
- Allumez l'appareil de l'humidificateur, régler le point de consigne à 65% HR (non-condensing), et attendre jusqu'à ce que le rhéomètre correspond au point de consigne. Placer la plaque de source dans l'adaptateur approprié.
- Déshydrater substrats hydrogel à 50 ° C pendant 15 min et le placer dans l'adaptateur approprié. Le microarrayer a des adaptateurs pour les deux lames de microscope et microplaques. Pour 35 mm rangeant plats à fond de verre de Pétri, charger la vaisselle dans une microplaque de 6 puits et placer la microplaque dans l'adaptateur de microplaques sur le arrayer.
- Réglez les paramètres du programme pour refléter fidèlement la mise en page de la plaque source, la conception de réseau, et le format désiré (par exemple, lame de microscope ou microplaque contenant 35 mm des boîtes de Pétri). Inclure les étapes de lavage utilisant à la fois l'eau et le diméthylsulfoxyde (DMSO) entre chaque état afin d'éviter de report et de contamination croisée.
- Commencez tableau de fabrication, de contrôle pas moins d'une fois une heure que l'humidité n'a pas chuté en dessous de 65% HR (sans condensation) et que les broches ne sont pas obstrués. Si l'humidité a chuté de façon inattendue, une pause rangeant pour remplir l'humidificateur et effacer tubes associés condensation. Si les broches sont obstrués, pause rangeant pour nettoyer les broches ou remplacer autrement avec des broches pré-nettoyés. A noter qu'il est possible de matrices multiples types de biomolécules successivement sur les mêmes substrats munis temps de séchage suffisant (c. -à- 4 h à une nuit).
- Une fois que le programme est terminé, placer des tableaux fabriqués dans une boîte de diapositives ou microplaques recouverts d'une feuille d'aluminium à la température ambiante et 65% HR (sans condensation) pendant la nuit. Notez qu'il peut être nécessaire d'évaluer la qualité du réseau et de rétention en utilisant les taches de protéines générale ou immunofluorescence; voir Brafman et al. pour plus de détails 25.
3. Culture cellulaire et Assay Readout
- Le jour après la fabrication, placez des substrats disposés en 4-chambré plats (lames de microscope) ou 6 puits des microplaques (boîtes de Pétri) et plonger dans 1%v / v de la pénicilline / streptomycine dans du PBS; utiliser 4 ml pour diapositives et 3 ml pour les plats. Exposer aux rayons ultraviolets C pendant 30 min. Échange solution de pénicilline / streptomycine pour les milieux de culture cellulaire.
- Recueillir et compter les cellules. Semences sur des tableaux à 500 × 3 au 2 octobre × 10 6 cellules / matrice à 4 ml par lame de microscope et 3 ml par 35 mm boîte de Pétri. Incuber les cultures de tableau à 37 ° C et 5% de CO 2 pour 2 - 24 h ou jusqu'à ce que la formation d'îlots de cellules bien peuplées. Réglez la densité et le temps que nécessaire pour vos cellules et application particulière ensemencement. - Ensemencement (c. -à faible densité ou le temps ensemencement) peut entraîner la population pauvre du tableau et les résultats biologiques asymétriques. Sursemis (c. -à haute densité ou le temps ensemencement) peut entraîner l' intégrité du réseau réduite due au détachement de l' île.
- Après avoir laissé pour la formation d'îlots de cellules, se laver les cultures deux fois avec de matrice préchauffée milieux de culture de cellules; à nouveau utiliser 4 ml pour diapositives et 3 ml pour les plats. optionaajouter lly contrôles et des traitements appropriés (par exemple, les inhibiteurs de petites molécules, des facteurs de croissance, etc.) d'intérêt pour le système biologique. Changer les médias des tableaux tous les 1 - 2 d, afin de maintenir la concentration de tous les traitements. Évaluer l'expression du marqueur cellulaire et la fonction des cellules par immunofluorescence ou d'une cellule-substrat interactions par TFM dans 1 - 5 d d'initier des cultures de tableau - voir Option 1 et Option 2 ci-dessous.
- Option 1: Effectuer immunofluorescence extrémité. A noter que l'immunofluorescence de certaines protéines peut nécessiter perméabilisation plus strictes en utilisant du méthanol, de l'éthanol ou du HCl. En raison de dommages potentiels aux tableaux, évaluer et optimiser chaque protocole de perméabilisation avant utilisation dans des expériences à grande échelle.
- Aspirer milieux de culture cellulaire à partir de diapositives de tableau en 4-chambré plats et ajouter 4 mL / slide fraîchement préparé 4% v / v paraformaldehyde (PFA) dans du PBS. Incuber pendant 15 min à température ambiante.
ATTENTION: Exposure de PFA peut entraîner une toxicité aiguë et peut également irriter ou corroder la peau au contact. Porter des gants, des vêtements et des lunettes de protection et utiliser uniquement dans une hotte chimique. - solution et PFA Aspirer laver chaque diapositive 3 fois avec 4 ml de PBS. À ce stade, les lames fixées peuvent être conservés à 4 ° C pendant 1 semaine. Il est conseillé, cependant, de continuer par immunomarquage et montage sur le même jour que la fixation afin d'assurer l'intégrité du réseau.
- Aspirer le PBS et on ajoute 4 ml / glissement de 0,25% v / v de Triton X-100 dans du PBS. Incuber pendant 10 min à température ambiante.
- Aspirer Triton X-100 solution et laver chaque diapositive 3 fois avec 4 ml de PBS. Ajouter 4 ml / glissement de 5% v / v de sérum adapté à l'espèce de l'anticorps secondaire (par exemple, du sérum d'âne pour des anticorps secondaires , des ânes) dans du PBS et on incube à température ambiante pendant 1 h.
- Retirer soigneusement la solution de blocage de chaque diapositive. Ajouter 500 ul / glissement de l'anticorps primaire dilué dans 5% v / v de sérum dans du PBS. Ce volume est suffisant pour couvrir les tableaux pour les 1 h incubations à température ambiante ainsi que les incubations d'une nuit à 4 ° C.
- Laver chaque lame de matrice 3 fois avec 4 ml de PBS. Retirer soigneusement le lavage final et ajouter 500 pi / slide de l'anticorps secondaire approprié dilué dans 5% v / v de sérum dans du PBS.
- Laver chaque lame de matrice 3 fois avec 4 ml de PBS. Laver brièvement avec dH 2 O avant de retirer soigneusement les diapositives de tableau de solution en utilisant une pince. Utilisez un tissu de laboratoire pour évacuer ou dH résiduel sec 2 O.
- Pipeter 100 ul de solution de DAPI à monter sur le coulisseau, tout en confirmant visuellement une couverture complète de l'ensemble du réseau.
- Placer un mm lamelle 22 × 60 sur la lame pour monter. Sceller les bords de la lamelle avec du vernis à ongles transparent. A conserver dans l'obscurité à 4 ° C jusqu'à ce que l'imagerie, au plus tôt le lendemain.
- toute l'image à l'aide de tableaux, soit un scanner de puces à ADN ou microscope inversé equipp fluorescented avec une scène robotique. Scanners de biopuces donnent une lecture plus rapide , mais peuvent nécessiter Cy3 ou Cy5 compatibles fluorophores et sont souvent de la résolution limitée à l'ordre de cellules individuelles ( par exemple, 1 - 10 um). microscopes fluorescents offrent la possibilité d'utiliser une variété de canaux fluorescentes et à une résolution plus élevée (<1 pm, ~ 100 × grossissement total), mais donnent une lecture plus lente en fonction de la qualité de la scène robotique et l'agrandissement / objectif.
- Sauvegarder les images capturées de tableaux entiers de deux méthodes sous forme de fichiers TIFF afin d'éviter la compression ou la perte de données associée à d' autres formats de fichiers (par exemple, JPG).
- Aspirer milieux de culture cellulaire à partir de diapositives de tableau en 4-chambré plats et ajouter 4 mL / slide fraîchement préparé 4% v / v paraformaldehyde (PFA) dans du PBS. Incuber pendant 15 min à température ambiante.
- Option 2: Effectuer l'évaluation en direct des interactions cellule-substrat à l'aide de TFM.
- Préparer une solution de 1% v / v de sérum-albumine bovine (BSA) et 1% v / v de dodécylsulfate de sodium (SDS) dans du PBS pour dissocier les cellules des substrats pendant TFM.
- Déplacer 35 mm des boîtes de Petri contenant des cultures de tableau àun incubée (37 ° C, 5% CO 2), microscope à fluorescence inversée avec un étage robotique pour les mesures TFM.
- Dans un plat, marquer les positions (coordonnée X, coordonnée Y) et de se concentrer plans (coordonnée Z) des îlots de cellules individuelles en utilisant la microscopie à contraste de phase.
- Passer à la microscopie à fluorescence rouge lointain pour visualiser les perles. Revenir à chacune des positions enregistrées à l'étape précédente et de corriger la coordonnée Z du plan de focalisation de sorte que seule la première couche de billes en dessous de l'île de la cellule est mise au point. Enregistrer les nouvelles coordonnées et procéder à l'imagerie automatisée de toutes les îles de la cellule pour capturer pré-dissociation contraste de phase et des images fluorescentes rouges loin.
- Soigneusement ajouter 150 pi de solution de BSA / SDS à l'antenne et attendre 5 min pour permettre la dissociation complète des cellules du substrat; surveiller la dissociation des cellules en utilisant la microscopie à contraste de phase.
- Après les îles de cellules ont été dissociées du substrat, revenir à til a marqué des positions et vérifier que la première couche de perles sont encore au point. Si ces billes sont hors du plan du fait de la déformation provoquée par la traction générée cellulaire, puis à corriger la coordonnée Z du plan de mise au point afin qu'ils soient à nouveau mis au point. Enregistrer les coordonnées Z corrigées et répéter l'imagerie automatisée de toutes les îles pour capturer post-dissociation des images fluorescentes bien rouges.
- Répétez les étapes 3.5.1 - 3.5.4 pour les plats restants.
4. Analyse des données
- L'analyse des données d'immunofluorescence.
- Processus images acquises de tableau. Divisez images matricielles composites dans des fichiers contenant des canaux individuels (ie, rouge, bleu ou vert) et convertir en 8 bits images TIFF 43, 44. Appliquer binning (par exemple, 2 × 2 ou 4 × 4) afin de réduire la taille de l' image pour ~ 32 mégapixels par canal afin de réduire les besoins en mémoire lors de l' aval analyse unicellulaire de entire images de tableau. Voir fichier code supplémentaire intitulé "array_processing.ijm" pour une ImageJ macro mise en œuvre de ces étapes de traitement de tableau.
- Notez les coordonnées en pixels du haut à gauche, en bas à gauche, et des marqueurs dextran en bas à droite rhodamine conjuguée ou conditions disposées. Utilisez ces coordonnées pour faire pivoter les images 8-bit TIFF pour être parfaitement vertical et, plus tard, à annoter sortie de l'analyse unicellulaire avec des conditions spécifiques déployées. Voir Fichiers de code supplémentaire intitulé "rb_array_rotater.ijm", "rg_array_rotater.ijm", "rgb_array_rotater.ijm", et "array_gridding.ijm" pour les implémentations de ces tableau tournant et gridding étapes.
- Effectuer l' analyse unicellulaire de binned, rotation des images TIFF 8 bits dans CellProfiler (version 2.1.1) 45 en utilisant les modules suivants: IdentifyPrimaryObjects, IdentifySecondaryObjects et MeasureObjectIntensity. IdentifyPrimaryObjects identifie les noyaux, IdentifySecondaryObjects identifie immunolabels associés à chaque noyau cellulaire, et fournit MeasureObjectIntensity quantifications pour les étiquettes et immunolabels nucléaires.
- les données une seule cellule de sortie de tous les trois modules sous forme de fichier CSV par canal en utilisant le module ExportToSpreadsheet pour faciliter une analyse ultérieure en aval. Voir Fichiers de code supplémentaire intitulé "b_array_image_analysis.cppipe", "gb_array_image_analysis.cppipe", "rb_array_image_analysis.cppipe", et "rgb_array_image_analysis.cppipe" pour les pipelines de CellProfiler mettant en œuvre ces étapes pour les ensembles d'images contenant des canaux rouge, vert ou bleu.
- Pour transformer les données pour tenir compte de la variabilité expérimentale et distributions unicellulaires non gaussiennes, appliquer la normalisation quantile par répliquée biologique 46. Ce processus génère une distribution partagée entre répétitions et permet des comparaisons impartiales sur les changements d'intensité immunolabel. En outre, unlike Z-score et d'autres méthodes paramétriques, la normalisation quantile est non-paramétrique et ne suppose pas une distribution particulière des données, permettant une plus représentative analyse du comportement à cellule unique en fonction de l'état rangé.
- Tracer des données et à interpréter. Selon le système biologique et hypothèses, calculer et tracer une ou plusieurs des mesures d'ensemble suivantes pour chaque condition rangé:
- Calculer et tracer les cellules par île comme une mesure combinée de l'adhérence et de la survie au cours de l'expérience.
- Calculer et tracer l'intensité de immunolabel quantile-normalisée en tant que mesure du destin ou de la fonction cellulaire.
- Calculer et tracer le pourcentage de cellules positives pour un immunolabel tel que déterminé par l'intensité supérieure à un seuil compatible, généralement 2 sd-dessus de l'intensité moyenne d'un contrôle négatif.
- Alternativement, les distributions de parcelles d'intensité immunolabel afin d'examiner et de classer le comportement à cellule uniqueen fonction de l'état rangé. Ces distributions peuvent en outre être caractérisés par des mesures de tendance centrale (moyenne, médiane, mode) et de la variation (variance, coefficient de variation, le facteur Fano) et les méthodes de test d'hypothèses telles que le test de Kolmogorov-Smirnov.
- L'analyse des données TFM. Ici est décrit une approche intégrant un algorithme développé précédemment par Butler et al. et Wang et al. 40, 47.
- Utilisez ImageJ pour convertir par lots les images en fichiers TIFF 8-bit. Appliquer binning pixel en moyenne (par exemple, 2 × 2) pour réduire le coût et le temps de calcul de l' analyse en aval. Comme algorithmes de TFM ont porté essentiellement sur l' analyse unicellulaire, la grande interface cellule-substrat des îles (~ 17,5 × 10 3 pm 2) par rapport à l'interface cellule-substrat d'une seule cellule (75 pm 2) necessiTates l'étape de binning.
- Entrez le contraste de phase capturé et images loin rouges fluorescentes ( à la fois pré-dissociation et post-dissociation) dans un environnement de programmation scientifique tels que MATLAB et processus en utilisant les algorithmes développés précédemment de Butler et al. et Wang et al. 40, 47.
- Sélectionnez trois régions éloignées de l'île de la cellule. Ces régions sont utilisées pour tenir compte des déplacements dus à l'image ou la dérive de l'échantillon.
- Fournir le facteur de conversion de pixels pour micromètres (par exemple 0.454 pixels / um), le module de Young du substrat (par exemple, 13 kPa) et le coefficient de Poisson (par exemple, 0,48 pour les gels de Polyacrylamide décrits ici).
- Pour chaque île, tracer une limite autour de la périphérie pour définir les contraintes géométriques; toutes les forces en dehors de cette limite sont mis à zéro. Ce système contraint est raisonnable compte tenu de la grande distance (ie, 450 um) entre les îles.
- Calculer la contrainte de traction racine carrée moyenne et le moment contractile pour chaque île. Le moment contractile est une mesure de la contrainte résiduelle dans l'îlot de cellules et a été montré pour tenir compte de la force des interactions cellule-cellule 48. Pour chaque condition parée, moyenne des valeurs racine carrée moyenne sur plusieurs îles et répétitions biologiques et calculer associés variance pour les tests d'hypothèses. Il est également possible de la moyenne de la répartition des contraintes ou des moments plus nombreuses îles de fournir une carte représentant des deux mesures en fonction de la géométrie, par exemple, la distance du centre de l'île.
Résultats
L' utilisation de cette plate - forme, nous avons étudié le rôle des deux indices biochimiques et biophysiques dans la spécification du sort des progéniteurs du foie 34, 35. / Notch G conjuguée à des ligands de protéine A ont amélioré la rétention et le regroupement dans l'hydrogel de polyacrylamide (figure 3A) et sont en outre capables de différenciation des progéniteurs du foie conduisant vers une destinée des cellules des canaux biliaires (figure 3B). En utilisant une analyse à cellule unique, nous avons quantifié la réponse à des ligands Notch pour les protéines d'ECM de collagène I, le collagène III, le collagène IV, la fibronectine et la laminine (figure 3C), estimant que la réponse des progéniteurs du foie au ligand dépend également de la contexte ECM. Enfin, nous avons utilisé shRNA knockdown pour générer progéniteurs du foie sans les ligands DLL1 et JAG1. La réponse au ligand de Notch revêtu variait en fonction de la prEsence soit un ligand, ce qui confirme que la réponse au ligand cellulaire extrinsèque est également fonction du ligand expression de cellule intrinsèque (figure 3D). De plus, nous avons observé une sous - population distincte de la double-positive (ALB + / OPN +) cellules dans le knockdown DLL1 (Figure 3D). Ensemble, ces résultats représentatifs montrent: (1) les capacités combinatoires du format de tableau, comme en témoigne l'appariement de multiples protéines revêtu d'ECM et des ligands Notch avec le knockdown de ligands individuels; (2) la fonctionnalité non seulement revêtit protéines ECM, mais aussi revêtit ligand cellule-cellule par l'intermédiaire de la protéine A / G à médiation par conjugaison; et (3) la mise en œuvre de notre analyse unicellulaire et sa capacité à discerner les sous-populations uniques.
Nous avons également observé que la différenciation des progéniteurs du foie dépend à la fois de la rigidité du substrat et la composition de l' ECM (figure 4A ong>), trouver spécifiquement que le collagène IV est favorable à la différenciation des deux substrats souples et rigides alors que la fibronectine ne supporte que la différenciation sur des substrats rigides (figure 4B). Cartes de chaleur représentatifs de mesures TFM ont suggéré que le stress de traction soutenue à faible rigidité du substrat sur le collagène IV une différenciation dans les cellules des canaux biliaires (figure 4C), une constatation confirmée par des valeurs racine carrée moyenne moyenne (Figure 4D). Ensemble, ces résultats représentatifs montrent: (1) l'intégration réussie de TFM avec des microréseaux de cellules sur des substrats avec une rigidité accordable pour évaluer à la fois le phénotype cellulaire et la contrainte de traction; (2) la coordination du sort de progéniteurs du foie à la fois avec la composition de matrice et la rigidité du substrat; et (3) la mise en œuvre de nos analyses et typiques profils de contraintes de traction TFM en microréseaux cellulaires.
e 1 "src =" / files / ftp_upload / 55362 / 55362fig1.jpg "/>
Figure 1: Vue d' ensemble schématique montrant les trois premières sections expérimentales. Dans la section 1, des substrats en verre sont nettoyés et silanisées pour faciliter la fabrication d'hydrogels de polyacrylamide. Dans la section 2, les combinaisons de biomolécules d'intérêt sont préparés dans une source microplaques 384 puits. Un arrayer robotique est ensuite chargé avec des épingles propres, la microplaque source, et les hydrogels de polyacrylamide et initialisée, la fabrication des tableaux sur les hydrogels. Dans la section 3, les cellules sont ensemencées sur des domaines disposés en réseau et on les laisse adhérer, après quoi le protocole de culture d'intérêt est effectuée. Au point final, les cellules sont soit fixes pour immunocytochimie / immunofluorescence ou analysées en utilisant TFM. Les barres d'échelle sont de 75 um. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
dans les pages = "1"> 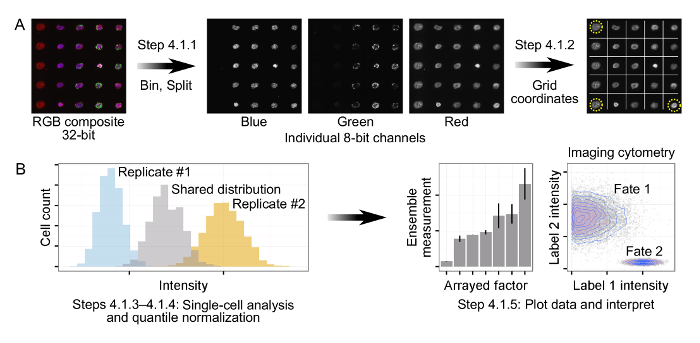
Figure 2: Traitement et analyse des données immunofluorescence de tableaux. (A) sol, composites 32 bits des images RVB sont d' abord mis en cellule et ensuite divisé en différents canaux de 8 bits. En utilisant une combinaison de marqueurs fluorescents disposés et îlots de cellules, trois coins de la matrice sont identifiés pour permettre l'orientation automatique et maillage des réseaux. (B) les données à l' unité de cellules est généré pour chaque canal des tableaux d'entrée. Afin de tenir compte de la dérive expérimentale, la normalisation quantile est appliquée par répliquée biologique, produisant une distribution unique partagée entre toutes les répétitions. Quantile données normalisées est ensuite tracée et interprétée par le calcul des mesures d'ensemble (par exemple, les cellules / île, intensité moyenne, les cellules de pourcentage positives pour une étiquette) ou d' analyse directe des distributions unicellulaires.m / files / ftp_upload / 55362 / 55362fig2large.jpg "target =" _ blank "> S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
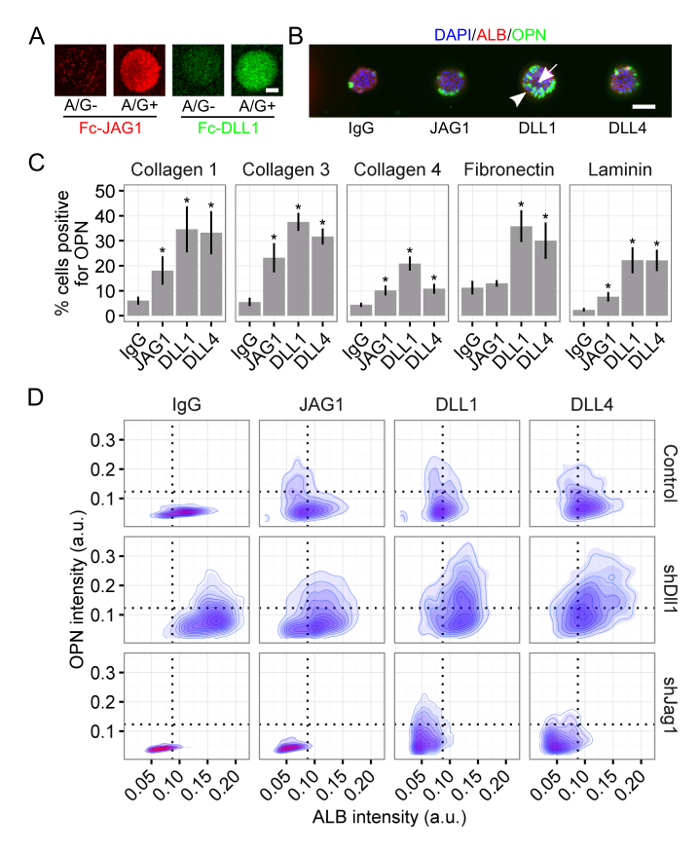
Figure 3: Notch Ligand Présentation du foie Progéniteur Différenciation à titre de médiateur. (A) Fc recombinant Notch ligands Jagged-1 (JAG1) et Delta-like 1 (DLL1) présentaient une meilleure rétention et le regroupement lorsque revêtit avec de la protéine A / G. La barre d'échelle est de 50 um. Progéniteurs (B) , du foie différenciées en cellules du canal cholédoque , sur présentation d' un ligand Notch. 4 ', 6-diamidino-2-phénylindole (DAPI) est un marqueur nucléaire, l'albumine (ALB) est un marqueur cellulaire hépatique, et l'ostéopontine (OPN) est un marqueur des cellules du canal cholédoque. La barre d'échelle est de 150 um. (C) Quantification du pourcentage de cellules positives pour OPN pour les ligands de Notch JAG1, DLL1 et Delta-like 4 (DLL4) sur les protéines ECM collagène I, collagen III, le collagène IV, la fibronectine et la laminine. T Les -Tests de Student ont été effectuées contre le contrôle IgG pour chaque ligand Notch rangé dans chaque protéine ECM avec P-valeurs indiquées pour P <0,05 (*). (D) Imaging cytométrie de ALB et OPN pour les cellules sur le collagène III présentées avec les ligands de Notch JAG1, DLL1 et DLL4. Progéniteurs hépatiques sans Notch ligands DLL1 et JAG1 (ie, shDll1 et shJag1) ont été générés en utilisant shRNA knockdown. Les données de (C) présentés sous forme de moyenne ± ETM Ce chiffre a été modifié à partir de Kaylan et al. 34. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
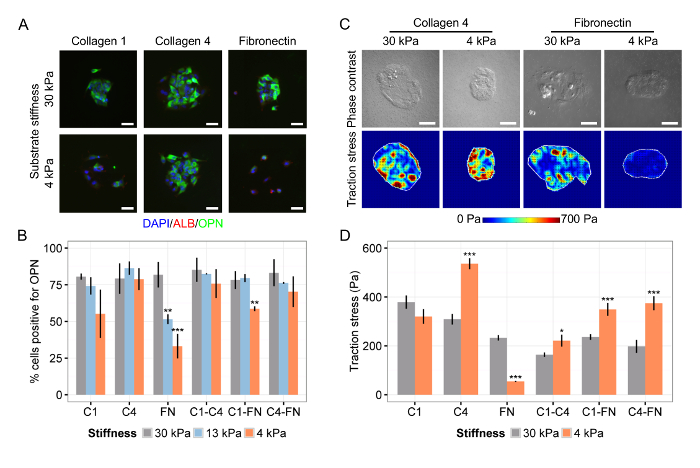
Figure 4: Matrix Composition et Substrat Rigidité Coordinate Liver Progéniteur Différenciation. (A) la différenciation hépatique progéniteurs biliaires cellules de canal dépend à la fois la composition de l' ECM et la rigidité du substrat. DAPI est un marqueur nucléaire, ALB est un marqueur cellulaire hépatique, et OPN est un marqueur des cellules du canal cholédoque. (B) Quantification du pourcentage de cellules positives pour l' OPN sur des substrats de module d'Young 30 kPa, 13 kPa et 4 kPa pour le collagène I (C1), le collagène IV (C4), la fibronectine (FN), et toutes bidirectionnelles combinaisons de ces protéines de la MEC. (C) Contrainte de traction cellulaire dépend à la fois la rigidité du substrat et de la composition ECM. (D) Quantification des valeurs racine carrée moyenne de stress de traction sur des substrats de module de Young 30 kPa et 4 kPa pour le collagène I (C1), le collagène IV (C4), la fibronectine (FN), et toutes les combinaisons dans les deux sens de ceux protéines ECM. En (B) et (D), les données ont été présentées sous forme de moyenne ± t les -Tests de MEB et étudiantsont été réalisées contre 30 kPa pour chaque combinaison d'ECM avec les valeurs P indiquées pour P <0,05 (*) P <0,01 (**), et P <0,001 (***). Les barres d'échelle sont de 50 um. Ce chiffre a été modifié depuis Kourouklis et al. 35. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
| Section | Problème | Causes possibles | Solution |
| 1. Fabrication de polyacrylamide Substrat. | Coverglass ne peut pas être retiré de l'hydrogel. | Surpolymérisation. | Réduire le temps de polymérisation à <10 minutes (4 W / m 2). Vérifier que crossli UVsortie nker est à portée attendue. |
| Une mauvaise polymérisation d'hydrogel de polyacrylamide. | Underpolymerization. | Augmenter le temps de 10 minutes (> 4 W / m 2) de polymérisation. Vérifiez que la sortie de réticulation UV est à portée attendue. | |
| hydrogels polyacrylamide sont endommagés après le retrait de lamelle. | hydrogels de polyacrylamide souples sont faciles à endommager. | Nous observons rendement décroissant hydrogel de fabrication (~ 50%) pour le plus doux (soit 4 kPa) hydrogels en particulier. Manipuler hydrogels doucement et augmenter le nombre de départ pour atteindre le rendement souhaité. | |
| 2. Fabrication de tableaux. | Pauvre ou incohérente morphologie du spot. | fonction de l'humidificateur Incohérence. | Vérifier que l'humidificateur et RHÉOMÈTRE fonctionnel tout au long de chaque cycle d'impression et de maintenir 65% d'humidité relative. |
| Pins coincé dans la tête d'impression ou de sabotged. | Nettoyez la tête d'impression pour permettre le mouvement de broche libre. broches Nettoyer soigneusement avant ou après chaque cycle d'impression pour éliminer les agrégats de canaux de broches. | ||
| 3. Culture cellulaire et test d'exécution. | détachement de la cellule ou de mort sur les tableaux après fixation initiale. | Sursemis et la prolifération excessive. | Réduire la densité de semis initiale et le temps. Utilisez "maintenance" ou "différenciation" des médias lors de la culture de réseau pour réduire la prolifération cellulaire. |
| Libération de toxique acrylamide monomère hydrogel. | Faire tremper les hydrogels dans dH 2 O pendant au moins 3 jours pour permettre la diffusion / libération de monomère d'acrylamide et de réduire la toxicité cellulaire. | ||
| Les cellules ne fixent pas les tableaux. | -Ensemencement. | Augmenter la densité de semis initiale et le temps. Utilisez un type plus fortement adhérente de cellules. | |
| Mauvais dépôt dela matrice ou de l'état de biomolécules. | Repères propres des particules et des agrégats, confirment les paramètres d'impression, et évaluer détachage des marqueurs fluorescents, par exemple, le dextrane conjugué à la rhodamine. | ||
| Spécificité des interactions cellule-matrice. | Différents types de cellules adhèrent spécifiquement à certains mais pas d'autres protéines de la MEC. Testez plusieurs protéines ECM différentes avec vos cellules. | ||
| stockage réseau Suboptimal après fabrication. | Nous recommandons le stockage des tableaux fabriqués nuit à 65% d'humidité relative et de la température ambiante, en partie pour éviter des changements de phase lors de la congélation. L'adhésion cellulaire est sensible à la fois l'humidité, la température et le temps de stockage; assurez-vous que ces paramètres sont compatibles / optimisé pour vos expériences. | ||
| Détachement d'hydrogel à partir du substrat de verre au cours de la culture cellulaire. | Pauvre diapositive nettoyage et silanisation. | Remplacer les solutions de travail pour le nettoyage de diapositives etsilanisation. | |
| hydrogel Overdehydrated. | Ne pas laisser hydrogels déshydratant sur une plaque chauffante pendant plus de 15-30 min. | ||
| 4. Analyse des données. | La forte variabilité entre les taches et les diapositives répliqués. | Variabilité dans le tableau de fabrication. | Vérifiez que les broches et les têtes d'impression sont propres. Confirmer la fonction de l'humidificateur. Visualiser et quantifier place et réseau de qualité en utilisant des marqueurs fluorescents. réseaux de magasins comme recommandé ci-dessus. |
Tableau 1: Dépannage.
Discussion
Dans nos expériences, nous avons constaté que les défaillances les plus courantes sont liées à la qualité des matrices fabriquées et mal caractérisés réponse dans le système biologique d'intérêt. Nous renvoyons le lecteur au tableau 1 pour les modes de défaillance communs dans les expériences de puces à ADN cellulaire et les étapes de dépannage associées. En ce qui concerne la qualité des réseaux, en particulier, nous recommandons ce qui suit. Confirmer la qualité technique et la robustesse des programmes, paramètres rangeant, et des tampons en utilisant des molécules marquées par fluorescence tels que la rhodamine conjuguée dextran. broches Nettoyer soigneusement avant ou après les rangeant par les instructions du fabricant et en outre vérifier visuellement que les canaux de broches sont claires des débris à l'aide d'un microscope optique. Confirmer la rétention de biomolécules Arrayed utilisant les taches de protéines générales ou immunomarquage. A noter que les biomolécules ayant un poids moléculaire inférieur à 70 kDa sont souvent pas conservés dans 23 l'hydrogel, sup> 31. Valider biomolécules Arrayed cellule-fonctionnalité en utilisant plusieurs types de cellules. Notez que seules les cellules adhérentes sont compatibles avec les tableaux; En outre, l' adhésion à des réseaux dépend à la fois des propriétés spécifiques à des cellules (par exemple, le profil d'expression de l' intégrine) et les protéines d'ECM sélectionnées.
En raison de l' espace limité, nous avons pas fourni un traitement extensif de conception de réseau, mise en page, et la fabrication ici et renvoyer le lecteur aux travaux antérieurs 23, 25. Nous utilisons généralement 100 sous - réseaux au comptant (150 diamètre um spot, 450 um de distance de centre à centre) composé de 10-20 conditions de biomolécules uniques (c. -à- 5-10 points / état). Le nombre de sous-réseaux dans un réseau varie en fonction du nombre de conditions biomolécules d'intérêt, qui peut être confortablement étendu pour 1.280 sur un 25 × 75 mm lame de microscope (~ 6400 points dans 64 sous-réseaux)xref "> 25 31 Les paramètres ci - dessus en outre varier en fonction de la taille du motif d'intérêt;. broches capables de générer des modèles de 75 à 450 um sont facilement disponibles.
expériences de tableau sont mieux complétés par la validation de haute notation conditions disposées d'intérêt en utilisant d'autres formats de culture, des lectures d'essai, et des systèmes de modèles biologiques. Plus précisément, nous vous recommandons de valider davantage les effets des conditions choisies disposées en utilisant des cultures en vrac conjointement avec des techniques standard de biologie moléculaire (par exemple, qRT-PCR, immunoblotting) ou la norme TFM. La manipulation génétique (par exemple, knockdown ou surexpression) du facteur d'intérêt dans un système modèle biologique approprié peut également servir à confirmer les effets observés dans des tableaux. In vivo des modèles animaux représentent un autre moyen de validation et ont été récemment utilisés, par exemple, de confirmer le rôle central de la galectine-3 et la galectine-8le cancer du poumon métastatiques créneau, comme initialement identifié par puce à ADN de la cellule 31, 49.
Un certain nombre d'autres méthodes ont été utilisées pour sonder la régulation microenvironnement de fonctions cellulaires, y compris une variété de bidimensionnelles systèmes micro - usinés 18, 50, 51, 52, 53, 54, 55 et des systèmes tridimensionnels biomatériau d' ingénierie 56, 57, 58 , 59, 60, 61. En comparaison avec d'autres méthodes, les avantages particuliers de la plate-forme de puces à ADN cellulaire décrits ici comprennent: (1) Débit jusqu'àdes centaines ou des milliers de différentes combinaisons de facteurs, ce qui permet l'analyse des effets de l'interaction; (2) accessible, l'imagerie et l'analyse automatisée; (3) l'intégration des deux lectures biochimiques et biophysiques avec présentation contrôlée de facteurs disposés; (4) l'aptitude à faire varier les propriétés du matériau du substrat; et (5) à haute teneur en analyse unicellulaire du destin et de la fonction cellulaire.
En résumé, la combinaison des microréseaux cellulaires avec TFM sur des substrats de rigidité du substrat accordable permet la caractérisation complète des deux indices biochimiques et biophysiques. Tel que présenté ici, cette plate-forme est généralisable et peut être facilement appliquée à une variété de types de cellules adhérentes et des contextes de tissus vers une meilleure compréhension de la régulation de microenvironnement combinatoire de la différenciation cellulaire et la mécanotransduction.
Déclarations de divulgation
Les auteurs déclarent qu'ils ont aucun intérêt financier concurrents.
Remerciements
Nous reconnaissons Austin Cyphersmith et Mayandi Sivaguru (Carl R. Institut Woese génomique de la biologie, Université de l'Illinois à Urbana-Champaign) pour l'assistance à la microscopie et généreusement accueillir l'écran et la capture vidéo au cœur de la microscopie.
matériels
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | Pall Corporation | 4433 | Match with appropriately-sized Luer lock plastic syringes. |
| 100 × penicillin–streptomycin solution | Fisher Scientific | SV30010 | |
| 22 × 60 mm coverglasses | Electron Microscopy Sciences | 63765 | |
| 3-(trimethoxysilyl)propyl methacrylate (3-TPM) | Sigma-Aldrich | 440159 | Store under inert gas per manufacturer's instructions. Exposure of 3-TPM to air could compromise silanization of glass substrates. CAUTION: 3-TPM is a combustible liquid. Keep away from heat, sparks, open flames, and hot surfaces and use only in a chemical fume hood. |
| 3-[(3-Cholamidopropyl)dimethylammonio]-1-propanesulfonate hydrate (CHAPS) | Sigma-Aldrich | C3023 | |
| 35 mm glass-bottom Petri dishes | Cell E&G | GBD00002-200 | 13 mm well consisting of #1.5 coverglass. Enables TFM and live-cell imaging. |
| 384-well polypropylene V-bottom microplate, non-sterile | USA Scientific | 1823-8400 | |
| 6-well polystyrene microplates | Fisher Scientific | 08-772-1B | 35 mm glass-bottom Petri dishes fit into wells of microplate, easing array fabrication. |
| Acetone | Sigma-Aldrich | 179973 | |
| Acrylamide | Sigma-Aldrich | A3553 | CAUTION: Exposure to acrylamide can result in acute toxicity and irritation. Wear protective gloves, clothing, and eye protection. |
| Collagen I, rat tail | EMD Millipore | 08-115MI | |
| Collagen III, human | EMD Millipore | CC054 | |
| Collagen IV, human | EMD Millipore | CC076 | |
| Crosslinker, 365 nm | UVP | CL-1000 | |
| Dextran, rhodamine B-conjugated, 70 kDa | ThermoFisher Scientific | D1841 | Used as a marker for array location. |
| Dimethyl sulfoxide | Fisher Scientific | BP231 | |
| Dulbecco's phosphate-buffered saline (PBS) | Fisher Scientific (HyClone) | SH3001302 | |
| Ethyl alcohol | Decon Labs | 2701 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | ED | |
| Fc-recombinant DLL1, mouse | R&D Systems | 5026-DL-050 | |
| Fc-recombinant DLL4, mouse | AdipoGen | AG-40A-0145-C050 | |
| Fc-recombinant JAG1, rat | R&D Systems | 599-JG-100 | |
| Fibronectin, human | Sigma-Aldrich | F2006 | |
| Fluorescent microscope, inverted | Zeiss | Axiovert 200M | Ensure microscope is equipped with a robotic stage for both automated fluorescent imaging and TFM. Environmental control (i.e., 37 °C and 5% CO2) is highly advisable for TFM. |
| Fluoromount G with DAPI | SouthernBiotech | 0100-20 | |
| Glacial acetic acid | Sigma-Aldrich | 695092 | CAUTION: Acetic acid is flammable and corrosive. Wear protective gloves, clothing, and eye protection. |
| Glycerol | Sigma-Aldrich | M6145 | |
| Irgacure 2959 | BASF Corporation | 55047962 | |
| Laminin, mouse | EMD Millipore | CC095 | |
| Methanol | Sigma-Aldrich | 179957 | |
| Microarray scanner | GenePix | 4000B | Fluorophores must be Cy3- or Cy5-compatible. |
| Microarrayer | Digilab | OmniGrid Micro | Other microarrayers of similar or greater capability can readily be substituted. |
| Microscope slides, 25 × 75 mm | Sigma-Aldrich | CLS294775X25 | ~0.9 – 1.1 mm thickness. |
| N,N′-Methylenebisacrylamide (bisacrylamide) | Sigma-Aldrich | M7279 | CAUTION: Exposure to acrylamide can result in acute toxicity and irritation. Wear protective gloves, clothing, and eye protection. |
| Paraformaldehyde (PFA), 16% v/v | Electron Microscopy Sciences | RT15710 | Prepare PFA fresh (do not store) for optimal fixation. CAUTION: Exposure to PFA can result in acute toxicity and can also irritate or corrode skin on contact. Wear protective gloves, clothing, and eye protection and use only in a chemical fume hood. |
| Protein A/G, recombinant | ThermoFisher Scientific | 21186 | |
| Pyrex drying tray, 2,000 mL | Fisher Scientific | 15-242B | |
| Rectangular 4-chambered culture dish | Fisher Scientific (Nunc) | 12-565-495 | For cell culture on arrayed microscope slides. |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | 415413 | CAUTION: NaOH is highly caustic and can cause severe skin burns and eye damage. Wear protective gloves, clothing, and eye protection. |
| Stealth pin for arraying | ArrayIt | SMP3 | Clean pins after each array run using the instructions of the manufacturer. Produces 150 micron domains; purchase other pin sizes (75–450 microns) as suited to your particular application. |
| Triton X-100 | Sigma-Aldrich | X100 |
Références
- Joyce, J. A., Pollard, J. W. Microenvironmental regulation of metastasis. Nat Rev Cancer. 9 (4), 239-252 (2009).
- Hsu, Y. C., Fuchs, E. A family business: stem cell progeny join the niche to regulate homeostasis. Nat Rev Mol Cell Biol. 13 (2), 103-114 (2012).
- Whiteside, T. L. The tumor microenvironment and its role in promoting tumor growth. Oncogene. 27 (45), 5904-5912 (2008).
- Jones, D. L., Wagers, A. J. No place like home: anatomy and function of the stem cell niche. Nat Rev Mol Cell Biol. 9 (1), 11-21 (2008).
- Discher, D. E., Mooney, D. J., Zandstra, P. W. Growth factors, matrices, and forces combine and control stem cells. Science. 324 (5935), 1673-1677 (2009).
- Trappmann, B., et al. Extracellular-matrix tethering regulates stem-cell fate. Nat Mater. 11 (7), 642-649 (2012).
- Ivanovska, I. L., Shin, J. W., Swift, J., Discher, D. E. Stem cell mechanobiology: diverse lessons from bone marrow. Trends Cell Biol. 25 (9), 523-532 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Chaudhuri, O., et al. Hydrogels with tunable stress relaxation regulate stem cell fate and activity. Nat Mater. 15 (3), 326-334 (2016).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Dev Cell. 6 (4), 483-495 (2004).
- Legate, K. R., Wickstrom, S. A., Fassler, R. Genetic and cell biological analysis of integrin outside-in signaling. Genes Dev. 23 (4), 397-418 (2009).
- Kessenbrock, K., Plaks, V., Werb, Z. Matrix metalloproteinases: regulators of the tumor microenvironment. Cell. 141 (1), 52-67 (2010).
- Underhill, G. H. Stem cell bioengineering at the interface of systems-based models and high-throughput platforms. Wiley Interdiscip Rev Syst Biol Med. 4 (6), 525-545 (2012).
- Underhill, G. H., Galie, P., Chen, C. S., Bhatia, S. N. Bioengineering methods for analysis of cells in vitro. Annu Rev Cell Dev Biol. 28, 385-410 (2012).
- Zorlutuna, P., et al. Microfabricated biomaterials for engineering 3D tissues. Adv Mater. 24 (14), 1782-1804 (2012).
- Ruiz, S. A., Chen, C. S. Microcontact printing: A tool to pattern. Soft Matter. 3 (2), 168-177 (2007).
- Guillotin, B., Guillemot, F. Cell patterning technologies for organotypic tissue fabrication. Trends Biotechnol. 29 (4), 183-190 (2011).
- Théry, M. Micropatterning as a tool to decipher cell morphogenesis and functions. J. Cell Sci. 123 (Pt 24), 4201-4213 (2010).
- Ranga, A., Lutolf, M. P. High-throughput approaches for the analysis of extrinsic regulators of stem cell fate. Curr. Opin. Cell Biol. 24 (2), 236-244 (2012).
- Kobel, S., Lutolf, M. High-throughput methods to define complex stem cell niches. Biotechniques. 48 (4), ix-xxii (2010).
- Fernandes, T. G., Diogo, M. M., Clark, D. S., Dordick, J. S., Cabral, J. M. S. High-throughput cellular microarray platforms: applications in drug discovery, toxicology and stem cell research. Trends Biotechnol. 27 (6), 342-349 (2009).
- Montanez-Sauri, S. I., Beebe, D. J., Sung, K. E. Microscale screening systems for 3D cellular microenvironments: platforms, advances, and challenges. Cell Mol Life Sci. 72 (2), 237-249 (2015).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat Methods. 2 (2), 119-125 (2005).
- Underhill, G. H., Flaim, C. J., Bhatia, S. N. Methods in Bioengineering: Stem Cell Bioengineering Artech House Methods in Bioengineering. Parekkadan, B., Yarmush, M. , Artech House Publishers. Boston, MA. 63-73 (2009).
- Brafman, D. A., Chien, S., Willert, K. Arrayed cellular microenvironments for identifying culture and differentiation conditions for stem, primary and rare cell populations. Nat Protoc. 7 (4), 703-717 (2012).
- Brafman, D. A., et al. Investigating the role of the extracellular environment in modulating hepatic stellate cell biology with arrayed combinatorial microenvironments. Integr Biol. 1 (8-9), 513-524 (2009).
- Soen, Y., Mori, A., Palmer, T. D., Brown, P. O. Exploring the regulation of human neural precursor cell differentiation using arrays of signaling microenvironments. Mol Syst Biol. 2, 37(2006).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integr Biol. 1 (1), 70-79 (2009).
- Anderson, D. G., Levenberg, S., Langer, R. Nanoliter-scale synthesis of arrayed biomaterials and application to human embryonic stem cells. Nat Biotechnol. 22 (7), 863-866 (2004).
- Brafman, D. A., Shah, K. D., Fellner, T., Chien, S., Willert, K. Defining long-term maintenance conditions of human embryonic stem cells with arrayed cellular microenvironment technology. Stem Cells Dev. 18 (8), 1141-1154 (2009).
- Reticker-Flynn, N. E., et al. A combinatorial extracellular matrix platform identifies cell-extracellular matrix interactions that correlate with metastasis. Nat Commun. 3, 1122(2012).
- Wood, K. C., et al. MicroSCALE screening reveals genetic modifiers of therapeutic response in melanoma. Sci Signal. 5 (224), rs4(2012).
- Braga Malta, D. F., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kaylan, K. B., Ermilova, V., Yada, R. C., Underhill, G. H. Combinatorial microenvironmental regulation of liver progenitor differentiation by Notch ligands, TGFbeta, and extracellular matrix. Sci Rep. 6 (23490), 23490(2016).
- Kourouklis, A. P., Kaylan, K. B., Underhill, G. H. Substrate stiffness and matrix composition coordinately control the differentiation of liver progenitor cells. Biomaterials. 99, 82-94 (2016).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integr. Biol. , (2016).
- Mann, C., Leckband, D. Measuring Traction Forces in Long-Term Cell Cultures. Cellular and Molecular Bioengineering. 3 (1), 40-49 (2010).
- Heisenberg, C. P., Bellaiche, Y. Forces in tissue morphogenesis and patterning. Cell. 153 (5), 948-962 (2013).
- Schwarz, U. S., Soine, J. R. Traction force microscopy on soft elastic substrates: A guide to recent computational advances. Biochim Biophys Acta. 1853 (11 Pt B), 3095-3104 (2015).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology-Cell Physiology. 282 (3), C595-C605 (2002).
- Wen, J. H., et al. Interplay of matrix stiffness and protein tethering in stem cell differentiation. Nat Mater. 13 (10), 979-987 (2014).
- Knoll, S. G., Ali, M. Y., Saif, M. T. A novel method for localizing reporter fluorescent beads near the cell culture surface for traction force microscopy. J Vis Exp. (91), e51873(2014).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Bolstad, B. M., Irizarry, R. A., Astrand, M., Speed, T. P. A comparison of normalization methods for high density oligonucleotide array data based on variance and bias. Bioinformatics. 19 (2), 185-193 (2003).
- Wang, N., et al. Cell prestress. I. Stiffness and prestress are closely associated in adherent contractile cells. Am J Physiol Cell Physiol. 282 (3), C606-C616 (2002).
- Krishnan, R., et al. Substrate stiffening promotes endothelial monolayer disruption through enhanced physical forces. Am J Physiol Cell Physiol. 300 (1), C146-C154 (2011).
- Reticker-Flynn, N. E., Bhatia, S. N. Aberrant glycosylation promotes lung cancer metastasis through adhesion to galectins in the metastatic niche. Cancer Discov. 5 (2), 168-181 (2015).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric control of cell life and death. Science. 276 (5317), 1425-1428 (1997).
- Kilian, K. A., Bugarija, B., Lahn, B. T., Mrksich, M. Geometric cues for directing the differentiation of mesenchymal stem cells. Proc Natl Acad Sci USA. 107 (11), 4872(2010).
- Nelson, C. M., Chen, C. S. Cell-cell signaling by direct contact increases cell proliferation via a PI3K-dependent signal. FEBS Lett. 514 (2-3), 238-242 (2002).
- Hui, E. E., Bhatia, S. N. Micromechanical control of cell-cell interactions. Proc. Natl. Acad. Sci. U. S. A. 104 (14), 5722-5726 (2007).
- Lutolf, M. P., Blau, H. M. Artificial stem cell niches. Adv Mater. 21 (32-33), 3255-3268 (2009).
- Gobaa, S., et al. Artificial niche microarrays for probing single stem cell fate in high throughput. Nat. Methods. 8 (11), 949-955 (2011).
- DeForest, C. A., Anseth, K. S. Cytocompatible click-based hydrogels with dynamically tunable properties through orthogonal photoconjugation and photocleavage reactions. Nat Chem. 3 (12), 925-931 (2011).
- Nelson, C. M., VanDuijn, M. M., Inman, J. L., Fletcher, D. A., Bissell, M. J. Tissue geometry determines sites of mammary branching morphogenesis in organotypic cultures. Sci. STKE. 314 (5797), 298(2006).
- Liu Tsang, V., et al. Fabrication of 3D hepatic tissues by additive photopatterning of cellular hydrogels. FASEB J. 21 (3), 790-801 (2007).
- Albrecht, D. R., Underhill, G. H., Mendelson, A., Bhatia, S. N. Multiphase electropatterning of cells and biomaterials. Lab. Chip. 7 (6), 702-709 (2007).
- Chan, V., Zorlutuna, P., Jeong, J. H., Kong, H., Bashir, R. Three-dimensional photopatterning of hydrogels using stereolithography for long-term cell encapsulation. Lab. Chip. 10 (16), 2062-2070 (2010).
- Boghaert, E., et al. Host epithelial geometry regulates breast cancer cell invasiveness. Proc. Natl. Acad. Sci. U. S. A. 109 (48), 19632-19637 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon