Method Article
Protocole de différenciation cérébelleux 3D simplifiée avec Modification 2D facultative
Dans cet article
Résumé
Nous décrivons un support de protocole de hPSCs, à l’aide de défini différenciation 3D simplifié et réduit les facteurs de croissance, capables de produire des agrégats cellulaires avec les premières structures neuroépithéliales et positifs pour les marqueurs associés à cervelet, ainsi qu’une option Modification 2D afin de différencier les cellules comme une monocouche pour générer des neurones fonctionnels.
Résumé
Réduire la complexité et le coût des protocoles de différenciation est important pour les chercheurs. Cet intérêt s’inscrit dans les préoccupations au sujet de possibles effets inattendus que facteurs extrinsèques structuration pourraient introduire dans les cellules souches pluripotentes humaines (hPSC) des modèles de développement du cerveau ou de la physiopathologie, comme masquant le phénotype de la maladie. Nous présentons ici deux protocoles de différenciation cérébelleux pour hPSCs, conçu avec la méthode de démarrage plus simple, moins des facteurs de répétition et moins les exigences matérielles que les protocoles antérieurs. Récemment, nous avons développé les méthodes de culture, qui génèrent flottants 3 Dimensions (3D) produits compatibles avec d’autres protocoles « organoïde » cerveau, y compris les morphologies rhombiques et pertinents à la modélisation de développement du cerveau tels que zone ventriculaire/sub- structures ressemblant à des lèvres. La seconde utilise une procédure de monocouche adhérentes, 2D de différenciation complète, ce qui est montrée capable de générer des neurones cérébraux fonctionnels, comme les produits sont positifs pour les marqueurs associés à cervelet et pièce influx calciques neuron-comme. Ensemble, ces protocoles offrent aux scientifiques un choix d’options adaptées à des fins de recherche différents, mais aussi un modèle de base pour tester d’autres types de différenciation neurale simplifiée.
Introduction
In vitro les protocoles permettant de différencier les hPSCs vers les lignées cérébelleuses initialement exploités sur le principe d’imiter en vivo développement cérébelleux1,2,3,4. À ce titre, ils exigeaient une succession de facteurs a présenté à des moments précis pour piloter la maturation et structuration pro-cérébelleux. Parmi ceux-ci étaient WNT, OS protéines morphogénétiques (PGB) et facteurs de croissance fibroblastiques (FGF) avec des rôles connus dans le cerveau Mid-postérieur développement et formation de l’organisateur Isthmiques5,6,7. Bien sûr, chaque étape supplémentaire et le facteur signifie une augmentation des manipulations fastidieuses et une plus grande dépense pour le chercheur, et ainsi développer des protocoles plus simples capables d’atteindre des résultats d’equal est intéressant. Cette question pratique concorde bien avec la question hypothétique, si les cellules ont besoin de ce contrôle serré, externe au cours de leur développement in vitro.
Pour une différenciation cérébelleuse, un protocole publié en 2015 adressée la nécessité d’utiliser un grand nombre de facteurs de croissance en utilisant seulement FGF2, FGF19 et facteur de culture cellulaire stromal 1 (SDF1) pour des fins8le patterning. Cette étude diffère également des protocoles cérébelleux antérieurs, en utilisant un système de culture 3D flottant. En plus de produire des cellules positifs pour les marqueurs cérébelleux, les cerveau « organoïdes » générés par leur technique se sont révélés pièce morphologie pertinente, non disponible dans certaines cultures traditionnelles monocouche 2D, comme rhombiques structures ressemblant à des lèvres. Bien que moins complexe et coûteux en ce qui concerne les facteurs de croissance, autres fonctionnalités telles que la formation de corps embryoïdes uniformes (EBs) et de la culture dans les plaques à 96 puits (96WPs), fait sur le plan procédural complexe au cours des étapes initiales. Un autre protocole 3D publiée la même année, a signalé la différenciation réussie aux lignées neurones à l’aide de commun et peu coûteux de cellule culture techniques9. Bien que ce groupe enquêtait corticaux plutôt que de différenciation cérébelleuse, application de leur concept de différenciation cérébelleuse ne pourrait pas être écartée.
Nous avons récemment rapporté un protocole de différenciation cérébelleux 3D à l’aide d’un nombre réduit de facteurs de structuration (à savoir, FGF2, 4 et 8), ainsi qu’une configuration simplifiée en gardant les cellules en plaques 6 puits (6WPs) tout au long de minimiser les exigences moyennes10. Pour faciliter la production de cellules étoilées, lissé agoniste (SAG) a été utilisé au cours de l’étape finale de la maturation. SAG est une alternative moins chère chimique à sonic hedgehog (SHH), qui avait été utilisée dans les protocoles antérieurs cérébelleux, en raison de son rôle dans la promotion de la croissance de granule cellules précurseurs (BPC) in vivo1,2, 11,12,13. Différenciation des produits étaient conformes à ceux d’autres protocoles 3D, y compris la présence de marqueurs associés à cervelet en structures morphologiquement concernées8,9. Ces résultats renforcent le message précédent qui détaille le mimétisme de l' in vivo environnement est peut-être pas nécessaire pour les protocoles de différenciation complexe 3D in vitro .
Outre le protocole 3D, ce rapport décrit un protocole de 2D, conçu avec la même configuration rapide, matériaux de base et réduit le nombre de facteurs de croissance. Il est capable de produire des cellules souches embryonnaires humaines (CSEh) ou induite par les cellules souches pluripotentes (hiPSCs), positifs pour les marqueurs associés à tôt neuronaux, cérébelleux et granule cell identités. En outre, imagerie calcique indique la présence de neurones humains fonctionnels. La possibilité de choisir entre les protocoles, ajoute un niveau de flexibilité pour les chercheurs, pour ceux qui souhaitent soit : (1) génération de cellule spécifique tape, le développement du cerveau humain (2) modélisation et associés structures, (3) l’analyse optimisée en monocouche paramètres (p. ex., enregistrements de patch clamp), ou les interactions cellule-cellule (4) dans des cultures mixtes neurales. Leur nature simple et à faible coût les rend accessible pour les chercheurs qui sont nouveaux dans le domaine de la hPSC, ou besoin de procédures de base hPSC d'où vous pourrez explorer davantage les options de différenciation.
Protocole
1. les préparatifs
Remarque : Pour toutes les étapes, voir Table des matières pour des éléments spécifiques.
-
Préparer 500 mL défini hPSC milieu de culture pour la culture de hPSC
Remarque : Utilisez le moyen pour obtenir la procédure 2,1 à 2,6.- Dégel hPSC moyenne supplément pendant la nuit (o/n) à 4 ° C. Retirer 12,5 mL de milieu de base hPSC de bouteille moyen, puis ajouter 10 mL de supplément et 2,5 mL (faire 100 U/L) pénicilline/streptomycine (Pen/Strep) à la bouteille.
- Conserver à 4 ° C et utiliser dans les 2 semaines.
NOTE : Le support de hPSC peut servir à congeler les cellules en ajoutant 10 µM ROCK inhibiteur (RI) et 10 % DMSO.
-
Préparer 1 L entretien neural milieu (NMM) pour la culture de la différenciation
Remarque : Utilisez le moyen pour obtenir la procédure 3.1-4.4.- Mix glutamine enrichie DMEM/F12 et milieu de base neurale (ratio 1:1) dans un 1 L bouteille, puis supplément avec supplément de N2 (1 x), (1 x) B27 supplément, 5 µg/mL d’insuline, 1,5 mM de L-glutamine, 100 µM non essentiels des acides aminés (NEAA), 100 U/L Pen/streptocoque et 10 µM bêta-mercaptoéthanol.
- Stocker un milieu à 4 ° C et utiliser dans les 3 semaines.
Remarque : Avant d’ajouter des suppléments au milieu basal mixte, retirer le volume approprié pour ajuster le volume requis de composants ajoutés, basé sur les concentrations de stock.
-
Préparer 500 mL de solution diluée de 0,5 mM EDTA pour passage hPSCs
Remarque : Utilisez le moyen pour obtenir la procédure 2.4 et 2.5.- Sous une hotte à flux, transférer 49 mL de Phosphate Buffered Saline (PBS) provenant d’une bouteille 500 mL de PBS stérile dans un tube de 50 mL. Ajouter 0,5 mL d’EDTA 0,5 M et 0,9 g de NaCl dans le tube de 50 mL. Mélanger doucement pour dissoudre.
- Solution de filtre-stériliser à l’aide d’un filtre à 0,22 µm et le transfert à la bouteille 500 mL de PBS stérile. Conserver à température ambiante (RT).
-
Préparer hPSC plaques de culture pour la culture de hPSC
Remarque : Utiliser les plaques pour obtenir la procédure 2.1-2.6.- Faites 50 x solution de revêtement adhérent adaptée à l’hPSC (PAAC) : Décongeler un flacon du PAAC o/n à 4 ° C. Diluer PAAC en rapport 1:1 avec DMEM/F12 et le transfert de 400 µL d’extraits dans des tubes de 1,5 mL. Magasin 50 x solution de travail PAAC à-80 ° C.
NOTE : (Important) PAAC se solidifie rapidement à la RT, il est donc nécessaire que tous les composants (DMEM, tubes, etc.) sont conservés sur la glace (ou à 4 ° C). - Décongeler le tube de 50 x solution de travail PAAC à 4 ° C, puis diluer 50 x en froid DMEM/F12. Ajouter 750 µL/puits du PAAC dilué à un 6WP. Incuber les plaques pendant au moins 1 h à 37 ° C.
Remarque : Les plaques PAAC peuvent être conservés pendant 1 semaine à 4 ° C, en enveloppant la plaque après la période d’incubation de 1 h. Assiette chaude à 37 ° C avant de l’utiliser.
- Faites 50 x solution de revêtement adhérent adaptée à l’hPSC (PAAC) : Décongeler un flacon du PAAC o/n à 4 ° C. Diluer PAAC en rapport 1:1 avec DMEM/F12 et le transfert de 400 µL d’extraits dans des tubes de 1,5 mL. Magasin 50 x solution de travail PAAC à-80 ° C.
-
Préparer les plaques anti-adhésives (AA) pour la culture de la différenciation
NOTE : Utiliser les plaques pour obtenir la procédure 3.1-4.1.- Faire 5 mg/mL Poly (méthacrylate de 2-hydroxyéthyle) solution de (poly-HEMA) à 95 % éthanol. Secouer o/n à 37 ° C jusqu'à obtention d’une solution claire. Magasin à température ambiante.
Remarque : Filtrant à travers un filtre à 0,22 µm peut retirer non dissous poly-HEMA. - Ajouter poly-HEMA à la plaque de culture afin qu’elle recouvre le fond de chaque puits. Incuber les plaques à 37 ° C pendant 2 jours et d’inspecter la plaque afin d’assurer l’évaporation complète du liquide et des revêtement de puits. AA plaques peuvent être emballés et stockés à température ambiante.
- Faire 5 mg/mL Poly (méthacrylate de 2-hydroxyéthyle) solution de (poly-HEMA) à 95 % éthanol. Secouer o/n à 37 ° C jusqu'à obtention d’une solution claire. Magasin à température ambiante.
-
Préparer les plaques Poly-L-Ornithine/laminine (PLO/LAM) pour la culture de la différenciation
NOTE : Utiliser les plaques pour obtenir la procédure 4.2-4.4.- Enduire la surface du puits, à l’aide de 20 µg/mL PLO, dissoute dans du PBS stérile. Incuber la plaque o/n à 37 ° C. Aspirer les PLO et rincer 3 fois avec du PBS.
Remarque : (Facultatif) Incubated plaque avec OLP peut être enveloppé et stocké à 4 ° C jusqu'à ce que nécessaire. - Enduire les surfaces des puits PLO-enduit, à l’aide de 10 µg/mL LAM dissoute dans du PBS stérile. Incuber pendant au moins 2 h à 37 ° C ou o/n à 4 ° C. Supprimer le LAM et laver les puits 2-3 x avec du PBS, puis immédiatement ajouter des milieux approprié et/ou des cellules.
NOTE : Solution enlevé LAM (facultatif) peut être stockée à 4 ° C et réutilisée jusqu'à 2 fois. (Important) Ne permettent pas de surfaces recouvertes LAM se dessécher ; pour empêcher ceci ajoutez immédiatement PBS ou milieu approprié.
- Enduire la surface du puits, à l’aide de 20 µg/mL PLO, dissoute dans du PBS stérile. Incuber la plaque o/n à 37 ° C. Aspirer les PLO et rincer 3 fois avec du PBS.
2. Protocole 1 : HPSC Feeder-free Culture
NOTE : CSEh proviennent d’une organisation non commerciale (ligne H01, voir Table des matières). Trois lignes de contrôle de hiPSC (hvs51, 60 et 88) ont été générés par la reprogrammation des fibroblastes de trois patients humains sains (fibroblastes ont été dérivées de donneurs anonymes et non identifiables et donc exemptés de l’approbation de l’IRB)10, 17.
- Maintenir les hPSCs en culture sans chargeur
- Après décongélation et hPSCs sur des plaques de PAAC dans un milieu hPSC de placage (Voir l’étape 2.2), maintenir hPSCs à 37 ° C, avec 5 % de CO2. Actualiser hPSC moyen par jour (Voir l’étape 2.3), sauf le jour après la décongélation ou passage et d’examiner les cellules au microscope (objectifs : 2.5x/0.06, 5 x / 0.12 Ph0, 10 x / 0.25 Ph1) d’observer la croissance de note et de cerner les domaines de la différenciation (Figure 3 , panneau gauche supérieur illustre exemple de différenciation).
- Passage de hPSCs tous les 3-4 jours, ou lorsque la culture atteint > confluence de 80 %. Si moins de 5 % de cellules pièce différenciation, utilisation normale hPSC passage de méthode (voir étape 2,4), sinon, utilisez la méthode douce (voir étape 2.5). Quand n’est plus nécessaire dans la culture, les hPSCs peuvent être congelés pour le stockage à long terme (Voir l’étape 2.6).
- Dégel hPSCs en milieu hPSC
- Transférer le volume requis de hPSC moyen de tubes stériles pour la décongélation (9 mL/cryogéniques tube) et de la plaque PAAC préparée à recevoir les cellules décongelées. Compléter le support dans les deux tubes avec 10 µM RI.
- Récupérer le cryotube de stockage2 LN et le déposer directement dans un bain d’eau (37 ° C). Quand seulement un petit reste de cristal de glace, retirez du bain-marie et transférer le contenu du tube cryogénique dans le tube pour le processus de dégel (volume total 10 mL). Centrifuger le tube à 290 x g pendant 5 min à température ambiante.
- Utiliser une pipette sérologique pour enlever le PAAC solution provenant des puits de la plaque PAAC (Voir l’étape 1.4) destiné à recevoir des cellules et ajouter le milieu hPSC avec 10 µM RI.
NOTE : (Important) ne pas aspirer solution PAAC avec une aiguille d’aspiration, ou il peut se solidifier et boucher les lignes vers la pompe à vide. - Éliminer le surnageant du tube et remettre en suspension des cellules dans un milieu hPSC avec 10 µM RI. Distribuer les cellules jusqu'à la tôle de la destination au ratio de 1 tube cryogénique/puits de 6WP. Incuber à 37 ° C, avec 5 % de CO2et ne pas actualiser moyen pendant 1 nuit.
Remarque : Mise en service des cellules à 5 % O2 peut augmenter la survie des cellules.
- HPSC moyen de rafraîchir
- Réchauffer le volume nécessaire de hPSC milieu dans un tube stérile à la droite ou dans un bain d’eau ; 2 mL/puits de 6WP est suggéré.
NOTE : (Facultatif) : en ajoutant une quantité supplémentaire de moyen de hPSC, le hPSCs peut rester une journée supplémentaire sans actualiser ; Toutefois, ne l’autorisent pas plus d’une fois par semaine. - Aspirer au moyen de puits contenant des hPSCs et ajouter des frais hPSC moyen.
- Culture de la hPSCs dans un incubateur à 37 ° C et 5 % de CO2.
- Réchauffer le volume nécessaire de hPSC milieu dans un tube stérile à la droite ou dans un bain d’eau ; 2 mL/puits de 6WP est suggéré.
- HPSCs de passage dans un milieu hPSC
- Transférer le volume requis de hPSC moyen à tubes stériles, pour le processus de passage et préparation de la plaque PAAC à recevoir des cellules de subcultures. Compléter le support de la plaque de destination avec 10 µM RI. Chauffer les tubes moyens à ta ou dans un bain d’eau.
NOTE : Préparation et manipulation diffèrera si en utilisant un matériau de revêtement autre que celle figurant dans la Table des matières. - Utiliser une pipette sérologique pour enlever le PAAC solution provenant des puits de la plaque PAAC (Voir l’étape 1.4) destiné à recevoir des cellules et ajouter le milieu hPSC avec 10 µM RI.
NOTE : (Important) ne pas aspirer la solution PAAC avec une aspiration d’aiguille, qu’il peut se solidifier et boucher les lignes vers la pompe à vide. - Aspirez le milieu des puits avec hPSCs pour être repiquées, laver deux fois avec 0,5 mM EDTA, les cellules puis ajouter 0,5 mM EDTA et incuber pendant 2 à 5 min à 37 ° C.
NOTE : 1 mL/puits de 6WP est un volume suffisant d’EDTA pour le lavage et l’incubation. - Vérifier les puits sous le microscope (objectifs : 2.5x/0.06, 5 x / 0.12 Ph0, 10 x / 0.25 Ph1). Si les cellules commencent à se détacher, aspirer la solution d’EDTA et cellules chasse libre utilisant le milieu hPSC.
NOTE : (Important) attention de ne pas enlever toute hPSC colonies en aspirant de l’EDTA (n’attendez pas jusqu'à ce que des colonies entières sont détacher). Ne pas rincer les cellules plus de 5 fois car cela peut nuire à hPSCs et affectent la pluripotence. Aussi, ne pas laisser les cellules dans un milieu hPSC avec RI avant rinçage des cellules provenant de puits, comme ils peuvent ré-adhérer à la plaque. - Basée sur la détermination empirique (habituellement associée à confluence, taille des colonies et des taux de croissance), transfert de hPSCs au puits de la plaque de destination un fendage ratio de 1:4-1:16 (c.-à-d., 1 puits de la plaque d’origine de 4 puits de la destination plaque). Incuber à 37 ° C, avec 5 % de CO2et ne pas actualiser moyen pendant 1 nuit.
NOTE : Fractionnement des ratios CTscan 01:16-01:20 sont possibles prévenir le surpeuplement et améliorer l’apparence des colonies.
- Transférer le volume requis de hPSC moyen à tubes stériles, pour le processus de passage et préparation de la plaque PAAC à recevoir des cellules de subcultures. Compléter le support de la plaque de destination avec 10 µM RI. Chauffer les tubes moyens à ta ou dans un bain d’eau.
- Passage hPSCs à l’aide de la méthode douce (méthode-G)
- Transférer le volume requis de hPSC moyen à tubes stériles, pour le processus de passaging et pour la préparation de plaque PAAC à recevoir des cellules de subcultures. Compléter le support de plaque de destination avec 10 µM RI. Tubes moyens chauds au RT, ou au bain-marie à 37 ° C.
- Pipette sérologique utilisation à supprimer PAAC solution de puits de la plaque PAAC (Voir l’étape 1.4) destiné à recevoir des cellules et ajouter le milieu hPSC avec 10 µM RI.
NOTE : (Important) ne pas aspirer PAAC avec une aiguille d’aspiration, ou il peut se solidifier et boucher des lignes à la pompe à vide. - Aspirer la moyenne provenant des puits avec hPSCs à être repiquées et laver les cellules deux fois à l’aide de 0,5 mM EDTA. Sur le deuxième lavage, attendre 30 s avant aspiration EDTA, puis ajouter 1 mL de PBS et incuber pendant 4-9 min à 37 ° C. En attendant, préparer un tube stérile avec 4 mL de PBS.
NOTE : 1 mL/puits de 6WP est un volume suffisant d’EDTA pour le lavage. - Vérifier les puits sous microscope (objectifs : 2.5x/0.06, 5 x / 0.12 Ph0, 10 x / 0.25 Ph1). Si les cellules commencent à se détacher, tapez doucement sur les côtés de la plaque pour aider les colonies libres. Lorsque > 50 % des colonies sont libres flottant, utiliser une pipette sérologique de 5 mL pour transférer des colonies dans 1 mL de PBS dans le tube contenant 4 mL de PBS (triturer pas).
NOTE : (Important) puisque le but est de nettoyer la culture hPSC, cocher pour déterminer si différencié cellules restent fixé à la plaque. En outre, il n’est pas nécessaire au passage de toutes les colonies dans un bien à l’aide de ce processus, afin que les colonies qui restent attachées peuvent être laissés. - Attendre 5-10 min à ta pour s’installer dans le tube, les cellules (centrifuger pas). Aspirer les PBS du tube, en prenant soin de ne pour supprimer hPSCs sédentaire. Soigneusement Resuspendre les cellules dans un milieu hPSC (triturer pas) et les cellules de transfert sur la plaque de destination un fendage ratio de 1:4-1:16. Incuber à 37 ° C, avec 5 % de CO2et ne pas actualiser moyen pendant 1 nuit.
- Gel vers le bas de hPSCs
- Selon la confluence, utiliser 1 bien de hPSCs, à 2-3 jours (maximum) dans la culture, à remplir 1-2 flacons de cryoconservation pour stockage à LN2.
- À la fin du passage (étape 2.5), utilisez 500 µL ou 1 mL de milieu hPSC pour transférer des cellules de 1 puits de 6WP pour 1 ou 2 flacons de cryoconservation, respectivement (500 µL/flacon). Dans chaque tube, ajouter 500 µL de 2 x gel moyen contenant hPSC moyens, 20 µM RI et 20 % DMSO.
Remarque : Les cellules (en option) peuvent être transférées directement dans 1 x gel médium à 1 mL/flacon. - Placer les tubes cryogéniques dans un récipient cryogénique (contenant l’isopropanol) préalablement refroidies à 4 ° C et le magasin immédiatement à-80 ° C.
- Le lendemain, transfert les tubes cryogéniques à un réservoir de2 LN pour le stockage à long terme.
3. Protocole 2 : 3D « organoïde » différenciation
-
Installation de différenciation avec G-méthode modifiée de passage des hPSCs
- Transférer le volume requis de NMM pour le nombre de puits de destination dans un tube stérile. Compléter avec 4 FGF2 ng/mL et 10 µM RI. Chauffer le tube moyen à ta, ou au bain-marie à 37 ° C.
Remarque : Selon la confluence des puits origination, hPSCs sont concentrés lors de la distribution à la plaque de destination en 2:1 ou 3:1 ratio (c.-à-d., 2 puits de la plaque d’origine 1 puits de la plaque de destination), avec un volume de fin de 2,5 mL/puits de 6WP. - Aspirer le milieu de puits contenant des hPSCs être différenciées et laver les cellules deux fois à l’aide de 0,5 mM EDTA. Sur le deuxième lavage, attendre 30 s avant aspiration EDTA, puis ajouter 1 mL de PBS et incuber pendant 4-9 min à 37 ° C. En attendant, préparer un tube stérile avec 4 mL de PBS.
NOTE : 1 mL/puits de 6WP est un volume suffisant d’EDTA pour le lavage. (Important) Il est préférable d’utiliser hPSCs qui n’étaient pas plus de 3 jours de culture après le dernier passage et au moins 1-2 passages après décongélation. - Vérifier les puits sous le microscope (objectifs : 2.5x/0.06, 5 x / 0.12 Ph0, 10 x / 0.25 Ph1). Si les cellules commencent à se détacher, libérer les cellules en tapant doucement sur les côtés de la plaque. Transférer les cellules dans un tube contenant 4 mL de PBS avec une pipette de 5 mL.
NOTE : Rinçage léger (Important) et la trituration est autorisé à démanteler les colonies et prélever des cellules lâches, mais ignore les cellules qui restent collés sur la plaque. - Laisser le tube de s’asseoir pendant 10 min à la droite, pour la séparation par gravité. Éventuellement, centrifuger les cellules légèrement (pas plus de 200 g à la droite, pendant 5 min) si nécessaire.
- Aspirer le PBS du tube, en prenant soin de ne pas pour supprimer hPSCs sédentaire, remettre en suspension les cellules en NMM avec 4 ng/mL FGF2 et 10 µM RI, puis de distribuer à la plaque de revêtement AA dans un ratio de 2:1 ou 3 / 1. Incuber à 37 ° C, avec 5 % de CO2et ne pas actualiser le milieu pendant 3 jours, sauf si requis (Voir l’étape 3.2.1).
NOTE : (Facultatif) pour des raisons pratiques de transfert de cellules, ajouter une partie du milieu de culture au puits de la plaque de destination avant d’être diffusés et remettre en suspension les cellules dans un plus petit volume. En outre, hPSCs peuvent être colorées et compté pour définir la densité de cellules départ exact. Toutefois, le dernier volume de la plaque de destination devrait être de 2,5 mL/puits de 6WP.
- Transférer le volume requis de NMM pour le nombre de puits de destination dans un tube stérile. Compléter avec 4 FGF2 ng/mL et 10 µM RI. Chauffer le tube moyen à ta, ou au bain-marie à 37 ° C.
-
Maintenir la différenciation flottant culture à 37 ° C (5 % CO2)
- Vérifier les plaques tous les jours pour les changements de couleur moyenne, accumulation de cellules mortes, agglutination et une adhésion bien à fond.
- En option : Quel que soit l’horaire de changement médiatique, rafraîchir (y compris les 3 premiers jours de no rafraîchissante) le support si elle s’est transformée jaune et suivez les instructions pour le changement moyen/mise à jour (étape 3.3). Si la majorité des cellules semble morte, suivez les instructions pour moyen changement/mise à jour avec séparation par gravité (étape 3.4).
NOTE : Formation d’EBs et la croissance en agrégats de grandes cellules sont attendus, mais les cellules et agrégats cellulaires peuvent s’agglutiner ensemble en grandes masses pas en raison de la croissance individuelle/prolifération. Si c’est observé, lumière de trituration pour briser les masses est admissibles. Si cellules commencent à adhérer à la surface de la plaque-AA, le restant flottant contenu du puits peut être transféré directement aux nouveaux puits ou transféré au cours du processus de changement moyen/actualisation. N’essayez pas de transférer des cellules qui ont adhéré à la plaque.
- En option : Quel que soit l’horaire de changement médiatique, rafraîchir (y compris les 3 premiers jours de no rafraîchissante) le support si elle s’est transformée jaune et suivez les instructions pour le changement moyen/mise à jour (étape 3.3). Si la majorité des cellules semble morte, suivez les instructions pour moyen changement/mise à jour avec séparation par gravité (étape 3.4).
- Le jour 3, modifier le milieu NMM avec 4 ng/mL FGF2. Actualiser le support de tous les autres jours.
- Jour 7, modifier le milieu NMM avec 1 µM d’acide rétinoïque (RA) et 100 ng/mL FGF8B 4 ng/mL FGF2. Actualiser le support de tous les autres jours.
NOTE : RA (Important) est sensible à la lumière. Protéger les échantillons de culture de RA de la lumière. - Le 14e jour, remplacez le milieu NMM avec 100 ng/mL FGF8B, 100 ng/mL FGF4 et 20 ng/mL FGF2. Actualiser le support de tous les autres jours.
- Jour 17, modifier le milieu NMM avec 100 ng/mL FGF8B. Actualiser le support de tous les autres jours.
- Au jour 21, remplacez le milieu NMM avec 100 ng/mL cerveau dérivée Neurotrophic Factor (BDNF) et 10 ng/mL gliales dérivé Neurotrophic Factor (GDNF). Actualiser le support de tous les autres jours.
- Le 28e jour, remplacez le milieu NMM avec 100 ng/mL BDNF et 10 ng/mL GDNF, 3 ng/mL SAG, 100 ng/mL facteur neurotrophique 3 (NT3) et 25 mM KCl. actualiser le support de tous les autres jours.
- Jour 35, recueillir les organoïdes 3D pour analyse.
- Vérifier les plaques tous les jours pour les changements de couleur moyenne, accumulation de cellules mortes, agglutination et une adhésion bien à fond.
-
Modification/actualisation moyen de différenciation pour la culture 3D
- Transférer le volume requis de NMM avec les composants appropriés (voir étapes 3.2.2-3.2.7 pour partie annexe) dans un tube stérile. Chaud dans un bain-marie à 37 ° C.
- Astuce de la plaque et agiter doucement jusqu'à ce que les cellules s’installer sur les bords du fond des puits. Soigneusement, prélever 2 mL de milieu ancien à l’aide d’une pipette sérologique, évitant les cellules, puis ajouter 2 mL de milieu frais. Incuber à 37 ° C, avec 5 % de CO2.
Remarque : Le volume (Important), la fin doit être 2,5 mL/puits de 6WP. En cas d’évaporation, ne pas enlever les 2 mL du vieux moyen car cela serait plus sécher les cultures cellulaires ; au lieu de cela, ajoutez moyen supplémentaire.
-
Modification/actualisation moyen de différenciation avec séparation par gravité
- Transférer le volume requis de NMM avec les composants appropriés dans un tube stérile. Chaud dans un bain-marie à 37 ° C.
- Transférer le contenu des puits dans un tube stérile et laisser le tube de s’asseoir pendant 10 min à la droite, pour la séparation par gravité.
- Utiliser une pipette pour retirer l’ancien milieu du tube, prenant soin de ne pas supprimer se sont installés des cellules, remettre en NMM avec les composants appropriés et ensuite distribuer de nouveaux puits AA-enduit. Incuber à 37 ° C, avec 5 % de CO2.
NOTE : (Facultatif) pour plus de commodité, une partie du milieu de culture peuvent être ajoutées à la destination puits avant la distribution, avec la différenciation des cellules resuspendues dans un volume plus faible. Le volume de fin doit être de 2,5 mL/puits de 6WP.
4. Protocole n° 3 : Différenciation 2D Alternative Culture
-
Commencer et maintenir la culture en utilisant les étapes conformément à l’article 3 pour le protocole 3D par jour 12
- Suivez les étapes 3.1-3.2.3 et changement/rafraîchissement moyen comme par les Etapes 3.3 et 3.4.
-
Basculez vers et maintenir la culture monocouche 2D
- Jour 13 de différenciation, suivez les instructions pour changement/rafraîchissement moyen avec séparation par gravité (étape 3.4), de distribuer uniquement les cellules/agrégats à PLO/LAM enduit plaque (voir étape 1.6) avec un volume de fin de 2,5 mL/puits de 6WP.
NOTE : Le support peut être complété avec 10 µM RI pendant les ensemencements initiaux pour aider à l’adhérence et la survie des cellules. (Important) Il est souhaitable de répartir uniformément les cellules dans les puits, afin d’éviter de faible densité ou encombrement sur les plaques et au passage (voir étape 4.4) si nécessaire. La taille par défaut du PLO/LAM revêtu de plaques(p. ex., 6WP, 12WP, etc.) doit être déterminée empiriquement, basé sur le taux de prolifération de la lignée cellulaire et le but du produit. Donneront des volumes pour 6WP instructions et peuvent être converties par moitié pour chaque doublement du nombre de puits (c.-à-d., 2 mL/puits pour 6WP, 1 mL/bien pour 12WP, etc.) - Le 14e jour, remplacez le milieu NMM avec 100 ng/mL FGF8B, 100 ng/mL FGF4 et 20 ng/mL FGF2. Actualiser le support de tous les autres jours comme indiqué au point 4.3.
- Jour 17, modifier le milieu NMM avec 100 ng/mL FGF8B. Actualiser le support de tous les autres jours comme indiqué au point 4.3.
- Au jour 21, changer le milieu à NMM avec 100 ng/mL BDNF et 10 ng/mL GDNF. Actualiser le support de tous les autres jours comme indiqué au point 4.3.
- Le 28e jour, remplacez le milieu NMM avec 100 ng/mL BDNF et 10 ng/mL GDNF, 3 ng/mL SAG, 100 ng/mL NT3 et 25 mM KCl. actualiser le support de tous les autres jours comme indiqué au point 4.3.
- Jour 35, prélever des cellules pour l’analyse, ou de maintenir dans le même milieu comme étape 4.2.5 pour culture prolongée (limite potentielle non testé).
- Jour 13 de différenciation, suivez les instructions pour changement/rafraîchissement moyen avec séparation par gravité (étape 3.4), de distribuer uniquement les cellules/agrégats à PLO/LAM enduit plaque (voir étape 1.6) avec un volume de fin de 2,5 mL/puits de 6WP.
-
Modification/actualisation moyen de différenciation pour la culture 2D
- Transférer le volume requis de NMM avec les composants appropriés (voir étapes 4.2.2-4.2.5 pour partie annexe) dans un tube stérile. Chaud dans un bain-marie à 37 ° C.
- Aspirez le milieu du puits, puis ajouter 2 mL de milieu nouveau. Incuber à 37 ° C, avec 5 % de CO2.
NOTE : Volume (en option) la fin peut être maintenu à 2,5 mL/puits de 6WP, en utilisant une pipette pour enlever les 2 mL du milieu de l’ancienne et ajouter 2 mL de milieu frais. Réservant une partie de l’ancien milieu dans les puits et empêcher les cellules de contact avec l’air, peut diminuer choc aux cellules durant les étapes de changement.
-
Culture de différenciation 2D de passage
- Transférer le volume requis de NMM avec les composants appropriés (voir étapes 4.2.2-4.2.5 pour partie annexe) dans des tubes stériles, pour le passage des processus et, séparément, pour préparer le PLO/LAM enduit plaque pour recevoir des cellules de subcultures. Compléter le support de la plaque de destination avec 10 µM RI. Chauffer les tubes moyens à ta, ou dans un bain-marie à 37 ° C. Pour économiser sur les composants, il est possible d’utiliser NMM seul pour le lavage des cellules pendant le processus de passaging.
Remarque : Les expériences (Important) si les produits seront utilisés dans l’imagerie calcique, assurer aux cellules de passage entre 2 à 6 jours avant la fin de la différenciation. - Aspirez le milieu des puits à être repiquées. Ajouter 300 µL/puits d’agent de dissociation axée sur la trypsine (voir Table des matières), tourbillon plaque pour recouvrir les puits, puis retirer immédiatement l’agent de dissociation.
- Laissez reposer pendant 2 min à ta la plaque, puis desserrez les cellules en tapant sur les côtés de la plaque. Ajouter 600 inhibiteur de la trypsine définis µL/puits (DTI) et transférer les cellules DTI dans un tube stérile avec 5 mL NMM.
- Centrifuger le tube à 290 x g pendant 15 min à RT. aspirer le milieu et ajouter un autre 5 mL NMM dans le tube.
- Centrifuger le tube à 290 x g pendant 15 min à RT. aspirer le milieu et remettre en suspension les cellules dans le milieu approprié contenant RI.
- Distribuer les cellules sur la plaque de PLO/LAM un fendage ratio de 1:1-1:12, selon la confluence initiale, taux de prolifération et taille différentielle à l’origine de la plaque de la destination et maintenir à 37 ° C, avec 5 % de CO2.
- Transférer le volume requis de NMM avec les composants appropriés (voir étapes 4.2.2-4.2.5 pour partie annexe) dans des tubes stériles, pour le passage des processus et, séparément, pour préparer le PLO/LAM enduit plaque pour recevoir des cellules de subcultures. Compléter le support de la plaque de destination avec 10 µM RI. Chauffer les tubes moyens à ta, ou dans un bain-marie à 37 ° C. Pour économiser sur les composants, il est possible d’utiliser NMM seul pour le lavage des cellules pendant le processus de passaging.
Résultats
Aperçu visuel de la réduction de la croissance Factor protocoles de différenciation cérébelleux 2D et 3D
La figure 1 montre la chronologie globale pour les protocoles de différenciation cérébelleux 2D et 3D, identification des facteurs extrinsèques et temps du bordé. La progression typique de hPSCs en cours de différenciation cérébelleuse 3D est représentée à la Figure 2: ligne de CSEh H01 à partir des colonies de culture sans chargeur au jour 0 (coin supérieur gauche de la figure) ; en cours de formation de l’EB par jour 2 (moyen supérieur) ; devenant plus grands agrégats cellulaires avec lumen apparente après induction neurale avec RA et FGF8 au jour 14 (en haut à droite) ; formant des agrégats de taille variable et de forme à 28 jours (en bas à gauche) ; développer en complexité avec les différentes structures d’un agrégat unique indiqué au jour 28 (milieu bas) ; et a poursuivi des changements morphologiques aux structures mêmes à jour 35 (en bas à droite). La progression typique de hPSCs en cours de différenciation cérébelleuse 2D est représentée à la Figure 3: des CSEh ligne H01 comme colonies en culture sans chargeur au jour 0 (coin supérieur gauche de la figure, avec cercle indique la zone de cellules différenciées entre les colonies de CSEh) ; en cours de formation de l’EB par jour 2 (moyen supérieur) ; devenant plus grands agrégats cellulaires avec lumen apparente après induction neurale avec RA et FGF8 au jour 13 (en haut à droite) ; la prolifération des cellules adhérentes après ensemencement au jour 14 (en bas à gauche) ; et puis comme une monocouche de cellules avec une morphologie complexe/mature plus à jour 35 sous faible (inférieure au milieu) et fort grossissement (en bas à droite).
Marqueurs d’exposition de produits 3D et les Structures du neuroépithélium précoce
Figure 2 (image du bas gauche) et la Figure 4 montre l’hétérogénéité de la morphologie globale 3D vue à travers la mise en culture, en raison de variables de croissance et/ou taux de maturation, ainsi que la fusion stochastique ou breaking apart des agrégats. Malgré l’hétérogénéité, chaque différenciation produit des agrégats présentant des marqueurs précoces de neuronales et neurones, y compris granule cérébelleux marqueur ZIC1, comme indiqué par immunocytochimie (ICC), coloration à la Figure 5. Plus important encore, Figure 5 et Figure 6 donnent à penser qu’une simple culture 3D, avec des facteurs de croissance réduites, est capable de produire des agrégats avec des structures complexes liés au développement du cerveau comme le neuroépithélium précoce et lèvre rhombique.
Les produits 2D pièce marqueurs cérébelleux et l’activité neuronale fonctionnelle
Alors que les cultures 2D ne peut pas reproduire des structures 3D complexes, ils sont capables de générer des cellules montrant des marqueurs précoces de neuronales et neurones, y compris de marqueur de cellules de granules cérébelleux ZIC1, comme indiqué par ICC coloration à la Figure 7. Analyse de l’expression génique par RT-PCR, comme on le voit dans la Figure 8, prend en charge les résultats de coloration de ICC, bien que la présence du marqueur précoce de cellules granule ATOH1 est variable entre les expériences et les lignes. Imagerie calcique est géré plus facilement dans la culture 2D. Comme on le voit dans la Figure 9, supplémentaire vidéo 1et 2 vidéo supplémentaire, stimulées électriquement les cellules montrent des influx calciques qui sont typiques des patrons de décharge neuronale, suggérant la génération de neurones fonctionnels.
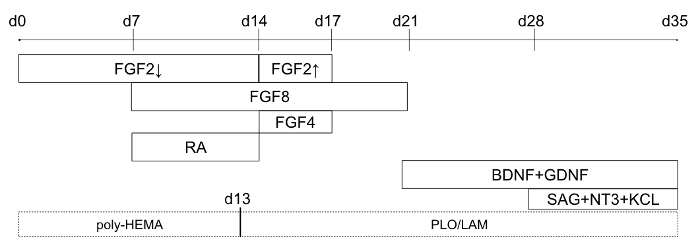
Figure 1 : chronologie du protocole de différenciation (commençant au jour J0 de différenciation). Boîtes de trait plein indiquent quand des facteurs particuliers sont ajoutés au milieu de culture, et boîtes en pointillé indiquent plaque-revêtement pour modification 2D facultative. Pour FGF2, la flèche vers le bas se réfère à des concentrations plus faibles (4 ng/mL), et la flèche vers le haut se réfère à des concentrations plus élevées (20 ng/mL). Ce chiffre a été modifié par Holmes et Heine10. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
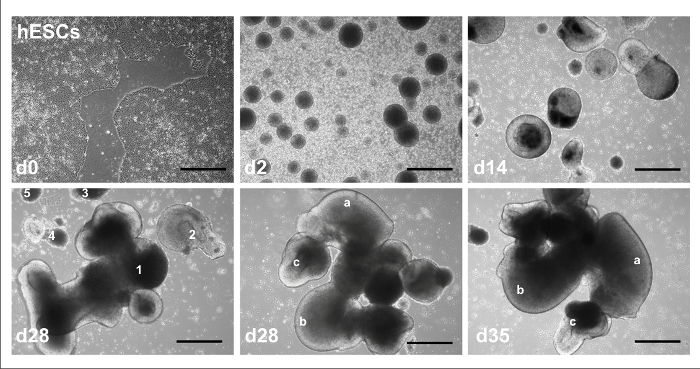
Figure 2 : images de fond clair représentant du protocole 3D. colonies de CSEh en d0, EBs à d2, agrégats après induction à d14, agrégats de taille différente et la morphologie (numérotés de 1-5) à J28, un agrégat unique avec des caractéristiques uniques et identifiables (indiqués par les lettres a–c) à J28, et changements visibles dans les mêmes fonctions à d35. Echelle = 100 µm. Ce chiffre a été modifié par Holmes et Heine10. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3 : images de fond clair représentant du protocole 2D. CSEh colonies une d0, EBs à d2, agrégats après induction à d13, après placage agrégats à d14, après maturation à d35, comme on le voit à 5 x grossissement et un grossissement de 20 x. Le cercle blanc dans le panneau de gauche montre la zone de cellules différenciées entre les colonies de CSEh. Echelle = 50 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
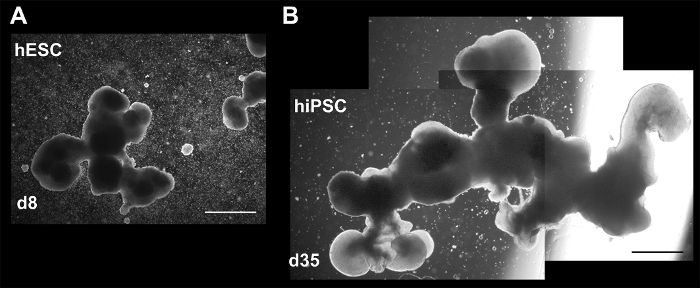
Figure 4 : images de fond clair montrant différentes dimensions et la complexité dans la culture 3D. Les agrégats à (A), jour 8 (CSEh) et (B) d35 (hiPSCs). L’image de ce dernier est composé de trois images séparées pour montrer l’ensemble global. Les deux agrégats ont été touchés par la fusion de petits agrégats ou perte (rupture) des structures. Echelle = 200 µm. Ce chiffre est republié de Holmes et Heine10. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
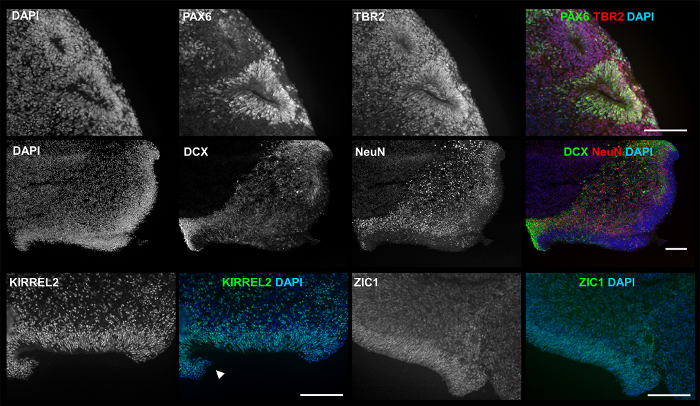
Figure 5 : ICC images de produits 3D montrent des marqueurs pertinents et structures. À la culture d35, pièce 3D produits : PAX6 (vert) et TBR2 (rouge) de lumen de rosette-comme la formation neurale (1er rang) ; DCX (vert) et NeuN (rouge) s’étalant de la zone ventriculaire de bord extérieur (VZ)-comme la structure (seconde ligne) ; KIRREL2, un marqueur associé neuroépithélium cérébelleux (troisième rangée, gauche) ; et les cellules ZIC1 un marqueur associé à granule cérébelleux (3e rangée, droite). L’expérience a été réalisée plusieurs fois à l’aide de quatre hPSC différentes lignes : ligne de CSEh H01 (n = 5) et hvs88 lignes iPSC (n = 4), hvs60 (n = 3) et hvs51 (n = 1). Flèches pointent vers la lèvre rhombique (RL)-comme la structure. Echelle = 100 µm. Ce chiffre est republié de Holmes et Heine10. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 6 : structure en forme de zone ventriculaire à grande échelle de produits 3D. Culture d35, un agrégat de CSEh dérivé est positif pour PAX6 (vert) et TBR2 (rouge), souvent associées à des neurones précoces dans les zones ventriculaires (VZs) et sous-ventriculaire (SVZs), in vivo. (En haut) Un astérisque (*) marque le côté apical d’une région de VZ-like qui longe le bord de l’agrégat, avec parenthèses indiquant la profondeur/division de VZ / SVZs. (milieu) : fusion des signaux montrent des tronçons épars de PAX6 + / TBR2-cellules augmente dans la taille vers l’extrémité supérieure droite de la VZ. (En bas) Image de grossissement plus élevé de la section indiquée par un rectangle dans le panneau central. Echelle = 100 µm. Ce chiffre a été modifié par Holmes et Heine10. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
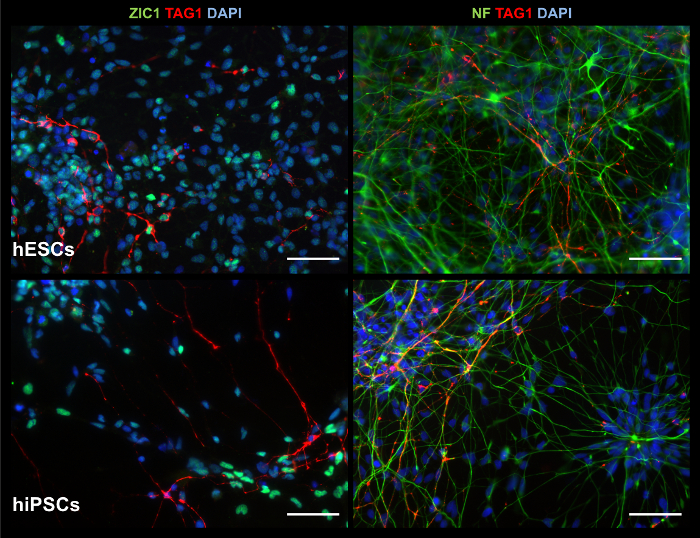
Figure 7 : ICC images 2D produits montrent des marqueurs pertinents. À la culture d35, exposition de produits 2D, de CSEh (rangée du haut) et hiPSCs (rangée du bas), cellules positives pour : marqueur de cellules de granules ZIC1 et migrateurs neurones cérébelleux marqueur TAG1 (colonne de gauche) ; et les neurofilaments (NF) de la marqueur neuronal et TAG1 (colonne de droite). Echelle = 50 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
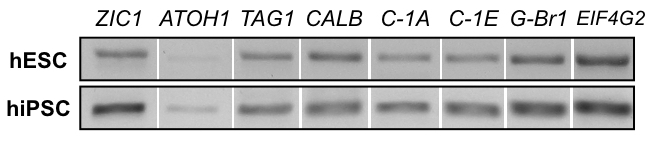
Figure 8 : RT-PCR de produits 2D. analyse de l’expression ARNm de CSEh ligne H01 (rangée du haut) et hiPSC ligne hvs60 (rangée du bas) à la fin de la 2D Protocole montre produits avec électrophorèse pour : marqueur de cellules de granules ZIC1, marqueur de cellules de granules ATOH1, neurones cérébelleux migrateurs marqueur TAG1, Purkinje marqueur de cellules Calbindin (CALB), canaux calciques voltage-dépendants de CACNA1A (C-1 a), CACNA1E (C-1F), récepteur de l’acide gamma - aminobutyrique (GABA) B 1 (G-Br1) et gène de ménage EIF4G2).
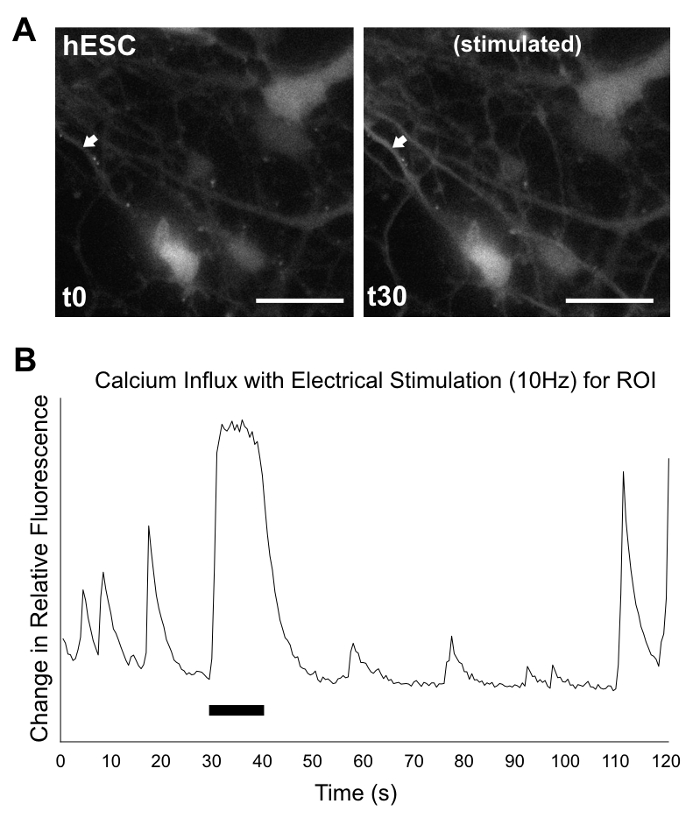
Figure 9 : Calcium imagerie/analyse des produits 2D. À la culture d35, hPSC produits de différenciation ont été enregistrés pendant 2 min au microscope, après incubation avec de la teinture de fluor5. À 30 s, les cellules ont été stimulés électriquement pour 10 s à 10 Hz. (A) des images fixes Voir la CSEh à 0 s (à gauche) et après le début de la stimulation électrique à 30 s (à droite). Les flèches indiquent la région d’intérêt (ROI) pour l’analyse des influx de calcium. Affichage de graphiques d’analyse (B) changement de fluorescence relative par rapport au temps de retour sur investissement (changer en fluorescence = (F-F,0) / f0, où F0 = (∑F1-n) / n), avec des pointes de neurone-comme se produisant avant, pendant et après stimulation. Barre noire indique la durée de la stimulation électrique. L’échelle bar (blanc) = 50 µm. complet d’enregistrement pour CSEh (comme ici) et une ligne de hiPSC (non illustré) sont disponibles en supplémentaire vidéo 1 et 2 de vidéo supplémentaire, respectivement. Les enregistrements sont au format AVI, à la vitesse 4 x. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Supplémentaire 1 vidéo : Calcium imagerie vidéo 2D produit de CSEh ligne H01. À la culture d35, produits de différenciation de la ligne de CSEh H01 ont été enregistrés pendant 2 min au microscope, après incubation avec fluor5 teignent. À 30 s, les cellules ont été stimulés électriquement pour 10 s à 10 Hz. enregistrements ont été réalisés à 2 images/s et transformé en vidéo AVI à ~ 7 images/s, produisant une vidéo d’une durée d’environ 30 s, à ~ 4 x la vitesse. S’il vous plaît cliquez ici pour télécharger ce fichier.
2 vidéo supplémentaire : Calcium imagerie vidéo 2D produit de hiPSC ligne hvs51. À la culture d35, produits de différenciation de hiPSC ligne hvs51 ont été enregistrés pendant 2 min au microscope, après incubation avec de la teinture de fluor5. À 30 s, les cellules ont été stimulés électriquement pour 10 s à 10 Hz. enregistrements ont été réalisés à 2 images/s et transformé en vidéo AVI à ~ 7 images/s, produisant une vidéo d’une durée d’environ 30 s, à ~ 4 x la vitesse. S’il vous plaît cliquez ici pour télécharger ce fichier.
Discussion
Complexité et les coûts sont des facteurs pertinents pour les chercheurs de cellules souches lorsque choisir ou élaborer des protocoles de différenciation. Cela est particulièrement vrai, car c’est une question ouverte du degré de contrôle externe est nécessaire pour générer des types de cellules désiré, ou — il poser différemment — comment hPSCs compétentes sont à produire leur propre environnement de développement, si laissés à eux-mêmes avec suffisamment éléments nutritifs. Introduction de facteurs extrinsèques in vitro peut produire très bien produits cellule désirée, mais ils pourraient aussi interférer avec les capacités de développement intrinsèques des cellules seraient ont exposés en vivo. Ces considérations sont importantes, surtout si l’objectif est utilisation de dérivés de patient CISP pour la modélisation de la maladie. Utilisation extensive des patrons ou des facteurs de croissance pourrait masquer les phénotypes de la maladie. Les protocoles détaillés dans le présent rapport suivent la tendance des études antérieures pour réduire la complexité, coût, ou l’utilisation de motifs extrinsèques facteurs8,9.
Selon les résultats fournis par Bertin et al. et notre propre étude récente, il semble qu’il est possible d’atteindre la différenciation vers cérébelleux destins sans efforts concertés visant à reproduire des conditions in vivo , comme les études antérieures ont fait 1 , 2 , 3 , 4 , 8 , 10. la partie intéressante est que les deux études utilisé différents ensembles de facteurs de croissance, ce qui suggère que ni ensemble était nécessaire, bien que les deux utilisés FGF2. Nous avons effectué des tests supplémentaires, où FGFs ont été sélectivement exclus du protocole et a montré que les cellules sont capables de générer les mêmes produits sans extrinsèque FGFs10. Différences entre nos études ont été qualifiés par le fait que nous avons utilisé des lignes de hPSC différents et des méthodes de culture, induit la différenciation neuronale avec RA et comprenaient des éléments étayant la survie des cellules granule et maturation (BDNF, GDNF, SAG et le KCL)11 –14. En outre, une méthode de démarrage moins complexe, comparée à Bertin et al., a été employée. Leur protocole a commencé en générant EBs uniforme en 96WPs, qui eux isolés physiquement et chimiquement les uns des autres. Le protocole ici a tous les PSC relativement entassés dans 6WPs lors de la formation EB, qui leur a permis d’interagir librement. Comment cela peut avoir affectées différemment l’environnement physique et chimique de l’EBs et organoïdes plus tard (y compris la production intrinsèque des composés de signalisation) sont inconnue et pourraient être explorées. Aussi, tandis que nous montrons l’expression des gènes associés — et si évocatrice de — origine cérébelleuse, situé au sein de structures morphologiquement similaires à ceux rapportés par Bertin et al., nous ne pouvons exclure génération neuronale-comme des structures qui sont d’identité non-cérébelleux. Des études futures, à l’aide d’un large panel d’anticorps comme ceux signalés par Bertin et al. (c.-à-d., ATOH1, CALB, etc.) rendrait ces affectations et comparer entre les produits de ces deux protocoles, plus concluantes.
Au sein du protocole 3D, il est important pour commencer et maintenir un nombre suffisant de cellules en culture pour assurer un nombre suffisant de produits finis pour l’analyse. Compte tenu des importantes mortalités massives tôt dans le protocole, nous recommande de commencer avec plus de 500 EBs/puits pendant les 3 premiers jours en culture (Figure 1). Cela ne devrait pas être difficile d’atteindre des tailles de colonie donnée pour hPSCs en culture sans chargeur, mais peut-être pas aussi facile pour ceux qui utilisent encore des méthodes axées sur le conducteur. Étant donné le grand nombre de cellules, il est important de regarder pour le changement de couleur dans un milieu (indiquer les changements de pH) et l’accumulation de cellules mortes. Les deux doivent être corrigées pour éviter l’effondrement de la culture. Il peut y avoir également agglutination des cellules et des agrégats en structures massives. Même s’il peut encore entraîner les agrégats qui peuvent être analysés, quantité de produit sera grandement réduite, afin de les briser en petits agrégats avec trituration douce peut être utile. Toutefois, éviter des agrégats normaux inquiétantes, qui eux-mêmes peuvent se développer de grandes tailles (Figure 4). Si agrégats deviennent trop clairsemées, il est recommandé de combiner les puits pour que les agrégats ne sont pas complètement isolés. Variabilité du produit (en nombre, la taille et la morphologie) est un problème bien connu dans la culture cellulaire 3D, y compris pour les protocoles à partir isolé, uniforme pas formation de EB, suggérant qu’une procédure de démarrage moins complexe (tels que le protocole décrit ici ) pourrait être plus pratique de8,15. Alors que cette hétérogénéité est quelque chose que les chercheurs doivent garder à l’esprit, en particulier pendant l’analyse, le protocole déclaré générée produits conformes à ceux trouvés dans d’autres protocoles 3D8,9,15. Basé sur la taille et la morphologie, ils entraient dans le cadre de rosette neuronaux à organoïde cérébrale, comme décrit dans une étude récente de15Kelava et Lancaster, le plus souvent mise en place au classement de sphéroïde. Particulièrement remarquables, sont la présence de structures 3D suggestif de rosettes neurales avec lumen, les zones ventriculaires (void) et rhombique-lèvre comme caractéristiques (Figure 5 et Figure 6) tels que définis par d’autres groupes8 , 15 , 16 , 17. étant donné que chaque expérience a produit au moins un agrégat avec putatifs VZ/SVZs et marqueurs associés à cervelet (ZIC1, KIRREL2), ceux sont des critères utiles pour déterminer le succès d’une différenciation 3D à l’aide de notre protocole, avec RL-comme caractéristiques fournit une aide supplémentaire. Prolonger la durée de la culture depuis 35 jours n’a pas testé, mais susceptibles d’être appliquée pour déterminer les limites de la croissance, la complexité et maturité permise par cette technique.
Le protocole 2D utilise la même formation de EB non adhérents et le processus d’induction neurale comme le protocole 3D et donc des observations qui précèdent s’appliquent également. Une fois plaqué, on doit considérer un ensemble différent de considérations. L’EBs devraient adhérer rapidement pour les cellules à proliférer vers l’extérieur sur la plaque. S’il y a des problèmes avec l’adhérence, adjonction de RI (si pas déjà utilisé), réduit le volume de milieu, ou les variations expérimentales de PLO/LAM concentration peuvent être appliquées. Il est important de garder les cellules de plus en plus trop dense ou éparse (préférence cultivé entre confluence de 20 à 80 %) dans les puits ; surveillance quotidienne et passage en temps opportun est important, pour éviter des cellules flottantes ou over-confluence. Contrairement au protocole 3D, il faut éviter une mortalité significative durant la culture, bien qu’il peut y avoir des zones de faible croissance, ou un ralentissement du taux de prolifération. Passage affecte l’état de maturation des cellules (par exemple, suppression des prolongements cellulaires et les réseaux développés entre les cellules) et devrait garder à l’esprit lorsque vous approchez des points où les cellules seront collectées ou analysés en quelque sorte. Par exemple, pour le calcium imaging il est très important de cellules de passage entre 2 à 6 jours avant l’analyse. Passage trop près d’analyse peut signifier les cellules n’ont pas eu le temps de se connecter ou matures et trop loin peuvent entraîner dans les cellules, la surpopulation, ce qui rend difficile l’imagerie. Bien que la variabilité entre les expériences peut-être exister, les résultats concordent avec ceux rapportés dans les premiers protocoles cérébelleux 2D1,2. Analyse d’expression de la coloration et le gène ICC corroborent la présence de cellules positives pour marqueur de cellules de granules ZIC1, tout en identifiant également les marqueurs associés aux autres identités neurones et cérébelleuses (Figure 7 et Figure 8). Imagerie calcique, qui implique la stimulation électrique des cellules incubées avec le colorant de fluor5, a indiqué l’activité neuronale fonctionnelle (Figure 9, supplémentaire Figure 1et la Figure 2), bien qu’il n’est pas confirmé si ces sont des cellules étoilées. On peut soutenir que, en donnant des cellules plus de temps pour mûrir en prolongeant la durée de la culture depuis 35 jours, le montant de l’activité neuronale fonctionnelle devrait augmenter. Ce potentiel pourrait être envisagée à l’avenir.
En plus des voies de recherche proposé ci-dessus, il serait intéressant pour déterminer les différences dans les identités de produit (quantité et qualité) entre les protocoles 2D et 3D. L’importance de la FGF extrinsèque n’a pas été testé dans le protocole de 2D, et il serait utile de savoir si l’absence de structure 3D après ensemencement, et donc les voies de signalisation associés, ferait des cultures 2D plus ou moins dépendantes sur ceux tôt le patterning composés. Des protocoles plus allégées (p. ex., non RA, le BDNF, le SAG) sont tout aussi plausibles lignes pour complément d’enquête. Enfin, les études futures pourraient bénéficier de nouveaux outils de recherche pour mieux caractériser (et évaluer l’efficacité de la production de) sous-types neuronales cérébelleux spécifiques à l’homme.
Avec les mises en garde donnés à l’esprit, ont tous deux signalé protocoles peuvent être utilisés pour les différenciations cérébelleuses, avec produits adaptés à des fins différentes. Elles peuvent servir de point de départ pratique pour les chercheurs qui effectuent des études pilotes, viabilité des lignées cellulaires pour ces différenciations, ou comme un modèle de base pour d’autres types de différenciation neurale ciblée.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous sommes reconnaissants envers Gerbren Jacobs et Jurjen Broeke pour leur aide technique d’experts, Prisca Leferink pour contribuer à la génération et la caractérisation de deux lignes de commande iPSC et de Lisa Gasparotto pour démontrer nos procédures.
matériels
| Name | Company | Catalog Number | Comments |
| DMEM/F12+Glutamax | Gibco | 31331-028 | glutamine fortified DMEM/F12 |
| Neurobasal medium | Gibco | 21103-049 | neural basic medium |
| N2 supplement | Gibco | 17502-048 | |
| B27 supplement | Gibco | 17504001 | |
| Insulin | Imgen | PT468-B | |
| L-glutamine | Gibco | 25030-024 | |
| Non-essential Amino Acids (NEAA) | Gibco | 11140-035 | |
| beta-mercaptoethanol | Gibco | 21985-023 | |
| Poly-L-Ornithine | Sigma | P3655 | |
| Poly (2-hydroxyethyl methacrylate) | Sigma | P3932 | aka Poly-Hema |
| Laminin | Sigma | L2020 | |
| E8 medium and supplement | Gibco | A1517001 | hPSC medium and supplement |
| Penicillin/Streptomycin | Gibco | 15140-122 | |
| Sodium Chloride | Sigma | S-5886 | |
| y-27632 (ROCK inhibitor) | SelleckChem | S1049-10mg | |
| DMSO | Sigma | D-2650 | |
| Geltrex | Gibco | A1413302 | hPSC-appropriate adherent coating (PAAC) |
| 0,5M EDTA | Gibco | 15575-020 | |
| 0.2 um filter | VWR | 28145-77 | |
| 1.5 mL Eppendorf tube | VWR | 525-0130 | |
| DMEM/F12 | Gibco | 21331-020 | |
| Ethanol | VWR | 83804360 | |
| Parafilm | Sigma | PM996 | wrap for culture plates |
| cryotubes | ThermoFisher | 368632 | |
| TrypLE | Gibco | 12563-029 | trypsin-based dissociation agent |
| Defined Trypsin Inhibitor (DTI) | Gibco | R-007-100 | |
| FGF-2 | Peprotech | 100-18B | |
| FGF-4 | R&D Systems | 100-31 | |
| FGF-8B | Peprotech | 100-25 | |
| Retinoic Acid | Sigma | R2625 | |
| Brain Derived Neurotrophic Factor | Peprotech | 450-02 | |
| Glial Derived Neurotrophic Factor | Peprotech | 450-10 | |
| Potassium Chloride | Sigma | P5405 | |
| Neurotrophic Factor 3 | Peprotech | 450-03 | |
| Smoothened Agonist (SAG) | Cayman | 11914 | CAS 912545-86-9 |
| Axiovert 40C microscope | Zeiss | Brightfield imaging microscope | |
| Axiocam | Zeiss | Brightfield imaging - image aquisition | |
| Eppendorf Centrifuge 5810 | Eppendorf | 521-0996 | centrifuge for cell culture |
| PBS (gebufferde natrium oplossing) | Braun Medical | 3623140 | |
| 5 ml Serological pipets | VWR | 612-4950 | |
| 10 ml Serological pipets | VWR | 612-4951 | |
| 6-wells culture plates | VWR | 734-2323 | |
| 12-wells culture plates | VWR | 734-2324 | |
| hESCs | WiCELL | line H01 |
Références
- Erceg, S., et al. Efficient differentiation of human embryonic stem cells into functional cerebellar-like cells. Stem Cells Dev. 19, 1745-1756 (2010).
- Erceg, S., Lukovic, D., Moreno-Manzano, V., Stojkovic, M., Bhattacharya, S. S. Derivation of cerebellar neurons from human pluripotent stem cells. Curr Protoc Stem Cell Biol. , (2012).
- Su, H. L., Muguruma, K., Matsuo-Takasaki, M., Kengaku, M., Watanabe, K., Sasai, Y. Generation of cerebellar neuron precursors from embryonic stem cells. Dev Biol. 290, 287-296 (2006).
- Salero, E., Hatten, M. E. Differentiation of ES cells into cerebellar neurons. Proc Natl Acad Sci U S A. 104, 2997-3002 (2007).
- Joyner, A. L. Engrailed, Wnt and Pax genes regulate midbrain--hindbrain development. Trends Genet. 12, 15-20 (1996).
- Joyner, A. L., Liu, A., Millet, S. Otx2, Gbx2 and Fgf8 interact to position and maintain a mid-hindbrain organizer. Curr Opin Cell Biol. 12, 736-741 (2000).
- Tam, E. W. Y., Benders, M. J. N. L., Heine, V. M. Cerebellar Development-The Impact of Preterm Birth and Comorbidities. Fetal and Neonatal Physiology. , (2017).
- Muguruma, K., Nishiyama, A., Kawakami, H., Hashimoto, K., Sasai, Y. Self-organization of polarized cerebellar tissue in 3D culture of human pluripotent stem cells. Cell Rep. 10, 537-550 (2015).
- Pasca, A. M., et al. Functional cortical neurons and astrocytes from human pluripotent stem cells in 3D culture. Nat Methods. 12, 671-678 (2015).
- Holmes, D. B., Heine, V. M. Simplified 3D protocol capable of generating early cortical neuroepithelium. Biology Open. 6, 402-406 (2017).
- Chen, J. K., Taipale, J., Cooper, M. K., Beachy, P. A. Inhibition of Hedgehog signaling by direct binding of cyclopamine to Smoothened. Genes Dev. 16, 2743-2748 (2002).
- Chen, J. K., Taipale, J., Young, K. E., Maiti, T., Beachy, P. A. Small molecule modulation of Smoothened activity. Proc Natl Acad Sci U S A. 99, 14071-14076 (2002).
- Dahmane, N., Ruiz-i-Altaba, A. Sonic hedgehog regulates the growth and patterning of the cerebellum. Development. 126, 3089-3100 (1999).
- Borghesani, P. R., et al. BDNF stimulates migration of cerebellar granule cells. Development. 129, 1435-1442 (2002).
- Kelava, I., Lancaster, M. A. Stem Cell Models of Human Brain Development. Cell Stem Cell. 18, 736-748 (2016).
- Kadoshima, T., et al. Self-organization of axial polarity, inside-out layer pattern, and species-specific progenitor dynamics in human ES cell-derived neocortex. Proc Natl Acad Sci U S A. 110, 20284-20289 (2013).
- Englund, C., et al. Pax6, Tbr2, and Tbr1 are expressed sequentially by radial glia, intermediate progenitor cells, and postmitotic neurons in developing neocortex. J Neurosci. 25, 247-251 (2005).
- Warlich, E., et al. Lentiviral vector design and imaging approaches to visualize the early stages of cellular reprogramming. Mol Ther. 19, 782-789 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon