Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Microscopie à fluorescence pour l’internalisation de l’ATP médiée par la macropinocytose dans les cellules tumorales humaines et les souris xénogreffées
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous avons développé une méthode reproductible pour visualiser l’internalisation de l’adénosine triphosphate fluorescente non hydrolysable (ATP), un substitut de l’ATP, avec une résolution cellulaire élevée. Nous avons validé notre méthode en utilisant des tests indépendants in vitro et in vivo - des lignées de cellules tumorales humaines et des souris immunodéficientes xénogreffées avec du tissu tumoral humain.
Résumé
Il a été démontré que l’adénosine triphosphate (ATP), y compris l’ATP extracellulaire (eATP), joue un rôle important dans divers aspects de la tumorigenèse, tels que la résistance aux médicaments, la transition épithélio-mésenchymateuse (EMT) et les métastases. L’eATP intratumoral est 10 à 104 fois plus élevée en concentration que dans les tissus normaux. Alors que l’eATP fonctionne comme un messager pour activer la signalisation purinergique pour l’induction EMT, il est également internalisé par les cellules cancéreuses par macropinocytose régulée à la hausse, un type spécifique d’endocytose, pour effectuer une grande variété de fonctions biologiques. Ces fonctions comprennent la fourniture d’énergie aux réactions biochimiques nécessitant de l’ATP, le don de groupes phosphate pendant la transduction du signal et la facilitation ou l’accélération de l’expression des gènes en tant que cofacteur transcriptionnel. L’ATP est facilement disponible, et son étude dans le cancer et d’autres domaines augmentera sans aucun doute. Cependant, l’étude eATP en est encore à un stade précoce et les questions non résolues restent sans réponse avant que les activités importantes et polyvalentes de l’eATP et de l’ATP intracellulaire internalisé puissent être complètement démêlées.
Les contributions des laboratoires de ces auteurs à ces premières études eATP comprennent l’imagerie microscopique de l’ATP fluorescent non hydrolysable, associée à un dextrans fluorescent de haut et de bas poids moléculaire, qui sert de traceurs de macropinocytose et d’endocytose, ainsi que divers inhibiteurs de l’endocytose, pour surveiller et caractériser le processus d’internalisation de l’eATP. Cette modalité d’imagerie a été appliquée à des lignées cellulaires tumorales et à des souris immunodéficientes, xénogreffées avec des tumeurs cancéreuses humaines, pour étudier l’internalisation de l’eATP in vitro et in vivo. Cet article décrit ces protocoles in vitro et in vivo, en mettant l’accent sur la modification et l’ajustement des conditions de dosage afin que les tests d’internalisation de l’eATP médiés par la macropinocytose / endocytose puissent être effectués avec succès dans différents systèmes.
Introduction
L’absorption opportuniste des nutriments extracellulaires intratumoraux (c’est-à-dire) a récemment été nommée une marque clé du métabolisme du cancer1. L’un de ces nutriments importants est l’ATP, car la concentration d’ieATP est 103 et10 4 fois plus élevée que celle trouvée dans les tissus normaux, dans la gamme de plusieurs centaines de μM à faible mM2,3,4,5. En tant que molécule clé d’énergie et de signalisation, l’ATP joue un rôle central dans le métabolisme cellulaire dans les cellules cancéreuses et saines6,7,8. L’ATP extracellulaire n’est pas seulement impliqué dans la croissance des cellules cancéreuses, mais il favorise également la résistance aux médicaments9. Des fonctions auparavant non reconnues de l’ATP, telles que l’activité hydrotrope, ont récemment été identifiées, impliquant ainsi l’implication de l’ATP dans des maladies telles que la maladie d’Alzheimer10. En effet, il semble que notre compréhension de l’ATP et de ses fonctions dans les cellules cancéreuses, les cellules saines et d’autres cellules malades soit loin d’être complète. Cependant, en raison de l’instabilité de l’ATP et des taux de renouvellement élevés dans les cellules, il est techniquement difficile de surveiller le mouvement de l’ATP à travers la membrane cellulaire et dans la cellule.
Pour résoudre ce problème et répondre au besoin de ce domaine de recherche, une méthode a été développée dans laquelle l’ATP fluorescent non hydrolysable (NHF-ATP)(Figure 1)a été utilisé comme substitut pour visualiser l’internalisation de l’ATP et observer la localisation spatiale intracellulaire de l’ATP internalisé, à la fois in vitro et in vivo11,12 . Il a été démontré que le NHF-ATP remplace l’ATP endogène pour étudier le mouvement de l’ATP à travers les membranes cellulaires animales, à la fois dans les lignées cellulaires cancéreuses et dans les tissus tumoraux humains xénogreffés sur des souris immunodéficientes11,12. De plus, l’administration d’inhibiteurs de la macropinocytose aux cellules a bloqué l’internalisation de l’eATP, suggérant que l’absorption intracellulaire de l’eATP implique un mécanisme macropinocytotique9,11,12. Ce protocole permet le comarquage immunosourcé contre des protéines spécifiques à la cellule et donc l’identification du type de cellule qui internalise le NHF-ATP. En utilisant des xénogreffes tumorales in vivo et la microscopie à haute résolution, le NHF-ATP peut être visualisé spatialement à travers l’échantillon de tissu et même dans une seule cellule. Ces méthodes permettent également une analyse quantitative, telle que le pourcentage d’absorption cellulaire, le nombre de vésicules macropinocytotiques et la cinétique d’internalisation. Cet article décrit en détail comment le NHF-ATP, travaillant seul ou avec le dextrans fluorescent dextrans13 , 14 , 15,16, peut être utilisé dans différents contextes expérimentaux pour étudier l’internalisation et la localisation intracellulaire de l’ATP, après l’internalisation dans les cellules.

Figure 1: Structures de l’ATP fluorescent non hydrolysable et de la tétraméthylrhodamine marquées de dextran fluorescent de haut poids moléculaire. (A) Structure de NHF-ATP. (B) Représentation schématique du HMWFD. Abréviations : ATP = adénosine triphosphate; NHF-ATP = ATP fluorescent non hydrolysable; TMR = tétraméthylrhodamine; HMWFD = dextran fluorescent de haut poids moléculaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les procédures rapportées ici ont été effectuées conformément à l’IACUC de l’Université de l’Ohio et au NIH.
1. Sélection de l’ATP fluorescent non hydrolysable (NHF-ATP) et du dextrans
- Sélectionner un NHF-ATP conjugué au fluorophore(Figure 1A)et des traceurs d’endocytose, un dextrans fluorescent de haut et de bas poids moléculaire (TMR-HMWFD et TMR-LMWFD)(Figure 1B),en fonction des longueurs d’onde d’émission préférées (p. ex., système d’imagerie équipé de filtres appropriés) et du processus d’endocytose spécifique à étudier.
2. Études de localisation de l’ATP, in vitro (Figure 2)
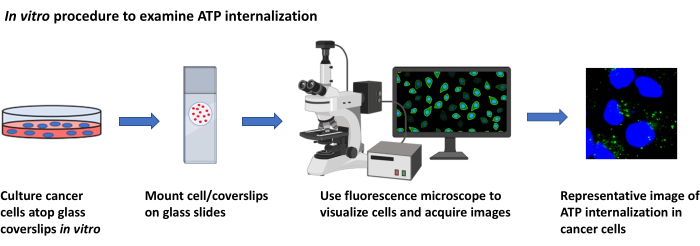
Figure 2: Procédure in vitro pour examiner l’internalisation de l’ATP. Représentation schématique du protocole pour visualiser l’internalisation de l’ATP extracellulaire dans les cellules cancéreuses en culture à l’aide de la microscopie à fluorescence. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Culture cellulaire et préparation de cellules
REMARQUE: Effectuer une culture cellulaire dans des conditions stériles dans une hotte de culture tissulaire.- Préparer le milieu Eagle modifié (DMEM) de Dulbecco, contenant 10 % (v/v) de sérum fœtal bovin (FBS) et 1 % (v/v) de pénicilline/streptomycine (ci-après appelé DMEM/FBS), une solution saline stérile tamponnée au phosphate (PBS) et 0,25 % de trypsine/éthylènediaminetétraacétique (EDTA), dans un bain-marie à 37 °C.
- Cultiver des cellules cancéreuses humaines dans du DMEM/FBS dans une boîte de culture tissulaire de 100 mm. Maintenez les cellules dans un incubateur réglé à 37 °C avec une atmosphère de CO2 à 5 %.
- Lorsque les cellules atteignent la confluence, passez les cellules en retirant d’abord le milieu de culture. Ensuite, rincez le plat avec 5 mL de PBS stérile, retirez le PBS et ajoutez 3 mL de trypsine à 0,25%. Incuber à 37 °C dans une atmosphère à 5 % deCO2 pendant 5 min.
- Récupérez la parabole, puis ajoutez 6 mL de DMEM/FBS pour arrêter la trypsinisation. Transférer les cellules en suspension dans un tube conique de 15 mL et centrifuger à 800 × g pendant 5 min pour granuler les cellules.
- Après centrifugation, aspirer le surnageant et utiliser 10 mL de DMEM/FBS pour remettre en suspension la pastille cellulaire par pipetage.
- Comptez la densité cellulaire et la viabilité à l’aide d’un hémocytomètre. Utilisez DMEM/FBS pour diluer la suspension cellulaire à une densité d’environ 7,5 × 104 cellules/mL.
- Préparation des couvertures et des cellules d’ensemencement
- Lavez les couvercles de 12 mm avec de l’éthanol à 70% et essuyez-les soigneusement avec des lingettes de travail délicates. Stérilisez les couvercles et une paire de pinces par autoclavage.
- Dans une hotte de culture tissulaire, utilisez une pince pour placer un couvercle dans chaque puits d’une plaque de culture tissulaire de 24 puits.
REMARQUE: Plus tard, le couvercle, avec des cellules, sera monté directement sur une lame de microscope pour l’imagerie. - Distribuer 300 μL de la suspension cellulaire (cellules en DMEM/FBS), à une densité d’ensemencement d’environ 2,5 × 104 cellules par puits, dans la plaque de 24 puits contenant les couvercles stérilisés. Incuber dans des conditions stériles à 37 °C avec un débit de CO 2 de5 %.
- Famine des cellules
- Vingt-quatre heures après l’ensemencement, retirez le DMEM/FBS de chaque puits. Ajoutez immédiatement 300 μL de DMEM sans sérum préavertissé dans chaque puits pour affamer les cellules pendant 15 à 18 h afin d’induire l’absorption des nutriments extracellulaires.
REMARQUE: La période de famine de 15 à 18 heures est un paramètre critique.
- Vingt-quatre heures après l’ensemencement, retirez le DMEM/FBS de chaque puits. Ajoutez immédiatement 300 μL de DMEM sans sérum préavertissé dans chaque puits pour affamer les cellules pendant 15 à 18 h afin d’induire l’absorption des nutriments extracellulaires.
- Préparation de solutions NHF-ATP et HMWFD/LMWFD
- Utilisez une balance analytique pour peser le TMR-dextran fluorescent de haut poids moléculaire (70 kDa) (TMR-HMWFD, 1 mg/mL), un traceur pour visualiser les macropoinosomes, ou NHF-ATP (10 μmol/L) dans le DMEM sans sérum dans un tube de microcentrifugation de 1,5 mL. Placez les tubes, à l’abri de la lumière, dans un bain-marie à 37 °C pendant 15 min.
- Centrifuger à 12 000 × g pendant 5 min à température ambiante. Transférez soigneusement le surnageant clair dans un nouveau tube de microcentrifugation de 1,5 mL, en laissant intacts les granulés ou débris pour éliminer les cristaux indissolubles.
- Ajouter les solutions de l’étape 2.4.1 aux cellules de chaque puits et incuber les cellules pendant 30 min à 37 °C.
REMARQUE: Si les solutions HMWFD et NHF-ATP doivent être mélangées pour co-incubation avec les cellules, préparer les deux solutions à 2x les concentrations finales. Les solutions seront mélangées plus tard à un rapport de 1: 1 pour obtenir les concentrations de travail précises finales. Évitez la lumière car les réactifs sont sensibles à la lumière.
- Traitement des cellules et fixation
- Dans une plaque fraîche de 24 puits, distribuer 500 μL de PBS préavertissé dans chacun des cinq puits.
- Après l’incubation cellulaire, ramassez soigneusement chaque couvercle à l’aide d’une pince. Rincez chaque couvercle en le trempant dans 500 μL de PBS préavertis. Répétez cinq fois en utilisant les cinq puits remplis de PBS.
REMARQUE: Un lavage en douceur des cellules sur les couvercles est essentiel au succès de cette expérience. - Après le lavage final du PBS, tapotez le couvercle sur une lingette de tâche délicate pour absorber le PBS supplémentaire et transférez immédiatement le couvercle à froid (4 ° C) 3,7% de formaldéhyde, préchargé dans une plaque de 24 puits. Fixez les cellules pendant 15 min à température ambiante.
- Pendant que les cellules sont fixées, pré-nettoyez les lames de microscope avec 70% d’éthanol. Retirez les couvercles des puits et montez-les sur les lames, en utilisant 5 μL de milieu de montage aqueux contenant la tache nucléaire 4′,6-diamidino-2-phénylindole (DAPI), par couvercle. Épongez délicatement l’excès de PBS avec une serviette en papier ou une lingette de tâche délicate.
- Microscopie à fluorescence et acquisition d’images
- Deux à 24 heures après les étapes ci-dessus, capturez des images de cellules et de HMWFD et/ou de NHF-ATP internalisés à l’aide d’un système d’imagerie par épifluorescence et d’un logiciel d’acquisition de données.
REMARQUE : cette sous-section décrit les étapes à suivre pour acquérir des images à l’aide d’un microscope Nikon NiU, équipé d’une capacité d’imagerie par épifluorescence et du logiciel Nikon NIS Elements. Cependant, d’autres systèmes d’imagerie et logiciels d’acquisition comparables peuvent être utilisés. Suivez les instructions d’utilisation du fabricant.- Placez la lame sur la scène d’un microscope à épifluorescence verticale en mode binoculaire. Accédez au programme d’imagerie.
- Sélectionnez l’objectif 10x, ajustez la scène pour définir la mise au point et scannez la diapositive de gauche à droite de manière serpentine pour identifier les régions d’intérêt.
REMARQUE : L’identification des régions d’intérêt variera d’un type cellulaire à l’autre, certaines lignées cellulaires ou certains types de cancer présentant des degrés divers et distincts d’absorption de TMR-HMWFD et/ou de NHF-ATP. - Sélectionnez l’objectif 40x et passez du mode binoculaire au mode de capture d’image à l’aide de la bascule du microscope.
- Cliquez sur l’icône Qualité en direct du programme d’imagerie pour afficher et ensuite acquérir des images.
- À l’aide du panneau OC de la barre d’outils Annotations et mesures, définissez les paramètres d’exposition pour chaque cube filtrant ou canal fluorescent.
REMARQUE: Sélectionnez le temps d’exposition approprié pour chaque canal, car les intensités du signal sont différentes. Par exemple, sélectionnez un temps d’exposition de 200 ms pour DAPI, 2 s pour HMWFD et 4 s pour NHF-ATP. Une fois le temps d’exposition déterminé par couche, utilisez ce paramètre pour toutes les images, par couche, avec un traitement ou des conditions différentes. - Une fois les paramètres d’exposition définis pour chaque couche, utilisez la barre d’outils d’acquisition multicanal pour acquérir une image à 3 couches avec les paramètres d’exposition définis.
REMARQUE: L’acquisition d’images via le mode d’acquisition ND multicanal permet la capture automatique d’images pour chaque canal du même champ de vision. L’obturateur se ferme automatiquement entre les changements de tourelle. - Vous pouvez également acquérir manuellement des images multicanaux en basculant entre les cubes de filtre, en réglant le temps d’exposition, en fermant / ouvrant l’obturateur entre l’acquisition de l’image pour chaque canal et en superposant chaque image prise pour des canaux individuels.
REMARQUE: Le mode d’acquisition ND automatise ce processus et fournit des images fusionnées. - Enregistrez l’image en tant que fichier .nd2 (le format Nikon Elements enregistre les métadonnées). Enregistrez les fichiers TIF, y compris l’image de canal fusionnée et les images de canal individuelles.
REMARQUE: Les fichiers TIF peuvent être utilisés avec une plus grande sélection d’applications logicielles. - Utilisez la fonction Nombre d’objets de la barre d’outils Analyse pour compter le nombre de cellules NHF-ATP-, TMR-HMWFD- et/ou TMR-LMWFD-positives sur un fichier image .nd2 enregistré.
- Exportez les données dans une feuille de calcul via le programme d’analyse.
- Deux à 24 heures après les étapes ci-dessus, capturez des images de cellules et de HMWFD et/ou de NHF-ATP internalisés à l’aide d’un système d’imagerie par épifluorescence et d’un logiciel d’acquisition de données.
- Quantification et analyse des données
- Pour chaque condition testée, imagez 50 à 100 cellules pour la quantification. À l’aide du logiciel d’analyse de données (logiciel inclus dans le système d’imagerie par épifluorescence ou un autre logiciel), comptez et calculez le nombre moyen de vésicules fluorescentes par cellule.
- Utilisez des méthodes statistiques appropriées pour analyser les résultats quantifiés.
3. Internalisation de l’ATP dans les tumeurs, ex vivo (Figure 3)

Figure 3: Procédure in vivo pour examiner l’internalisation de l’ATP. Représentation schématique du protocole pour visualiser l’internalisation de l’ATP extracellulaire dans les xénogreffes tumorales en utilisant la cryosection et la microscopie à fluorescence. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Préparation de cultures cellulaires pour l’implantation
- Cultiver les cellules cancéreuses à 80 % de confluence à 37 °C dans une fiolede 225 cm 2, en utilisant du DMEM complété par FBS, jusqu’à une concentration finale de 10 % (v/v) et de la pénicilline/streptomycine à 1 % (v/v).
- Lavez les cellules deux fois avec 10 mL de PBS. Préchauffer 0,25 % de trypsine/EDTA à 37 °C. Ajouter 8 mL de trypsine/EDTA et incuber à 37 °C pendant 2 min.
- Une fois que les cellules commencent à se détacher du fond de la fiole, utilisez une pipette sérologique stérile de 10 mL pour ajouter 8 mL de DMEM/FBS. Aspirez deux fois pour déloger les cellules adhérentes. Utilisez la pipette pour transférer les cellules détachées de la fiole dans un tube conique de 50 mL.
- Ajouter 10 mL de DMEM/FBS à l’aide d’une pipette de 10 mL et recueillir toutes les cellules flottantes restantes dans le même tube conique de 50 mL.
- Centrifuger la suspension de la cellule à 600 × g,4 °C pendant 4 min. Retirez le surnageant et remettez les cellules en suspension dans 1 mL de PBS glacé.
- Comptez la densité cellulaire à l’aide d’un hémocytomètre. Gardez la suspension cellulaire sur la glace pendant le comptage.
- Centrifuger la suspension de la cellule à 600 × g,4 °C pendant 4 min. Retirez le surnageant et suspendez les cellules dans du PBS glacé de telle sorte que la densité cellulaire devienne de 5 × 106 cellules pour 100 μL de PBS. Transférer la suspension cellulaire dans un tube de microcentrifugation de 1,5 mL.
- Injection sous-cutanée de cellules cancéreuses pour le développement de tumeurs xénogreffes
- Utilisez une seringue sans latex (1 mL) avec une aiguille de glisse de précision (aiguille de 27 G) pour l’injection de cellules cancéreuses.
- Transférer la suspension cellulaire (5 × 106 en 100 μL de PBS) dans un tube de microcentrifugation de 1,5 mL. Aspirez les cellules dans la seringue.
- Sélectionnez un site d’injection sur le flanc d’une souris immunodéficiente (nue) et nettoyez doucement la peau à l’aide d’éthanol à 75%. Essuyez l’excès d’éthanol avec une tâche délicate.
- Pour l’injection sous-cutanée, maintenez l’aiguille à un angle d’environ 10° par rapport à la peau. Insérez la pointe de l’aiguille, côté biseauté vers le haut, juste sous la peau, de sorte que seulement 1-2 mm de l’aiguille soit visible à l’extérieur de la peau. Distribuer les cellules de la seringue lentement pendant environ 10 s.
- Après avoir injecté tout le volume, continuez à maintenir l’aiguille en place pendant 3 à 5 s, puis retirez l’aiguille et utilisez un doigt pour appliquer une pression douce mais ferme sur le site d’injection pendant 3 à 5 secondes supplémentaires pour éviter toute fuite du contenu injecté.
- Surveillez et mesurez la croissance tumorale à l’aide d’étriers vernier jusqu’à ce que les tumeurs atteignent un volume de 200-500 mm3.
- Préparation de solutions HMWFD et NHF-ATP à utiliser après la résection tumorale
- Dissoudre 300 μL de 16 mg/mL de HMWFD dans du DMEM (milieu de culture) sans sérum, incuber au bain-marie à 37 °C pendant 30 min et centrifuger à 12 000 × g pendant 5 min comme décrit ci-dessus. Transférer la solution dans un tube de microcentrifugation de 1,5 mL.
- Ajouter 40 μL de matière analogique NHF-ATP (1 mM) à 160 μL de DMEM sans sérum pour préparer une solution de NHF-ATP de 0,2 mM.
- Préparation de puits expérimentaux
NOTE: Cette conception expérimentale testera l’internalisation intracellulaire de HMWFD + NHF-ATP, indiquant l’absorption par les macropinosomes.- Préparer les puits comme suit : Puits no 1, Contrôle : 200 μL de DMEM sans sérum; Puits #2, Contrôle: 100 μL de 16 mg/mL de MMAM + 100 μL de DMEM sans sérum = 200 μL de 8 mg/mL de MDF; Puits #3, Contrôle: 100 μL de 0,2 mM NHF-ATP + 100 μL de DMEM sans sérum = 200 μL de 0,1 mM NHF-ATP; Puits #4; Expérimental : 100 μL de 16 mg/mL HMWFD + 100 μL de 0,2 mM NHF-ATP = 200 μL de 0,1 mM NHF-ATP et 8 mg/mL HMWFD.
- Préparation des tissus tumoraux
- Euthanasier la souris par luxation cervicale ou selon le protocole approuvé par l’IACUC.
- Utilisez un scalpel de taille 10 pour trancher les tumeurs isolées à une épaisseur d’environ 500-1 000 μm.
- Incuber les tranches tumorales dans du DMEM sans sérum complété par 100 μM de NHF-ATP et/ou 8 mg/mL H/LMWFD dans des tubes de microcentrifugation pendant 40 min à 37 °C avec un débit de CO2 de 5 %.
REMARQUE: Après l’incubation, le métabolisme des tissus tumoraux provoque un changement de couleur du milieu. - Rincer les tissus dans du PBS préaverti à 37 °C (2 mL pour chaque rinçage dans une plaque de 24 puits).
- Transférer le tissu dans une nouvelle plaque de 24 puits avec du PBS frais préavertissé, rincer et répéter quatre fois en agitant doucement.
- Cryo-intégration (préparation de blocs de tissus congelés)
- Préparer des étiquettes d’identification pour chaque tumeur à récolter. Coupez un morceau de ruban de laboratoire de 2 cm et pliez-le en deux, côtés adhésifs ensemble, dans le sens de la longueur. Utilisez un stylo de marquage pour étiqueter l’étiquette, par exemple, avec un numéro d’identification de souris / tumeur.
- Préparez les moules d’encastrement en plaçant les moules en tissu en acier inoxydable directement sur la glace carbonique.
REMARQUE: La glace carbonique peut causer des engelures, des brûlures et de l’asphyxie. Portez des gants isolés lorsque vous manipulez de la glace carbonique. Utilisez de la glace carbonique dans un endroit bien ventilé. Ne conservez pas la glace carbonique dans un contenant hermétiquement fermé. Au lieu de cela, stockez dans un récipient (comme un refroidisseur en polystyrène) qui permet au gaz de s’échapper. - Pendant que la moisissure refroidit, placez un petit bassin de milieu de congélation des tissus dans une plaque de culture tissulaire de 10 mm. Assurez-vous que le volume est suffisant pour submerger le tissu tumoral qui sera récolté.
- Utilisez une cuillère perforée pour ramasser le tissu tumoral réséqué et placez immédiatement le tissu dans un milieu de congélation, en veillant à ce que le tissu soit submergé. À l’aide de la cuillère perforée, roulez doucement le tissu dans le milieu de congélation, en vous assurant que le milieu baigne toutes les surfaces des tissus.
- Déplacez soigneusement le tissu dans le moule d’encastrement contenant le milieu de congélation. Placez l’étiquette correspondante verticalement dans le milieu de congélation/moule pour congeler en place. Assurez-vous que l’étiquette écrite est visible à l’extérieur du support.
- Lorsque la congélation est terminée (le milieu de congélation devient blanc opaque), retirez le bloc de tissu du moule, placez-le sur de la glace sèche et répétez pour chaque tumeur. Conservez les blocs de tissus à -80 °C pendant plusieurs mois avant la procédure de cryosection.
- Préparation de lames d’échantillons de tissus
- Pour maximiser les chances de trouver des cellules à internalisation positive et d’avoir des régions tissulaires plus représentatives, collectez des cryosections en série à -18 à -20 °C à l’aide d’un cryostat.
- Préchiller les outils cryostat (lame, lame de rasoir, plaque anti-roulis, porte-mandrin en tissu, pinceau) et équilibrer les blocs de tissu tumoral en les plaçant dans une chambre de cryostat à -18 à -20 °C. Réglez l’angle du support de lame sur 5-10°. Coupez soigneusement le bloc de tissu, au besoin, avec une lame de rasoir, et montez-le sur le porte-mandrin en utilisant un milieu de congélation de tissu comme « colle ».
- Verrouillez le porte-mandrin en position verticale sur l’unité de microtome, qui avance à la distance définie (par exemple, 10 μm) à chaque tour de manivelle. Positionnez la plaque anti-roulis pour qu’elle repose juste au-dessus de la hauteur de la lame. Pour éviter le curling des tissus avant de faire avancer le microtome, faites glisser soigneusement le pouce sur le bord inférieur du bloc tissulaire.
- Au fur et à mesure que le microtome avance et que la section de tissu tombe sur la plaque métallique, utilisez un pinceau pour guider la section de tissu et déroulez le tissu, si nécessaire.
- Passez la lame du microscope sur la section de tissu sans la toucher afin que la section soit attirée par la lame.
REMARQUE: Les lames cryostat (hautes profils, jetables) sont extrêmement tranchantes et peuvent causer des blessures graves. Faites preuve de prudence lorsque vous manipulez des lames et utilisez le cryostat. Utilisez un protecteur de lame, le cas échéant. Une formation appropriée est requise. - Découpez la tumeur en sections de 10 μm d’épaisseur. Transférez immédiatement les sections tranchées sur une lame de microscope en verre chargée positivement.
REMARQUE: Pour les sections en série, collectez d’abord une section de 10 μm d’épaisseur dans le coin supérieur gauche de chacune des huit lames chargées positivement. Avancez le cryostat à travers 100 à 200 μm de tissu et jetez le tissu. Transférez immédiatement toutes les sections tranchées sur des lames de microscope en verre. - Ensuite, collectez une autre section de 10 μm d’épaisseur, à côté de la section de tissu précédemment placée, pour chacune des huit lames. Répétez ce processus de collecte en série jusqu’à ce que chacune des huit lames contienne huit sections de tissu, chacune espacée de 100 à 200 μm. Gardez les sections de tissus dans l’obscurité pour préserver la fluorescence.
REMARQUE: Les sections de tissus sur les lames peuvent être stockées dans une boîte à glissière à -80 ° C pendant plusieurs mois.
- Pour maximiser les chances de trouver des cellules à internalisation positive et d’avoir des régions tissulaires plus représentatives, collectez des cryosections en série à -18 à -20 °C à l’aide d’un cryostat.
- Fixation des lames tissulaires
- ÉTAPE CRITIQUE: Fixez les coupes de tissu dans de l’éthanol à 95% à -18 à -20 ° C à pendant 5 min.
- Lavez la section fixe pendant 5 minutes avec du PBS à température ambiante, puis montez les sections tumorales fixes sous un couvercle en verre à l’aide de 10 μL d’un milieu de montage aqueux avec DAPI.
- Douze à 24 heures après le montage, examinez les sections tumorales fixes par microscopie à fluorescence et acquérez des images, comme décrit pour les cellules cultivées ci-dessus.
- Microscopie à fluorescence et acquisition d’images
- Identifier les régions d’intérêt et acquérir des images, comme décrit à la section 2.6.
- Quantification et analyse des données
- Quantifier les cellules et appliquer les analyses statistiques appropriées, comme à la section 2.7.
4. Internalisation de l’ATP dans les tumeurs, in vivo
- Préparer les cultures cellulaires pour l’implantation comme décrit à la rubrique 3.1.
- Injection sous-cutanée de cellules cancéreuses pour le développement de tumeurs xénogreffes
- Générer des tumeurs xénogreffées comme décrit à la rubrique 3.2.
- Injection d’ATP et/ou de dextran dans les tumeurs de xénogreffe
- Préparer les solutions de traitement de DMEM (véhicule) ou de 8 mg/mL HMWFD ou LMWFD, avec ou sans NHF-ATP (100 μM) dans DMEM, comme décrit ci-dessus.
- Utilisez une seringue de 1 mL pour prélever 50 μL d’une solution de traitement et injecter la solution directement dans chaque tumeur de xénogreffe. Répétez la procédure pour quatre répliques biologiques de chaque traitement.
- Prélèvement de tissus et cryo-intégration
- Préparer des étiquettes d’identification pour chaque tumeur à récolter. Coupez un morceau de ruban de laboratoire de 2 cm et pliez-le en deux, côtés adhésifs ensemble, dans le sens de la longueur. Utilisez un stylo de marquage pour étiqueter l’étiquette, par exemple, avec un numéro d’identification de souris / tumeur.
- Environ 5 min après l’injection, euthanasier la souris par luxation cervicale ou selon le protocole approuvé par l’IACUC.
- À l’aide d’un scalpel de taille 10, faites une incision adjacente à la tumeur et approximativement perpendiculaire à la direction de l’injection d’aiguille. Utilisez des pinces et des ciseaux chirurgicaux pour réséquer le tissu tumoral du tissu environnant.
- Divisez la tumeur en deux à quatre pièces de 1 cm2, en fonction de la taille totale de la tumeur.
- Préparez les moules d’encastrement et incorporez le tissu, comme décrit ci-dessus à la section 3.6. Assurez-vous que le temps de récolte, de l’injection intratumorale de dextran à la cryo-intégration, ne dépasse pas 7 à 8 minutes.
- Préparation de lames d’échantillons de tissus
- Prélever des coupes tumorales en série, comme décrit à la rubrique 3.7.
- Fixation des lames tissulaires
- Fixez le tissu, comme décrit à la rubrique 3.8.
- Microscopie à fluorescence et acquisition d’images
- Identifier les régions d’intérêt et acquérir des images, comme décrit à la section 2.6.
- Quantification et analyse des données
- Quantifier les cellules et appliquer les analyses statistiques appropriées, comme décrit à la section 2.7.
Access restricted. Please log in or start a trial to view this content.
Résultats
Étude in vitro
L’internalisation intracellulaire de NHF-ATP a été démontrée par colocalisation de NHF-ATP avec HMWFD ou LMWFD (Figure 4). Le succès de cette procédure repose principalement sur l’utilisation de concentrations appropriées de NHF-ATP et de dextrans et sur la détermination du ou des types appropriés de dextrans (poly-lysine vs neutre). Par exemple, pour étudier la macropinocytose, HMWFD a été choisi car il n’est ...
Access restricted. Please log in or start a trial to view this content.
Discussion
Une méthode a été développée pour l’analyse spatiale, temporelle et quantitative de l’internalisation cellulaire de l’ATP non hydrolysable. Cette méthode est largement applicable pour une utilisation dans divers systèmes biologiques, y compris divers modèles tumorigènes, pour lesquels nous fournissons des instructions techniques et des données représentatives. Pour acquérir des données interprétables lors d’études in vivo d’internalisation de l’ATP (section 4 du protocole), il est esse...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt concurrent.
Remerciements
La cryosection a été réalisée sur place au ohio University Histopathology Core. Ce travail a été soutenu en partie par des fonds de démarrage (Ohio University College of Arts & Sciences) à C Nielsen; Subvention NIH R15 CA242177-01 et prix RSAC à X Chen.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| A549 cells, human lung epithelial, carcinoma | National Cancer Institute | n/a | Less expensive source |
| Acetone | Fisher Scientific | S25904 | |
| Aluminum foil, Reynolds | Grainger | 6CHG6 | |
| Aqueous Mounting Medium, ProLong Gold Anti-fade Reagent | ThermoFisher | P36930 | |
| ATP analog | Jena Biosciences | NK-101 | |
| Autoclave, sterilizer | Grainger | 33ZZ40 | |
| Blades, cryostat, high profile | C. L. Sturkey, Inc. | DT554550 | |
| Calipers, vernier | Grainger | 4KU77 | |
| Cell medium, Ham's Nutrient Mixture F12, serum-free | Millipore Sigma | 51651C-1000ML | |
| Centrifuge, refrigerated with swinging bucket rotor | Eppendorf | 5810R | |
| Chloroform | Acros Organics | 423555000 | |
| Conical tube, 15 mL | VWR | 21008-216 | |
| Conical tube, 50 mL | VWR | 21008-242 | |
| Coverslips, glass, 12 mm | Corning | 2975-245 | |
| Cryostat, Leica CM1950 | Leica Biosystems | CM1950 | |
| Delicate task wipe, Kim Wipes | Kimberly-Clark | 34155 | |
| Dextran, Lysine fixable, High Molecular Weight (HMW) | Invitrogen | D1818 | MW = 70,000, Tetramethylrhodamine |
| Dextran, Neutral, High Molecular Weight (HMW) | Invitrogen | D1819 | |
| Dulbecco's Modified Eagle Medium (DMEM), serum-free | Fisher Scientific | 11885076 | |
| Dry ice | Local delivery | Custom order | |
| Epifluorescent imaging system, Nikon NiU and Nikon NIS Elements acquisition software | Nikon | Custom order | |
| Ethanol | Fisher Scientific | BP2818-4 | |
| Fetal bovine serum (FBS) | ThermoFisher | 16000044 | |
| Forceps, Dumont #7, curved | Fine Science Tools | 11274-20 | |
| Forceps, Dumont #5, straight | Fine Science Tools | 11254-20 | |
| Gloves (small, medium, large) | Microflex | N191, N192, N193 | |
| Gloves, MAPA Temp-Ice 700 Thermal (for handling dry ice) | Fisher Scientific | 19-046-563 | |
| Hemocytometer | Daigger | EF16034F EA | |
| Incubator, cell culture | Eppendorf | Galaxy 170 S | |
| Labelling tape | Fisher Scientific | 159015R | |
| Marking pen, Sharpie (ultra-fine) | Staples | 642736 | |
| Mice, immunodeficient (Nu/J) | Jackson Laboratory | 2019 | |
| Microcentrifuge, accuSpin Micro17 | Fisher Scientific | 13-100-675 | |
| Microcentrifgue tubes, Eppendorf tubes (1.5 mL) | Axygen | MCT-150-C | |
| Microscope slide box | Fisher Scientific | 50-751-4983 | |
| Needle, 27 gauge | Becton-Dickinson | 752 0071 | |
| Paintbrush | Grainger | 39AL12 | |
| Paper towels | Staples | 33550 | |
| Paraformaldehyde | Acros Organics | 416785000 | |
| Penicillin/Streptomycin | Gibco | 15140122 | |
| Perforated spoon, 15 mm diameter, 135 mm length | Roboz Surgical Instrument Co. | RS-6162 | |
| Phosphate buffered saline (PBS) | Fisher Scientific | BP3991 | |
| Pipet tips (10 μL) | Fisher Scientific | 02-707-438 | |
| Pipet tips (200 μL) | Fisher Scientific | 02-707-411 | |
| Pipet tips (1000 μL) | Fisher Scientific | 02-707-403 | |
| Pipets, serological (10 mL) | VWR | 89130-910 | |
| Pippetor, Gilson P2 | Daigger | EF9930A | |
| Pipettor Starter Kit, Gilson (2-10 μL, 20-200 μL, 200-1000 μL) | Daigger | EF9931A | |
| Platform shaker - orbital, benchtop | Cole-Parmer | EW-51710-23 | |
| Positively-charged microscope slides, Superfrost | Fisher Scientific | 12-550-15 | |
| Scalpel, size 10, Surgical Design, Inc. | Fisher Scientific | 22-079-707 | |
| Scissors, surgical - sharp, curved | Fine Science Tools | 14005-12 | |
| Software for image analysis, Nikon Elements | Nikon | Custom order | |
| Software for image analysis, ImageJ (FIJI) | National Institutes of Health | n/a | Download online (free) |
| Specimen disc 30 mm (chuck holder), cryostat accessory | Leica Biosystems | 14047740044 | |
| Staining tray, 245 mm BioAssay Dish | Corning | 431111 | |
| Syringe, 1 cc | Becton-Dickinson | 309623 | |
| Tape, laboratory, 19 mm width | Fisher Scientific | 15-901-5R | |
| Timer | Fisher Scientific | 14-649-17 | |
| Tissue culture dish, 100 x 15 mm diameter | Fisher Scientific | 08-757-100D | |
| Tissue culture flask, 225 cm2 | ThermoFisher | 159933 | |
| Tissue culture plate, 24-well | Becton-Dickinson | 353226 | |
| Tissue embedding mold, stainless steel | Tissue Tek | 4161 | |
| Tissue Freezing Medium, Optimal Cutting Temperature (OCT) | Fisher Scientific | 4585 | |
| Trypsin-EDTA (ethylenediaminetetraacetic acid), 0.25% | Gibco | 25200072 | |
| Water bath, Precision GP 2S | ThermoFisher | TSGP2S |
Références
- Pavlova, N. N., Thompson, C. B. The emerging hallmarks of cancer metabolism. Cell Metabolism. 23 (1), 27-47 (2016).
- Pellegatti, P., et al. Increased level of extracellular ATP at tumor sites: in vivo imaging with plasma membrane luciferase. PLoS ONE. 3, 25992008(2008).
- Falzoni, S., Donvito, G., Di Virgilio, F. Detecting adenosine triphosphate in the pericellular space. Interface Focus. 3 (3), 20120101(2013).
- Michaud, M., et al. Autophagy-dependent anticancer immune responses induced by chemotherapeutic agents in mice. Science. 334, 1573-1577 (2011).
- Wilhelm, K., et al. Graft-versus-host disease is enhanced by extracellular ATP activating P2X7R. Nature Medicine. 16, 1434-1438 (2010).
- Vander Heiden, M. G., Cantley, L. C., Thompson, C. B. Understanding the Warburg effect: the metabolic requirements of cell proliferation. Science. 324, 1029-1033 (2009).
- Cairns, R. A., Harris, I. S., Mak, T. W. Regulation of cancer cell metabolism. Nature Reviews Cancer. 11, 85-95 (2011).
- Chen, X., Qian, Y., Wu, S. The Warburg effect: evolving interpretations of an established concept. Free Radical Biology & Medicine. 79, 253-263 (2015).
- Wang, X., et al. Extracellular ATP, as an energy and phosphorylating molecule, induces different types of drug resistances in cancer cells through ATP internalization and intracellular ATP level increase. Oncotarget. 8 (5), 87860-87877 (2017).
- Patel, A., et al. ATP as a biological hydrotrope. Science. 356, 753-756 (2017).
- Qian, Y., et al. Extracellular ATP is internalized by macropinocytosis and induces intracellular ATP increase and drug resistance in cancer cells. Cancer Letters. 351, 242-251 (2014).
- Qian, Y., Wang, X., Li, Y., Cao, Y., Chen, X. Extracellular ATP a new player in cancer metabolism: NSCLC cells internalize ATP in vitro and in vivo using multiple endocytic mechanisms. Molecular Cancer Research. 14, 1087-1096 (2016).
- Commisso, C., et al. Macropinocytosis of protein is an amino acid supply route in Ras-transformed cells. Nature. 497, 633-637 (2013).
- Li, L., et al. The effect of the size of fluorescent dextran on its endocytic pathway. Cell Biology International. 39, 531-539 (2015).
- Yanagawa, Y., Matsumoto, M., Togashi, H. Enhanced dendritic cell antigen uptake via alpha2 adrenoceptor-mediated PI3K activation following brief exposure to noradrenaline. Journal of Immunology. 185, 5762-5768 (2010).
- Hoppe, H. C., et al. Antimalarial quinolines and artemisinin inhibit endocytosis in Plasmodium falciparum. Antimicrobial Agents & Chemotherapy. 48, 2370-2378 (2004).
- Chaudry, I. H. Does ATP cross the cell plasma membrane. Yale Journal of Biology & Medicine. 55, 1-10 (1982).
- Pant, H. C., Terakawa, S., Yoshioka, T., Tasaki, I., Gainer, H. Evidence for the utilization of extracellular [gamma-32P]ATP for the phosphorylation of intracellular proteins in the squid giant axon. Biochimica et Biophysica Acta. 582, 107-114 (1979).
- Chaudry, I. H., Baue, A. E. Further evidence for ATP uptake by rat tissues. Biochimica et Biophysica Acta. 628, 336-342 (1980).
- Koppenol, W. H., Bounds, P. L., Dang, C. V. Otto Warburg's contributions to current concepts of cancer metabolism. Nature Reviews Cancer. 11, 325-337 (2011).
- Dang, C. V. Links between metabolism and cancer. Genes & Development. 26, 877-890 (2012).
- Israelsen, W. J., Vander Heiden, M. G. ATP consumption promotes cancer metabolism. Cell. 143, 669-671 (2010).
- Koster, J. C., Permutt, M. A., Nichols, C. G. Diabetes and insulin secretion: the ATP-sensitive K+ channel (K ATP) connection. Diabetes. 54, 3065-3072 (2005).
- Szendroedi, J., et al. Muscle mitochondrial ATP synthesis and glucose transport/phosphorylation in type 2 diabetes. PLoS Medicine. 4, 154(2007).
- Miyamoto, S., et al. Mass spectrometry imaging reveals elevated glomerular ATP/AMP in diabetes/obesity and identifies sphingomyelin as a possible mediator. EBioMedicine. 7, 121-134 (2016).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon