Method Article
Criblage multiparamétrique de médicaments organoïdes tumoraux à l’aide de l’imagerie de cellules vivantes à grand champ pour l’analyse en vrac et mono-organoïde
Dans cet article
Résumé
Ce protocole décrit une méthode semi-automatisée pour les criblages de médicaments organoïdes à débit moyen à élevé et un logiciel d’analyse d’images automatisé indépendant du microscope pour quantifier et visualiser les réponses multiparamétriques et mono-organoïdes aux médicaments afin de capturer l’hétérogénéité intratumorale.
Résumé
Les organoïdes tumoraux dérivés du patient (PDTO) sont très prometteurs pour la recherche préclinique et translationnelle et la prédiction de la réponse thérapeutique du patient à partir de criblages de médicaments ex vivo . Cependant, les tests actuels de dépistage de médicaments basés sur l’adénosine triphosphate (ATP) ne saisissent pas la complexité d’une réponse médicamenteuse (cytostatique ou cytotoxique) et l’hétérogénéité intratumorale qui s’est avérée retenue dans les PDTO en raison d’une lecture globale. L’imagerie de cellules vivantes est un outil puissant pour surmonter ce problème et visualiser les réponses aux médicaments plus en profondeur. Cependant, les logiciels d’analyse d’images ne sont souvent pas adaptés à la tridimensionnalité des PDTO, nécessitent des colorants de viabilité fluorescents ou ne sont pas compatibles avec un format de microplaque à 384 puits. Cet article décrit une méthodologie semi-automatisée pour ensemencer, traiter et imager des PDTO dans un format à haut débit de 384 puits à l’aide de systèmes d’imagerie conventionnels à champ large et à cellules vivantes. En outre, nous avons développé un logiciel d’analyse d’images sans marqueur de viabilité pour quantifier les mesures de réponse aux médicaments basées sur le taux de croissance qui améliorent la reproductibilité et corrigent les variations de taux de croissance entre les différentes lignées PDTO. En utilisant la mesure de réponse normalisée aux médicaments, qui évalue la réponse au médicament en fonction du taux de croissance normalisé à une condition de contrôle positive et négative, et un colorant de mort cellulaire fluorescent, les réponses cytotoxiques et cytostatiques aux médicaments peuvent être facilement distinguées, améliorant considérablement la classification des répondeurs et des non-répondeurs. En outre, l’hétérogénéité de la réponse aux médicaments peut être quantifiée à partir de l’analyse de la réponse aux médicaments organoïde unique pour identifier des clones potentiels et résistants. En fin de compte, cette méthode vise à améliorer la prédiction de la réponse thérapeutique clinique en capturant une signature multiparamétrique de réponse au médicament, qui comprend l’arrêt de la croissance cinétique et la quantification de la mort cellulaire.
Introduction
Au cours des dernières années, la découverte in vitro de médicaments contre le cancer, le dépistage de médicaments et la recherche fondamentale sont passés de l’utilisation de modèles de cancer bidimensionnels traditionnels (2D) avec des lignées cellulaires immortalisées à des modèles de cancer tridimensionnels (3D) plus pertinents sur le plan physiologique. Cela a stimulé l’adoption de sphéroïdes tumoraux avec des lignées cellulaires cancéreuses établies, qui recréent des interactions et des structures de cellule à cellule plus complexes présentes dans les tumeurs solides. Actuellement, les organoïdes tumoraux dérivés de patients (PDTO) sont le modèle de cancer 3D le plus avancé et physiologiquement pertinent disponible pour la recherche in vitro sur le cancer, car ils offrent des avantages supplémentaires par rapport aux sphéroïdes tumoraux, à savoir l’hétérogénéité trouvée chez les patients cancéreux1. Les PDTO sont établis à partir de tissus tumoraux provenant de patients cancéreux et conservent donc à la fois le phénotype tumoral et le génotype. En tant que tels, les PDTO deviennent inestimables pour la recherche fondamentale et translationnelle sur le cancer et ont le potentiel d’améliorer considérablement la précision de l’oncologie2.
Malgré leur potentiel prometteur, ces modèles 3D sophistiqués de cancer in vitro sont souvent sous-utilisés en raison d’un manque de méthodes d’analyse avancées. Le test le plus couramment utilisé détermine le nombre de cellules viables dans le PDTO via la quantification de l’ATP3 intracellulaire. Ces tests sont normalement des analyses globales à point temporel unique, négligeant ainsi les réponses critiques dépendantes du temps et négligeant les réponses clonales. Plus précisément, la capacité de surveiller la croissance des PDTO (taux de croissance) et leur réponse à des thérapies spécifiques présente un grand intérêt 4,5. La réponse normalisée aux médicaments (NDR), qui évalue la réponse au médicament en fonction du taux de croissance normalisé à une condition positive (ctrl+) et de contrôle négatif (ctrl-), a également été récemment considérée comme une mesure cruciale pour évaluer la sensibilité aux médicaments anticancéreux avec le dépistage cellulaire, bien que cela ait été principalement fait pour les lignées cellulaires2D 6. Par conséquent, des méthodes d’analyse plus sophistiquées sont nécessaires pour tirer pleinement parti de ces modèles de cancer 3D plus représentatifs et plus complexes sur le plan clinique. La microscopie est considérée comme une approche puissante pour étudier la complexité de ces modèles organoïdes7.
Cet article décrit une méthode de surveillance des réponses cinétiques aux médicaments dans des modèles 3D de cancer, à l’aide de microscopes à grand champ conventionnels et de systèmes d’imagerie de cellules vivantes. Des adaptations ont été apportées au protocole décrit par Driehuis et al.4 pour être compatible avec l’automatisation à l’aide d’un robot de pipetage, d’un distributeur numérique de médicaments et d’un système d’imagerie de cellules vivantes afin d’augmenter la reproductibilité et de réduire le nombre d’heures de travail « pratique ». Cette méthode permet le criblage médicamenteux à débit moyen à élevé des sphéroïdes tumoraux avec des lignées cellulaires cancéreuses établies (voir le tableau supplémentaire S1 pour les lignées cellulaires testées), ainsi que des PDTO, dans un format microplaque et multi-organoïde à 384 puits. En utilisant un processus d’apprentissage automatique en réseau convolutionnel, l’identification et le suivi automatisés des sphéroïdes tumoraux individuels ou PDTO pourraient être effectués uniquement à partir de l’imagerie en fond clair et sans l’utilisation de colorants fluorescents pour le marquage des cellules vivantes8. Ceci est très avantageux, car la plupart des identifications avec l’imagerie en fond clair nécessitent une annotation manuelle (ce qui est laborieux et prend beaucoup de temps) ou nécessitent l’ajout de colorants fluorescents, ce qui peut confondre les réponses médicamenteuses liées au stress oxydatif induit par la photoxicité9.
Le logiciel d’analyse d’images qui en résulte, développé en interne, étend les fonctionnalités des systèmes d’imagerie de cellules vivantes conventionnels, car les modules d’analyse d’images 3D ne sont pas disponibles, limités par la plate-forme ou ne sont pas compatibles avec les microplaques 384 puits et l’imagerie de puits entiers. De plus, ces modules sont souvent très chers et offrent des lectures organoïdes en vrac limitées. Par conséquent, cette méthode est très pertinente pour les scientifiques qui ont accès à des systèmes d’imagerie de cellules vivantes largement disponibles et visent à extraire plus d’informations sur une réponse médicamenteuse par rapport au test de référence mais rudimentaire basé sur l’ATP. Avec l’ajout d’indicateurs spécifiques de mort cellulaire, les réponses cytostatiques aux médicaments peuvent être distinguées des réponses cytotoxiques, fournissant ainsi un aperçu supplémentaire des actions mécanistes des médicaments actuellement inaccessibles à partir d’une analyse ponctuelle unique. Enfin, l’imagerie de cellules vivantes permet un suivi organoïde individuel pour obtenir des mesures de réponse aux médicaments organoïdes uniques afin de saisir l’hétérogénéité de la réponse et d’identifier les sous-clones potentiellement résistants.
L’objectif de cette méthode et du logiciel d’analyse d’images associé est de mettre en œuvre une automatisation à faible coût dans le dépistage de médicaments organoïdes afin de limiter l’intervention des utilisateurs et de réduire la variabilité dans la manipulation, l’analyse d’images et l’analyse de données. Pour mettre ce logiciel à la disposition des chercheurs, il est indépendant du microscope et de la plate-forme, et une application basée sur le cloud est mise à disposition. Ainsi, en prenant en charge les systèmes conventionnels d’imagerie de cellules vivantes, nous visons également à améliorer leur fonctionnalité pour les applications et l’analyse de culture 3D.
Protocole
Des organoïdes dérivés de patients atteints d’adénocarcinome canalaire pancréatique humain (PDAC) ont été utilisés. Des fragments de résection tissulaire ont été obtenus chez des patients subissant une chirurgie curative à l’hôpital universitaire d’Anvers. Le consentement éclairé écrit a été obtenu de tous les patients et l’étude a été approuvée par le comité d’éthique de l’UZA (réf. 14/47/480). Les détails relatifs à tous les matériaux, réactifs, équipements et logiciels utilisés dans ce protocole sont fournis dans le tableau des matériaux. Une vue d’ensemble du flux de travail est présentée à la figure 1. Des exemples de données sont fournis dans le matériel supplémentaire pour reproduire le protocole.
1. Jour 0 : Préparation des organoïdes âgés de 2 ou 3 jours
- Préchauffer les microplaques à 37 °C pendant une nuit et décongeler la matrice extracellulaire (MEC) à 4 °C.
- Préparer le milieu de culture organoïde PDAC complet : supplément ADF+++ (DMEM/F12 avancé, supplément de glutamine à 1 %, HEPES à 1 % et 1 % de pénicilline/streptomycine) avec 0,5 nM de protéine de substitution WNT-Fc-Fusion, milieu conditionné à la protéine de fusion Noggin-Fc à 4 %, milieu conditionné par protéine de fusion Rpso3-Fc, 1x B27, 1 mM N-acétylcystéine (NAC), nicotinamide 5 mM, 500 nM A83-01, 100 ng/mL FGF10 et 10 nM Gastrin).
- Établir les PDTO selon la méthode choisie.
REMARQUE: Un protocole détaillé est fourni par Driehuis et al., qui décrit la méthode conventionnelle pour établir, mettre en culture et passer des PDTO dans les dômes ECM4. - Dissocier enzymatiquement les organoïdes dans les dômes ECM.
- Aspirer le milieu et laver 1x avec une solution saline tamponnée au phosphate (PBS). Ajouter l’enzyme de dissociation (p. ex., 2 mL dans une microplaque à 6 puits) et pipeter de haut en bas 10x avec une pipette de 1 mL pour dissocier mécaniquement les organoïdes et les dômes ECM.
- Incuber pendant 10 min à 37 °C, pipeter de haut en bas et vérifier si les organoïdes sont dissociés en cellules individuelles. Répétez cette étape si nécessaire.
- Recueillir la suspension cellulaire dans un tube de 15 mL, ajouter ADF+++ à un volume de 10 mL, centrifuger pendant 5 min à 450 × g à température ambiante et aspirer le surnageant avec une pipette Pasteur et une pompe d’aspiration.
- Remettez la pastille en suspension dans 100-200 μL de milieu complet selon la taille de la pastille et comptez le nombre de cellules en utilisant la méthode choisie. Par exemple : mélangez 10 μL de suspension cellulaire + 10 μL de Trypan Blue et comptez avec un compteur de cellules automatisé.
- Plaquez des cellules individuelles dans des dômes ECM.
- Diluer la suspension cellulaire et ajouter 2/3 ECM conformément au tableau 1. Pipeter jusqu’à dix gouttelettes de 20 μL par puits dans une plaque préchauffée de 6 puits. Retourner la plaque et incuber pendant 30 min à 37 °C.
- Superposition avec milieu complet complété par 10 μM Y-27632 et incuber pendant 2-3 jours dans un incubateur.
REMARQUE: Dix dômes contenant 75 000 cellules chacun sont généralement suffisants pour remplir une microplaque de 384 puits à une concentration de 200 organoïdes / puits, à l’exclusion des puits en bord.
2. Jours 2 - 3: Récolte et semis d’organoïdes âgés de 2 ou 3 jours
- Collectez les organoïdes intacts des dômes ECM.
REMARQUE : Les organoïdes ont tendance à adhérer aux surfaces en plastique (p. ex. tubes, pointes de pipettes). Pour éviter cela, les articles en plastique peuvent être prérincés avec une solution d’albumine sérique bovine (BSA)/PBS à 0,1%.- Aspirer le milieu et laver 1x avec du PBS. Ajouter 1 à 2 mL de solution de récolte organoïde froide (4 °C) dans une plaque de 6 puits, selon le nombre de dômes ECM, et incuber sur de la glace sur une plate-forme agitée pendant 10 minutes.
- Pipette de haut en bas avec une pipette de 1 mL pour dissocier les dômes ECM, incuber pendant 10 minutes supplémentaires sur la glace et vérifier visuellement au microscope si l’ECM est dissocié.
- En option : si une distribution granulométrique plus uniforme est préférable, filtrer la suspension à travers une crépine cellulaire de 70 μm avant la centrifugation.
- Recueillir les organoïdes dans un tube de 15 mL préduit de BSA/PBS à 0,1 %, ajouter ADF+++ jusqu’à 10 mL et centrifuger pendant 5 min à 200 × g à 4 °C. Aspirer le surnageant et remettre en suspension la pastille dans un maximum de 1 000 μL de milieu organoïde PDAC complet selon la taille de la pastille pour obtenir une concentration de >6 000 organoïdes/mL.
- Comptez les organoïdes en utilisant n’importe quelle méthode de comptage de votre choix, de préférence basée sur l’image.
- Ensemencer les organoïdes.
REMARQUE : Voir le tableau des matériaux pour connaître le volume minimal ou le puits pour deux types différents de microplaques à 384 puits utilisés dans ce protocole.- Prérefroidir tous les articles en plastique à -20 °C ou sur de la glace pendant au moins 20 minutes avant utilisation pour éviter la solidification de l’ECM.
- Préparer la solution d’ensemencement à partir de 1 mL de solution mère organoïde (étape 2.1.6) en utilisant le milieu complet pour ensemencer ~200 organoïdes par puits dans 50 μL, le volume minimum utilisé pour remplir un puits. Utilisez le fichier supplémentaire 1 pour calculer la quantité de solution d’ensemencement organoïde. Ajouter un volume résiduel de 1 500 μL lors de l’utilisation d’un réservoir de 25 mL et d’une pipette multicanal ou d’un robot de pipetage.
- Ensemencer les organoïdes à l’aide d’un robot de pipetage.
NOTE: La solution d’ensemencement et la microplaque doivent être refroidies à 4 °C pendant le pipetage pour éviter la solidification de l’ECM. Par conséquent, un réservoir de 25 mL et un porte-microplaque ont été imprimés en 3D pour être utilisés en combinaison avec le robot de pipetage pouvant contenir les éléments de refroidissement énumérés dans le tableau des matériaux. Les fichiers STL pour l’impression 3D du matériel de laboratoire personnalisé (fichier supplémentaire 2 et fichier supplémentaire 3) et les fichiers JSON de laboratoire personnalisés pour le robot de pipetage (fichier supplémentaire 4 et fichier supplémentaire 5) sont fournis.- Concevez le protocole de distribution à l’aide de l’outil de conception de protocole en ligne. Un exemple de fichier JSON (fichier supplémentaire 6) est fourni, dans lequel le matériel de laboratoire personnalisé est déjà chargé et une pipette p300 (Gen2) à huit canaux avec les pointes de pipette correspondantes est utilisée.
- Ouvrez l’application de contrôle du robot de pipetage, sélectionnez les protocoles, cliquez sur Importer et faites glisser et déposez le fichier supplémentaire 6 dans le champ désigné.
- Sélectionnez le protocole importé et placez tous les articles de laboratoire, y compris les éléments de refroidissement et les articles en plastique, dans les platines conformément à la disposition indiquée dans le champ Configuration du pont . Utilisez la fente gauche pour le réservoir de 25 mL et l’élément de refroidissement, comme indiqué dans le dossier supplémentaire 7.
- Cliquez sur Exécuter le protocole et passez à la configuration. Ouvrez l’onglet Labware Setup (Configuration du logiciel Labware), cliquez sur Run Labware Position Check (Exécuter la vérification de la position du Labware) et suivez les instructions pour calibrer le robot de pipetage sur le nouveau matériel.
REMARQUE : les données de décalage du logiciel de laboratoire peuvent être stockées pour plus tard, mais il est recommandé d’exécuter la vérification de la position du logiciel de laboratoire avant chaque exécution. - Remplissez le réservoir de 25 mL (placé sur le dessus de l’élément de refroidissement) avec la solution d’ensemencement organoïde refroidie et cliquez sur Start Run (Démarrer l’exécution).
REMARQUE: Les puits supérieur et inférieur sont également remplis de solution de suspension organoïde en raison de l’utilisation de la pipette à huit canaux. - Centrifuger la microplaque pendant 1 min à 100 × g à 4 °C.
- Incuber à 37 °C pendant au moins 30 min.
- Remplir les puits blancs extérieurs avec au moins 50 μL deH2Opour éviter l’évaporation.
- Incuber à 37 °C pendant une nuit pour éliminer les bulles dans le puits qui peuvent interférer avec l’analyse de l’image.
3. Jour 4 : Traitement médicamenteux et distribution de réactifs avec distributeur numérique de médicaments
- Créez le protocole de distribution de médicaments à l’aide du logiciel de contrôle numérique des distributeurs de médicaments.
- Passez la souris sur la plaque 1 au-dessus de la disposition de la plaque, sélectionnez Modifier les attributs de la plaque et remplissez le type de plaque : 384 puits, volume supplémentaire (μL) : 50 et limite DMSO (%) : 1.
- Ajoutez des fluides en cliquant sur le bouton + à côté de Fluides. Double-cliquez sur le fluide nouvellement créé et nommez-le; sélectionner la classe (à base de DMSO ou aqueux + Tween 20) et la concentration.
REMARQUE: Tous les médicaments et réactifs doivent être dissous dans 100% DMSO ou 0,3% Tween-20. Une solution mère de 1 à 10 mM peut être utilisée, en tenant compte d’une concentration maximale de DMSO de <1%. Le tableau 2 donne des exemples des dilutions requises pour les réactifs fluorescents courants et les thérapies. - Disposition des plaques
- Pour le titrage des médicaments, sélectionnez les puits et cliquez sur Titrage. Pour le liquide, sélectionnez le médicament d’intérêt, choisissez la concentration la plus élevée (p. ex. 2 000 nM) et la concentration la plus faible (p. ex. 10 nM); Pour les répétitions, choisissez un minimum de 2 et choisissez le modèle de titrage souhaité.
REMARQUE : Le schéma de titrage dépendra de nombreux facteurs, y compris la quantité de composé qui doit être insérée dans une seule plaque, si les puits doivent être randomisés et le nombre de répétitions et de témoins. - Pour le contrôle positif, sélectionnez trois puits, cliquez sur Définir la valeur et remplissez 2 μM de staurosporine à partir de 10 mM de stock dans DMSO, ce qui induira une mort cellulaire maximale.
- Pour Cytotox Green, sélectionnez tous les puits utilisés, cliquez sur Set Value (Définir la valeur) et entrez 60 nM/puits.
REMARQUE: La coloration fluorescente Cytotox Green indique que les cellules sont mortes et n’interférera donc pas avec la surveillance de la réponse aux médicaments. Ici, aucun marqueur fluorescent pour les cellules vivantes n’est requis. - Pour le contrôle négatif et la normalisation DMSO, sélectionnez tous les puits avec quatre puits supplémentaires pour la commande du véhicule, cliquez avec le bouton droit de la souris, sélectionnez la normalisation, sélectionnez normaliser la classe de fluide: basé sur DMSO, et normaliser au volume de classe le plus élevé pour obtenir une concentration égale de DMSO dans chaque puits.
REMARQUE: Les concentrations de DMSO doivent être de <1%. Un exemple de fichier de titrage des médicaments ATS (dossier supplémentaire 8) est fourni. - Cliquez sur la flèche sous Exécuter dans le coin supérieur gauche, sélectionnez Toujours simuler, puis cliquez sur Simuler pour identifier les erreurs et obtenir les volumes de chaque médicament à préparer.
REMARQUE : Pour surmonter un avertissement lorsque le volume de distribution initial est trop faible, « Un puits d’avertissement de distribution de 30 nL ou plus est recommandé pour chaque fluide sur chaque plaque », sélectionnez deux puits sur le bord qui sont remplis d’eau, sélectionnez Définir la valeur et entrez 10 μM du médicament pour lequel l’avertissement se produit. Cela amorce la cartouche de médicament avec un volume supérieur à 30 nL. Ces mêmes puits peuvent être utilisés pour amorcer la cartouche de DMSO en définissant une normalisation à % de la valeur du volume total (par exemple, 0,5%).
- Pour le titrage des médicaments, sélectionnez les puits et cliquez sur Titrage. Pour le liquide, sélectionnez le médicament d’intérêt, choisissez la concentration la plus élevée (p. ex. 2 000 nM) et la concentration la plus faible (p. ex. 10 nM); Pour les répétitions, choisissez un minimum de 2 et choisissez le modèle de titrage souhaité.
- Décochez Toujours simuler sous le bouton Exécuter ; cliquez sur Exécuter pour démarrer le protocole de distribution du médicament et suivez les instructions.
- Appliquez la membrane d’étanchéité sur la microplaque pour éviter l’évaporation.
- Incuber le colorant vert Cytotox 1-2 h à 37 °C dans l’incubateur et passer à l’étape 4.
4. Acquérir des images avec l’imageur de cellules vivantes
REMARQUE: Pour le taux de croissance et la NDR, une scintigraphie au point de temps 0 (T0 = début du traitement) doit être acquise 1-2 h après l’ajout de Cytotox Green.
- Ouvrez le logiciel de contrôle de l’imageur de cellules vivantes, sélectionnez Éditeur de méthode Nouveau, accédez au fichier > importation, puis sélectionnez l’exemple de fichier XML de méthode (fichier supplémentaire 9). Vous pouvez également créer un nouveau fichier et sélectionner Plate: (CORE384fb_OpticalImaging) - Corning 384 Flat Black (Corning #4588), No lid and No moisture cassette; Application: Images seulement; Objectif : 4x; Modèle : Central; Vérifiez les canaux Brightfield et Green (intensité de la LED (%) = 40; Temps d’exposition (ms) = 200).
REMARQUE: Les paramètres du canal vert fonctionnent bien pour une concentration de 60 nM Cytotox Green. L’option Live viewer peut être utilisée pour ajuster le décalage de mise au point et/ou les paramètres de led en temps réel. - Cliquez sur Démarrer pour lancer l’analyse à T0.
- Répétez l’analyse toutes les 24 heures pendant 5 jours maximum en utilisant la même méthode. Sinon, pour exécuter automatiquement la mesure timelapse, ajustez la méthode dans le logiciel de contrôle de l’imageur de cellule sous tension à une expérience cinétique en cliquant et en faisant glisser l’onglet Boucle cinétique dans le champ de la méthode . De même, les onglets Température et Gaz doivent être glissés dans le champ de la méthode pour régler le système à 37 °C et 5% de CO2 pour garantir les conditions correctes dans l’imageur de cellule vivante pendant l’expérience.
5. Analyse des images et des données
- Fusion et compression des données
- Le logiciel de contrôle de l’imageur de cellules vivantes génère un dossier pour chaque numérisation à chaque point de temps. Créez un nouveau dossier, copiez les dossiers d’expérience individuels dans ce nouveau dossier parent et ajoutez _0h, _24h, _48h, _72h, _96h et _120h aux noms de dossier d’expérience correspondants.
- Préparer une carte de plaque XLSX à partir du logiciel de contrôle numérique des distributeurs de médicaments en cliquant avec le bouton droit de la souris sur la disposition de la carte des plaques à partir du protocole de distribution du médicament et copier tous les puits; coller les données dans un fichier XLSX. Supprimez les données de Cytotox Green et de staurosporine et ajoutez une matrice pour la lignée cellulaire et la réplication. Entrez ctrl- et ctrl+. Voir le fichier supplémentaire 10 pour un exemple de carte de plaque.
- Ouvrez l’outil de compression de données, cliquez sur Parcourir, sélectionnez le dossier parent, puis cliquez sur Exécuter pour lancer la compression des données d’image. Tous les fichiers image TIFF pour les différents points de temps sont compressés en un seul HDF5 pour chaque puits dans un nouveau dossier de jeux de données dans le dossier parental.
- Analyse d’images
- Accédez à la plateforme d’analyse d’images, connectez-vous et cliquez sur Ajouter un nouveau projet dans l’onglet Accueil . Entrez le nom du projet, continuez, sélectionnez Ajouter une nouvelle expérience et téléchargez le dossier des jeux de données contenant les fichiers HDF5.
- Après le téléchargement, accédez au dossier du projet et de l’expérience et cliquez sur Télécharger la carte pour obtenir des fonctionnalités supplémentaires. Cliquez sur Exécuter l’analyse, sélectionnez Analyse multi-organoïde, Paramètres par défaut, puis cliquez sur Analyser pour lancer l’analyse de l’image.
- Cliquez sur Télécharger les résultats pour télécharger les tableaux de données brutes qui contiennent les mesures pour chaque puits (par exemple, la surface totale en fond clair, la zone verte de fluorescence totale, etc.) et les images / vidéos segmentées pour confirmer l’exactitude de l’analyse et du traitement ultérieur des données.
- Paramètres de réponse aux médicaments basés sur le taux de croissance et réponse normalisée aux médicaments
- Sélectionnez le fichier Raw_NDR.xlsx dans le dossier de résultats (carte de plaque requise) (fichier supplémentaire 11) et chargez-le dans le script R Official_NDR_7point (fichier supplémentaire 12) pour générer automatiquement des tables de valeurs GR (normalisé en ctrl-) et NDR (normalisé en ctrl- et ctrl+) (fichier supplémentaire 13, fichier supplémentaire 14, fichier supplémentaire 15 et fichier supplémentaire 16 ). Les valeurs GR et NDR sont calculées à partir du paramètre comme indiqué dans l’équation (1) à l’aide du script R (fichier supplémentaire 12).
Surface de survie totale = Surface totale en fond clair - Superficie verte totale (1)
Où 0 < NDR <1 = effet cytostatique (arrêt de croissance) et NDR < 0 = réponse cytotoxique (mort cellulaire).
NOTE: Le script R a été adapté de Gupta et al.6. - À partir de la table clonal_data.xlsx, récupérez les données de réponse d’un seul organoïde et tracez-les sous forme de graphique à bulles.
- Utilisez le facteur Z10 pour évaluer la qualité du dépistage d’une série (voir l’équation (2)). Rejeter une expérience avec un facteur Z < 0,5.
 (2)
(2)
- Sélectionnez le fichier Raw_NDR.xlsx dans le dossier de résultats (carte de plaque requise) (fichier supplémentaire 11) et chargez-le dans le script R Official_NDR_7point (fichier supplémentaire 12) pour générer automatiquement des tables de valeurs GR (normalisé en ctrl-) et NDR (normalisé en ctrl- et ctrl+) (fichier supplémentaire 13, fichier supplémentaire 14, fichier supplémentaire 15 et fichier supplémentaire 16 ). Les valeurs GR et NDR sont calculées à partir du paramètre comme indiqué dans l’équation (1) à l’aide du script R (fichier supplémentaire 12).
Résultats
Le protocole de pipetage automatisé assure une répartition uniforme des PDTO PDAC_060 dans toutes les colonnes de la microplaque à 384 puits (Figure 2A). Comme prévu, une variation du nombre et de la superficie moyenne des PDTO a été observée entre les puits (figure 2A, B). La surface de survie totale (surface totale en fond clair - surface verte totale) combine la segmentation organoïde sans marquage avec le signal de mort cellulaire basé sur la fluorescence et constitue, selon notre expérience, le paramètre le plus robuste pour étudier les réponses aux médicaments au fil du temps (Figure 2C)8. Pour tenir compte des variations dans l’ensemencement cellulaire et la taille des organoïdes, des paramètres basés sur le taux de croissance devraient être utilisés pour réduire les variations entre les répétitions, comme le montrent les barres d’erreur réduites de la figure 2D par rapport à la figure 2C, et un facteur Z plus élevé indiquant une qualité de dépistage des médicaments fortement améliorée (figure 2E).
La courbe dose-réponse NDR (figure 2G), normalisée en ctrl- et ctrl+, est nettement supérieure à la courbe dose-réponse GR (figure 2F), normalisée en ctrl-, car elle augmente la séparation des courbes de réponse au médicament et représente plus précisément les réponses cytotoxiques aux médicaments. Un exemple des images associées pour le PDTO traité par ctrl, ctrl+ et 400 nM gemcitabine/80 nM de paclitaxel est présenté à la figure 3. Une observation intéressante est que l’effet cytotoxique de la gemcitabine était dominant dans la thérapie combinée car aucune valeur ajoutée du paclitaxel n’a été observée.
Ensuite, deux lignes PDTO supplémentaires, PDAC_052 et PDAC_087, ont été utilisées. Une nette différence de taux de croissance entre ces lignes a été observée (figure 4A), ce qui appuie l’utilisation de paramètres GR. Encore une fois, les courbes dose-réponse NDR (Figure 4C) ont entraîné une augmentation de la plage dynamique et de la séparation entre les trois patients différents par rapport aux courbes GR (Figure 4B). De plus, le protocole permet de déterminer le NDR au fil du temps et montre que PDAC_052 et PDAC_060 avaient une réponse cytostatique très similaire à une faible dose de gem-pac (Figure 4D), tandis qu’une réponse cytostatique par rapport à cytotoxique différentielle claire pouvait être observée pour les doses moyennes (Figure 4E) et élevées (Figure 4F) de gem-pac. Ces réponses médicamenteuses étaient cohérentes avec les réponses cliniques observées chez les patients (Figure 4G).
Enfin, un avantage majeur de l’approche et du logiciel est que les réponses aux médicaments organoïdes uniques peuvent être quantifiées pour étudier l’hétérogénéité de la réponse et identifier les sous-clones potentiellement résistants. La figure 5 donne un aperçu clair de la dynamique clonale des différents patients et montre que PDAC-087 avait les sous-clones les plus résistants après traitement, ce qui est cohérent avec la maladie agressive et hautement résistante observée chez le patient. Fait intéressant, ce patient était également le moins sensible à la staurosporine ctrl +.
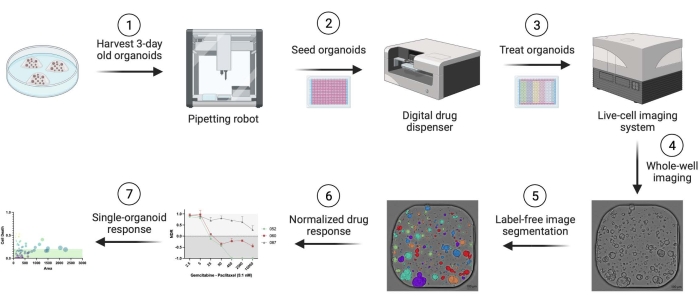
Figure 1 : Vue d’ensemble du flux de travail. Veuillez cliquer ici pour voir une version agrandie de cette figure.
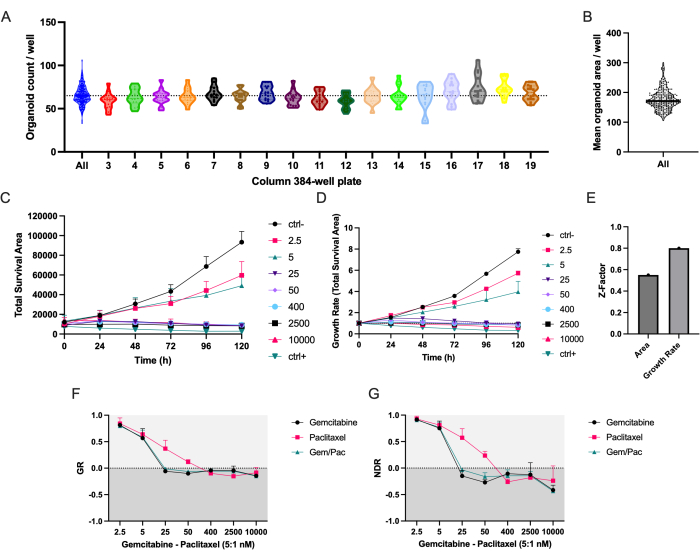
Figure 2 : Précision de l’ensemencement et mesures de réponse aux médicaments. (A) Dénombrement organoïde/puits de PDAC_060 PDTO ensemencés dans une microplaque de 384 puits à l’aide du robot de pipetage. Chaque point représente le nombre dans un seul puits et les tracés sont séparés par les colonnes de microplaques de 384 puits. (B) Superficie ou puits moyen de PDTO. (C) Surface de survie totale (surface totale en fond clair - surface verte totale) et (D) taux de croissance (surface de survie totale normalisée à T0 = 1) des PDTO PDAC_060 traités avec un rapport 5:1 gemcitabine/paclitaxel. (E) Facteur Z comme mesure de la qualité des essais. (F) Courbe taux de croissance-dose-réponse normalisée à ctrl- et (G) courbe normalisée de réponse au médicament normalisée à ctrl- et ctrl+. Les barres d’erreur indiquent la moyenne ± SD de deux puits. Abréviations : PDAC = adénocarcinome canalaire pancréatique; PDTO = organoïde tumoral dérivé du patient; GR = taux de croissance; NDR = réponse médicamenteuse normalisée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
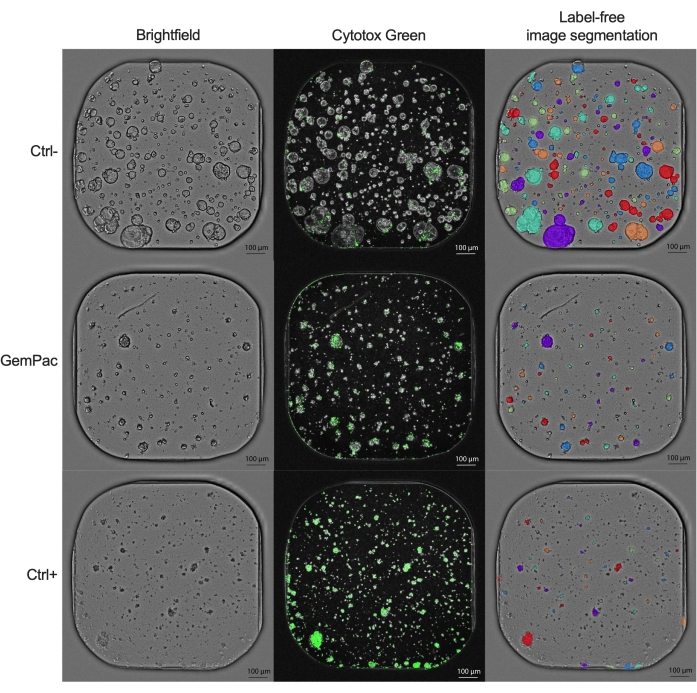
Figure 3 : Exemples d’images. Images représentatives d’PDAC_060 PDTO traitées avec du véhicule (ctrl-), 400 nM gemcitabine/80 nM paclitaxel et 2 μM staurosporine (ctrl+). La colonne de gauche montre les images en fond clair, la colonne du milieu montre le signal fluorescent Cytotox Green et la colonne de droite montre les images de fond clair annotées sans étiquette à l’aide du module d’analyse organoïde. Barres d’échelle = 100 μm. Abréviations : PDAC = adénocarcinome canalaire pancréatique; PDTO = organoïde tumoral dérivé du patient; GemPac = gemcitabine/paclitaxel. Veuillez cliquer ici pour voir une version agrandie de cette figure.
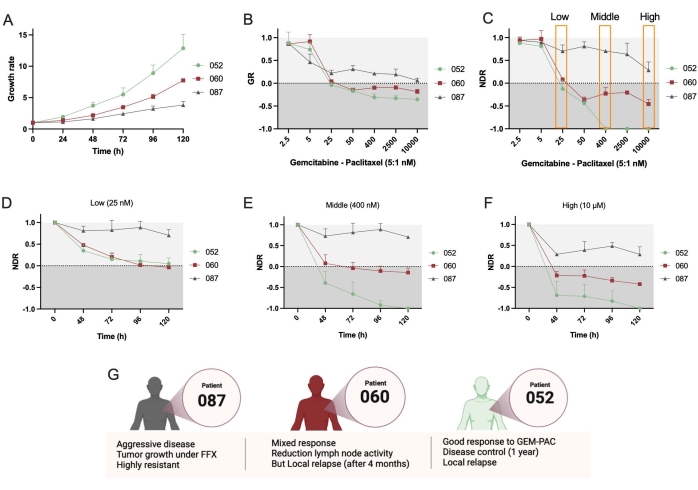
Figure 4 : Comparaison de la réponse interpatiente aux médicaments. (A) Comparaison du taux de croissance (basé sur la surface de survie totale) des lignées PDTO PDAC_052, PDAC_060 et PDAC_087. (B) Courbe taux de croissance-dose-réponse normalisée à ctrl- et (C) courbe normalisée de réponse au médicament normalisée à ctrl- et ctrl+. NDR cinétique d’une dose (D) faible, (E) moyenne et (F) élevée de gemcitabine/paclitaxel (rapport 5:1). (G) les caractéristiques cliniques des patients PDAC. Les barres d’erreur indiquent la moyenne ± SD de deux puits. Abréviations : PDAC = adénocarcinome canalaire pancréatique; PDTO = organoïde tumoral dérivé du patient; GR = taux de croissance; NDR = réponse normalisée au médicament; FFX = folfirinox. Veuillez cliquer ici pour voir une version agrandie de cette figure.
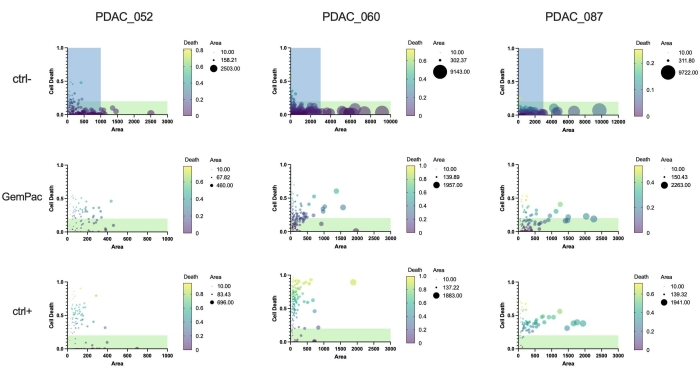
Figure 5 : Mesures organoïdes uniques. Dose-réponse organoïde unique basée sur la mort cellulaire (zone verte/zone de fond clair) et l’aire (fond clair) des PDTO PDAC_052, PDAC_060 et PDAC_087 traitées avec du véhicule (ctrl-), 400 nM gemcitabine/80 nM de paclitaxel et 2 μM de staurosporine (ctrl+). Les régions vertes indiquent des organoïdes viables; les régions bleues indiquent la plage x-as des tracés GemPac et ctrl+. Abréviations : PDAC = adénocarcinome canalaire pancréatique; PDTO = organoïde tumoral dérivé du patient; GemPac = gemcitabine/paclitaxel. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Stock de suspension cellulaire | Cellules/goutte | # Gouttes (20 μL) | Stock (1/3) | ECM (2/3) |
| 1,13 × 107 cellules/ml | 75,000 | 10 | 75 uL | 150 μL |
| 1,13 × 107 cellules/ml | 75,000 | 5 | 40 uL | 80 μL |
Tableau 1 : Dilution pour le placage dans les dômes ECM. Abréviation : ECM = matrice extracellulaire.
| Composé | Concentration des stocks | Dilution | Concentration de travail | Solvant | Concentration du puits | Commentaires |
| Cytotox Vert | 1 mM (DMSO) | 1/10 | 10 μM | DMSO | 60 nM | Marqueur de mort cellulaire |
| Cytotox Rouge | 1 mM (DMSO) | 1/10 | 10 μM | DMSO | 250 nM | Marqueur de mort cellulaire |
| Caspases 3/7 Vert | 5 mM (DMSO) | 1/2 | 2,5 mM | DMSO | 2,5 μM | Marqueur apoptotique |
| Hoechst | 20 mM (H2O) | 1/200 | 100 μM | 0,33 % préadolescents/PBS | 50 nM | Marqueur nucléaire |
| Staurosporin | 10 mM (DMSO) | / | 1 - 10 mM | / | 2 – 5 μM | Contrôle positif |
| Gemcitabine | 10 mM (DMSO) | / | 1 - 10 mM | / | Titrage | Chimiothérapie |
| Paclitaxel | 10 mM (DMSO) | / | 1 - 10 mM | / | Titrage | Chimiothérapie |
| Cisplatine | 5 mM (0,9 % NaCl) | 1/2 | 2,5 mM | 0,6 % préadolescents/PBS | Titrage | Chimiothérapie |
Tableau 2 : Exemples de dilutions de médicaments et de réactifs fluorescents fréquemment utilisés. Chaque composé doit être dissous dans 100% DMSO ou 0,3% Tween/PBS.
Tableau supplémentaire S1 : Vue d’ensemble des lignées cellulaires cancéreuses compatibles. Statique : les sphéroïdes ne sont pas migrateurs. Fusionner : les sphéroïdes migrent les uns vers les autres et fusionnent ensemble. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 1 : Outil de calcul de solution d’ensemencement organoïde. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 2: Fichier STL pour l’impression 3D de matériel de laboratoire personnalisé 'Microplate Holder'. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 3 : Fichier STL pour l’impression 3D de matériel de laboratoire personnalisé '2 x 25 mL Reservoir Holder'. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 4: Fichier JSON pour robot de pipetage de matériel de laboratoire personnalisé 'Microplate Holder'. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 5 : Fichier JSON pour robot de pipetage de laboratoire personnalisé '2 x 25 mL Reservoir Holder_WithCooler'. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 6 : fichier JSON pour le protocole de robot de pipetage 'Plating_ PDO_384well_Cooled_Row2-23'. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 7 : Vue d’ensemble de la configuration du bureau du robot pipeteur. (A) Éléments de refroidissement et (B) réservoir et microplaque. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 8 : Fichier TDD pour le protocole du distributeur numérique de médicaments. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 9 : Fichier XML pour le protocole de l’imageur de cellule vivante pour l’imagerie en fond clair et en fluorescence. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 10 : Exemple de carte de plaques. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 11 : Exemple de fichier d’entrée pour le script NDR R. Abréviation : NDR = réponse médicamenteuse normalisée. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 12 : Script NDR normalisé de la réponse médicamenteuse. Abréviation : NDR = réponse médicamenteuse normalisée. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 13 : Exemple de fichier de sortie des valeurs GR du script NDR R. Abréviations : GR = taux de croissance; NDR = réponse médicamenteuse normalisée. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 14 : Exemple de fichier de sortie de script NDR R avec les valeurs GR transposées. Abréviations : GR = taux de croissance; NDR = réponse médicamenteuse normalisée. Veuillez cliquer ici pour télécharger ce fichier.
Supplementary File 15 : Exemple de fichier de sortie des valeurs NDR du script NDR R. Abréviation : NDR = réponse médicamenteuse normalisée. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 16 : Exemple de fichier de sortie de script NDR avec des valeurs NDR transposées. Abréviation : NDR = réponse médicamenteuse normalisée. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le dépistage des médicaments PDTO à débit moyen à élevé repose souvent sur des lectures qui n’extraient qu’une fraction des informations que les organoïdes pourraient potentiellement fournir. Il est devenu de plus en plus clair que, pour que la technologie organoïde en évolution rapide réalise un plus grand potentiel scientifique et clinique, des tests 3D, des lectures et des méthodes d’analyse plus avancés sont absolument nécessaires. Ici, un pipeline de criblage avancé est décrit, ce qui non seulement augmente la reproductibilité, mais améliore également considérablement la translatabilité clinique en intégrant une lecture d’imagerie de cellules vivantes pilotée par l’IA. En plus du logiciel d’analyse développé à l’interne, l’utilisation de la mesure de réponse normalisée aux médicaments (NDR) est mise en œuvre, ce qui démontre clairement sa capacité à définir les différences spécifiques au patient dans la réponse au traitement6.
L’inclusion de cette mesure de normalisation sera sans aucun doute d’une valeur considérable, si l’on se souvient que de nombreuses études visent à délimiter les réponses au traitement en fonction de différences mineures d’aire sous la courbe (ASC) ou de concentration inhibitrice demi-maximale (IC50) (car la plupart des courbes dose-réponse se chevauchent/sont situées à proximité les unes des autres)11,12 . Des mesures du taux de croissance ont déjà été mises en œuvre dans des protocoles de criblage de médicaments organoïdes utilisant le test basé sur l’ATP, mais reposent sur la normalisation des puits de référence lysés au point temporel 04. En revanche, cette méthode permet la normalisation du taux de croissance intrapuits, ce qui tient compte non seulement des différences interpatients dans le taux de croissance PDTO, mais aussi des différences entre puits résultant des variations de la densité de semis et des effets dépendants de l’emplacement des plaques pour augmenter la reproductibilité. De plus, nous avons adapté la NDR pour augmenter davantage la séparation de la réponse PDTO interpatiente en incluant un contrôle positif pour la normalisation 6,8.
En outre, l’analyse, qui est compatible avec les formats à haut débit et d’automatisation, peut détecter avec précision les réponses organoïdes individuelles, permettant de quantifier la résistance sous-clonale, la principale force motrice de la rechute et de la progression tumorales13. Par exemple, bien que PDAC052 et PDAC060 aient montré une bonne réponse au traitement in vitro (basée sur le NDR), l’analyse mono-organoïde supplémentaire a permis de détecter une petite population (plus grande population avec PDAC060) de sous-clones qui ne répondent pas au traitement. Fait intéressant, cela correspondait fortement à l’observation clinique, étant donné que PDAC052 et PDAC060 avaient une réponse durable (aucune activité tumorale détectée) mais ont finalement été diagnostiqués avec une progression tumorale locale (en raison de la présence de clones résistants). Par rapport aux lectures 3D conventionnelles (test basé sur l’ATP et taille/nombre), ce pipeline de dépistage avancé devrait augmenter les performances prédictives en extrayant des informations plus pertinentes sur le plan clinique de ces « patients en laboratoire ». Cette hypothèse est actuellement testée en criblant des échantillons cliniques PDTO dans le laboratoire des auteurs avec cette méthode pour corréler ex vivo avec la réponse in vivo et les résultats cliniques.
Pour mieux comprendre les mécanismes d’une réponse aux médicaments, les réactifs d’imagerie de cellules vivantes fluorescentes conventionnels, en plus des colorants de cytotoxicité, sont compatibles avec cette méthode pour étudier les mécanismes de la mort cellulaire. Nous avons précédemment montré la compatibilité de cette méthode avec le réactif vert Sartorius Caspase 3/7 pour étudier l’induction de l’apoptose dépendante de la caspase après un traitement au cisplatine8. La compatibilité avec d’autres colorants pour étudier le stress oxydatif (réactifs CellROX) ou l’hypoxie (réactifs Image-iT Hypoxia) reste à tester. Cependant, ces réactifs ont déjà été utilisés avec succès dans des modèles in vitro 3D14,15.
Le logiciel d’analyse d’images est également compatible avec d’autres formats de plaques ou méthodes de culture (par exemple, plaques à microcavités, dômes ECM) si des images claires et nettes des organoïdes peuvent être capturées. Ceci est souvent difficile pour les organoïdes cultivés dans des dômes car ils poussent dans différents plans z, ce qui nécessite une fonctionnalité d’empilement z du microscope qui n’est pas toujours disponible. Par conséquent, nous conseillons l’utilisation de microplaques ULA 384 puits à fond plat pour garantir des images de qualité suffisante.
De plus, l’analyse est compatible avec d’autres systèmes d’imagerie de cellules vivantes, comme indiqué précédemment pour les images à contraste de phase capturées avec un système IncuCyte ZOOM8. Une limitation du système d’imagerie de cellules vivantes Spark Cyto qui a été utilisé dans ce manuscrit est la capacité d’une plaque pour les mesures cinétiques. Cependant, l’extension Spark Motion augmente sa capacité jusqu’à 40 microplaques qui peuvent être criblées en vrac. La compatibilité du logiciel développé en interne sera étendue à ces systèmes et à d’autres afin d’offrir une solution indépendante de la plate-forme, dans le but de normaliser et d’automatiser les pipelines d’analyse d’images et de données. L’application Web comprendra également des outils graphiques interactifs et des calculs automatisés de mesures des médicaments, comme le montre le présent document, afin de réduire le temps d’analyse manuelle.
L’algorithme de segmentation PDTO sans marquage a été formé et testé sur divers modèles sphéroïdes et PDTO cultivés en interne avec des différences morphologiques distinctes (solide, semi-solide, kystique), et peut par conséquent les détecter avec une grande précision8. Une limite du modèle est que l’inclusion de PDTO kystiques a augmenté la détection indésirable des bulles présentes dans le puits suivant l’ensemencement. Cependant, l’incubation nocturne a été suffisante pour éliminer la plupart de ces bulles, permettant un balayage qualitatif du point de temps 0. La précision de la segmentation de l’image organoïde et de la méthode doit être validée par d’autres utilisateurs, et en fonction de leurs commentaires, le logiciel peut être formé davantage pour obtenir un algorithme d’analyse d’image robuste et automatisé. En outre, nous visons à obtenir plus de données cliniques pour corréler la réponse ex vivo au médicament quantifiée par cette méthode à la réponse clinique chez le patient afin d’identifier les meilleurs paramètres pour prédire la réponse thérapeutique et de développer davantage cette méthode pour la médecine fonctionnelle de précision du cancer16.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Une partie de cette recherche a été financée par des dons de différents donateurs, dont Dedert Schilde vzw et M. Willy Floren. Ce travail est partiellement financé par la Fondation flamande pour la recherche, 12S9221N (A.L.), G044420N (S.V., A.L., E.G), 1S27021N (M.L), et par le Fonds de recherche industrielle de l’Université d’Anvers, PS ID 45151 (S.V., A.L., C.D.). La figure 1 a été créée avec BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 6-well plate | Greiner | 657160 | |
| 8-Channel p300 (GEN 2) pipette | Opentrons | ||
| 300 µL Tips | Opentrons | ||
| 384-well flat-bottom ULA microplate | Corning | 4588 | minimum volume 50 µL |
| 384-well flat-bottom ULA Phenoplate | Perkin Elmer | 6057802 | minimum volume 75 µL |
| A8301 | Tocris Bioscience | 2939 | |
| ADF+++ | Advanced DMEM/F12, 1% GlutaMAX, 1% HEPES, 1% penicillin/streptomycin | ||
| Advanced DMEM/F-12 | ThermoFisher Scientific | 12634 | |
| B27 | ThermoFisher Scientific | 17504044 | |
| Breathe easy sealing membrane | Sigma-Aldrich | Z380059 | |
| Caspase 3/7 Green | Sartorius | 4440 | |
| Cell Counting Slides for TC10/TC20 | Bio-Rad Laboratories | 1450017 | |
| CellTiter-Glo 3D | Promega | G9681 | ATP-assay |
| Cooler for 25 mL reservoir | VWR (Diversified Biotech) | 490006-908 | |
| Cooling element 12 x 8 x 3 cm | Bol.com | 9200000107744702 | For custom microplate holder OT-2 |
| Cultrex Organoid Harvesting Solution | R&D systems | 3700-100-01 | |
| Cultrex PathClear Reduced Growth Factor BME, Type 2 | R&D systems | 3533-010-02 | extracellular matrix (ECM) |
| Cytotox Green | Sartorius | 4633 | |
| Cytotox Red | Sartorius | 4632 | |
| D300e | Tecan | Digital drug dispenser | |
| D300e Control v3.3.5 | Tecan | Control software D300e | |
| FGF10 | Peprotech | 100-26 | |
| Full Medium | ADF+++ supplemented with 0.5 nM WNT surrogate-Fc-Fusion protein, 4% Noggin-Fc Fusion Protein conditioned medium, 4% Rpso3-Fc Fusion Protein conditioned medium, 1x B27, 1 mM N-acetyl cysteine (NAC), 5 mM nicotinamide, 500 nM A83-01, 100 ng/mL FGF10, and 10 nM Gastrin | ||
| Gastrin | Sigma-Aldrich | G9145 | |
| Gemcitabine | Selleck Chemicals | S1714 | |
| GlutaMAX | ThermoFisher Scientific | 35050 | |
| HEPES | ThermoFisher Scientific | 15630056 | |
| Hoechst 33342 Solution (20 mM) | ThermoFisher Scientific | 62259 | |
| Human pancreatic ductal adenocarcinoma (PDAC) patient-derived organoids | Biobank@uza (Antwerp, Belgium; ID: BE71030031000; Belgian Virtual Tumorbank funded by the National Cancer Plan) | ||
| N-acetyl-cysteine | Sigma-Aldrich | A9165-25G | |
| Nicotinamide | Sigma-Aldrich | N0636-100G | |
| Noggin-Fc Fusion Protein conditioned medium | Immunoprecise | N002 | |
| Opentrons App v6.0.1 | Opentrons | OT-2 control software | |
| Opentrons Protocol Designer Tool | Opentrons | https://designer.opentrons.com/ | |
| Orbits data compression tool | www.orbits-oncology.com or contact corresponding author | ||
| Orbits image analysis webapp | University of Antwerp | www.orbits-oncology.com or contact corresponding author | |
| OT-2 | Opentrons | Pipetting robot | |
| Paclitaxel | Selleck Chemicals | S1150 | |
| Pasteur Pipette 230 mm | Novolab | A33696 | |
| Peniciline-Streptomycin | ThermoFisher Scientific | 15140 | |
| Prism 9 | GraphPad | ||
| Rspo3-Fc Fusion Protein conditioned medium | Immunoprecise | N003 | |
| Spark Cyto 600 | Tecan | Live-cell imaging and multi-mode platereader | |
| SparkControl v3.1 | Tecan | Spark Cyto control software | |
| Staurosporine | Tocris Bioscience | 1285 | |
| Sterile 25 mL reservoir | VWR (Diversified Biotech) | 10141-922 | |
| T8 plus cassette | Tecan | ||
| TC20 | Bio-Rad Laboratories | automated cell counter | |
| TrypLE | ThermoFisher Scientific | 12604-021 | dissociation enzyme |
| Tween-20 | Acros Organics | 233360010 | |
| WNT Surrogate-Fc-Fusion protein | Immunoprecise | N001 | |
| Y-27632 | Selleck Chemicals | S1049 |
Références
- Drost, J., Clevers, H. Organoids in cancer research. Nature Reviews Cancer. 18 (7), 407-418 (2018).
- Veninga, V., Voest, E. E. Tumor organoids: Opportunities and challenges to guide precision medicine. Cancer Cell. 39 (9), 1190-1201 (2021).
- Le Compte, M., et al. Patient-derived organoids as individual patient models for chemoradiation response prediction in gastrointestinal malignancies. Critical Reviews in Oncology/Hematology. 157, 103190(2021).
- Driehuis, E., Kretzschmar, K., Clevers, H. Establishment of patient-derived cancer organoids for drug-screening applications. Nature Protocols. 15 (10), 3380-3409 (2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nature Methods. 13 (6), 521-527 (2016).
- Gupta, A., Gautam, P., Wennerberg, K., Aittokallio, T. A normalized drug response metric improves accuracy and consistency of anticancer drug sensitivity quantification in cell-based screening. Communications Biology. 3 (1), 42(2020).
- Rios, A. C., Clevers, H. Imaging organoids: a bright future ahead. Nature Methods. 15 (1), 24-26 (2018).
- Deben, C., Cardenas, E., et al. OrBITS: label-free and time-lapse monitoring of patient derived organoids for advanced drug screening. Cellular Oncology. , (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), (2017).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Engel, R. M., et al. Patient-derived colorectal cancer organoids upregulate revival stem cell marker genes following chemotherapeutic treatment. Journal of Clinical Medicine. 9 (1), 128(2020).
- Hennig, A., et al. Detecting drug resistance in pancreatic cancer organoids guides optimized chemotherapy treatment. The Journal of Pathology. 257 (5), 607-619 (2022).
- D'Alterio, C., Scala, S., Sozzi, G., Roz, L., Bertolini, G. Paradoxical effects of chemotherapy on tumor relapse and metastasis promotion. Seminars in Cancer Biology. 60, 351-361 (2020).
- Njoroge, R. N., et al. Organoids model distinct Vitamin E effects at different stages of prostate cancer evolution. Scientific Reports. 7 (1), 16285(2017).
- Godet, I., et al. Fate-mapping post-hypoxic tumor cells reveals a ROS-resistant phenotype that promotes metastasis. Nature Communications. 10 (1), 4862(2019).
- Letai, A. Functional precision cancer medicine-moving beyond pure genomics. Nature Medicine. 23 (9), 1028-1035 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon