Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Culture en vrac non fractionnée de muscles squelettiques de souris pour récapituler la niche et la quiescence des cellules souches
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Le muscle squelettique comprend plusieurs types de cellules, y compris les cellules souches résidentes, chacune ayant une contribution particulière à l’homéostasie et à la régénération musculaires. Ici, la culture 2D de cellules souches musculaires et la niche des cellules musculaires dans un environnement ex vivo qui préserve de nombreuses caractéristiques physiologiques, in vivo et environnementales sont décrites.
Résumé
Le muscle squelettique est le plus grand tissu du corps et remplit de multiples fonctions, de la locomotion au contrôle de la température corporelle. Sa fonctionnalité et sa récupération après des blessures dépendent d’une multitude de types de cellules et de signaux moléculaires entre les cellules musculaires centrales (myofibres, cellules souches musculaires) et leur niche. La plupart des contextes expérimentaux ne préservent pas ce microenvironnement physiologique complexe, et ne permettent pas non plus l’étude ex vivo des cellules souches musculaires en quiescence, un état cellulaire crucial pour elles. Ici, un protocole est décrit pour la culture ex vivo de cellules souches musculaires avec des composants cellulaires de leur niche. Grâce à la dégradation mécanique et enzymatique des muscles, on obtient un mélange de types cellulaires, qui est mis en culture 2D. L’immunomarquage montre qu’en l’espace d’une semaine, plusieurs cellules de niche sont présentes en culture aux côtés de myofibres et, surtout, de cellules Pax7-positives qui présentent les caractéristiques des cellules souches musculaires au repos. Ces propriétés uniques font de ce protocole un outil puissant pour l’amplification cellulaire et la génération de cellules souches de type quiescent qui peuvent être utilisées pour répondre à des questions fondamentales et translationnelles.
Introduction
Le mouvement, la respiration, le métabolisme, la posture corporelle et le maintien de la température corporelle dépendent tous du muscle squelettique, et des dysfonctionnements du muscle squelettique peuvent ainsi provoquer des pathologies débilitantes (c’est-à-dire des myopathies, des dystrophies musculaires, etc.) 1. Compte tenu de ses fonctions essentielles et de son abondance, le muscle squelettique a attiré l’attention des laboratoires de recherche du monde entier qui s’efforcent de comprendre les aspects clés qui soutiennent une fonction musculaire normale et peuvent servir de cibles thérapeutiques. De plus, le muscle squelettique est un modèle largement utilisé pour étudier la régénération et la fonction des cellules souches, car un muscle sain peut s’autoréparer complètement après une blessure et une dégénérescence complètes, principalement en raison de ses cellules souches résidentes2 ; Celles-ci sont également appelées cellules satellites et sont localisées sous la lame basale à la périphérie des fibres musculaires3.
Les cellules centrales du muscle squelettique adulte sont les myofibres (longues cellules multinucléées syncytiales) et les cellules satellites (cellules souches à potentiel myogénique qui sont au repos jusqu’à ce qu’une blessure les active). Ces dernières cellules sont les cellules centrales de la régénération musculaire, et ce processus ne peut pas se produire en leur absence 4,5,6,7. Dans leur microenvironnement immédiat, il existe de multiples types de cellules et de facteurs moléculaires qui leur signalent. Ce créneau s’établit progressivement tout au long du développement et jusqu’à l’âge adulte8. Le muscle adulte contient plusieurs types de cellules (cellules endothéliales, péricytes, macrophages, progéniteurs fibro-adipogènes-FAP, lymphocytes T régulateurs, etc.) 9,10 et composants de la matrice extracellulaire (laminines, collagènes, fibronectine, fibrillines, périostine, etc.) 11 qui interagissent entre elles et avec les cellules satellites dans le contexte de la santé, de la maladie et de la régénération.
La préservation de ce créneau complexe dans des contextes expérimentaux est fondamentale, mais difficile. Il est tout aussi difficile de maintenir ou de revenir à la quiescence, un état cellulaire critique pour les cellules satellites9. Plusieurs méthodes ont été introduites pour relever partiellement ces défis, chacune ayant ses avantages et ses inconvénients (détaillés dans la section de discussion). Nous présentons ici une méthode qui permet de surmonter partiellement ces deux barrières. Les muscles sont d’abord prélevés, puis décomposés mécaniquement et enzymatiquement avant que le mélange de cellules hétérogènes ne soit mis en culture. Au cours de la culture, de nombreux types de cellules de la niche sont détectés et des cellules satellites qui sont revenues à la quiescence sont observées. Comme dernière étape du protocole, les étapes d’immunofluorescence qui permettent la détection de chaque type de cellule grâce à l’utilisation de marqueurs universellement acceptés, sont présentées.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les expérimentations ont été conformes à la réglementation animale française et européenne de l’Institut Mondor de Recherche Biomédicale (INSERM U955), notamment la directive 2010/63/UE. Les animaux ont été gardés dans un environnement contrôlé et enrichi dans les animaleries portant les numéros de certification A94 028 379 et D94-028-028 ; Ils n’ont été manipulés que par des chercheurs et des soigneurs d’animaux autorisés, et ils ont été inspectés visuellement par le personnel de l’élevage des animaux pour détecter tout signe d’inconfort au cours de leur vie. Ils ont été euthanasiés par luxation cervicale avant la dissection. Aucune intervention n’a été effectuée au cours de la vie des animaux ; Il n’était donc pas nécessaire d’obtenir l’approbation d’un comité d’éthique et du ministère de l’Enseignement supérieur, de la Recherche et de l’Innovation pour la procédure. En effet, aucune attestation éthique n’est requise pour l’euthanasie et la dissection post-mortem selon la directive 2010/63/UE. Les résultats présentés dans ce manuscrit proviennent de la lignée C57BL/6NRj de type sauvage (voir Tableau des matériaux) et de la lignée transgénique Tg :Pax7-nGFP 12 (sélectionnée par notre équipe). Le protocole a été appliqué à des souris mâles et femelles âgées de 8 à 12 semaines.
1. Préparation des réactifs et de l’équipement avant la digestion
- Vaporisez les outils de dissection (ciseaux droits et courbes, pinces, voir le tableau des matériaux) avec de l’éthanol à 70 % et séchez-les avec du papier. Enduisez une assiette en liège de papier d’aluminium et gardez des boîtes de Pétri de 10 cm (une par animal) à proximité. Ayez du papier et de l’éthanol à 70 % à portée de main.
REMARQUE : À la fin de la dissection, rincez les outils de dissection à l’eau, puis vaporisez-les avec de l’éthanol à 70% et séchez-les avec du papier. - Régler un bain-marie rotatif à 37 °C et préparer le mélange de digestion (20 mL/animal) en combinant le DMEM avec 1 % de pénicilline-streptomycine, 0,5 U/mL de collagénase, 3 U/mL de dispase (voir le tableau des matériaux) et 0,2 % de BSA dans un tube de 50 mL.
- Faire passer le mélange de digestion à travers un filtre de 0,22 μm dans une hotte de culture cellulaire.
REMARQUE : Il est recommandé de préparer le mélange de digestion frais à chaque fois.
2. Préparation des réactifs et de l’équipement après la digestion
- Après la digestion, le mélange peut être congelé ou cultivé. Pour la congélation, préparez 10 % de DMSO : 90 % de sérum de veau fœtal (FBS), ainsi qu’un ensemble de cryotubes (1 mL de suspension cellulaire par 2 mL de cryotube). Pour la culture, préparez un milieu de culture (DMEM complété par 1 % de pénicilline-streptomycine, 4 ng/mL de bFGF et 20 % de FBS) et un ensemble de plaques à 8 puits. Les plaques doivent être revêtues avant le placage des cellules (les détails sont fournis à l’étape 7.1).
- Pour la coloration, préparer du paraformaldéhyde (PFA) à 4 % dans une solution saline tamponnée au phosphate (PBS) (0,15 mL/puits de la plaque à 8 puits) et une solution bloquante (Albumine sérique bovine [BSA] à 5 % sans IgG dans le PBS ; 0,15 mL/puits de la plaque à 8 puits).
ATTENTION : Ne pas respirer dans la poudre de PFA ; Préparez-le et manipulez-le sous une hotte chimique.
3. Dissection
- Vaporisez l’animal euthanasié avec 70 % d’éthanol. Faites une incision horizontale (du côté gauche du corps vers le côté droit) avec de gros ciseaux au niveau de l’abdomen et coupez autour de la taille. Retirez la peau des membres postérieurs pour révéler les muscles (Figure 1A).
- Placez l’animal sur la plaque de liège recouverte de papier d’aluminium et épinglez les membres antérieurs et postérieurs opposés. Retirez rapidement tous les muscles des membres postérieurs (avant et arrière) dans une boîte de Pétri de 10 cm placée sur de la glace (Figure 1B,C). Faites particulièrement attention à retirer le tissu adipeux des zones autour des quadriceps et des muscles postérieurs. Les fascias, les nerfs et les tendons peuvent également être retirés à ce stade si cela ne compromet pas le temps total consacré à la dissection.
REMARQUE : Un temps de dissection optimal pour les deux membres postérieurs devrait être d’environ 15 à 20 minutes. Il est conseillé que le temps de dissection ne dépasse pas 30 min. - Ajoutez de temps en temps des gouttes de DMEM aux muscles pour les garder humides, mais pas trop, car cela rendra le hachage difficile. Répétez l’opération pour l’autre membre postérieur. Une fois que tous les muscles d’un animal sont dans la boîte de Pétri (Figure 1D), hachez-les finement avec des ciseaux pendant 7 à 10 min pour obtenir un homogénat lisse (Figure 1E).
REMARQUE : Dans ce protocole, le DMEM complété par de la L-glutamine, du pyruvate et 4,5 g / L de D-glucose est utilisé.
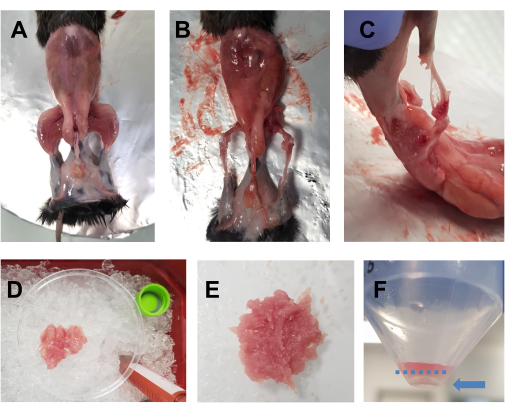
Figure 1 : Préparation musculaire en pré-culture. (A) La peau est enlevée pour révéler les muscles des membres postérieurs, comme décrit à l’étape 3.1. (B,C) Tous les muscles des membres postérieurs sont prélevés (B) autour et (C) entre les os, comme décrit à l’étape 3.2. (D) Les muscles prélevés sont placés dans une boîte de Pétri de 10 cm sur de la glace avec des gouttes de DMEM pour les maintenir humides, comme décrit à l’étape 3.3. (E) Les muscles sont finement hachés avec des ciseaux jusqu’à l’obtention d’une pâte lisse avec la consistance représentée sur cette image. (F) Une image de la pastille après la centrifugation finale ; La flèche bleue met en évidence la pastille, qui se trouve contre le tube, sous la ligne bleue en pointillés. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. La digestion
REMARQUE : À la fin de la digestion, une centrifugeuse à 4 °C, un seau de glace, trois crépines cellulaires (100 um, 70 um, 40 um) et trois tubes de 50 ml (par animal) sont nécessaires pour la section 5.
- Préparez et filtrez le mélange de digestion comme décrit à l’étape 1.2. Gardez le mélange sur de la glace.
- Une fois que tous les muscles sont hachés, placez l’homogénat dans un tube de 50 mL avec 20 mL de mélange de digestion. Enveloppez les bords du couvercle d’un film souple pour éviter les fuites et placez le tube dans un bain-marie à 37 °C à vitesse faible à moyenne (50 tr/min).
- Après 1 h à 37 °C, ouvrez le couvercle et mélangez en pipetant doucement sept fois de haut en bas avec une pipette de 10 mL pour obtenir un mélange homogène. Appliquez un nouveau film autour du couvercle et remettez-le dans le bain-marie. Après 1 h, retirez le tube et éteignez le bain.
REMARQUE : Pour la culture, utilisez ce temps d’incubation pour enrober les plaques comme décrit à l’étape 7.1 avant de passer à la section 5.
5. Filtration
- Remplir le tube digestif avec du DMEM froid (complété par 1 % de pénicilline-streptomycine) jusqu’à 50 mL. Mélangez en retournant le tube trois fois. Conservez le DMEM dans un seau à glace pour les étapes suivantes.
- Placez une crépine de 100 μm sur un nouveau tube de 50 ml. Passez le mélange digéré à travers la passoire cellulaire dans le nouveau tube. Centrifuger à 600 x g pendant 5 min à 4 °C. Versez le surnageant dans un récipient à déchets liquides.
- Remettre la granule en suspension dans 1 mL de DMEM froid (complété par 1 % de pénicilline-streptomycine). Remplissez le tube jusqu’à 50 mL avec le même DMEM. REMARQUE : Si la centrifugation est sautée, la pastille suivante sera plus difficile à identifier et à entretenir.
- Placez une crépine de 70 μm sur un nouveau tube de 50 ml. Passez le mélange centrifugé/remis en suspension à travers le tamis cellulaire dans le nouveau tube. Centrifuger à 80 x g pendant 5 min à 4 °C.
REMARQUE : Cette étape n’est pas obligatoire mais est recommandée pour éliminer les débris cellulaires. - Placez une crépine de 40 μm sur un nouveau tube de 50 ml. Passez le surnageant à travers le tamis cellulaire dans le nouveau tube. Centrifuger à 600 x g pendant 5 min à 4 °C, verser le surnageant dans un récipient à déchets liquides et remettre en suspension le granulé dans du FBS sous la hotte de culture. La pastille est très petite à cette étape (figure 1F).
REMARQUE : Le filtrage à travers la crépine de 40 μm élimine les débris, ce qui donnerait un signal non spécifique lors de la coloration ultérieure des cultures.
6. (Facultatif) Congélation
REMARQUE : La section 6 est facultative. Le protocole peut être mis en pause après le filtrage, mais cela peut réduire la survie des cellules et le succès de la culture.
- Ajouter du DMSO pour obtenir un rapport DMSO :90 % FBS, puis transférer dans des cryotubes (1 mL de pastille remise en suspension par 2 mL de cryotube).
- Placez le cryotube à −80 °C dans une boîte en polystyrène pendant la nuit. Passer à −150 °C le lendemain pour un stockage à long terme.
REMARQUE : Un stockage à court terme à −80 °C est également possible. - Au début de la culture, décongeler rapidement le cryotube au bain-marie à 37 °C jusqu’à ce que la suspension cellulaire soit décongelée. Mélanger avec 4 mL de DMEM sous la hotte de culture. Essorer à 600 x g pendant 5 min à 4 °C. Pipeter le surnageant et continuer comme décrit à l’étape 7.2.
7. Culture
REMARQUE : On peut s’attendre à ce que les suspensions de cellules congelées ou fraîches remplissent 24 à 32 puits de trois à quatre plaques de 8 puits.
- Enduire les plaques à 8 puits avec la solution d’enrobage, qui doit être décongelée à 4 °C ou sur de la glace (la solution d’enrobage mère est normalement conservée à −20 °C). Ajouter 0,4 mL de solution d’enrobage dans un puits et pipeter d’un puits à l’autre. Après avoir transféré la solution d’enrobage dans tous les puits, elle peut être collectée et recongelée pour de futures cultures. Maintenez les plaques revêtues à 37 °C pendant 30 min avant de plaquer les cellules.
- Ajouter du DMEM (complété par 1 % de pénicilline-streptomycine) complété par 4 ng/mL de bFGF (voir tableau des matériaux) à la suspension de cellules FBS pour obtenir un rapport FBS :80 % de DMEM.
REMARQUE : Même si l’ajout de bFGF peut être bénéfique dans les cultures primaires de myoblastes et dans la production de cellules de type satellite dans les cultures en vrac, son ajout est facultatif, car son omission dans les cultures en vrac de ~7 jours ne compromet pas gravement les rendements cellulaires. - Plaquer 0,4 mL de suspension par puits (à partir de l’étape 7.2) dans les plaques revêtues à 8 puits.
REMARQUE : Calculer 30 cm2 de culture par animal pour les préparations congelées et fraîches. - Incuber les cultures à 37 °C avec 5 % de CO2 pendant 10 jours maximum, en changeant le milieu tous les jours après que la culture commence à prendre une couleur jaunâtre (généralement 5 à 7 jours).
REMARQUE : Pour quantifier les cellules dans la phase S du cycle cellulaire13, ajouter 10 μM EdU 2 h avant la fixation. Pour capturer la première phase S, ajouter 10 μM d’EdU du placage et fixer à 40 h de culture.
8. Fixation
REMARQUE : Les sections 8 à 10 doivent être conduites à température ambiante, sauf indication contraire.
- Pipeter le milieu de culture et fixer les cellules avec 4 % de PFA (0,15 ml/puits).
ATTENTION : Ajouter du PFA sous une hotte chimique.
REMARQUE : Si tous les puits sont fixés en même temps, incuber avec du PFA à température ambiante pendant 10 minutes. Si les puits sont fixés à des moments différents, ajoutez de l’APF dans les puits à fixer et maintenez la plaque dans l’incubateur à 37 °C pendant 5 min. - Pipeter le PFA et ajouter le PBS pendant 10 s (0,15 ml/puits). Pipeter le PBS et ajouter du PBS frais pendant 5 min (0,15 ml/puits).
REMARQUE : Si tous les puits sont fixés en même temps, incuber avec du PBS à température ambiante. Si les puits sont fixés à des moments différents, ajoutez du PBS aux puits fixes et maintenez la plaque dans l’incubateur à 37 °C pendant 5 minutes. Ensuite, ajoutez 0,4 mL de PBS et conservez l’assiette dans l’incubateur jusqu’à 1 semaine.
9. Perméabilisation et blocage
- Lorsque vous êtes prêt à colorer, pipeter le PBS et perméabiliser avec 0,5 % de TritonX 100 dans du PBS (0,15 ml/puits) pendant 8 min. Pipeter le TritonX 100, rincer avec du PBS pendant 10 s (0,15 mL/puits), pipeter le PBS et laver avec du PBS pendant 5 min (0,15 mL/puits).
- Bloquer avec 5 % de BSA sans IgG dans du PBS pendant 30 à 60 min (0,15 mL/puits).
10. Coloration
- Pipeter le BSA et ajouter le mélange d’anticorps primaires dilué dans du PBS (0,15 mL/puits) (voir le tableau des matériaux ; dilutions : anti-CD31 1 :100, anti-FOSB 1 :200, anti-GFP 1 :1 000, anti-KI67 1 :1 000, anti-MyHC 1 :400, anti-MYOD 1 :200, anti-MYOG 1 :150, anti-PAX7 1 :100, anti-PDGFRa 1 :50) pour une incubation nocturne à 4 °C.
REMARQUE : Après l’incubation de l’anticorps, recueillir le mélange d’anticorps, ajouter de l’azoture de sodium et conserver à 4 °C ou −20 °C (selon les instructions du fabricant de l’anticorps) pour une réutilisation ultérieure. - Pipeter le mélange d’anticorps, rincer avec du PBS pendant 10 s (0,15 mL/puits), pipeter le PBS et laver avec du PBS pendant 5 min (0,15 mL/puits).
- Pipeter le PBS de lavage, ajouter le mélange d’anticorps secondaires (Alexa Fluor 488 anti-souris de chèvre, Alexa Fluor 555 anti-lapin de chèvre, Alexa Fluor 647 anti-rat de chèvre, Alexa Fluor 555 anti-souris de chèvre, Alexa Fluor 488 anti-poulet de chèvre, tous utilisés à des dilutions de 1 :500 à 1 000) et le marqueur de noyau (par exemple, DAPI) dilué dans du PBS (0,15 mL/puits) (voir le tableau des matériaux), et incuber pendant 1 h à température ambiante, à l’abri de la lumière.
- Pipeter le mélange d’anticorps secondaire, rincer avec du PBS pendant 10 s (0,15 mL/puits), pipeter le PBS, laver avec du PBS pendant 5 min (0,15 mL/puits), pipeter le PBS et le monter.
REMARQUE : Si des plaques à 8 puits avec des séparateurs amovibles sont utilisées, décollez les séparateurs avant le montage.
Access restricted. Please log in or start a trial to view this content.
Résultats
Ce protocole permet la culture de cellules musculaires tout en préservant les cellules satellites et la plupart des cellules de leur niche endogène. La figure 2 résume les principales étapes du protocole, tandis que les parties essentielles de la dissection et de la digestion sont présentées à la figure 1. La dissection de la musculature des membres postérieurs est recommandée (Figure 1A-C)...
Access restricted. Please log in or start a trial to view this content.
Discussion
La fonction des muscles squelettiques adultes est sous-tendue par un ensemble finement orchestré d’interactions cellulaires et de signaux moléculaires. Ici, une méthode est présentée qui permet d’étudier ces paramètres dans un cadre ex vivo qui ressemble beaucoup au microenvironnement physiologique.
Plusieurs groupes ont rapporté des méthodes in vitro pour la culture de cellules myogéniques. Ces méthodes visaient à isoler des cellules satellites afin d’étud...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Pour la figure 2, des modèles de Servier Medical Art (https://smart.servier.com/) ont été utilisés. Le laboratoire FR est soutenu par l’Association Française contre les Myopathies - AFM via TRANSLAMUSCLE (subventions 19507 et 22946), la Fondation pour la Recherche Médicale - FRM (EQU202003010217, ENV202004011730, ECO201806006793), l’Agence Nationale pour la Recherche - ANR (ANR-21-CE13-0006-02, ANR-19-CE13-0010, ANR-10-LABX-73), et la Ligue Contre le Cancer (IP/SC-17130). Les bailleurs de fonds susmentionnés n’ont joué aucun rôle dans la conception, la collecte, l’analyse, l’interprétation ou la rédaction de cette étude ou dans la rédaction de ce manuscrit.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| anti-CD31 | BD | 550274 | dilution 1:100 |
| anti-FOSB | Santa Cruz | sc-7203 | dilution 1:200 |
| anti-GFP | Abcam | ab13970 | dilution 1:1000 |
| anti-Ki67 | Abcam | ab16667 | dilution 1:1000 |
| anti-MyHC | DSHB | MF20-c | dilution 1:400 |
| anti-MYOD | Active Motif | 39991 | dilution 1:200 |
| anti-MYOG | Santa Cruz | sc-576 | dilution 1:150 |
| anti-Pax7 | Santa Cruz | sc-81648 | dilution 1:100 |
| anti-PDGFRα | Invitrogen | PA5-16571 | dilution 1:50 |
| b-FGF | Peprotech | 450-33 | concentration 4 ng/mL |
| Bovine serum albumin (BSA) – used for digestion | Sigma Aldrich | A7906-1006 | concentration 0.2% |
| BSA IgG-free, protease-free – used for staining | Jackson ImmunoResearch | 001-000-162 | concentration 5% |
| Cell strainer 40 um | Dominique Dutscher | 352340 | |
| Cell strainer 70 um | Dominique Dutscher | 352350 | |
| Cell strainer 100 um | Dominique Dutscher | 352360 | |
| Collagenase | Roche | 10103586001 | concentration 0.5 U/mL |
| Culture plate | Sarstedt | 94.6140.802 | |
| Dimethyl sulfoxide (DMSO) | Euromedex | UD8050-05-A | |
| Dispase | Roche | 4942078001 | concentration 3 U/mL |
| Dissection forceps size 5 | Fine Science Tools | 91150-20 | |
| Dissection forceps size 55 | Fine Science Tools | 11295-51 | |
| Dissection scissors (big, straight) | Fine Science Tools | 9146-11 | ideal for chopping |
| Dissection scissors (small, curved) | Fine Science Tools | 15017-10 | |
| Dissection scissors (small, straight) | Fine Science Tools | 14084-08 | |
| Dulbecco's Modified Eagle's Medium (DMEM) | ThermoFisher | 41966-029 | |
| EdU Click-iT kit | ThermoFisher | C10340 | |
| Fetal bovine serum – option 1 | Eurobio | CVF00-01 | |
| Fetal bovine serum – option 2 | Gibco | 10270-106 | |
| Matrigel | Corning Life Sciences | 354234 | coating solution |
| Parafilm | Dominique Dutscher | 090261 | flexible film |
| Paraformaldehyde – option 1 | PanReac AppliChem ITW Reagents | 211511.1209 | concentration 4% |
| Paraformaldeyde – option 2 | ThermoFisher | 28908 | concentration 4% |
| Penicillin streptomycin | Gibco | 15140-122 | |
| Shaking water bath | ThermoFisher | TSSWB27 | |
| TritonX100 | Sigma Aldrich | T8532-500 ML | concentration 0.5% |
| Wild-type mice | Janvier | C57BL/6NRj |
Références
- Frontera, W. R., Ochala, J. Skeletal muscle: A brief review of structure and function. Calcified Tissue International. 96 (3), 183-195 (2015).
- Forcina, L., Cosentino, M., Musarò, A. Mechanisms regulating muscle regeneration: Insights into the interrelated and time-dependent phases of tissue healing. Cells. 9 (5), 1297(2020).
- Mauro, A. Satellite cell of skeletal muscle fibers. Journal of Biophysical and Biochemical Cytology. 9 (2), 493-495 (1961).
- Lepper, C., Partridge, T. A., Fan, C. -M. An absolute requirement for Pax7-positive satellite cells in acute injury-induced skeletal muscle regeneration. Development. 138 (17), 3639-3646 (2011).
- McCarthy, J. J., et al. Effective fiber hypertrophy in satellite cell-depleted skeletal muscle. Development. 138 (17), 3657-3666 (2011).
- Murphy, M. M., Lawson, J. A., Mathew, S. J., Hutcheson, D. A., Kardon, G. Satellite cells, connective tissue fibroblasts and their interactions are crucial for muscle regeneration. Development. 138 (17), 3625-3637 (2011).
- Sambasivan, R., et al. Pax7-expressing satellite cells are indispensable for adult skeletal muscle regeneration. Development. 138 (17), 3647-3656 (2011).
- Hicks, M. R., Pyle, A. D. The emergence of the stem cell niche. Trends in Cell Biology. 33 (22), 112-123 (2022).
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nature Communications. 12 (1), 692(2021).
- Gama, J. F. G., et al. Role of regulatory T cells in skeletal muscle regeneration: A systematic review. Biomolecules. 12 (6), 817(2022).
- Loreti, M., Sacco, A. The jam session between muscle stem cells and the extracellular matrix in the tissue microenvironment. NPJ Regenerative Medicine. 7 (1), 16(2022).
- Sambasivan, R., et al. Distinct regulatory cascades govern extraocular and pharyngeal arch muscle progenitor cell fates. Developmental Cell. 16 (6), 810-821 (2009).
- Pereira, P. D., et al. Quantification of cell cycle kinetics by EdU (5-ethynyl-2'-deoxyuridine)-coupled-fluorescence-intensity analysis. Oncotarget. 8 (25), 40514-40532 (2017).
- Bismuth, K., Relaix, F. Genetic regulation of skeletal muscle development. Experimental Cell Research. 316 (18), 3081-3086 (2010).
- Yin, H., Price, F., Rudnicki, M. A. Satellite cells and the muscle stem cell niche. Physiological Reviews. 93 (1), 23-67 (2013).
- Lertkiatmongkol, P., Liao, D., Mei, H., Hu, Y., Newman, P. J. Endothelial functions of platelet/endothelial cell adhesion molecule-1 (CD31). Current Opinion in Hematology. 23 (3), 253-259 (2016).
- Scholzen, T., Gerdes, J. The Ki-67 protein: From the known and the unknown. Journal of Cellular Physiology. 182 (3), 311-322 (2000).
- Abou-Khalil, R., Le Grand, F., Chazaud, B. Human and murine skeletal muscle reserve cells. Stem Cell Niche. 1035, 165-177 (2013).
- Pasut, A., Oleynik, P., Rudnicki, M. A. Isolation of muscle stem cells by fluorescence activated cell sorting cytometry. Methods in Molecular Biology. 798, 53-64 (2011).
- Liu, L., Cheung, T. H., Charville, G. W., Rando, T. A. Isolation of skeletal muscle stem cells by fluorescence-activated cell sorting. Nature Protocols. 10 (10), 1612-1624 (2015).
- Montarras, D., et al. Direct isolation of satellite cells for skeletal muscle regeneration. Science. 309 (5743), 2064-2067 (2005).
- Qu, Y., Edwards, K., Barrow, J. Isolation, culture, and use of primary murine myoblasts in small-molecule screens. STAR Protocols. 4 (2), 102149(2023).
- Danoviz, M. E., Yablonka-Reuveni, Z. Skeletal muscle satellite cells: Background and methods for isolation and analysis in a primary culture system. Methods in Molecular Biology. 798, 21-52 (2011).
- Saclier, M., Theret, M., Mounier, R., Chazaud, B. Effects of macrophage conditioned-medium on murine and human muscle cells: analysis of proliferation, differentiation, and fusion. Methods in Molecular Biology. 1556, 317-327 (2017).
- Giordani, L., et al. High-dimensional single-cell cartography reveals novel skeletal muscle-resident cell populations. Molecular Cell. 74 (3), 609-621 (2019).
- Tabula Muris Consortium et al. Single-cell transcriptomics of 20 mouse organs creates a Tabula Muris. Nature. 562 (7727), 367-372 (2018).
- Brunetti, J., Koenig, S., Monnier, A., Frieden, M. Nanopattern surface improves cultured human myotube maturation. Skeletal Muscle. 11 (1), 12(2021).
- Denes, L. T., et al. Culturing C2C12 myotubes on micromolded gelatin hydrogels accelerates myotube maturation. Skeletal Muscle. 9 (1), 17(2019).
- LaFramboise, W. A., et al. Effect of muscle origin and phenotype on satellite cell muscle-specific gene expression. Journal of Molecular and Cellular Cardiology. 35 (10), 1307-1318 (2003).
- Azhar, M., Wardhani, B. W. K., Renesteen, E. The regenerative potential of Pax3/Pax7 on skeletal muscle injury. Journal of Generic Engineering and Biotechnology. 20 (1), 143(2022).
- Hardy, D., et al. Comparative study of injury models for studying muscle regeneration in mice. PLoS One. 11 (1), e0147198(2016).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon