Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Application de l’analyse multifactorielle multi-omique non supervisée pour découvrir les modèles de variation et les processus moléculaires liés aux maladies cardiovasculaires
Dans cet article
Résumé
Nous présentons un flux de travail flexible et extensible basé sur Jupyter-lab pour l’analyse non supervisée d’ensembles de données multi-omiques complexes qui combine différentes étapes de prétraitement, l’estimation du modèle d’analyse factorielle multi-omique et plusieurs analyses en aval.
Résumé
Les mécanismes de la maladie sont généralement complexes et régis par l’interaction de plusieurs processus moléculaires distincts. Les ensembles de données complexes et multidimensionnels sont une ressource précieuse pour générer davantage d’informations sur ces processus, mais l’analyse de ces ensembles de données peut être difficile en raison de la grande dimensionnalité résultant, par exemple, de différentes conditions de maladie, de points temporels et de données omiques capturant le processus à différentes résolutions.
Ici, nous présentons une approche permettant d’analyser et d’explorer un ensemble de données multiomiques aussi complexe de manière non supervisée en appliquant l’analyse factorielle multi-omique (MOFA) à un ensemble de données généré à partir d’échantillons de sang qui capturent la réponse immunitaire dans les syndromes coronariens aigus et chroniques. L’ensemble de données se compose de plusieurs tests à différentes résolutions, y compris des données sur les cytokines au niveau de l’échantillon, la protéomique plasmatique et le premier-seq des neutrophiles, et les données de séquençage de l’ARN sur cellule unique (scRNA-seq). À cela s’ajoute la nécessité de mesurer plusieurs points temporels différents par patient et plusieurs sous-groupes de patients.
Le flux de travail d’analyse décrit comment intégrer et analyser les données en plusieurs étapes : (1) Prétraitement et harmonisation des données, (2) Estimation du modèle MOFA, (3) Analyse en aval. L’étape 1 décrit comment traiter les caractéristiques des différents types de données, filtrer les caractéristiques de faible qualité et les normaliser afin d’harmoniser leurs distributions pour une analyse plus approfondie. L’étape 2 montre comment appliquer le modèle MOFA et explorer les principales sources de variance au sein de l’ensemble de données pour toutes les caractéristiques omiques. L’étape 3 présente plusieurs stratégies pour l’analyse en aval des motifs capturés, en les reliant aux conditions de la maladie et aux processus moléculaires potentiels régissant ces conditions.
Dans l’ensemble, nous présentons un flux de travail pour l’exploration non supervisée de données multi-omiques complexes afin de permettre l’identification des principaux axes de variation composés de différentes caractéristiques moléculaires qui peuvent également être appliquées à d’autres contextes et ensembles de données multi-omiques (y compris d’autres tests présentés dans le cas d’utilisation exemplaire).
Introduction
Les mécanismes de la maladie sont généralement complexes et régis par l’interaction de plusieurs processus moléculaires distincts. Déchiffrer les mécanismes moléculaires complexes qui conduisent à des maladies spécifiques ou régissent l’évolution d’une maladie est une tâche d’une grande pertinence médicale car elle pourrait révéler de nouvelles perspectives pour la compréhension et le traitement des maladies.
Les progrès technologiques récents permettent de mesurer ces processus à une résolution plus élevée (par exemple, au niveau de la cellule unique) et à diverses couches biologiques (par exemple, l’ADN, l’ARNm, l’accessibilité de la chromatine, la méthylation de l’ADN, la protéomique) en même temps. Cela conduit à la génération croissante de grands ensembles de données biologiques multidimensionnelles, qui peuvent être analysés conjointement pour générer plus d’informations sur les processus sous-jacents. Dans le même temps, la combinaison et l’analyse des différentes sources de données d’une manière biologiquement significative restent une tâche difficile1.
Les différentes limites technologiques, les bruits et les plages de variabilité entre les différentes chambres omiques posent un défi. Par exemple, les données de séquençage de l’ARN sur cellule unique (scRNA-seq) sont très rares et souvent influencées par des effets techniques ou de lots importants. De plus, l’espace des caractéristiques est souvent très grand, couvrant plusieurs milliers de gènes ou de protéines mesurés, tandis que la taille des échantillons est limitée. Cela est encore compliqué par des conceptions complexes, qui peuvent inclure plusieurs maladies, facteurs de confusion, points temporels et résolutions. Par exemple, dans le cas d’utilisation présenté, différents types de données étaient disponibles au niveau de la cellule unique ou de l’échantillon (en vrac). En outre, les données peuvent être incomplètes et toutes les mesures peuvent ne pas être disponibles pour tous les sujets analysés.
En raison de ces défis, les différentes omiques et les caractéristiques incluses sont encore souvent analysées uniquement séparément2, même si la réalisation d’une analyse intégrée peut non seulement fournir une image complète du processus, mais les bruits biologiques et techniques d’une omique peuvent également être compensés par d’autres omiques 3,4. Plusieurs méthodes différentes ont été proposées pour effectuer une analyse intégrée de données multi-omiques, notamment les méthodes bayésiennes, les méthodes basées sur le réseau 5,6, l’apprentissage profond multimodal7 et les méthodes de réduction de la dimensionnalité par factorisation matricielle 8,9. Pour ces derniers, les résultats d’une vaste étude comparative10 ont montré que la méthode MOFA9 (analyse multi-omique factorielle) est l’un des outils les mieux adaptés lorsque les données doivent être liées à des annotations cliniques.
En particulier dans les environnements complexes, les méthodes de factorisation matricielle non supervisée sont une approche utile pour réduire la complexité et extraire des signaux partagés et complémentaires à partir de différentes sources de données et fonctionnalités. En décomposant l’espace complexe en représentations latentes de rang inférieur, les principales sources de variance au sein des données peuvent être rapidement explorées et liées à des covariables connues. Dans le cas où le même modèle de variation est partagé entre plusieurs caractéristiques (par exemple, des gènes ou des protéines), cela peut être agrégé à quelques facteurs tout en réduisant le bruit. La régularisation peut être utilisée pour augmenter la rareté des coefficients du modèle, ce qui rend l’approche bien adaptée aux contextes où l’espace des caractéristiques est grand alors que le nombre d’échantillons est limitéà 9.
Ce protocole présente un flux d’analyse flexible qui utilise le modèle MOFA pour montrer comment explorer rapidement un ensemble de données multi-omiques complexe et distiller les principaux modèles de variation qui caractérisent cet ensemble de données. Le flux de travail se compose de trois étapes principales. Dans un premier temps, le prétraitement et l’harmonisation des données, différentes stratégies de prétraitement des données basées sur différents types de données d’entrée (scRNA-seq, protéomique, cytokine, données cliniques) sont présentées. Le protocole explique comment traiter les caractéristiques des différents ensembles de données d’entrée, filtrer les caractéristiques de faible qualité et les normaliser pour harmoniser leurs distributions. Nous montrons également comment ces décisions de prétraitement peuvent affecter les résultats en aval. Dans la deuxième étape, le modèle MOFA est appliqué aux données, et la décomposition de la variance résultante peut être utilisée pour évaluer l’intégration des différents ensembles de données. La troisième étape montre comment relier les facteurs capturés aux covariables et découvrir les programmes moléculaires qui définissent ces facteurs. Avec le flux de travail présenté, nous avons pu extraire plusieurs facteurs latents liés à des covariables cliniques dans un ensemble de données de patients souffrant de syndromes coronariens et identifier des programmes immunitaires multicellulaires sous-jacents potentiels d’un projet précédent11. Nous utiliserons cet ensemble de données ici, mais le protocole peut facilement être appliqué à d’autres contextes, y compris d’autres omiques.
L’ensemble de données se compose d’échantillons provenant de patients atteints de syndromes coronariens chroniques stables (SCC), de syndromes coronariens aigus (SCA) et d’un groupe témoin atteint de coronaires saines (non-SCC) (Figure 1). Le SCA est causé par une rupture de plaque dans un SCC préexistant, entraînant une perturbation aiguë du flux sanguin vers le myocarde et une lésion ischémique ultérieure du cœur. Cette lésion provoque une réponse inflammatoire du système immunitaire suivie d’une phase réparatrice, qui dure jusqu’à plusieurs jours après l’événement aigu12. Pour pouvoir caractériser cette réponse immunitaire chez les patients atteints de SCA, des échantillons de sang ont été prélevés à quatre moments différents : aiguë (TP1) ; après recanalisation (14 [± 8] h) (TP2) ; 60 [± 12] h plus tard (TP3) ; avant le congé (6,5 [±1,5] jours) (TP4) (figure 1A). Pour la SCC et les patients ayant des coronaires saines, un seul point temporel était disponible (TP0). Pour tous les patients et à tous les points temporels, différents dosages basés sur les échantillons de sang ont été mesurés : marqueurs cliniques de l’inflammation (créatine-kinase (CK), CK-MB, TROPONINE, PROTÉINE C-réactive (CRP)), scRNA-seq des cellules mononucléées du sang périphérique (PBMC), analyse des cytokines, protéomique plasmatique et données prime-seq13 des neutrophiles.
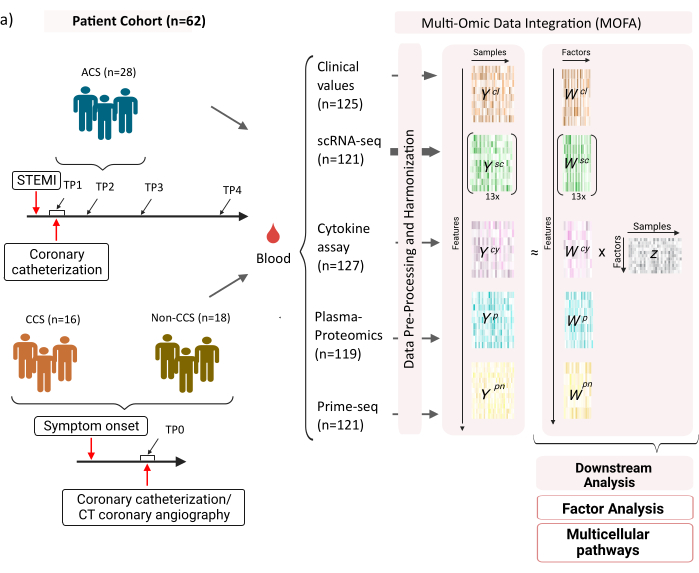
Figure 1 : Ensemble de données d’entrée multi-omiques sur l’infarctus du myocarde. Ensemble de données d’entrée : Les données analysées comprennent des échantillons de sang de patients (n = 62) atteints de syndrome coronarien aigu (SCA), de syndromes coronariens chroniques (SCC) et de patients atteints de coronaires saines (non-SCC). Pour les patients atteints de SCA, des échantillons de sang ont été inclus à quatre moments différents (TP1-4), pour les patients atteints de SCC et les patients non SCC à un seul point temporel (TP0). Chaque combinaison de patient et de point temporel est traitée comme un échantillon distinct dans l’analyse. Différents tests omiques ont été mesurés sur les échantillons : tests sanguins cliniques (n = 125), scRNA-seq (n = 121), plasma-protéomique (n = 119), test de cytokines (n = 127) et neutrophiles prime-seq (n = 121). Par la suite, le protocole décrit a été appliqué pour intégrer les données dans toutes les omiques et les explorer à l’aide du modèle MOFA et d’une analyse en aval plus poussée (analyse factorielle, enrichissement de la voie). Veuillez cliquer ici pour voir une version agrandie de cette figure.
En tant qu’entrée pour le flux de travail présenté ici, nous prenons des comptages bruts à partir des données scRNA-seq après traitement avec cellranger et le contrôle qualité (QC), comme indiqué, par exemple, dans le tutoriel de prétraitement scanpy14 . Pour l’annotation de type cellule, nous avons utilisé le pipeline automatisé Azimuth15 . Les nombres sont ensuite agrégés au niveau de l’échantillon pour chaque type de cellule en prenant la moyenne de toutes les cellules pour chaque échantillon et type de cellule (agrégation pseudo-en bloc). La protéomique plasmatique est incluse en tant qu’intensités normalisées et centrées sur la médiane, et pour les neutrophiles, nous prenons le nombre d’exons de l’identifiant moléculaire unique (UMI) de l’estimateur premier. Sur les cytokines et les valeurs cliniques, aucun prétraitement préalable n’a été appliqué. De plus amples détails sur la génération de données (expérimentales) sont décrits dans le manuscrit correspondant11. Comme les résultats présentés ici sont basés sur l’utilisation de l’annotation azimutale automatisée pour les types de cellules dans les données de séquençage de l’ARNc par rapport à la stratégie basée sur les marqueurs qui a été utilisée dans la publication référencée, les résultats présentés ici sont similaires mais pas exactement les mêmes que ceux présentés dans la publication. Dans le manuscrit, il a été démontré que la stratégie d’annotation de type cellulaire ne modifie pas les principaux modèles et interprétations biologiques de l’analyse, mais que de petits changements dans les valeurs exactes résultant du modèle peuvent varier. Dans l’ensemble, les données d’entrée étaient un ensemble de données multidimensionnelles complexe comprenant différents points temporels et niveaux de mesure (cellules uniques ou en vrac) de plus de 10 000 caractéristiques différentes (gènes, protéines, valeurs cliniques). Une stratégie stricte de prétraitement et d’harmonisation des données, suivie d’une analyse MOFA, s’est avérée être un outil utile et rapide pour explorer les données et extraire le programme immunitaire pertinent. Chaque combinaison de point temporel et de patient est traitée comme un échantillon indépendant dans l’analyse MOFA. Chaque type de données et type de cellule est considéré comme une vue distincte dans l’analyse MOFA.
Ce protocole fournit des instructions pour préparer les données d’entrée pour le flux de travail, exécuter les différentes étapes du flux de travail, personnaliser les configurations, interpréter les figures résultantes et ajuster de manière itérative les configurations en fonction des interprétations. Une vue d’ensemble des différentes étapes du protocole, des ensembles de données d’entrée requis à chaque étape, ainsi que des figures et des ensembles de données qui en résultent est donnée par la vue d’ensemble du flux de travail technique (Figure 2).
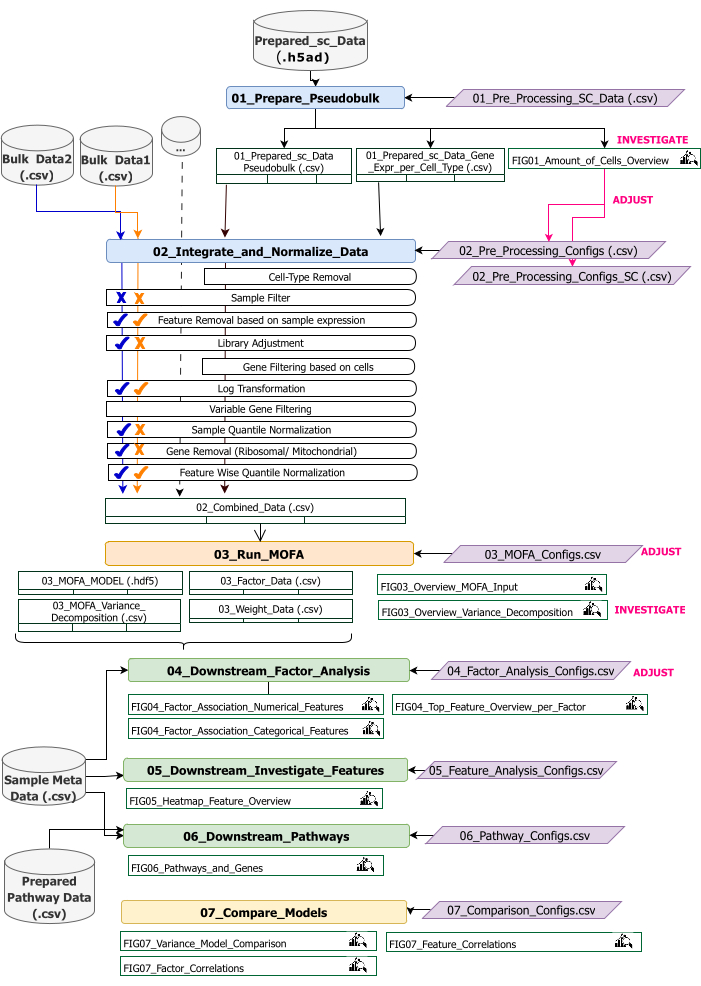
Figure 2 : Vue d’ensemble du flux de travail technique. Aperçu du flux de travail pour l’analyse de l’ensemble de données multi-omiques. Les différents éléments sont mis en valeur par des couleurs et des symboles différents. Les notebooks Jupyter appartenant à l’étape Prétraitement et harmonisation des données (1) sont colorés en bleu. Les carnets Jupyter appartenant à l’étape 'MOFA Model' (2) sont colorés en orange. Les notebooks Jupyter appartenant à l’étape 'Analyse aval' (3) sont colorés en vert. Un Jupyter Notebook à utiliser pour la comparaison des résultats est coloré en jaune. Les fichiers de configuration où les paramètres d’exécution du flux de travail peuvent être modifiés sont surlignés en violet. Les jeux de données d’entrée nécessaires à l’exécution du flux de travail sont indiqués par le symbole du jeu de données et mis en surbrillance en gris. Toutes les sorties de figures générées pendant l’exécution du flux de travail sont indiquées par le symbole de la loupe. Les jeux de données générés lors de l’exécution du workflow sont indiqués sous forme de tables. En général, le flux de travail est exécuté de manière séquentielle : (1) Le prétraitement et l’harmonisation des données se composent de deux étapes : la première génération d’une table pseudo-bulk basée sur les données d’entrée scRNA-seq (01_Prepare_Pseudobulk) et l’intégration et la normalisation ultérieures de ces données avec toutes les autres entrées au niveau de l’échantillon (en vrac) (02_Integrate_and_Normalize_Data). Dans le cadre de cette étape, via les fichiers de configuration, il est possible de configurer séparément pour chaque ensemble de données les étapes de prétraitement et de normalisation indiquées (par exemple, le filtre d’échantillonnage) à appliquer. (2) « Modèle MOFA » : exécute le modèle MOFA sur l’entrée générée de la première étape avec les configurations spécifiées dans le fichier de configuration (03_MOFA_configs.csv) (3) « Analyse en aval » : se compose de trois blocs-notes différents qui peuvent être exécutés indépendamment les uns des autres pour générer des informations sur les résultats MOFA générés et les associer à des exemples de métadonnées (covariables) fournies en entrée via le fichier « Exemple de méta-Data.csv ». (4) « Comparaison de modèles » : il s’agit d’une petite étape distincte qui peut être utilisée pour comparer différents modèles générés à l’étape 2. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Le flux de travail se compose de plusieurs blocs-notes Jupyter écrits en R et Python (la connaissance des langages R et Python n’est pas requise pour exécuter le flux de travail, mais peut être utile en cas d’apparition d’erreurs). Dans les différentes étapes du protocole, les paramètres sont modifiés via des fichiers de configuration (fichiers '.csv' contenant le postfix '_Configs' dans le nom). Dans le protocole, nous ne décrivons que les paramètres qui doivent être modifiés à partir de la configuration par défaut.
Plusieurs autres paramètres peuvent également être modifiés, par exemple pour personnaliser le pré-traitement. Une documentation de ces paramètres et des explications est donnée dans le fichier 'Documentation_Config_Parameter', qui est inclus dans le référentiel téléchargé.
Protocole
1. Préparatifs : Configuration technique et installation
REMARQUE : Pour exécuter ce programme, ayez wget, git et Apptainer préinstallés sur l’appareil. Un guide pour l’installation d’Apptainer sur différents systèmes (Linux, Windows, Mac) est donné ici : https://apptainer.org/docs/admin/main/installation.html. Les informations d’installation sur git peuvent être trouvées ici : https://git-scm.com/book/en/v2/Getting-Started-Installing-Git. En fonction de la taille des différents jeux de données d’entrée, il est recommandé d’exécuter le flux de travail sur une machine appropriée (16 CPU, 64 Go de mémoire). Un test de fumée avec les données d’exemple fournies peut être exécuté sur la machine locale. Les instructions et les résultats attendus de l’exécution du protocole sur les données d’exemple sont donnés dans le fichier supplémentaire 1. Reportez-vous au fichier vidéo supplémentaire 1 pour les étapes importantes du protocole qui sont exécutées sur l’ensemble de données décrit ci-dessus.
- Ouvrez la console et choisissez ou créez un dossier dans lequel tout le code d’analyse et les sorties seront stockés. Accédez au dossier en tapant la commande : cd path_to_folder dans le terminal.
- Téléchargez ou clonez le dépôt de code à partir de Github (https://github.com/heiniglab/mofa_workflow) ou en tapant git clone https://github.com/heiniglab/mofa_workflow.git dans la fenêtre du terminal.
- Téléchargez l’image qui contient toutes les installations requises de Zenodo en tapant wget https://zenodo.org/records/11192947/files/mofa_image.sif dans la fenêtre du terminal.
- Générez un dossier dans lequel toutes les données de résultat seront stockées en tapant mkdir results dans la fenêtre du terminal.
- Générez un dossier dans lequel toutes les données d’entrée à utiliser dans l’analyse seront ajoutées en tapant mkdir input_data dans la fenêtre du terminal.
- Exécutez le conteneur qui démarrera une session JupyterLab en tapant la commande suivante dans le terminal : apptainer run mofa_image.sif. Copiez l’URL renvoyée par la commande dans le navigateur, ce qui ouvrira une session Jupyter-lab (plus d’informations sur Jupyter-lab peuvent être trouvées dans la documentation du logiciel16).
REMARQUE : Lorsque le flux de travail est exécuté localement sur un ordinateur portable, il est recommandé d’utiliser la commande apptainer exec mofa_image.sif jupyter-lab à la place, qui renverra directement une adresse d’hôte locale. Dans le cas où le conteneur est exécuté dans un environnement informatique en cluster, il peut être nécessaire de configurer la redirection de port, qui peut être effectuée via ssh.
2. Initialisation et préparation des données
- Dans la session Jupyter-Lab, utilisez le menu de navigation sur le côté gauche. Accédez au dossier input_data en double-cliquant sur input_data.
- Copiez tous les jeux de données qui seront utilisés comme entrée pour l’analyse dans le répertoire input_data à l’aide du glisser-déposer. Faites glisser le fichier à partir du dossier où il se trouve actuellement et déposez-le dans la session Jupyter-lab dans la zone située sous le dossier input_data.
REMARQUE : Tous les ensembles de données doivent être au format .csv ou .h5ad (dans le cas de données à cellule unique). Tous les fichiers .csv doivent contenir une colonne sample_id correspondante (des ID identiques doivent être utilisés dans tous les ensembles de données). Toutes les autres colonnes seront utilisées comme fonctionnalités. Dans le fichier h5ad-, l’annotation de cellule doit contenir deux identificateurs spécifiant le sample_id et le cluster_id. Ceux-ci seront utilisés pour l’agrégation et la correspondance. Les jeux de données omiques dans d’autres formats doivent être convertis au format .csv spécifié avant d’être utilisés (Figure 3). Les ensembles de données scRNA-seq donnés au format .h5seurat peuvent être convertis en .h5ad en exécutant le Jupyter-notebook : 00_Data_Conversion.ipynb. - Accédez au dossier configurations en cliquant sur le symbole du dossier , puis en double-cliquant sur les dossiers mofa_workflow, scripts et configurations. Dans le dossier, ouvrez le fichier Data_configs.csv en double-cliquant dessus.
- Dans la colonne valeur , ajoutez les chemins d’accès aux dossiers des dossiers input_data (data_path) et results (result_path). Ajoutez un nom qui sera ajouté en tant qu’extension de fichier à tous les fichiers enregistrés dans la colonne de valeur du configuration_name (ce protocole utilisé MI_v1 [Infarctus du myocarde version1]) (Figure 4).
- Enregistrez les modifications en cliquant sur Fichier > Enregistrer le fichier CSV dans le menu en haut.
- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier scripts en cliquant sur scripts. Ouvrez le notebook d’initialisation en double-cliquant sur 00_Configuration_Update.ipynb. Exécutez le script en cliquant sur le bouton Redémarrer le noyau et exécuter toutes les cellules en haut, puis en cliquant sur Redémarrer dans la fenêtre contextuelle (Figure 5).
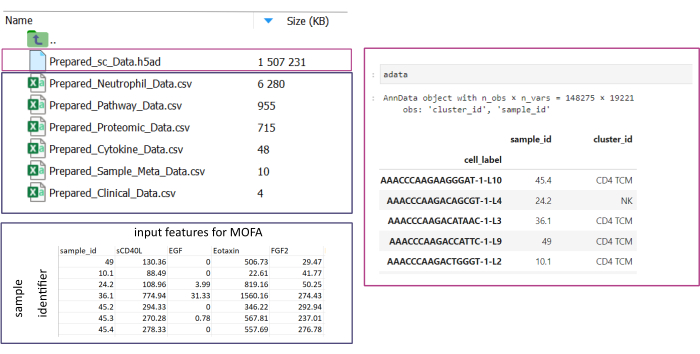
Figure 3 : Saisie et configuration des données. Pour l’exécution du flux de travail, toutes les données doivent être stockées dans un dossier input_data spécifié. Pour chaque jeu de données d’entrée, un fichier distinct doit être fourni. Les données à cellule unique doivent être fournies sous forme de .h5ad contenant l’annotation de cellule sur le cluster_id (résultant, par exemple, des étapes précédentes d’annotation de type de cellule) et une colonne sample_id (identifiant de manière unique chaque échantillon distinct qui doit être analysé). Tous les autres ensembles de données d’entrée doivent être présentés au format « .csv », y compris une colonne spécifiant les sample_id (correspondant à la colonne correspondante des données à cellule unique) et les caractéristiques à utiliser dans l’analyse MOFA dans toutes les autres colonnes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
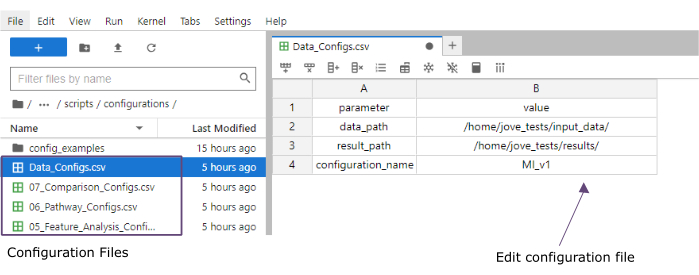
Figure 4 : Fichiers de configuration de Jupyter-lab. Lors de l’exécution du flux de travail, les modifications des paramètres (par exemple, l’ajustement des options de filtrage, etc.) sont spécifiées via des fichiers de configuration .csv. Dans le référentiel cloné, les fichiers de configuration par défaut pour chaque étape sont inclus. Ils peuvent être modifiés directement dans la console jupyter-lab, de la même manière que dans une feuille de calcul. Veuillez cliquer ici pour voir une version agrandie de cette figure.
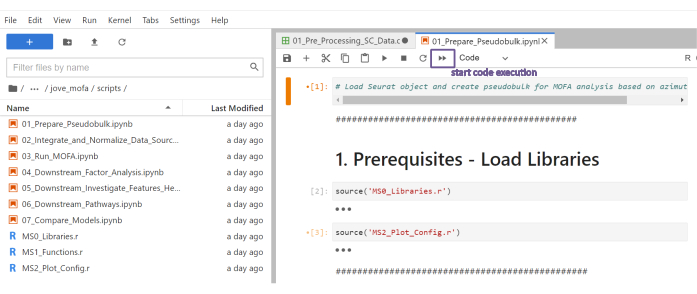
Figure 5 : Scripts Jupyter-notebooks. Le flux de travail complet se compose d’une série de notebooks Jupyter qui seront exécutés séquentiellement une fois que les fichiers de configuration correspondants auront été modifiés. En double-cliquant sur le carnet Jupyter sur le côté gauche, le fichier correspondant s’ouvrira sur le côté droit. L’exécution complète du fichier peut être lancée à l’aide du bouton mis en évidence en haut. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Prétraitement et harmonisation des données
- Prétraitement - Convertissez les données sc en pseudobulk.
REMARQUE : Cette étape ne doit être exécutée que si des données à cellule unique sont utilisées dans l’analyse.- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier configurations en double-cliquant sur configuration. Ouvrez le fichier 01_Preprocessing_SC_Data.csv en double-cliquant. Vérifiez les valeurs renseignées automatiquement dans le fichier et, si nécessaire, ajustez les valeurs de la colonne data_name pour qu’elles correspondent aux noms de fichiers des jeux de données à cellule unique dans le dossier input_data qui sera utilisé pour l’analyse.
REMARQUE : Par défaut, tous les noms des fichiers .h5ad dans le dossier de données d’entrée seront ajoutés au fichier de configuration dans le script d’initialisation. Si certains ensembles de données ne doivent pas être utilisés pour l’analyse, ils peuvent être supprimés ici. - Enregistrer les modifications apportées en cliquant sur Fichier > Enregistrer le fichier CSV dans le menu en haut.
- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier scripts en cliquant sur scripts. Ouvrez le bloc-notes 01_Prepare_Pseudobulk.ipynb en double-cliquant dessus. Exécutez le script en cliquant sur le bouton Redémarrer le noyau et exécuter toutes les cellules en haut, puis en cliquant sur Redémarrer dans la fenêtre contextuelle.
- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier figures en double-cliquant d’abord sur les figures , puis sur 01_figures. Ouvrez le FIG01_Amount_of_Cells_overview de tracé nouvellement généré en double-cliquant dessus.
REMARQUE : L’exécution du bloc-notes peut prendre plusieurs minutes. Lorsque le bloc-notes a été exécuté avec succès, une fenêtre contextuelle s’affiche et le FIG01_Amount_of_Cells_Overview de fichiers est soit mis à jour par le carnet, soit généré à nouveau. La colonne Dernière modification peut indiquer quand le fichier a été généré afin d’évaluer s’il s’agit d’un nouveau ou d’un ancien fichier. - Examinez le graphique pour identifier les groupes de types cellulaires avec un très faible nombre de cellules par échantillon. Notez les noms de ces cluster_ids pour les exclure dans les étapes suivantes (Figure 6).
- Utilisez le menu de navigation sur le côté gauche pour revenir au dossier des configurations en cliquant sur ... puis en double-cliquant sur les configurations. Ouvrez le fichier 02_Preprocessing_Configs_SC.csv en double-cliquant dessus.
- Vérifiez les valeurs dans les colonnes configuration_name et data_name et ajustez-les si nécessaire.
REMARQUE : Dans le script d’initialisation, ces valeurs sont pré-remplies avec tous les noms des fichiers .h5ad dans le dossier de données d’entrée et la valeur configuration_name définie précédemment dans le fichier Data_Configs.csv . Si des fichiers doivent être exclus de l’analyse ou si une autre extension pour les noms de fichiers doit être utilisée, cela peut être ajusté ici. - Ajustez la valeur de la colonne cell_type_exclusion et ajoutez tous les cluster_id qui ont été identifiés à exclure à l’étape précédente, séparés par ', '.
- Enregistrez les modifications en cliquant sur Fichier > Enregistrer le fichier CSV dans la barre de navigation en haut.
- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier configurations en double-cliquant sur configuration. Ouvrez le fichier 01_Preprocessing_SC_Data.csv en double-cliquant. Vérifiez les valeurs renseignées automatiquement dans le fichier et, si nécessaire, ajustez les valeurs de la colonne data_name pour qu’elles correspondent aux noms de fichiers des jeux de données à cellule unique dans le dossier input_data qui sera utilisé pour l’analyse.
- Prétraitement - Harmonisez et intégrez d’autres sources de données omiques.
- Ouvrez le fichier 02_Preprocessing_Configs.csv en double-cliquant dessus et ajustez la configuration de prétraitement pour chacun des jeux de données qui seront inclus et qui sont stockés dans le dossier data_input (une ligne par jeu de données).
- Vérifiez les valeurs dans les colonnes configuration_name et data_name et ajustez-les si nécessaire.
- Ajustez les autres paramètres des colonnes en conséquence, en fonction des étapes de prétraitement à appliquer.
REMARQUE : Des valeurs par défaut sont ajoutées pour chaque ensemble de données qui se trouve dans le dossier input_dataset, mais ne sont pas spécifiques aux types de données uniques des données. Par conséquent, des ajustements seront nécessaires. Une documentation détaillée des paramètres est fournie dans le fichier Documentation_Config_Parameter.doc . - Enregistrez les modifications en cliquant sur Fichier > Enregistrer le fichier CSV.
- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier scripts en cliquant sur scripts. Ouvrez le notebook 02_Integrate_and_Normalized_Data_Sources.ipynb en double-cliquant dessus. Exécutez le script en cliquant sur le bouton Redémarrer le noyau et exécuter toutes les cellules en haut, puis en cliquant sur Redémarrer dans la fenêtre contextuelle.
- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier 02_results généré en cliquant sur le symbole du dossier , puis en double-cliquant sur les résultats et 02_results. Vérifiez s’il inclut le fichier 02_Combined_data_'configuration_name'_Integrated.csv contenant le fichier d’entrée de données prétraité combiné.
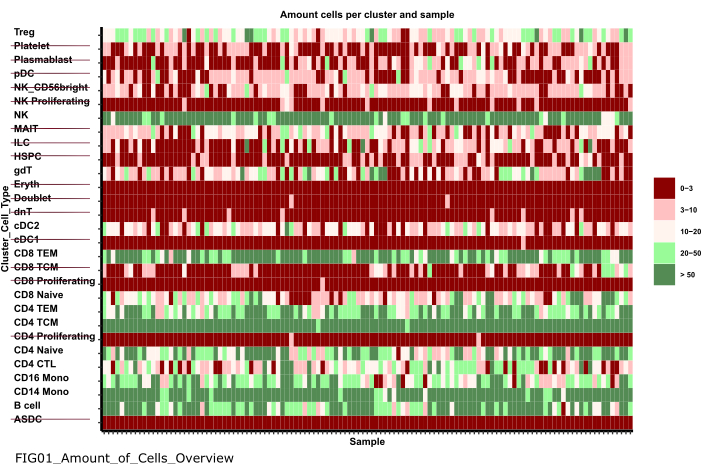
Figure 6 : Prétraitement et harmonisation des données. L’une des sorties de l’étape '01_Prepare_Pseudobulk' est le tracé 'Fig01_Amount_of_Cells_Overview'. Ici, pour chaque cluster_id (axe des y indiquant le type de cellule des étapes d’annotation de type de cellule précédentes), le nombre de cellules par échantillon ('sample_id') est indiqué. Dans les résultats présentés, les types de cellules avec un faible nombre de cellules par échantillon sont exclus de l’analyse ultérieure (indiquée par le barré). Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Exécution de MOFA
- Dans Jupyter-Lab, utilisez le menu de navigation sur le côté gauche pour accéder au dossier des configurations en cliquant sur le symbole du dossier , puis en double-cliquant sur mofa_workflow, puis en double-cliquant sur les scripts et les configurations. Ouvrez le fichier 03_MOFA_Configs.csv en double-cliquant dessus.
- Vérifiez les entrées des colonnes configuration_name et mofa_result_name et ajustez les entrées si d’autres noms doivent être utilisés.
REMARQUE : Le mofa_result_name sera ajouté en tant qu’extension de fichier à tous les fichiers de résultats générés sur la base du MOFA. Cela peut être différent de la valeur configuration_name , car différentes configurations MOFA peuvent être exécutées avec les mêmes données d’entrée (ce protocole utilise MI_v1_MOFA). - Entrez le nombre de facteurs qui doivent être estimés dans le modèle MOFA (colonne amount_of_factors ) et définissez si la pondération et la mise à l’échelle doivent être appliquées (colonnes weighting_of_views et scale_views ) en ajustant les valeurs dans le fichier.
- Enregistrez les modifications en cliquant sur Fichier > Enregistrer le fichier CSV.
- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier scripts en cliquant sur 'scripts'. Ouvrez le notebook 03_Run_MOFA.ipynb en double-cliquant sur le fichier. Exécutez le script en cliquant sur le bouton Redémarrer le noyau et exécuter toutes les cellules en haut et en cliquant sur Redémarrer dans la fenêtre contextuelle.
- Naviguez jusqu’au dossier 03_figures en double-cliquant sur les chiffres , puis 03_figures. Ouvrez le tracé généré FIG03_Overview_Variance_Decomposition_'mofa_result_name et examinez le résultat du modèle (Figure 7A).
- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier 03_results généré en cliquant sur le symbole du dossier , puis en double-cliquant sur les résultats et 03_results. Vérifiez s’il inclut le fichier de valeur de facteur d’échantillonnage 03_Factor_Data_'mofa_result_name'.csv et le fichier de pondération de facteur de caractéristique 03_Weight_Data_'mofa_result_name'.csv.
5. Analyse en aval
- Interprétation des facteurs.
- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier input_data en cliquant sur le symbole du dossier , puis en double-cliquant sur input_data.
- Préparez un fichier .csv (Prepared_Sample_Meta_Data.csv) qui contient toutes les métadonnées (covariables) des échantillons qui seront analysés en association avec les facteurs générés. Copiez le fichier dans le dossier input_data à l’aide du glisser-déposer , en déposant le fichier dans la vue d’ensemble des dossiers du dossier input_data .
REMARQUE : Il doit contenir la colonne sample_id pour le faire correspondre aux données précédemment utilisées et des colonnes supplémentaires pour chaque caractéristique qui doit être analysée. - Dans Jupyter-Lab, utilisez le menu de navigation sur la gauche pour revenir au dossier des configurations en cliquant sur le symbole du dossier , puis en double-cliquant sur mofa_workflow, puis sur scripts et configuration. Ouvrez le fichier 04_Factor_Analysis.csv en double-cliquant dessus.
- Vérifiez que les entrées du configuration_name et du mofa_result_name contiennent les noms des résultats de configuration et de MOFA qui seront analysés dans le script et ajustez-les si nécessaire.
- Dans la colonne numeric_covariates , ajoutez le nom de toutes les colonnes numériques du fichier Prepared_Sample_Meta_Data.csv qui seront examinées par rapport aux facteurs MOFA séparés par une virgule (ce protocole utilise CRP,CK).
- Dans la colonne categorical_covariates', ajoutez le nom de toutes les colonnes catégorielles du fichier Prepared_Sample_Meta_Data.csv qui seront examinées par rapport aux facteurs MOFA séparés par une virgule (Ce protocole utilise la mesure).
- Enregistrez les modifications en cliquant sur Fichier > Enregistrer le fichier CSV.
- Utilisez le menu de navigation à gauche pour accéder au dossier 'scripts' en cliquant sur scripts. Ouvrez le bloc-notes 04_Downstream_Factor_Analysis.ipynb en double-cliquant dessus. Exécutez le script en cliquant sur le bouton Redémarrer le noyau et exécuter toutes les cellules en haut et en cliquant sur redémarrer dans la fenêtre contextuelle.
- Utilisez le menu de navigation de gauche pour accéder au dossier 04_figures en double-cliquant sur les chiffres , puis en 04_figures. Ouvrez les graphiques générés en double-cliquant dessus et recherchez les facteurs pour trouver des motifs et des associations intéressants : FIG04_Factor_Association_with_numeric_features_
'mofa_result_name.pdf (figure 7B). FIG04_Factor_Association_
with_categorical_features_'mofa_result_name.pdf (figure 7C). FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf (figure 8A).
- Analyse des caractéristiques
- Utilisez le menu de navigation à gauche pour revenir au dossier des configurations en cliquant sur ... puis en double-cliquant sur les configurations. Ouvrez le fichier 05_Feature_Analysis_Configs.csv en double-cliquant dessus.
- Vérifiez que les entrées des colonnes configuration_name et mofa_result_name correspondent aux noms de la configuration et du résultat MOFA généré qui seront utilisés pour l’analyse en aval et ajustez-les si nécessaire.
- Dans la colonne facteur , ajoutez le facteur pour lequel les principales caractéristiques seront tracées dans le script suivant.
- Dans la colonne faceting_variable, ajoutez le nom d’une colonne catégorielle dans le Prepared_Sample_Meta_Data.csv qui sera utilisée pour regrouper les échantillons dans le graphique (Ce protocole utilise la mesure)
- Enregistrez les modifications en cliquant sur Fichier > Enregistrer le fichier CSV.
- Utilisez le menu de navigation de gauche pour accéder au dossier scripts en cliquant sur scripts. Ouvrez le bloc-notes 05_Downstream_Investigate_Features_Heatmap.ipynb en double-cliquant dessus. Exécutez le script en cliquant sur le bouton Redémarrer le noyau et exécuter toutes les cellules en haut, puis en cliquant sur Redémarrer dans la fenêtre contextuelle.
- Utilisez le menu de navigation de gauche pour accéder au dossier 05_figures en double-cliquant d’abord sur les figures , puis sur 05_figures. Ouvrez et examinez le graphique généré FIG05_Heatmap_Feature_Overview__ 'mofa_result_name.pdf en double-cliquant sur le fichier (Figure 8B).
REMARQUE : En fonction du nombre de fonctionnalités qui seront affichées dans le graphique, il peut être nécessaire d’ajuster les paramètres plot_width et plot_height dans le 05_Feature_Analysis_Configs.csv et de réexécuter le script pour s’assurer que tout rentre dans le graphique.
- Analyse des parcours
- Utilisez le menu de navigation sur le côté gauche pour accéder au dossier input_data en cliquant sur le symbole du dossier , puis en double-cliquant sur input_data.
- Préparez un fichier .csv (Prepared_Pathway_Data.csv) qui contient une liste de chemins qui seront testés pour l’enrichissement. Copiez le fichier dans le dossier input_data à l’aide du glisser-déposer, en déposant le fichier dans la vue d’ensemble des dossiers du dossier input_data.
REMARQUE : Il doit contenir trois colonnes : ID (un identifiant unique de la voie), gène (les gènes donnés par leur nom de gène (SYMBOL) appartenant à la voie, une ligne par gène), pathway_name (un nom/une description textuelle des voies). - Dans la session Jupyter-Lab, utilisez le menu de navigation à gauche pour naviguer jusqu’au dossier des configurations en cliquant sur ... puis en double-cliquant sur les configurations. Ouvrez le fichier 06_Pathway_Configs.csv en double-cliquant dessus.
- Vérifiez l’entrée de la colonne mofa_result_name et assurez-vous qu’elle correspond au nom du résultat MOFA généré qui sera utilisé pour le calcul de l’enrichissement du parcours.
- Vérifiez l’entrée dans la colonne types et supprimez les entrées de la colonne types qui ne contiennent pas de caractéristiques correspondant à la colonne gène dans le fichier Prepared_Pathway_Data.csv .
REMARQUE : Par défaut, toutes les différentes vues qui ont été utilisées dans le modèle MOFA sont ajoutées à ce fichier lors de l’exécution du flux de travail. S’il existe des vues qui ne contiennent pas de caractéristiques correspondant à au moins un parcours, elles doivent être supprimées ; Sinon, l’exécution échouera. Par exemple, le fichier de chemin ne contient que des annotations de chemin pour les gènes, mais il existe une vue contenant des noms de protéines. - Enregistrez les modifications en cliquant sur Fichier > Enregistrer le fichier CSV.
- Utilisez le menu de navigation pour accéder au dossier scripts en cliquant sur scripts. Ouvrez le bloc-notes 06_Downstream_Pathways.ipynb en double-cliquant dessus. Exécutez le script en cliquant sur le bouton Redémarrer le noyau et exécuter toutes les cellules en haut, puis en cliquant sur Redémarrer dans la fenêtre contextuelle.
- Utilisez le menu de navigation de gauche pour accéder au dossier 06_figures en double-cliquant d’abord sur les figures , puis en 06_figures. Ouvrez le graphique généré FIG06_Pathways_and_Genes_ 'mofa_result_name en double-cliquant dessus et examinez les chemins visualisés (Figure 8C).
REMARQUE : La façon dont les chemins visualisés sont sélectionnés peut être configurée via le fichier de configuration. Pour plus de détails, reportez-vous à la documentation des paramètres. - Utilisez le menu de navigation sur le côté gauche pour accéder au dossier 06_results généré en cliquant sur le symbole du dossier , puis en double-cliquant sur les résultats et 06_results. Vérifiez s’il inclut le fichier contenant les parcours enrichis 06_Pathway_enrichment__'mofa_result_name'.
6. Comparaison de différentes configurations et versions (figure supplémentaire 1, figure supplémentaire 2, figure supplémentaire 3, figure supplémentaire 4)
- Pour comparer l’effet de l’utilisation de différents paramètres/configurations tout au long du flux de travail, réexécutez les sections 3 à 5, en modifiant les paramètres dans les fichiers de configuration et en utilisant différents identificateurs de configuration_name et de mofa_result_name .
REMARQUE : Les nouveaux résultats seront stockés avec ces noms pour être utilisés pour comparer différentes exécutions. - Dans Jupyter-Lab, utilisez le menu de navigation sur la gauche pour accéder au dossier des configurations . Ouvrez le fichier 07_Comparison_Configs.csv en double-cliquant dessus.
- Dans la colonne mofa_result_name , ajoutez les noms de toutes les exécutions MOFA précédentes qui seront comparées (une ligne par nom/configuration, par exemple, MI_v1_MOFA, MI_v2_MOFA).
- Dans la colonne compare_factors , ajoutez les facteurs qui seront comparés entre les modèles. Par défaut, il s’agit de Factor1,Factor2,Factor3. (Figure supplémentaire 2A).
REMARQUE : Dans ce script, les valeurs de caractéristiques et de facteurs des différents modèles seront comparées en les corrélant. Cela ne fonctionne que pour les modèles basés sur les mêmes échantillons (indiqués par sample_id) et le même ensemble de fonctionnalités. Si les échantillons ou les fonctionnalités ne correspondent pas entre les versions comparées, ils seront exclus de la comparaison. - Enregistrez les modifications en cliquant sur Fichier > Enregistrer le fichier CSV.
- Utilisez le menu de gauche pour accéder au dossier scripts en cliquant sur scripts. Ouvrez le bloc-notes 07_Compare_Models.ipynb en double-cliquant dessus. Exécutez le script en cliquant sur le bouton Redémarrer le noyau et exécuter toutes les cellules , puis en cliquant sur Redémarrer dans la fenêtre contextuelle.
- Utilisez le menu de gauche pour accéder au dossier 06_figures en double-cliquant d’abord sur les chiffres , puis sur 06_figures. Ouvrez les tracés générés en double-cliquant sur les fichiers pour analyser la similitude des différentes versions :
FIG07_Variance_Model_Comparison.pdf (Figure supplémentaire 2B)
FIG07_Factor_Correlations.pdf
(Figure supplémentaire 2C)
FIG07_Feature_Correlations.pdf
(Figure supplémentaire 3C)
7. Extension du flux de travail : ajout d’autres paramètres et configurations
REMARQUE : Outre les paramètres actuellement configurables dans les fichiers de configuration, d’autres ajustements dans le code ou d’autres paramètres peuvent être inclus. Par exemple, le modèle MOFA lui-même offre plusieurs autres paramètres d’entraînement17 qui peuvent être soit modifiés directement dans le code, soit rendus ajustables via les fichiers de configuration. La section suivante du protocole présentera un exemple de la façon de procéder pour des paramètres d’entraînement de modèle MOFA supplémentaires. Pour cette partie, des connaissances en programmation R sont requises.
- Dans Jupyter-Lab, utilisez le menu de navigation sur la gauche pour accéder au dossier scripts . Ouvrez le bloc-notes 03_Run_MOFA.ipynb en double-cliquant dessus.
- Cliquez sur l’onglet Table des matières sur le côté gauche, puis accédez à la sous-section 4.3 Définir les options d’entraînement MOFA et exécutez l’entraînement du modèle en cliquant dessus. Faites défiler vers le bas pour voir la sortie imprimée du modèle MOFA des paramètres configurables dans l’ordinateur portable.
- Dans la boucle R pour du code sous l’en-tête, toutes les données MOFA, le modèle et les options d’entraînement sont définis. Sous la ligne model_opts$num_factors = mofa_configs$amount_of_factors[i], ajoutez une autre ligne avec le code ci-dessous
model_opts$likelihoods['data_type'] = 'poisson'.
REMARQUE : Cela modifiera la distribution que le modèle prend en entrée pour la vue spécifiée par le nom data_type pour toutes les exécutions MOFA. Lorsque vous spécifiez poisson pour un type de données, le modèle ne s’exécute que lorsque les caractéristiques de ce type de données sont des entiers (par exemple, le nombre de lectures à partir du séquençage de l’ARN). Pour obtenir plus d’informations sur les données, l’entraînement et les options de modèle MOFA, vous pouvez également consulter les tutoriels et la documentation MOFA17. - Enregistrez les modifications dans le carnet en cliquant sur le bouton Enregistrer en haut.
- Pour transférer de nouveaux paramètres via les fichiers de configuration .csv , utilisez la navigation sur le côté gauche pour accéder au dossier des configurations en double-cliquant sur les configurations et ouvrez le fichier 03_MOFA_Configs.csv en double-cliquant.
- Ajoutez une nouvelle colonne spécifiant le nom du paramètre, par exemple, number_iterations et entrez une valeur, par exemple, 1000. Enregistrez les modifications en cliquant sur Fichier > Enregistrer le fichier CSV.
- Utilisez le menu de navigation pour naviguer dans le dossier scripts en cliquant sur scripts. Ouvrez le bloc-notes 03_Run_Mofa.ipynb en double-cliquant dessus. Cliquez sur l’onglet Table des matières sur le côté gauche, puis accédez à la sous-section 4.3 Définir les options d’entraînement MOFA et exécutez l’entraînement du modèle en cliquant dessus.
- Remplacez la ligne train_opts$maxiter = 50000 par train_opts$maxiter = mofa_configs$column_name[i] (lorsque le nom de la colonne ajoutée est number_of_iterations il est train_opts$maxiter = mofa_configs$number_of_iterations[i])).
REMARQUE : Le fichier de configuration 03_MOFA_Configs.csv dans ce notebook est lu au début du notebook (sous-section : Configurations et paramètres préalables) comme mofa_config data.frame dans la session et, par conséquent, dans cette ligne de code, cet objet et la colonne correspondante nouvellement générée sont référencés. Comme plusieurs configurations peuvent être exécutées en même temps, le i identifie la ligne du data.frame lorsque l’estimation du modèle est exécutée dans une boucle for sur toutes les différentes lignes du fichier .csv . Le principe de lecture dans le fichier de configuration au début du notebook dans la section 'Configurations et paramètres prérequis' est le même pour tous les notebooks, et d’autres modifications peuvent être apportées de cette manière. - Enregistrez les modifications dans le carnet en cliquant sur le bouton Enregistrer .
Résultats
Après l’exécution réussie du flux de travail, plusieurs tableaux et figures sont générés, comme l’indique la figure 2. Les figures sont placées dans le dossier /figures (Figure 6, Figure 7, Figure 8, Figure supplémentaire 1, Figure supplémentaire 2, Figure supplémentaire 3, Figure ...
Discussion
Avec le protocole décrit, un flux de travail modulaire et extensible basé sur un bloc-notes Jupyter qui peut être utilisé pour explorer rapidement un ensemble de données multi-omiques complexe est présenté. Les principales parties du flux de travail consistent en la partie prétraitement et harmonisation des données (offrant différentes étapes standard pour le filtrage et la normalisation des données), l’estimation du modèle MOFA9 et une analyse en a...
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
C.L. est soutenu par l’Association Helmholtz dans le cadre de l’école de recherche commune « Munich School for Data Science - MUDS ».
matériels
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
Références
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31 (2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535 (2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125 (2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22 (2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806 (2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124 (2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124 (2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88 (2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15 (2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121 (2022).
- . Get Started - JupyterLab 4.1.0a4 documentation Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024)
- . MOFA2: training a model in R Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020)
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272 (2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68 (2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111 (2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA Available from: https://biofam.github.io/MOFA2/ (2024)
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon