Method Article
זרימת עבודה עבור גבוהה תוכן, כימות תא יחידה של פלורסנט סמנים מUniversal מיקרוסקופים נתונים, הנתמכים על ידי תוכנות קוד פתוחות
In This Article
Summary
מוצגים הוא עבודה אינפורמטיקה גמישה המאפשרת ניתוח מבוסס תמונת ריבוב של תאים שכותרתו fluorescently. העבודה מכמתת סמנים גרעיניים וcytoplasmic ומחשבה טרנסלוקציה סמן בין תאים אלה. נהלים מסופקים ההפרעות של תאים באמצעות siRNA ומתודולוגיה אמינה לגילוי סמן על ידי immunofluorescence העקיף בפורמטי 96-היטב.
Abstract
התקדמות בהבנת מנגנוני הבקרה המסדירים את התנהגותם של תאים בתרבית רקמת דגמי יונקים חסיד הופכות תלויה יותר ויותר במצבים של ניתוח מתא בודד. שיטות אשר מספקות נתונים מרוכבים המשקפים את הערכים הממוצעים של סמנים ביולוגיים מאוכלוסיות תאים עלולות לאבד דינמיקה תת-אוכלוסייה המשקפת את ההטרוגניות של המערכת הביולוגית למדה. בקנה אחד עם זו, גישות מסורתיות מוחלפות על ידי, או נתמכת עם, צורות מתוחכמות יותר של assay הסלולרי שפותחו כדי לאפשר הערכה על ידי מיקרוסקופ גבוה תוכן. מבחני אלה פוטנציאל לייצר מספר רב של תמונות של סמנים ביולוגיים ניאון, אשר מופעלים על ידי המלווה את חבילות תוכנת קניינית, מאפשר למדידות רבות פרמטרית לכל תא. עם זאת, עלויות הון גבוהות יחסית והתמחות יתר של רבים מהתקנים אלה מנעו את נגישותם לחוקרים רבים.
שתואר כאן הואזרימת עבודה אוניברסלית לכימות של עוצמות סמן פלואורסצנטי מרובות מאזורי subcellular הספציפיים של תאים בודדים מתאימים לשימוש עם תמונות מרוב מיקרוסקופי ניאון. מפתח לזרימת עבודה זו הוא יישום תוכנת Profiler הסלולרי זמינה באופן חופשי 1 להבחין בין תאים בודדים בתמונות האלה, קטע אותם לאזורי subcellular המוגדרים ולספק עוצמת הקרינה סמן הערכים ספציפי לאזורים אלה. חילוץ ערך עוצמת תא בודד מנתוני תמונה הוא המטרה של זרימת עבודה זו המרכזית ויהיה מאויר עם הניתוח של נתונים שליטה ממסך siRNA לרגולטורי מחסום G1 בתאים אנושיים חסיד. עם זאת, העבודה שהוצגה כאן יכולה להיות מיושמת על ניתוח של נתונים מאמצעים אחרים של הפרעת תא (לדוגמא, מסכי מתחם) וצורות אחרות של סמנים סלולריים הקרינה מבוססת וכך צריכים להיות שימושיים למגוון רחב של מעבדות.
Introduction
העבודה מוצגת כאן מתארת את השימוש בתוכנת Profiler הסלולרי הזמינים באופן חופשי לבצע פילוח מודרך אלגוריתם של תמונות מיקרוסקופ פלואורסצנטי של תאים חסיד לזהות תאים בודדים ואזורי subcellular מוגדרים. גישה זו, המכונה פילוח תמונה, מאפשרת הניתוח רב-פרמטרית הבא של תאי הדמיה על ידי כימותי סמנים שכותרתו fluorescently מקומיים לכל תא או אזור subcellular (המכונה אובייקטים מפולחים כ). עבודה זו מהווה בסיס למאפשר ניתוח גבוה תוכן, והוא נועד לשמש ככלי שיכול להיות מפותח יותר ושונה כדי שיתאים לרב-פרמטרית, תא בודד מנתח במעבדות ללא גישה למכשירים גבוהים תוכן מיוחדים או תוכנת קניינית. הקבצים מצורפים לכתב יד זה כוללים מערך בדיקה של הנתונים רלוונטיים תמונת גלם, הגדרות אלגוריתם ותסריטים התומכים על מנת ליצור את הניתוח שתואר. F הגדרות אלגוריתם מסופקתאו Profiler הסלולרי מותאם לנתונים הדוגמא להגדיר ופרטי סעיף הדיון מה התאמות עשויות להיות נחוצות כדי לאפשר שימוש בנתוני תמונה ממחקרים אחרים.
ברגע שנתונים כמותיים כבר הופקו באמצעות Profiler הסלולרי, ייתכן שיש לי מעבדות שונות יש דרישות שונות לאופן שימוש במידע שהוצג על ידי ערכי תא הבודדים בנתונים הגולמיים; המוצג כאן הוא גישה אחת על ידי שעיר מוחלים על הנתונים הגולמיים עבור כל assay. שימוש בשערים אלה, נתונים הופכים למונחים בינארי של תגובה, המאפשרים הדמיה של מגמות המקשרות טיפולים שונים עם תת-האוכלוסיות של תאים עוברים תגובה שהוגדרה על ידי השערים. השערים נקבעים בהתבסס על תצפיות של הפצות הנתונים המתקבלות לבקרות שליליות וחיוביות מתאימות לכל מדידה רלוונטית. השימוש בשערים הוא רק דוגמא אחת של איך לנהל את מדידות גלם, המבוסס על התאים. כמו כן מוצג כאן הוא השימוש בעוצמת DNA הגרעיניasurements בצורתם הגולמית כטווח רציף של ערכים בשילוב עם נתונים מגודרות. גישות אחרות לניהול נתונים ניתוח תמונה צריכה להיחשב, בהתאם לאופי של המחקר; חלופות סטטיסטיות לשימוש בשערים לשיוך תאים לאוכלוסיות דווחו 2 ושיטתי השוואות של אסטרטגיות כדי לסכם נתונים גבוה תוכן במספר גדול של פרמטרים דווח 3.
ניתוחים גבוהים תוכן של נתוני תמונה מצאו שימוש במחקרים סלולריים של סמים-תגובה, הפוך גנטיקה ולחץ סביבתי איתות 4-6. הכשרון של ניתוח גבוה תוכן נובע מהעובדה שהניתוח האלגוריתמי של נתוני מיקרוסקופ פלואורסצנטי מאפשר פרמטרים כמותיים ומרחב כדי להיחשב בו-זמנית על פני תאים בודדים 7. בדרך זו, תוצאות סלולריות למבחנים מרובים יכולות להיות התנהגות מוצלבים, ההפרש של assay-דניתן לעקוב תת-אוכלוסיות תאי efined בתוך תנאים ומבחנים ניסיוניים יכולים לכלול בחינה של משתנים מורפולוגיים. העבודה האסטרטגיות וניתוח שנדונה כאן, כמו לגישות גבוהה תוכן אחרים, הם מסוגלים לספק נתונים מרובבים המצליבים לתאים בודדים. מחקרי חליפת שיטות גבוהים תוכן שיוצרים תמונות מיקרוסקופ פלואורסצנטי וחלים על ניתוח של נתונים הנעים בין עשרות תמונות שהופקו במיקרוסקופיה הקרינה מבוססת קונבנציונלית תפוקה נמוכה ועד לאלף תמונות שהופק באמצעות פלטפורמות גבוהות הקרנת תוכן אוטומטית.
העבודה באה לידי ביטוי כאן עם נתוני דוגמא שממבחנים נפרדים הנמדדים במונחים של שני עוצמות גרעיניות ניאון סמן או טרנסלוקציה הגרעינית / cytoplasmic של חלבון כתב ניאון, בהתאמה. העבודה היא גמישה שבמבחנים אלה יכולים להיחשב בנפרד או בשילוב בהתאם EACh נתון שאלת מחקר על ידי חוקרים שונים. נתוני הדוגמא מיוצרים כחלק מניסוי התערבות RNA (RNAi) (איור 1). oligonucleotides הקטן מפריע RNA (siRNA) משמש למציאת חלבונים ספציפיים בתאי סרטן מעי גס האנושי HCT116 שגורמים לשינויים לשני כתבי ניאון של פעילות cyclin-dependent kinase (CDK). זירחון CDK6 תלוי של החלבון הגרעיני רטינובלסטומה בסרין 780 (RB1 P-S780) מוערך על ידי צביעת נוגדן. באותם תאים, כתב חלבון מתויג ניאון ירוק של פעילות CDK2 (כתב GFP-CDK2) מוערך על ידי הגרעין שלה ליחס cytoplasmic בי בהעדר פעילות CDK2 הכתב מתגורר בגרעין ועל הסעות הפעלת CDK2 לתוך הציטופלסמה 8. בנוסף, ה- DNA הגרעיני של כל תא מוכתם באמצעות צבע intercalating-DNA, Bisbenzimide, המשמש כאמצעי לזיהוי תאים ולהגדיר גבולות גרעינים בתמונות, כמו גםמדד sa של שפע DNA מתן מידע על עמדת מחזור התא של התא (איור 2).
הפעילויות של CDK6 וCDK2 הן לגילוי תאי מעבר מG1 לשלב S של מחזור התא 5 ולהצליח בכל 9,10 אחר, וככזה, קרובה התאמה בין שני הכתבים בתאים בודדים צפויה. בסיס נתוני ההפגנה משמשים כאן כדוגמא מנתח את ההשפעה של siRNA מטרות CDK6, חלבון רטינובלסטומה (RB1) ושליטה ללא מיקוד שלילית (טבלת 1). מציאה של CDK6 צריכה לעורר שתי ירידה של epitope RB1 P-S780 והצטברות של תאים בשלב G1 של מחזור התא. מציאה RB1 משמשת כביקורת מגיב להספציפיות של נוגדן phospho-S780. תמונות מיקרוסקופ פלואורסצנטי מפורמלין קבועות 11, תאים בתרבית רקמת HCT116 מוכתמים fluorescently משמשים לניתוח תמונה אלגוריתמי. נתונים המספריים שנוצר לאחר מכן נעשה שימוש להפניה מקושרת הכתבים ולאמוד את ההשפעה של המדינות השונות מציאה.
גודל הפוטנציאל של נתונים המיוצרים על ידי סוג זה של ניתוח יכול להוות אתגר לכלי ניתוח רגילים. לדוגמא, את נתוני תא הבודדים יכולים להיות גדולים יותר מאשר כמה תוכנות גיליון אלקטרוני תהיה להתאים. הוצאות כוללים סקריפטים Perl אשר מבצעים עיבוד פשוט, מאוד-חוזר על עצמו, בפיקוח של הנתונים כדי לסייע ניתוח של מערכי נתונים גדולים. סקריפטים Perl נכתבים במיוחד עבור קבצי פלט המיוצרים על ידי התאים Profiler, בעת עיבוד קבצי תמונה עם אמנת קובץ שמות ספציפית (איור 3), ולאפשר למספרים משתנה של שדות לכל גם לשימוש בניתוח. היא לעתים קרובות חשובה לנתונים assay תא בודד שער כדי לעקוב אחר מגמות בתת אוכלוסיות של תאי 5 ומוצגים כאן הוא השימוש בתסריט פרל לדגל כל תא המבוסס על שער שנקבע מראש סט לכל סוג assay. כמו כן, נכללים סקריפטים Perl אופציונלייםשיסכם את תוצאת נתונים לבארות בודדות (או תנאים), ומספק: אחוז התאים בתוך שער הסט והערכים הממוצעים של ציוני assay גלם. הדרך השנייה, הומוגנית יותר של הצגת הנתונים, תקפה שבו תגובות רוב התאים בתוך היטב או כל להשפיע. כאמור לעיל, הערכה כזו היא פחות שימושית מזה מוענק על ידי gating נתוני תא הבודד שבי התגובה מוגבלת לקבוצת משנה של תאים בתוך אוכלוסייה.
השירות של זרימת העבודה המתוארת אינו מוגבל להפרעות על ידי siRNA או מבחני הסמן תיארו. מחקרים השתמשו בגישה זו לassay תגובות בניסויים בתרבית רקמה באמצעות שילובים של siRNA, מעכבים כימיים וטיפול בקרינה, ולהערכה של סמנים אחרים מאשר CDK6 ופעילות CDK2 5.
מבחינה מושגית, האסטרטגיה הניסיונית מאפשרת מגוון רחב של אזורי subcellular ביולוגיים שימושיים להיות רשום באופן אוטומטיבתאים בודדים הנמצאים בתמונות מיקרוסקופ פלואורסצנטי. ככזה, גישה זו יכולה להניב מידע כמותי, נתונים ריבוב חושף ביולוגי שעלולים להחמיץ באמצעות טכניקות המתמקדות באוכלוסיות ולא תאים בודדים. עם שינויים קלים, העבודה הגישה וניתוח שתוארה יכולה להניב נתונים כמותיים תא, פרט לכל יציאות הקרינה מבוססת assay ותגובות תא-ביולוגי, שבו הערכה כמותית של תוכן DNA, כימות של הקרינה גרעינית או cytoplasmic או הטלטלות של סמנים בין אלה שני תאים בנפרד או באופן ריבוב הוא עניין. דרישות הוצאה לאור נוטות יותר ויותר לכיוון הגשת נתונים בגלוי הנגיש גלם, גישה ולהיכרות עם כלים חינמיים לניתוח תמונה מיקרוסקופית כגון אלה שתוארו כאן גם יהיו עניין ישיר למעבדות מחפשים לנתח מחדש נתונים שפורסמו.
Protocol
1. ניסיוני הפרעות ותיוג תא לסמני תגובה (מסך ההפוך Transfection siRNA)
- בפיפטה ברדס רקמת סטרילית תרבות 70 μl של 200 ננומטר siRNA במאגר 1x siRNA בבארות של צלחת סטרילי, רגיל 96 גם. לדלל שומנים transfection לתוך 40 כרכים של תקשורת סרום ללא DMEM ולוותר 105 μl לכל siRNA המכיל גם.
הערה: דילול 262.5 שומנים μl ל10.5 מיליליטר של סרום ללא DMEM מניבה תערובת אמן מתאימה לצלחת גם 96 כל siRNA, ומספקת 2.6 μl של שומנים בדם בכל טוב. שימוש של 200 ננומטר ריכוז מתחיל siRNA בשלב זה יספק ריכוז עבודה של 20 ננומטר בשלב 1.3, אבל נהלים יעבדו לעבודת ריכוזים עד 5 ננומטר, עם הריכוז מתחיל המותאם בהתאם (כלומר 50 ננומטר). ריכוז עבודה נמוך יותר עשוי להפחית ציונים חיוביים כוזבים מחוץ היעד, למרות שהם יכולים להפחית את עוצמת תגובה על-היעד, שהוביל לגידול של-targe בt שיעורים שליליים כוזבים. - מערבבים את הצלחת על ידי רטט עדין לעשר דקות בטמפרטורת חדר. תת-לחלק את 175 μl וכתוצאה מכך לשלוש 50 μl משכפל ליעד על תרבות שטופלה, 96-גם צלחת עם בסיס שקוף אטומה, רקמה.
- transfect הפוך על ידי מחלק 8,000 תאים לכל גם ב150 DMEM μl המכיל 10% בסרום ישירות על מתחמי שומני siRNA 50 μl. תאי מעי גס אנושיים השימוש HCT116 יציבות להביע סמן GFP מתויג דיווח על פעילות CDK2 5,8. ללא ערבוב נוסף הוא הכרחי. חותם את הצלחת עם קרום לנשימה סטרילי, דבק לשלוט לחות ולמנוע 'קצה הלוואי' הצלחת ומניח את הצלחת לתוך חממת humidified על 37 מעלות צלזיוס, 5% CO 2 במשך 48 שעות.
- לשאוב את התקשורת באופן שסכום שייר קטן של תקשורת נשאר בבארות. תקן את התאים על ידי הוספה של 4% פורמלדהיד נאגר 100 μl לכל היטב דגירה במנדף במשך 10 דקות בחדר temperature.
- הסר את פתרון התיקון על ידי aspirating הצלחת. בשלב זה או להפסיק את הניסוי על ידי שטיפת הצלחת שלוש פעמים עם 100 מלוח μl פוספט שנאגרו (PBS) ולאחר מכן לאחסן אטום, תחת של PBS 100 μl בחושך על 4 מעלות צלזיוס עד שבוע, או להמשיך ב permeabilization של התאים.
הערה: אנו ממליצים צלחות עיבוד בהקדם האפשרי לאחר קיבוע, ובאופן כללי מעדיפים אחסון של צלחות מעובד באופן מלא. חומרים משמרים Biocidal כגון תימרוסל, אזיד הנתרן, או חלופות מסחריות ניתן להוסיף כדי למנוע צמיחת micoroganismal. תוספת של מעכבי phosphatase עוזרת לשמר phospho-epitopes, ואמצעים אחרים כדי לשמר מדינות שינוי החלבון עשוי להיות שימושי בהקשרים רלוונטיים assay - הסר PBS מהצלחת וpermeabilize התאים על ידי הוספה של פתרון permeabilization 100 μl. דגירה של 10 דקות בטמפרטורת חדר בלי לרעוד. לשאוב את פתרון permeabilization באמצעות multichפיפטה annel. חזור על פעולה זו שלוש פעמים.
- חסום את התאים על ידי הוספת 100 פתרון לחסום μl לכל גם במשך 30 דקות בטמפרטורת חדר. הסר את הפתרון לחסום ידי aspirating הצלחת, אז לחקור עם 50 μl של נוגדן RB1 P-S780 אנטי דילול 500 פי בפתרון לחסום עבור שעה 2 בחושך בטמפרטורת חדר.
- לשטוף את הצלחת שלוש פעמים עם 100 פתרון לשטוף צלחת μl, עוזב את הפתרון בצלחת במשך 5 דקות בכל פעם. לחקור את צלחת הלילה בחושך על 4 מעלות צלזיוס עם 50 נוגדנים משני 1000 פי דילול μl fluorescently מתויג בפתרון לחסום בתוספת 2 מיקרומטר של צבע DNA הכרומטין הספציפי Bisbenzimide. לשטוף את הצלחת שלוש פעמים כמו קודם וחנות אטומה, מתחת ל -100 μl PBS בחושך על 4 מעלות צלזיוס. תמונת הצלחת בתוך שבועיים.
2. הדמיה וסגמנטציה
- השתמש במיקרוסקופ פלואורסצנטי confocal או ספינינג-הדיסק עם מטרת 20X לקחת נפרד 16-בתים, תמונות TIFF בגווני אפור בשלושה ערוצים המקבילים לצבע DNA, GFP וfluorophores חיסוני מכתים. ללכוד רבים סטי תמונה שאינן חופפים, המכונה כאן מסגרות, לתמונה כ 1,000-2,000 תאים לכל טוב.
- שם קבצי התמונה באופן שיטתי, כך שכל שם קובץ הוא שילוב ייחודי של 'שם ניסוי "," גם את כתובת', 'מספר מסגרת' ו 'מזהה ערוץ', בסדר זה (איור 3). נתוני דוגמא הסט משתמש "כחול" (צביעת הכרומטין DNA) או "הירוק" (GFP) או "אדום" (fluorophore המוכתם החיסונית) כמזהי ערוץ. מזהה את כתובת היטב, מספר מסגרת וערוץ הנוסף על המכונה תמונה מטה. השתמש בסמל התחתון כדי למנוע metadata היטב והמסגרת מבלבל.
- שם קבצים עם אלמנטי metadata הבאים לפי הסדר שצוין. זה הכרחי על מנת להבטיח שהצעדים התוכנה הבאים לקורערכות קבוצת ctly של תמונות לצורך הניתוח.
- להוריד ולהתקין את Profiler נייד התוכנה החופשית, פעיל פרל Edition הקהילה, סביבת R סטטיסטית תכנות וRStudio. קבל את כל אפשרויות ברירת המחדל במהלך התקנה; משתמשי PC התקנת Perl Active צריכים לאפשר לכל האפשרויות הנוגעות לPATH, עמותת סיומת קובץ ומיפוי תסריט שבו תתבקש לעשות זאת. פרל הפעיל הוא אופציונאלי עבור משתמשי מקינטוש, אבל הם אחרת יצטרכו להפעיל את תסריט פרל בשלב 3.2 משורת פקודת המסוף ולא באמצעות לחיצה על סמל.
- פתח את תוכנת Profiler הסלולרי, לחץ על 'קובץ', 'יבוא צינור' אז 'מקובץ' ובחר את קובץ 3_channels_pipeline.cppipe (האיורים S1A & S1B). הקובץ מכיל הוראות הדרושות לתוכנה לפרש metadata קובץ תמונה משם הקובץ מתואר הכנס. Profiler תא עכשיו מתייחס תמונות, תמציות DNA גרעיני ועוצמות נוגדן מהESE ומשתמש בערוץ ה- GFP כדי לחשב את היחס של גרעין לעומת עוצמת ציטופלסמה עבור כל תא זוהו (איורים 4 ו -5).
- לחץ על "הגדרות הפלט צפה בכפתור בפינה השמאלית התחתונה של חלון Profiler הסלולרי. בחלק העליון של המסך החדש הן תיבות טקסט שכותרתו "תיקיית ברירת מחדל של קלט 'ו' תיקיית פלט ברירת מחדל '. אחד בכל פעם, לחץ על התיקייה-סמלים בצד הימין של תיבות אלה ובחר את המיקום של קבצי תמונה לניתוח והיעד לנתונים שחולצו, בהתאמה (איור S1C).
- בגין ניתוח תמונה על ידי לחיצה על הכפתור "התמונות לנתח" בפינה השמאלית התחתונה של Profiler הסלולרי. בחלק התחתון של המסך לצפות בזמן שנותר להפקת נתונים, 'ניתוח Stop' ולחצני 'השהה'. אם נדרש להשהות את הניתוח על ידי בחירה בלחצן 'השהה' בכל עת, אשר היא שימושיבעת צפייה בתמונות שנתחה (מתואר בשלב 2.8).
- לחלופין, לפתוח את החלונות לכל אחד משלבי ניתוח תמונה על ידי לחיצה על עין-הסמלים בפנל על השמאל הקיצוני של חלון התכנית (איור S1D). שים לב לחלון 'IdentifyPrimaryObjects' ואלה עבור 'אובייקטים תיכונית' 'משני' ולבדוק שההגדרות הנוכחיות בProfiler הסלולרי לבצע פילוח תמונה מתאים (ראו תרשים 1 ודיון לקבלת ייעוץ על שינוי הגדרות אלה).
- לחץ על 'אישור' בתיבת ההודעה שתופיע כאשר הניתוח הוא מלא. עבור אל 'תיקיית ברירת המחדל של הפלט' המיקום שבו כל קבצי נתונים עם התוצאות נשמרים כמופרדים בפסיקים-ערך (csv) קבצים (איור S2A).
3. הפקת נתונים
- מצא את הקובץ החדש 'Nuclei.csv', אשר נכלל ביןפלט מProfiler הסלולרי. קובץ זה מכיל נתונים תא יחידים לעוצמת ניאון נוגדן גרעיני, עוצמת DNA גרעינית וערכי GFP-CDK2 יחס כתב (איורים 6 א & S2A).
הערה: מעבדות שונות רוצה לעבד את זה סוג של נתונים כדי שיתאימו לאופי של מבחני שלהם. הציע לנתונים הנוכחיים הוא gating של תאים מכל מצב טיפול על פי נתוני הנוגדן וערכי כתב GFP-CDK2 באמצעות סקריפט Perl מסופק 2_gate_classifier.pl '. - העתק את קובץ התסריט פרל מסופק 2_gate_classifier.pl 'לאותה התיקייה כקובץ נתונים' Nuclei.csv '(איור S2A). לחץ לחיצה כפולה על הסמל לתסריט פרל ו, כאשר תתבקשו לעשות זאת, הקלד את השם המלא של קובץ הנתונים ואחריו את שם קובץ '.csv' לקובץ שבו התאים הם להיות מגודרות ולבסוף ערכי השער לנוגדןהקרינה ונתונים כתב GFP-CDK2.
הערה: כיצד לקבוע את הגדרות בעיקר שער ולהחיל אלה לניתוח של הנתונים בהמשך בסעיף נציג נתונים ואיור 6 (כדי לנתח את נתוני שימוש מסופק 0.004 'ו' 1.5 ', בהתאמה). משתמשי מקינטוש צריכים להריץ את הסקריפט פרל משורת פקודת המסוף על ידי הקלדה: "פרל 2_gate_classifier.pl '. - שים לב לקובץ החדש שנוצר המשלב את ערכי assay התא בודדים גלם מנתוני Profiler הסלולרי המקוריים עם תוויות תת-אוכלוסייה שמראים איך כל תא מכל הטוב מבצע נגד שני השערים (איור 6 ג).
- העלילה נתונים עבור כל תנאי ניסוי באמצעות התוויות תת-אוכלוסיית תא בודדת על ידי פתיחת תוכנת RStudio. לחץ על 'קובץ' ו 'פתח קובץ', ולאחר מכן בחר את הקובץ 'analysis.r' סיפק. שים לב לפקודות לעלילת 6B דמויות, 7 ו -8 בחלון השמאלי העליון של RStudio (איור S2B). בחלון השמאלי העליון, בין סימנים ציטוט כפולים על קווים 5 ו -6, הקלד את כתובת המחשב של התיקייה המכילה את הנתונים מגודרות. מוסיף את אות כונן ואת שמו של הקובץ עצמו, בהתאמה (לדוגמא, "C: / תיקיית ניתוח / פלט ניתוח" ו- "nuclei_gated.csv").
הערה: אם RStudio משמש בפעם הראשונה על מחשב נתון, חבילת גרפיקת ר 'ggplot2' תצטרך להיות מותקנת ראשון. זהו צעד רק פעם אחת להתקנה חדשה של RStudio, לאחר שצעד זה הופך להיות מיותר. כדי להתקין 'ggplot2', לחץ על הכרטיסייה בשם 'חבילות' מעל החלון בפינה הימנית התחתונה של RStudio, לחץ על הכפתור 'חבילות התקנה' שמופיע מתחת לזה. חלון חדש יופיע. "Ggplot2 'סוג (ציטוטי השמטה) לים' חבילות 'קצב בחלון חדש זה, ולבסוף לחץ על הכפתור 'תקן' כדי לסגור את החלון, להתקין את פונקציות ggplot2 הכרחיות ולחזור לחלון הראשי RStudio להמשיך מצעד 3.6. - קווי גולת כותרת 1 עד 17 בחלון השמאלי העליון של RStudio, לאחר מכן לחצו על הכפתור "הפעל". זה ייכנס לנתוני הניסוי, ערכי סף ופרטי מיקום טוב לR (איור S2C). R כעת באופן זמני להחזיק את הנתונים הרלוונטיים לקשירת קשר.
- סמן בלוקים בודדים של הקוד שנותר מתחת לקו 17 וליצור עלילות המקבילה על ידי לחיצה על הכפתור "הפעל" כמו קודם. שים לב למגרשים בחלון בפינה הימנית התחתונה של RStudio ולשמור את המספר של פורמטים ידי לחיצה על הכפתור "היצוא" (איור S2D).
- בעת סגירת RStudio, לחץ על 'אל תשמור' כאשר תתבקשו לעשות זאת. זה מונע בלבול בשימוש הבא של RStudio, אשר אחרת יקיים נתוניםמהפגישה הקודמת.
תוצאות
דוגמא סט של תמונות שנוצרה באמצעות פרוטוקול הקרנת siRNA ההפוך-transfection הוכן ולנותחה באמצעות תוכנת Profiler הסלולרי. הנתונים הגולמיים המספריים וכתוצאה מכך הוא כזה שכל תא מיוצג בנפרד, למעקב חזרה לתמונה שלה וגם ממוצא ונמדד במשך כמה פרמטרים עוצמת הקרינה (איור 6 א). עבור כל תא זיהה את עוצמת הקרינה גרעינית הממוצעת לנוגדן RB1 P-S780 ועוצמת DNA המשולבת למסכות הגרעיניות מוגדר צבע DNA נקבעים. אומר ערכי עצמת GFP לאזורי גרעין והציטופלסמה של כל תא נרשמים גם מאפשר חישוב גרעיני לעומת הקרינה cytoplasmic של כתב ה- GFP-CDK2. במורד הזרם של שימוש הקרינה האלגוריתמי אלה מדידות עוצמת עשוי מנתונים אלה תאים הבודדים להגדיר שערים לשני מבחני, מכתים נוגדן גרעיני וכתב GFP-CDK2. הביאור הבא של התאים על הבסיס דואר של תוצאת assay ושימוש בתוויות אלה כדי לאפשר תת-אוכלוסיות ספציפיות להתאפיין נוסף על ידי מדידה שלישית (תוכן DNA גרעיני) מתוארת.
חלקות היסטוגרמה של נתוני עוצמת הקרינה גלם שנאספו עבור כל assay הן דרך יעילה להערכה כיצד תת-אוכלוסיות תאים מתנהגות בתנאים שונים. היסטוגרמות באיור 6 מראות את התפלגות האוכלוסייה של נתוני תא בודדים מבארות בשלושה עותקים עבור כל תנאי מציאה RNAi. משמאל הם נתונים לעצמת נוגדן גרעיני ועל זכותם נתונים המקביל לכתב GFP-CDK2. נתונים נוגדן RB1 P-S780 מגלים כי התאים קיימים באופן כללי בשתי אוכלוסיות ביחס לשינוי שלאחר translational זה ושאוכלוסיות תאים עם הפסד של RB1 phosphorylated על S780 ניתן להבחין כשמאלי שיא של עוצמה גרעינית שהועשר כאשר CDK6 הוא דפק על ידי siRNA. אותו שמאלי שיא זהאפשר לראות כאשר RB1 עצמו הוא יעד RNAi, המשקף את ההסרה המוחלטת של החלבון ובכך מכתים RB1 P-S780. בניגוד לכך, אותם התנאים ניסיוניים לאותם תאים, כאשר נצפו באמצעות assay כתב GFP-CDK2, להראות דינמי שונה בנתונים תא הבודדים. הפצה רציפה הוא ציין, עם רק שיא אחד, אבל siRNA שמפריע מחזור התא (siCDK6) וגורם להצטברות בתוצאות שלב G1 בהארכת יד ימין הכתף של הפצה ש( כלומר המצביע על נוכחות מוגברת של תאים המראים עלייה ביחס GFP הגרעיני / ציטופלסמה, זמם על ציר ה- X).
כמו כן מוצג על היסטוגרמות של איור 6 הם ערכי השער (הקווים האנכיים) שנבחרו על בסיס החלוקה של שני הקבוצות של נתונים assay. כלל משמש לנתונים נוגדן RB1 P-S780 הוא להגדיר את עמדת השער כחצי הגובה: עמדה מרבי רוחב על הכתף השמאלית של ראשי(מימין) לשיא כאשר בוחן את נתוני תא בקרה השליליים (ללא מיקוד siRNA). הנתונים מודגשים אדום הם תאים עם RB1 P-S780 מופחת ונעדר, שמזוהים עם השער הזה. שער דומה ממוקם על הכתף הנגדית של הפצת ערך היחס משמש לכתב GFP-CDK2. תאי וכתוצאה מכך גבוה היחס תת-האוכלוסייה, שבו חוסר או פעילות CDK2 תכונה מופחתת, מוצגים בצבע ירוק. כדי להמחיש ניתוח ריבוב של שני מבחני איור 6 ג מציג את היישום של שני ערכי השער באמצעות סקריפט Perl 2_gate_classifier.pl להמיר את הנתונים הגולמיים (איור 6 א) לקובץ המבואר להלן. קובץ חדש זה כולל את הנתונים המקוריים לצד טור חדש של תוויות כיתה לכל תא וערכי שער שני משמשים כדי להבדיל ביניהם (במקרה זה של שערים 0.004 לנתונים הנוגדן ו -1.5 לכתב GFP-CDK2 שמשו, בהתאמה) .
לאחר שסיווג את התאים הבודדים from כל תנאי מציאה על הבסיס של שני מבחני ניתן כיום להשתמש בתוויות כיתה אלה כדי לסייע ליאור של חלקות של נתוני assay. איור 7 מראה לפזר חלקות את נתוני תא הבודדים עבור P-S780 RB1 וGFP- מבחני CDK2 מנתוני הדוגמא שנקבעו לכל שלושת תנאי RNAi. מספרי ביאורי הרביעים בscatterplots להראות האחוזים היחסי של כל תת-אוכלוסייה מגודרת לכל שלהקשר מציאה ונוצרים בR באמצעות תוויות הכיתה שתוארו לעיל. חלקות אלה עולים כי, בהשוואה לתאי transfected עם שאינו במיקוד siRNA (איור 7), תאי transfected עם siCDK6 לחשוף הפצת הנתונים נטו עברו שני כלפי מטה על ציר ה- Y (המצביע על העדר זירחון RB1 בסרין 780) וימינה על ציר ה- X (המצביע על פעילות CDK2 נמוכה, איור 7C). שתי משמרות אלו צפויים למציאה של יעד זה. בניגוד לכך, נתונים מאדוניםתאי transfected B1 (איור 7 א) להציג הפסד של מכתים הנוגדן בקנה אחד עם אובדן epitope, אך השפעה קטנה בחלוקת נתונים לכתב CDK2 השוואה לקבוצת ביקורת transfected עם ללא מיקוד siRNA, המצביע על שום השפעה רבה על GFP כתב -CDK2 נובע ממציאת RB1.
כדי לחקור את השימוש בנתוני תאים בודדים, סיווג תת-אוכלוסייה ואיור ריבוב assay 8 נוספים מציג את עלילת הפיזור לנתונים siCDK6 מפרופילי היסטוגרמה איור 7C לזווג צד לעצמת DNA משולב. הזוגות של היסטוגרמות מתייחסים לחצי מנוגדים של האוכלוסייה כולה, מחולקים על הבסיס או עוצמת נוגדנים (מימין של scatterplot) או ערכי GFP-CDK2 כתב יחס (מעל scatterplot). כימות של עוצמת DNA הגרעינית לאוכלוסיות אלו מראה שתי פסגות אופייניות לתוכן DNA 2N ו4N כמו פסגות ימין ועל שמאל, בהתאמה. Intentions של השערים מוצג איורים 6, 7 ו -8 הם כאלה שתאים זוהו כנמוכים לP-S780 RB1 (שכותרתו: P-S780-) או עם ערך יחס גבוה מכתב GFP-CDK2 (שכותרתו: G1) יהיה להיות בשלב G1 של מחזור התא. ואכן, היסטוגרמות פרופיל DNA לאוכלוסיות מזוהות עם אף אחד ממבחנים אלה בעיקר מכילה תאים עם תוכן 2N DNA. פרופילי DNA של האוכלוסייה מגודרת ההפוך (שכותרתו: P-S780 + או ללא G1) מכיל תאים עם הפצות החל 2N ל4N, בקנה אחד עם תאים כאלה אימוץ מגוון של מחזור התא ממקם שלב פוסט-G1.
למרות ההתמקדות כאן כבר הדור והניתוח של נתונים תא יחידים מתמונות צבעוניות fluorescently, זה גם שימושי כדי להיות מסוגל לקחת את הנתונים האלה ולסכם כל assay על בסיס על ידי היטב היטב כדי לפקח על שונות בין משכפל והביצועים של כל הבארות לassay נתון על פני צלחת שלמה של נתונים. איור 9 מציג את הנתונים מכל טיפול siRNA לסכם ערכים הממוצעים מבארות בשלושה עותקים לתאי אחוזים בשערים פנו ל) נתונים RB1 P-S780 ו- B) נתונים כתב GFP-CDK2. הערכים להתוות A ו- B מיוצרים על ידי שני תסריטי פרל נוספים הניתנים בכתב היד הזה; "Antibody_fluorescence_summary.pl 'ו' G1assay_summary.pl ', בהתאמה. תסריטים אלה להשתמש בנתונים הגולמיים שנוצרו על-ידי תא Profiler (Nuclei.csv) ונתוני דו"ח ולi) תאים כולל שנמדדו לכל טוב, ii) מספר התאים בתוך השער, iii) תאי אחוזים בתוך השער וiv) החשבון אומר של הנתונים שנמדדו, גלם שלכן. זה נכלל כאופציה מתאימה למחפש על פני קבוצות גדולות של נתונים assay, לפני התמקדות בנתוני טיפול פרטניים באמצעות הערכת ריבוב של נתוני תא בודדים כפי שמודגם ב איורים 7 ו -8. התרשימים המוצגים כאן עלילה 'iii) תאי אחוזים בתוך השער' עבור שני מבחני, שיתאימו להפצות נתונים שאינם רגילים לראות בנתוני P-S780 RB1 וGFP-CDK2 בהיסטוגרמות באיור 6. תסריטים אלה גם לחשב 'iv) ממוצע אריתמטי של הנתונים שנמדדו, גלם שלגם', שיתאימו ניתוח של נתונים לתגובות אוכלוסייה הומוגנית והפצת נתונים נורמלית לפני ואחרי ההפרעות ניסיוניות.
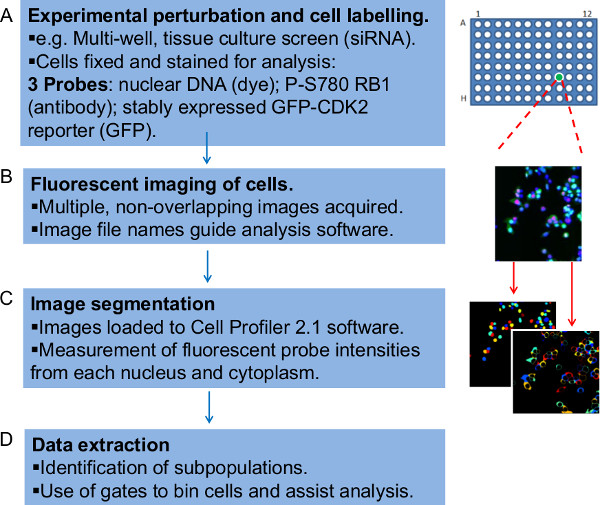
איור 1:. סקירה של השלבים בזרימת העבודה כדי לנתח כמותית נתוני תמונת מיקרוסקופ שכותרתו fluorescently העבודה מיוצגת כאן כארבעה שלבים () ראשית יש צורך להכין את ניסוי תאי הדמיה ניאון.. הדוגמא שתוארה כאן היא זה שלמסך שבו תאים אנושיים חסיד טופל siRNA גידול גדלו במשך 48 שעות, קבועים ומוכתמים על צלחת תרבית רקמת 96-היטב. תנאי RNAi שונים נמצאים בשלושה עותקים בבארות נפרדות בתוך הצלחת. התאים הם מוכתמים צבע DNA, נוגדנים ספציפיים לRB1 phosphorylated על cdk4 ו -6 (RB1 P-S780) סלקטיבית אתר היעד Serine-780 והם גם יציבות להביע GFP-CDK2-כתב, דיווח יציאת מחזור תא G1. באופן קולקטיבי בדיקות ניאון אלה מהווים שני מבחני העריכו בנפרד בתוך זרימת העבודה. (ב) תמונות מיקרוסקופ במקביל עבור כל בדיקה ניאון (ערוץ) נוצרות ושם כגון לכלול פרטים שבו תוכנת ניתוח תמונה יכולה לארגן את הנתונים. (C) קבצי תמונה נטענים לתוך תוכנת Profiler הסלולרי, אשר אלגוריתמי מזהה תאים בודדים והזוגות הקשורים של גרעינים וציטופלסמה לפני מניב מדידות עוצמת במשך שלוש בדיקות הניאון לזהותed בכל. (ד) לבסוף, תסריט פרל משמש לארגן נתונים כמותיים הגלם מיוצר. צעד זה חל שערים לנתונים עוצמת הקרינה לכל תא, ביעילות binning התאים לאוכלוסיות משנה, אשר ניתן להתוות, מעקב וחקירה נגדית. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
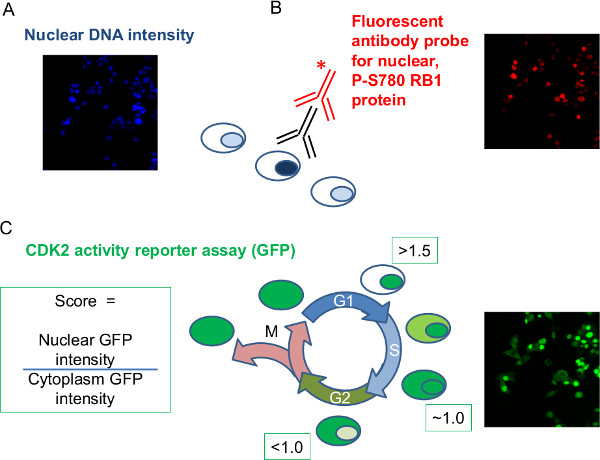
איור 2: נתונים מחקריים שיתקבל על ידי ניתוח תמונת התאים הקבועים, שטופל ב- siRNA, שכותרתו fluorescently מנתוני הדוגמא להגדיר הם צילמו ומדידות עוצמת מקבילה נלקחו לתא.. נתוני תמונת נציג מוצגים לכל פרמטר שנרשם במהלך ניתוח תמונת עוצמה () הגרעיני DNA:. עוצמת הכתמים של צבע DNA הגרעיני משמשת ל תשואת מדד של DNA לגרעין (B) עוצמה גרעינית של phospho-RB1:. ספציפי לRB1 P-S780 באמצעות נוגדן ראשוני (שחור) ומתויג fluorescently נוגדנים משני (אדום) מאפשר מדידת עוצמת זירחון RB1 במכתים-חיסונית . S780 לגרעין (C) כתב GFP-CDK2: התאים המשמשים ביציבות להביע חלבון כתב GFP מתויג שtranslocates בין הגרעין וציטופלסמה בדפוס קבוע עם מחזור התא. מדידה כפולה של עוצמת GFP הגרעינית וcytoplasmic לזווג לכל תא המאפשרת חישוב יחס לכל תא שיכול לשמש כדי להבחין שלב G1 משאר מחזור התא. שלושה יעדי siRNA ישמשו כדי להמחיש את הניתוח; ללא מיקוד siRNA שליטה השלילי; CDK6 siRNA כביקורת חיובית בperturbing זירחון RB1 והתקדמות מחזור התא; RB1 siRNA להקים סגוליות נוגדן.k "> לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.

איור 3: ארגון קבצי התמונה לפני ניתוח תמונת התמונות שצולמו מצלחת תרבית הרקמה נקראות באופן שיטתי על מנת לאפשר לתוכנת ניתוח התמונה להתייחס נתוני תמונה חזרה להקשר הניסיוני המקורי.. מידע זה ממוקם בתוך הקובץ עבור כל תמונה. (א) כפי שכל טוב בצלחת הניסוי עשוי מתאימות למטרות שונות RNAi או טיפולים, חלק צורות כתובת גם של שם הקובץ. מספר המסגרת (B) הוא חלק משם הקובץ . כמו כל גם הוא הדמיה לאסוף מרובה, שאינם חופף מסגרות (C) בדיקות ניאון מכל מסגרת הם צילמו בנפרד; וכתוצאה מכך גם שמות הקבצים צריכים לשקף בי ערוץ כל תמונה מתייחסת ל. (D) שמות קבצים דוגמא, הנוגעים לטוב (G12), Frame (2) עם כל תמונה המייצגת את אחד הערוצים (כחול, אדום, ירוק). קווים מקווקווים לקשר אלמנטי שם הקובץ לייצוגים סכמטי הרלוונטיים לובכן, מסגרת וערוץ, בהתאמה. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.

איור 4:. השימוש בProfiler הנייד למדידה DNA הגרעיני ומכתים נוגדן עם ההגדרות בקובץ הצינור סיפק (3_channels_pipeline.cppipe), ערכי עוצמת הקרינה אמצעי תוכנת ניתוח תמונת Profiler הסלולרי לDNA וגרעיני נוגדן מחייב הנוגע לתאים בודדים . (א) גרעינים שזוהו בתמונה 'הכחולה' הערוץ של DNA המוכתם. (ב) < / Strong> העמדות של גרעיני DNA מוכתמים מתקיימות באופן זמני ב'מסכת גרעינים ". מסכת הגרעינים אז מעולף על גבי (C) תמונות הכחולות ואדומות הערוץ (נתוני DNA וקרינת נוגדן, בהתאמה), וערכי הקרינה ממגזרי תמונה חופפות עם המסכה נרשמות כנגד כל תא מזוהה. זיהוי מוצלח של גרעינים נפרדים, השכנה ניתן להעריך מבחינה ויזואלית במראה של מסכת הגרעינים. לצורך ההמחשה, המוצג בעיגול תמונת מסכה זו, הן דוגמאות בן ההגדרות שנבחרו לאלגוריתם יש לי מזוהה mis גרעינים שכנים גרעין יחיד. שינוי גדרות האלגוריתם כדי למזער את האירועים הללו הוא הציג בסעיף הדיון. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
/ftp_upload/51882/51882fig5highres.jpg "/>
איור 5: שימוש בProfiler הנייד למדידת עוצמות GFP גרעיניות וcytoplasmic כתב CDK2 מתויג GFP translocates בין הגרעין וציטופלסמה ביחס לעמדת מחזור התא של התאים.. בה בעת שProfiler הסלולרי מחשבת את ה- DNA ונוגדני עוצמות גרעיניות לכל תא (איור 4), זה גם מחשבים את הגרעין לציטופלסמה יחס של עוצמות GFP לכל תא. (א) נתונים לצבוע DNA עבור כל תמונה משמש להפקה מסכת גרעינים. Profiler הסלולרי (B) משתמשת בגרעינים להסוות בשיתוף עם תמונת GFP מכתב GFP-CDK2 זרע המיקום של כל תא ולאחר מכן מתרחבת כדי היקפי של כל תא להעריך את כל טביעת הרגל של כל תא. זה הופך להיות "Cell מסכה 'חדשה,. (C) מסכת הגרעינים יורדת ממסכת Cell להניב סדרה כמו סופגנייה-של ציטופלסמה מתאר, שהפכה"מסכת הציטופלסמה '. (ד') מסכת המסכה וציטופלסמה הגרעינים משמשות Profiler הנייד למדידת זוגות של ערכי GFP גרעיניים וcytoplasmic. ערכים לזווג אלה משמשים לאחר מכן על ידי Profiler הסלולרי לחישוב יחסים, שיודיעו על העמדה של כל תא במחזור התא. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
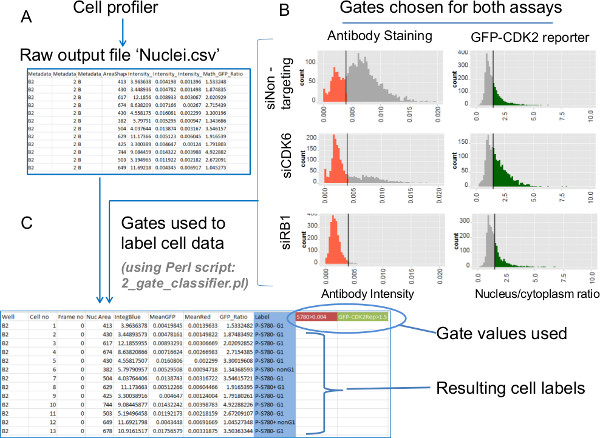
איור 6:. חילוץ נתונים - עיבוד נתונים תא בודדים גלם על ידי הטלת שערים על ערכי assay מגמות ביולוגיות מנתוני התא הבודדים עבור מכתים הנוגדן ומבחני כתב GFP-CDK2 מופק תוך שימוש בנתונים מגודר. היסטוגרמות של נתונים הגולמיים מאפשרת זיהוי של ערכי שער מתאימים. אלו מכן שהוטלו עם תסריט פרל. ()התוצר הסופי של ניתוח קבצי תמונה עם ההגדרות הניתנות לProfiler הסלולרי הם מופרד בפסיקים-ערך (csv) קבצים. קבצים אלה ג ontain נתוני תא בודדים הנוגעים לכל אחד ממגזרי משנה הסלולר השונים. הקובץ 'Nuclei.csv' מכיל את כל המדידות שנבחרו מתייחסות לשימוש במסכת הגרעינים. מדידות אלה כוללים עוצמה גרעינית נוגדן, עוצמת DNA גרעינית ויחס GFP (גרעין / ציטופלסמה). (ב) Histograms של עוצמה גרעינית נוגדן (משמאל) ויחסי כתב GFP-CDK2 (מימין) זמם מנתוני תא בודדים עבור כל מצב מציאה siRNA . הברים על היסטוגרמות המוצגות להראות עמדות שער הרצויות למבחנים אלה. נתונים צבעוניים על היסטוגרמות לציין את תת-אוכלוסיות מגודרות. (C) השערים לשני מבחני שמודגמים בB מוחלים על נתונים הגולמיים באמצעות '2_gate_classifier.pl' פרל סקריפט. התסריטיוצר עותק שונה של קובץ פלט Profiler הסלולרי המקורי (Nuclei.csv) כדי לסייע למזימות שלאחר מכן. שני ערכי השער נרשמים בקובץ החדש (מסומן בצבע כאן) ועמודה 'לייבל' חדשה נוספה. תוויות הסל כל תא לאחת מארבע תת-קבוצות אפשריות המבוסס על שני ערכי assay מגודר לכל תא. תוויות אלה משמשות בחלקות שלאחר מכן איזו תכונת חישובים של התרומות של כל תת-אוכלוסייה, כמו גם ההצלבה של פרמטרים נוספים שנוצרו בProfiler הסלולרי. לחצו כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
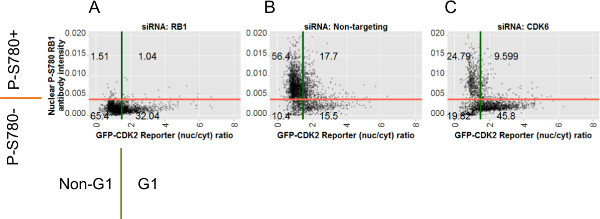
איור 7:. מגרשי פיזור לכל מצב siRNA המתאר את נתוני גלם לתאים בודדים ועמדות שער מפזר חלקות indiviנתוני תא כפולים מכל התמונות לתנאי siRNA מצויינים: siRB1 (); ביקורת שלילית מיקוד siNon (B); (ג) siCDK6. זממו נגד Y-הצירים הם ערכים של הקרינה גרעינית מהכתמת RB1 אנטי-P-S780. זממו נגד X-הצירים הם ערכי היחס המקביל מחושבים מכתב GFP-CDK2. הברים האדומים וירוקים מציינים את עמדותיהם של השערים לשער RB1 P-S780 והשערים כתב GFP-CDK2, בהתאמה. שני השערים לחלק את התאים לארבע תת-אוכלוסיות והמספרים על הרביעים וכתוצאה מכך הם מספר אחוזים מתאים מכל אחד מאלה. הסברים סביב הצירים למצביעים ארבעה תווית-אלמנטים אפשריים יחולו על כל תא על ידי תסריט פרל 2_gate_classifier.pl. תוויות אלו הוצגו ביחס לשער assay שלהם ונמצאות בשימוש בR-סקריפט (analysis.r) כדי ליצור את המגרשים באיורים 6, 7 ו 8. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
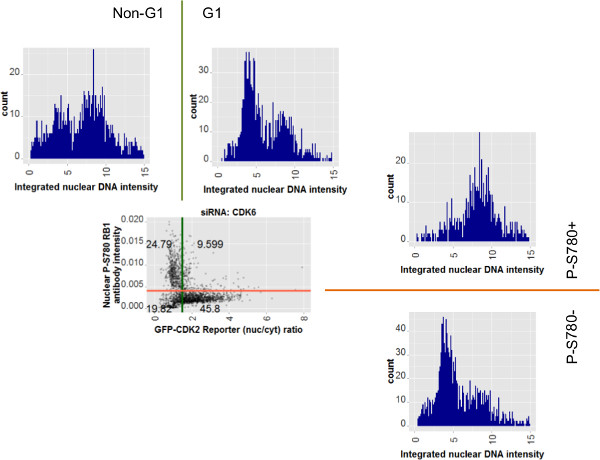
איור 8: תת-אוכלוסיות תאים שהוגדרו על ידי שני מבחני מעבר G1 להראות פרופילי 2N ו4N DNA בקנה אחד עם תוצאות assay עלילת הפיזור של נתונים לתאי siCDK6 חוזרת על עצמו מהאיור 7C.. המקיף את עלילת הפיזור היא היסטוגרמות לעצמת DNA הגרעיני משולבת המתייחסת לתת קבוצות של האוכלוסייה. אלה מעל scatterplot מתייחסים לassay כתב GFP-CDK2. אלה בצד הימין של scatterplot מתייחסים למדידות נוגדן phospho-RB1 הגרעיני לבד. קווי השער הצבעוניים הרחיבו להראות הקשר שלהם להיסטוגרמות. תוויות השער שבו את נתוני התא נבחרו לחלקות נוספות אלה מוצגות גם. תאים עם הפסד של RB1 phosphorylated על סרין 780 (P-S780-) או שהם בגרעין כתב גבוה GFP-CDK2 יחס cytoplasmic (המצביעים על פעילות CDK2 נמוכה) מראים פרופילי DNA כמו-2N בעיקר, ואילו עמיתיהם הפוכים שלהם עבור כל assay המתאים להראות הפצה של 2N ו4N, אופייני לאוכלוסייה מעורבת, לאחר G1-שלב של תאים. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
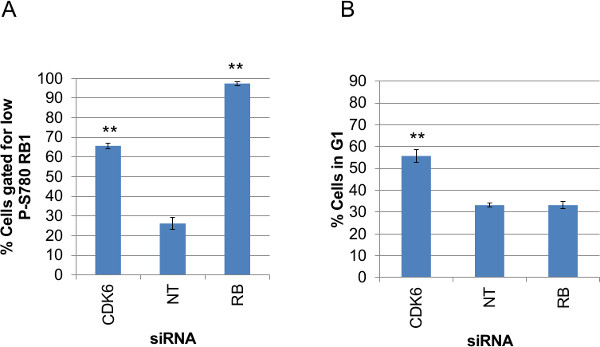
איור 9: חלקות סיכום של ערכי assay מגודר עבור כל siRNA מצב חלקות נתוני סיכום של (A) נתונים מגודר P-S780 RB1 ונתונים (B) GFP-CDK2 מבארות בשלושה עותקים עבור כל תנאי מציאה siRNA.. ערכים חושבו מתפוקת Profiler הסלולרי הגלם (Nuclei.csv) באמצעותסקריפטים Perl, 'antibody_fluorescence_summary.pl' () או 'G1assay_summary.pl' (B). הערכים זממו הם אמצעי תאי אחוזים בתוך השער יחול על כל assay. ברים לציין סטיות התקן מחושבים מבארות בשלושה עותקים. ערכים מזווגים, homoscedastic T-Test P עבור כל תנאי מציאה בהשוואה ללא-מיקוד siRNA מוצגים מעל הנתונים זממו בי P <0.001 (**) וP <0.05 (*). לחצו כאן כדי לצפות בגרסה גדולה יותר של זה דמות.
איור S1. הגדרת תוכנת Profiler הסלולרי לניתוח תמונה. (א) צילום המסך של Profiler הנייד לפני כל הגדרות ניתוח תמונה מוזנות. (ב) צילום מסך של Profiler הסלולרי לאחר שהעמיסו את פרטי האלגוריתם כלולים ב'3_channels_pipeline.cppipe '. הגבוה כרטיסייה מוארת בפינה השמאלית העליונה של מסך מצביעה על כך שזה מראה את הפרמטרים לבמה 'LoadImages' של הניתוח. לחיצה על החלקים האחרים של הרשימה שלהלן זה יהיה לחשוף את הפרטים לשלבים הבאים בניתוח. (C) צילום מסך של סלולארי Profiler עם פרטים לתיקיית קלט ופלט התיקייה נכנסה. (ד) צילום מסך של Profiler Cell אחרי 'לנתח הכפתור 'תמונות כבר לחץ להתחיל ניתוח. גבי שלושה חלונות חדשים הממחישים את המסכות אלגוריתמית בייצור שנוצרו על ידי התוכנה מהתמונות בניתוח. חלונות אלה ניתן לגשת על ידי לחיצה על הסמלים "עין" למצב הפתוח ליד המדרגות הרלוונטיות בניתוח, בפינה השמאלית העליונה של חלון Profiler הסלולרי העיקרי. נופים אלה מסייעים למשתמש לבדוק אם הגדרות יצירת מסכות קונפטי בצבע מסכימים עם המידע הנלווה, המקורי, בגווני אפור.
אף אוזן גרון "> איור S2. השימוש בפרל וRStudio לנתוני תא בודד שער ואת העלילה תת-האוכלוסיות של תאים וכתוצאה מכך. () הפנל הימני מראה את התיקייה שנבחרה לקבל קבצי ה- csv הפלט (סמלים ירוקים) מניתוח Profiler הסלולרי. סקריפטים Perl מסופקים עם כתב היד (סמלים כחולים) מועתקים לתיקייה זו. מודגש הוא תסריט פרל '2_gate_classifier.pl', אשר כבר עם העכבר כדי לייצר את תיבת הדו-שיח בפנל השמאלי-לחץ כפולה. מוצגים הם הנחיות ותשובות המתאימות הקלדת צורך השער את נתוני תא הבודדים מהקובץ 'Nuclei.csv'. (ב) צילום מסך של RStudio מייד לאחר טעינת התסריט 'analysis.R'. מודגש הם פקודות כדי להעלות את הנתונים מגודרות ל התוכנה לפני קשירת קשר (פרטים בפתק קווים 5 ו -6 תצטרך להיות מותאמת על פי שבו נתונים מגודרות נמצאים על המחשובr המשמש לניתוח). (C) צילום המסך של RStudio פעם נתונים הועלה. (ד) צילום מסך של הקרנת RStudio הדגיש את הבלוק של קוד הנדרש כדי לייצר העלילה שמוצגת בחלון הימני התחתון. קודים לכל חלקה מופרדים על ידי שורות ריקות ומקובצים לפי סוג של עלילה.| יעד siRNA | כתובות גם צלחת |
| ללא מיקוד (NT) | E5, F5, G5 |
| רטינובלסטומה (RB) | E7, F7, G7 |
| קינאז התלוי cyclin 6 (CDK6) | B2, C2, D2 |
טבלת 1: ובכן כתובות ותנאי siRNA המתאימים בשימוש בנתונים הדוגמא להגדיר.
Discussion
העבודה המתוארת מהווה הליך perturbance multiwell של תאים באמצעות siRNA, איתור סמן לאחר מכן ולבסוף להשתמש בשורה של צעדים-נתמך תוכנה כדי להקל על חילוץ של נתונים כמותיים מתמונות מיקרוסקופ פלואורסצנטי וכתוצאה מכך. הגישה מתמקדת באספקת ערך עוצמת גרעינית וcytoplasmic לתאים בודדים, שבה יש יישום מעשי נרחב ביישומים מבוססי תאים רבים. נתוני הדוגמא משמשים כאן נוצרו בהגדרת מסך siRNA שבו שני מבחני ניאון למעבר מחזור תא שלב G1 נבדקים וקורלציה חזרה למדד biophysical ישיר יותר של תוכן DNA הגרעיני.
השימוש בכתם DNA ניאון כדי DNA הגרעיני תמונה הוא צעד הכרחי בתהליך פילוח תמונה כפי שהיא מאפשרת זיהוי של תאים בודדים ו'מסכת הגרעינים "וכתוצאה מכך משמשת כנקודת ההתחלה לזהות cytoplasmi המקבילאזורי ג. כתב CDK2 מתויג GFP, אשר באו לידי ביטוי ביציבות בתאים, מעניק משתנה עדיין גבוה באופן עקבי מאשר אות רקע בציטופלסמה שבאמצעותו ניתן התווה תא זה. אותו צינור הניתוח צריך להיות ישים לניתוח של אירועי טרנסלוקציה החלבון באמצעות כתבים אחרים מתאימים צמוד הקרינה ותגובתם לperturbance. כמו כן, החלפת כתב GFP-CDK2 עם צבעי ניאון ציטופלסמה הספציפית תאפשר השימוש האלטרנטיבי של אלגוריתם זה כדי למדוד את הממדים של ציטופלסמה וגדלים היחסי של תאים בתמונות.
שיקול נוסף בעיצוב אסטרטגית פילוח התמונה המתוארת כאן הוא השימוש בProfiler הסלולרי כדי לספק ערכי עוצמה משולבים לכימות DNA. שילוב של עוצמת ערכים עבור DNA הגרעיני נתונים צביעה מאפשרים לשינויים אפשריים בגודל גרעין, ומייצג התאמה קרובה לפרופילי הכימות ראולpropidium יודיד מוכתם FACS נתונים. עם זאת, עוצמת משולבת לא יכולה לספק אמצעים מתאימים כדי להעריך את תפקוד חלבון שבו ריכוז ממוצע, שהודגם על ידי עוצמת הקרינה ממוצעת של אנטיגן, הוא יותר מבחינה ביולוגית רלוונטי מהסכום הכולל המשולב של חלבון (וקרינה קשורה) בתוך תא תא. לכן אומר ערכי עוצמה שמשו לP-S780 RB1 ונתונים GFP. האפשרות לשנות בין שני המצבים (כלומר או משולב) של הערכת נתונים נמצא בלוח 'ExportToSpreadsheet' של תוכנת Profiler הסלולרי.
הגדרות הניתוח בקובץ 3_channels_pipeline.cppipe מותאמות לתמונות בערכת נתוני דוגמא. ניתוח של ערכות תמונות חדשות עם פרוטוקול זה יחייב את שמות הקבצים לאמץ את המוסכמה למתן שמות שתוארה לעיל (איור 3). כמו כן, רגישות ערכים כדי להתאים את הבהירות של הכתמה גרעינית DNA ומפתנים לרקעעוצמות בסטי התמונה החדשות ייתכן שתצטרכנה להיות מותאמות בתוך הגדרות Profiler הסלולרי. בהתחשב בתפקיד המרכזי צביעת DNA מחזיק לבניית מסכות פילוח תמונה השונות, היישום של הגדרות רגישות נכונות לערוץ זה הוא מפתח לניתוח המוצלח של נתוני תמונה חדשים עם תוכנת Profiler הסלולרי. הגדרות Profiler הסלולרי סיפק קובץ (3_channels_pipeline.cppipe) מכיל הערות על הפרמטרים הכי שימושי לעתים קרובות להתאמת הניתוח לנתונים חדשים. הערות אלו הן בתיבת הטקסט בחלק העליון של המסך בחלון הראשי של תא Profiler ותכלולנה הדרכה על שינוי הגדרות הרגישות והתאמת מספר הערוצים להיות מנותח. כנגדו כתב אישום בסעיף 2.8 לפרוטוקול, כדי לבדוק את הגדרות לנתוני תמונה חדשות זה עשוי להיות נחוץ כדי לבחון את פילוח התמונה במהלך ניתוח תמונה על ידי לחיצה פתוחה הסמלים 'עין' לכל אחת מ'זיהוי אובייקטים ... "צעדי פרוטוקול (איור S1D ). בפרט, להדמיה של נתוני תמונה באמצעות 'IdentifyPrimaryOjects "תציג אם מסכת הגרעינים מזוהה בצורה נכונה מתמונות של צביעת DNA. על Profiler Cell דף התוכנה למודול 'IdentifyPrimaryOjects' הוא גורם תיקון סף. התאמת ניסוי והטעייה של ערך זה תהיה לתקן את רוב השגיאות בזיהוי גרעיניות. הערכים לאזן את ערוץ DNA נגד עוצמת הרקע לכל תמונה. ערכי גורם תיקון סף הציר סביב 1, שבו גדול יותר מזה הוא יותר מחמיר (טוב לתמונות ברורות) ופחות מ -1 מקל (מתאימה לתמונות עם פחות ניגוד בין מכתים ורקע).
תפוקת הגלם של נתוני תא בודדים מProfiler Cell ניתן לנתח במובנים שונים כדי להתאים לצרכימים של מחקרים אחרים. מוצג כאן הוא השימוש בתסריט פרל ליישם שערים לשניים מהפרמטרים שנמדדו לכל תא במטרה לסייע לחילוץ מגמות ביולוגיות מהנתוניםהיתר ד הצלבה של תת-האוכלוסיות המזוהות עם מדידות נוספות. למרות שזה אפשרי במידה שווה לכלול אלמנטים של gating במסגרת Profiler הסלולרי, התוואי החלופי המשמש כאן מספק גמישות ומהירות גדולות יותר, במיוחד אם ערכות נתונים גדולות צריכים להיות מוערכות. השלב האיטי ביותר בשלבי רכישת פוסט דימוי של הפרוטוקול הנוכחי הוא הריצה של תוכנת Profiler הסלולרי. מאבחן תא כאן מנוהל ללא הטלת שערים לייצר סט נתונים גולמי מגודרת un אשר ניתן reanalyzed עם תסריט פרל שלאחר מכן במהירות רבה יותר ובמידת צורך, איטרטיבי עם ערכי שער שונים. לא כל המחקרים תדעו מראש את ערכי השער המתאימים כמו זה עשויים להשתנות עם חומרים כימיים על כל קבוצת נתונה של נתונים, ובאופן פוטנציאלי לאורך זמן. זהו, אם כן, מומלץ ליצור היסטוגרמות המתארת את התפלגות הנתונים הגולמית המתקבלת מהתא Profiler עבור פקדים חיוביים ותאי דגם מוטרדים על מנת לזהות valu שער המתאיםes לפרמטרים של עניין.
סקריפטים Perl נכתבים לקבל מבנה טור מוגדר בנוקשות של נתונים מProfiler הסלולרי ועלולים להפסיק לעבוד אם משתמש משנה את מספר פלט פרמטרים על ידי Profiler הסלולרי באמצעות ההגדרות 'ExportToSpreadsheet'. כדי לעזור ליישם שינוי של הגדרות הערות כלולות בתוך קבצי תסריט פרל. כדי לראות אלה להציג את התסריט בעורך טקסט, רצוי עורך הטקסט של מתכנת מוגדר אלמנטי פרל קוד צבע (למשל, http://www.activestate.com/komodo-edit). הערות אלו מציינות היכן להתאים את התסריט להסתגל לשינויים בנתונים בפורמט. בדומה לתסריטי Perl, קובץ R-הקוד שסופק (analysis.r), המכיל את ההוראות לקשירת קשר הדמויות מנתונים ניתוח התמונה, ניתן לקרוא בעורך טקסט או תוכנת RStudio לראות הערות נוספות על שימוש והסתגלות. ניתן להוסיף הערות אלו עם פרטים על ביטויים רגילים ופרל 12 והחבילה ggplot2 13 לR, אשר שניהם מהווים את הבסיס לאופן שבו נתונים הוא לקריאה, מבואר וזממו, בהתאמה.
מחקרים חדשים באמצעות מיקרוסקופ פלואורסצנטי, כמו גם הנתונים גולמיים שהופקדו עם פרסומי קוד פתוחים נוחות לשיטות ניתוח כגון אלה שתוארו כאן. טבעו של נתונים גבוה תוכן משאיל את עצמו לניתוח רקורסיבית עם דגשים אנליטית שונים בהתאם לתחומי המחקר של כל צופה נתון. למרות השאלות שיכולים להישאל של נתונים מוגבלים על ידי הבדיקות המשמשות במקור, נתוני תמונה יכולים להיות לעתים קרובות reanalyzed משמעותי מעבר להיקף של המחקרים שחוללו אותם.
Disclosures
יש לי המחברים אין לחשוף
Acknowledgements
עבודה זו נתמכה על ידי מענקי CRUK 15,043 וCRUK 14251.
אנו מודים דניאל Wetterskog וקה Kei הו לקבלת סיוע טכני וקריאה ביקורתית של כתב היד.
Materials
| Name | Company | Catalog Number | Comments |
| AllStars negative control siRNA | Qiagen | 1027280 | Negative control siRNA. |
| CDK6 siRNA | Dharmacon/ Custom synthesis | NA | Antisense sequence: 5' CUCUAGGCCAGUCUUCUUCUU |
| RB1 siRNA | Dharmacon/ Custom synthesis | NA | Antisense sequence: 5' GGUUCAACUACGCGUGUAATT |
| 5x siRNA buffer | Thermo Scientific | B-002000-UB-100 | To be diluted with nuclease-free, non-DEPC treated water. |
| Nulcease-free water (non-DEPC) | Applied Biosystems | AM9937 | For dilution of siRNA buffer. |
| Hiperfect | Qiagen | 301705 | Transfection lipid. |
| DMEM | Life Technologies | 41966052 | Pyruvate and high-glucose supplemented tissue culture media. |
| 96 well tissue culture plate | Falcon | 3072 | Plain, 96 well tissue culture plate for the parallel, compartmentalized mixing of transfection complexes. |
| Packard Viewplate | Perkin Elmer LAS | 6005182 | 96 well TC plate with optical base on which cells are transfected, grown, fixed and eventually imaged. Supplied with opaque, adhesive plate seals, which are used during storage of used plates. |
| Breathable membrane | Alpha Labs | LW2783 | Sterile, gas-permeable, adhesive membrane. |
| Neutral buffered formalin, 10% | Sigma-Aldrich | HT5012-1CS | 10% Formalin (4% formaldehyde) fixative used neat in protocol. |
| Triton X-100 | Sigma-Aldrich | X100PC | Non-ionic detergent |
| [header] | |||
| Tris | Sigma-Aldrich | T1503 | Trizma base (tris(hydroxymethyl)aminomethane) |
| Tween 20 | Sigma-Aldrich | P2287 | For plate wash solution. |
| Anti-P-S780 RB1 antibody | Abcam | ab32513 | Rabbit monoclonal |
| AlexaFluor647 Anti-rabbit | Invitrogen | A21245 | Highly cross-adsorbed, fluorescently labelled secondary antibody. |
| Hoechst 33342 (Bisbenzimide) | Sigma-Aldrich | B2261 | Fluorescent, chromatin-intercalating, DNA dye. |
| Permeabilization solution | NA | NA | 0.1% Triton X-100 in 50 mM Tris-buffered saline, pH 8.0. |
| Plate wash solution | NA | NA | Tris-buffered saline containing 0.1% Tween-20. |
| Block solution | NA | NA | 5% powdered milk in Tris-buffered saline and 0.1% Tween-20. For blocking plate prior to immuno-staining and dilution of antibodies. |
| Cell Profiler 2.1 | Broad Institute | http://www.cellprofiler.org/download.shtml | |
| Active Perl Community Edition | ActiveState | http://www.activestate.com/activeperl/downloads | |
| R programming environment | The R Foundation | http://www.r-project.org | |
| Rstudio | Rstudio | http://www.rstudio.com/ | |
References
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), 100 (2006).
- Khan, A., Eldaly, H., Rajpoot, N. A gamma-gaussian mixture model for detection of mitotic cells in breast cancer histopathology images. J Pathol Inform. 4, 11 (2013).
- Selzer, P., Beibel, M., Gubler, H., Parker, C. N., Gabriel, D. Comparison of multivariate data analysis strategies for high-content screening. J Biomol Screen. 16 (3), 338-347 (2011).
- Lyman, S. K., et al. High content, high-throughput analysis of cell cycle perturbations induced by the HSP90 inhibitor XL888. PLoS One. 6 (3), e17692 (2011).
- Richardson, E., Stockwell, S. R., Li, H., Aherne, W., Cuomo, M. E., Mittnacht, S. Mechanism-based screen establishes signalling framework for DNA damage-associated G1 checkpoint response. PLoS One. 7 (2), e17692 (2012).
- Heynen-Genel, S., Pache, L., Chanda, S. K., Rosen, J. Functional genomic and high-content screening for target discovery and deconvolution. Expert Opin Drug Discov. 7 (10), 955-968 (2012).
- Krausz, E. High-content siRNA screening. Mol Biosyst. 3 (4), 232-240 (2007).
- Gu, J., Xia, X., et al. Cell Cycle-dependent Regulation of a Human DNA Helicase That Localizes in. DNA Damage Foci. Mol Biol Cell. 15 (7), 3320-3332 (2004).
- Mittnacht, S. Control of pRB phosphorylation. Curr Opin Genet Dev. 8 (1), 21-27 (1998).
- Mittnacht, S. The retinoblastoma protein--from bench to bedside. Eur J Cell Biol. 84 (2-3), 97-107 (2005).
- Nybo, K. GFP imaging in fixed cells. BioTechniques. 52 (6), 359-360 (2012).
- Schwartz, R. L., Foy, B. D., Phoenix, T. . Learning Perl - Making Easy Things Easy and Hard Things Possible. , 1-363 (2011).
- Wickham, H. . ggplot2: Elegant Graphics for Data Analysis. , 978-970 (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved