Method Article
高コンテンツ、オープンソースソフトウェアでサポートされているユニバーサル顕微鏡データからの蛍光マーカー、個々の細胞定量のワークフロー
要約
蛍光標識された細胞の多重化された画像ベースの分析を可能に柔軟インフォマティクスワークフローがある発表。ワークフローは、核と細胞質のマーカーを定量化し、これらの区画の間のマーカー座を計算します。手順は、96ウェルフォーマットで、間接免疫蛍光法によってマーカーを検出するためのsiRNAで信頼性の高い方法論を用いた細胞の摂動のために提供される。
要約
接着性の哺乳動物組織培養モデルにおける細胞の挙動を支配する制御機構を理解することにおける進歩は、単一細胞分析のモードにますます依存するようになってきている。細胞集団からのバイオマーカーの平均値を反映して、複合データを配信する方法は研究され、生物学的システムの不均一性を反映した亜集団のダイナミクスを失う危険性があります。これを踏まえて、従来の手法により置き換えられている、またはハイコンテント顕微鏡による評価を可能にするために開発された細胞アッセイの、より洗練された形でサポート。これらのアッセイは、潜在的にプロプライエタリソフトウェアパッケージに付属することで使用可能な蛍光バイオマーカーの大量の画像を生成、細胞あたりのマルチパラメトリック測定が可能になります。しかしながら、比較的高い資本コストと、これらのデバイスの多くのoverspecializationは多くの研究者に彼らのアクセス可能性を妨げてきた。
ここに記述されているほとんどの蛍光顕微鏡からの画像との使用に適した個々の細胞の特定の細胞内領域からの複数の蛍光標識強度の定量化のために普遍的に適用可能なワークフロー。このワークフローの鍵は、これらのイメージ内の個々の細胞を区別定義された細胞内領域に分割して、これらの領域に固有の蛍光マーカー強度値を提供するために自由に利用可能な細胞プロファイラソフトウェア1の実装です。画像データから個々のセルの強度値の抽出は、このワークフローの主な目的であり、接着性ヒト細胞におけるG1チェックポイントレギュレータのsiRNAスクリーニングからの制御データを解析して説明する。しかし、ここに提示ワークフローは、細胞摂動の他の手段からのデータの分析に適用することができる( 例えば 、化合物スクリーニング)このようにして、蛍光ベースの細胞マーカーの他の形態とは、研究室の広い範囲のために有用であるべきである。
概要
ここで紹介する作品は、個々の細胞と定義された細胞内領域を同定する接着細胞の蛍光顕微鏡画像のアルゴリズム誘導内訳を実行するために自由に利用できるソフトウェアセルプロファイラの使用が記載されている。このアプローチは、画像セグメンテーションと呼ばれる(セグメント化オブジェクトと呼ぶ)は、各細胞または細胞内領域に局在蛍光標識マーカーを定量することによって画像化された細胞のその後のマルチパラメトリック分析が可能になる。このワークフローは、高含有量の分析を可能にするための基礎を構成し、さらに開発され、マルチパラメータに合わせて変更する、個々の細胞は特殊な高コンテンツ器具または独自のソフトウェアにアクセスすることなく、実験室で分析することができるツールとして役立つことを意図している。本稿に付属ファイルについて分析を生成するために、関連する原画像データ、アルゴリズム設定支持スクリプトのテストセットを含む。提供アルゴリズム設定fはまたは細胞プロファイラは、他の研究からの画像データの利用を可能にするために必要であるかもしれないものの調整例のデータセットとディスカッションセクションの詳細のために最適化される。
定量的データは、Cellプロファイラを使用して抽出された後、別の研究室は、生データ内の個々のセルの値によって提示された情報を使用する方法のための異なる要件を有していてもよい。ここに示されているゲートは、各アッセイのために生データに適用されることにより、一つのアプローチである。これらのゲートを使用して、データがゲートによって定義された応答を受けた細胞の亜集団と異なる処理を結ぶ傾向の可視化を可能にする、応答のバイナリ条件に変換される。ゲートは、それぞれの関連測定のための適切な陰性および陽性対照について得られたデータ分布の観察に基づいて設定されている。ゲートの使用は、生、細胞ベースの測定を管理する方法のほんの一例である。また、ここに示した核DNA強度の使用である私にゲートされたデータと組み合わせて、連続した範囲の値としての生の形でasurements。画像解析データを管理するための他のアプローチは、調査の性質に応じて、考慮されるべきである。部分集団に細胞を割り当てるためのゲートを用いて統計的選択肢が2報告されており、パラメータの多数にわたって高いコンテンツデータを要約するための戦略の系統的比較は3に報告されている。
6 -画像データの高含有量分析は、薬物応答の細胞研究における用途が見出されている、遺伝学および4シグナリング環境ストレスを逆転。高含有量の分析の利点は、蛍光顕微鏡データのアルゴリズム的解析は、定量的および空間的なパラメータは、個々のセル7で同時に考慮されることを可能にするという事実から生じる。このように、複数のアッセイのための細胞の結果は、アッセイdの相互参照、差動動作することができefined細胞亜集団は、形態学的な変数を考慮を含むことができ、実験条件およびアッセイ内で追跡することができる。戦略、ここで説明する解析ワークフローは、他の高含量のアプローチのように、個々のセルへの相互参照された多重化データを配信することが可能である。蛍光顕微鏡画像を生成する画像の数千までの低スループットは従来の蛍光ベースの顕微鏡検査で生成された画像の数の範囲のデータの分析に適用可能な高含量の方法スーツ研究自動化ハイコンテンツスクリーニングプラットフォームを用いて製造。
ワークフローは、別のアッセイは、核蛍光マーカー強度またはそれぞれの蛍光レポータータンパク質の細胞質/核移行、のいずれかの点で測定されるデータ例をここに図示されている。これらのアッセイは、EACに応じて、別々にまたは組み合わせて考慮することができるという点で、ワークフローが柔軟であるhは異なる研究者によって研究課題を与えられた。例えばデータは、RNA干渉(RNAi)実験( 図1)の一部として製造される。低分子干渉RNAオリゴヌクレオチド(siRNA)は、サイクリン依存性キナーゼ(CDK)の活性の二つの蛍光レポーターのための変化をもたらすHCT116ヒト結腸直腸癌細胞中の特定のタンパク質をノックダウンするために使用される。セリン780(P-S780のRB1)における核網膜芽細胞腫タンパク質のCDK6依存性リン酸化は、抗体染色によって評価される。同じ細胞では、CDK2活性(GFP-CDK2レポーター)の緑色蛍光タンパク質タグ付きレポーターはCDK2活性の非存在下でレポーターは細胞質に核及びCDK2活性シャトル上に存在する細胞質比にその核によって評価される8。加えて、各細胞の核DNAは細胞を同定し、同様に画像内の核の境界線を定義するための手段として機能するDNAインターカレート色素、ビスベンズイミドを用いて染色されDNAの存在量のsaの尺度は、( 図2)細胞の細胞周期位置に関する情報を提供する。
CDK6およびCDK2の活性は、細胞周期5のG1からS期への細胞の通過として検出可能であるような、個々の細胞内の2つのレポーターの間の緊密な一致が予想されるように、互いに9,10成功し。ここで使用されるデモデータセットを例として分析siRNAの効果は、CDK6、網膜芽細胞腫タンパク質(RB1)及び非標的化ネガティブコントロール( 表1)を標的とする。 CDK6のノックダウンは、P-S780のRB1エピトープの減少および細胞周期のG1期における細胞の蓄積の両方を引き出すべきである。 RB1のノックダウンは、リン酸化S780抗体の特異性のための試薬対照として役立つ。ホルマリン固定された11からの蛍光顕微鏡画像は、蛍光染色されたHCT116組織培養細胞は、アルゴリズムの画像解析のために使用される。得られた数値データは、その後に使用されるクロスリファレンス記者や、異なるノックダウン状態の影響を評価。
このタイプの分析によって生成されたデータの潜在的な大きさは通常の解析ツールへの課題を提示することができます。例えば、個々のセルデータは、いくつかの表計算ソフトを収容するよりも大きくすることができる。大きなデータセットの分析を支援するためのデータの単純な、高度に反復的な、教師の処理を行うのPerlスクリプトが含まれる。 Perlスクリプトは、特定のファイルの命名規則( 図3)で画像ファイルを処理する際に細胞プロファイラ、によって生成される出力ファイルのために特別に書かれた、と分析に使用するウェル当たりのフィールド変数番号を可能にしている。これは、細胞亜集団5の動向を追跡するためにゲート個々の細胞アッセイデータに頻繁に重要であり、ここに示されている各アッセイタイプについて、所定の設定ゲートに基づいて、各セルは、フラグにPerlスクリプトを使用することである。また、オプションのPerlスクリプトも含まれていますその実現、個々のウェル(または条件)のためのデータの結果を要約:セットのゲート内の細胞の割合と生の検定スコアの平均値を。応答はすべて、またはウェル内の細胞の大部分に影響を与える場所データを表示するの後者の、より均質な方法は、有効である。上述のように、そのような評価は、応答が、集団内の細胞のサブセットに限定される個々のセルデータゲーティングによってもたらされるものよりもあまり有用である。
記載されたワークフローの有用性は、siRNA又は説明マーカーアッセイによって摂動に限定されるものではない。研究は、siRNAの組み合わせ、化学阻害剤および放射線治療を用いて組織培養実験およびCDK6以外のマーカーの評価及びCDK2活性5に対する応答をアッセイするためにこのアプローチを使用している。
概念的には、実験的な戦略は、生物学的に有用な細胞内領域が多様に自動的に登録されることを可能にする蛍光顕微鏡画像中に存在する個々の細胞である。このように、このアプローチは、集団ではなく、個々のセルに焦点技術によって見逃され得る生物学的情報を明らかにし、定量的、多重化されたデータを得ることができる。軽微な変更で、説明されたアプローチおよび分析ワークフローは、任意の蛍光ベースのアッセイ出力および細胞生物学的応答、DNA含有量の定量的評価、核または細胞質の蛍光またはこれらの間のマーカーの往復の定量のための定量的な、個々のセルのデータを得ることができる二つの区画個別にまたは多重化方式では重要である。パブリッシングの要件はますます公然とアクセス可能な生データの提出に向けて傾向があるように、このようなここに記載されているような顕微鏡画像解析のためにアクセスし、無料ツールに精通も公開されたデータを再分析するために探してラボへの直接対象としています。
プロトコル
応答マーカーの1。実験摂動と細胞標識(リバーストランスフェクションのsiRNA画面)
- 無菌の組織培養フードピペットで無菌の、無地96ウェルプレートのウェル中に1×siRNAの緩衝液中で200nMのsiRNAを70μlの。無血清DMEM培地の40ボリュームにトランスフェクション脂質を希釈し、各ウェルsiRNAを含むに105μlずつ分注する。
注:無血清DMEMの10.5ミリリットルに262.5μlの脂質を希釈ウェル当たり脂質の2.6μLを提供し、siRNAの全体の96ウェルプレートに適しマスターミックスを生み出す。この段階で200 nMのsiRNAを開始濃度の使用は、ステップ1.3で20nmの作業濃度をお届けしますが、手順はそれに応じて調整開始濃度( すなわち 、50 nMで)で、5 nMのダウン濃度の作業のために動作します。これらはオンターゲット応答の大きさを減少させることができるが、より低い使用濃度は、オンタージの増加につながる、オフターゲット偽陽性のスコアを減少させることができる偽陰性率をトン。 - 室温で10分間穏やかに振動板を混ぜる。 350μlの中に生じた175μlのサブ分割する透明基材との不透明な、組織培養処理、96ウェルプレート上にターゲットごとに複製します。
- 50μlの脂質-siRNA複合体上に直接10%の血清を含有する150μlのDMEM中にウェルあたり8000細胞を分配することによってリバーストランスフェクション。使用HCT116ヒト結腸直腸細胞が安定的にCDK2活性5,8を報告GFPタグ付きマーカーを発現する。これ以上の混合は必要ありません。湿度を制御し、プレート「エッジ効果」を防止し、48時間37℃、5%CO 2の加湿インキュベーターにプレートを配置し、滅菌、接着剤通気性の膜でプレートを密封する。
- メディアの小さな残留量がウェル内に残っている媒体は、吸引除去する。各ウェルに、4%緩衝ホルムアルデヒド100μlを加えることにより細胞を固定し、部屋トンで10分間ドラフト内でインキュベートemperature。
- プレートを吸引することにより、定着液を除去します。この時点で、週間まで4℃で暗所に100μlのPBS下で、100μlのリン酸緩衝生理食塩水(PBS)でプレートを3回洗浄した後、ストア密封することによって、実験を停止か、を続行細胞の透過化。
注:私たちは、固定後できるだけ早く処理プレートを推奨し、一般的には完全に処理プレートのストレージを好む。チメロサール、アジ化ナトリウム、または商業的代替物として殺生防腐剤はmicoroganismal成長を防止するために添加することができる。ホスファターゼ阻害剤の添加は、リン酸化エピトープを維持するために役立ち、タンパク質修飾の状態を維持する他の手段は、当該アッセイの文脈において有用であり得る - プレートからPBSを除去し、透過処理溶液100μlを添加して細胞を透過性。振盪せずに室温で10分間インキュベートする。マルチチャンネルを使用して透過処理液を吸引annelピペット。このステップを3回繰り返します。
- 室温で30分間、ウェルあたり100μlのブロック溶液を添加して細胞をブロックする。抗P-S780 RB1抗体を50μlのプローブ次に、プレートを吸引し、ブロック溶液を除去し、室温で暗所で2時間ブロック溶液中で500倍に希釈した。
- 5分間、プレート上の各時間溶液を残し、100μlのプレート洗浄溶液でプレートを3回洗浄する。プローブは、50μlの蛍光標識二次抗体と4℃で一晩暗所でのプレートは、クロマチン特異的DNA染料ビスベンズイミドの2μMで補充ブロック溶液中で1000倍に希釈した。 3回以前のように、店舗のプレートを洗浄し、4℃で暗所に100μlのPBSの下で、密封された。画像2週間以内のプレート。
2.画像撮影·画像セグメンテーション
- 個別の16を取るために20Xの目的で、共焦点または回転ディスク蛍光顕微鏡を使用して、ビットの、DNA色素、GFPおよび免疫染色の蛍光団に対応する3つのチャンネルでグレースケールTIFFイメージ。多くの非オーバーラップ画像セットをキャプチャし、画像にフレームを、ウェルあたり約1,000〜2,000細胞とここで言う。
- 各ファイル名は「実験名'、'よくアドレス」、の順で「フレーム番号」と「チャネル識別子」、( 図3)のユニークな組み合わせになるように体系的に画像ファイルに名前を付けます。例えばデータセットは、チャネル識別子として「青」(クロマチンDNA染色)、または「グリーン」(GFP)または「赤」(免疫染色されたフルオロフォア)を使用する。ウェルアドレス、フレーム番号およびチャネル識別子はさらに上の画像のメタデータと呼ばれる。紛らわしいウェルおよびフレームメタデータを避けるために、アンダースコア記号を使用してください。
- 指定された順序でこれらのメタデータ要素を持つファイルに名前を付けます。これにより、後続のソフトウェア工程がカレことを保証する必要がある分析のための画像のCTLYグループを設定します。
- フリーウェアの携帯プロファイラをダウンロードしてインストールし、アクティブなPerlのコミュニティ版、R統計プログラミング環境とRStudio。インストール時にすべてのデフォルトオプションを受け入れ、アクティブPerlをインストールするPCのユーザーは、プロンプトが表示パス、ファイル拡張子の関連付け、スクリプトマッピングに関連するすべてのオプションを有効にする必要があります。アクティブPerlはMacユーザーのためのオプションですが、彼らはそれ以外の端末コマンドラインではなく、アイコンのクリックを使用してから、ステップ3.2でPerlスクリプトを実行する必要があります。
- セル·プロファイラソフトウェアを開き、「ファイル」をクリックし、[インポート]パイプライン」、次に「ファイルから」とファイル3_channels_pipeline.cppipe( 図S1A&S1B)を選択します。ファイルには記述されているファイル名規則から画像ファイルのメタデータを解釈するためのソフトウェアのために必要な命令が含まれています。細胞プロファイラは今番目から核DNAと抗体の強度を抽出し、画像を関連ESEとセル毎に検出( 図4および5)は、細胞質強度に対する核の比率を計算するためにGFPチャネルを使用する。
- 細胞プロファイラウィンドウの左下隅にあるボタン「表示出力の設定]をクリックします。新しい画面の上部にある[既定の入力フォルダ」と「デフォルト出力フォルダ」というラベルの付いたテキストボックスがある。当時の一つは、これらのボックスの右にあるフォルダのアイコンをクリックして、(S1C図 )をそれぞれ抽出したデータ、の分析のための画像ファイルの場所と目的地を選択します。
- 細胞プロファイラの左下隅に「分析イメージ」ボタンを押すと、画像解析を開始します。画面の下部にデータ抽出、「ストップ分析」と「一時停止」ボタンの残り時間を観察する。必要な便利である、任意の時点で「一時停止」ボタンを選択して、分析を一時停止した場合分析される画像を見たときに(ステップ2.8参照)。
- 必要に応じて、プログラムウィンドウ( 図S1D)の左端のパネルに目をアイコンをクリックすると、画像解析ステップのいずれかのためのウィンドウを開きます。画像分割を実行するために、細胞プロファイラで現在の設定は、(これらの設定を変更する上でのアドバイスについては、図1および考察を参照のこと)に適していることを確認するために「IdentifyPrimaryObjects」ウィンドウと「セカンダリ」のものを観察し、「第三紀Objectsの。
- 分析が完了したときに表示されるメッセージボックスで「OK」をクリックします。その結果、すべてのデータファイルは、コンマ区切り値(.csv)にファイル( 図S2A)として保存されている場所」のデフォルトの出力フォルダ」に移動します。
3.データの抽出
- 中に含まれている新しいファイル'Nuclei.csv'を検索細胞プロファイラーからの出力。このファイルには、蛍光核抗体の強さ、核DNA強度とGFP-CDK2レポーター比の値( 図6A&S2A)のための個々のセルデータが含まれています。
注:別の研究室は、独自のアッセイの性質に合わせて、このタイプのデータを処理するであろう。現在のデータのために提案、提供Perlスクリプト」2_gate_classifier.pl」を使用して抗体データとGFP-CDK2レポーター値に応じてそれぞれの処理条件から細胞のゲーティングである。 - 「Nuclei.csv「データファイル( 図S2A)と同じフォルダに提供されるPerlスクリプトファイル'2_gate_classifier.pl'をコピーする。 Perlスクリプトのアイコンをダブルクリックして、プロンプトが表示されたとき、細胞はゲートされ、最終的に抗体のためのゲートの値されるべきで、ファイルの「.CSV」ファイル名が続くデータファイルの完全な名前を入力します蛍光とGFP-CDK2レポーターデータ。
注:主にゲート設定を決定し、データの分析のためにこれらを適用する方法が代表的なデータセクション及び図6に、以下に議論される(データ提供利用「0.004」をそれぞれ「1.5」を、分析する)。 「perlの2_gate_classifier.pl ':Macユーザーは次のように入力して、ターミナルコマンドラインからPerlスクリプトを実行する必要があります。 - 各ウェルからの各セルは両方のゲート( 図6C)に対して実行する方法を示し、サブ母集団のラベルを元の細胞プロファイラデータから生の個々の細胞アッセイ値を組み合わせ、新しく作成したファイルを確認します。
- RStudioソフトウェアを開くことによって、個々の細胞亜集団のラベルを使用して、各実験条件についてのデータをプロットします。 「ファイル」と「ファイルを開く」をクリックし、提供「analysis.r」ファイルを選択します。 図6Bをプロットするためのコマンドを観察し、 図S2B)の左上のウィンドウで·チョン> 7と8。左上のウィンドウでは、線5,6上の二重引用記号の間に、ゲートされたデータを含むフォルダのコンピュータアドレスを入力します。 ( ":/分析フォルダ/分析出力C」と「nuclei_gated.csv」など 、)は、それぞれドライブ文字とファイル自体の名前を含めます。
注:RStudioは、与えられたコンピュータ上で初めて使用されている場合は、Rグラフィックパッケージ 'ggplot2'を最初にインストールする必要があります。これは、このステップは、冗長になるの後RStudioの新規インストールのための一度だけのステップです。 「ggplot2」をインストールするには、RStudioの右下にウィンドウの上に「パッケージ」と呼ばれるタブをクリックし、この下に表示される「パッケージのインストール」ボタンをクリックします。新しいウィンドウが表示されます。 'パッケージの中にタイプ' ggplot2 '(省略さ引用)この新しいウィンドウでペース、最後にステップ3.6から継続して、ウィンドウを閉じる必要ggplot2機能をインストールし、メインRStudioウィンドウに戻り「インストール」ボタンをクリックしてください。 - ハイライトラインRStudioの左上のウィンドウで17を1、それから「ファイル名を指定して実行]ボタンをクリックします。これはR( 図S2C)に実験データ、しきい値およびウェル場所の詳細を入力します。 Rは今一時的にプロットするための関連データを保持します。
- 17行の下に残りのコードの個々のブロックをハイライト表示し、以前のように「ファイル名を指定して実行」ボタンをクリックすることで、対応するプロットを作成します。 RStudioの右下隅にウィンドウにプロットを観察し、「エクスポート」ボタン( 図S2D)をクリックすることにより、フォーマットの数を保存します。
- RStudioを閉じながら、プロンプトが表示されたら、「保存しない」をクリックします。これはそうでなければ、データを保持するRStudioの次の使用上の混乱を防ぎます前のセッションから。
結果
リバーストランスフェクションsiRNAスクリーニングプロトコルを使用して生成された画像のセットの例は、のために準備し、細胞プロファイラソフトウェアを用いて分析した。得られた数値の生データは、各セルを個別に追跡可能戻す画像によく起源の、表現およびいくつかの蛍光強度パラメータ( 図6A)について測定されるようなものである。 P-S780のRB1抗体およびDNA染料定義核マスクの組み込まれたDNA強度の平均核蛍光強度を特定し、各セルに対して決定される。各細胞の核および細胞質領域に対するGFP強度値の平均はまた、GFP-CDK2レポーターの細胞質蛍光対核の計算を可能に記録されている。これらのアルゴリズム的蛍光強度測定用の下流には、2つのアッセイ、核抗体染色およびGFP-CDK2レポーターのためのゲートを定義するために、これらの個々のセル·データで構成されている。目の上の細胞のその後の注釈アッセイの結果と第3の測定(核DNA含有量)によって特徴付けられる特定の亜集団を有効にするためにこれらの標識の使用者の電子単位について説明する。
各アッセイのために収集された生の蛍光強度データのヒストグラムプロットは、細胞亜集団は、異なる条件下でどのように振る舞うかを評価する有効な方法である。 図6Bのヒストグラムは、各RNAiのノックダウン状態のための三連ウェルから個々のセルデータの人口分布を示している。左側には核抗体の強さのために、右側のデータは、GFP-CDK2レポーターのための対応するデータがされている。 P-S780 RB1抗体データは、細胞が広く、この翻訳後修飾に関してつの集団内に存在し、S780がリン酸化RB1の損失でその細胞集団が富化される核強度の左側のピークとして識別することができることを明らかにCDK6は、siRNAによってノックダウンされたとき。これと同じ左側のピークRB1自体は完全にタンパク質を除去することにより、P-S780のRB1染色を反映して、RNAi標的である場合に見られる。対照的に、同じセルに同じ実験条件は、GFP-CDK2レポーターアッセイで観察したときに、個々のセルデータの異なるダイナミックを示す。連続分布は、単一のピークが観察されるが、細胞周期(siCDK6)を乱し(その分布の右側の肩の延長G1期結果の蓄積を引き起こすsiRNAを、すなわち示す細胞の増強された存在を示す核/細胞質GFP率の増加、)X軸にプロット。
図6Bのヒストグラム分析データの両方のセットの分布に基づいて選択されたゲート値(垂直バー)であるにも示す。メインの左肩上の最大幅の位置:P-S780 RB1抗体データに使用される規則は、ハーフハイトのようなゲート位置を定義することです陰性対照細胞データを考慮した場合(右)のピーク(siRNAの非標的)。データは、赤を強調し、このゲートで識別され減少したと不在P-S780 RB1、との細胞である。比の値の分布の反対の肩に位置する同様のゲートは、GFP-CDK2レポーターのために使用される。欠くまたは機能CDK2の活性が低下し、得られた高倍率の亜集団の細胞は、緑色で示されている。 2アッセイ図6Cの多重化された分析を説明するために、以下の注釈付きのファイルに生データ( 図6A)を変換するために2_gate_classifier.pl Perlスクリプトを用いたゲート値の両方の実装を示しています。この新しいファイル(この場合GFP-CDK2レポーターに対する抗体データについて0.004と1.5のゲートは、それぞれ、使用された)各セルのクラスラベルと区別するために使用される2つのゲートの値の新しい列並んで元のデータを含む。
個々のセルFに分類したROMは、アッセイデータのプロットの注釈を支援するために、これらのクラスラベルを使用することができるようになりました2アッセイに基づいて、各ノックダウン状態。7に示す。図 P-S780 RB1とGFP-のための個々のセルデータのプロットを散乱すべての3つのRNAiの条件に設定されたサンプルデータからCDK2アッセイ。散布図に象限に注釈をつける数字は、そのノックダウンコンテキストの全体に各ゲートさ亜集団の相対的な割合を示し、上記のクラスラベルを使用してRで生成されます。これらのプロットはsiCDK6でトランスフェクトされた細胞は、正味のデータ分布(セリン780でのRB1のリン酸化の非存在を示す)、Y軸上と右下がりの両方にシフト明らかにする、非標的とするsiRNA( 図7B)でトランスフェクトされた細胞と比較して、ことを明らかにしたX軸上に( 図7C、低CDK2活性を示す)。これらのシフトの両方が、このターゲットのノックダウンのために期待されている。 SIRからこれとは対照的に、データB1トランスフェクトした細胞( 図7A)は 、エピトープの損失に保つのに抗体染色の喪失を示しているが、非標的化siRNAでトランスフェクトした対照と比較して、CDK2レポーターのデータ分布にほとんど影響、GFPには大きな効果を示唆していない-CDK2記者はRB1のノックダウンから生じる。
さらに、個々のセルデータ、亜集団の分類と分析多重図8の使用を探求するには、組み込まれたDNA強度については、図7(c)と一緒にペアになったヒストグラムプロファイルからsiCDK6データ用の散布図を示している。ヒストグラムの組は、抗体強度(散布図の右側)またはGFP-CDK2レポーター比の値(散布上記)のいずれかに基づいて分割された全人口の対向する半分に関連する。これらの集団のための核DNA強度の定量化は、それぞれ、左および右のピークが2Nと4N DNA含量の特徴二つのピークを示している。私またはGFP-CDK2レポーター(標識:G1)より高い比の値で: 図6、図7および図8に示すゲートntentions細胞としてP-S780 RB1、低(P-S780-標識)が同定されるようである意志細胞周期のG1期にある。実際に、これらのアッセイのいずれかで識別亜集団のためのDNAプロファイルヒストグラムは、主に、2N DNA含量を有する細胞を含む。逆にゲートさ集団のDNAプロファイルは(ラベル:P-S780 +または非G1)、2Nから4Nまでの範囲の分布を有する細胞を含む、そのような細胞は、細胞周期の範囲を採用することと一致してポストG1期を位置決めする。
ここでの焦点は、蛍光染色された画像から、個々のセルデータの生成と分析されているが、これらのデータを取得し、反復と性能との間の変動を監視するために、よくによってウェルに基づいて、各アッセイをまとめることができることも有用であるデータのプレート全体にわたる所与のアッセイのための全てのウェルの。 図9はAに印加ゲート内のパーセント細胞のための三連ウェルからの平均値としてまとめられ、各siRNA処理からデータ)P-S780のRB1データおよびB)GFP-CDK2レポーターデータを示している。 AとBにプロットされた値は、この原稿で設けられた2つの追加のPerlスクリプトによって生成されます。それぞれ「antibody_fluorescence_summary.pl 'と' G1assay_summary.pl」、。これらのスクリプトは、ウェル)iと当たりの測定された全細胞をウェルあたりの生細胞プロファイラ(Nuclei.csv)によって作成されたデータとレポートデータを使用し、II)ゲート内の細胞数、ⅲ)ゲート内のパーセント細胞およびiv)算術よくそのための測定された、生のデータの平均。これに示すように、前の個々のセルデータの多重化された評価を使用して、個々の治療データに着目し、アッセイデータの大きな集合にわたって検索するための適切なオプションとして含まれている 7及び8図。チャートは、 図6Bのヒストグラム中のP-S780 RB1とGFP-CDK2データに見られる非正規データ分布に合わせて両アッセイ、のための「ゲート内III)パーセント細胞を「ここプロットを示した。これらのスクリプトは、また実験的摂動前と後の均質な集団応答と正常なデータ配信のためのデータの分析に合うだろう」だけでなく、そのために測定し、生データのIV)算術平均を」、計算する。
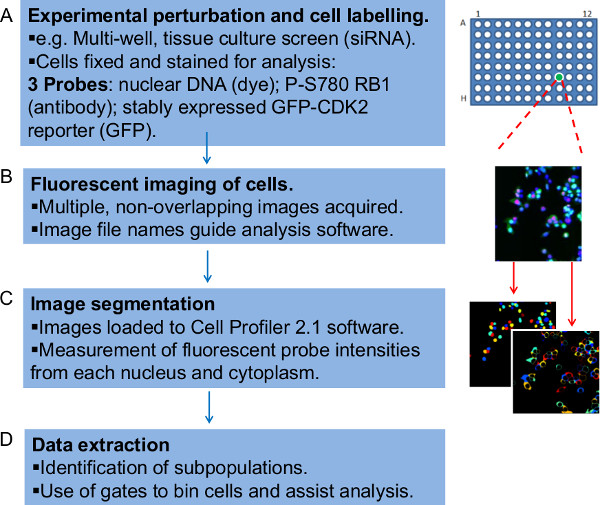
図1:定量的に蛍光標識された顕微鏡画像データを解析するためのワークフローの手順の概要ワークフローは、4つのステップとしてここで表される(A)は、第一に、それは実験的に蛍光イメージングのための細胞を調製する必要がある。ここで説明する例でのものであるsiRNAで処理された接着性ヒト腫瘍細胞を96ウェル組織培養プレートに、48時間増殖させ、固定し、染色された画面。異なるRNAiの条件は、プレート内の別々のウェルに三重で存在している。細胞は、DNA色素、CDK4及び6選択的標的部位セリン-780(P-S780のRB1)がリン酸化RB1に特異的な抗体で染色され、それらはまた、安定的にG1細胞周期の終了を報告し、GFP-CDK2レポーターを発現する。集合的に、これらの蛍光プローブは、2つのアッセイは、別々にワークフロー内で評価され構成されている。(B)の各蛍光プローブ(チャネル)のための並列の顕微鏡画像を生成し、画像解析ソフトウェアは、データを整理することが可能な内容を含むようにそのような名前が付けられています。(C)画像ファイルは、アルゴリズム的に検出3つの蛍光プローブの強度測定値が得られる前に、個々の細胞および核と細胞質の関連するペアを特定する細胞プロファイラソフトウェアにロードされている各ED。(D)最後に 、Perlスクリプトが生成生定量的データを整理するために使用されている。このステップは、効果的にプロットすることができ亜集団、に細胞をビニング、各セルの蛍光強度データにゲートを適用し、追跡し、クロスを検討した。 この図の拡大版をご覧になるにはこちらをクリックしてください。
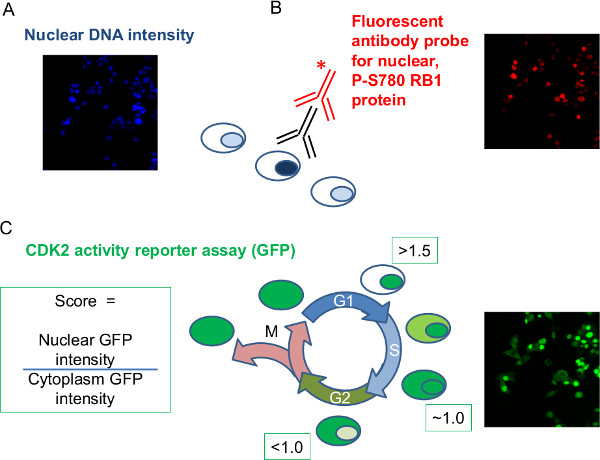
図2:画像解析によって得られる実験データの例からの固定、siRNA治療、蛍光標識された細胞は、データセットを画像化し、対応する強度測定値は、セルごとに採取した。代表画像データは、画像解析の間に記録された各パラメータについて示される(A)核DNA強度:核DNA色素の染色の強度は、に使用され核あたりのDNAの測定値を得た(B)リン酸化RB1の核強度:免疫染色一次(黒)抗体を用いて蛍光二次抗体(赤色)をタグ付きでRB1リン酸化の強度測定を可能にするP-S780のRB1に特異的な。核あたりのS780(C)GFP-CDK2レポーター:使用される細胞は、安定的に細胞周期とセットパターンの核と細胞質の間で移行したGFPタグ付きレポータータンパク質を発現する。各セルの対の核および細胞質GFP強度の二重の測定は、細胞周期の残りの部分からG1期を区別するために使用することができ、細胞あたりの比率を計算することができる。三つのsiRNA標的分析を説明するのに使用される。非標的ネガティブコントロールsiRNA。 RB1のリン酸化および細胞周期進行を乱すにおける陽性対照としてCDK6のsiRNA。 RB1 siRNAは、抗体の特異性を確立する。K ">この図の拡大版をご覧になるにはこちらをクリックしてください。

図3:画像ファイルの編成前の画像解析 、 組織培養プレートから採取された画像は、元の実験的背景に画像データを関連付けるための画像解析ソフトウェアを可能にするために、体系的に命名される。この情報は、各画像のファイル名内に配置されている。(A)ウェル実験プレート上のそれぞれのように異なるのRNAi標的または治療は、ファイル名のウェルアドレス形式の一部分に対応することができる。(B)フレーム番号をファイル名の一部である各ウェルは、複数の重複しないフレームを収集するために撮像されるように、各フレームから(C)蛍光プローブを別々に結像される。その結果、ファイル名には、各画像がに関し、どのチャンネルに反映する必要があります。 (D)例ファイル名、フレーム(2)チャネル(青、赤、緑)のいずれかを表す各画像である。点線はさて、フレームおよびチャネルに関連する概略図に、ファイル名の要素をリンク、それぞれ。 この図の拡大版をご覧になるにはこちらをクリックしてください。

図4:細胞プロファイラの使用は、核DNAと抗体染色を測定するために設けられ、パイプファイル(3_channels_pipeline.cppipe)の設定では、細胞プロファイラ画像解析ソフトウェア対策個々のセルに関連する核DNAとの抗体結合についての蛍光強度値。 (A)核が染色されたDNAの「青」チャンネル画像において識別される。(B)< / strong>のDNA染色された核の位置が一時的に「核マスク」で開催されている。核マスクが次に(C)(それぞれ、DNAおよび抗体の蛍光データ)、青と赤のチャネル画像上にオーバーレイされ、マスクと重複する画像セグメントからの蛍光値は、識別された各セルに対して記録されている。別個の隣接核の同定の成功は、視覚的に核マスクの外観を評価することができる。説明のために、このマスク画像の丸で囲ん示し、アルゴリズムの選択した設定は、単一の核として誤って識別され、隣接する核を持っている例がある。ディスカッションセクションで紹介されているこれらのイベントを最小化するアルゴリズムの設定を調整する。 この図の拡大版をご覧になるにはこちらをクリックしてください。
/ftp_upload/51882/51882fig5highres.jpg "/>
図5:セルプロファイラの使用は、核および細胞質GFPの強度を測定するためには、GFPタグ付きCDK2レポーターは、細胞の細胞周期位置に関連して、核と細胞質との間で移動する。細胞プロファイラは細胞あたりDNAおよび抗体の核強度を算出するのと同時に( 図4)では、各セルのためのGFP強度の細胞質比に核を計算する。(A)各画像のDNA色素データを生成するために使用される核マスク。(b)細胞Profilerは、核が各セルの位置をシードするGFP-CDK2レポーターからのGFPの画像と一緒にマスクが使用した後、各セルの全体フットプリントを推定するために、各セルの境界に展開されます。これは、核マスクが細胞質のドーナツ状シリーズを生成するためにセル·マスクから減算される(C)。新しい、「セル·マスク」になるとなっている、アウトライン「細胞質マスク」。(D)核および細胞質マスクマスクは、核および細胞質GFP値のペアを測定するために細胞プロファイラによって使用される。これらのペア値は、細胞周期の各セルの位置に関して知らせる比率を計算するために細胞プロファイラによって使用されている。 この図の拡大版をご覧になるにはこちらをクリックしてください。
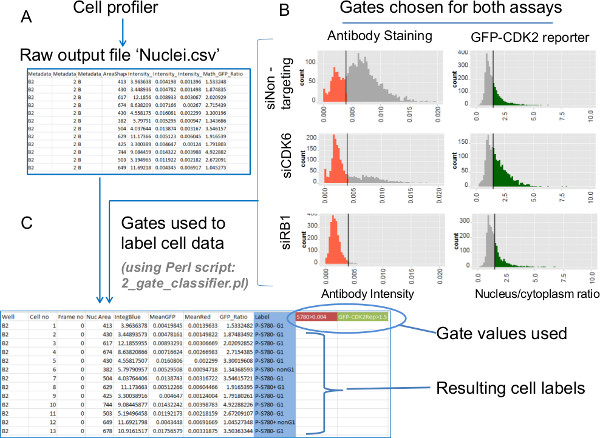
図6:データ抽出-アッセイ値にゲートをかけることによって、生の個々のセル·データの処理抗体染色とGFP-CDK2レポーターアッセイのための個々のセルデータから生物学的な傾向は、抽出されたゲート制御データを使用している。生データのヒストグラムは、適切なゲート値の識別を可能にする。次に、これらをPerlスクリプトで課されている。(A)細胞プロファイラのために提供設定で画像ファイルを分析の最終製品は、カンマ区切り値(.csv)にファイルです。異なるサブセルラーセグメントのそれぞれに関連する個々のセルデータontainこれらのファイルをC。ファイル'Nuclei.csvは「核マスクの使用に関する選択したすべての測定結果が含まれています。これらの測定は、核抗体強度(B)のヒストグラム(左)。核抗体強度、核DNA強度及びGFP比(核/細胞質)を含み、GFP-CDK2レポーター比(右)は、各siRNAのノックダウン条件の個々のセルデータからプロット。表示されたヒストグラム上のバーは、これらのアッセイのために所望のゲート位置を示している。ヒストグラム上の着色されたデータは、ゲートさ亜集団を示している。(C)Bに示した2アッセイ用のゲートはPerlのスクリプト」2_gate_classifier.pl」を使用して生データに適用されます。スクリプトその後のプロットを支援するために元の細胞プロファイラ出力(Nuclei.csv)ファイルの変更されたコピーが作成されます。二つのゲートの値(ここでは色で強調表示)新しいファイルに記録され、新たな「ラベル」列が追加される。ラベルビン各セルのための2つのゲーテッドアッセイ値に基づいて4つの可能なサブグループの1つに各セル。これらのラベルは、各亜集団ならびに細胞プロファイラで生成された追加のパラメータの相互参照の貢献の計算を特徴とその後のプロットに使用されている。 この図の拡大版をご覧になるにはこちらをクリックしてください。
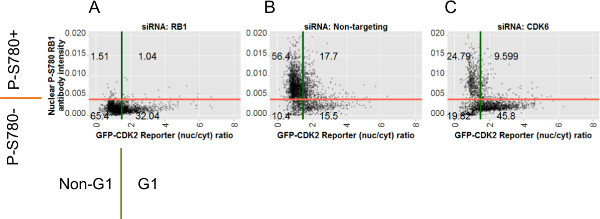
図7:個々のセルとゲート位置の生データを描いたそれぞれのsiRNA条件の散布図 INDIVIのプロットを散布siRNAの条件のすべての画像からのデュアルセルのデータが示されている:(A)siRB1;(B)シノーンターゲティング陰性対照(C)siCDK6。抗P-S780のRB1染色から核蛍光の値は、Y軸に対してプロットされている。 X軸に対してプロットは、GFP-CDK2レポーターから算出し、対応する比の値である。赤と緑のバーは、それぞれ、P-S780のRB1ゲート用のゲートおよびGFP-CDK2レポーターゲートの位置を示す。 2つのゲートが4亜集団に細胞を分割し、その結果の象限上の数字は、これらのそれぞれからの細胞のパーセント数である。 Aの軸回りの注釈は2_gate_classifier.pl Perlスクリプトによって各セルに適用可能な4つのラベル要素を示す。これらのラベルは、それぞれのアッセイゲートに関連して示され、 図6、図7のプロットを生成するために、R-スクリプト(analysis.r)で使用され 8。 この図の拡大版をご覧になるにはこちらをクリックしてください。
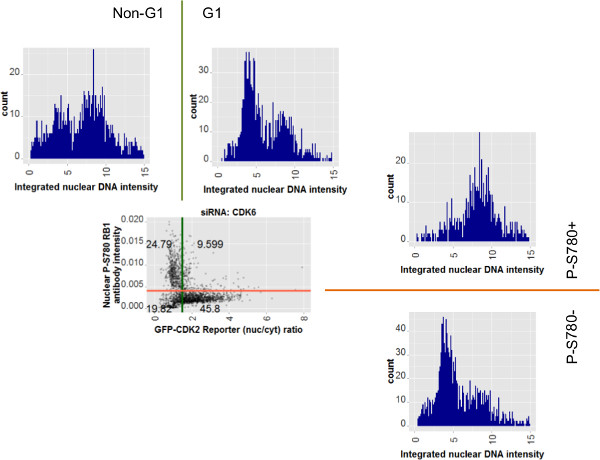
図8は、2つのG1輸送アッセイによって定義された細胞の亜集団は、アッセイの結果と一致して、2Nと4N DNAプロファイルを示す siCDK6細胞のためのデータの散布図を図7Cから繰り返される。散布図を囲むのは人口のサブセットに関連する統合された核DNA強度のヒストグラムである。散布上記のものは、GFP-CDK2レポーターアッセイに関連しています。散布図の右側にあるものだけでは、核リン酸化RB1抗体の測定に関連しています。着色されたゲート線はヒストグラムとの関係を示すために拡張される。セルデータは、これらの追加のプロットのために選択されたことにより、ゲート·ラベルも示されている。それぞれのアッセイのためにそれらの逆の対応に対し、細胞質比(低CDK2活性を示す)のセリン780(P-S780-)または高GFP-CDK2レポーター核を有するものでリン酸化RB1の損失を有する細胞、主に2N状DNAプロファイルを示し、 2Nと4N、細胞の混合、ポストG1期の人口の特性の分布を示している。 この図の拡大版をご覧になるにはこちらをクリックしてください。
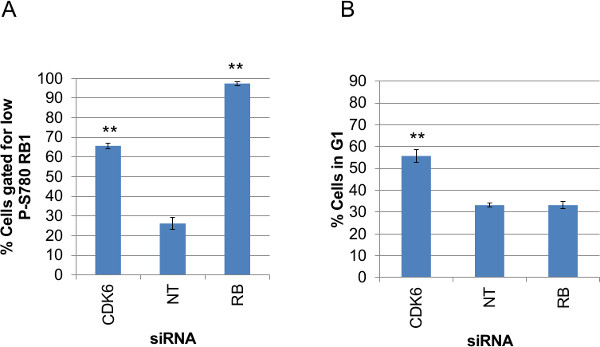
図9:。ゲートさ(A)P-S780のRB1データおよびそれぞれのsiRNAノックダウン状態のための三連ウェルから(B)GFP-CDK2データの各siRNAの条件の概要データプロットのためのゲーテッドアッセイ値の概要プロット 。値は、使用して、生細胞プロファイラ出力(Nuclei.csv)から算出したPerlのスクリプト、「antibody_fluorescence_summary.pl '(A)または' G1assay_summary.pl '(B)。プロットされた値は、各アッセイに印加されるゲート内のパーセント細胞の手段です。バーは三連ウェルから計算された標準誤差を示している。対になっていないが、非標的とするsiRNAと比較した各ノックダウン条件の等分散T検定P値はP <0.001(**)とP <0.05(*)が。プロットされたデータの上に表示されているこのの拡大版を表示するには、こちらをクリックしてください図。
図S1。画像解析のための細胞プロファイラソフトウェアをセットアップする。細胞プロファイラの(A)のスクリーンショットは、任意の画像解析の設定が入力される前に。(B)スクリーンショット細胞プロファイラの「3_channels_pipeline.cppipe」に含まれているアルゴリズムの詳細がロードされた後。高い左上隅に点灯タブには、この画面は、分析の「LoadImages」舞台のパラメータを示していることを示します。この下のリストの他の部分をクリックすると、分析で後続のステップの詳細が明らかになります。入力フォルダとフォルダが入った出力の詳細と細胞プロファイラの(C)のスクリーンショット。(D)細胞プロファイラのスクリーンショットの後」の分析画像」ボタンを押して分析を開始するためにクリックされた。分析中の画像からソフトウェアによって生成アルゴリズム的に生産マスクを示す3新しい窓が重畳されている。これらのウィンドウはメインセルプロファイラウィンドウの左上隅に、次の分析の関連するステップに開いた位置に「目」のアイコンをクリックしてアクセスされている。これらのビューは、紙吹雪色のマスクを生成する設定が添付、オリジナル、グレースケールのデータと一致するかどうかを確認するためにユーザーを助ける。
ゲート個々のセルデータに ENT PerlとRStudioの "> 図S2。使用し、得られた細胞亜集団をプロットします。(A)右側のパネルには、セル·プロファイラ分析から出力.CSVファイル(緑のアイコン)を受信するように選択したフォルダを示しています。原稿(青いアイコン)で提供するPerlスクリプトは、このフォルダにコピーされます。ハイライトは、左側のパネルで、ダイアログボックスを生成するために、マウスでダブルクリックされた「2_gate_classifier.pl「Perlスクリプトです。表示されますすぐに「analysis.R 'スクリプトをロードした後RStudioのゲート「Nuclei.csv'ファイルから個々のセルのデータに必要なプロンプトと入力した対応する回答。(B)スクリーンショット。ハイライトは、へのゲートさのデータをアップロードするコマンドです事前の線5,6におけるノートの詳細(プロットへのソフトウェアは、ゲートされたデータは、計算に配置されている場所に応じて調整する必要があります分析に使用R)。RStudioの(C)スクリーンショットデータは一度はアップロードされた。(RStudioを示すのD)スクリーンショットは右下のウィンドウに表示されるプロットを生成するために必要なコードのブロックを強調した。各プロットのためのコードは、空行で分離され、プロットの種類によってグループ化されています。| siRNA標的 | プレートウェルアドレス |
| ノンターゲッティング(NT) | E5、F5、G5 |
| 網膜芽細胞腫(RB) | E7、F7、G7 |
| サイクリン依存性キナーゼ6(CDK6) | B2、C2、D2 |
表1:まあアドレスと例のデータセットで使用される対応するsiRNA条件。
ディスカッション
記載されたワークフローは、siRNA、その後のマーカー検出を用いた細胞のウェル摂動する手順を構成し、最終的に得られた蛍光顕微鏡画像から定量的データの抽出を容易にするために、ソフトウェアでサポートされている一連のステップの使用。アプローチは、多くの細胞ベースのアプリケーションにおいて幅広い実用的用途を有する個々の細胞の核および細胞質強度値の送達に焦点を当てている。ここで使用される例示的なデータは、G1期の細胞周期の通過のための2つの蛍光アッセイは、試験され、バック核DNA含有量のより直接的な生物物理学的測定値に相関されたsiRNAの画面の設定で生成された。
それは個々の細胞の同定を可能にし、得られた「核マスクが「対応cytoplasmiを識別するための出発点として機能するように、画像の核DNAに蛍光DNA染色の使用は、画像分割プロセスに不可欠なステップであるC領域。安定的に細胞内で発現されたGFPタグCDK2の記者は、まだこのコンパートメントは描写することが可能な細胞質中のバックグラウンドシグナルよりも一貫して高い変数を提供します。同じ分析パイプラインは、他の適当な蛍光連結記者や摂動に対する応答を使用してタンパク質転位事象の分析に適用可能であるべきである。また、細胞質特異的蛍光色素を用いてGFP-CDK2レポーターを代入すると、このアルゴリズムの代替的な使用は、細胞質の次元画像中の細胞の相対的な大きさを測定することを可能にする。
ここで説明する画像分割戦略の別の設計考慮事項は、DNAの定量化のための統合された強度値を提供するセルプロファイラを使用することである。核DNAの染色データに対する強度値の積分核のサイズの可能なバリエーションを可能と見られる定量化プロファイルの近い一致を表すヨウ化プロピジウムのためのFACSデータを染色した。しかし、積分強度は、抗原の蛍光の平均強度によって例示平均濃度は、細胞コンパートメント内でより生物学的に関連するタンパク質の集積総量(および関連蛍光)よりもタンパク質の機能を評価するための適切な手段を提供することができない。したがって、強度値は、P-S780 RB1及びGFPのデータのために使用したことを意味する。データ評価の2つのモードを変更するオプションが(平均または統合)細胞プロファイラソフトウェアの「ExportToSpreadsheet」パネルに記載されています。
3_channels_pipeline.cppipeファイル内の解析設定は、例えば、データセット内の画像のために最適化される。このプロトコルを使用して新しいイメージのセットの分析は、ファイル名が命名規則は、上記( 図3)に記載採用することを必要とします。また、感度は、バックグラウンド核DNA染色としきい値の明るさに応じた値新しい画像セットにおける強度は、Cellプロファイラの設定内で調整する必要があるかもしれない。 DNA染色は、様々な画像セグメンテーションマスクを作成するための保持に重要な役割を考えると、このチャネルの正しい感度設定の適用は、細胞プロファイラソフトウェアと新たな画像データの解析に成功の鍵である。提供される細胞プロファイラ設定ファイル(3_channels_pipeline.cppipe)は、新しいデータに分析を適合させるための最も頻繁に有用なパラメータに関する注意事項が含まれています。これらのノートは、Cellプロファイラーのメインウィンドウで、画面上部のテキストボックスにあり、感度の設定を変更し、分析するチャネル数を調整する上でのガイダンスが含まれています。新たな画像データの設定をテストするために、プロトコルのセクション2.8で起訴されたように、プロトコルステップ'...オブジェクトを識別する」をそれぞれについて「目」のアイコンを開く]をクリックして画像解析時の画像分割を観察する必要があるかもしれない( 図S1D )。核マスクが正しくDNA染色の画像から特定された場合、特に、「IdentifyPrimaryOjects 'を介して画像データの視覚化は、表示される。 「IdentifyPrimaryOjects」モジュールのセル·プロファイラソフトウェアページで閾値補正係数である。この値の試行錯誤調整はほとんどの核認識エラーを修正します。値は、各画像のバックグラウンド強度に対するDNAのチャネルのバランスをとる。閾値補正係数値の周りの1、これよりも大きいが、より厳しい(クリアな画像のために良い)で、1未満(染色と背景の少ないコントラストの画像に適した)寛大ですヒンジ。
細胞プロファイラからの個々のセルデータの生の出力は、他の研究者のニーズに合わせて様々な方法で分析することができる。ここに示したデータanから生物学的な傾向を抽出することを支援するために、セルごとに測定されたパラメータのうちの2つにゲートを適用するためのPerlスクリプトの使用である追加の測定で識別部分集団のD許可相互参照。それは、Cellプロファイラの枠組みの中でゲーの要素を含むことも同様に可能ではあるが、ここで使用される代替ルートは、大規模なデータセットを評価する必要がある場合には特に、より高い柔軟性とスピードを提供します。現在のプロトコルの後の画像取得の段階で最も遅い段階は、Cellプロファイラソフトウェアの実行されています。ここで細胞プロファイラは、必要に応じて反復的に異なるゲート値で、より迅速に、その後のPerlスクリプトを再分析することができ、未ゲーテッド生データセットを生成するためにゲートをかけることなく実行されます。すべての研究は、このように適切なゲート値はデータの任意のセットで、潜在的に時間をかけての試薬と異なる場合があり、事前に知っているわけではありません。したがって、適切なゲートバリューを識別するために、陽性対照およびモデル摂動細胞について細胞プロファイラから得られた生データ配信を示すヒストグラムを生成することをお勧めです興味のあるパラメータのためのES。
Perlスクリプトは、Cellプロファイラからのデータの厳密に定義された列構造を受け入れるために、ユーザーが「ExportToSpreadsheet」設定を用いて細胞プロファイラによってパラメータ出力の数を変更する場合は、作業を停止することが書かれています。ノートはPerlのスクリプトファイル内に含まれる設定の変更を実装するのに役立ちます。これらはテキストエディタでスクリプトを表示する表示するには、好ましくは、プログラマのテキストエディタ( 例えば 、http://www.activestate.com/komodo-edit)カラーコードPerlの要素に設定されています。これらのノートは、データフォーマットの変化に適応するためのスクリプトを調整する場所を示す。 Perlスクリプト、(analysis.r)提供R-コードファイルと同様に、画像解析データから数値をプロットするための命令を含む、使用および適応に追加したメモを参照し、テキストエディタやRStudioソフトウェアで読み取ることができます。これらのノートは、正規表現とPerlについての詳細を補足することができる<> 12(商標)、それぞれ、データは、読み込みアノテートしてプロットされている方法の基礎を形成し、どちらもR用ggplot2 13パッケージ。
蛍光顕微鏡を用いて新たな研究と同様のオープンソースの出版物に寄託生データは、ここに記載されるような分析方法に適している。高コンテンツデータの本質は、任意の観察者の研究関心に応じて、異なる分析的な強調を持つ再帰的な分析に適しています。データの求められることができる質問はもともと使用されるプローブによって制限されていますが、画像データは、多くの場合、意味のある、それを生成した研究の範囲を超えて再分析することができます。
開示事項
著者らは、開示することは何もない
謝辞
この作品は、助成金CRUK 15043とCRUK 14251によってサポートされていました。
私たちは、技術支援や原稿の重要な読書のためのダニエルWetterskogおよびKa圭ホーに感謝。
資料
| Name | Company | Catalog Number | Comments |
| AllStars negative control siRNA | Qiagen | 1027280 | Negative control siRNA. |
| CDK6 siRNA | Dharmacon/ Custom synthesis | NA | Antisense sequence: 5' CUCUAGGCCAGUCUUCUUCUU |
| RB1 siRNA | Dharmacon/ Custom synthesis | NA | Antisense sequence: 5' GGUUCAACUACGCGUGUAATT |
| 5x siRNA buffer | Thermo Scientific | B-002000-UB-100 | To be diluted with nuclease-free, non-DEPC treated water. |
| Nulcease-free water (non-DEPC) | Applied Biosystems | AM9937 | For dilution of siRNA buffer. |
| Hiperfect | Qiagen | 301705 | Transfection lipid. |
| DMEM | Life Technologies | 41966052 | Pyruvate and high-glucose supplemented tissue culture media. |
| 96 well tissue culture plate | Falcon | 3072 | Plain, 96 well tissue culture plate for the parallel, compartmentalized mixing of transfection complexes. |
| Packard Viewplate | Perkin Elmer LAS | 6005182 | 96 well TC plate with optical base on which cells are transfected, grown, fixed and eventually imaged. Supplied with opaque, adhesive plate seals, which are used during storage of used plates. |
| Breathable membrane | Alpha Labs | LW2783 | Sterile, gas-permeable, adhesive membrane. |
| Neutral buffered formalin, 10% | Sigma-Aldrich | HT5012-1CS | 10% Formalin (4% formaldehyde) fixative used neat in protocol. |
| Triton X-100 | Sigma-Aldrich | X100PC | Non-ionic detergent |
| [header] | |||
| Tris | Sigma-Aldrich | T1503 | Trizma base (tris(hydroxymethyl)aminomethane) |
| Tween 20 | Sigma-Aldrich | P2287 | For plate wash solution. |
| Anti-P-S780 RB1 antibody | Abcam | ab32513 | Rabbit monoclonal |
| AlexaFluor647 Anti-rabbit | Invitrogen | A21245 | Highly cross-adsorbed, fluorescently labelled secondary antibody. |
| Hoechst 33342 (Bisbenzimide) | Sigma-Aldrich | B2261 | Fluorescent, chromatin-intercalating, DNA dye. |
| Permeabilization solution | NA | NA | 0.1% Triton X-100 in 50 mM Tris-buffered saline, pH 8.0. |
| Plate wash solution | NA | NA | Tris-buffered saline containing 0.1% Tween-20. |
| Block solution | NA | NA | 5% powdered milk in Tris-buffered saline and 0.1% Tween-20. For blocking plate prior to immuno-staining and dilution of antibodies. |
| Cell Profiler 2.1 | Broad Institute | http://www.cellprofiler.org/download.shtml | |
| Active Perl Community Edition | ActiveState | http://www.activestate.com/activeperl/downloads | |
| R programming environment | The R Foundation | http://www.r-project.org | |
| Rstudio | Rstudio | http://www.rstudio.com/ | |
参考文献
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), 100 (2006).
- Khan, A., Eldaly, H., Rajpoot, N. A gamma-gaussian mixture model for detection of mitotic cells in breast cancer histopathology images. J Pathol Inform. 4, 11 (2013).
- Selzer, P., Beibel, M., Gubler, H., Parker, C. N., Gabriel, D. Comparison of multivariate data analysis strategies for high-content screening. J Biomol Screen. 16 (3), 338-347 (2011).
- Lyman, S. K., et al. High content, high-throughput analysis of cell cycle perturbations induced by the HSP90 inhibitor XL888. PLoS One. 6 (3), e17692 (2011).
- Richardson, E., Stockwell, S. R., Li, H., Aherne, W., Cuomo, M. E., Mittnacht, S. Mechanism-based screen establishes signalling framework for DNA damage-associated G1 checkpoint response. PLoS One. 7 (2), e17692 (2012).
- Heynen-Genel, S., Pache, L., Chanda, S. K., Rosen, J. Functional genomic and high-content screening for target discovery and deconvolution. Expert Opin Drug Discov. 7 (10), 955-968 (2012).
- Krausz, E. High-content siRNA screening. Mol Biosyst. 3 (4), 232-240 (2007).
- Gu, J., Xia, X., et al. Cell Cycle-dependent Regulation of a Human DNA Helicase That Localizes in. DNA Damage Foci. Mol Biol Cell. 15 (7), 3320-3332 (2004).
- Mittnacht, S. Control of pRB phosphorylation. Curr Opin Genet Dev. 8 (1), 21-27 (1998).
- Mittnacht, S. The retinoblastoma protein--from bench to bedside. Eur J Cell Biol. 84 (2-3), 97-107 (2005).
- Nybo, K. GFP imaging in fixed cells. BioTechniques. 52 (6), 359-360 (2012).
- Schwartz, R. L., Foy, B. D., Phoenix, T. . Learning Perl - Making Easy Things Easy and Hard Things Possible. , 1-363 (2011).
- Wickham, H. . ggplot2: Elegant Graphics for Data Analysis. , 978-970 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved