Method Article
EPA שיטת המדידה של 1615. Enterovirus ומופע norovirus במים על ידי תרבות וRT-qPCR. חלק שלישי. איתור וירוסים על ידי RT-qPCR
In This Article
Summary
Here we present a procedure to quantify enterovirus and norovirus in environmental and drinking waters using reverse transcription-quantitative PCR. Mean virus recovery from groundwater with this standardized procedure from EPA Method 1615 was 20% for poliovirus and 30% for murine norovirus.
Abstract
EPA Method 1615 measures enteroviruses and noroviruses present in environmental and drinking waters. This method was developed with the goal of having a standardized method for use in multiple analytical laboratories during monitoring period 3 of the Unregulated Contaminant Monitoring Rule. Herein we present the protocol for extraction of viral ribonucleic acid (RNA) from water sample concentrates and for quantitatively measuring enterovirus and norovirus concentrations using reverse transcription-quantitative PCR (RT-qPCR). Virus concentrations for the molecular assay are calculated in terms of genomic copies of viral RNA per liter based upon a standard curve. The method uses a number of quality controls to increase data quality and to reduce interlaboratory and intralaboratory variation. The method has been evaluated by examining virus recovery from ground and reagent grade waters seeded with poliovirus type 3 and murine norovirus as a surrogate for human noroviruses. Mean poliovirus recoveries were 20% in groundwaters and 44% in reagent grade water. Mean murine norovirus recoveries with the RT-qPCR assay were 30% in groundwaters and 4% in reagent grade water.
Introduction
PCR כמותית (QPCR, ראו חומרים נוספים להגדרות של מונחים המשמשים בכתב היד הזה) ולהפוך שעתוק-qPCR (RT-qPCR) הם כלים רבי ערך לאיתור וכימות וירוסי מעיים אנושיים בסביבה ושתיית מים, ובמיוחד לוירוסים רבים שעושים לא לשכפל או לשכפל בצורה גרועה במערכות תרבית תאים. שני הכלים הוכיחו כי סוגים רבים וירוס נמצאים במי שתייה סביבתיים ובעולם 1-6. השימוש בם בשילוב עם רצף של שברים הגנומי מוגברים במהלך חקירות פרוץ מחלה סיפק ראיות להעברת נגיף המועברות במים, כפי שהם הראו כי הנגיף נמצא במי השתייה הוא זהה לסככה שעל ידי חולי פרוץ 7-10.
שני qPCR וRT-qPCR הם כלים בריאות ציבור שימושיים. לדוגמא, נתונים ממחקרים שנערכו על ידי הסוכנות להגנת הסביבה בארה"ב (EPA) הראו קשר חזק להיותמדידות מחוון tween ידי qPCR והשפעות בריאותיות במים ופנאי. כתוצאה מכך, קריטריוני איכות מים קלים של EPA הסופיים 2,012 כוללים שיטת qPCR לניטור חופי פנאי 11,12. Borchardt ועמיתים מצאו גם קשר חזק בין קיבה ומעיים חריפים בקהילות באמצעות מי תהום ווירוס שלא טופלה בתהום כפי שנמדדו על ידי RT-qPCR 1.
מטרת מאמר זה היא לתאר את רכיב assay המולקולרי של 13,14 EPA השיטה 1,615. assay זה משתמש RT-qPCR לספק הערכה כמותית של enterovirus ועותקי norovirus הגנומי (GC) לליטר המבוסס על הנפח המקורי של מי שתייה או הסביבתי עבר סינון electropositive. סקירה של ההליך המולקולרי מוצגת באיור סעיף 1. פרוטוקול פרטים 1 הנהלים להכנת העקומה סטנדרטית. תקנים אלה הוכנו ממגיבים המכיל RNעותק של רצף המטרה לכל קבוצות פריימר / הבדיקה. סעיף 2 מתאר את הליך ריכוז שלישוני. סעיף 3 נותן את ההליך להפקת RNA מדגימות מים ושליטה מרוכזות. RNA מכל מדגם בדיקה הפוך עיבד באמצעות מבחני בשלושה עותקים ופריימרים אקראיים לראש השעתוק (סעיף 4). CDNA מכל תגובת שעתוק לאחור מחולק לחמישה מבחני נפרדים וירוס ספציפי שמנותחים בשלושה עותקים על ידי qPCR (סעיף 5; איור 2). Assay משתמש פריימרים ובדיקות מהספרות המדעית (טבלת 1) שנועדו לזהות enteroviruses רב וnoroviruses ומגיב מכיל צהבת G RNA לזהות דגימות בדיקה שמעכבות עד 15 RT-qPCR.
Protocol
הערה: גיליונות נתונים השתמשו כדי לעקוב אחר כל השלבים של הפרוטוקול; ניתנים גיליונות דוגמא נתונים בלוחות החומרים המשלימים S2-S4.
1. תקן Curve הכנה
- הכן מלאי עבודה של מגיב העקומה סטנדרטית (למשל, שריון RNA EPA-1,615) על ידי דילול אותו מהריכוז שסופק על ידי היצרן לריכוז של 2.5 x 10 8 מיליליטר / חלקיקים (2.5 x 10 8 GC / מיליליטר) באמצעות TSM מאגר III. מחלקים את המניה עובדת לaliquots 250 μl באמצעות 1.5 מיליליטר צינורות microcentrifuge וחנות ב -20 ° C.
הערה: ראה שלב פרוטוקול חומרים משלים S1 להוראות המופיעה על מניות הכנת עבודה של וירוס ופלסמידים לשימוש כחומרים כימיים עקומה סטנדרטית חלופיים. - להפשיר אחד או יותר ממניות aliquots עובד. הכן חמישה דילולים סדרתי של פי 10 באמצעות 1.5 מיליליטר צינורות microcentrifuge, נותן ריכוזים של 2.5 x 10 7, 2.5 x 10 6, 2.5 xx x 10 5, 2.5 10 4, ו -2.5 10 3 GC / מיליליטר.
- הכן את הדילול הראשון על ידי הוספת 25 μl של 2.5 x 10 8 GC / מיליליטר המניה עובדת 225 μl של חיץ TSM III. מערבבים במשך 5-15 שניות בעזרת מערבל מערבולת.
- הכן את הדילול הבא על ידי הוספת 25 μl של הדילול מוכן בשלב 1.2.1 225 μl של חיץ TSM III. מערבבים שוב ולהמשיך בתהליך דומה להכנת שלושה דילולים של פי 10 הבאים.
- חלץ את RNA ממניות עקומת סטנדרט עבודה ודילולי חמישה באמצעות ההליך מייד בסעיף 3.
2. ריכוז תיכונית
- הכן רכז צנטריפוגלי (30,000 הפסקת משקל מולקולרית) עבור כל דגימה שנאסף על ידי הוספה לפחות 10 מיליליטר של 1x PBS, 0.2% אלבומין בסרום שור (BSA) לתא המדגם העליון. להבטיח פתרון שמלא את תא ריכוז ערוץ הדק, ולאחר מכן להחזיק O / N ב 4 ° C.
- מחק את הנוזל. יש לשטוף את רכז פעם אחת עם לפחות 10 מיליליטר של מים כיתה מגיב סטרילי כדי להסיר BSA עודף ואז להשליך את המים.
- להוסיף סכום של תרכיז מים משני מכל מדגם בדיקה שווה לS, הנפח לדוגמא Assay לריכוז צנטריפוגלי נפרד.
- לחשב S באמצעות משוואת 1,
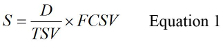
כאשר D היא הנפח של דגימת מים המקורית assayed, TSV הוא נפח המדגם וFCSV הוא נפח הדוגמה מרוכז הסופי. ראה S2 חומרים נוספים לדוגמא של חישוב ס
- לחשב S באמצעות משוואת 1,
- צנטריפוגה מדגם זה מבחן ב3,000-6,000 XG ב 4 מעלות צלזיוס בהרוטור דלי מתנדנד במשך 20 - 30 דקות. בדוק את הנפח בתא ריכוז ערוץ הדק.
- אם הנפח גדול מ -400 μl, צנטריפוגות שוב במשך 20 דקות או יותר. המשך צנטריפוגה עד המדגם בהתא ריכוז ערוץ דק דואר צומצם פחות מ -400 μl. אל תסיר את supernatant.
- שטוף את הצדדים של רכז צנטריפוגלי עם 1 מיליליטר של פוספט 0.15 מ 'נתרן סטרילי, pH 7-7.5 להגדיל התאוששות וירוס. צנטריפוגה שוב ב3,000-6,000 XG ו -4 ° C עד המדגם צומצמה פחות מ -400 μl. חזור על שלב זה לשטוף פעם אחת נוספת.
- באמצעות micropipette 100-200 μl, למדוד בזהירות ולהעביר כל דגימה מרוכזת לצינור microcentrifuge 1.5 מיליליטר (כלומר, להעביר 200 μl לצינור microcentrifuge ולאחר מכן למדוד את התרכיז שנותר על ידי התאמת micropipette עד הנוזל שנותר ניתן להסיק לגמרי לתוך קצה pipettor). להוסיף 0.15 M פוספט נתרן, pH 7-7.5, להתאים את הנפח הסופי עד 400 ± 2 μl.
- לחלץ חומצות גרעין מייד על ידי שתמשיך לשלב 3. החזיקו כל דגימות מרוכזות שלא ניתן processeד מייד בC ° 4 ללא יותר מ -24 שעות.
בידוד 3. חומצות גרעין
- הוסף 200 μl של חיץ חילוץ מוכן כפי שתואר בשלב 3.3 ו -200 μl של התרכיז שיישונים מכל מדגם בדיקה מהשלב 2.5 או דילול עקומה סטנדרטי משלב 1.3 ללהפריד כותרת 1.5 מיליליטר צינורות microcentrifuge. להקפיא נותרו תרכיזים שלישוני או מתחת -70 ° C. לחלץ את חומצות גרעין מכל מדגם על פי הוראות יצרן ערכת חילוץ חומצות הגרעין לפרוטוקול הספין לדגימות דם עם החריגים הבאים.
- אל תוסיף פרוטאז לדגימות מים או להשתמש בחיץ החילוץ מצורף לערכת חילוץ חומצות הגרעין.
- הכן חיץ חילוץ עם RNA המוביל
- להוסיף 310 μl של חיץ דילול RNA המוביל לבקבוקון המכיל 310 מיקרוגרם של רנ"א המוביל. מערבבים להמסה ולאחר מכן מתחלק ל6 aliquots המכיל כ -50 μl. חנות ב -20מעלות צלזיוס.
- להוסיף 28 μl של רנ"א המוביל המופשר לכל מיליליטר של חיץ החילוץ להשיג ריכוז RNA המוביל של 0.027 מיקרוגרם / μl. השתמש בחיץ החילוץ-תוקן RNA המוביל במקום שמסופק עם ערכת החילוץ.
- הכן פתרון אדון של חיץ elution ידי הוספת ribonuclease (RNase) מונעי לריכוז סופי של 400 יחידות / מיליליטר למאגר elution מסופק עם ערכת החילוץ.
- Elute RNA מחומצות הגרעין מחייבות טור ספין על ידי הצבת 50 μl של חיץ elution עם מעכב RNase לטור. חכה דקות 1, ואז צנטריפוגות ב6,000 XG דקות 1 ב RT.
- חזור על שלב 3.4.1 ואז להסיר ולסלק את העמודה.
- הכן aliquots של תמציות RNA מהשלב 3.4.2. הכן 6 aliquots של מניית עקומת סטנדרט עבודה וכל דילול עקומה סטנדרטי המכיל לפחות 15 μl כל אחד. הכן 4 aliquots של כל דגימות RNA האחרות המכילות לפחות22 μl כל אחד. אחסן aliquot אחד של כל פקדי מדגם ועקומה סטנדרטית ב 4 ° C אם הם יכולים להיות מעובד על ידי שעתוק לאחור תוך 4 שעות; אחרת, לאחסן את כל aliquots או מתחת -70 ° C.
4. הפוך תמלול (RT)
- הכן 100 מיקרומטר פתרונות מניות של כל צבע יסוד oligonucleotide ובדיקה המפורט בטבלה 1 על ידי הוספת נפח מים בדרגת PCR לכל בקבוקון באמצעות סכום (במיקרוליטר) שווה לעשר פעמים מספר nanomoles (nmol) של oligonucleotide הנוכחי ב בקבוקון (כפי שמוצג על תווית הבקבוקון או בדף המפרט של היצרן (לדוגמא, resuspend פריימר המכיל 36.3 nmol ב363 μl). מערבבים להמסה.
- לדלל את 100 מיקרומטר הפתרונות 1:10 עם מים הכיתה PCR להכין 10 מיקרומטר עובדים פתרונות.
- הכן RT מיקס מאסטר 1 ו -2 בחדר נקי תוך שימוש במדריך בטבלה 2. פיפטה 16.5 μl של RT master מיקס 1 לכל צלחת PCR גם באמצעות פיפטה רבה.
- הפשרה, אם קפוא, תמציות חומצות גרעין מכל מדגם שדה והמעבדה מטריקס מחוזק לדוגמא (LFSM; כלומר, מדגם מטריקס מים זרע).
- לדלל כל שדה ומדגם LFSM 1: 5 ו01:25 במאגר elution המכיל 400 / מיליליטר יחידות של מעכב RNase.
- הפשרה, אם קפוא, אבל לא לדלל מעבדה מבוצרת בלנק (LFB; כלומר, בקרת איכות חיובית באמצעות מים כיתה מגיב זרע), המעבדה מגיב בלנק (LRB; כלומר, שליטה שלילית איכות שימוש במי כיתה מגיב), הערכת ביצועים (PE ; כלומר, דגימות מים כיתה מגיב זרע המשמשות להערכת ביצועי מעבדה לפני תחילת מחקר), מבחן ביצועים (PT, כלומר, דגימות מים כיתה מגיב זרע עם titers ידוע לאנליסט המשמשים להערכת ביצועי מעבדה במהלך מחקר ), שליטה אצווה NA שלילי חילוץ, או RNA שחולץ מסט העקומה הסטנדרטי.
- הנח 6.7 μl של רנ"א מכל מדגם בדיקה, בקרה, ועקומה סטנדרטית לתוך בארות צלחת PCR נפרדות, באמצעות בארות בשלושה עותקים עבור דגימות בדיקה ובקרה ולשכפל בארות לעקומות סטנדרטיים (ראה איור S1 לדוגמא צלחת RT).
- הנח 6.7 μl של חיץ elution לתוך בארות צלחת PCR נפרדות ללא שולט תבנית (NTC). כולל 2-8 NTC לכל צלחת RT, באמצעות שני למדגם הראשון ולאחר מכן עוד שני לכל רביעי מדגם נוסף.
- להפיץ חילוץ השלילי ובקרות NTC לאורך הצלחת.
- חותם את צלחת PCR עם אוטם צלחת עמיד בחום. מערבבים את הדגימות למשך 5-10 שניות ואז צנטריפוגות ב≥ 500 בקצרה XG.
- דגירה את הצלחת למשך 4 דקות ב -99 מעלות צלזיוס ולאחר מכן לקרר במהירות עד 4 מעלות צלזיוס בCycler תרמית. צנטריפוגה שוב ב≥ 500 בקצרה XG.
- מוציא בזהירות את חותם הצלחת ולאחר מכן להוסיף 16.8 μl של RT מיקס מאסטר 2 היטב כל אחד. Seאל הצלחת שוב עם אוטם צלחת עמידה בחום, ואחרי הערבוב וצנטריפוגה קצרה ב≥ 500 x גרם.
- מניחים את הצלחת בCycler תרמית ולהפעיל במשך 15 דקות במהירות של 25 מעלות צלזיוס, ואחריו 60 דקות ב 42 מעלות צלזיוס, 5 דקות ב -99 מעלות צלזיוס, ולאחר מכן על ידי מחזור אחיזת 4 ° C.
- תהליך באופן מיידי או בתוך 8 שעות על ידי qPCR (שלב 5), או דגימות חנות או מתחת -70 ° C עד שהם יכולים להיות מעובד. חנות דגימות שיכולים להיות מעובד בתוך 8 שעות על 4 מעלות צלזיוס.
5. בזמן אמת PCR (qPCR)
- לקבוע את הצהבת הממוצעת ערך G CQ לכל מנה של מגיב G צהבת לפני הפעלת כל דגימות בדיקה.
- הפעל assay RT באמצעות 10 משכפל מוכן כפי שתואר עבור פקדי NTC (שלב 4.1.1). הפעל את assay G qPCR הצהבת כמתואר להלן (שלבי 5.2 ל5.5.3). לחשב את ערך CQ הממוצע של 10 חזרות.
- התאם את צהבת כמות G מגיב בRT מיקס מאסטר 1 (טבלת 2), במידת צורך, לקבלת ערך CQ ממוצע בין 25 ל 32 יחידות. לפצות את הסכום שגויס או הוריד על ידי שינוי נפח מים הוסיף לשמור על הנפח הסופי RT מיקס מאסטר 1 ב16.5 μl לassay.
- לאשר את התאמות על ידי חזרה על שלבי 5.1.1 ל5.1.2 ושינוי טבלה 2 כדי לשקף את הכמויות המותאמת.
- הכן תערובת אב qPCR בחדר נקי באמצעות המדריכים בטבלה 3 לenterovirus, לוח 4 ולוח 5 לgenogroup norovirus אני, לוח 6 לgenogroup norovirus השני, לוח 7 לnorovirus עכברי (V genogroup norovirus), ולוח 8 לצהבת ג 'מיקס כל תערובת אמן ואז צנטריפוגות ב≥ 500 בקצרה XG.
- מוסיף את תערובת אב PCR לבארות המתאימות צלחת תגובה אופטית שכותרתו, באמצעות 14 μl לכל גם וצלחות נפרדות לכל assay qPCR (ראה איור S2 לפריסה אפשרית עבור assay qPCR מבוסס על פריסת RT באיור S1).
- להפשיר את צלחת RT משלב 4.8 ב RT, אם קפוא. לערבב בעזרת מערבל צלחת ואז צנטריפוגות ב≥ 500 בקצרה XG.
- לוותר 6 μl של cDNA המתאים לבארות המתאימות של צלחת התגובה האופטית. מערבבים את הדגימות בצלחת התגובה האופטית וצנטריפוגות ב≥ 500 בקצרה XG.
- הפעל את צהבת assay G qPCR בתחום חי ומדולל ודגימות LFSM לפני הפעלת כל מבחני qPCR האחרים. השתמש בדילול הנמוך ביותר של שדה או מדגם LFSM שהוא <ערך 1 CQ גדול מאומרים צהבת ערך G CQ למבחני qPCR enterovirus וnorovirus.
- להגדיר את תוכנת Cycler תרמית PCR כמו על פי הוראות היצרן. זהה את דגימות עקומה סטנדרטי כסטנדרטים ולכל דילול עקומה סטנדרטי, הזן את ערכי עותק הגנומי מוצגים בטבלה 9.
- הפעל את הצלחת בCycler תרמית PCR כמו 10 דקות ב 95 מעלות צלזיוס, ואחריו 45 מחזורים של 15 שניות על 95 מעלות צלזיוס ודקות 1 ב 60 ° C.
- לקבוע אם כל עקומה סטנדרטית עומדת בערכים המקובלים הניתנים בלוח 10. ראה סעיף חומרים משלימים S3 לדוגמאות.
- לחשב את סטיית התקן הכוללת (סטייתתקן) לעקומה סטנדרטית באמצעות משוואת 2,
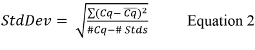
שם CQ הוא הערך המדווח עבור כל לשכפל עקומה סטנדרטית, CQ הוא ממוצע הערך עבור כל קבוצה של משכפל, #Cq הוא המספר הכולל של ערכי CQ לכל משכפל השליטה הסטנדרטי שיש לי ערכים חיוביים (כלומר, לא נקבע), ו# מחלות מין הוא מספר הפקדים סטנדרטיים שיש ערכים חיוביים. - אם תוכנת Cycler תרמית כמותי PCR לא לחשב את השיפוע לכל עקומה סטנדרטית, לחשב את השיפוע באמצעות משוואת 3,60;
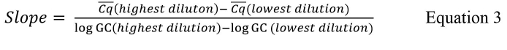
שם CQ הוא הערך הממוצע של דילולים הגבוהים ביותר והנמוכים ביותר בשימוש ולהיכנס GC הוא היומן של עותק הערך הגנומי לדילולים הגבוהים ביותר והנמוכים ביותר בשימוש מלוח 9. - לחשב את ערך R 2 באמצעות 4 משוואה.
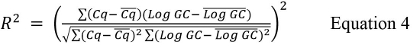
שם CQ הוא ממוצע של כל ערכי CQ והתחבר GC הוא ערך יומן GC הממוצע עבור כל לשכפל. - לחשב את יעילות% באמצעות המשוואה 5:
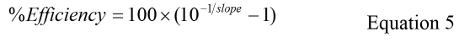
- לחשב את סטיית התקן הכוללת (סטייתתקן) לעקומה סטנדרטית באמצעות משוואת 2,
- רשום את ערכי GC מחושבים על ידי תוכנת Cycler תרמית לכל דגימות הבדיקה מבוססות על עקומות סטנדרטיים שעומדות בקריטריונים שנקבעו בלוח 10 וערכי GC הממוצעים עבור כל דגימה. שידור חוזר כל דגימות עם עקומות סטנדרטיים שאינו עומדים בקריטריונים ב לוח 10 או שבו כל פקדים שליליים (LRB, שליטת חילוץ שלילית אצווה NA, או NTC) הן חיוביות. לעבד מחדש כל דגימות שאינן עומדים בקריטריונים או בקרות חיוביות כוזבות בשידור החוזר.
- לקבוע GC לליטר (L GC) עבור כל דגימת בדיקה באמצעות משוואת 6:
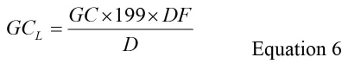
שם GC הוא מספר העותק הגנומי הממוצע מהצעד 5.7, הגורם '199' הוא הגורם לדילול הכולל להפחתת הנפח המתרחשות במהלך הריכוז שלישוני, הפקת RNA וצעדי RT-qPCR, DF הוא הגורם לדילול שמפצה על עיכוב , ו- D הוא הנפח של דגימת מים המקורית assayed בליטרים. ראה S4 סעיף חומרים נוספים לדוגמא של חישוב L GC. - לחשב את GC הכולל של LFB וLRB דגימות על ידי הכפלת ערך GC הממוצע משלב 5.5 על ידי 199 וחלוקת ב -0.3.
תוצאות
התאוששות וירוס בסך הכל נקבעה באמצעות שדה מותאם ודגימות מים קרקע LFSM. בסך הכל שבע קבוצות מדגם נותח באמצעות שתי מערכות שנאספו בהזדמנויות נפרדות משלושה מפעלים ציבוריים טיפול, וקבוצת מדגם אחד שנאסף מהבאר הפרטית. רמות זרע לדגימות LFSM היו 3 x 10 6 MPN של סבין נגיף הפוליו סרוטיפ 3 ו -5 x 10 6 PFU של norovirus עכברי. norovirus Murine שימש כפונדקאי בהערכת השיטה בשל חוסר מניות norovirus אנושיים עם ריכוז וירוס מספיק לדגימות LFSM. לדגימות מי תהום התאוששות נגיף הפוליו הממוצעת הייתה 20%, עם סטיית תקן של 2%, ואילו 14 אומר התאוששות norovirus עכברית הייתה 30%, עם סטיית תקן של 3% (איור 3). מדגם תהום שדה הרגיל לכל LFSM לא היה enterovirus או norovirus לזיהוי.
דגימות LFB וLRB נמדדו באמצעות ואט כיתה מגיב זרע וunseeded אה. כל LRB הדגימות היו שליליות (מידע לא מוצג). התאוששות נגיף הפוליו בממוצע 44% עם סטיית תקן של 1% (איור 3), ואילו התאוששות norovirus עכברית בממוצע 4% עם סטיית תקן של 0.5%.
RT-qPCR מחייב השימוש בחומרים כימי עקומה סטנדרטית נאותים. איור 4 מראה עקומת סטנדרט אופיינית לenterovirus וnorovirus Gii. עקומת Gii norovirus עומדת בקריטריונים סטנדרטי ביצועים עקום (לוח 10) עם R 2 ערך של .9987, סטיית תקן כוללת של 0.14, ויעילות 101%. Norovirus GIA ועקומות GIB (לא מוצג) הם כמעט זהים לזה של Gii norovirus. עקומת enterovirus עומדת בקריטריוני ביצועי שיטה עם R 2 ערך של .9874, סטיית תקן כוללת של 0.58, ויעילות 103%, אבל יש כמאה פי פחות רגישות ובכך להגביל את גילוי גבוה מעקומות norovirus.
דף = "תמיד"> 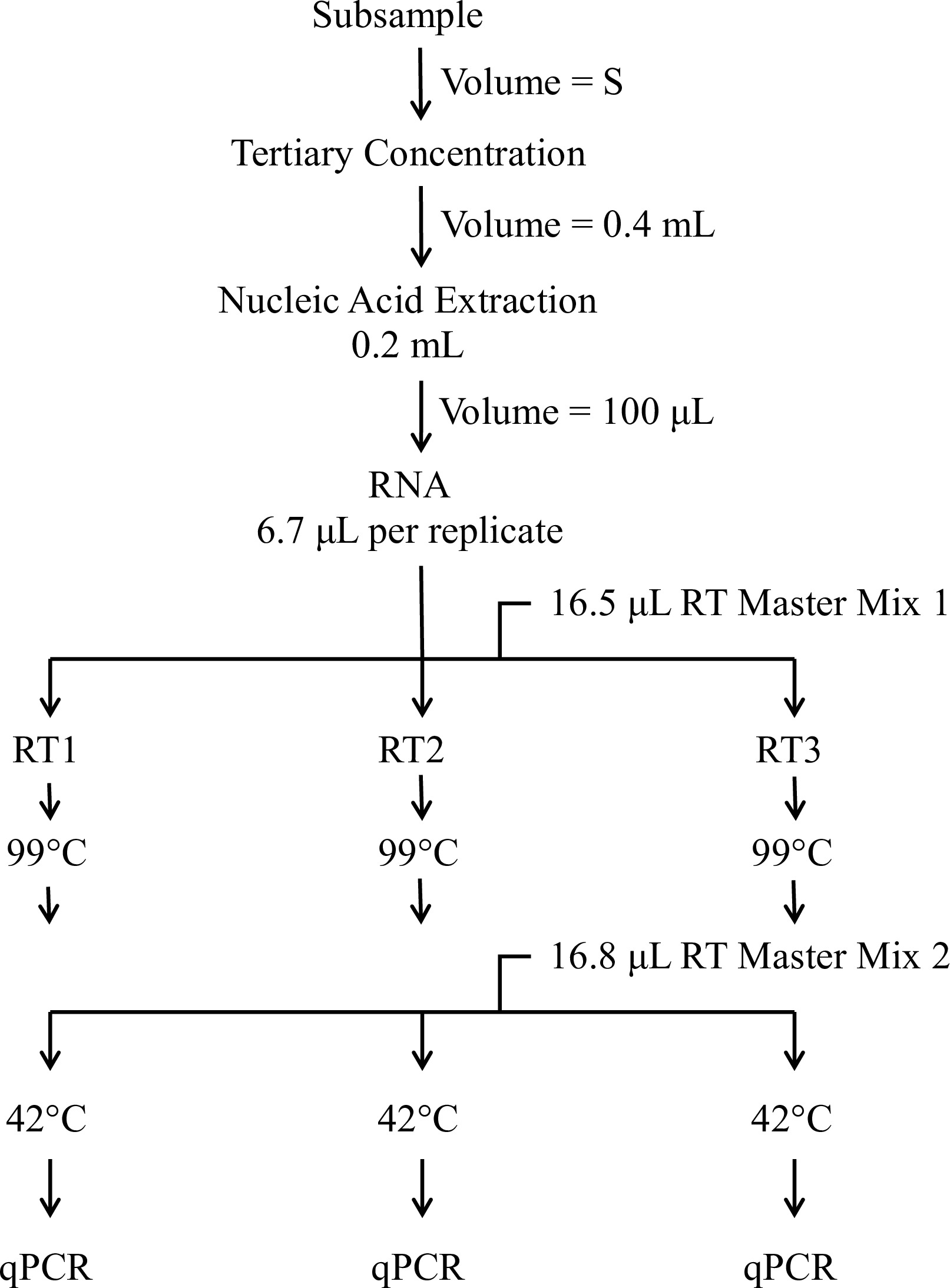
איור 1. סקירה של ההליך המולקולרי. ההליך המולקולרי כולל ריכוז מדגם נוסף מעבר לזה שבוצע למדידת וירוס מדבק, חילוץ של חומצות גרעין, שני שלבים הפוכים שעתוק פרוטוקול (RT), וPCR כמותית (QPCR). ההתחלה (S) הנפח מייצג חלק מוגדר שיטה של דגימת המים המקורית.
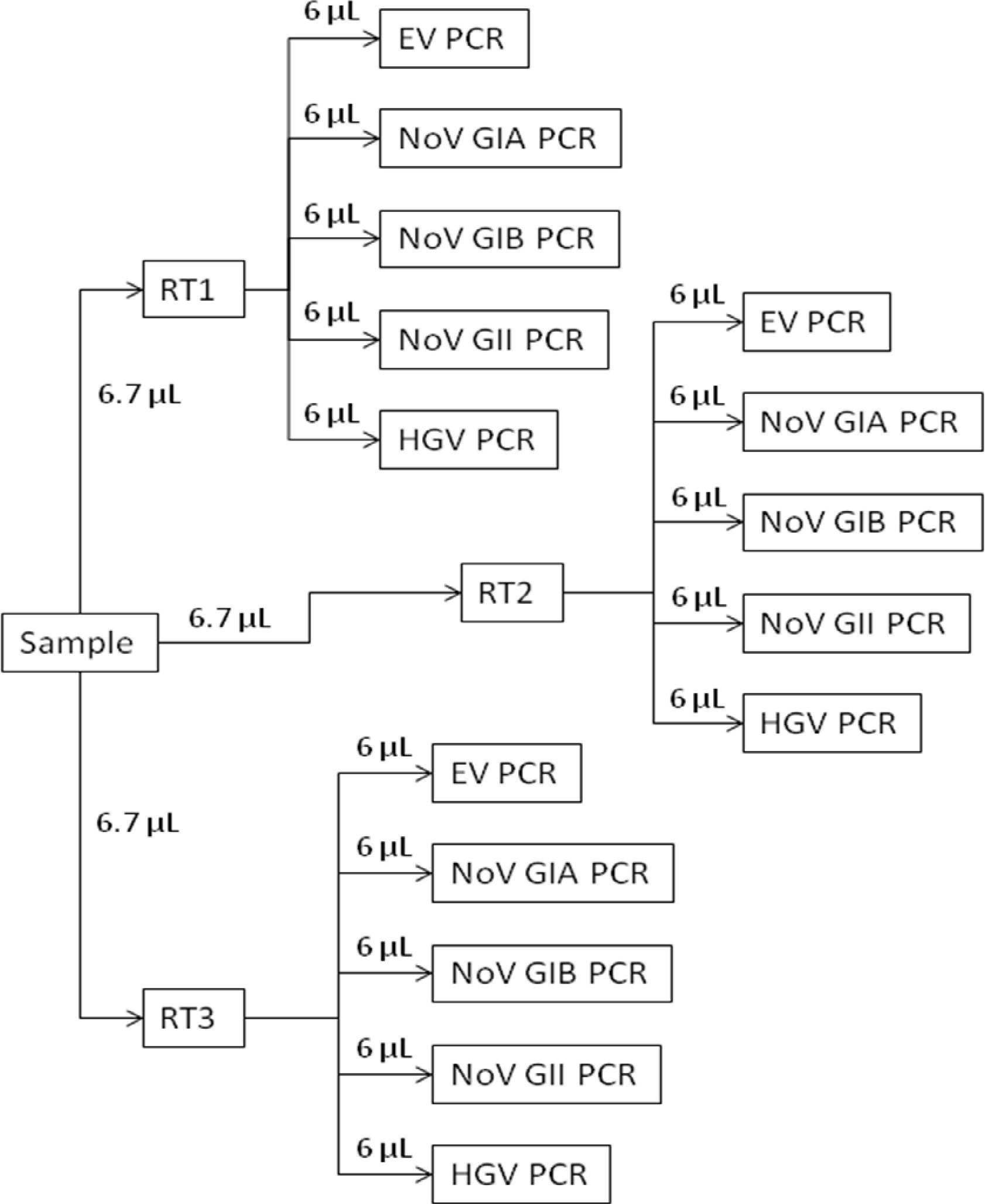
איור 2. סכמטית סקירת RT-qPCR. כל RNA מדגם בדיקת חילוץ הפוך עיבד באמצעות מבחני שלושה עותקים (RT1, RT2, וRT3). CDNA מכל אחד מהמבחנים בשלושה עותקים RT אז הוא ניתח לוירוסים ספציפיים באמצעות enterovirus הנפרד (EV PCR), genogroup norovirus אני (נו GIA PCR ונו GIB PCR), genogroup norovirus השני (NOV Gii PCR), וצהבת G (HGV PCR) מבחני.
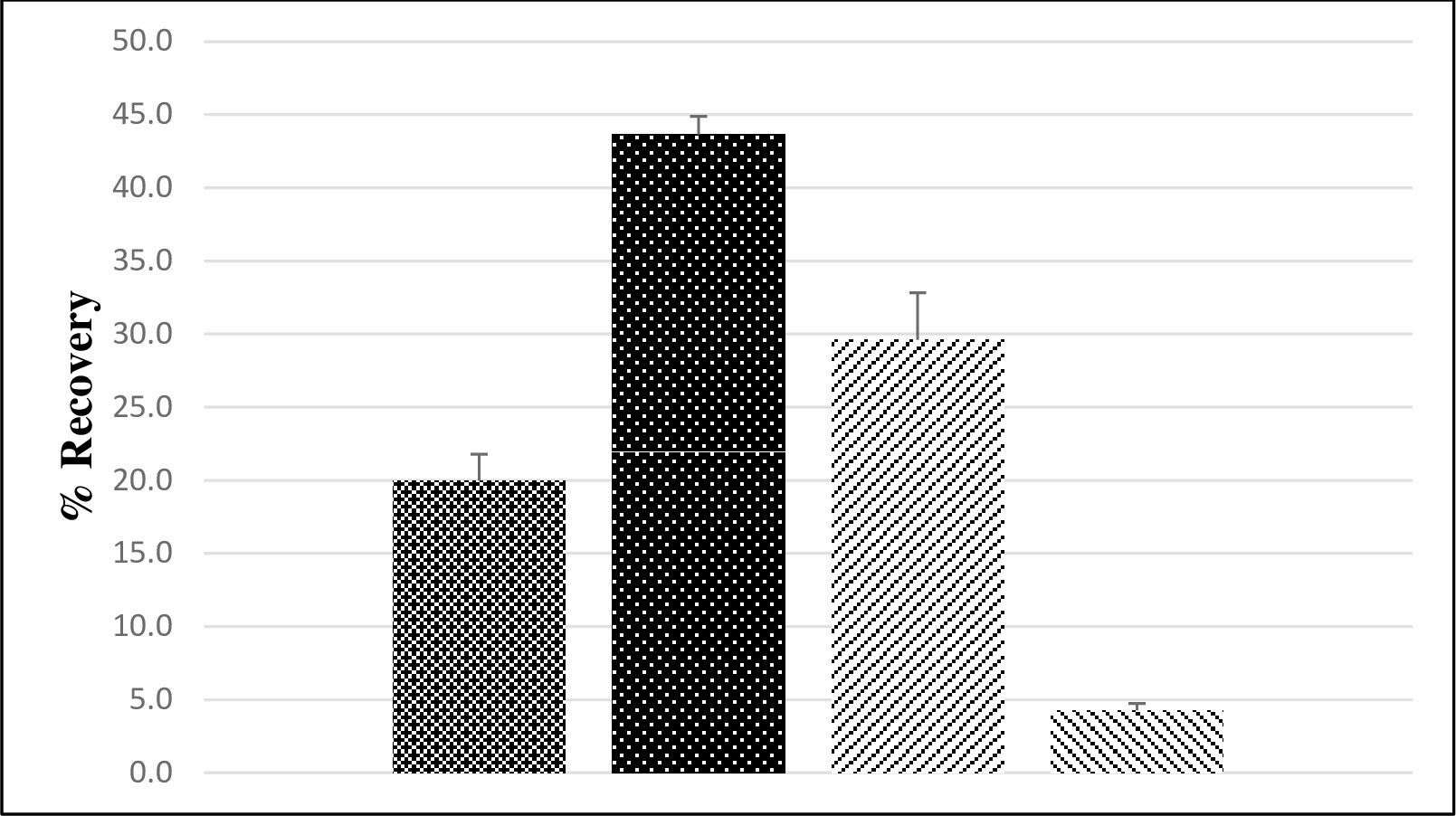
איור 3. ממוצע נגיף הפוליו וMurine norovirus שחזור (%) מקרקע והמים. התאוששות אחוזים הממוצע מגיב-כיתה מוצג לנגיף פוליו מהקרקע (  ; n = 7) ומהכיתה מגיב (
; n = 7) ומהכיתה מגיב (  ; n = 12) מים ולnorovirus עכברי מהקרקע (
; n = 12) מים ולnorovirus עכברי מהקרקע (  ; n = 7) ומהכיתה מגיב (
; n = 7) ומהכיתה מגיב (  ; n = 12) מים (1), שבו "n" הוא מספר דגימות מים נפרדים מעובד. ברים שגיאה מייצגים סטיית התקן.
; n = 12) מים (1), שבו "n" הוא מספר דגימות מים נפרדים מעובד. ברים שגיאה מייצגים סטיית התקן.

איור 4. Enterovirus וnorovirus Gii תקן עקום. עקומות סטנדרטי אופייניות לenterovirus וnorovirus Gii מוצגים. נוסחות נותנים מדרון וR 2 ערכים עבור כל עקומה מחושבות על ידי Cycler התרמית.
1. קובץ נוסף אנא לחץ כאן כדי להוריד את הקובץ.
| קבוצת וירוס | פריימר / בדיקה שם (1) | רצף (2) | הַפנָיָה | |
| Enterovirus | ||||
| EntF (EV-L) | CCTCCGGCCCCTGAATG | 20 | ||
| Entr (EV-R) | ACCGGATGGCCAATCCAA | 20 | ||
| EntP (EV-בדיקה) | 6FAM-CGGAACCGACTACTTTGGGTGTCCGT-טמרה | 21 | ||
| Norovirus GIA | ||||
| NorGIAF (JJV1F) | GCCATGTTCCGITGGATG | 22 | ||
| NorGIAR (JJV1R) | TCCTTAGACGCCATCATCAT | 22 | ||
| NorGIAP (JJV1P) | 6FAM-TGTGGACAGGAGATCGCAATCTC-טמרה | 22 | ||
| Norovirus GIB | ||||
| NorGIBF (QNIF4) | CGCTGGATGCGNTTCCAT | 23 | ||
| NorGIBR (NV1LCR) | CCTTAGACGCCATCATCATTTAC | 23 | ||
| NorGIBP (NV1LCpr) | 6FAM-TGGACAGGAGAYCGCRATCT-טמרה | 23 | ||
| Norovirus Gii | ||||
| NorGIIF (QNIF2d) | ATGTTCAGRTGGATGAGRTTCTCWGA | 25 | ||
| NorGIIR (COG2R) | TCGACGCCATCTTCATTCACA | 25 | ||
| NorGIIP (QNIFS) | 6FAM-AGCACGTGGGAGGGCGATCG-טמרה | 25 | ||
| Norovirus GV | ||||
| MuNoVF1 | AGATCAGCTTAAGCCCTATTCAGAAC | 14 | ||
| MuNoVR1 | CAAGCTCTCACAAGCCTTCTTAAA | 14 | ||
| MuNoVP1 | ויק-TGGCCAGGGCTTCTGT-מג"ב | 14 | ||
| צהבת G | ||||
| HepF (5'-NCR פריימר קדימה) | CGGCCAAAAGGTGGTGGATG | 19 | ||
| HepR (5'-NCR פריימר ההפוכה) | CGACGAGCCTGACGTCGGG | 19 | ||
| הפ (Probe G TaqMan צהבת | 6FAM-AGGTCCCTCTGGCGCTTGTGGCGAG-טמרה | 1 | ||
טבלה 1. Primers וTaqMan בדיקות לאיתור וירוסים על ידי RT-qPCR.
(1) פריימר השיטה 1,615 שמות ובדיקה שלוש האותיות הראשונות של שם הווירוס בשרשור לF, R, או P לקדימה, אחורה, ובדיקה. Genogroup norovirus מיועד על ידי הוספת GI וGii לשמות. שתי קבוצות פריימר GI norovirus גם נבדלות באמצעות A ו- B. פריימר ובדיקת שמות מהאזכור הראשוני ניתנות בהורהheses.
(2) הנטייה של רצפי פריימר ובדיקה היא 5 'ל 3'. מדדי הבסיס המנוונים הבאים משמשים: N-תערובת של כל ארבעת נוקלאוטידים; R-+ G; Y-T + C; W-+ T; ואני-inosine.
| מַרכִּיב | נפח לכל תגובה (μl) (2) | ריכוז סופי | נפח למיקס מאסטר (μl) (3) |
| RT מיקס מאסטר 1 | |||
| פריימר אקראי | 0.8 | 10 ng / μl (ג. 5.6 מיקרומטר) | 84 |
| צהבת G שריון RNA (4) | 1 | 105 | |
| מים כיתה PCR | 14.7 | 1,543.5 | |
| לטל | 16.5 | 1,732.5 | |
| RT מיקס מאסטר 2 | |||
| 10x PCR הצפת השני | 4 | 10 מ"מ טריס, pH 8.3, 50 מ"מ KCl | 420 |
| 25 מ"מ MgCl 2 | 4.8 | 3 מ"מ | 504 |
| dNTPs 10 מ"מ | 3.2 | 0.8 מ"מ | 336 |
| 100 מ"מ DTT | 4 | 10 מ"מ | 420 |
| RNase מונעי | 0.5 | 0.5 יחידות / μl | 52.5 |
| עילי השני RT | 0.3 | 1.6 יחידות / μl | 31.5 |
| סה"כ | 16.8 | 1,764 | |
טבלת 2. RT מיקס מאסטר 1 ו -2 (1).
(1) הכן RT מאסטר תערובות בCLחדר EAN, כלומר, חדר שבו נהלים מולקולריים ומיקרוביולוגית אינם מבוצע.
(2) הסופי RT נפח assay הוא 40-μl.
(3) הכרכים להראות מבוססים על 105 מבחני. זה מספיק לצלחת PCR 96-היטב עם מבחני נוספים המתווספים לחשבון להפסדים. הסכום עשוי להיות מדורגים למעלה או למטה בהתאם למספר הדגימות ובקרות שינותח.
(4) לקבוע את כמות מגיב G צהבת לכלול בRT מיקס מאסטר 1 כפי שתואר בשלב S4 חומרים משלימים.
| מַרכִּיב | נפח לכל תגובה (μl) (2) | ריכוז סופי | נפח למיקס מאסטר (μl) (3) |
| 2x LightCycler 480 בדיקות מיקס מאסטר | 10 | קניינית | 1,050 |
| התייחסות רוקסצבע (4) | 0.4 | 0.5 מ"מ | 42 |
| מים כיתה PCR | 1 | 105 | |
| EntF 10 מיקרומטר | 0.6 | 300 ננומטר | 63 |
| 10 מיקרומטר entr | 1.8 | 900 ננומטר | 189 |
| 10 מיקרומטר EntP | 0.2 | 100 ננומטר | 21 |
| סה"כ | 14 | 1,470 |
טבלה 3. PCR מיקס מאסטר לEnterovirus (EV) Assay (1).
(1) הכן את כל תערובות מאסטר PCR בחדר נקי.
(2) הסופי qPCR נפח assay הוא 20 μl.
(3) הכרכים להראות מבוססים על 105 מבחני. זה מספיק לצלחת PCR 96-היטב עם מבחני נוספים המתווספים לחשבון להפסדים. הסכום עשוי להיות מדורגים למעלה או למטה בהתאם למספר הדגימות ובקרות שתהיינהלהיות מנותח.
(4) תחליף המים PCR כיתה למגיב זה בעת שימוש במכשירים שאינם דורשים את זה.
| מַרכִּיב | נפח לכל תגובה (μl) (2) | ריכוז סופי | נפח למיקס מאסטר (μl) (3) |
| 2x LightCycler 480 בדיקות מיקס מאסטר | 10 | קניינית | 1,050 |
| צבע רוקס התייחסות (4) | 0.4 | 0.5 מ"מ | 42 |
| מים כיתה PCR | 1.4 | 147 | |
| 10 מיקרומטר NorGIAF | 1 | 500 ננומטר | 105 |
| 10 מיקרומטר NorGIAR | 1 | 500 ננומטר | 105 |
| 10 מיקרומטר NorGIAP | 0.2 | 100 ננומטר | 21 |
| סה"כ | 14 | 1,470 |
לוח 4. PCR מיקס מאסטר לnorovirus GIA (נו GIA) Assay (1).
ראה טבלה 3 להערות שוליים (1) - (4).
| מַרכִּיב | נפח לכל תגובה (μl) (2) | ריכוז סופי | נפח למיקס מאסטר (μl) (3) |
| 2x LightCycler 480 בדיקות מיקס מאסטר | 10 | קניינית | 1,050 |
| צבע רוקס התייחסות (4) | 0.4 | 0.5 מ"מ | 42 |
| מים כיתה PCR | 0.3 | 31.5 | |
| 10 מיקרומטר NorGIBF | 1 | 500 ננומטר | 105 |
| 10 מיקרומטר NorGIBR | 1.8 | 900 ננומטר | 189 |
| 10 מיקרומטר NorGIBP | 0.5 | 250 ננומטר | 52.5 |
| סה"כ | 14 | 1,470 |
לוח 5: PCR מיקס מאסטר לnorovirus GIB (נו GIB) Assay (1).
ראה טבלה 3 להערות שוליים (1) - (4).
| מַרכִּיב | נפח לכל תגובה (μl) (2) | ריכוז סופי | נפח למיקס מאסטר (μl) (3) |
| 2x LightCycler 480 בדיקות מיקס מאסטר | 10 | קניינית | 1,050 |
| צבע רוקס התייחסות (4) | 0.4 | 0.5 מ"מ | 42 |
| מים כיתה PCR | 0.3 | 31.5 | |
| 10 מיקרומטר NorGIIF | 1 | 500 ננומטר | 105 |
| 10 מיקרומטר NorGIIR | 1.8 | 900 ננומטר | 189 |
| 10 מיקרומטר NorGIIP | 0.5 | 250 ננומטר | 52.5 |
| סה"כ | 14 | 1,470 |
לוח 6. PCR מיקס מאסטר לnorovirus Gii (נו Gii) Assay (1).
ראה טבלה 3 להערות שוליים (1) - (4).
| מַרכִּיב | נפח לכל תגובה (μl) (2) | ריכוז סופי | נפח למיקס מאסטר (μl) (3) |
| 2x LightCycler 480 בדיקות מיקס מאסטר | 10 | קניינית | 1,050 |
| צבע רוקס התייחסות (4) | 0.4 | 0.5 מ"מ | 42 |
| מים כיתה PCR | 0.3 | 31.5 | |
| 10 מיקרומטר MuNoVF1 | 1 | 500 ננומטר | 105 |
| 10 מיקרומטר MuNoVR1 | 1.8 | 900 ננומטר | 189 |
| 10 מיקרומטר MuNoVP1 | 0.5 | 250 ננומטר | 52.5 |
| סה"כ | 14 | 1,470 |
לוח 7: PCR מיקס מאסטר לMurine norovirus Assay (1).
ראה טבלה 3 להערות שוליים (1) - (4).
| מַרכִּיב | כֶּרֶךלכל תגובה (μl) (2) | ריכוז סופי | נפח למיקס מאסטר (μl) (3) |
| 2x LightCycler 480 בדיקות מיקס מאסטר | 10 | קניינית | 1,050 |
| צבע רוקס התייחסות (4) | 0.4 | 0.5 מ"מ | 42 |
| מים כיתה PCR | 1.4 | 147 | |
| 10 מיקרומטר HepF | 1 | 500 ננומטר | 105 |
| 10 מיקרומטר HepR | 1 | 500 ננומטר | 105 |
| 10 מיקרומטר הפ | 0.2 | 100 ננומטר | 21 |
| סה"כ | 14 | 1,470 |
לוח 8: PCR מיקס מאסטר לצהבת מהסוג G (HGV) Assay (1).
לִרְאוֹת לוח 3 להערות שוליים (1) - (4).
| ריכוז עקומת סטנדרט | עותקים הגנומי לRT-qPCR Assay (1, 2) |
| 2.5 x 10 8 | 502500 |
| 2.5 x 10 7 | 50,250 |
| 2.5 x 10 6 | 5025 |
| 2.5 x 10 5 | 502.5 |
| 2.5 x 10 4 | 50.25 |
| 2.5 x 10 3 | 5.025 |
9. עותקים רגילים Curve הגנום שולחן.
(1) זהה את בארות עקומה סטנדרטי כתקנים ולמקם את העותקים הגנומי לערכי assay RT-qPCR במקום המתאים בתוכנת thermocycler.
(2) עקומת סטנדרט מקובלת תהיה יעילות של 70% -110%, ערך R 2> 0.97, וסטיית תקן כללית של <0.5 לnorovirus ו< 1.0 לenterovirus.
| קריטריונים | ערך מקובל | |
| Norovirus | Enterovirus | |
| סטיית תקן בסך הכל | <0.5 | <1.0 |
| R 2 | > 0.97 | > 0.97 |
| יְעִילוּת | 70% ל -115% | 70% ל -115% |
שולחן 10. קריטריונים רגילים Curve קבלה (1).
(1) עקומות סטנדרטיות עם יעילות% של 70% -110% מקובלות, אבל ערכים בטווח של 90% -115% הם אידיאליים. ערכים פחות מ -90% עשויים להצביע על שגיאות pipetting או דילול.
| QA רכיב | אומר טווח שחזור (%) | מקדם שונה (%) |
| מעבדה מגיב בלנק; דגימות PT או PE שליליים | 0 | N / A (1) |
| מעבדה מחוזק בלנק; מטריקס דוגמא מחוזק מעבדה | 5-200 | N / |
| דגימות החיוביות PT וPE | 15-175 | ≤130 |
שולחן 11. שיטה 1,615 קריטריוני ביצועים.
(1) לא ישים.
| כְּלֵי תִקְשׁוֹרֶת | הֶרכֵּב |
| 0.15 מ 'פוספט נתרן, pH 7.0-7.5 | הכן פוספט 0.15 מ 'נתרן על ידי המסת 40.2 גרם של נתרן פוספט, dibasic (Na 2 4 HPO · 7H 2 O) בנפח סופי של DH 1 ליטר 2 O. התאם את ה- pH ל7.0-7.5 עם HCl. החיטוי ב 121 מעלות צלזיוס, 15 psi במשך 15 דקות. פתרון חנות פוספט נתרן ב RT עד 12 חודשים. |
| 5% BSA | הכן על ידי המסת 5 גרם של BSA בשל DH 2 א '100 מיליליטר לעקר על ידי עובר את הפתרון דרך פילטר חיטוי 0.2 מיקרומטר. |
| PBS, 0.2% BSA | הכן על ידי הוספת 4 מיליליטר של BSA 5% ל- 96 מיליליטר של PBS. לעקר על ידי עובר את הפתרון דרך פילטר חיטוי 0.2 מיקרומטר. |
| מאגר TSM III | ממיסים 1.21 בסיס g Trisma, 5.84 גר 'NaCl, .203 גרם MgCl 2, ג'לטין Prionex 1 מיליליטר, 3 מיליליטר וMicrocide שלישי ב950 מיליליטר מים כיתה מגיב. התאם את ה- pH 7.0 ולאחר מכן להביא את הנפח הסופי עד 1 ל לעקר על ידי עובר את הפתרון דרך פילטר חיטוי 0.2 מיקרומטר. |
| .525% Hypochlorite נתרן (NaClO) | הכן .525% פתרון NaClO על ידי דילול אקונומיקה 1:10 ב דH 2 O. אחסן פתרונות NaClO .525% בשבוע עד 1 ב RT. |
| טטרתיונט נתרן 1-M (Na 2 S 2 O 3) pentahydrate | הכן פתרון 1 M על ידי המסת 248.2 גרם של Na 2 S 2 O 3 ב1 ליטר של DH 2 א ' Thiosulfate נתרן חנות לתקופה של עד 6 חודשים בRT. |
שולחן 12. טבלת מדיה.
Discussion
מחקרים לאומיים בקנה מידה גדולה של זיהום הנגיפי של מים מקור ושתייה דורשים השימוש במעבדות אנליטיות מרובות. בתנאים אלה יש צורך בשיטה סטנדרטית כדי להבטיח כי הנתונים שנוצרו על ידי מעבדות הרבות הוא דומים. ישנן שיטות רבות שפורסמו מולקולריות לאיתור וירוסים, אבל מעט מאוד שיטות מולקולריות אחידות. EPA שיטה 1,615 היא שיטה סטנדרטית שתוכננה במיוחד עבור זיהוי של enterovirus וnorovirus במטריצות מים על ידי RT-qPCR. שיטות מולקולריות סטנדרטיות זמינות לאיתור וירוסים במזונות (CEN / ISO TS 15,216-1 וCEN / ISO TS 15,216-2; 7 אפריל 2013) 16,17 ויושמו לזיהוי של צהבת וירוס וnorovirus באביב מים 16. כל השיטות סטנדרטי חייבת לכלול בקרת ביצועי איכות וקריטריונים כדי למזער בינלאומי ופנים-וריאציה מעבדה והנתונים חיוביים כוזבים עקב זיהום במעבדה. כדי לצמצם עוד יותר נתונים כוזבים, Eרשות שיטה 1,615 כדלקמן ההדרכה של המשרד לאיכות הסביבה על שיטות מולקולריות, 18 הקובע הפרדת העבודה במהלך עיבוד ודרך אחת זרימת עבודה. הוא כולל G צהבת 1,19 שליטה ונהלים כדי למזער תוצאות שליליות שגויות בשל מעכבים של 15 RT-qPCR פנימיים. היא משתמשת במבחנים כמותיים יחד עם כמויות סטנדרטיות של שני המים שנדגמו והמים ניתחו כך שכל נתוני השדה מתבטאים בעותקים הגנומי לליטר של השדה או שתיית נדגמו מים. למרות שצינור יעיל יחיד (צעד אחד) מבחני RT-PCR זמין מסחרי, בשיטה במכוון משתמשת מבחני נפרדים. זה יש את החסרון של צמצום כמות המדגם שניתן assayed בכל תגובה, אבל נותן גמישות רבה יותר בשימוש בערכות פריימר מרובים. מבחני RT-qPCR מוגבלים על ידי ורק טוב כמו פריימרים ובדיקות בשימוש וסביר לא קבוצת פריימר תהיה לזהות את כל גרסאות הווירוס בתוך קבוצה. סט פריימר enterovirus נבחר בגללהיא פוגעת באזור 5'-שאינו נשמרים קידוד, 20,21 מזהה מגוון רחב של זנים אל enterovirus, ווירוסים שאותרו על ידי זה קשורים להשפעות בריאותיות מצריכה של groundwaters מטופל 1. שתי קבוצות פריימר משמשות לזיהוי של genogroup אני noroviruses 22,23. הראשון נבחר בשל המתאם החזק בין השפעות הבריאותיות בילדים צעירים וזוהה וירוס 1. Genogroup השני אני פריימר להגדיר ולהגדיר פריימר משמש לgenogroup השני noroviruses נבחרו משום שהם מזהים את המגוון הרחב ביותר של זני 24,25.
למרות היתרונות הגדולים של נהלי qPCR וRT-qPCR לאיתור רנ"א הנגיפי במים, יש כמה מגבלות. ראשית, שני חלקיקי הווירוס מדבקים ומידבקים, כוללים אלה מומתים על ידי חומרי חיטוי, יכולים להיות מוגברים על ידי נהלים אלה. התוצאות מצביעות על כך שBorchardt זה פחות בעיה לF groundwaters שלא טופלהאקוויפרים ROM דומים לאלה שביישובים שנבדקו בהשוואה למים עיליים חיטוי 1. לוירוסים culturable בעיה זו ניתן להתגבר באמצעות PCR בשילוב עם תרבות 26,27. הבעיה גם טופלה במשך כמה וירוסים באמצעות שימוש בסוכני cross-linking חומצות גרעין 28-30. גישה זו אחרונה היא יעילה יותר לוירוסים מומת על ידי היפוכלוריט ולא יעיל למי מומת על ידי UV.
מגבלה שנייה של נהלים המולקולריים אלה היא כי היקף המדגם מרוכז שניתן assayed בדרך כלל הוא הרבה יותר קטן מזה המשמש לנוהלי תרבות 6,31. בעיה זו מטופלת לעתים קרובות על ידי שתי החלפת הליך המבוסס על פוליאתילן גליקול לריכוז הסטנדרטי המשני על ידי הפתתה אורגנית, המאפשר את המדגם כדי להיות resuspended בנפח קטן יותר, או על ידי התוספת של מדגם שיישוני צעד ריכוז 6,32,33 . שיטה 1,615 משתמשת centrifאולטרה ugal לספק ריכוז שלישוני. אולטרה סינון צנטריפוגלי מסיר מים ורכיבים פחות מ -30,000 Daltons וכתוצאה מכך גם הריכוז של כל וירוס בדגימות בדיקה וירידה במשקל מולקולרי קטנים מעכבים של מבחני מולקולריים. תוצאות זה שיישוני צעד ריכוז בגורם ריכוז כולל של> 10 5 לכל וירוס שהיה נוכח במים נבדקות.
מגבלה שלישית היא נוכחותם של מעכבי הליכים מולקולריים בדגימות סביבתיות. למרות מספר רב של גישות להסרת מעכבים פותחו, אין גישה יעילה לכל מטריצות המים וסוגי וירוס 6,34,35, מה שהופך את השימוש בבקרות פנימיות שנועדו להעריך את הרמה של עיכוב חיוני. מגיב G הצהבת בשימוש בשיטה זו עונה על דרישה זו על ידי מתן על רמה קבועה של רנ"א הנגיפי בכל התגובות וassay RT-qPCR להערכת עיכוב. כאשר הטוב ביותר של tהוא מעכב גישות ההסרה להיכשל כדי להסיר עיכוב, יכולים להיות מדוללים תרכיזי מדגם עוד ריכוזי וירוס הם גבוהים מריכוזי מעכב 14,15.
יש ההליך העקום הסטנדרטי המתואר במסמך זה גם יתרונות ומגבלה עיקרית. יתרון הוא שמגיב משמש אספקה כל המרכיבים הדרושים במגיב אחד, המאפשר שליטה יחידה שתשמש לכל מבחני. מגיב הדבר נכון במיוחד יתרון עבור מבחני norovirus. ניתן להשיג חלקיקי norovirus רק מאנשים נגועים ולכן קשה מאוד להשיג חלקיקים נגיפיים לשימוש כסטנדרטים. יתרון חשוב נוסף הוא שהיא מספקת סטנדרטית RNA לכל וירוסי RNA הממוקדים במגיבה אחד, כבעל סטנדרטי RNA הוא חיוני לכימות מדויק של RNA 36. עם זאת, יכולתו לכמת במדויק וירוס היא מוגבלת על ידי העובדה שהשפעות המטריצה אינן נלקחות בחשבון. משמעות הדבר היא כי עותק הגנומיערכי מספר אינו יכולים להיחשב מוחלטים וצריכים להיחשב רק במונחים יחסי. מומלץ מספר מספיק של מניות aliquots עובדות עקומה סטנדרטית (שלב 1.2) להיות מוכן לכיסוי לימודים מלאים. לדוגמא, כל aliquot μl 250 מספק מגיב מספיק ל6 צלחות RT. אם ידוע שמחקר דורש ניתוח של 500 דגימות, מינימום של 12 aliquots יהיה צורך (500 דגימות / 7 דגימות לכל צלחת RT / 6 צלחות RT לaliquot).
EPA שיטה 1,615 היא שיטה מבוססת ביצועים. יצרנים רבים להפוך ריאגנטים שווי ערך לאלה המפורטים ובניתן להחליף ריאגנטים אלה כל עוד קריטריוני ביצועים הם נפגשו. העקומה סטנדרטית, המשמשת גם כביקורת חיובית RT-qPCR, היא בעל ערך בבעיות ביצועים לפתרון בעיות. ביצועים יכולים לרדת עקב הידרדרות של רנ"א, חיי מדף מגיב, כישלון של מקפיאים, כיול מכשיר, וטעות טכנית. בעיות ביצועים צריכים להיות חשודותאד אם עקומות סטנדרטי שונה שממוצגים באיור 4 או אם הם אינם עומדים במפרט ביצועים לעקומות סטנדרטיים. Assay RT-qPCR הוא די חזק; כישלון מוחלט הוא כנראה עקב טיפול לא נכון של RNA או טעות טכנית (למשל, מגיב חסר). בזהירות רבה יש לנקוט בטיפול בדגימות RNA בין החילוץ וצעד RT להפחית השפלה RNA מribonucleases.
החלמה מנגיף פוליו המים קרקע וכיתה מגיב והחלמת norovirus עכברית מתהום פגשה EPA שיטת 1,615 קריטריונים לקבלת ביצועים (לוח 11) ודומים לאלה שדווחו על ידי אחרים 33,37,38. ההחלמה norovirus Murine מדגימות LFB היו נמוכה בהרבה מאלו של נגיף הפוליו ולא עמדה בקריטריוני קבלת נגיף הפוליו ספציפי. הסיבות להתאוששות norovirus עכברית נמוכה ממי כיתה מגיב אינן ידועות. collea דומה לתוצאות במסמך זה, כרים וגואז דיווח התאוששות לGI.1 norovirus של 4% ממים ברז 39. לי ואח '. 37 דיווחו החלמה ממוצעת לnorovirus עכברי וGII.4 norovirus האנושי של 18% ו -26% ממים מזוקקים באמצעות מסנני דיסק, בהתאמה. שימוש בתנאים דומים ללי ועמיתים, קים וKo נצפו החלמה של 46% ו -43% לוירוסים אלה, בהתאמה 38. גיבונס et al. 40 הושג סביב התאוששות 100% מGII.4 norovirus האנושי ממי ים, אבל קים וKo 38 מצאו כי התוספת של מלח למים מזוקקים בריכוזים דומים או גבוהים יותר מאשר מי ים באופן משמעותי החלמה מופחתת של הווירוס עכברי והביאה לירידה כפולה בהתאוששות GII.4.
Disclosures
The authors have nothing to disclose.
Acknowledgements
The authors thank Dr. Eric Rhodes for preparing the clones used in the development of Standard curve reagent, Brian McMinn for assistance in sample processing, Larry Wymer for statistical analysis, Dr. Mark Borchardt, U.S. Department of Agriculture, Marshfield, WI, for supplying the Sabin poliovirus serotype 3 used in this study and Dr. H. W. Virgin, Washington University, St. Louis, MO, for murine norovirus. The authors also acknowledge Gretchen Sullivan for assistance in preparation of stock laboratory reagents, Dr. Mohammad Karim for propagation of murine norovirus stocks, and local private well owners and utilities for allowing us to collect water samples. Although this work was reviewed by EPA and approved for publication, it may not necessarily reflect official Agency policy. Mention of trade names or commercial products does not constitute endorsement or recommendation for use.
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 ml tube chamber | Diversified Biotech | CHAM-3000 | |
| 1 °C cool brick | Diversified Biotech | BRIK-2501 | |
| 10x PCR Buffer II and 25-mM MgCl2 | Life Technologies | N8080130 | |
| -20 °C freezer | VWR | 97043-346 | Must be a manual defrost freezer |
| -70 °C or colder freezer | Thermo Scientific | MBF700LSAO-E | |
| 96-well chamber | Diversified Biotech | CHAM-1000 | |
| Absolute ethanol | Fisher Scientific | BP2818-100 | |
| Armored RNA EPA-1615 | Asuragen | Custom order | Used for quantifying the RT-qPCR assay |
| Armored RNA Hepatitis G virus | Asuragen | 42024 | |
| Autoclave | Steris | Amsco Lab Series | |
| Biosafety cabinet | NuAir Laboratory Equipment Supply | Labgard 437 ES | |
| Bovine serum albumin (BSA) | Affymetrix | 10856 | Crystalline grade or better |
| Buffer AVE | Qiagen | 1026956 | Carrier RNA dilution buffer |
| Buffer AVL | Qiagen | 19073 | Extraction buffer |
| Carrier RNA | Qiagen | Not applicable | Use carrier RNA supplied with Buffer AVL |
| Centrifuge bottles | Fisher Scientific | 05-562-23 or 05-562-26 | |
| Centrifuge rotors | Beckman Coulter | 339080, 336380 | |
| Cool safe box | Diversified Biotech | CSF-BOX | |
| Dithiothreitol (DTT) | Promega | P1171 | |
| LightCycler® 480 Probes Master kit | Roche Diagnostics | 4707494001 | |
| Microcentrifuge tubes | Fisher Scientific | 02-682-550 | Use ribonuclease- and deoxyribonuclease-free tubes with snap caps |
| Microcide III | Fitzgerald | 99R-103 | |
| Microseal 'A' film | Bio-Rad Laboratories | HSA5001 | Heat resistant |
| Microseal 'F' film | Bio-Rad Laboratories | MSA1001 | Freezer resistant |
| Mini-plate spinner | Labnet International | MPS1000 | |
| Multichannel pipette | Rainin | L8-20 | |
| Multi-tube chamber | Diversified Biotech | CHAM-5000 | |
| Optical reaction plate | Life Technologies | 4314320 | |
| PCR nucleotide mix | Promega | U1515 | |
| PCR plate | Bio-Rad Laboratories | HSS9601 | |
| PCR-grade water | Roche | 3315932001 | |
| Phosphate buffered saline (PBS) | U.S. Biological | D9820 | |
| Plate mixer | Scientific Industries | MicroPlate Genie | |
| Prionex gelatin | Sigma Aldrich | G0411 | |
| QIAamp DNA Blood Mini Kit | Qiagen | 51104 | Includes Buffers AW1 (first wash buffer), AW2 (second wash buffer), AE (elution buffer); ethanol must be added to Buffers AW1 and AW2 before use; Do not use Buffer AL supplied with the kit |
| Quantitative PCR thermal cycler | Life Technologies | 4351405 | |
| Random primer | Promega | C1181 | |
| Reagent Reservoir | Fisher Scientific | 21-381-27E | |
| Refrigerated centrifuge | Beckman Coulter | 367501 | |
| RNase Inhibitor | Promega | N2515 or N2615 | |
| ROX reference dye | Life Technologies | 12223 | |
| SuperScript II or III Reverse Transcriptase | Life Technologies | 18064-022 or 18080044 | |
| Surgical gloves | Fisher Scientific | 19-058-800 | |
| Thermal cycler | Life Technologies | 4314879 | |
| Trisma base | Sigma Aldrich | T1503 | |
| Vivaspin 20 centrifugal concentrator units | Sartorius-Stedim | VS2022 |
References
- Borchardt, M. A., Spencer, S. K., Kieke, B. A., Lambertini, E., Loge, F. J. Viruses in nondisinfected drinking water from municipal wells and community incidence of acute gastrointestinal illness. Environ. Health Perspect. 120 (9), 1272-1279 (2012).
- Ye, X. Y., et al. Real-time PCR detection of enteric viruses in source water and treated drinking water in Wuhan, China. Curr. Microbiol. 65 (3), 244-253 (2012).
- Williamson, W. M., et al. Enteric viruses in New Zealand drinking-water sources. Water Sci. Technol. 63 (8), 1744-1751 (2011).
- Lambertini, E., Borchardt, M. A., Kieke, B. A., Spencer, S. K., Loge, F. J. Risk of viral acute gastrointestinal illness from nondisinfected drinking water distribution systems. Environ. Sci. Technol. 46 (17), 9299-9307 (2012).
- Chigor, V. N., Okoh, A. I. Quantitative RT-PCR detection of hepatitis A virus, rotaviruses and enteroviruses in the Buffalo River and source water dams in the Eastern Cape Province of South Africa. Int. J. Environ. Res. Public Health. 9 (11), 4017-4032 (2012).
- Fout, G. S., Martinson, B. C., Moyer, M. W., Dahling, D. R. A multiplex reverse transcription-PCR method for detection of human enteric viruses in groundwater. Appl. Environ. Microbiol. 69 (6), 3158-3164 (2003).
- Hewitt, J., Bell, D., Simmons, G. C., Rivera-Aban, M., Wolf, S., Greening, G. E. Gastroenteritis outbreak caused by waterborne norovirus at a New Zealand ski resort. Appl. Environ. Microbiol. 73 (24), 7853-7857 (2007).
- Jack, S., Bell, D., Hewitt, J. Norovirus contamination of a drinking water supply at a hotel resort. New Zealand Med. J. 126 (1387), 98-107 (2013).
- Anderson, A. D., et al. A waterborne outbreak of Norwalk-like virus among snowmobilers-Wyoming, 2001. J. Infect. Dis. 187 (2), 303-306 (2003).
- Parshionikar, S. U., et al. Waterborne outbreak of gastroenteritis associated with a norovirus. Appl. Environ. Microbiol. 69 (9), 5263-5268 (2003).
- U.S. Environmental Protection Agency Office of Water. . 820-F-12-058: Recreational Water Quality Criteria. , 1-63 (2012).
- Wade, T. J., et al. Rapidly measured indicators of recreational water quality and swimming-associated illness at marine beaches: a prospective cohort study. Environ. Health. 9, 66 (2010).
- Fout, G. S., et al. . Method 1615: Measurement of enterovirus and norovirus occurrence in water by culture and RT-qPCR (EPA/600/R-10/181). , 1-91 (2012).
- Cashdollar, J. L., et al. Development and Evaluation of EPA Method 1615 for Detection of Enterovirus and Norovirus in Water. Appl. Environ. Microbiol. 79 (1), 215-223 (2013).
- Gibson, K. E., Schwab, K. J., Spencer, S. K., Borchardt, M. A. Measuring and mitigating inhibition during quantitative real time PCR analysis of viral nucleic acid extracts from large-volume environmental water samples. Water Res. 46 (13), 4281-4291 (2012).
- Fuentes, C., et al. Standardized multiplex one-step qRT-PCR for hepatitis A virus, norovirus GI and GII quantification in bivalve mollusks and water. Food Microbiol. 40, 55-63 (2014).
- Coudray, C., Merle, G., Martin-Latil, S., Guillier, L., Perelle, S. Comparison of two extraction methods for the detection of hepatitis A virus in lettuces using the murine norovirus as a process control. J. Virol. Methods. 193 (1), 96-102 (2013).
- Sen, K., et al. . EPA 815-B-04-001: Quality assurance/quality control guidance for laboratories performing PCR analyses on environmental samples. , 1-56 (2004).
- Schlueter, V., Schmolke, S., Stark, K., Hess, G., Ofenloch-Haehnle, B., Engel, A. M. Reverse transcription-PCR detection of hepatitis G virus. J. Clin. Microbiol. 34 (11), 2660-2664 (1996).
- De Leon, R., Shieh, C., Baric, R. S., Sobsey, M. D. Detection of enteroviruses and hepatitis A virus in environmental samples by gene probes and polymerase chain reaction. Proc. Water Qual. Technol. Conf. , 833-853 (1990).
- Monpoeho, S., et al. Quantification of enterovirus RNA in sludge samples using single tube real-time RT-PCR. BioTechniques. 29 (1), 88-93 (2000).
- Jothikumar, N., et al. Rapid and sensitive detection of noroviruses by using TaqMan-based one-step reverse transcription-PCR assays and application to naturally contaminated shellfish samples. Appl. Environ. Microbiol. 71 (4), 1870-1875 (2005).
- da Silva, A. K., et al. Evaluation of removal of noroviruses during wastewater treatment, using real-time reverse transcription-PCR: different behaviors of genogroups I and II. Appl. Environ. Microbiol. 73 (24), 7891-7897 (2007).
- Butot, S., et al. Evaluation of various real-time RT-PCR assays for the detection and quantitation of human norovirus. J. Virol. Methods. 167 (1), 90-94 (2010).
- Loisy, F., et al. Real-time RT-PCR for norovirus screening in shellfish. J. Virol. Methods. 123 (1), 1-7 (2005).
- Reynolds, K. A., Gerba, C. P., Pepper, I. L. Detection of infectious enteroviruses by an integrated cell culture-PCR procedure. Appl. Environ. Microbiol. 62 (4), 1424-1427 (1996).
- Ming, H. X., Zhu, L., Zhang, Y. Rapid quantification of infectious enterovirus from surface water in Bohai Bay, China using an integrated cell culture-qPCR. Mar. Pollut. Bull. 62 (10), 2047-2054 (2011).
- Parshionikar, S., Laseke, I., Fout, G. S. Use of propidium monoazide in reverse transcriptase PCR to distinguish between infectious and noninfectious enteric viruses in water samples. Appl. Environ. Microbiol. 76 (13), 4318-4326 (2010).
- Sanchez, G., Elizaquivel, P., Aznar, R. Discrimination of infectious hepatitis A viruses by propidium monoazide real-time RT-PCR. Food Environ. Virol. 4 (1), 21-25 (2012).
- Kim, S. Y., Ko, G. Using propidium monoazide to distinguish between viable and nonviable bacteria, MS2 and murine norovirus. Lett. Appl. Microbiol. 55 (3), 182-188 (2012).
- Fout, G. S., Schaefer, F. W., Messer, J. W., Dahling, D. R., Stetler, R. E. . EPA/600/R-95/178: ICR Microbial Laboratory Manual, I.1-ApD-23. , (1996).
- Abbaszadegan, M., Stewart, P., LeChevallier, M. A strategy for detection of viruses in groundwater by PCR. Appl. Environ. Microbiol. 65 (2), 444-449 (1999).
- Lambertini, E., et al. Concentration of enteroviruses, adenoviruses, and noroviruses from drinking water by use of glass wool filters. Appl. Environ. Microbiol. 74 (10), 2990-2996 (2008).
- Rodriguez, R. A., Thie, L., Gibbons, C. D., Sobsey, M. D. Reducing the effects of environmental inhibition in quantitative real-time PCR detection of adenovirus and norovirus in recreational seawaters. J. Virol. Methods. 181 (1), 43-50 (2012).
- Iker, B. C., Bright, K. R., Pepper, I. L., Gerba, C. P., Kitajima, M. Evaluation of commercial kits for the extraction and purification of viral nucleic acids from environmental and fecal samples. J. Virol. Methods. 191 (1), 24-30 (2013).
- Fey, A., et al. Establishment of a real-time PCR-based approach for accurate quantification of bacterial RNA targets in water, using Salmonella as a model organism. Appl. Environ. Microbiol. 70 (6), 3618-3623 (2004).
- Lee, H., et al. Evaluation of electropositive filtration for recovering norovirus in water. J. Water Health. 9 (1), 27-36 (2011).
- Kim, M., Ko, G. Quantitative characterization of the inhibitory effects of salt, humic acid, and heavy metals on the recovery of waterborne norovirus by electropositive filters. J. Water Health. 11 (4), 613-622 (2013).
- Karim, M. R., Rhodes, E. R., Brinkman, N., Wymer, L., Fout, G. S. New electropositive filter for concentrating enteroviruses and noroviruses from large volumes of water. Appl. Environ. Microbiol. 75 (8), 2393-2399 (2009).
- Gibbons, C. D., Rodriguez, R. A., Tallon, L., Sobsey, M. D. Evaluation of positively charged alumina nanofibre cartridge filters for the primary concentration of noroviruses, adenoviruses and male-specific coliphages from seawater. J. Appl. Microbiol. 109 (2), 635-641 (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved