Method Article
エンテロウイルスと文化およびRT-qPCRにより水中のノロウイルス発生のEPAメソッド1615測定。パートIII。 RT-qPCRによりウイルス検出
要約
Here we present a procedure to quantify enterovirus and norovirus in environmental and drinking waters using reverse transcription-quantitative PCR. Mean virus recovery from groundwater with this standardized procedure from EPA Method 1615 was 20% for poliovirus and 30% for murine norovirus.
要約
EPA Method 1615 measures enteroviruses and noroviruses present in environmental and drinking waters. This method was developed with the goal of having a standardized method for use in multiple analytical laboratories during monitoring period 3 of the Unregulated Contaminant Monitoring Rule. Herein we present the protocol for extraction of viral ribonucleic acid (RNA) from water sample concentrates and for quantitatively measuring enterovirus and norovirus concentrations using reverse transcription-quantitative PCR (RT-qPCR). Virus concentrations for the molecular assay are calculated in terms of genomic copies of viral RNA per liter based upon a standard curve. The method uses a number of quality controls to increase data quality and to reduce interlaboratory and intralaboratory variation. The method has been evaluated by examining virus recovery from ground and reagent grade waters seeded with poliovirus type 3 and murine norovirus as a surrogate for human noroviruses. Mean poliovirus recoveries were 20% in groundwaters and 44% in reagent grade water. Mean murine norovirus recoveries with the RT-qPCR assay were 30% in groundwaters and 4% in reagent grade water.
概要
定量的PCR(定量PCR;この原稿で使用される用語の定義については、補足資料を参照)、逆転写定量PCR(RT-qPCRを)は、環境や飲料水の中に人間の腸内ウイルスを検出し、定量化するため、特に行う多くのウイルスのための貴重なツールであります複製または細胞培養系では不十分複製しません。どちらのツールは、多くのウイルス型は世界1-6全体の環境や飲料水の中に存在していることを実証しました。彼らは飲料水で見つかったウイルスが流行患者7-10によって流されたものと同一であることが示されているような疾患のアウトブレイク調査の間に増幅されたゲノム断片の塩基配列決定と相まってこれらの使用は、水性ウイルス伝播の証拠を提供してきました。
定量PCRおよびRT-qPCRの両方が便利な公衆衛生上のツールです。例えば、米国環境保護庁(EPA)によって行われた研究からのデータは強い関係があることが示されました定量PCRやレクリエーション水域における健康影響によりトゥイーン指標の測定。その結果、EPAの最終2012レクリエーション水質基準は、レクリエーションビーチ11,12を監視するための定量PCR法を含んでいます。 RT-qPCRの1によって測定されるようにBorchardtらはまた、地下水に未処理の地下水やウイルスを使用して、地域社会における急性胃腸炎との間に強い関連性を発見しました。
本稿の目的は、EPAメソッド1615 13,14の分子アッセイ成分を記述することです。このアッセイは、陽電フィルタを通過し、環境や飲料水の元の体積に基づいてリットル当たりエンテロウイルスとノロウイルスゲノムコピー(GC)の定量的な推定値を提供するために、RT-qPCRのを使用しています。分子の手順の概要を図1に示されている。プロトコル部1は、標準曲線を調製するための手順を詳述します。これらの規格は、RNが含まれている試薬から調製されます全てのプライマー/プローブセットの標的配列のコピー。第2節では、第三級濃度の手順を説明します。第3節では、濃縮水と対照試料からRNAを抽出するための手順を説明します。各試験サンプルからのRNAを逆三重アッセイおよびプライム転写(第4)のランダムプライマーを使用して転写されます。 (; 図2第5項)、各逆転写反応からcDNAをqPCRにより三重に分析されている5つの別々のウイルス特異的なアッセイに分割されます。アッセイは、多くのエンテロウイルスとノロウイルスおよびRT-qPCRを15に阻害されている試験サンプルを同定するためのG型肝炎RNAを含む試薬を検出するように設計されている科学文献( 表1)からのプライマーおよびプローブを使用します。
プロトコル
注:使用データシートには、プロトコルのすべてのステップを追跡するために、例えばデータシートは、補足材料表S2、S4に示されています。
1.標準曲線の準備
- TSMを使用して、2.5×10 8粒子/ mlの濃度になるように、製造業者によって供給された濃度から、それを希釈することにより標準曲線試薬( 例えば 、アーマードRNA EPA-1615)の作業ストックを準備します(/ mlの2.5×10 8 GC) IIIバッファ。 -20℃で1.5 mlのマイクロ遠心チューブとストアを使用して250μlのアリコートに作業用ストックを分割します。
注 :代替標準曲線の試薬 として使用するための準備作業のウイルスの株式およびプラスミドの手順については、補足材料プロトコルステップS1を参照してください。 - 作業ストック一定分量の一つ以上を解凍します。 、2.5×10 7の濃度を与え、2.5×10 6、2.5×1.5 mlのマイクロチューブを使用して5〜10倍連続希釈液を調製します10 5、2.5×10 4、2.5×10 3、GC / mlです。
- TSM IIIバッファの225μlに2.5×10 8 GC / mlの作業ストックの25μLを追加することで、最初の希釈を準備します。ボルテックスミキサーを使用して、5-15秒間混合します。
- TSM IIIバッファの225μlにステップ1.2.1で製造した希釈液の25μlのを追加することで、次の希釈を準備します。再び混合し、次の3つの10倍希釈液を準備するために同様のプロセスを続行します。
- 標準曲線作業ストックからのRNAとすぐに第3の手順を用いて5希釈液を抽出します。
2.第三紀濃度
- 上部試料室に、1×PBS、0.2%ウシ血清アルブミン(BSA)の少なくとも10ミリリットルを添加することによって収集された各試料についての遠心濃縮器(分子量30,000カットオフ)を準備します。そのソリューションは、薄いチャネル濃度室を満たしていることを確認した後、4℃でO / Nを保持します。
- 流体を捨てます。過剰のBSAを除去した後、水を廃棄し、滅菌試薬グレードの水の少なくとも10mlでコンセントレータ1時間をすすぎます。
- 別々の遠心濃縮にSに等しい各試験サンプル、アッセイのサンプル量から二水濃縮物の量を追加します。
- 式1を使用して、Sを計算します
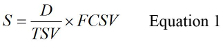
Dをアッセイオリジナル水試料の容量である場合には、TSVは、総サンプル量で、FCSVは最終濃縮サンプルボリュームです。 Sの計算の例については、補足材料S2を参照してください。
- 式1を使用して、Sを計算します
- 遠心20スイングバケットローターで4℃で3,000-6,000×gで各試験サンプル - 30分。薄いチャネル濃度室でボリュームを確認してください。
- ボリュームは、20分以上再度400μlの、遠心分離機よりも大きい場合。番目のサンプルまで遠心分離し続けます電子薄いチャネル濃度室未満400μlに減少しました。上清を除去しないでください。
- ウイルス回収を増加させるための無菌0.15 Mリン酸ナトリウム、pHは7〜7.5の1mlで遠心濃縮の側面を洗ってください。サンプルが400未満μlに減少されるまで、3,000-6,000×gで、4℃で再び遠心分離。この洗浄工程をさらに1回繰り返します。
- 、100〜200μlのマイクロピペットを用いて慎重に測定し、1.5 mlのマイクロ遠心チューブ( すなわち 、マイクロチューブに200μlのを転送し、残りの流体が完全に描画することができますまで、マイクロピペットを調整することで、残りの濃縮物を測定するために、各濃縮サンプルを転送)ピペット先端に。 0.15 Mリン酸ナトリウム、pHは7〜7.5は400±2μlに最終容量を調整するために、追加します。
- processeすることはできません任意の濃サンプル3.保留に進むことで、すぐに核酸を抽出dはすぐに超えない24時間4℃で。
3.核酸単離
- ラベルされた1.5mlの微小管を分離するために、ステップ1.3からステップ2.5または標準曲線希釈から各試験サンプルから三次濃縮のステップ3.3で説明し、200μlのように調製した抽出緩衝液200μlを追加します。 -70℃以下に残りの三次濃縮液を凍結。以下の例外を除いて、血液試料のスピンプロトコルの核酸抽出キットを製造者の指示に従って、各試料から核酸を抽出します。
- 水試料にプロテアーゼを追加したり、核酸抽出キット付属の抽出緩衝液を使用しないでください。
- キャリアRNAを抽出緩衝液を準備
- キャリアRNAの310μgの含有するバイアルにキャリアRNA希釈緩衝液の310μLを加えます。溶解した後、約50μLを含む6アリコートに分割する混ぜます。 -20で保存°C。
- 0.027μgの/μlとキャリアRNAの濃度を得るための抽出緩衝液1ml当たり解凍したキャリアRNAの28μLを追加します。抽出キットに付属のものの代わりにキャリアRNA-改正抽出緩衝液を使用してください。
- リボヌクレアーゼを添加することにより溶出バッファーのマスター溶液を調製する(RNアーゼ)阻害剤抽出キットで提供される溶出緩衝液への400単位/ mlの最終濃度まで。
- カラムにRNaseインヒビターで溶出緩衝液50μlを配置することにより、スピンカラムを結合核酸から溶出RNA。 1分間待ってから、室温で1分間、6,000×gで遠心します。
- ステップ3.4.1を繰り返し、その後、列を削除し、廃棄します。
- ステップ3.4.2からのRNA抽出物のアリコートを準備します。 6標準曲線作業ストックの一定分量と、少なくとも15μlの各を含む各標準曲線の希釈を準備します。少なくとも含む他のすべてのRNAサンプルの4アリコートを準備22μlの各。それらは4時間以内に逆転写することによって処理することができる場合、4℃で各サンプル、標準曲線コントロールの1つのアリコートを保存し、それ以外の場合は、-70℃以下にすべてのアリコートを保存します。
4.逆転写(RT)
- 10倍に等しい(マイクロリットル中)に存在するオリゴヌクレオチドのナノモル(ナノモル)の数量を用いて各バイアルにPCRグレードの水の量を添加することにより、表1にリストされた各オリゴヌクレオチドプライマーおよびプローブの100μMストック溶液を調製しますバイアル(バイアルラベルにか、製造元の仕様書に示されるように(例えば、)363μlの36.3ナノモルを含むプライマーを懸濁します。溶解するために混ぜます。
- ソリューションの作業10μMのを準備するために、PCRグレードの水で100μMのソリューションを1:10に希釈します。
- 表2のガイドを使用して、クリーンルーム内でのRTマスターミックス1と2を準備します。RT Masteのピペット16.5μLウェルマルチチャンネルピペットを用いて各PCRプレートにミックス1 R。
- 解凍、凍結した場合、各フィールドのサンプルとラボ強化さサンプルマトリックスからの核酸抽出物(LFSM; すなわち 、播種水マトリックス試料)。
- RNase阻害剤の400単位/ mlを含有する溶出緩衝液中で5と1:25:各フィールドとLFSMサンプル1を希釈します。
- 、研究室試薬ブランク(LRB、すなわち、試薬グレードの水を使用して、負の品質管理)、性能評価(PE;解凍、凍結した場合、しかし、ラボ要塞はブランク( すなわち 、シードされた試薬グレードの水を使用して、ポジティブな品質管理LFB)を希釈していません; すなわち 、播種試薬グレードの水サンプルは、試験の開始前に実験室の性能)、パフォーマンステスト(PTを評価するために使用される; すなわち 、試験中に実験室での性能を評価するために使用される分析者に未知の力価を有する播種試薬グレードの水サンプル)、NAバッチネガティブ抽出制御、または標準曲線のセットからRNAを抽出しました。
- (RTプレート例については、図S1を参照)試験サンプルおよびコントロールの三重井戸を使用して、別々のPCRプレートのウェルに、すべての試験サンプル、コントロール、および標準曲線からのRNAの6.7μLを置 き、標準曲線のために井戸を複製します。
- テンプレートを含まないコントロール(NTC)のための独立したPCRプレートのウェルに溶出緩衝液の6.7μLを置きます。 2-8 RTプレートあたりNTC、最初のサンプルのための2つを使用して、すべての第四の追加のサンプルについて、2つ以上を含みます。
- プレート全体に負の抽出とNTCコントロールを配布します。
- 耐熱プレートシーラーでPCRプレートをシール。 5-10秒間のサンプルを混合した後、≥500×gで簡単に遠心します。
- 99℃で4分間プレートをインキュベートした後、サーマルサイクラーで4℃に急速に冷却します。 ≥500 XG簡単で再び遠心分離。
- 慎重にプレートシールを除去し、各ウェルにRTマスターミックス2の16.8μlを添加します。セアル≥500×gで混合し、短時間の遠心分離に続いて、耐熱性のプレートシーラー、と再びプレート。
- サーマルサイクラーでプレートを置き、42℃、99℃で5分、で60分間続いた後、4℃の保持サイクルにより、25℃で15分間実行します。
- プロセス直ちにまたは定量PCR(ステップ5)、またはストアサンプルで8時間内で以下を-70℃で、それらを処理することが可能になるまで。 4℃で8時間以内に処理することができる店舗のサンプル。
5.リアルタイム定量PCR(定量PCR)
- いずれの試験サンプルを実行する前にG型肝炎試薬のロットごとに肝炎G Cqの値を意味するかを決定します。
- NTCコントロール(ステップ4.1.1)のために記載したように調製10の反復を用いてRTアッセイを実行します。 (5.5.3までの流れ5.2)以下に説明するように、肝炎GのqPCRアッセイを実行します。 10回の平均はCq値を計算します。
- RTマスターミックス1中のG型肝炎試薬量を調整した (表2)、必要に応じて、25及び32単位の間の平均Cqの値を取得します。水の容積を変化させることにより上昇または下降量を補償することは、アッセイあたり16.5μLでのRTマスターミックス1最終容量を維持するために追加されました。
- 調整された量を反映するために、5.1.2および変更表2にステップ5.1.1を繰り返して調整を確認してください。
- 私は肝炎のためのノロウイルスgenogroup II、マウスノロウイルスについては、表7(ノロウイルスgenogroupのV)、 および表8 は 、表6、ノロウイルスgenogroupためエン テロウイルスについては、表3のガイドを使用して、クリーンルーム内でのqPCRマスターミックスを調製し、 表4 および表5 G.ミックスは、各マスターミックスして、≥500×gで簡単に遠心します。
- 各定量PCRアッセイのためによくし、別々のプレートあたり14μLを使用して、標識された光学反応プレートの適切なウェルにPCRマスターミックスを追加します(図S2 図S1におけるRTのレイアウトに基づいて、定量PCRアッセイのための可能なレイアウトのためのG>)。
- 凍結した場合、室温でステップ4.8からRTプレートを解凍します。プレートミキサーを使用して混合した後、≥500×gで簡単に遠心します。
- 光学反応プレートの適切なウェルに適切なcDNAの6μLを分注します。 ≥500 XG簡単で光反応プレートと遠心機でサンプルを混ぜます。
- 他のすべてのqPCRアッセイを実行する前に希釈されていないと希釈フィールドとLFSMサンプルの肝炎GのqPCRアッセイを実行します。フィールドまたはあるLFSMサンプル<エンテロウイルスやノロウイルス定量PCRアッセイのための意味肝炎G Cqの値よりも大きい1 Cqを値の最低希釈を使用してください。
- メーカーの指示に従って定量PCRサーマルサイクラーソフトウェアをセットアップします。標準として標準曲線サンプルを識別し、それぞれの標準曲線希釈に、 表9に示したゲノムコピー値を入力します。
- は95℃と60℃で1分で15秒間の45サイクル、続いて95℃で10分間定量PCRサーマルサイクラーでプレートを実行します。
- 各標準曲線は表10に与えられた許容可能な値を満たしているかどうかを決定します。例については、補足材料セクションS3を参照してください。
- 式2を使用して、標準曲線の全体的な標準偏差(STDDEV)を算出
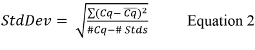
Cqの各標準曲線の複製について報告された値であり、Cqのは複製の各セットの平均値であり、#Cqは正の値( すなわち 、未確定ではない)を持っているすべての標準コントロールの複製のためのCqを値の合計数であり、#性感染症は、正の値を持っている標準コントロールの数です。 - 定量PCRサーマルサイクラー・ソフトウェアは、各標準曲線の傾きを計算していない場合は、式3を用いて勾配を計算し、60;
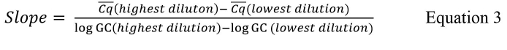
Cqには、使用される最高と最低の希釈液の平均値であり、GCのログをどこ表9から使用される最高と最低の希釈のためのゲノムコピー値のログです。 - 式4を用いて、R 2の値を計算します。
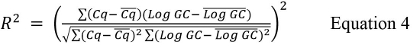
Cqには、すべての Cqを値の平均であるとログGCは各反復の平均ログのGC値です。 - 式(5)使用して%の効率を計算します。
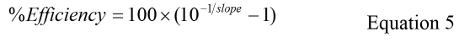
- 式2を使用して、標準曲線の全体的な標準偏差(STDDEV)を算出
- 表10に指定された条件と、各サンプルの平均GC値を満たす標準曲線に基づいて、すべてのテストサンプル用のサーマルサイクラーソフトウェアによって計算されたGC値を記録します。で基準を満たしていない標準的な曲線を持つ任意のサンプルを再実行表10または任意の負のコントロールは(LRB、NAバッチネガティブ抽出制御、またはNTC)が陽性です。再実行する際に判定基準を満たすために失敗したり、偽陽性のコントロールを持っている任意のサンプルを再処理。
- 式6を用いて、各試験サンプルのためリットル当たりGC(GCのL)を決定します。
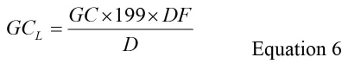
GCは、ステップ5.7からの平均のゲノムコピー数であり、係数「199」は、第三級濃度の間に発生する容量削減の総希釈率で、RNA抽出およびRT-qPCRのステップ、DFは阻害を補償する希釈係数であります、およびDは、リットルでアッセイオリジナル水試料の体積です。 GC Lの計算の例については、補足資料のセクションS4を参照してください。 - 199により、ステップ5.5からの平均GC値を乗算し、0.3で割って合計LFBのGCおよびLRBのサンプルを計算します。
結果
全体のウイルス回収は、対のフィールドとLFSM地下水サンプルを用いて決定しました。 7のサンプルセットの合計は、別々の3つの公開処理プラントからの機会、およびプライベートウェルから収集つのサンプルセットを収集する二組を使用して分析しました。 LFSMサンプルの種子レベルはセービンのポリオウイルス血清型マウスノロウイルスの3と5×10 6 PFUの6 MPN 3×10でした。マウスノロウイルスが原因LFSMサンプルに十分なウイルス濃度でヒトノロウイルス株の欠如にメソッド評価で代用として使用しました。地下水のサンプルの平均ポリオウイルス回収率は2%の標準誤差で、14しばらくは、マウスノロウイルスの回復は3%( 図3)の標準誤差で、30%であった平均は20%でした。各LFSMの通常のフィールドの地下水サンプルは、検出可能なエンテロウイルスやノロウイルスがありませんでした。
LFBとLRBのサンプルを播種し、播種していない試薬グレードワットを使用して測定しましたえー。すべてのサンプルは、LRB(データは示していない)陰性でした。マウスノロウイルスの回復は0.5%の標準誤差で4%を平均しながら、ポリオウイルスの回復は、1%の標準誤差( 図3)で44%を平均しました。
RT-qPCRのは、適切な標準曲線試薬の使用を必要とする。 図4は、エンテロウイルスやノロウイルスGIIのための典型的な標準曲線を示します。ノロウイルスGII曲線は0.9987のR 2値、0.14の全体的な標準偏差、101%の効率で標準曲線の性能基準( 表10)を満たしています。ノロウイルスGIAとGIB曲線(図示せず)は、ノロウイルスGIIのとほぼ同じです。エンテロウイルス曲線は0.9874のR 2値、0.58の全体的な標準偏差、および103%の効率で法の性能基準を満たしているが、約百倍低い感度とノロウイルス曲線よりもより高い検出限界を持っています。
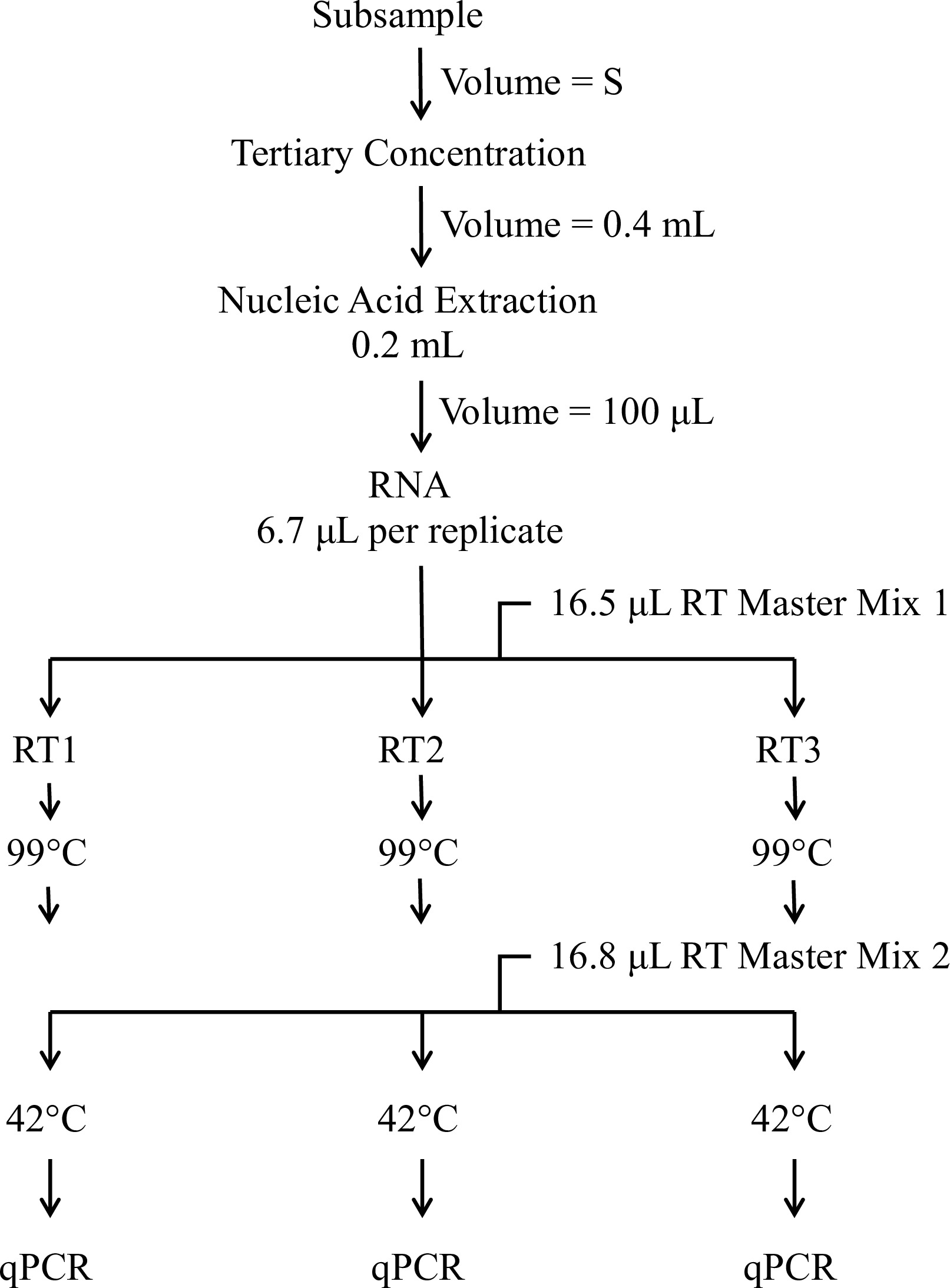
分子の手順の概要を図1。分子の手順は感染性ウイルスを測定するために実行されるものを超えた追加のサンプルの濃度、核酸の抽出を含む、二段階は、転写(RT)プロトコル、および定量PCR(定量PCR)を逆転。出発体積(S)は、元の水試料の方法に定義された割合を表します。
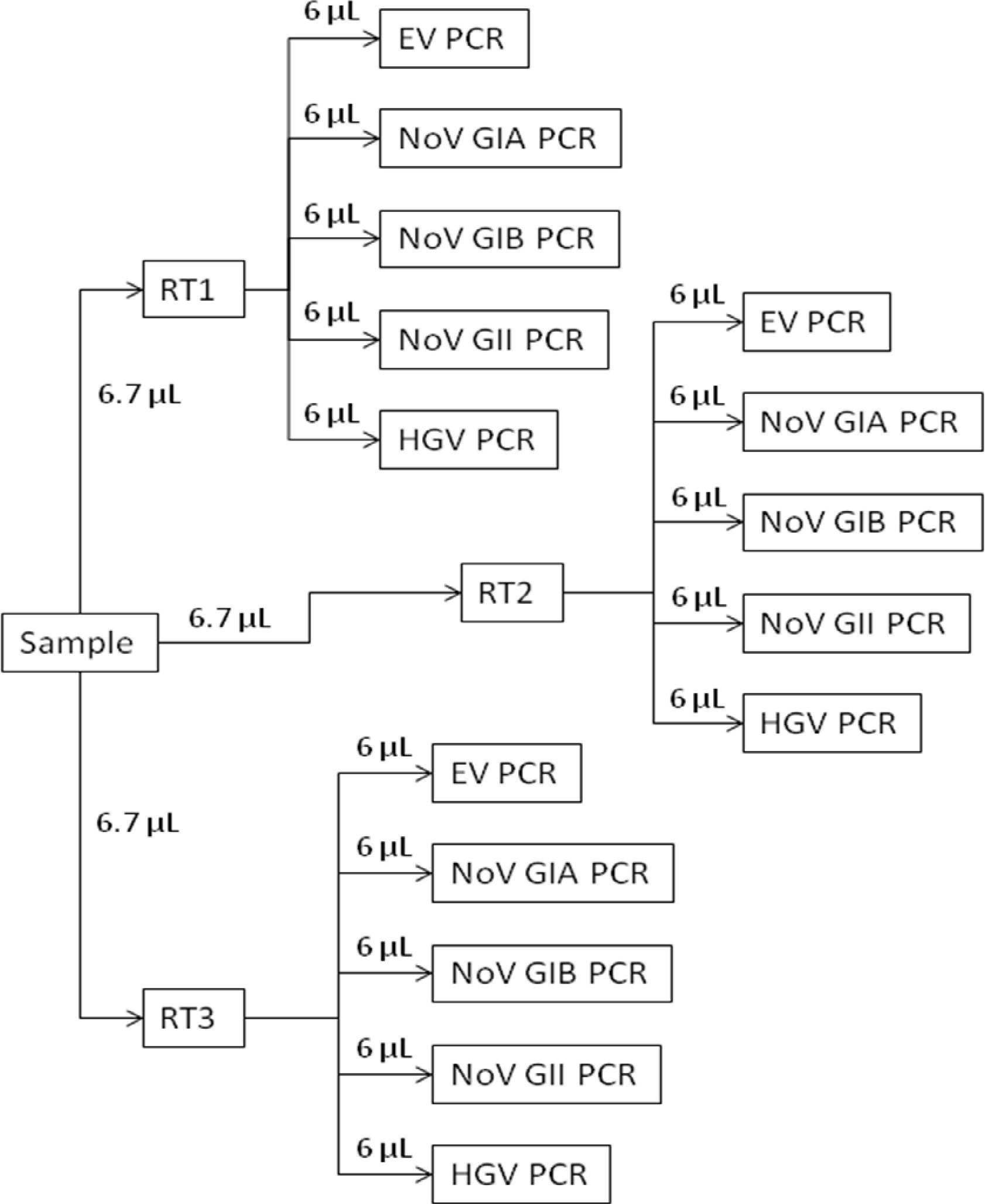
図2 RT-qPCRの概要概略図。各抽出試験サンプルのRNAを逆三重アッセイ(RT1、RT2、RT3と)を使用して転写されます。三重RTアッセイのそれぞれからのcDNAは、その後独立したエンテロウイルス(EV PCR)を使用して、特定のウイルス、ノロウイルスgenogroup I(NOV GIA PCRおよびNOV GIB PCR)、ノロウイルスgenogroup II(Nについて分析されますOV GII PCR)、およびG型肝炎(HGV PCR)アッセイ。
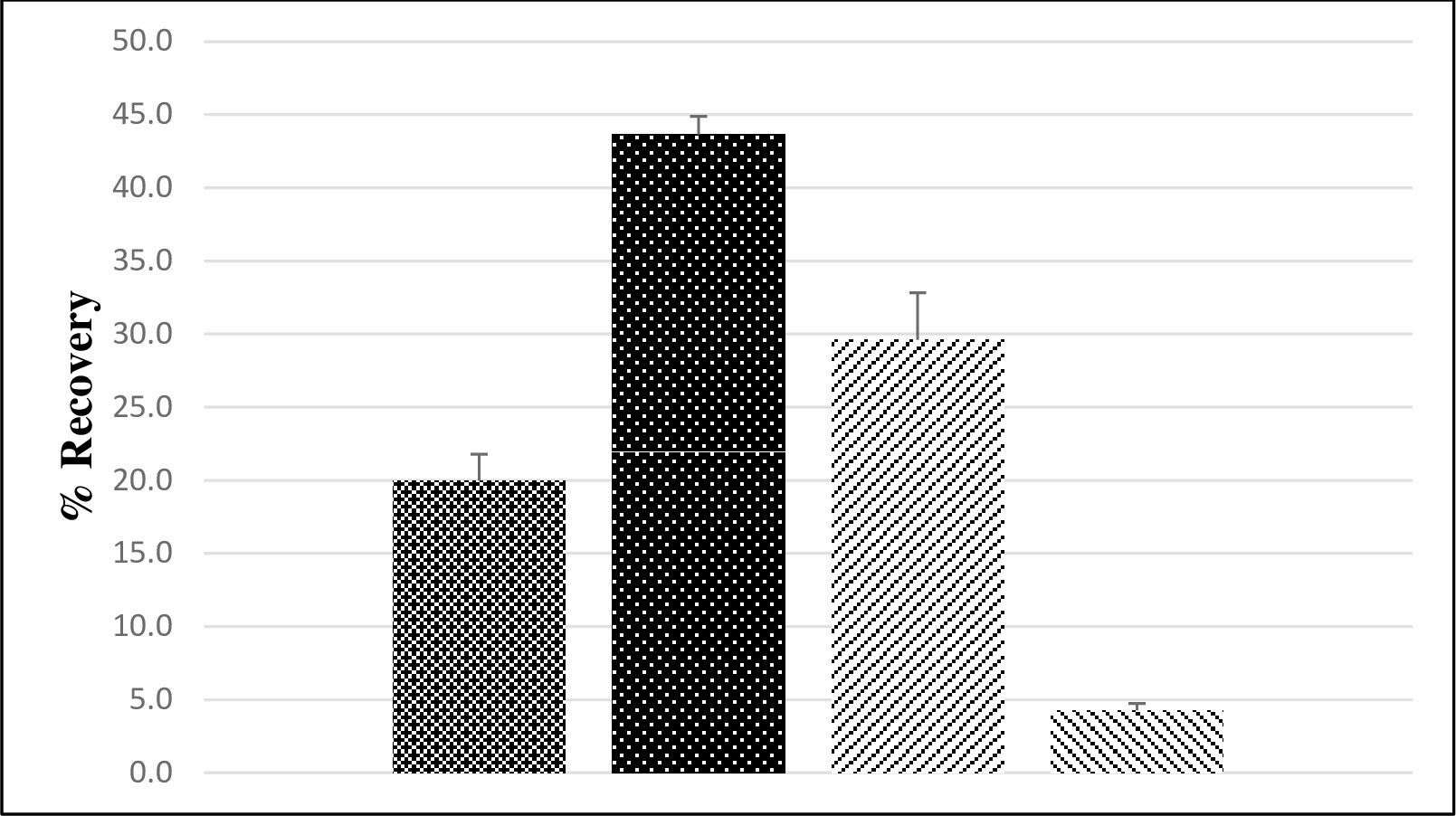
図3は、平均ポリオウイルスと地上からのマウスノロウイルスの回復(%)および試薬グレードの水。平均回収率は、地上からのポリオウイルス(のために示されています ; N = 7)および試薬グレードから(
; N = 7)および試薬グレードから(  ; N = 12)は、水と地面からのマウスノロウイルスのため(
; N = 12)は、水と地面からのマウスノロウイルスのため(  ; N = 7)および試薬グレードから(
; N = 7)および試薬グレードから(  ; N = 12)の水(1)ここで、「n」は独立した処理済みの水のサンプルの数です。エラーバーは標準誤差を表します。
; N = 12)の水(1)ここで、「n」は独立した処理済みの水のサンプルの数です。エラーバーは標準誤差を表します。

図4.エンテロウイルスとノロウイルスGII標準曲線。エンテロウイルスやノロウイルスGIIのための典型的な標準曲線が示されています。式は、各曲線の2つの値は、サーマルサイクラーで計算される傾きとRを与えます。
補足ファイル1. このファイルをダウンロードするにはこちらをクリックしてください。
| ウイルス群 | プライマー/プローブ名(1) | シーケンス(2) | リファレンス | |
| エンテロウイルス属 | ||||
| EntF(EV-L) | CCTCCGGCCCCTGAATG | 20 | ||
| ENTR(EV-R) | ACCGGATGGCCAATCCAA | 20 | ||
| ENTP(EV-プローブ) | 6FAM-CGGAACCGACTACTTTGGGTGTCCGT-TAMRA | 21 | ||
| ノロウイルスGIA | ||||
| NorGIAF(JJV1F) | GCCATGTTCCGITGGATG | 22 | ||
| NorGIAR(JJV1R) | TCCTTAGACGCCATCATCAT | 22 | ||
| NorGIAP(JJV1P) | 6FAM-TGTGGACAGGAGATCGCAATCTC-TAMRA | 22 | ||
| ノロウイルスGIB | ||||
| NorGIBF(QNIF4) | CGCTGGATGCGNTTCCAT | 23 | ||
| NorGIBR(NV1LCR) | CCTTAGACGCCATCATCATTTAC | 23 | ||
| NorGIBP(NV1LCpr) | 6FAM-TGGACAGGAGAYCGCRATCT-TAMRA | 23 | ||
| ノロウイルスGII | ||||
| NorGIIF(QNIF2d) | ATGTTCAGRTGGATGAGRTTCTCWGA | 25 | ||
| NorGIIR(COG2R) | TCGACGCCATCTTCATTCACA | 25 | ||
| NorGIIP(QNIFS) | 6FAM-AGCACGTGGGAGGGCGATCG-TAMRA | 25 | ||
| ノロウイルスGV | ||||
| MuNoVF1 | AGATCAGCTTAAGCCCTATTCAGAAC | 14 | ||
| MuNoVR1 | CAAGCTCTCACAAGCCTTCTTAAA | 14 | ||
| MuNoVP1 | VIC-TGGCCAGGGCTTCTGT-MGB | 14 | ||
| G型肝炎 | ||||
| HepF(5'-NCRフォワードプライマー) | CGGCCAAAAGGTGGTGGATG | 19 | ||
| HEPR(5'-NCRリバースプライマー) | CGACGAGCCTGACGTCGGG | 19 | ||
| ヘップ(肝炎G TaqManプローブ | 6FAM-AGGTCCCTCTGGCGCTTGTGGCGAG-TAMRA | 1 | ||
表1のプライマーおよびRT-qPCRによりウイルス検出用のTaqManプローブ。
(1)方法1615プライマーおよびプローブ名が逆に、前進のためのF、R、またはPに連結されたウイルス名の最初の3文字であり、プローブ。ノロウイルスgenogroupは名にGIとGIIを追加することで指定されています。また、主な参考文献からAとBプライマーおよびプローブ名を使用して区別されている2つのノロウイルスGIのプライマーセットは、親に与えられていますheses。
(2)プライマーおよびプローブ配列の向きは、5 'から3'です。次の縮重塩基指示薬が使用されています。すべての4つのヌクレオチドのN混合物; R-A + G; Y-T + C; W-A + T;そして、I-イノシン。
| 成分 | 反応あたり容量(μL)(2) | 最終濃度 | マスターミックスあたりの容量(μL)(3) |
| RTマスターミックス1 | |||
| ランダムプライマー | 0.8 | 10 NG /μL(C。5.6μM) | 84 |
| G型肝炎アーマードRNA(4) | 1 | 105 | |
| PCRグレードの水 | 14.7 | 1543.5 | |
| にタル | 16.5 | 1732.5 | |
| RTマスターミックス2 | |||
| 10倍のPCR緩衝液II | 4 | 10mMのトリス、pHが8.3、50mMの塩化カリウム | 420 |
| 25-のMgCl 2 | 4.8 | 3 mMの | 504 |
| 10-MMのdNTP | 3.2 | 0.8 mMの | 336 |
| 100-のDTT | 4 | 10 mMの | 420 |
| RNase阻害剤 | 0.5 | 0.5単位/μlの | 52.5 |
| スーパースクリプトII RT | 0.3 | 1.6単位/μL | 31.5 |
| トータル | 16.8 | 1764 | |
表2 RTマスターミックス1と2(1)。
(1)CLにRTマスターミックスを準備しますEAN部屋、 すなわち 、分子および微生物学的手順が行われていない部屋。
(2)最後のRTアッセイ容量は40μLです。
(3)ボリュームは105アッセイに基づいている示しています。これは、損失を考慮するために追加の余分なアッセイと、96ウェルPCRプレートのために十分です。量は、分析されるサンプルおよびコントロールの数に応じて拡大または縮小することができます。
補足資料に記載されているように(4)RTマスターミックス1に含めるG型肝炎試薬の量を決定するステップS4。
| 成分 | 反応あたり容量(μL)(2) | 最終濃度 | マスターミックスあたりの容量(μL)(3) |
| 2倍速ライトサイクラー480プローブマスターミックス | 10 | 所有権の | 1050 |
| ROXリファレンス染料(4) | 0.4 | 0.5 mMの | 42 |
| PCRグレードの水 | 1 | 105 | |
| 10μMのEntF | 0.6 | 300 nMの | 63 |
| 10μMのENTR | 1.8 | 900 nMの | 189 |
| 10μMのENTP | 0.2 | 100 nMの | 21 |
| トータル | 14 | 1470 |
表3. PCRマスターミックスエンテロウイルス(EV)アッセイのために (1)。
(1)クリーンルーム内のすべてのPCRマスターミックスを準備します。
(2)最終のqPCRアッセイ容量は20μlです。
(3)ボリュームは105アッセイに基づいている示しています。これは、損失を考慮するために追加の余分なアッセイと、96ウェルPCRプレートのために十分です。量はサンプルとなりますコントロールの数に応じてスケールアップまたはスケールダウンすることができます分析すること。
(4)本試薬の代替PCRグレードの水を、それを必要としない機器を使用するとき。
| 成分 | 反応あたり容量(μL)(2) | 最終濃度 | マスターミックスあたりの容量(μL)(3) |
| 2倍速ライトサイクラー480プローブマスターミックス | 10 | 所有権の | 1050 |
| ROXリファレンス色素(4) | 0.4 | 0.5 mMの | 42 |
| PCRグレードの水 | 1.4 | 147 | |
| 10μMのNorGIAF | 1 | 500 nMの | 105 |
| 10μMのNorGIAR | 1 | 500 nMの | 105 |
| 10μMのNorGIAP | 0.2 | 100 nMで | 21 |
| トータル | 14 | 1470 |
表4.ノロウイルスGIAのためのPCRマスターミックス(11月GIA)アッセイ (1)。
脚注については、表3を参照してください(1) - (4)。
| 成分 | 反応あたり容量(μL)(2) | 最終濃度 | マスターミックスあたりの容量(μL)(3) |
| 2倍速ライトサイクラー480プローブマスターミックス | 10 | 所有権の | 1050 |
| ROXリファレンス色素(4) | 0.4 | 0.5 mMの | 42 |
| PCRグレードの水 | 0.3 | 31.5 | |
| 10μMのNorGIBF | 1 | 500 nMの | 105 |
| 10μMのNorGIBR | 1.8 | 900 nMの | 189 |
| 10μMのNorGIBP | 0.5 | 250 nMの | 52.5 |
| トータル | 14 | 1470 |
表5.ノロウイルスGIBためのPCRマスターミックス(11月GIB)アッセイ (1)。
脚注については、表3を参照してください(1) - (4)。
| 成分 | 反応あたり容量(μL)(2) | 最終濃度 | マスターミックスあたりの容量(μL)(3) |
| 2倍速ライトサイクラー480プローブマスターミックス | 10 | 所有権の | 1050 |
| ROXリファレンス色素(4) | 0.4 | 0.5 mMの | 42 |
| PCRグレードの水 | 0.3 | 31.5 | |
| 10μMのNorGIIF | 1 | 500 nMの | 105 |
| 10μMのNorGIIR | 1.8 | 900 nMの | 189 |
| 10μMのNorGIIP | 0.5 | 250 nMの | 52.5 |
| トータル | 14 | 1470 |
表6ノロウイルスGIIのためのPCRマスターミックス(11月GII)アッセイ (1)。
脚注については、表3を参照してください(1) - (4)。
| 成分 | 反応あたり容量(μL)(2) | 最終濃度 | マスターミックスあたりの容量(μL)(3) |
| 2倍速ライトサイクラー480プローブマスターミックス | 10 | 所有権の | 1050 |
| ROXリファレンス色素(4) | 0.4 | 0.5 mMの | 42 |
| PCRグレードの水 | 0.3 | 31.5 | |
| 10μMのMuNoVF1 | 1 | 500 nMの | 105 |
| 10μMのMuNoVR1 | 1.8 | 900 nMの | 189 |
| 10μMのMuNoVP1 | 0.5 | 250 nMの | 52.5 |
| トータル | 14 | 1470 |
表7. PCRマスターミックスマウスノロウイルスアッセイのために (1)。
脚注については、表3を参照してください(1) - (4)。
| 成分 | ボリューム反応当たり(μL)(2) | 最終濃度 | マスターミックスあたりの容量(μL)(3) |
| 2倍速ライトサイクラー480プローブマスターミックス | 10 | 所有権の | 1050 |
| ROXリファレンス色素(4) | 0.4 | 0.5 mMの | 42 |
| PCRグレードの水 | 1.4 | 147 | |
| 10μMのHepF | 1 | 500 nMの | 105 |
| 10μMのHEPR | 1 | 500 nMの | 105 |
| 10μMのヘップ | 0.2 | 100 nMの | 21 |
| トータル | 14 | 1470 |
表8. G型肝炎のためのPCRマスターミックス(HGV)アッセイ (1)。
見る脚注については、表3(1) - (4)。
| 標準曲線濃度 | RT-qPCRのアッセイあたりゲノムコピー(1、2) |
| 2.5×10 8 | 502500 |
| 2.5×10 7 | 50250 |
| 2.5×10 6 | 5025 |
| 2.5×10 5 | 502.5 |
| 2.5×10 4 | 50.25 |
| 2.5×10 3 | 5.025 |
表9.標準曲線ゲノムコピー。
(1)標準として標準曲線ウェルを特定し、サーモサイクラーソフトウェアの適切な場所に、RT-qPCRのアッセイ値あたりゲノムコピーを配置します。
(2)許容可能な標準曲線は70%-110%の効率を持つことになります、R 2の値 > 0.97、およびノロウイルスのための全体的な標準偏差<0.5およびエンテロウイルスのための<1.0。
| 基準 | 許容値 | |
| ノロウイルス | エンテロウイルス属 | |
| 全体的な標準偏差 | <0.5 | <1.0 |
| R 2 | > 0.97 | > 0.97 |
| 効率 | 115%70% | 115%70% |
表10.標準曲線許容基準 (1)。
(1)70%-110%の%の効率で標準曲線は許容されるが、90%-115%の範囲内の値が理想的です。以下、90%以上の値は、ピペッティングまたは希釈エラーを示すことがあります。
| QAコンポーネント | 平均回収レンジ(%) | 変動係数 (%) |
| ラボ試薬ブランク。負のPTまたはPEのサンプル | 0 | N / A(1) |
| ラボブランク要塞。ラボ要塞サンプルマトリックス | 5-200 | N / A |
| ポジティブPTとPEサンプル | 15から175 | ≤130 |
表11.メソッド1615の性能基準。
(1)該当事項はありません。
| メディア | 組成 |
| 0.15 Mリン酸ナトリウム、pHが7.0〜7.5 | 1 LのdHの最終体積の二塩基性リン酸ナトリウムの40.2グラムを溶解し、0.15Mリン酸ナトリウム、し(Na 2 HPO 4・7H 2 O)を調製 2 O HClで7.0〜7.5にpHを調整します。 121℃、15分間、15 psiでオートクレーブ。最大12ヶ月間室温で保存リン酸ナトリウム溶液を得ました。 |
| 5%BSA | dH 2 Oで100mlにBSAを5g溶解することにより調製0.2μmの滅菌フィルターに溶液を通すことにより滅菌します。 |
| PBS、0.2%BSA | PBS 96 mlに、5%BSAを4mlを添加することにより準備します。 0.2μmの滅菌フィルターに溶液を通すことにより滅菌します。 |
| TSM IIIバッファ | 1.21グラムTrismaベース、5.84グラムのNaCl、0.203グラムのMgCl 2を、1ミリリットルPrionexゼラチン、および950ミリリットルの試薬 グレード水で3ミリリットルMicrocide IIIを溶解します。 pHを7.0に調整した後、0.2μmの滅菌フィルターに溶液を通すことにより、1 Lの滅菌するために、最終的なボリュームをもたらします。 |
| 0.525パーセントの次亜塩素酸ナトリウム(NaClOを) | (d)に家庭用漂白剤を1:10に希釈することにより、0.525パーセントのNaClO溶液を調製しますH 2 O.室温で1週間まで0.525パーセントのNaClOソリューションを保管してください。 |
| 1-Mチオ硫酸ナトリウム( の Na 2 S 2 O 3)五水和物 | dH 2 Oを 1 L中のNa 2 S 2 O 3の248.2グラムを溶解して、1M溶液を調製します室温で6ヶ月までのStoreチオ硫酸ナトリウム。 |
メディアの表12表。
ディスカッション
ソースと飲料水のウイルス汚染の大規模国家の研究では、複数の分析ラボを使用する必要があります。これらの条件下での標準的な方法は、複数の研究室で生成されたデータが比較可能であることを保証するために必要とされます。ウイルス検出のための多くの公表された分子的方法が、非常に少数の標準化された分子の方法があります。 EPAメソッド1615は、具体的には、RT-qPCRにより水マトリックス中エンテロウイルスやノロウイルスの検出のために設計された標準化された方法です。 16,17と春に肝炎の検出ウイルスとノロウイルスに適用されている。標準化された分子法は、食品(2013年4月7日CEN / ISO TS 15216から1とCEN / ISO TS 15216から2)でウイルス検出のために利用可能です水16。すべての標準的な方法は、実験室による汚染に間およびイントラ実験室変動や偽陽性のデータを最小限にするために品質性能のコントロールや基準を含める必要があります。さらに、Eを誤ったデータを低減するために、PA法1615は、処理し、一方通行の作業の流れの間に仕事の分離を定めた分子法のEPAの指導、18の後に続きます 。これは、G型肝炎1,19内部統制およびRT-qPCRの15の阻害剤に起因する偽陰性の結果を最小限に抑えるための手順が含まれています。これは、すべてのフィールドデータがフィールド1リットル当たりのゲノムコピーで表現または飲料水をサンプリングするようにサンプリングし、水を分析し、両方の水の標準化されたボリュームと一緒に定量的アッセイを使用しています。効率的な単一の管(ワンステップ)RT-PCRアッセイは、市販されているが、この方法は、意図的に独立したアッセイを使用します。これは、各反応でアッセイすることができる試料の量を最小化するという欠点を有しているが、複数のプライマーセットを使用することでより大きな柔軟性を与えます。 RT-のqPCRアッセイは、によって制限され、のみ使用プライマーおよびプローブと同じくらい良い、おそらく何のプライマーセットは、グループ内のすべてのウイルスの亜種を検出しませんされています。エンテロウイルスプライマーセットがあるため選ばれましたそれは、20,21はエンテロウイルス血清型の多種多様を検出し、それによって検出されたウイルスは、未処理の地下1の消費による健康への影響に関連している、保存された5 '非コード領域を標的とします。二つのプライマーセットは、Iが22,23 genogroupノロウイルスの検出のために使用されます。最初は、若い子供の健康への影響との間には強い相関関係に起因して選択し、ウイルス1を検出しました。彼らは株24,25の広い多様性を検出するので、私はプライマーセット第genogroupとgenogroup IIノロウイルスに使用するプライマーセットを選択しました。
水の中のウイルスRNAを検出するための定量PCRおよびRT-qPCRの手順の主な利点にもかかわらず、いくつかの制限があります。まず、消毒剤によって不活性化されるものを含む感染性および非感染性両方のウイルス粒子は、これらの方法によって増幅することができます。 Borchardtの結果は、これが未処理の地下水fの問題のあまりであることを示唆しています地域社会と同様のROMの帯水層は、消毒地表水1の場合よりも勉強しました。培養可能なウイルスのため、この問題は、培養物26,27との組み合わせでPCRを使用して克服することができます。この問題はまた、核酸架橋剤28-30の使用を介していくつかのウイルスの対処されてきました。この後者のアプローチは、ウイルス、次亜塩素酸により不活性化し、UVにより不活性化にとって有効ではないため、より効果的です。
これらの分子の手順の第2の制限は、典型的にアッセイすることができる濃縮試料の量は、培養手順6,31に使用されるものよりもはるかに小さいことです。この問題は、多くの場合、いずれかのサンプルは小さい体積中に再懸濁し、又は第三のサンプル濃縮工程6,32,33を添加することによってすることが可能な有機凝集による二次標準濃度のためのポリエチレングリコールベースの手順を置換することによって処理されます。方法1615は、centrifを使用していますugal限外ろ過は、第三級の濃度を提供します。遠心限外濾過、水と試験サンプル中の任意のウイルスの両方の濃度および分子アッセイの小分子インヒビターが低下コンポーネント30,000未満ダルトンを除去します。水中に存在した任意のウイルスのための> 10 5の全体的な濃縮係数のこの三次濃縮工程の結果がテストされています。
第三の制限は、環境試料中の分子の手順の阻害剤の存在です。阻害剤を除去するための多くのアプローチが開発されてきたが、何のアプローチは、本質的な阻害のレベルを推定するように設計された内部対照を利用して、すべての水マトリックスおよびウイルス型6,34,35のために有効ではありません。この方法で使用される肝炎G試薬は、すべての反応および抑制を推定するためのRT-qPCRのアッセイにおいて、ウイルスRNAの一定のレベルを提供することによって、この要件を満たします。とき、tの最高の彼は、サンプル濃縮物は限りウイルス濃度は、濃度14,15阻害剤よりも高くなっているように希釈することができ、除去アプローチは阻害を削除することができない阻害剤。
本明細書中に記載の標準曲線の手順は、利点と主要な制限の両方を持っています。利点は、試薬が、単一の制御は、全てのアッセイに使用されることを可能にする、単一の試薬に供給するに必要なすべてのコンポーネントを使用することです。この試薬は、特にノロウイルスアッセイのために有利です。ノロウイルス粒子は、それが非常に困難な標準として使用するためのウイルス粒子を得ることができる感染した個体から得ることができます。多くの重要な利点は、RNA標準は、RNA 36の正確な定量化のために不可欠であるものとして、それは、一つの試薬 のすべての標的のRNAウイルスのRNA標準を提供することです。しかし、正確にウイルスを定量する能力は、マトリックス効果は考慮されないという事実によって制限されます。これは、ゲノムのコピーを意味します数値は、絶対的とみなすことができず、相対的な用語でのみ考慮されるべきです。これは、標準曲線ワーキングストックのアリコートの十分な数(ステップ1.2)は、完全な研究をカバーするために準備することをお勧めします。例えば、各250μlのアリコートを6 RTプレートに十分な試薬を提供します。それは研究が500サンプルの分析が必要になることが知られている場合、12のアリコートの最小値は、(アリコートあたりRTプレート/ 6 RTプレートあたり500サンプル/ 7サンプル)を必要とされるであろう。
EPAメソッド1615は、パフォーマンスベースの方法です。多くのメーカーは、本明細書で指定されたものと同等の試薬を作り、これらの試薬があれば、性能基準が満たされているように置換され得ます。また、正のRT-qPCRのコントロールとして機能し、標準曲線は、パフォーマンスの問題のトラブルシューティングを行うには価値があります。性能は、RNAの分解、試薬貯蔵寿命、冷凍庫の故障、機器のキャリブレーション、および技術的なエラーに辞退することができます。パフォーマンスの問題が容疑者でなければなりませんこれらは標準曲線のための性能仕様を満たさない場合、EDは、標準曲線を図4に示したものと異なる場合、または。 RT-のqPCRアッセイは、非常に堅牢です。完全な失敗は、RNAまたは技術的なエラー(たとえば、欠けている試薬)の不適切な取り扱いに起因する可能性が高いです。細心の注意は、リボヌクレアーゼからRNAの分解を低減するために、抽出し、RT工程間のRNAサンプルの取り扱いに注意が必要です。
地下から地上と試薬グレードの水およびマウスノロウイルスの回収からポリオウイルスの回収率は、EPAメソッド1615の性能受け入れ基準( 表11)を満たし、他33,37,38によって報告されたものと類似しています。 LFBサンプルからのマウスノロウイルスの回収率は、ポリオウイルスのものよりはるかに低かったとポリオウイルス固有の合格基準を満たしていないだろう。試薬グレードの水から下のマウスノロウイルスの回復の理由は不明です。本明細書の結果と同様に、カリムとcolleaゲスは、水道水39から4%のノロウイルスGI.1の回復を報告しました。リーら 37は、マウスノロウイルスと18%の人間ノロウイルスGII.4それぞれ、ディスクフィルターを用いて蒸留水から26%の平均回収率を報告しました。リーや同僚と同様の条件を用いて、金とコはそれぞれ38、これらのウイルスのために46%と43%の回収率を観察しました。ギボンズら 40海水からのヒトノロウイルスGII.4の回収率100%を中心に得られるが、キムとコ38は、類似または海水よりも高い濃度で蒸留水に塩を添加すると、著しくマウスウイルスの回収率を低下させ、得られたことがわかりましたGII.4回復で約2倍の減少です。
開示事項
The authors have nothing to disclose.
謝辞
The authors thank Dr. Eric Rhodes for preparing the clones used in the development of Standard curve reagent, Brian McMinn for assistance in sample processing, Larry Wymer for statistical analysis, Dr. Mark Borchardt, U.S. Department of Agriculture, Marshfield, WI, for supplying the Sabin poliovirus serotype 3 used in this study and Dr. H. W. Virgin, Washington University, St. Louis, MO, for murine norovirus. The authors also acknowledge Gretchen Sullivan for assistance in preparation of stock laboratory reagents, Dr. Mohammad Karim for propagation of murine norovirus stocks, and local private well owners and utilities for allowing us to collect water samples. Although this work was reviewed by EPA and approved for publication, it may not necessarily reflect official Agency policy. Mention of trade names or commercial products does not constitute endorsement or recommendation for use.
資料
| Name | Company | Catalog Number | Comments |
| 1.5 ml tube chamber | Diversified Biotech | CHAM-3000 | |
| 1 °C cool brick | Diversified Biotech | BRIK-2501 | |
| 10x PCR Buffer II and 25-mM MgCl2 | Life Technologies | N8080130 | |
| -20 °C freezer | VWR | 97043-346 | Must be a manual defrost freezer |
| -70 °C or colder freezer | Thermo Scientific | MBF700LSAO-E | |
| 96-well chamber | Diversified Biotech | CHAM-1000 | |
| Absolute ethanol | Fisher Scientific | BP2818-100 | |
| Armored RNA EPA-1615 | Asuragen | Custom order | Used for quantifying the RT-qPCR assay |
| Armored RNA Hepatitis G virus | Asuragen | 42024 | |
| Autoclave | Steris | Amsco Lab Series | |
| Biosafety cabinet | NuAir Laboratory Equipment Supply | Labgard 437 ES | |
| Bovine serum albumin (BSA) | Affymetrix | 10856 | Crystalline grade or better |
| Buffer AVE | Qiagen | 1026956 | Carrier RNA dilution buffer |
| Buffer AVL | Qiagen | 19073 | Extraction buffer |
| Carrier RNA | Qiagen | Not applicable | Use carrier RNA supplied with Buffer AVL |
| Centrifuge bottles | Fisher Scientific | 05-562-23 or 05-562-26 | |
| Centrifuge rotors | Beckman Coulter | 339080, 336380 | |
| Cool safe box | Diversified Biotech | CSF-BOX | |
| Dithiothreitol (DTT) | Promega | P1171 | |
| LightCycler® 480 Probes Master kit | Roche Diagnostics | 4707494001 | |
| Microcentrifuge tubes | Fisher Scientific | 02-682-550 | Use ribonuclease- and deoxyribonuclease-free tubes with snap caps |
| Microcide III | Fitzgerald | 99R-103 | |
| Microseal 'A' film | Bio-Rad Laboratories | HSA5001 | Heat resistant |
| Microseal 'F' film | Bio-Rad Laboratories | MSA1001 | Freezer resistant |
| Mini-plate spinner | Labnet International | MPS1000 | |
| Multichannel pipette | Rainin | L8-20 | |
| Multi-tube chamber | Diversified Biotech | CHAM-5000 | |
| Optical reaction plate | Life Technologies | 4314320 | |
| PCR nucleotide mix | Promega | U1515 | |
| PCR plate | Bio-Rad Laboratories | HSS9601 | |
| PCR-grade water | Roche | 3315932001 | |
| Phosphate buffered saline (PBS) | U.S. Biological | D9820 | |
| Plate mixer | Scientific Industries | MicroPlate Genie | |
| Prionex gelatin | Sigma Aldrich | G0411 | |
| QIAamp DNA Blood Mini Kit | Qiagen | 51104 | Includes Buffers AW1 (first wash buffer), AW2 (second wash buffer), AE (elution buffer); ethanol must be added to Buffers AW1 and AW2 before use; Do not use Buffer AL supplied with the kit |
| Quantitative PCR thermal cycler | Life Technologies | 4351405 | |
| Random primer | Promega | C1181 | |
| Reagent Reservoir | Fisher Scientific | 21-381-27E | |
| Refrigerated centrifuge | Beckman Coulter | 367501 | |
| RNase Inhibitor | Promega | N2515 or N2615 | |
| ROX reference dye | Life Technologies | 12223 | |
| SuperScript II or III Reverse Transcriptase | Life Technologies | 18064-022 or 18080044 | |
| Surgical gloves | Fisher Scientific | 19-058-800 | |
| Thermal cycler | Life Technologies | 4314879 | |
| Trisma base | Sigma Aldrich | T1503 | |
| Vivaspin 20 centrifugal concentrator units | Sartorius-Stedim | VS2022 |
参考文献
- Borchardt, M. A., Spencer, S. K., Kieke, B. A., Lambertini, E., Loge, F. J. Viruses in nondisinfected drinking water from municipal wells and community incidence of acute gastrointestinal illness. Environ. Health Perspect. 120 (9), 1272-1279 (2012).
- Ye, X. Y., et al. Real-time PCR detection of enteric viruses in source water and treated drinking water in Wuhan, China. Curr. Microbiol. 65 (3), 244-253 (2012).
- Williamson, W. M., et al. Enteric viruses in New Zealand drinking-water sources. Water Sci. Technol. 63 (8), 1744-1751 (2011).
- Lambertini, E., Borchardt, M. A., Kieke, B. A., Spencer, S. K., Loge, F. J. Risk of viral acute gastrointestinal illness from nondisinfected drinking water distribution systems. Environ. Sci. Technol. 46 (17), 9299-9307 (2012).
- Chigor, V. N., Okoh, A. I. Quantitative RT-PCR detection of hepatitis A virus, rotaviruses and enteroviruses in the Buffalo River and source water dams in the Eastern Cape Province of South Africa. Int. J. Environ. Res. Public Health. 9 (11), 4017-4032 (2012).
- Fout, G. S., Martinson, B. C., Moyer, M. W., Dahling, D. R. A multiplex reverse transcription-PCR method for detection of human enteric viruses in groundwater. Appl. Environ. Microbiol. 69 (6), 3158-3164 (2003).
- Hewitt, J., Bell, D., Simmons, G. C., Rivera-Aban, M., Wolf, S., Greening, G. E. Gastroenteritis outbreak caused by waterborne norovirus at a New Zealand ski resort. Appl. Environ. Microbiol. 73 (24), 7853-7857 (2007).
- Jack, S., Bell, D., Hewitt, J. Norovirus contamination of a drinking water supply at a hotel resort. New Zealand Med. J. 126 (1387), 98-107 (2013).
- Anderson, A. D., et al. A waterborne outbreak of Norwalk-like virus among snowmobilers-Wyoming, 2001. J. Infect. Dis. 187 (2), 303-306 (2003).
- Parshionikar, S. U., et al. Waterborne outbreak of gastroenteritis associated with a norovirus. Appl. Environ. Microbiol. 69 (9), 5263-5268 (2003).
- U.S. Environmental Protection Agency Office of Water. . 820-F-12-058: Recreational Water Quality Criteria. , 1-63 (2012).
- Wade, T. J., et al. Rapidly measured indicators of recreational water quality and swimming-associated illness at marine beaches: a prospective cohort study. Environ. Health. 9, 66 (2010).
- Fout, G. S., et al. . Method 1615: Measurement of enterovirus and norovirus occurrence in water by culture and RT-qPCR (EPA/600/R-10/181). , 1-91 (2012).
- Cashdollar, J. L., et al. Development and Evaluation of EPA Method 1615 for Detection of Enterovirus and Norovirus in Water. Appl. Environ. Microbiol. 79 (1), 215-223 (2013).
- Gibson, K. E., Schwab, K. J., Spencer, S. K., Borchardt, M. A. Measuring and mitigating inhibition during quantitative real time PCR analysis of viral nucleic acid extracts from large-volume environmental water samples. Water Res. 46 (13), 4281-4291 (2012).
- Fuentes, C., et al. Standardized multiplex one-step qRT-PCR for hepatitis A virus, norovirus GI and GII quantification in bivalve mollusks and water. Food Microbiol. 40, 55-63 (2014).
- Coudray, C., Merle, G., Martin-Latil, S., Guillier, L., Perelle, S. Comparison of two extraction methods for the detection of hepatitis A virus in lettuces using the murine norovirus as a process control. J. Virol. Methods. 193 (1), 96-102 (2013).
- Sen, K., et al. . EPA 815-B-04-001: Quality assurance/quality control guidance for laboratories performing PCR analyses on environmental samples. , 1-56 (2004).
- Schlueter, V., Schmolke, S., Stark, K., Hess, G., Ofenloch-Haehnle, B., Engel, A. M. Reverse transcription-PCR detection of hepatitis G virus. J. Clin. Microbiol. 34 (11), 2660-2664 (1996).
- De Leon, R., Shieh, C., Baric, R. S., Sobsey, M. D. Detection of enteroviruses and hepatitis A virus in environmental samples by gene probes and polymerase chain reaction. Proc. Water Qual. Technol. Conf. , 833-853 (1990).
- Monpoeho, S., et al. Quantification of enterovirus RNA in sludge samples using single tube real-time RT-PCR. BioTechniques. 29 (1), 88-93 (2000).
- Jothikumar, N., et al. Rapid and sensitive detection of noroviruses by using TaqMan-based one-step reverse transcription-PCR assays and application to naturally contaminated shellfish samples. Appl. Environ. Microbiol. 71 (4), 1870-1875 (2005).
- da Silva, A. K., et al. Evaluation of removal of noroviruses during wastewater treatment, using real-time reverse transcription-PCR: different behaviors of genogroups I and II. Appl. Environ. Microbiol. 73 (24), 7891-7897 (2007).
- Butot, S., et al. Evaluation of various real-time RT-PCR assays for the detection and quantitation of human norovirus. J. Virol. Methods. 167 (1), 90-94 (2010).
- Loisy, F., et al. Real-time RT-PCR for norovirus screening in shellfish. J. Virol. Methods. 123 (1), 1-7 (2005).
- Reynolds, K. A., Gerba, C. P., Pepper, I. L. Detection of infectious enteroviruses by an integrated cell culture-PCR procedure. Appl. Environ. Microbiol. 62 (4), 1424-1427 (1996).
- Ming, H. X., Zhu, L., Zhang, Y. Rapid quantification of infectious enterovirus from surface water in Bohai Bay, China using an integrated cell culture-qPCR. Mar. Pollut. Bull. 62 (10), 2047-2054 (2011).
- Parshionikar, S., Laseke, I., Fout, G. S. Use of propidium monoazide in reverse transcriptase PCR to distinguish between infectious and noninfectious enteric viruses in water samples. Appl. Environ. Microbiol. 76 (13), 4318-4326 (2010).
- Sanchez, G., Elizaquivel, P., Aznar, R. Discrimination of infectious hepatitis A viruses by propidium monoazide real-time RT-PCR. Food Environ. Virol. 4 (1), 21-25 (2012).
- Kim, S. Y., Ko, G. Using propidium monoazide to distinguish between viable and nonviable bacteria, MS2 and murine norovirus. Lett. Appl. Microbiol. 55 (3), 182-188 (2012).
- Fout, G. S., Schaefer, F. W., Messer, J. W., Dahling, D. R., Stetler, R. E. . EPA/600/R-95/178: ICR Microbial Laboratory Manual, I.1-ApD-23. , (1996).
- Abbaszadegan, M., Stewart, P., LeChevallier, M. A strategy for detection of viruses in groundwater by PCR. Appl. Environ. Microbiol. 65 (2), 444-449 (1999).
- Lambertini, E., et al. Concentration of enteroviruses, adenoviruses, and noroviruses from drinking water by use of glass wool filters. Appl. Environ. Microbiol. 74 (10), 2990-2996 (2008).
- Rodriguez, R. A., Thie, L., Gibbons, C. D., Sobsey, M. D. Reducing the effects of environmental inhibition in quantitative real-time PCR detection of adenovirus and norovirus in recreational seawaters. J. Virol. Methods. 181 (1), 43-50 (2012).
- Iker, B. C., Bright, K. R., Pepper, I. L., Gerba, C. P., Kitajima, M. Evaluation of commercial kits for the extraction and purification of viral nucleic acids from environmental and fecal samples. J. Virol. Methods. 191 (1), 24-30 (2013).
- Fey, A., et al. Establishment of a real-time PCR-based approach for accurate quantification of bacterial RNA targets in water, using Salmonella as a model organism. Appl. Environ. Microbiol. 70 (6), 3618-3623 (2004).
- Lee, H., et al. Evaluation of electropositive filtration for recovering norovirus in water. J. Water Health. 9 (1), 27-36 (2011).
- Kim, M., Ko, G. Quantitative characterization of the inhibitory effects of salt, humic acid, and heavy metals on the recovery of waterborne norovirus by electropositive filters. J. Water Health. 11 (4), 613-622 (2013).
- Karim, M. R., Rhodes, E. R., Brinkman, N., Wymer, L., Fout, G. S. New electropositive filter for concentrating enteroviruses and noroviruses from large volumes of water. Appl. Environ. Microbiol. 75 (8), 2393-2399 (2009).
- Gibbons, C. D., Rodriguez, R. A., Tallon, L., Sobsey, M. D. Evaluation of positively charged alumina nanofibre cartridge filters for the primary concentration of noroviruses, adenoviruses and male-specific coliphages from seawater. J. Appl. Microbiol. 109 (2), 635-641 (2010).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved