È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Spettroscopia di Risonanza Magnetica dal vivo Drosophila melanogaster Utilizzando Magic Angle Spinning
In questo articolo
Riepilogo
Questa tecnica consente l'utilizzo di alta risoluzione magic angle spinning spettroscopia RM del protone (1H-MRS HRMAS) per la caratterizzazione molecolare di vivere Drosophila melanogaster Con un tradizionale 14,1 tesla spettrometro dotato di una sonda HRMAS.
Abstract
Ad alta risoluzione magico angolo Spinning (HRMAS) spettroscopia di risonanza magnetica protonica (
Protocollo
Parte 1: Preparazione della Drosophila per le misure HRMAS
- Utilizzando le normali procedure flylab 1, raccogliere nuovi eclosing vola per 3 giorni e trasferirli a volare flaconi contenenti prodotti alimentari freschi volare. Incubare le mosche raccolti per 5 giorni in modo che le mosche diventerà 5-8 giorni vecchio appena prima dell'esperimento. Solo sesso (in genere maschi) viene utilizzato per gli esperimenti.
- Uso sano, integro tipo selvatico vola da confrontare con trattati esempio traumatizzati o geneticamente modificati vola 1.
- Luogo vola sola in provette da 2 ml contenente un pezzo (~ 0,2 ml) di volare cibo 24 ore prima dell'esperimento. Questi tubi sopportare un buco nella coppa effettuata utilizzando un tubo di fiamma riscaldato ago da insulina.
- Pesare i tubi immediatamente prima e dopo l'inserimento volo usando una bilancia ad alta precisione e definire il peso di ogni volo s sottraendo il primo valore da quest'ultima. Una mosca singolo maschio pesa generalmente 0,7-1 mg.
Parte 2: Preparazione del rotore HRMAS.
- Posizionare un unico tubo in ghiaccio per meno di un minuto per anestetizzare la parte interna volare.
- Posare il volo su uno strato di foglio di alluminio posti su ghiaccio e spingere il volano dolcemente nello spazio vuoto semisferica dell'inserto rotore NMR con una spazzola morbida per evitare un inserimento completo volare evitando lesioni volare.
- Collocare l'inserto in ossido di zirconio (ZrO 2) tubo rotore (4 mm di diametro, 50 ml) per individuare le mosche tra l'inserto del rotore e la parte inferiore del rotore (vedi figura 1).
- Chiudere l'inserto con una vite e coprire con parafilm (vedi figura 1) per evitare il contatto tra la mosca e la soluzione TSP standard (TSP: trimetilsilil-propionico-2 ,2,3,3-d4 acido, Mw = 172, δ = 0,00 ppm, 50 mm in deuterato acqua-D 2 O, che funzionano come un punto di riferimento per entrambi i turni di risonanza chimica e quantificazione).
- Aggiungere 8 ml di soluzione standard di TSP in cima al rotore inserto. (Vedi figura 1).
- Fissare e serrare il tutto allestito nella rotori con un tappo superiore (vedi figura 1).
Parte 3: HRMAS Data Acquisition
- Introdurre il rotore nella sonda HRMAS e posizionarlo in un angolo magico 54,7 ° (vedi figura 1).
- Impostare la temperatura a 4 ° C da un BTO-2000 unità in combinazione con il pneumatico MAS. Le mosche sono conservati a 4 ° C, mentre nello spettrometro per mantenere l'anestesia.
- Impostare il HRMAS 1 H MRS velocità di rotazione a 2 MAS kHz.
- Stabilizzare la rotazione MAS frequenza a 2,0 ± 0,001 kHz da un regolatore di velocità MAS (frequenza di rotazione).
- Per la preparazione magnete: sintonia e corrispondenza della bobina per ottimizzare prestazioni e spessore il magnete per una qualità ottimale degli spettri.
- Acquistare una dimensione 1 spettri H con rotore sincronizzato Carr-Purcell-Meiboom-Gill (CPMG) sequenza di spin echo impulsi 2, [90 ° - (τ-180 ° - τ) n-acquisizione], che funziona come un filtro a 2 T rimuovere l'ampliamento dello spettro 1 H NMR singolo volo di spettri acquisiti unidimensionale dati su tutti i campioni. Sincronizzare l'inter-impulso ritardo (τ = 500μs) per la frequenza di rotazione MAS (2 kHz). Impostare il numero dei transitori a 256 con 32.768 (32k) punti di dati. Tempo di acquisizione 9 min.
- Acquisire bidimensionali (2D) 1 H-1 H NMR HRMAS singolo volo spettri su tutti i campioni utilizzando una sequenza TOBSY con adiabatico impulsi 3. Parametri di acquisizione sono: dimensione 2k punti dati diretto (11 ppm larghezza spettrale), acqua 1 s pre-saturazione durante il ritardo di rilassamento, 8 scansioni per incremento, 128 incrementi, tempo di ripetizione totale di 2 s, 45 ms di tempo di miscelazione, e un acquisizione totale tempo di 29 min.
Parte 4: elaborazione dei dati / Analisi
- Analizzare la MRspectra di campioni utilizzando MestReC software (Mestrelab Ricerca, www.mestrec.com)
- Utilizzare una linea-ampliamento funzione apodizzazione di 0,5 Hz e si applicano a tutti HRMAS FID 1 H prima trasformata di Fourier.
- Gli spettri di riferimento MR rispetto a TSP a δ = 0,0 ppm (standard esterno).
- Fase di spettri e di applicare manualmente uno stimatore di base Whittaker per sottrarre i componenti generali della linea di base prima del calcolo delle aree dei picchi.
- Stima delle aree dei picchi utilizzando il software MestReC. Scala di altezze del picco rispetto a TSP per ogni spettro acquisito (TSP picco altezza = 1). Usa t-test (a due code, p <0,05) per confrontare all'interno del gruppo per i rapporti tra i gruppi.
- 2D TOBSY parametri di processo sono: QSINE = 2 funzione finestra in entrambe le dimensioni, FT con punti 2k nella dimensione diretta e priva di riempimento al 1k nella seconda dimensione, la correzione di fase in entrambe le dimensioni e la correzione al basale nella seconda dimensione.
- Produrre gli spettri 2D utilizzando il programma Sparky (TD Goddard e DG Kneller, SPARKY 3, USCF, http://www.cgl.ucsf.edu/home / Sparky /)
Parte 5: Spectra Rappresentante da Live Drosophila melanogaster Flies
Le procedure sopra descritte consentono di raccogliere spettri riproducibile dal vivo le mosche Drosophila melanogaster. Figure 2 e 3 mostrano rappresentante MR spettri acquisiti nel wild-type (WT) Oregon-R mosche. La figura 2 presenta 1D 1 H HRMAS spettri CPMG. Componenti lipidici principali [C H 3 (0,89 ppm), (C H 2) n (1,33 ppm), C H 2 C-CO (1.58ppm), C H 2 C = C (2,02 ppm), C H 2 C = O (2,24 ppm), C H = C H (5,33 ppm)], glicerolo (1,3-C H 4,10 ppm e 4,30 ppm; 2-CH 2 5,24 ppm), e piccoli metaboliti: β-Alanina (β-Ala , 2,57 ppm), acetato (At, 1,97 ppm), phosphocholine (PC, 3.22 ppm) e phophoetanolamine (PE, 3.23ppm) sono stati rilevati e assegnate in base ai rapporti prima del 4, 5. Segnali a 2,02 ppm sono stati assegnati ai protoni del metilene H2 C-CH = CH porzione di acidi grassi mono-insaturi (cioè palmitoleico). Gli acidi grassi insaturi sono stati identificati da un segnale a 5,33 ppm prodotta da protoni del-CH = CH-porzione. Metaboliti piccole dimensioni che non potevano essere assegnati o non erano visibili utilizzando lo spettro 1D sono stati rilevati utilizzando 2D 1 H-1 H TOBSY HRMAS (vedi figura 3).

Figura 1. Sperimentale messa a punto di in vivo HRMAS 1H MRS per lo studio della Drosophila vivere al 14,1 T. esterna di serie trimetilsilil-propionico-2 ,2,3,3-d4 acido in acqua deuterato (TSP / D 2 O). Nella piazza: posizione del rotore con un angolo magico nel HRMAS sonda.
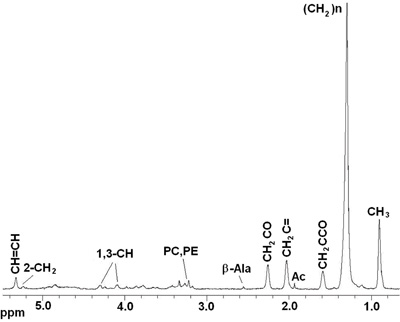
Figura 2. In vivo 1D HRMAS 1 spettri H CPMG di un live mosca Drosophila melanogaster peso. Componenti lipidici: C H 3 (0,89 ppm), (C H 2) n (1,33 ppm), C H 2 C-CO (1.58ppm), acetato (Ac), C H 2 C = C (2,02 ppm), C H 2 C = O (2,24 ppm), β-Alanina (β-Ala), phosphocholine (PC) e phophoetanolamine (PE), glicerolo (1,3-C H 4,10, 4,30 ppm; 2-C H 2 5,22 ppm) , C H = C H (5,33 ppm).
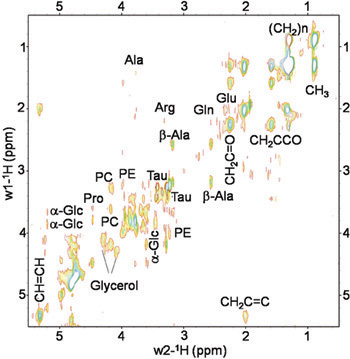
Figura 3. In vivo 2D 1H-1H TOBSY HRMAS spettro di un moscerino Drosophila melanogaster peso vivo al 14,1 metaboliti T. Piccolo e componenti lipidici sono stati identificati. Metaboliti: alanina (Ala), β-Alanina (β-Ala), arginina (Arg), glutamina (Gln), glutammato (Glu), phosphocholine PC (PC), phophoetanolamine (PE), Taurina (Tau), α-glucosio (α-Glc) e glicerolo. Componenti Lipidi: C H 3 (0,89 ppm), (C H 2) n (1,33 ppm), C H 2 C-CO (1,58 ppm), C H 2 C = C (2,02 ppm), C H 2 C = O (2,24 ppm), C H = C H (5,33 ppm).
Discussione
Con l'eccezione del recente rapporto della fattibilità di risonanza magnetica in vivo nei moscerini della frutta 6, studi in vivo MRS in Drosophila non sono ancora stati segnalati. Nel presente protocollo, si descrive l'implementazione di un romanzo in vivo HRMAS 1 H-MRS approccio per la rilevazione di molecole biologicamente importanti. In particolare, abbiamo rilevato lipidi e metaboliti piccoli vivono Drosophila vola al 14,1 T in circa 45 min...
Divulgazioni
Riconoscimenti
Questo lavoro è stato sostenuto in parte da una National Institutes of Health (NIH) a concedere AI063433 Laurence G. Rahme, una Istituti National Institute of Health (NIH) Centro Grant (P50GM021700) di Ronald Tompkins G. (A. Tzika Aria, Direttore del il nucleo NMR), e un Ospedale per i bambini Shriner assegno di ricerca (# 8893) ad A. Aria Tzika. Ringraziamo Dionyssios Mintzopoulos Ph.D. per l'assistenza nelle fasi iniziali di sviluppo di questo protocollo e Ovidiu C. Andronesi Ph.D. per l'assistenza con la sequenza di impulsi TOBSY.
Materiali
| Name | Company | Catalog Number | Comments | |
| Deuterium oxide | Reagent | Sigma-Aldrich | 7789-20-0 | |
| 3-(trimethylsilyl)propionic-2,2,3,3-d4 acid | Reagent | Sigma-Aldrich | 24493-21-8 | |
| agar, sucrose, yeast, cornmeal | Food | Genesee Scientific | http://www.flystuff.com/ | |
| Oregon RS or Canton-S flies | Adult fly lines | Bloomington Stock center | http://flystocks.bio.indiana.edu/ | |
| Paintbrush | Equipment | (size 0) | ||
| 2ml tubes | Equipment | Fisher Scientific | K749521-1590 | |
| Fly incubators | Equipment | high humidity capacity (60-75%), adjustable temperature, and a 12 h:12 h light: dark cycle. | ||
| Bruker Bio-Spin Avance NMR spectrometer (600.13 MHz) 4mm triple resonance (1H, 13C, 2H) HRMAS probe | Equipment | Bruker Corporation | ||
| BTO-2000 unit in combination with a MAS pneumatic unit | Equipment | Bruker Corporation | ||
| 4mm zirconium oxide rotor (capacity 50 ul) | Equipment | Bruker Corporation | B3829 (Bruker store) | |
| MestReC (Mestrelab Research) | Software | 1D NMR spectra analysis http://mestrelab.com/ | ||
| SPARKY 3, USCF | Software | 2D NMR spectraanalysis http://www.cgl.ucsf.edu/home/sparky/ |
Riferimenti
- Apidianakis, Y., Rahme, L. G. Drosophila melanogaster as a model host for studying Pseudomonas aeruginosa infection. Nat Protoc. 4, 1285-1294 (2009).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation time. Rev Sci Instrum. 29, 688-691 (1958).
- Andronesi, O. C., Mintzopoulos, D., Struppe, J., Black, P. M., Tzika, A. A. Solid-state NMR adiabatic TOBSY sequences provide enhanced sensitivity for multidimensional high-resolution magic-angle-spinning 1H MR spectroscopy. J Magn Reson. 193, 251-258 (2008).
- Astrakas, L. G. Proton NMR spectroscopy shows lipids accumulate in skeletal muscle in response to burn trauma-induced apoptosis. Faseb J. 19, 1431-1440 (2005).
- Fan, T. W. M. Metabolite profiling by one- and two dimensional NMR analysis of complex mixtures. Prog Nuc Magn Reson Spec. 28, 161-219 (1996).
- Null, B., Liu, C. W., Hedehus, M., Conolly, S., Davis, R. W. High-resolution, in vivo magnetic resonance imaging of Drosophila at 18.8 Tesla. PLoS One. 3, e2817-e2817 (2008).
- Zektzer, A. S. Improved signal to noise in high-resolution magic angle spinning total correlation spectroscopy studies of prostate tissues using rotor-synchronized adiabatic pulses. Magn Reson Med. 53, 41-48 (2005).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon