Method Article
Hi-C: un metodo per studiare l'architettura tridimensionale dei genomi.
* Questi autori hanno contribuito in egual misura
In questo articolo
Erratum Notice
Riepilogo
L'Hi-C metodo consente l'identificazione imparziali, genoma delle interazioni della cromatina (1). Hi-C coppie legatura di prossimità e di sequenziamento massicciamente parallelo. I dati ottenuti possono essere usati per studiare architettura genomica alle scale multiple: risultati iniziali identificate caratteristiche come territori cromosoma, la segregazione della cromatina aperti e chiusi, e la struttura della cromatina alla scala megabase.
Abstract
Tridimensionale piegatura dei cromosomi del genoma e suddivide e può portare lontano elementi funzionali, come promotori e stimolatori, in stretta prossimità spaziale 2-6. Decifrare il rapporto tra organizzazione e l'attività dei cromosomi del genoma sarà di aiuto nella comprensione dei processi genomica, come la trascrizione e la replicazione. Tuttavia, poco si sa su come i cromosomi volte. Microscopia è in grado di distinguere un gran numero di loci contemporaneamente o in alta risoluzione. Ad oggi, il rilevamento di interazioni con conformazione cromosomica catturare cromosoma (3C) e la sua successivi adeguamenti necessari alla scelta di una serie di loci bersaglio, rendendo genome-wide studi impossibile 7-10.
Abbiamo sviluppato Hi-C, un'estensione della 3C che è in grado di identificare le interazioni a lungo raggio in un imparziali, genoma della moda. In Hi-C, le cellule vengono fissate con formaldeide, causando loci interagendo ad essere legati l'uno all'altro mediante covalente DNA-proteine cross-link. Quando il DNA è frammentato in seguito con un enzima di restrizione, questi loci rimangono collegati. Un residuo biotinilato è incorporato come il 5 'sbalzi sono riempiti in seguito, smussato-end legatura viene effettuata in condizioni di diluire gli eventi che favoriscono la legatura tra reticolato frammenti di DNA. Il risultato è un genoma libreria di prodotti legatura, corrispondenti a coppie di frammenti che erano originariamente in stretta vicinanza l'uno all'altro nel nucleo. Ogni prodotto legatura è contrassegnato con la biotina al sito della giunzione. La biblioteca è tosato, e le giunzioni sono tirato verso il basso con perline streptavidina. Le giunzioni purificato può essere successivamente analizzati utilizzando un elevato throughput sequencer, risultando in un catalogo di frammenti interagenti.
Analisi diretta della matrice contatto risultante rivela numerose funzioni di organizzazione genomica, come la presenza dei territori cromosomici e l'associazione preferenziale di piccoli geni ricchi cromosomi. Analisi di correlazione può essere applicata alla matrice di contatto, a dimostrazione che il genoma umano è suddivise in due comparti: un comparto meno densamente contenente cromatina aperta, accessibile e attivo e uno scomparto più denso contenente chiuso, regioni inaccessibili, e inattivi cromatina. Infine, l'analisi d'insieme della matrice di contatto, accoppiato con derivazioni teoriche e simulazioni computazionali, ha rivelato che su scala megabase Hi-C rivela caratteristiche coerenti con una conformazione globulo frattale.
Protocollo
Questo metodo è stato utilizzato nella ricerca riportata in Lieberman-Aiden et al. Science 326, 289-293 (2009) .
I. La reticolazione, digestione, Marcatura dei Ends DNA, e Blunt-end Legatura
- Hi-C inizia con reticolazione delle cellule, che è un filo comune tra tutti i metodi basati 3C. Per iniziare, crescere tra 2 x 10 7 e 2,5 x 10 7 cellule di mammifero, sia aderente o in sospensione, e crosslink le cellule. (Per i dettagli sulla reticolazione delle cellule, si veda: 11
- Lisare le cellule in 550 microlitri lysisbuffer (500 microlitri 10 mM Tris-HCl pH 8.0, 10 mM NaCl, 0,2% Igepal CA-630 e 50 gli inibitori della proteasi microlitri) utilizzando un omogeneizzatore. Spin la cromatina a 5.000 giri e lavare il pellet due volte con 500 microlitri 1x NEBuffer 2.
- Risospendere la cromatina in 1x NEBuffer 2, aliquote in 5 tubi numerati e aggiungere 1x NEBuffer 2 per un volume finale di 362 microlitri. Aggiungere 38 microlitri SDS 1%, mescolare con cura e incubare a 65 ° C per 10 minuti. Posizionare i tubi di nuovo sul ghiaccio subito dopo l'incubazione.
- Placare la SDS con l'aggiunta di 44 microlitri Triton X-100 e mescolare con cura. Digerire la cromatina con l'aggiunta di 400 Unità di HindIII e incubare a 37 ° C per una notte durante la rotazione.
- I passi successivi sono Hi-C specifico e comprendono la marcatura del DNA si conclude con la biotina e l'esecuzione di brusco-end legatura di frammenti reticolato. Questo passo permetterà giunzioni legatura di essere purificata in seguito. Tubo 1 non deve essere sottoposta al passo biotinilazione e dovrebbe invece essere tenuti separati e servire come controllo 3C per garantire che la digestione, e le condizioni di legatura sono stati ottimali.
- Per compilare frammento sovrasta la restrizione e segnare il DNA si conclude con biotina nei restanti 4 tubi, aggiungere 1,5 microlitri 10 mM dATP, 1,5 microlitri 10 mM dGTP, 1,5 microlitri 10 mM dTTP, 37,5 microlitri 0,4 mm biotina-14-dCTP, e 10 microlitri Klenow 5U/μl ai tubi 2-5. Mescolare con cura e incubare per 45 minuti a 37 ° C.
- Collocare le provette su ghiaccio. Per inattivare gli enzimi, aggiungere 86 microlitri SDS 10% ai tubi 1-5. Incubare le provette a 65 ° C per 30 minuti esatti e disporli sul ghiaccio subito dopo.
- La legatura è eseguita in condizioni estremamente diluite per favorire eventi legatura tra frammenti reticolato. Lavorando su ghiaccio, aggiungere 7,61 ml di mix legatura [745 microlitri 10% Triton X-100, 745 microlitri di buffer legatura 10x (500 mM Tris-HCl pH 7,5, 100 mM MgCl 2, 100 mM DTT), 80 microlitri 10 mg / ml BSA , 80 microlitri 100 mM ATP e 5,96 ml di acqua] per ciascuna delle cinque numerati provette da 15 ml. Trasferimento di ogni miscela cromatina digerito ad un corrispondente tubo 15 ml.
- Per regolare la legatura 3C, aggiungere 10 microlitri 1U/μl DNA ligasi T4 al tubo 1. Per smussato-end Hi-C legatura, aggiungere 50 microlitri 1U/μl DNA ligasi T4 ai tubi 2-5. Miscelare invertendo i tubi e incubare tutti e 5 i tubi per 4 ore a 16 ° C.
- Legami crociati sono invertiti e la proteina è degradata aggiungendo 50 microlitri 10 mg / ml di proteinasi K per tubo e incubando i tubi notte a 65 ° C. Aggiungi un ulteriore 50 microlitri 10 mg / ml di proteinasi K al tubo del giorno dopo e continuare l'incubazione a 65 ° C per altre 2 ore.
- Raffreddare le miscele di reazione a temperatura ambiente e trasferirli a cinque provette da 50 ml. Purificare il DNA in questi tubi eseguendo una estrazione fenolo. Aggiungere 10 ml di fenolo pH 8.0 e vortex per 2 minuti. Spin i tubi per 10 minuti a 3.500 giri e con attenzione trasferimento come gran parte della fase acquosa possibile ad un nuovo tubo da 50 ml.
- Ripetere l'estrazione con fenolo pH 8.0: cloroformio (1:1) e precipitare il DNA con etanolo. (Per i dettagli sulla purificazione del DNA, vedere: 11
- Dopo centrifugazione della etanolo precipitato DNA, sciogliere ogni pellet di DNA in 450 microlitri 1x TE (10 mM Tris-HCl pH 8.0, 1 mM EDTA). Trasferire la miscela di DNA in una provetta da 1,7 ml per centrifuga.
- Un altro giro di purificazione viene eseguita facendo 2 fenolo: cloroformio estrazioni. Aggiungere 500 microlitri fenolo pH 8.0: cloroformio (1:1) e vortex per 1 minuto. Centrifugare le provette per 5 minuti a 14.000 rpm e trasferire la fase acquosa in una nuova provetta. Dopo la seconda estrazione, precipitare il DNA con l'aggiunta di volumi di NaOAc 0.1x, 2x il volume del 100% di etanolo e incubare 30 minuti a -80 ° C.
- Dopo la fase di arresto il DNA precipitato, lavare ogni pellet di DNA con il 70% di etanolo e risospendere ogni pellet di DNA in 25 microlitri 1x TE. Degradare qualsiasi RNA che potrebbero essere presenti con l'aggiunta di 1 ml a 1 mg / ml RNasi Un tubo per l'incubazione e le provette per 30 minuti a 37 ° C. Piscina Hi-C contenuti di tubi 2-5, mantenendo separati tubo da 1 come controllo 3C.
- Ora è una buona occasione per esaminare la Hi-C marcatura e l'efficienza legatura. Questi controlli sono eccellenti indicatori di se un Hi-libreria C sta per avere successo.
- Per controllare la qualità e la quantità delle librerie, eseguire 2 microlitri e 6 aliquote ml di diluizioni 1:10 da entrambe le 3C e Hi-C librerie su un gel di agarosio 0,8%. (Vedi Figura 2A)
- Hi-C marcatura e Hi-C efficienza legatura è verificata da un test PCR digerire. Successo fill-in e legatura di un sito HindIII (AAGCTT) crea un sito per l'enzima di restrizione NheI (GCTAGC). Un prodotto particolare legatura formata da due frammenti di restrizione nelle vicinanze è amplificato con PCR (come in 3C 11 con 0,2 ml di ciascuna biblioteca come modello. I prodotti della PCR vengono successivamente digeriti con HindIII, NheI o entrambi. Dopo aver eseguito i campioni su un gel al 2% , il numero relativo di 3C e Hi-C eventi legatura può essere stimata quantificando l'intensità delle bande taglio e non tagliato (vedi Figura 2B).
- Alcuni frammenti non sarà stata legatura: per evitare di tirare giù dopo, rimuovere biotina da questi fini unligated con l'attività esonucleasica della DNA polimerasi T4.
- Biotina-14-dCTP a legatura non finisce DNA viene rimosso con l'attività esonucleasica della DNA polimerasi T4. Mescolare 5 mg di Hi-C biblioteca con 1 ml 10 mg / ml BSA, 10 microlitri NEBuffer 10x 2, 1 ml 10 mM dATP, 1 ml dGTP 10 mm e 5 unità di DNA polimerasi T4 in un volume totale di 100 l ed incubare la miscela a 12 ° C per 2 ore. Se possibile, le reazioni multiple di 5 mcg vengono eseguite.
- La reazione viene bloccata con l'aggiunta di 2 microlitri 0,5 M EDTA pH 8,0.
- Per purificare il DNA, a pH 8,0 fenolo: cloroformio (1:1) l'estrazione avviene seguita dalla precipitazione in etanolo.
- Il supernatante viene scartato e il pellet di DNA sono risospesi e raccolto in un volume totale di 100 microlitri di acqua.
II. Tranciatura e Selezione del formato
- Per rendere il DNA biotinilato adatto per high-throughput sequencing, il DNA deve essere tranciato a una dimensione di 300-500 paia di basi con uno strumento Covaris S2 (duty cycle 5, intensità 5, cicli / raffica 200, tempo 60 secondi per 4 cicli) .
- Per riparare le estremità del DNA tranciati, aggiungere 14 microlitri di buffer legatura 10x, 14 microlitri 2,5 mM dNTP mix, 5 microlitri di DNA polimerasi T4, 5 chinasi microlitri polinucleotide T4, 1 ml DNA polimerasi Klenow e 1 microlitri di acqua. Incubare per 30 minuti a temperatura ambiente.
- Dopo l'incubazione, utilizzare una colonna MinElute Qiagen per purificare il DNA in base alle raccomandazioni del fabbricante. Eluire il DNA due volte con 15 microlitri 1x Tris-Basso-EDTA (TLE: 10 mM Tris pH 8.0, 0.1 mM EDTA). Poi, allegare un dATP alle estremità 3 'del fine-riparazione del DNA aggiungendo 5 microlitri NEBuffer2 10x, 10 microlitri 1 mM dATP, 2 microlitri di acqua e 3 Klenow microlitri (eso-). Incubare la reazione per 30 minuti a 37 ° C.
- Per inattivare il frammento Klenow, incubare le reazioni per 20 minuti a 65 ° C e successivamente raffreddare le reazioni sul ghiaccio. Utilizzando un speedvac, ridurre i volumi di reazione a 20 l.
- Quindi, caricare il DNA in un 1,5% gel con 1X TAE e correre per 3,5 ore a 80-90V. Dopo la colorazione del gel con SYBR green, visualizzare il DNA su un DarkReader. Frammenti di DNA accise tra 300 e 500 paia di basi e li purifica con un kit di estrazione Qiagen gel con 2-4 colonne a seconda del peso del gel. Eluire il DNA con 50 microlitri TLE 1x.
- Combina il eluati dalle colonne QIAquick e portare il volume finale a 300 ml con 1x TLE. Infine, determinare la concentrazione di DNA con la Quant-iT test utilizzando il fluorimetro qubit e calcolare la quantità totale di DNA.
III. Biotina Pull-down e associati-end Sequencing
- In questa sezione del protocollo, giunzioni legatura sono purificati dalla piscina del DNA, consentendo l'identificazione efficiente di interagire frammenti di cromatina appaiati-end di sequenziamento. Eseguire tutti i passaggi successivi in tubi LoBind DNA.
- Preparare perle per la biotina pull-down con il lavaggio 150 microlitri risospeso sfere magnetiche streptavidina due volte con 400 microlitri Buffer Tween (TB: 5 mM Tris-HCl pH 8.0, 0.5 mM EDTA, 1 M NaCl, 0,05% Tween).
Lava questi e futuri consistono in cinque fasi:- Aggiungi tampone ai talloni
- Trasferire il composto in una nuova provetta
- Ruotare il campione per 3 minuti a temperatura ambiente
- Recuperare le perle con un concentratore di particelle magnetiche
- Rimuovere il surnatante
- Risospendere le sfere in 300 microlitri 2x Nessun buffer Tween (2x NTB: 10 mM Tris-HCl pH 8.0, 1 mM EDTA, 2 M NaCl) e si combinano con 300 ml Hi-C del DNA. Lasciare che la biotina etichettato Hi-C DNA di legarsi ai talloni streptavidina incubando la miscela a temperatura ambiente per 15 minuti con rotazione.
- Recuperare il DNA legato perline streptavidina con la Concentrazione di particelle magneticheator, e rimuovere il surnatante. Lavare le perline in 400 microlitri 1x NTB (5 mM Tris-HCl pH 8.0, 0.5 mM EDTA, 1 M NaCl), seguita da 100 microlitri di buffer legatura 1x. Risospendere le sfere in 50 microlitri di buffer legatura 1x e trasferire il composto in una nuova provetta.
- Per preparare il sequenziamento del DNA per Illumina Fine associati, prendere la quantità totale di DNA usato come input per la biotina pull-down, che è stato calcolato in precedenza al punto 2.6, e dividerlo per 20 a stimare la quantità di Hi-C del DNA che ha stato tirato giù ed è disponibile per la legatura. Aggiungere 6 picomoles di Illumina adattatori Fine associati per mg di Hi-C DNA di legatura. Usa 1200 unità T4 DNA ligasi per legare gli adattatori al DNA. Incubare per 2 ore a temperatura ambiente.
- Smontare il dispositivo anti-legatura adattatori End accoppiati bonificando l'Hi-C perle DNA legato e lavare le perle due volte con 400 TB microlitri 1x.
- Lavare le perline con 200 microlitri NTB 1x, seguiti da 200 ul e poi 50 microlitri 1x NEBuffer 2. Dopo l'ultimo lavaggio, risospendere le sfere in 50 microlitri 1x NEBuffer 2 e il trasferimento in una nuova provetta.
- Per determinare il numero di cicli necessari per generare prodotti a sufficienza per il sequenziamento della PCR, creato quattro test di reazioni PCR con cicli di 6, 9, 12 o 15. (Per i dettagli sulla PCR, si veda: 12 Determinare il numero ottimale ciclo eseguendo le reazioni PCR su un gel di poliacrilamide al 5% e colorazione con SYBR Green, assicurando l'assenza di bande spurie e la presenza di uno striscio tra i 400 -. 600 paia di basi, che è la lunghezza dei prodotti tranciate dopo la legatura agli adattatori.
- Amplifica il resto della Hi-C-library-bound perle streptavidina in un grande PCR con il numero ottimale di cicli di PCR. Piscina prodotti di PCR dai pozzetti separati e recuperare le sfere. Mantenere l'1% del prodotto su larga scala della PCR separato per l'esecuzione su un gel e purificare il resto del prodotto di PCR con 1,8 x perline Ampure il volume secondo le raccomandazioni del produttore.
- Eluire il DNA con 50 microlitri di buffer TLE 1x e confrontare l'1% del prodotto tallone Ampure purificato PCR per l'aliquota 1% del prodotto originale PCR su un gel di poliacrilamide al 5%, garantendo la rimozione di successo del primer PCR.
- Si consiglia inoltre di clonazione 1 microlitri della Hi-libreria C e determinare il prodotto di circa 100 cloni mediante sequenziamento Sanger. Questo vi permetterà di valutare il numero relativo di regolabile in Hi-C legge nella miscela PCR. Per i risultati tipici, vedi Figura 3B.
- Sequenza Hi-C biblioteca con Illumina sequenziamento fine appaiati. Allineare ciascuna estremità in modo indipendente utilizzando Maq (http://maq.sourceforge.net/) per identificare i frammenti di cromatina interagiscono.
IV. Rappresentante Hi-C Risultati
- I seguenti risultati sono attesi quando il protocollo di Hi-C è tecnicamente ben eseguito e può essere considerato standard di controllo di qualità.
- Fasi di controllo qualità dovrebbero rivelare che entrambe le librerie 3C e Hi-C a conduzione band, quanto piuttosto stretto superiore a 10 kb. Uno striscio DNA indica scarsa efficienza legatura. Tipicamente, l'efficienza legatura è leggermente inferiore a un impianto Hi-libreria C rispetto a un modello 3C (vedi Figura 2A).
- Hi-C, la marcatura e l'efficienza legatura può essere stimata dalla digestione di un prodotto di PCR generato usando primer 3C. Giunzioni 3C sono tagliati da HindIII e non da NheI. È vero il contrario per Hi-C giunzioni. Questo test PCR digerire mostra che il 70% di Hi-C ampliconi sono tagliati da NheI e non da HindIII, confermando la marcatura efficiente delle giunzioni legatura (vedi Figura 2B).
- Analisi della legge sequenza dovrebbe mostrare che legge da entrambe le interazioni intrachromosomal e interchromosomal, indicato dalle linee blu e rosso, allineare in modo significativo più vicino a siti di restrizione HindIII rispetto al generato a caso si legge, visualizzati in verde (vedi figura 3A).
- In un esperimento riuscito, il 55% delle coppie regolabile in leggere rappresentano interazioni interchromosomal. Quindici per cento rappresentano le interazioni tra intrachromosomal frammenti di meno di 20 kb a parte e il 30% sono coppie intrachromosomal letto che sono più di 20 kb a parte (vedi Figura 3B). Questa distribuzione può essere campionato prima di high-throughput sequencing, come una forma di controllo della qualità, la clonazione e sequenziamento Sanger di circa 100 cloni di solito è sufficiente.
- Le interazioni della cromatina può essere visualizzato come una heatmap, dove x e y assi rappresentano loci per genomica e ogni pixel rappresenta il numero di interazioni osservate tra di loro. Tipicamente, frammenti di DNA che sono molto vicini gli uni agli altri nel genoma lineare avrà la tendenza a interagire frequentemente con l'altro. Questo è visto nel heatmaps intrachromosomal come una diagonale di primo piano (vedi Figura 4A).
- I seguenti risultati mostrano diversi modi di analizzare i dati a rivelare i vari livelli di organizzazione del genoma. Tracciare la probabilit contattoy genomica rispetto alla distanza (vedere figura 5a) mostra che la probabilità di contatto diminuisce in funzione della distanza genomica, raggiungendo alla fine un plateau. Ad ogni distanza, interazioni intrachromosomal, mostrato in linea continua, sono arricchiti rispetto alle interazioni interchromosomal, rappresentato dalle linee tratteggiate. Questo implica direttamente la presenza dei territori cromosomici.
- Il calcolo del numero osservato / atteso di contatti interchromosomal tra tutte le coppie di cromosomi rivela associazione preferenziale tra coppie di cromosomi particolare. Cromosomi gene ricco di piccole preferenzialmente interagiscono tra loro, indicato dal colore rosso brillante (vedi Figura 5B).
- Cromosomi possono anche essere esaminato. Il heatmap crudo può essere regolata tramite un heatmap dovrebbe spiegare la distanza genomica tra coppie di loci, con un conseguente heatmap osservati / attesi. Poi, una matrice di correlazione può essere prodotto da correlare le righe e le colonne del heatmaps osservati / attesi. Usando l'analisi di correlazione, è dimostrato che il detentore distingua genoma umano in due compartimenti. Questo è illustrato dal plaid-pattern nel heatmaps di correlazione (Figura 4A-D). (Per i dettagli del Hi-C analisi dei dati, si veda: 1.
- Utilizzando Hi-C dati, sono state acquisite nuove conoscenze in ripiegamento cromatina alla scala megabase. Il modello classico di polimero di condensazione della cromatina pacchi suggerisce che in un globulo equilibrio. Probabilità di contatto tramando in funzione della distanza che illustra le scale contatto probabilità come una legge di potenza con la distanza genomica la cui pendenza è di circa -1 (Figura 6A). Questo non è coerente con il comportamento di un globulo equilibrio, ma non corrisponde alle aspettative di una struttura alternativa conosciuto come un globulo frattale (vedi Figura 6B).
- Qui, due strutture globulari sono mostrati. Colorazione corrisponde alla distanza da un punto finale, che vanno dal blu al ciano, verde, giallo, arancione e rosso (vedi figura in alto 6C). A differenza dei globuli equilibrio, globuli frattale mancanza coinvolgimenti. In un globulo frattale, loci che si trovano nelle vicinanze, lungo il contorno tendono ad essere vicina in 3D, che porta alla presenza di blocchi monocromatici (vedi Figura 6C centro). Tali blocchi non si trovano nel globulo equilibrio (vedi figura in basso 6C).
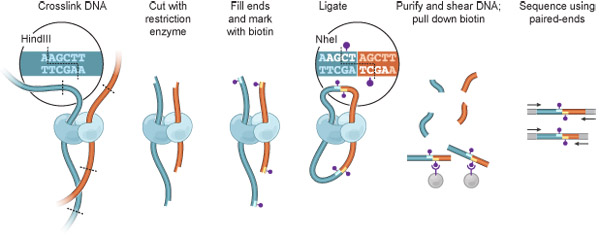
Figura 1. Hi-C panoramica. Le cellule sono reticolato con formaldeide, con conseguente legami covalenti tra i segmenti della cromatina spazialmente adiacenti (frammenti di DNA: blu scuro, rosso, proteine, in grado di mediare tali interazioni, sono mostrati in blu chiaro e ciano). Cromatina è digerito con un enzima di restrizione (qui, HindIII; sito di restrizione: linea tratteggiata, vedi riquadro). Le estremità risulta appiccicoso sono riempiti con nucleotidi, uno dei quali è biotinilato (dot viola). Legatura viene eseguita in condizioni estremamente diluite favorendo eventi legatura intramolecolare, il sito HindIII è persa e un sito NheI viene creato (nel riquadro). Il DNA è purificata e tosato, e incroci biotinilati sono isolati con perline streptavidina. Frammenti di interagire sono identificati da coppie-end di sequenziamento.

Figura 2. Hi-libreria C controlli di qualità. (A) quantità crescenti di un controllo 3C e Hi-libreria C sono stati risolti su un gel di agarosio 0,8%. Entrambe le biblioteche eseguito come una fascia piuttosto stretta superiore a 10 kb. Efficienza legatura tipica in un Hi-libreria C è leggermente inferiore rispetto a quanto osservato in un modello 3C, ed è indicato dal striscio nella Hi-C corsie. (B) PCR digerire controllo. Una giunzione legatura formata da due frammenti nelle vicinanze è amplificato utilizzando le normali condizioni di PCR 3C. Hi-C prodotti legatura possono essere distinti da quelli prodotti nel convenzionali 3C dalla digestione del sito legatura. Hi-C giunzioni sono tagliati da NheI non HindIII, il contrario è vero per giunzioni 3C. 70% di Hi-C ampliconi sono stati tagliati da NheI, confermando efficiente marcatura di giunzione legatura. Due repliche sono state eseguite per garantire la quantificazione affidabile.

Figura 3. Hi-C leggere controlli di qualità. (A) Legge da frammenti corrispondenti a due intrachromosomal (blu) e interchromosomal (rosso) interazioni allineare in modo significativo più vicino a siti di restrizione HindIII rispetto alla legge generati in modo casuale (verde). Sia il intrachromosomal legge e interchromosomal legge curve diminuiscono rapidamente aumenta la distanza dal sito del HindIII fino a un altopiano è raggiunta a una distanza di ~ 500 bp. Questo corrisponde alla dimensione del frammento massima utilizzata per il sequenziamento. (B) In genere, il 55% delle coppie regolabile in leggere rappresentano interazioni interchromosomal. Quindici per cento rappresentano le interazioni tra intrachromosomal frammenti di meno di 20 kba parte e il 30% sono coppie intrachromosomal letto che sono più di 20 kb di distanza. Questa distribuzione può essere campionato prima di high-throughput sequencing, come una forma di controllo della qualità, la clonazione e sequenziamento Sanger di circa 100 cloni di solito è sufficiente.

Figura 4. Analisi di correlazione dimostra che il nucleo è segregato in due compartimenti. (A) Heatmap corrispondenti alle interazioni intrachromosomal sul cromosoma 14. Ogni pixel rappresenta tutte le interazioni tra un 1-Mb e un altro locus 1-Mb locus; intensità corrisponde al numero totale di letture (range: 0-200 letture). Tacche appaiono ogni 10 Mb. Il heatmap mostre sottostruttura in forma di diagonale intenso e una costellazione di grandi blocchi. (Cromosoma 14 è acrocentrico;. Braccio corto non è indicato) Utilizzando la Hi-C set di dati per calcolare la probabilità media di contatto per un paio di loci a distanza genomica dato, una matrice aspettativa è prodotta (B) corrispondente a quello che sarebbe osservato se non ci fossero a lungo raggio strutture. Il quoziente di queste due matrici è una matrice osservati / attesi (C) in cui l'esaurimento è mostrato in blu e in rosso arricchimento [gamma: 0.2 (blu) a 5 (rosso)]. Il modello di blocco diventa più evidente. La matrice di correlazione (D) illustra la correlazione [range: -1 (blu) a 1 (rossa)] tra i profili di interazione intrachromosomal di ogni coppia di loci lungo del cromosoma 14. Il modello scozzese colpisce indica la presenza di due scomparti all'interno del cromosoma.

Figura 5. La presenza e l'organizzazione dei territori cromosomici. (A) Probabilità di contatto diminuisce in funzione della distanza genomico sul cromosoma 1, raggiungendo alla fine un altopiano a ~ 90Mb (blu). Il livello di contatto interchromosomal (linee nere) si distingue per diverse coppie di cromosomi; loci sul cromosoma 1 hanno più probabilità di interagire con i loci sul cromosoma 10 (trattini verdi) e meno in grado di interagire con i loci sul cromosoma 21 (linee rosse). Interazioni Interchromosomal sono esaurite relative alle interazioni intrachromosomal. (B) osservati / attesi numero di contatti interchromosomal tra tutte le coppie di cromosomi. Il rosso indica l'arricchimento e il blu indica l'esaurimento [range: 0,5 (blu) a 2 (rosso)]. Piccolo, ricco di cromosomi gene tendono ad interagire più uno con l'altro.

Figura 6. L'imballaggio locale della cromatina è coerente con il comportamento di un globulo frattale. (A) probabilità di contatto in funzione della distanza genomica in media in tutto il genoma (blu). Un importante ridimensionamento legge di potenza è visto tra i 500kb e 7Mb (regione ombreggiata) con una pendenza di -1,08 (in forma mostrato in ciano). (B) I risultati della simulazione per la probabilità di contatto in funzione della distanza di equilibrio (rosso) e frattale (blu) globuli. La pendenza di un globulo frattale è quasi -1 (ciano), confermando la nostra previsione romanzo teorica 1. La pendenza di un globulo equilibrio è -3 / 2, che corrisponde alle aspettative prima teorica. L'inclinazione per il globulo frattale ricorda da vicino la pendenza osservato nel Hi-C risultati, mentre la pendenza di un globulo equilibrio non è visto in Hi-C dati. (C) In alto: Una catena polimerica spiegato, 4000 monomeri lungo. Colorazione corrisponde alla distanza da un punto finale, che vanno dal blu al ciano, verde, giallo, arancione e rosso Oriente:. Tipico esempio di un globulo frattale che traiamo dalla nostra ensemble. Globuli frattale mancanza coinvolgimenti. Loci che si trovano nelle vicinanze, lungo il contorno tendono ad essere vicina in 3D, che porta alla presenza di grandi blocchi monocromatici che sono evidenti sulla superficie e in sezione trasversale inferiore:. Un globulo equilibrio. La struttura è molto aggrovigliati; loci che si trovano nelle vicinanze lungo il contorno (colore simile) non deve essere vicina in 3D.
Discussione
Vi presentiamo un metodo di studio del 3-dimensionale architettura del genoma con la mappatura delle interazioni della cromatina in un imparziali, genoma modo. La fase più critica sperimentale ciò che contraddistingue questa tecnologia a parte dal lavoro precedente - è l'incorporazione di nucleotidi biotinilato a limitare le estremità dei frammenti reticolato smussato-end prima di legatura. L'esecuzione di questo passo con successo permette il sequenziamento profondo di tutte le giunzioni legatura, e dà Hi-C la sua portata e la potenza.
Il numero di letture in ultima analisi, determina la risoluzione delle mappe di interazione. Qui, un 1 Mb per l'interazione mappa del genoma umano è presentato con ~ 30 milioni regolabile in legge. Al fine di aumentare la risoluzione 'per tutti gli usi' di un fattore di n, il numero di letture deve essere aumentato di un fattore di n 2.
L'Hi-C tecnica può essere facilmente combinata con altre tecniche, quali la cattura ibridi dopo generazione libreria (per indirizzare parti specifiche del genoma) e immunoprecipitazione della cromatina dopo la legatura (a esaminare l'ambiente cromatina delle regioni associate a specifiche proteine).
Divulgazioni
Riconoscimenti
Ringraziamo A. Kosmrlj per le discussioni e il codice; AP Aiden, XR Bao, M. Brennero, D. Cene di Gala, W. Gosper, A. Jaffer, A. Melnikov, A. Miele, G. Giannoukos, C. Nusbaum, AJM Walhout , L. Wood, e K. Zeldovich per le discussioni, e L. Gaffney e B. Wong aiuto per la visualizzazione.
Sostenuto da un Hertz Fannie e John Foundation laureato borsa di studio, una Difesa Nazionale della Scienza e Ingegneria laureato comunione, una comunione NSF laureato, il National Space Biomedical Research Institute, e non concede. T32 HG002295 dal National Human Genome Research Institute (NHGRI) (EL); i2b2 (Informatica per l'integrazione e la biologia Comodino), il NIH supportato Center for Biomedical Computing presso il Brigham and Women s Hospital (LAM), non concede. HG003143 dal NHGRI, e un Keck Foundation premio insigne studioso giovani (JD). Prime e mappati Hi-C dati di sequenza è stato depositato presso la banca dati GEO ( www.ncbi.nlm.nih.gov/geo/ ), l'adesione no. GSE18199. Visualizzazioni aggiuntive sono disponibili all'indirizzo http://hic.umassmed.edu .
Materiali
| Name | Company | Catalog Number | Comments |
| Protease inhibitors | Sigma-Aldrich | P8340-5ml | Step 1.2 |
| biotin-14-dCTP | Invitrogen | 19518-018 | Step 1.6 |
| Klenow | New England Biolabs | M0210 | Steps 1.6 and 2.2 |
| T4 DNA ligase | Invitrogen | 15224 | Step 1.9 |
| T4 DNA polymerase | New England Biolabs | M0203 | Steps 1.17 and 2.2 |
| 10x ligation buffer | New England Biolabs | B0202 | Steps 2.2 and 3.4 |
| T4 PNK | New England Biolabs | M0201 | Step 2.2 |
| Klenow (exo-) | New England Biolabs | M0212 | Step 2.3 |
| Dynabeads MyOne Streptavin C1 Beads | Invitrogen | 650.01 | Step 3.2 |
| T4 DNA ligase HC | Enzymatics | L603-HC-L | Step 3.5 |
| Phusion HF mastermix | New England Biolabs | F531 | Step 3.8 |
| Ampure beads | Beckman Coulter Inc. | A2915 | Step 3.9 |
Riferimenti
- Lieberman-Aiden, E., Van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I., Lajoie, B. R., Sabo, P. J., Dorschner, M. O., Sandstrom, R., Bernstein, B., Bender, M. A., Groudine, M., Gnirke, A., Stamatoyannopoulos, J., Mirny, L. A., Lander, E. S., Dekker, J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Kosak, S. T., Groudine, M. Form follows function: the genomic organization of cellular differentiation. Genes and Dev. 18, 1371-1384 (2004).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (2007).
- Dekker, J. Gene Regulation in the Third Dimension. Science. 319, 1793-1794 (2008).
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet. 2, 292-301 (2001).
- Sexton, T., Schober, H., Fraser, P., Gasser, S. M. Gene regulation through nuclear organization. Nat Struct and Mol Biol. 14, 1049-1055 (2007).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing Chromosome Conformation. Science. 295, 1306-1311 (2002).
- Zhao, Z., Tavoosidana, G., Sjölinder, M., Göndör, A., Mariano, P., Wang, S., Kanduri, C., Lezcano, M., Sandhu, K. S., Singh, U., Pant, V., Tiwari, V., Kurukuti, S., Ohlsson, R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347 (2006).
- Simonis, M., Klous, P., Splinter, E., Moshkin, Y., Willemsen, R., de Wit, E., van Steensel, B., de Laat, W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1348-1354 (2006).
- Dostie, J., Richmond, T. A., Arnaout, R. A., Selzer, R. R., Lee, W. L., Honan, T. A., Rubio, E. D., Krumm, A., Lamb, J., Nusbaum, C., Green, R. D., Dekker, J. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Res. 16, 1299-1309 (2006).
- Miele, A., Dekker, J. Mapping Cis- and Trans Chromatin Interaction Networks Using Chromosome Conformation Capture (3C). Methods Mol Biol. 464, 105-121 (2009).
- Maccallum, I., Przybylski, D., Gnerre, S., Burton, J., Shlyakhter, I., Gnirke, A., Malek, J., McKernan, K., Ranade, S., Shea, T. P., Williams, L., Young, S., Nusbaum, C., Jaffe, D. B. ALLPATHS 2: small genomes assembled accurately and with high continuity from short paired reads. Genome Biol . 10, R103-R103 (2009).
Erratum
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon