È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Quantificazione dei γH2AX Foci in risposta alle radiazioni ionizzanti
In questo articolo
Riepilogo
La quantificazione di DNA a doppio filamento striature formazione γH2AX utilizzando come marcatore molecolare è diventata uno strumento prezioso nel campo della biologia delle radiazioni. Qui mostriamo l'utilizzo di un test di immunofluorescenza per la quantificazione della γH2AX focolai dopo l'esposizione delle cellule alle radiazioni.
Abstract
DNA a doppio filamento (DSB), che sono indotti da processi metabolici sia endogena o da fonti esogene, sono una delle lesioni del DNA più critici in termini di sopravvivenza e la conservazione dell'integrità del genoma. Una risposta presto per l'induzione di DSB è la fosforilazione della variante dell'istone H2A, H2AX, alla serina-139 residui, altamente conservati nel C-terminale motivo SQEY, formando γH2AX
Protocollo
Preparazione delle cellule
- Cheratinociti umani (FEP-1811) sono state coltivate in cheratinociti-siero Media gratuito (K-SFM; Invitrogen) integrato con il fattore di crescita epidermico, estratto di ipofisi bovina e 20 mg / ml di gentamicina, a 37 ° C e 5% di CO 2.
- Una sospensione singola cella è stata preparata da staccare con tripsina-EDTA (0,05% v / v)
- Le cellule sono state seminate a 8-Tek ben Lab II diapositive microchamber (10.000 cellule / pozzetto) e le diapositive sono state incubate per 3 giorni a 37 ° C e 5% di CO 2.
Irradiazione
- Le cellule sono state irradiate su ghiaccio con 2 Gy utilizzando una sorgente di 137 Cs (Gammacell 1000 Elite irradiatore; Nordion internazionale, ON, Canada, 20,6 secondi / Gy)
- Controllo irradiato e 2 Gy cellule irradiate sono state incubate per 1 ora a 37 ° C e 5% di CO 2.
Immunofluorescenza
- Media è stata una soffiata e le cellule sono state lavate con 300μl di PBS (w / o Ca 2 + o Mg 2 +) per bene e sono stati ruotati su un mixer orbitale per 5 minuti.
- Il buffer è stata una soffiata e 100μl di preparati al momento 4% (v / v) è stato aggiunto paraformaldeide in ciascun pozzetto e diapositive sono state incubate a temperatura ambiente per 10 minuti.
Tutte le incubazioni sono state eseguite in una vasca colorazione umidificata - Le cellule sono state poi lavate con PBS (w / o Ca 2 + o Mg 2 +). I vetrini sono stati posti in una vaschetta Coplin e ruotata su un mixer orbitale per 5 minuti. Questa fase di lavaggio è stata ripetuta altre due volte.
- Il buffer è stata una soffiata e l'eccesso di PBS è stato gentilmente cancellato.
- Le cellule sono state permeabilizzate con 100ml Triton X-100 (0,1% v / v) per bene e una incubazione di 10 minuti a temperatura ambiente.
- Le cellule sono state lavate con PBS (w / o Ca 2 + o Mg 2 +), come descritto nei punti 3 e 4 di cui sopra.
- Non specifico legame con le proteine è stato bloccato con 100 ml di BSA (1% v / v) per bene e 20 minuti di incubazione a temperatura ambiente.
- L'eccesso di BSA è stata una soffiata e 100μl di mouse primario monoclonale anti-fosfo-istone H2AX anticorpi (diluito 1:500 in 1% BSA, Millipore), è stato aggiunto in ciascun pozzetto di una incubazione 1 ora a temperatura ambiente.
- Le cellule sono state lavate con PBS (w / o Ca 2 + o Mg 2 +), come descritto nei punti 3 e 4 di cui sopra e incubato con 100μl di anticorpo secondario (Alexa Fluor 488 di capra anti-IgG di topo diluito 1:500 in BSA 1% ; Invitrogen) per bene per 45 minuti a temperatura ambiente al buio. (Anticorpo diluito è stato tenuto all'oscuro durante tutta la procedura)
- Le cellule sono state lavate con PBS (w / o Ca 2 + o Mg 2 +), come descritto nei punti 3 e 4 di cui sopra. Tuttavia, l'esposizione alla luce è stato minimizzato usando pellicola.
- Di contrasto nucleare è stata eseguita con 100ml TOPRO3 (diluito 1:500; Invitrogen) per bene e una incubazione 10 minuti a temperatura ambiente
- Le cellule sono state lavate con PBS (w / o Ca 2 + o Mg 2 +) come descritto al punto 10.
- Le camere sono state accuratamente rimosse dalle guide di scorrimento, l'umidità in eccesso è stato cancellato e le diapositive sono stati lasciati asciugare all'aria.
- Una goccia di prolungare ORO anti-sbiadimento soluzione (Invitrogen) è stato aggiunto per bene e le diapositive sono state montate (22x50 millimetri coprioggetto) e il liquido in eccesso lungo i bordi di scorrimento è stata cancellata.
- I vetrini sono stati tenuti al buio per altri 30 minuti a temperatura ambiente prima di sigillare con smalto.
- I vetrini sono stati conservati durante la notte a 4 ° C al buio prima dell'analisi.
Microscopia / Analisi
- Meta confocale Zeiss LSM510 Microscpe utilizzato per acquisire immagini utilizzando la GFP standard (per γH2AX - Alexa Fluor 488 capra anti-topo IgG) e laser rosso lontano (per TOPRO-3). Tipicamente, un olio 63 x lente ad immersione obiettivo è utilizzato.
Le immagini sono acquisite in una serie Z tracciato con dimensione del passo di 0,5 micron. Un passo di 0,5 micron è stata scelta per minimizzare la perdita di focolai presenti nei diversi piani nei nuclei. Durante l'analisi, i piani individuali sono deconvoluted e sovrapposti per produrre una immagine proiettata al massimo per ridurre al minimo la sovrapposizione di focolai (Top-hat filtro applicato). - Metamorph (Molecular Devices, USA) è stato utilizzato per analizzare il numero di focolai.
Il programma quantitates il numero di focolai in ogni cella dopo la soglia è stata applicata per escludere sfondo. Le informazioni vengono registrate in un foglio di calcolo Microsoft Excel per ulteriori analisi.

Figura 1. Visualizzazione di immunofluorescenza γH2AX focolai (verde) non trattati in cheratinociti umani e in cellule irradiate con 2 Gy e incubate per circa 1 ora a 37 ° C, 5% di CO 2. Il DNA è stato macchiato con TOPRO-3 (blu). Immagini sono state acquisite utilizzando un Zeiss LSM 510 Meta microscopio confocale. Bar = 10 micron.
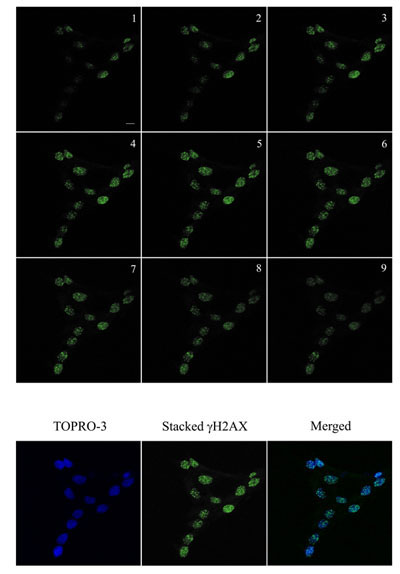
Figura 2. Visualizzazione di immunofluorescenza γH2AX focolai (verde) in cheratinociti umani e in cellule irradiate con 2 Gy e incubate per circa 1 ora a 37 ° C, 5% di CO 2. Immagini sono state acquisite utilizzando un Zeiss LSM 510 Meta microscopio confocale utilizzando 0,5 micron Z-sezionamento (1-9) per garantire che tutti focolai sono stati acquisiti. Le immagini è stata poi impilati per la quantificazione utilizzando Metamorph. Il DNA è stato macchiato con TOPRO-3 (blu). Il γH2AX impilati e le immagini blu erano accatastati per la visualizzazione. Bar = 10 micron.
Discussione
In seguito all'esposizione a radiazioni ionizzanti (raggi γ), γH2AX forma foci rapidamente e numeri focolai raggiungono un massimo tra 30-60 minuti 2. Pertanto, il nostro 1 ora dalla irradiazione punto di tempo riflette la formazione iniziale DSB. Abbiamo usato la dose di radiazioni clinicamente rilevante di 2 Gy per il nostro esperimento. Tuttavia, il metodo può essere utilizzato per dosi di radiazioni fino a 4 Gy per il rilevamento di formazione iniziale DSB; sovrapposizione significativa di focolai ...
Divulgazioni
Riconoscimenti
Il sostegno della Australian Institute of Nuclear Science and Engineering è riconosciuto. TCK è stato il destinatario di premi AINSE. Lab Medicina epigenomic è supportata dal National Health and Medical Research Council of Australia (566.559). LM è supportato da Melbourne di ricerca (Università di Melbourne) e borse di studio integrative Biomedical Imaging CRC. Il supporto di Monash Micro Imaging (Dr. Stephen Cody e Iska Carmichael) è stata preziosa per questo lavoro.
Materiali
| Name | Company | Catalog Number | Comments | |
| Keratinocyte-Serum Free Medium (K-SFM) | Media | Invitrogen | 17005042 | Keratinocyte media supplemented with human recombinant Epidermal Growth Factor 1-53 (EGF 1-53), and Bovine Pituitary Extract (BPE). |
| Lab Tek lI Chamber Slides (8-well) | Chamber Slides | Nalge Nunc international | NUN154534 | |
| Coverslips (22x50mm) | Coverslips | Menzel-Glaser | CS2250100 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A7906 | BSA (1%) is used to block any non-specific antibody binding. Primary and secondary antibodies are diluted in BSA. | |
| PBS (without Ca2+ and Mg2+) | Invitrogen | 17-517Q | ||
| 0.5% Trypsin-EDTA x10 | Invitrogen | 15400-054 | Trypsin-EDTA (0.05%) used to detach cells from culture flasks. | |
| Triton X-100 | Reagent | Sigma-Aldrich | T8787 | Triton X-100 (0.1%) used to permeabilise cells. |
| Paraformaldehyde | Reagent | Sigma-Aldrich | 158127 | Paraformaldehyde (4%) used to fix cells. |
| Mouse monoclonal anti-phospho histone-H2AX antibody | Primary Antibody | EMD Millipore | 16193 | Dilution of primary antibody (1:500), in 1% BSA. |
| Alexa Fluor 488 goat anti-mouse IgG (H+L) | Secondary Antibody | Invitrogen | 11029 | Dilution of secondary antibody (1:500), in 1% BSA. |
| TOPRO3 | DNA Stain | Invitrogen | T3605 | DNA stain commonly used: 4,6-diamidino-2-phenylindole dihydrochloride (DAPI). Can only be used with microscopes with the appropriate excitation laser. |
| ProLong Gold | Anti-fade solution | Invitrogen | P36930 | Refractive index of 1.42 at 20°C. |
| Tissue Culture Flask, Vented Cap | Culture Flask | BD Biosciences | 353112 | |
| Tissue Culture Dish (150x25mm) | Petridish | BD Biosciences | 353025 | |
| Coplin Jar, glass | Grale Scientific P/L | 1771-OG | ||
| Staining Trough | Grale Scientific P/L | V1991.99 | ||
| Gammacell 1000 Elite Irradiator | Gamma Irradiator | Nordion International Inc. | ||
| Zeiss LSM 510 Meta Confocal | Confocal Microscope | |||
| Metamorph | Software for Imaging analysis | Molecular Devices |
Riferimenti
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. J. Cell Biol. 146, 905-916 (1999).
- Bonner, W. M. Gamma H2AX and cancer. Nature Reviews Cancer. 8 (12), 957-967 (2008).
- Savic, V. Formation of Dynamic [gamma]-H2AX Domains along Broken DNA Strands Is Distinctly Regulated by ATM and MDC1 and Dependent upon H2AX Densities in Chromatin. Molecular Cell. 34 (3), 298-310 (2009).
- Downs, J. Binding of chromatin-modifying activities to phosphorylated histone H2A at DNA damage sites. Mol Cell. 16 (6), 979-990 (2004).
- Chowdhury, D. [gamma]-H2AX Dephosphorylation by Protein Phosphatase 2A Facilitates DNA Double-Strand Break Repair. Molecular Cell. 20 (5), 801-809 (2005).
- Nakada, S., Chen, G., Gingras, A., Durocher, D. PP4 is a gamma H2AX phosphatase required for recovery from the DNA damage checkpoint. EMBO Rep. 9 (10), 1019-1026 (2008).
- Altaf, M., Auger, A., Covic, M., Côté, J. Connection between histone H2A variants and chromatin remodeling complexes. Biochem Cell Biol. 87 (1), 35-50 (2009).
- Kusch, T. Acetylation by Tip60 Is Required for Selective Histone Variant Exchange at DNA Lesions. Science. 306, 2084-2087 (2004).
- Hurlin, P. Progression of human papillomavirus type 18-immortalized human keratinocytes to a malignant phenotype. Proc Natl Acad Sci U S A. 88 (2), 570-574 (1991).
- Dickey, J. H2AX: functional roles and potential applications. Chromosoma. , (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon