È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
DNA Stabile-isotopica Probing (DNA-SIP)
In questo articolo
Riepilogo
DNA-isotopo stabile sondaggio è un metodo di coltivazione-indipendente di identificare e caratterizzare le comunità di microrganismi attivi che sono in grado di utilizzare substrati specifici. L'assimilazione del substrato arricchito in isotopo pesante porta alla costituzione degli atomi indicati sull'etichetta in biomassa microbica. Ultracentrifugazione gradiente di densità recupera DNA etichettato per la valle analisi molecolari.
Abstract
DNA-isotopo stabile sondaggio (DNA-SIP) è una tecnica potente per identificare i microrganismi attivi che assimilare particolari substrati di carbonio e di sostanze nutritive in biomassa cellulare. Come tale, questa coltura-indipendente tecnica è stata una metodologia per l'assegnazione di importanti funzioni metaboliche per le diverse comunità che abitano una vasta gamma di ambienti terrestri e acquatici. Dopo l'incubazione di un campione ambientale con isotopi stabili-molecole marcate, estratto acido nucleico è sottoposto a ultracentrifugazione gradiente di densità e di frazionamento gradiente successive per separare gli acidi nucleici di quote differenziate. Purificazione del DNA da cloruro di cesio recupera etichettati e privi di etichetta del DNA per la successiva caratterizzazione molecolare (ad esempio le impronte digitali, microarray, biblioteche clone, metagenomica). Questo protocollo video JOVE fornisce visive passo-passo le spiegazioni del protocollo per il frazionamento gradiente ultracentrifugazione gradiente di densità, e il recupero del DNA marcato. Il protocollo include anche i dati del campione SIP ed evidenzia importanti suggerimenti e precauzioni che devono essere considerati per assicurare il successo del DNA-SIP analisi.
Protocollo
1. Preparazione dei reagenti
DNA-SIP richiede l'uso di reagenti che dovrebbe essere preparata in anticipo della procedura vera e propria. Le indicazioni per la preparazione di ogni reagente sono elencati in questa sezione e sono modificati da un precedente protocollo SIP 1.
- Cloruro di Cesio (CsCl), soluzione per la preparazione di gradienti SIP - Preparare una soluzione 7,163 M CsCl gradualmente sciogliendo 603,0 g di CsCl in acqua distillata e deionizzata (DDH 2 O) ad un volume finale di 500 mL. Fare attenzione a non superare i 500 ml! Il riscaldamento della soluzione leggermente mescolando aiuterà sciogliere tutti i CsCl. Aliquota della soluzione finale in aliquote sigillate. Nel nostro laboratorio, una pratica di storage comune è quello di preparare 100 mL aliquote in flaconi da 125 ml di siero, che sono poi crimpare sigillati con tappi di gomma butilica. Le aliquote sigillate possono essere conservati a tempo indeterminato a temperatura ambiente (20 ° C). Le guarnizioni aiutare a prevenire l'evaporazione e CsCl formazione "crosta". Determinare la densità della soluzione pesando triplice copia da 100 microlitri aliquote, oppure utilizzando un rifrattometro digitale (es. Reichert AR200) che è stato attentamente calibrato per le soluzioni di CsCl. Una volta calibrato con successo, il AR200 Reichert è coerente e fornisce letture accurate per diversi anni. A temperatura ambiente (20 ° C), la densità finale di questa soluzione varia tipicamente 1,88-1,89 g ml -1. La densità varia leggermente ogni volta che viene preparato un nuovo magazzino.
- Soluzione di cloruro di cesio per la preparazione di sfumature con etidio bromuro (EtBr) - Unire 250 g di CsCl con 250 ml di acqua sterile DDH 2 O. Aliquota questa soluzione in separata fiale di siero che sono stati crimpare sigillato con guarnizioni in gomma butilica, come descritto al punto 1.1.
- Buffer gradiente - Combinare 50 ml di 1 M Tris-HCl, 3,75 g di KCl e 1 ml di EDTA 0,5 M a 400 ml di acqua. Sciogliere il KCl, quindi aggiungere DDH 2 O a 500 ml. Filtro-sterilizzare e autoclave. La soluzione finale è 0,1 M Tris, 0,1 M KCl e 1 mM EDTA.
- Polietilene glicole (PEG) soluzione - Preparare la soluzione di PEG sciogliendo 150 g di polietilenglicole 6000 e 46,8 g di NaCl in acqua sterile DDH 2 O ad un volume totale di 500 mL (PEG 30%, 1,6 M NaCl). Autoclave.
Nota: questa soluzione si separa in due fasi con autoclave. Includono un ancoretta in bottiglia in autoclave in modo che la soluzione può essere adeguatamente miscelato quando questo si verifica. - TE Buffer - Preparare una soluzione di 10 mM Tris-HCl (pH 8,0) e 1 mM EDTA (pH 8,0) in sterili DDH 2 acqua O, utilizzando soluzioni stock in autoclave di 1 M Tris-HCl (pH 8,0) e 0,5 M EDTA ( pH 8,0). Filtrare e sterilizzare in autoclave.
- 70% di etanolo - Unire 350 ml di etanolo ad alta purezza con 150 ml di acqua sterile DDH 2 O.
2. Campione di incubazione e di estrazione del DNA
Per DNA-SIP incubazione, i campioni sono in genere incubati con pesante isotopo di carbonio (13 C) del substrato. Periodi di incubazione e le condizioni (ad esempio l'integrazione di nutrienti, umidità, luce) varia a seconda del tipo di campione che viene incubata e la natura del substrato. DNA-SIP esperimenti sono stati effettuati con successo utilizzando una varietà di composti del carbonio singolo 2,3, multi-composti del carbonio 4,5,6, e l'utilizzo di azoto o ossigeno etichettati 7,8 9. Tuttavia, uno svantaggio di utilizzare 15 N-o 18 O-etichettati composti è la separazione fisica ridotta di acido nucleico marcato, principalmente a causa della presenza di un minor numero di azoto e atomi di ossigeno nel DNA e relativi ad atomi di carbonio RNA.
Un controllo critico per il DNA-SIP esperimenti di incubazione è un identico stabilito con nativo (ad esempio 12 C) del substrato. Questo incubazione fornisce un confronto successivo per garantire che ogni etichettatura apparente di acido nucleico non era un artefatto del ultracentrifugazione o G + C differenze di contenuto densità nel DNA hanno contribuito alla separazione 10. E 'anche importante tenere materiale congelato del campione per il confronto di' luce 'e del DNA' pesanti ', e vale la pena anche un no-substrato di controllo per valutare i cambiamenti di fondo per tutta la popolazione di incubazione SIP.
- Incubare campioni ambientali in microcosmi contenenti substrato marcato (Figura 1). Nella nostra esperienza, abbiamo trovato che un minimo di incorporazione tra 500-500 mmol di carbonio 13 C per grammo di campione sarà adatto a campioni contenenti elevata biomassa come campioni di suolo 1. Per i campioni contenenti meno biomassa acquatica di terreni, di 100-100 mmol incorporata di carbonio 13 C per litro può produrre rilevabile pesante firma isotopica 1. La quantità di carbonio modifica, percentuale di carbonio incorporato nella biomassa e la necessità di supplementare per l'assimilazione dei nutrienti tutto dipenderà dalle caratteristiche dei campioni da analizzare e il bersaglio organisms di interesse. Un unico insieme di linee guida di incubazione del campione non sarà applicabile per tutti i campioni. Importante, la concentrazione del substrato utilizzato per l'incubazione SIP dovrebbero idealmente essere il più vicino possibile alla concentrazione normalmente riscontrate in situ; pregiudizi sperimentale può essere una conseguenza delle condizioni di coltura di arricchimento 10.
- Dopo l'incubazione del campione con substrato stabile-isotopo marcato, estrarre il DNA da microcosmi utilizzando un protocollo rigoroso di estrazione (per la PCR o la clonazione piccolo inserto) o una lisi enzimatica di fiducia per la clonazione di alto peso molecolare (ad esempio su larga inserire metagenoma). RNA co-estrazione generalmente non influenzano l'analisi, in modo da protocolli che producono RNA e DNA possono essere utilizzati. L'ultracentrifugazione del DNA estratto non sarà di taglio frammenti più piccoli di circa 50 coppie kb 1.
- Quantificare il DNA estratto prima installazione dei tubi CsCl ultracentrifugazione gradiente. Quantificare il DNA utilizzando uno spettrofotometro (ad esempio NanoDrop 2000) se il protocollo di estrazione produce solo DNA (per esempio kit a base di colonna). In alternativa, quantificare tramite elettroforesi su gel di agarosio.
3. Preparazione Soluzioni per ultracentrifugazione gradiente
Questa procedura comporta l'aggiunta di DNA per ultracentrifuga tubi. Ci sono più di un tipo di tubo e il rotore in modo che il protocollo esatto varierà e dipenderà da istruzioni del produttore. Detto questo, si consiglia l'uso di un bene rotore verticale per garantire la massima possibilità di una separazione del DNA leggeri e pesanti. Usiamo un Beckman-Coulter Vti 65,2 rotore con 16 pozzi per lo svolgimento di 5,1 ml tubi Polyallomer QUICKSEAL e il protocollo fornirà i passi e le considerazioni per queste condizioni.
- Utilizzando le concentrazioni di DNA determinato al punto 2.3, calcolare il volume richiesto di DNA estratto, che è tenuta a fornire 0,5 mg - 5 mg di DNA nei tubi ultracentrifuga.
- Combina DNA estratto (0,5 - 5 mg) con tampone di gradiente (vedi punto 1.3) e 4,8 ml di 7,163 M CsCl ad un volume totale di circa 6 ml in una sterile monouso da 15 ml tubo. Si noti che la densità della soluzione CsCl può variare anche a molarità stesso (vedi punto 1.1). La seguente equazione può essere utilizzata per determinare il volume di gradiente Buffer / DNA miscela che è necessario per generare un adeguato rapporto di miscelazione:
Gradiente di soluzione tampone e il DNA del volume (ml) = (densità magazzino CsCl soluzione - voluta densità finale) x volume di soluzione madre CsCl aggiunto x 1,52
Specificare il volume della soluzione madre CsCl a 4,80 ml. La densità desiderata finale dovrebbe essere 1,725 g ml -1. La densità della soluzione madre è stata determinata al punto 1.1.
Si noti inoltre che i volumi relativi CsCl e sfumatura Buffer / DNA si tradurrà in un volume complessivo superiore a 5,1 ml. Preparare volumi superiori alla capacità volume massimo dei tubi ultracentrifuga (superiore a 5,1 ml), farà in modo che ci sia una soluzione sufficiente per riempire completamente il tubo. - Miscelare capovolgendo 10 volte. Il DNA è stabile a temperatura ambiente in CsCl.
4. Creazione di un gradiente di controllo EtBr (opzionale)
Perché EtBr è un colorante che intercalando complessi con il DNA che lo rende visibile ai raggi UV, i gradienti di controllo contenenti EtBr sono utili perché forniscono immediata conferma visiva della formazione di pendenze prima di frazionamento di tubi campione (es. Figura 1). L'inclusione di una provetta di controllo contenente EtBr e una miscela di entrambi i 12 C-DNA e 13 C-DNA (o 14 N-DNA e 15 N-DNA) permette la visualizzazione immediata di formazione di gruppo all'interno dei tubi al termine di ultracentrifugazione. Questo è importante perché un tubo rotto durante ultracentrifugazione o condizioni di funzionare in modo improprio programmato può causare formazione di pendenze fallito. Legato al DNA, EtBr abbassa la densità del DNA e, di conseguenza, un protocollo diverso è seguito per la preparazione sfumature. Da notare che altre macchie di acido nucleico può essere usato al posto di EtBr 11 ma il protocollo richiede l'ottimizzazione con fluorofori altri.
- Il gradiente di controllo richiede due volumi di DNA genomico: una completamente marcato con isotopi stabili-e una senza etichetta. Noi di solito utilizzare meliloti Sinorhizobium colta in un mezzo contenente 13 C o 12 C-glucosio come unica fonte di carbonio, o Methylococcus Bagno ceppo capsulatus coltivate in presenza di 13 C o 12 C-metano, come i nostri controlli.
- Combina un 5 -10 mg quantità sia dei 12 C-DNA e 13 C-DNA con tampone sfumatura per un volume finale di 1,00 ml in un usa e getta da 15 ml con tappo a vite del tubo.
- Aggiungere 1,00 g di CsCl solido al tubo stesso. Mescolare per inversione.
- Aggiungere 110 microlitridi una soluzione da 10 mg ml -1 EtBr e 4,3 ml di una g 1 magazzino ml -1 soluzione CsCl allo stesso tappo a vite del tubo utilizzato nel passaggio 4.2. La densità finale della soluzione che approssimativa della soluzione originale magazzino CsCl.
- Un ulteriore soluzione "vuoto" di controllo contenenti EtBr, saranno necessari anche per controbilanciare la soluzione creata al punto 4.4. Combina 1,00 mL di tampone di gradiente, 1,00 g di CsCl, 110 microlitri di una soluzione di 10 mg ml -1 EtBr e 4,3 ml di una soluzione 1 g ml di brodo -1 CsCl in una finestra separata da 15 ml con tappo a vite del tubo e mescolare per inversione.
5. Ultracentrifugazione
- Usando una lampadina e pipetta Pasteur, compilare accuratamente tubi ultracentrifuga con soluzioni gradiente preparato nel passo 3.2 (o 4.4 nel caso passi la preparazione di un gradiente di controllo EtBr). Aggiungere con cautela le soluzioni per i tubi con una pipetta Pasteur. Etichettare le provette sulla spalla tubo con un pennarello sottile permanente. ATTENZIONE: Assicurarsi che i tubi si riempiono esattamente alla base del collo tubo. Tubi non sufficientemente riempito possono scoppiare durante ultracentrifugazione.
- Quando tutti i tubi necessari sono pieni di soluzioni del campione, registrare la massa esatta di ciascun tubo. Tubi di coppia e l'equilibrio li entro 0-10 mg. Per il bilanciamento, trovare le coppie quasi abbinati e aggiungere o rimuovere piccole quantità di soluzione fino a quando non sono in equilibrio, mantenendo il livello di soluzione il più vicino possibile alla base del collo del tubo il più possibile. Si noti che per la pesatura di tubi, usiamo un rovesciata da 15 ml con tappo a vite del tubo che è stato tagliato a metà come supporto tubo per l'equilibrio.
- Tenuta dei tubi con un 'tubo topper' secondo le istruzioni del produttore.
- Controllare che i tubi sono sigillati correttamente invertendo loro e applicando una pressione moderata. Pesare i tubi di nuovo per verificare che siano ancora in bilico dopo la sigillatura entro 0-10 mg.
- Controllare ogni rotore e con attenzione per assicurare che i pozzi siano puliti e privi di detriti o polvere che potrebbero forare i tubi durante ultracentrifugazione.
- Inserire i tubi nel rotore con le coppie bilanciate uno di fronte all'altro. Registrare la posizione del rotore di ciascun campione, perché il processo di ultracentrifugazione può causare etichette marcatore di essere danneggiati o cancellati. Attentamente tenuta dei pozzi rotore come indicato dal costruttore.
- Caricare il rotore in ultracentrifuga. Chiudere la porta ultracentrifuga e applicare il vuoto. Se si utilizza un Vti 65,2 rotore, impostare la velocità di rotazione di 44.100 giri al minuto (~ 177.000 xg av), la temperatura a 20 ° C il tempo, e ultracentrifugazione per 36-40 ore. Seleziona il vuoto, la massima accelerazione, e spegnere il freno (assicura gradiente non interrotta da decelerazione). Da notare che disattivando il freno comporterà un'aggiunta di 1-2 ore per la fase di esecuzione. Si noti inoltre che i tempi di esecuzione più breve non può raggiungere una risoluzione sufficiente banda. Corre lungo ultracentrifugazione è raccomandata, e conducono ad una maggiore risoluzione di distinti gruppi acidi nucleici.
- Immediatamente dopo il completamento della procedura di ultracentrifugazione, rimuovere il rotore con attenzione. Evitando qualsiasi inclinazione o urti del rotore, rimuovere delicatamente i tubi dal rotore per non disturbare i gradienti all'interno dei tubi. In rare circostanze, un tubo si romperà durante la corsa. Se è così, c'è una possibilità che i gradienti nei tubi altro non forma in modo corretto. Se un gradiente di controllo è stato incluso, controllare questo tubo con attenzione ai raggi UV per confermare la formazione di pendenze. Se il gradiente non si è formato correttamente nel tubo di controllo, si consiglia di ripetere tutta la fase 5. Si noti che la provetta di controllo EtBr e il suo controllo in bianco possono essere conservati al buio e riutilizzati fino a sei mesi. Aver cura di pulire accuratamente il rotore in base alle istruzioni del produttore, una volta scoppiato il tubo è stato rimosso. Non utilizzare spazzole metalliche o detergenti abrasivi per pulire i pozzi del rotore in modo da evitare graffi ai pozzetti rotore! Rotore specifici pennelli e soluzione di pulizia può essere acquistato da Beckman.
6. Gradiente di Frazionamento
Ci sono due metodi che sono attualmente utilizzati per recuperare DNA dai tubi ultracentrifuga: frazionamento ed estrazione ago. Questo protocollo solo descrivere il processo di estrazione del DNA con la tecnica del frazionamento. Questo perché per la maggior parte degli esperimenti SIP, il DNA marcato non può essere visualizzato con EtBr e deve invece essere rilevata mettendo a confronto la luce equivalenti e frazioni pesanti dalle provette multiple. Una pompa a siringa è altamente raccomandato per recuperare pari frazioni gradiente di densità da tubi ultracentrifuga. Noi usiamo una pompa di infusione modello BSP (Braintree Scientific Inc.). Un basso flusso pompa peristaltica o una pompa HPLC può essere utilizzata.
- Riempire un sterile 60 ml di siringa sterile con DDH 2 O contenente sufficiente colorante blu bromofenolo per fornire un colore blu scuro. Posizionare la siringa sul braccio di caricamento della pompa a siringa.Collegare il tubo della pompa dotata di 23-gauge 1 "ago e accendere la pompa fino a qualche DDH 2 O è arrivato fino alla fine dell'ago. Noti che eventuali bolle d'aria in questa fornitura DDH 2 O pregiudichi il processo di frazionamento.
- Fissare uno dei tubi ultracentrifugazione per uno stand morsetto. Assicurarsi che la pinza è sufficientemente stretto per evitare che il tubo di essere spostato, ma non in modo tale che la pressione sul tubo potrebbe causare un rilascio di soluzione CsCl quando il tubo è trafitto. Perforare il fondo del tubo lungo la giuntura del tubo con un nuovo 23 gauge 1 "ago. Per i migliori risultati, forare il tubo in modo controllato, veloce e sicuro. Questo è molto difficile da fare bene, pratica diverse volte prima questo è il primo tentativo con tubi campione.
- Per ogni campione, preparare 12 sterili provette da 1,5 ml microcentrifuga con etichette che indicano il numero del campione e la frazione (1-12; pesante alla luce). Utilizzando l'ago al tubo della pompa (punto 6.1), forare la parte superiore del tubo sulla spalla tubo superiore, lungo la cucitura. Raccogliere la soluzione gradiente utilizzando i tubi microcentrifuga. Per quanto eseguito per la parte inferiore del tubo, forare il tubo in modo rapido e controllato. Pratica in anticipo ed essere molto attenti a usare un movimento controllato per evitare che tirando l'ago forzata di passare attraverso il tubo e in un dito! Utilizzano una frequenza di pompa precedentemente tarato che produrrà 12 x 425 microlitri frazioni in 12 minuti (425 microlitri min -1).
- Utilizzare un rifrattometro digitale (es. Reichert AR200, consigliato) o una bilancia analitica per verificare la densità delle frazioni da un gradiente di confermare la formazione adeguata pendenza. Avrete bisogno di usare ~ 50 l di campione per questo test. Includiamo spesso DNA puro cultura in un tubo (come descritto per la preparazione dei gradienti di controllo EtBr) per servire come controllo per il frazionamento e di utilizzare per la determinazione della densità. Si aspettano le densità di spaziare da ~ 1,690-1,760 g ml -1, con una densità media di circa 1,725 g ml -1.
7. Precipitazione del DNA
- DNA precipitato di tutte le frazioni in primo luogo l'aggiunta di 20 mg di poliacrilammide lineare come vettore per le precipitazioni. Mescolare per inversione. Aggiungere 2 volumi di soluzione di PEG (vedi punto 1) e mescolare per inversione. Si noti che un vettore di precipitazione (es. glicogeno o poliacrilamide lineare) è fondamentale per il recupero quantitativo di DNA da parte delle fazioni gradiente, ma deve essere usata cautela quando il glicogeno è utilizzato come vettore per le precipitazioni di questo protocollo. Preparati glicogeno hanno dimostrato di essere contaminati da batteri acidi nucleici e la contaminazione può facilmente confondere l'interpretazione delle frazioni gradiente SIP 12.
- Lasciare le provette a temperatura ambiente per 2 ore per permettere al DNA di precipitare. Se lo si desidera, i tubi possono essere lasciati durante la notte a temperatura ambiente.
- Centrifugare a 13.000 g per 30 minuti con il dorso dei tubi verso l'esterno per un orientamento coerente nel tubo rotore. Con attenzione aspirare e scartare il surnatante. Il pellet deve essere visibile, ma può essere molto difficile da vedere in questa fase. Lavorare sotto una fonte di luce intensa (ad esempio scrivania lampada) per aiutare a visualizzare il pellet.
- Lavare il pellet con 500 ml di etanolo al 70%. Centrifugare a 13.000 g per 10 minuti. Con attenzione aspirare e scartare il surnatante. Il pellet di solito è più visibile di questo passo, ma si dissociano dalla parete del tubo più facilmente.
- Lasciare asciugare il pellet a temperatura ambiente per 15 minuti.
- Sospendere ogni pellet in 50 ml di tampone TE (vedi punto 1.5). Esegui 5 ml di ogni frazione di un gel di agarosio secondo protocolli standard di laboratorio.
8. Frazione Caratterizzazione
Il metodo utilizzato per caratterizzare le frazioni del gradiente per valutare il successo di un SIP incubazione può variare a seconda del laboratorio e la disponibilità di attrezzature. Utilizzando un metodo di rilevamento delle impronte digitali per il targeting del gene 16S rRNA è un approccio comune e metodi come il polimorfismo di restrizione lunghezza del terminale frammento (T-RFLP) o denaturazione elettroforesi su gel di pendenza (DGGE) sono appropriate (Figura 1). Seguendo il protocollo sopra descritto, si aspettano il DNA luce per essere associate con le frazioni 9-11 (~ 1,705-1,720 g ml -1) e le impronte digitali del DNA pesante per essere associate in frazioni 5-8 (~ 1,720-1,735 g ml -1 ). Impronte digitali univoco associato con le frazioni 5-8 del isotopi stabili-campioni incubati, ma non con nativo-substrato controlli incubati fornisce forti evidenze che collegano gli organismi specifici con il metabolismo di particolare substrato etichettati. Se insufficiente DNA marcato rimane per alcune applicazioni (ibridazione, metagenoma), l'amplificazione spostamento più possono essere utilizzati per produrre maggiori quantità di 13-15, ma questo può introdurre chimere nel DNA amplificato 14,16.
9. Risultati
Tipico DNA-SIP risultati dimostrano una separazione del DNA etichettati e privi di etichetta nel gradiente formato da ultracentrifugazione. Idealmente, la risoluzione completa di alto peso molecolare materiale genetico (ad esempio 13 C, 15 N) con materiali privi di etichetta sarà raggiunto. Risoluzione può essere testimoniata visivamente osservando la formazione di band in provette di controllo EtBr. Le concentrazioni di DNA genomico recuperate contenute nelle frazioni gradiente può anche essere usato per confermare la formazione adeguata pendenza.
Per questo protocollo ci sono risultati rappresentativi di ultracentrifugazione gradiente eseguite utilizzando acido nucleico da due colture pure (Figura 2). Il gradiente frazionato incluso qui è stato preparato utilizzando DNA genomico estratto da S. meliloti (ATCC 1021), e 13 C-marcato M. str capsulatus. Vasca da bagno. A seguito di ultracentrifugazione, il frazionamento e il recupero del DNA, etichettati e senza etichetta DNA genomico separati in frazioni gradiente rispettive quote differenziate (Figura 2A). Heavy-isotopo DNA marcate si osservano nelle frazioni 4-5, mentre il DNA senza etichetta si trova in alte concentrazioni in frazioni 9-10. Il DNA di ogni frazione è stata caratterizzata con elettroforesi su gradiente di gel denaturante 17 e la PCR-amplificato prodotti generati discreti modelli di bande corrispondenti alle due organismi inclusi nel gradiente (Figura 2B). La densità delle frazioni variava da ~ 1,580-1,759 g ml -1, e sono mostrati in ordine decrescente di densità da sinistra a destra.
Anche se la separazione tra puro e 13 C-12 C-DNA può essere pronunciata (Figura 2), incubazioni campione ambientale può essere più difficile da interpretare. Per esempio, abbiamo incubato tundra terreni da Resolute Bay (Nunavut, Canada) sia con C-marcato 12 C o 13 di glucosio per un periodo di 14 giorni a 15 ° C. Il gel di agarosio del DNA purificato gradiente frazione dimostrato che il DNA genomico è stato 'spalmato' tra frazioni 70-10 per entrambi i 12 C e 13 C-incubazione (Figura 3A e 3C, rispettivamente). In questo caso, 13 C-arricchimento di biomassa microbica da taxa particolare può essere determinata solo con un approccio quali DGGE dei geni rRNA 16S. Il 12 C-glucosio DNA suolo incubato genera modelli simili in tutte le frazioni del gradiente (Figura 3B), ma il 13 C-glucosio campione incubato generato DGGE impronte digitali che sono frutto dell'associazione unica con le frazioni 5-8 (Fig. 3D). Di particolare interesse sono le bande conservati indicato dalle frecce. Questo dominante 'phylotype' è coerente in tutte le frazioni del gradiente, ma si sposta verso le frazioni più pesanti del DNA ottenuto da 13 C-glucosio suolo incubate. Successivo sequenziamento del DNA di questa band e / o clonare analisi biblioteca confermerebbe l'identità di questo particolare gene 16S rRNA e guida metagenomica successivi o coltivazione approcci.
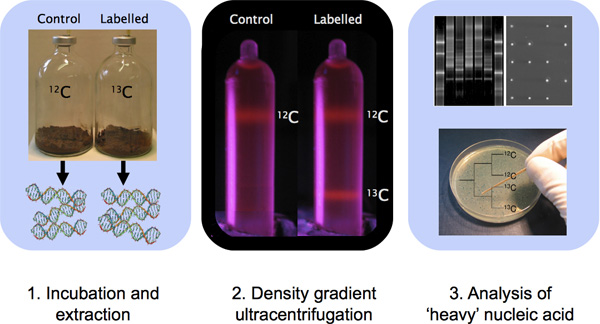
Figura 1. Schema di un DNA-SIP esperimento che coinvolge l'incubazione del campione, l'estrazione del DNA, CsCl ultracentrifugazione gradiente di densità e la caratterizzazione del DNA con tecniche di biologia molecolare.
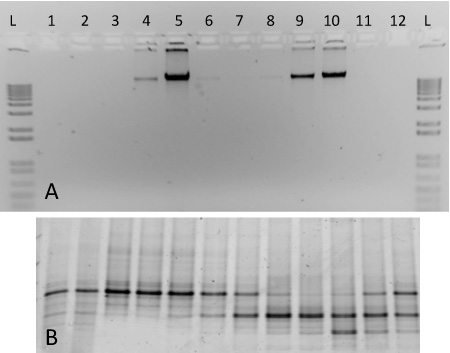
Figura 2. Risultati attesi per un frazionamento gradiente SIP compreso il DNA di due colture pure. (A) Aliquote di DNA da frazioni gradiente 1-12 sono state eseguite su un 1% gel da un gradiente contenente 13 C-marcato M. capsulatus Bagno ceppo (frazioni 4-6) e 12 C-marcato S. meliloti (frazioni 8-10). A 1-kb scaletta si effettua il confronto (B) PCR-DNA amplificato dalle frazioni stesse sono state eseguite su un gel DGGE 10%. Modelli di impronte digitali rivelano notevoli differenze tra 5 e 9 frazioni, per esempio.
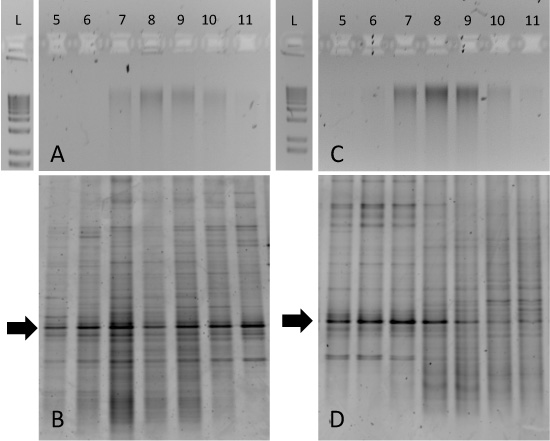
Figura 3. Risultati attesi per il frazionamento gradiente SIP da incubazioni campione di suolo. Aliquote delle frazioni gradiente sia da C-glucosio 12 suolo modificato (A) e 13 C-glucosio suolo modificato (C) sono state eseguite su gel di agarosio 1% e un 1-kb scaletta è incluso per il confronto. Impronte digitali DGGE corrispondente per ognuno di questi esempi sono mostrati in (B) e (D). Fingerprinting delle frazioni rivela arricchimento di particolari taxa batterici nel C-glucosio 13 campione modificato in frazioni 5-8 (D).
Discussione
Corretta progettazione di stabili-isotopo esperimenti sondaggio è di importanza critica per l'ottenimento di DNA marcato al di sopra della comunità sfondo senza etichetta. Considerazioni relative a tempi di incubazione del campione, le concentrazioni di substrato, condizioni di incubazione (per esempio nutrienti, umidità del suolo), alimentazione incrociata e la replicazione sono stati discussi altrove 10,18 e si raccomanda al lettore di consultare le pubblicazioni quando si progetta un incubazione SIP...
Divulgazioni
Riconoscimenti
Questo lavoro è stato sostenuto dal Progetto Strategico e borse di Discovery per JDN dalle scienze naturali e ingegneria Research Council del Canada (NSERC).
Materiali
| Name | Company | Catalog Number | Comments | |
| Bromophenol Blue | Reagent | Fisher Scientific | BP115-25 | |
| Cesium chloride | Reagent | Fisher Scientific | BP210-500 | |
| Ethanol, reagent grade | Reagent | Sigma-Aldrich | 652261 | |
| Ethidium bromide | Reagent | Sigma-Aldrich | E1510 | |
| Hydrochloric acid | Reagent | Fisher Scientific | 351285212 | |
| Linear polyacrylamide | Reagent | Applichem | A6587 | |
| Polyethylene Glycol 6000 | Reagent | VWR international | CAPX1286L-4 | |
| Potassium Chloride | Reagent | Fisher Scientific | AC42409-0010 | |
| Sodium Chloride | Reagent | Fisher Scientific | S2711 | |
| Sodium Hydroxide pellets | Reagent | Fisher Scientific | S3181 | |
| Tris base | Reagent | Fisher Scientific | BP1521 | |
| Dark Reader | Equipment | Clare Chemical | DR46B | |
| Microcentrifuge | Equipment | Eppendorf | 5424 000.410 | |
| Nanodrop 2000 | Equipment | Fisher Scientific | 361013650 | |
| Infusion pump | Equipment | Braintree Scientific, Inc. | N/A | Model Number: BSP See www.braintreesci.com for ordering details. |
| Tube sealer | Equipment | Beckman Coulter Inc. | 358312 | |
| Ultracentrifuge | Equipment | Beckman Coulter Inc. | ||
| Ultracentrifuge rotor | Equipment | Beckman Coulter Inc. | 362754 | |
| Ultraviolet light source | Equipment | UVP Inc. | 95-0017-09 | Any UV source will suffice |
| Ultraviolet light face shield | Equipment | Fisher Scientific | 114051C | |
| Butyl rubber stoppers, gray | Material | Sigma-Aldrich | 27232 | |
| Centrifuge tubes | Material | Beckman Coulter Inc. | 342412 | |
| Hypodermic needle, 23 gauge, 2” length | Material | BD Biosciences | 305145 | |
| Microfuge tubes, 1.5 mL | Material | DiaMed | AD151-N500 | |
| Open center seals, 20 mm diameter | Material | Sigma-Aldrich | 27230-U | |
| Pasteur pipettes, glass | Material | Fisher Scientific | 13-678-6C | |
| Pipet tips | Material | DiaMed | BPS340-1000 | Catalogue number is for 200 μl tips. 10 or 20 μl tips may be purchased from the same source |
| Pump tubing 1.5 mm bore x 1.5 mm wall | Material | Appleton Woods | ||
| Screw-cap tubes, 15 mL | Material | DiaMed | AD15MLP-S | |
| Serum vials, 125 mL volume | Material | Sigma-Aldrich | Z114014 | |
| Syringe, 60 mL | Material | BD Biosciences | 309653 |
Riferimenti
- Neufeld, J. D. DNA stable-isotope probing. Nat. Protocols. 2, 860-866 (2007).
- Neufeld, J. D., Boden, R., Moussard, H., Schäfer, H., Murrell, J. C. Substrate-specific clades of active marine methylotrophs associated with a phytoplankton bloom in a temperate coastal environment. Appl. Environ. Microbiol. 74, 7321-7328 (2009).
- Nercessian, O., Noyes, E., Kalyuzhnaya, M. G., Lidstrom, M. E., Chistoserdova, L. Bacterial populations active in metabolism of C1 compounds in the sediment of Lake Washington, a freshwater lake. Appl. Environ. Microbiol. 71, 6885-6899 (2005).
- Padmanabhan, P. Respiration of 13C-labelled substrates added to soil in the field and subsequent 16S rRNA gene analysis of 13C-labelled soil DNA. Appl. Environ. Microbiol. 69, 1614-1622 (2003).
- Bernard, L. Dynamics and identification of soil microbial populations actively assimilating carbon from 13C-labelled wheat residue as estimated by DNA- and RNA-SIP techniques. Environ. Microbiol. 9, 752-764 (2007).
- Haichar, e. l. Z. a. h. a. r., F, . Identification of cellulolytic bacteria in soil by stable isotope probing. Environ. Microbiol. 9, 625-634 (2007).
- Addison, S., McDonald, I., Lloyd-Jones, G. Stable isotope probing: Technical considerations when resolving 15N-labelled RNA in gradients. J. Microbiol. Meth. 80, 70-75 (2009).
- Buckley, D. H., Huangyutitham, V., Hsu, S. -. F., Nelson, T. A. Stable isotope probing with 15N achieved by disentangling the effects of genome G + C content and isotope enrichment on DNA density. Appl. Environ. Microbiol. 73, 3189-3195 (2007).
- Schwartz, E. Characterization of growing microorganisms in soil by stable isotope probing with H218O. Appl. Environ. Microbiol. 73, 2541-2546 (2007).
- Neufeld, J. D., Dumont, M. G., Vohra, J., Murrell, J. C. Methodological considerations for the use of stable isotope probing in microbial ecology. Microb. Ecol. 53, 435-442 (2007).
- Martineau, C., Whyte, L., Greer, C. Development of a SYBR safe technique for the sensitive detection of DNA in cesium chloride density gradients for stable isotope probing assays. J. Microbiol. Meth. 73, 199-202 (2008).
- Bartram, A. K., Poon, C., Neufeld, J. D. Nucleic acid contamination of glycogen used in nucleic acid precipitation and assessment of linear polyacrylamide as an alternative co-precipitant. Biotechniques. 47, 1019-1022 (2009).
- Chen, Y. Revealing the uncultivated majority: combining DNA stable-isotope probing, multiple displacement amplification and metagenomic analyses of uncultivated Methylocystis in acidic peatlands. Environ. Microbiol. 10, 2609-2622 (2008).
- Neufeld, J. D., Chen, Y., Dumont, M. G., Murrell, J. C. Marine methylotrophs revealed by stable-isotope probing, multiple displacement amplification and metagenomics. Environ. Microbiol. 10, 1526-1535 (2008).
- Kalyuzhnaya, M. High-resolution metagenomics targets specific functional types in complex microbial communities. Nat. Biotechnol. 26, 1029-1034 (2008).
- Binga, E. K., Lasken, R. S., Neufeld, J. D. Something from (almost) nothing: the impact of multiple displacement amplification on microbial ecology. ISME J. 2, 233-241 (2008).
- Green, S. J., Leigh, M. B., Neufeld, J. D., Timmis, K. N. . Microbiology of Hydrocarbon and Lipid Microbiology. , 4137-4158 (2010).
- Neufeld, J. D., Wagner, M., Murrell, J. C. Who eats what, where and when? Isotope-labelling experiments are coming of age. ISME J. 1, 103-110 (2007).
- Gallagher, E., McGuinness, L., Phelps, C., Young, L. Y., Kerkhof, L. J. DNA shortens the incubation time needed to detect benzoate-utilizing denitrifying bacteria by stable-isotope probing. Appl. Environ. Microbiol. 71, 5192-5196 .
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon