Method Article
PAR-Clip - un metodo per identificare trascrittoma livello i siti di legame delle proteine RNA binding
In questo articolo
Riepilogo
Trascrizioni di RNA sono soggetti ad ampie regolamentazione posttranscriptional che è mediato da una moltitudine di trans-acting RNA-binding proteins (RBPs). Qui vi presentiamo un metodo generalizzabile per identificare con precisione e trascrittoma su scala i siti di legame di RNA RBPs.
Abstract
Trascrizioni di RNA sono sottoposti a regolazione post-trascrizionale genica interagendo con centinaia di RNA-binding proteins (RBPs) e microRNA contenenti complessi ribonucleoproteina (miRNPs) che sono spesso espressi in una cellula di tipo dipendente. Per capire come l'interazione di questi RNA-binding fattori influisce sulla regolazione della trascrizione individuale, mappe ad alta risoluzione in vivo le interazioni proteina-RNA sono necessari 1.
Una combinazione di fattori genetici, biochimici e approcci computazionali sono generalmente applicati per individuare l'RNA-RBP o RNA-RNP interazioni. Profiling microarray di RNA associate a RBPs immunopurified (RIP-Chip) 2 definisce gli obiettivi a livello del trascrittoma, ma la sua applicazione è limitata alla caratterizzazione delle interazioni cineticamente stabile e solo in rari casi 3,4 permette di identificare l'elemento RBP riconoscimento (RRE ) all'interno del RNA bersaglio lungo. Più diretto obiettivo informazioni RBP sito è ottenuto dalla combinazione di reticolazione UV 5,6 vivo con immunoprecipitazione 7-9 seguito da l'isolamento reticolato di segmenti di RNA e sequenziamento del DNA (CLIP) 10. CLIP è stato utilizzato per identificare gli obiettivi di un certo numero di RBPs 11-17. Tuttavia, CLIP è limitata dalla scarsa efficienza dei raggi UV 254 nm RNA-proteina reticolazione, e la posizione della reticolazione non è facilmente individuabile all'interno dei frammenti sequenziati reticolato, il che rende difficile separare reticolati UV bersaglio segmenti di RNA da sfondo non reticolati anche frammenti di RNA presenti nel campione.
Abbiamo sviluppato una potente cellula approccio basato reticolazione a determinare in alta risoluzione e trascrittoma livello i siti di legame di RBPs cellulare e miRNPs che chiamiamo PAR-Clip (Photoactivatable-ribonucleosidi-Enhanced reticolazione e immunoprecipitazione) (vedi fig. 1A per uno schema del metodo). Il metodo si basa sulla costituzione di analoghi ribonucleosidi fotoreattivo, come thiouridine 4-(4-SU) e 6-thioguanosine (6-SG) nella nascente trascrizioni di RNA da cellule viventi. L'irradiazione delle cellule dalla luce UV di 365 nm induce la reticolazione efficiente delle fotoreattivo nucleosidi marcati RNA cellulare RBPs interagenti. Immunoprecipitazione della RBP di interesse è seguita da isolamento del RNA reticolato e coimmunoprecipitated. L'RNA isolato è trasformata in una libreria di cDNA e profondo in sequenza usando la tecnologia Solexa. Una caratteristica di librerie cDNA preparati da PAR-Clip è che la posizione precisa di reticolazione può essere identificata da mutazioni che risiedono nella sequenza del DNA. Quando si utilizza 4-SU, sequenze reticolato timidina di transizione citidina, mentre l'uso di 6-SG risultati in guanosina di adenosina mutazioni. La presenza di mutazioni in sequenze reticolato rende possibile la loro separazione dallo sfondo di sequenze derivate da abbondanti RNA cellulare.
Applicazione del metodo ad una serie di diverse proteine RNA binding è stata riportata in Hafner et al 18.
Protocollo
Il protocollo seguente descrive il PAR-Clip procedura per cellule HEK293 che esprimono FLAG / HA-tag IGF2BP1 su induzione con doxiciclina. Useremo un anti-FLAG anticorpo per immunoprecipitazione.
PAR-Clip funziona con qualsiasi linea di cellule che esprimono livelli rilevabili di endogeno, senza tag legame con le proteine RNA (RBP) di interessi se un anticorpo efficace per immunoprecipitazione è disponibile.
Le cellule in espansione
- Espandere FlpIn-HEK293/TO/FLAG/HA-IGF2BP1 cellule in terreno di crescita. Si consiglia di utilizzare tra 100-400 x 10 6 cellule (circa 15 centimetri 10-40 piastre di coltura cellulare) come punto di partenza. Crescere a circa l'80% confluenza.
- 14 h prima di aggiungere un reticolazione) 4-thiouridine ad una concentrazione finale di 100 mM (1:1000 v / v di un 1 M 4-thiouridine soluzione madre) direttamente al mezzo di coltura cellulare e b) inducono l'espressione del FLAG / HA tagged IGF2BP1 mediante aggiunta di 1 mg / ml di doxiciclina (1:10.000 v / v di 10 mg / ml soluzione madre doxiciclina). NOTA: al posto di 4-thiouridine è anche possibile utilizzare il 100 mM di 6-thioguanosine.
UV-reticolazione
- Lavare le cellule una volta con 10 ml ghiacciata PBS per piastra e rimuovere completamente PBS.
- Piastre posto su un vassoio con ghiaccio e irradiare scoperto con 0,15 J / cm 2 di 365 nm luce UV in un Stratalinker 2400 (Stratagene) o un dispositivo simile.
- Raschiare le cellule con un poliziotto in gomma in 1 ml di PBS per piastra, trasferimento in tubi da 50 ml centrifugazione e raccogliere per centrifugazione a 500 xg per 5 minuti a 4 ° C e scartare il surnatante. 100 x 10 6 cellule HEK293 (10 15 lastre cm) produrrà circa. 1 ml di pellet umido.
- (Opzionale) A meno che non si desidera continuare direttamente con la lisi cellulare, shock congelare il pellet di cellule in azoto liquido e conservare a -80 ° C. Pellet cellulare può essere conservato per almeno 12 mesi.
Lisi cellulare e RNaseT1 digerire
- Raccogliere pellet cellulare delle cellule reticolato in 3 volumi di tampone di lisi 1x NP40 e incubare in ghiaccio per 10 minuti.
- Lisato di cellule chiare per centrifugazione a 13.000 xg per 15 minuti a 4 ° C.
- Cancella il lisato ulteriormente filtrandola attraverso una siringa da 0,2 micron filtro a membrana (Acrodisc Pall o equivalente).
- Aggiungi RNasi T1 (Fermentas, 10.000 U / mL) a una concentrazione finale di 1 U / mL e incubare a bagnomaria per 15 minuti a 22 ° C. Reazione fredda successivamente per 5 minuti in ghiaccio prima di procedere.
Immunoprecipitazione ed il recupero di frammenti di RNA bersaglio reticolato
Utilizzando il separatore magnetico
Seguire queste linee guida per tutta la preparazione del campione per evitare che il sfere magnetiche si secchi.
- Collocare la provetta contenente le perline sul supporto magnetico per 1 2 minuti.
- Aggiungere la soluzione tampone al tubo mentre il tubo si trova sul separatore magnetico.
- Chiudere la provetta, rimuoverlo dal separatore magnetico, e risospendere le sfere. Si può risospendere le sfere muovendo il tubo con le dita o utilizzare un vortex fissato a 5 6.
- Centrifugare brevemente per raccogliere le sfere che possono rimanere nel tappo del tubo.
- Ripetere i passaggi da 1 a 4 come richiesto.
Preparazione di perline magnetiche
- Trasferire 10 ml di particelle di proteina G Dynabeads magnetico (Invitrogen) per cella ml di lisato (per un tipico esperimento che dovrebbe essere di ca. 40 50 ml di perline) in una provetta da microcentrifuga 1,5 ml. Lavare perle due volte con 1 ml di tampone citrato-fosfato.
- Risospendere in doppio del volume di tampone citrato-fosfato rispetto al volume originario di sospensione tallone.
- Aggiungi 0,25 mg di anti-FLAG anticorpi monoclonali M2 (Sigma) per ogni ml di sospensione e incubare su una ruota girevole per 40 minuti a temperatura ambiente.
- Lavare perle due volte in 1 ml di tampone citrato-fosfato per rimuovere l'anticorpo non legato.
- Risospendere le sfere in doppio del volume di tampone citrato-fosfato rispetto al volume originario di sospensione tallone.
Immunoprecipitazione (IP), secondo la digestione RNasi T1, e defosforilazione
- Aggiungere 20 ml di preparati al momento sfere magnetiche anticorpi coniugati per ml di lisato cellulare parziale RNasi T1 trattati e incubare in provette da 15 ml centrifugazione su una ruota girevole per 1 ora a 4 ° C.
- Raccogliere sfere magnetiche su un collettore di particelle magnetiche per 15 e 50 tubi di centrifugazione ml (Invitrogen) e trasferimento in tubi da 1,5 ml microcentrifuga.
- Lavare perle 3 volte in 1 ml di tampone di lavaggio IP.
- Aggiungi RNaseT1 (Fermentas, 10.000 U / mL) a una concentrazione finale di 100 U / mL e incubare la sospensione tallone a bagnomaria per 15 minuti a 22 ° C. Raffreddare successivamente sul ghiaccioper 5 min.
- Lavare perle 3 volte in 1 ml di alto sale tampone di lavaggio.
- Perline risospendere in 1 volume di tampone defosforilazione
- Aggiungi vitello fosfatasi alcalina intestinale (CIAP, NEB) ad una concentrazione finale di 0,5 U / mL, e incubare la sospensione per 10 minuti a 37 ° C.
- Lavare perle due volte in 1 ml di tampone di lavaggio fosfatasi
- Lavare perle due volte in polinucleotide chinasi (PNK) buffer senza digitale terrestre (DTT la concentrazione necessaria per la reazione enzimatica è abbastanza alta da danneggiare le sfere magnetiche).
- Perline risospendere in un unico volume originale cordolo di PNK tampone
Radiomarcatura reticolato di segmenti di RNA a proteine immunoprecipitato
- Per la sospensione tallone descritto sopra, aggiungere Υ-32 P-ATP ad una concentrazione finale di 0,5 μCi / mL e T4 PNK (ONA) a 1 U / mL in un unico volume originale tallone. Incubare la sospensione per 30 minuti a 37 ° C.
- Aggiungi non radioattivo ATP per ottenere una concentrazione finale di 100 mM e incubare per altri 5 minuti a 37 ° C.
- Lavare le sfere magnetiche 5 volte con 800 ml di PNK buffer senza DTT.
- Risospendere le sfere in 70 ml di SDS-PAGE tampone di caricamento.
SDS-PAGE e electroelution reticolato di RNA-proteina complessi da fette di gel
- Incubare la sospensione radiomarcato per 5 minuti in un blocco di calore a 95 ° C per denaturare e rilasciare il RBP immunoprecipitati con RNA reticolato e vortice.
- Rimuovere le sfere magnetiche sul separatore e trasferire il supernatante in una provetta da microcentrifuga pulita 1,5 ml.
- Carico massimo di 40 ml di supernatante per pozzetto di una Novex Bis-Tris 4-12% (Invitrogen) gel di poliacrilammide prefabbricati ed eseguire il gel per 55 min a 200 V.
- Smontare la camera di gel e smantellare il gel, lasciandolo montato su una piastra. Per facilitare l'allineamento del gel alla lista su carta phosphorimager, si consiglia di impiantare tre pezzi minuscoli gel radioattivi asimmetrico a tre dei quattro angoli del gel. Pezzi gel radioattivi possono essere raccolti dal gel che in precedenza erano utilizzate per purificare radiomarcati oligonucleotidi sintetici. Avvolgere il gel in film plastico (ad esempio pellicola trasparente) per evitare contaminazioni.
- Esporre il gel per uno schermo oscurato phosphorimager per 1 ora e visualizzare su un phosphorimager.
- Allineare il gel sulla parte superiore della stampa phosphorimager utilizzando i pezzi di gel impiantati per l'orientamento. Tagliare le bande che corrispondono alle dimensioni previste di RBP (IGF2BP1, ca. 75 kDa) e trasferirla in un D-Tube Tubo dializzatore Midi e aggiungere 800 microlitri 1x tampone SDS esecuzione.
- Electroelute il reticolato RNA-RBP complesso in 1x SDS gestione del buffer a 100 V per 2 ore escisse dal gel e electroeluted in un D-Tube dializzatore Midi (Novagen) in 800 microlitri di buffer SDS esecuzione secondo le istruzioni del produttore.
Proteinasi K digestione
- Aggiungere un volume equivalente di tampone 2x proteinasi K rispetto alla electroeluate, seguito con l'aggiunta di proteinasi K (Roche) ad una concentrazione finale di 1,2 mg / ml. Incubare per 30 minuti a 55 ° C.
- Recuperare l'RNA da acido fenolo / cloroformio / IAA di estrazione (25:24:1, pH 4,0) seguita da un'estrazione con cloroformio. Aggiungere 1 ml di glicogeno (10 mg / ml di brodo) precipitare l'RNA con l'aggiunta di 3 volumi di etanolo. Sciogliere il precipitato in acqua 10,5 microlitri.
biblioteca preparazione cDNA e sequenziamento profondo
Portare l'RNA recuperato attraverso un protocollo standard cDNA preparazione biblioteca originariamente descritta per la clonazione dei piccoli RNA normativo 19. Il primo passo, la legatura adattatore 3 ', è stata effettuata come descritto in una scala 20 l con 10,5 ml di RNA recuperato. Utilizzare il Solexa sequenziamento adattatore set descritto. A seconda della quantità di RNA recuperato, 5'-adattatore-3'-adattatore prodotti senza inserti possono essere rilevati dopo l'amplificazione del cDNA come ulteriori bande PCR. In tali casi, il prodotto più accise PCR delle dimensioni previste dal NuSieve 3% a basso punto di fusione su gel di agarosio, eluire il prodotto di PCR dai pezzi gel con il kit GelElute (Qiagen) e sequenza utilizzando la tecnologia Solexa. Un Solexa sequenziamento eseguire offre di solito tra i 6 ei 10 milioni di sequenza si legge che sono sufficienti per una copertura trascrittoma gamma di siti di legame delle proteine RNA vincolanti.
Analisi bioinformatica
Un'attenta analisi bioinformatica della sequenza di legge deve essere fatto per ottenere la intuizioni significative nei siti RNA vincolante per la RBP esame, come l'elemento di riconoscimento di RNA, le regioni preferito vincolante la RBP ha (exonic vs intronico, codifica sequenza vs non tradotte sequenza). La sequenza si legge devono essere allineati contro le banche dati del genoma e EST. Noi di solito utilizzare legge MAPPIng esclusivamente al genoma con fino a un disadattamento, inserimento o l'eliminazione di costruire gruppi di sequenza si legge che possono poi essere ulteriormente analizzato. La frequenza di mutazioni caratteristiche del cluster sequenziato legge, T alle transizioni C quando si usa 4-SU e G ad A transizioni quando si utilizzano 6-SG, sono indicativi di sequenze con successo reticolato. Nella nostra esperienza RNA uncrosslinked etichettati con 4-SU mostrano un tasso di mutazione sfondo di circa il 20%. Questo tasso è aumenta di ca. 50-80% su reticolazione.
Una descrizione dettagliata delle analisi bioinformatica può essere trovato nel materiale supplementare della pubblicazione da parte Hafner et al 18.
Passi opzionali
Determinazione dei livelli di incorporazione di 4-thiouridine in RNA totale
Isolare RNA totale dalla linea cellulare che esprimono stabilmente la RBP di interesse dopo essere cresciuto in media integrato con 100 mM 4SU 16 ore prima del raccolto. Come controllo, le cellule raccolta coltivati senza aggiunta 4SU. Isolare RNA totale con l'aggiunta di 3 volumi di reagente Trizol (Sigma) al pellet cellulari lavato seguendo le istruzioni del produttore s. è stata ulteriormente purificare l'RNA totale usando Qiagen RNeasy secondo il protocollo del produttore. Per prevenire l'ossidazione del 4SU durante l'isolamento di RNA e analisi, aggiungere 0,1 mM ditiotreitolo (DTT) a tamponi di lavaggio e successive reazioni enzimatiche. Digerire e defosforilata RNA totale di nucleosidi singolo per l'analisi HPLC come descritto in precedenza 20. In breve, in un volume di 30 microlitri, incubare a 40 mcg di RNA totale sono stati purificati per 16 ore a 37 ° C con 0,4 U fosfatasi alcalina batterica (Biochemical Worthington) e 0,09 veleno di serpente U fosfodiesterasi (Worthington Biochemical). Come standard di riferimento, utilizzare una sintesi di RNA 4SU marcato, (siamo in standard uso CGUACGCGGAAUACUUCGA (4SU) U) e anche soggetti per completare la digestione enzimatica. Separare le miscele risultante di ribonucleosides mediante HPLC su Discovery Supelco C18 (particelle di silice legata fase mM 5, 250 x 4,6 mm) colonna a fase inversa (Bellefonte PA, USA). Buffer HPLC sono 0,1 M TEAA nel 3% acetonitrile (A) e il 90% acetonitrile in acqua (B). Utilizzare un gradiente isocratica: 0% B per 15 minuti, da 0 a 10 B% per 20 minuti, dal 10 al 100% B per 30 min. Applicare un min 5 100% B lavare applicata tra le esecuzioni per pulire la colonna HPLC.
Rappresentante Risultati
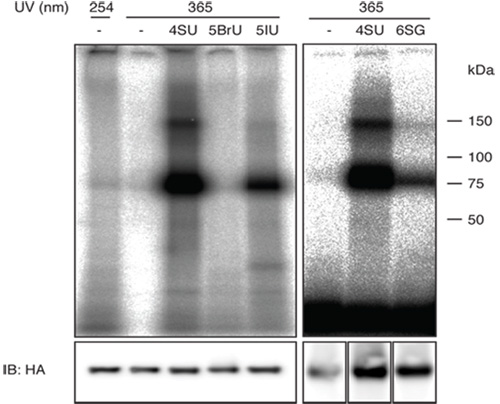
Figura 1 (pannello di destra) mostra un risultato rappresentativo di un PAR-Clip eseguita con linee cellulari che esprimono FLAG / HA-tag IGF2BP1 con 4-SU e 6-SG. Si noti che l'efficienza reticolazione di 6-SG per IGF2BP1 è inferiore al rendimento reticolazione per 4-SU. L'efficienza reticolazione inferiore si tradurrà in un contesto più elevato di sequenze derivate da abbondanti frammenti di RNA cellulare e quindi si dovrebbe considerare di scala l'esperimento quando si utilizzano nucleosidi fotoreattivo meno efficienti.
Il pannello di sinistra della figura 1 mostra un confronto di utilizzare diversi analoghi uridina fotoreattivo che potrebbero essere potenzialmente utilizzate per PAR-Clip rispetto ai tradizionali reticolazione UV 254 nm.
L'intensità della banda radioattiva della lunghezza corretta che dà una buona idea se il PAR-Clip esperimento ha funzionato e che è stato isolato l'RNA sufficiente per portare a termine una piccola sequenza di RNA protocollo (step-by-step descrizione per la preparazione di piccole librerie di cDNA sequenziamento RNA può essere trovato in 19). La frequenza di mutazioni caratteristiche della legge in sequenza, T alle transizioni C quando si usa 4-SU e G ad A transizioni quando si utilizzano 6-SG, sono indicativi di sequenze con successo reticolato. Nella nostra esperienza RNA uncrosslinked etichettati con 4-SU mostrano un tasso di mutazione sfondo di circa il 20%. Questo tasso è aumenta di ca. 50-80% su reticolazione.
Divulgazioni
Riconoscimenti
Ringraziamo i membri del laboratorio Tuschl per le discussioni utili. MH è supportato dal Deutscher Akademischer Austauschdienst (DAAD). Questo lavoro è stato sostenuto dal Fondo Nazionale Svizzero di Grant # 3100A0-114001 a MZ; TT è un investigatore HHMI, e il lavoro nel suo laboratorio è stato sostenuto da NIH GM073047 e MH08442 e la Fondazione Starr.
Materiali
Tamponi e reagenti
Crescita media cellule HEK293
- DMEM
- 10% FBS
- 2 mM L-Glutammina
- 100 U / ml di penicillina
- 100 U / ml streptomicina
- 100 mg / ml Hygromycin
- 15 mg / ml blasticidin
4-thiouridine soluzione madre (1 M)
- 260,27 mg 4-thiouridine
- 1 ml di DMSO
Doxycyclin magazzino (10 mg / ml)
- 10 mg doxycyclin
- 1 ml di DMSO
1x NP40 buffer di lisi
Preparare uno stock di tampone 5x senza DTT e inibitori della proteasi. Aggiungi DTT e inibitore della proteasi direttamente prima dell'esperimento.
- HEPES 50 mM, pH 7,5
- 150 mM KCl
- 2 mM EDTA
- 1 mM NaF
- 0,5% (v / v) NP40
- 0,5 mM DTT
- completa EDTA senza inibitore della proteasi cocktail (Roche)
Tampone citrato-fosfato
- 4,7 g / l di acido citrico
- 9,2 g / l Na 2 HPO 4
- pH 5,0
IP-tampone di lavaggio
- 50 mM HEPES-KOH, pH 7,5
- 300 mM KCl
- 0,05% (v / v) NP40
- 0,5 mM DTT (aggiungere direttamente prima dell'esperimento)
- completa EDTA senza inibitore della proteasi cocktail (Roche) (aggiungere direttamente prima dell'esperimento)
High-sale tampone di lavaggio
- 50 mM HEPES-KOH, pH 7,5
- 500 mM KCl
- 0,05% (v / v) NP40
- 0,5 mM DTT (aggiungere direttamente prima dell'esperimento)
- completa EDTA senza inibitore della proteasi cocktail (Roche) (aggiungere direttamente prima dell'esperimento)
Defosforilazione tampone
- 50 mM Tris-HCl, pH 7,9
- 100 mM NaCl
- 10 mM MgCl 2
- 1 mM DTT
Fosfatasi tampone di lavaggio
- 50 mM Tris-HCl, pH 7,5
- 20 mM EGTA
- 0,5% (v / v) NP40
Polinucleotide chinasi (PNK) buffer senza DTT
- 50 mM Tris-HCl, pH 7,5
- 50 mM NaCl
- 10 mM MgCl 2
PNK tampone
- 50 mM Tris-HCl, pH 7,5
- 50 mM NaCl
- 10 mM MgCl 2
- 5 mM DTT
SDS PAGE tampone di caricamento
- 10% glicerolo (v / v)
- 50 mM Tris-HCl, pH 6.8
- 2 mM EDTA
- 2% SDS (w / v)
- 100 mM DTT
- 0,1% bromofenolo blu
2x proteinasi K tampone
- 100 mM Tris-HCl, pH 7,5
- 150 mM NaCl
- 12,5 mm EDTA
- 2% (w / v) di SDS
Riferimenti
- Keene, J. D. RNA regulons: coordination of post-transcriptional events. Nat. Rev. Genet. 8 (7), 533-533 (2007).
- Tenenbaum, S. A. Identifying mRNA subsets in messenger ribonucleoprotein complexes by using cDNA arrays. Proc. Nat. Acad. Sci. 97 (26), 14085-14085 (2000).
- Gerber, A. P. Genome-wide identification of mRNAs associated with the translational regulator PUMILIO in Drosophila melanogaster. Proc. Nat. Acad. Sci. 103 (12), 4487-4487 (2006).
- Lopez de Silanes, I. Identification of a target RNA motif for RNA-binding protein HuR. Proc. Nat. Acad. Sci. 101 (9), 2987-2987 (2004).
- Greenberg, J. R. Ultraviolet light-induced crosslinking of mRNA to proteins. Nucl. Acids Res. 6 (2), 715-715 (1979).
- Wagenmakers, A. J. Cross-linking of mRNA to proteins by irradiation of intact cells with ultraviolet light. Eur. J. Biochem. 112 (2), 323-323 (1980).
- Mayrand, S. Structure of nuclear ribonucleoprotein: identification of proteins in contact with poly(A)+ heterogeneous nuclear RNA in living HeLa cells. The Journal of Cell Biology. 90 (2), 380-380 (1981).
- Dreyfuss, G. Characterization of heterogeneous nuclear RNA-protein complexes in vivo with monoclonal antibodies. Mol. Cell. Biol. 4 (6), 1104-11 (1984).
- Adam, S. A., Dreyfuss, G. Adenovirus proteins associated with mRNA and hnRNA in infected HeLa cells. J. Virol. 61 (10), 3276-3276 (1987).
- Ule, J. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1212 (2003).
- Licatalosi, D. D. HITS-CLIP yields genome-wide insights into brain alternative RNA processing. Nature. 456 (7221), 464-464 (2008).
- Yeo, G. W. An RNA code for the FOX2 splicing regulator revealed by mapping RNA-protein interactions in stem cells. Nat. Struct. Mol. Biol. 16 (2), 130-130 (2009).
- Sanford, J. R. Splicing factor SFRS1 recognizes a functionally diverse landscape of RNA transcripts. Genome Res. 19 (3), 381-381 (2009).
- Granneman, S. Identification of protein binding sites on U3 snoRNA and pre-rRNA by UV cross-linking and high-throughput analysis of cDNAs. Proc. Nat. Acad. Sci. , (2009).
- Guil, S., Caceres, J. F. The multifunctional RNA-binding protein hnRNP A1 is required for processing of miR-18a. Nat. Struct. Mol. Biol. 14 (7), 591-591 (2007).
- Chi, S. W. Argonaute HITS-CLIP decodes microRNA-mRNA interaction maps. Nature. 460 (7254), 479-479 (2009).
- Zisoulis, D. G. Comprehensive discovery of endogenous Argonaute binding sites in Caenorhabditis elegans. Nat. Struct. Mol. Biol. , (2010).
- Hafner, M. Transcriptome-wide identification of RNA-binding protein and microRNA target sites by PAR-CLIP. Cell. , (2010).
- Hafner, M. Identification of microRNAs and other small regulatory RNAs using cDNA library sequencing. Methods. 44 (1), 3-3 (2008).
- Andrus, A., Kuimelis, R. G. Base composition analysis of nucleosides using HPLC. Current Protocols in Nucleic Acid Chemistry. Chapter 10 (Unit 10.6), (2001).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon