È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
La produzione di C. elegans Transgeni attraverso recombineering con il GalK Marcatori selezionabili
In questo articolo
Riepilogo
La capacità di produrre transgeni per Caenorhabditis elegans Usando il DNA genomico portato da fosmids è particolarmente interessante come tutti gli elementi nativi normativo vengono conservati. Descritto è un procedimento semplice e robusto per la produzione di transgeni attraverso recombineering con il GalK Marcatore.
Abstract
La creazione di animali transgenici è largamente utilizzato in C. elegans ricerca compreso l'uso di proteine di fusione GFP per studiare la regolamentazione e pattern di espressione di geni di interesse o di generazione di purificazione in tandem affinità (TAP) tagged versioni di geni specifici per facilitare la loro purificazione. Tipicamente transgeni sono generati mettendo un promotore a monte di un gene reporter GFP o cDNA di interesse, e questo produce spesso un modello rappresentativo di espressione. Tuttavia, gli elementi critici della regolazione genica, come elementi di controllo nella regione non tradotta 3 'o promotori alternativa, potrebbe non essere raggiunto da questo approccio. Inoltre solo una variante di splicing singolo può essere in genere studiati con questo mezzo. Al contrario, l'uso del DNA genomico verme portato da cloni di DNA fosmid comprende probabilmente la maggior parte se non tutti gli elementi coinvolti nella regolazione genica in vivo che permette la maggiore capacità di catturare il modello autentico di espressione e la tempistica. Per facilitare la generazione di transgeni usando il DNA fosmid, ci descrivono un E. coli procedura basata recombineering per inserire GFP, un TAP-tag, o sequenze di interesse in qualsiasi posizione nel gene. La procedura utilizza il gene galK come marcatore di selezione per entrambe le fasi di selezione positiva e negativa in recombineering che si traduce ad ottenere la modifica desiderata con alta efficienza. Inoltre, plasmidi contenenti il gene galK affiancato da braccia omologia con GFP di uso comune e TAP geni di fusione sono disponibili che riducono il costo di oligo del 50% quando si genera un GFP o proteina di fusione TAP. Questi plasmidi utilizzare l'origine replica R6K che esclude la necessità di ampia purificazione dei prodotti PCR. Infine, dimostrano anche una tecnica per integrare il unc-119 marcatore al backbone fosmid che permette fosmid di essere iniettato direttamente o bombardati in vermi per generare animali transgenici. Questo video mostra le procedure coinvolte nella generazione di un transgene attraverso recombineering utilizzando questo metodo.
Protocollo
Panoramica
Transgeni molti utilizzati per la generazione di transgenico C. elegans costituiti da sequenze promotore e forse un gene cDNA clonato in uno dei vettori generati dal laboratorio del Dr. Andy Fire 1. Mentre questi transgeni hanno spesso successo per quanto riguarda la produzione di un gene reporter GFP o esprimere un cDNA in un modello desiderato, questi transgeni possono mancare i promotori alternativi, elementi enhancer, e 3 'regione non tradotta (UTR), elementi che giocano un ruolo importante nel controllo dell'espressione genica in vivo 2. Per esempio, sia il daf-12 e fah-1 geni sono importanti elementi enhancer che si trovano al di fuori del promotore prossimale che erano mancati nel promotore costruisce solo 3,4,5. Ulteriori costruisce transgene molti usano il unc-54 3'UTR che impedisce la regolamentazione da parte dei geni microRNA appropriato 6,7,8. Di conseguenza, generando transgeni con grandi segmenti di DNA genomico verme sarebbe l'ideale per catturare tutti i promotori, varianti di splicing, e 3 'UTR elementi di controllo. Recentemente un C. elegans biblioteca fosmid che consiste di circa 40 regioni kb di DNA genomico e copre quasi tutto il genoma è stato costruito. L'uso del DNA genomico verme portato da questi risultati fosmid cloni di DNA nella maggiore capacità di catturare il modello genuina espressione e la tempistica di specifici geni 2,8,9,10,11.
Tuttavia, lavorando con ampie regioni di DNA genomico pone problemi pratici come le grandi difficoltà in utilizzando le normali tecniche di biologia molecolare 12. Per superare queste limitazioni, le tecniche di modificare fosmids o cromosomi batterici artificiali mediante ricombinazione omologa in E. coli sono stati sviluppati e sono chiamati recombineering 12,13. Recombineering permette l'inserimento senza soluzione di continuità GFP, una purificazione affinità tandem (TAP)-tag, o sequenze di interesse in qualsiasi posizione nel gene trasportato dal C. elegans fosmid clone 2,10,14. Ricombinazione omologa avviene tra un prodotto di PCR affiancato da 50 regioni di omologia bp al sito di destinazione e il DNA bersaglio appositamente E. coli ceppi.
Abbiamo recentemente descritto una procedura in due fasi per la modifica di C. fosmids elegans da recombineering che consiste nell'inserire il gene galK nella posizione desiderata e quindi sostituendo questo gene con la sequenza desiderata 2. Il gene galK serve come un marcatore di selezione efficace per entrambe le fasi del processo in quanto può essere selezionata a favore e contro tramite l'uso del mezzo di crescita selettiva 15. Nella prima fase di modifica fosmid, il gene viene inserito galK tramite ricombinazione omologa nella posizione desiderata, e la fosmids correttamente modificati individuati da una selezione positiva per la capacità di utilizzare al galattosio come fonte di carbonio 2,15. Nella seconda fase, il gene galK è sostituita dalla sequenza desiderata, e la fosmids correttamente modificati vengono identificati attraverso la selezione negativa contro il gene galK attraverso l'uso dei derivati tossici deoxygalactose galattosio che uccide i batteri galK + 2,15. Un vantaggio del galK è la capacità di un singolo gene da utilizzare per le fasi di selezione positiva e negativa, invece di altri indicatori che hanno i geni separati per ogni passaggio, ed i risultati per ottenere la modifica desiderata con alta efficienza 2,15.
Per facilitare l'applicazione di questa tecnica a C. elegans ricerca, abbiamo fatto diverse modifiche alle risorse disponibili. In primo luogo, i tag GFP e TAP sono comunemente usati per generare transgeni verme, così abbiamo costruito in 50 regioni di omologia bp a ciascuno di questi tag nel pMOD4 galK-G e pMOD4 galK-GT plasmidi che servono come fonte del gene galK 2. Queste regioni permettono un unico insieme di oligonucleotidi da utilizzare per entrambe le fasi della modifica fosmid che salva la necessità di ordinare una seconda serie di oligo un po 'costoso. In secondo luogo, questi plasmidi utilizzare l'origine replica R6K che esclude la necessità di digerire il plasmide o estesa genitore purificazione dei prodotti PCR come il genitore plasmide non è in grado di replicare nei batteri utilizzati per recombineering, e può solo replicare in ceppi speciali come EC100 2 , 16 (Tabella 1 e Tabella 2). Infine, un modo comune di generare transgenici C. elegans è attraverso l'uso di bombardamenti biolistic seguita dalla selezione per i vermi transgenici attraverso il salvataggio del unc-119 mutazione 17. Per rendere il fosmids compatibile con i bombardamenti, abbiamo sviluppato il unc-119 pLoxP plasmide che può essere utilizzato per integrare il unc-119 marcatore al backbone fosmid 2.
I. Oligo Progettazione
Con recombineering le sequenze desiderato può essere inserito in qualsiasi sito all'interno del gene. Luoghi comuni sono al 5 'o 3' a seconda domini funzionali, varianti di splicing, o modificazioni post-traduzionali come scissione da proteasi. La GFP pMOD4 plasmide creato dal nostro laboratorio può essere utilizzato per inserire una FLAG-tag GFP in qualsiasi sito come il plasmide include un codone iniziatore e manca codone di stop a 3 '(Figura 1). Al contrario, il tag TAP ha versioni specifiche per 5 'e 3' fusioni a causa della scissione TEV utilizzato durante la purificazione 18,19.
- Pianificare il sito di inserimento tag all'interno del gene. Considerate promotori alternativi, domini funzionali, alternativa affettare, e modificazioni post-traduzionali se si considera il sito di inserimento. Siti di inserzione diversi potrebbe essere usato per codificare tutti, una o alcune isoforme di un gene specifico. Identificare le 50 regioni bp a monte ea valle del punto di inserimento.
- Progettare l'oligo (Tabella 1). O 100 nM scala - Gel oligo purificata o oligo Ultramer dalle tecnologie del DNA integrato può essere utilizzato per la procedura.
Per eseguire recombineering galK, è necessario progettare primer galK omologia con 50 bp in una zona che costeggia il sito desiderato da modificare e 3 'di questi primers si legano alla cassetta galK, che è presente in entrambe le pMOD4 galK-G e pMOD4 galK-GT (Figura 1). L'attaccante oligo sarà 5'------- ------- 50 bp omologia CCTGTTGACAATTAATCATCGGCA-3 'e l'inverso come 5'------- 50 omologia pb sul filamento complementare - ------ TCAGCACTGTCCTGCTCCT-3 '.
Per eseguire recombineering galK con GFP, è necessario progettare pMOD4 galK-G o pMOD4 galK-GT primer omologia con 50 bp in una zona che costeggia il sito desiderato da modificare (Figura 1). Essere sicuri di mantenere la proteina di fusione in frame. L'ATG può essere eliminata se lo si desidera. L'estremità 3 'di questi primer si legano alle regioni di omologia GFP fiancheggianti la cassetta galK. Nota: I codoni primo e l'ultimo di GFP sono sottolineate per dimostrare il quadro di lettura. L'oligo avanti sarà 5'------- ------- 50 bp omologia ATG GATTACAAGGACGATGACGATAAGATGAG -3 '3' e 5 'invertire una omologia ------- 50 pb sul complementari filamento ------- CAA AGCTTGTGGGCTTTTGTATAG-3 '
Per eseguire galK C-termine recombineering TAP, è necessario progettare pMOD4 galK-GT primer omologia con 50 bp in una zona che costeggia il sito desiderato da modificare (Figura 1). Essere sicuri di mantenere la proteina di fusione in frame. L'estremità 3 'di questi primer si legano alle regioni di omologia TAP fiancheggianti la cassetta galK. Nota: I codoni primo e l'ultimo della TAP sono sottolineate per dimostrare il quadro di lettura. L'oligo avanti sarà 5'------- ------ 50 bp omologia ATG GAAAAGAGAAGATGGAAAAAG - -3 'e invertire una omologia 5'------- 50 pb sul filamento complementare - ------ GGT TGACTTCCCCGC -3 '
Per eseguire galK N-termine recombineering TAP, è necessario progettare pMOD4 galK-GT primer omologia con 50 bp in una zona che costeggia il sito desiderato da modificare (Figura 1). Essere sicuri di mantenere la proteina di fusione in frame. L'estremità 3 'di questi primer si legano alle regioni di omologia TAP fiancheggianti la cassetta galK. Nota: I codoni primo e l'ultimo della TAP sono sottolineate per dimostrare il quadro di lettura. L'oligo avanti sarà 5'------- ------ 50 bp omologia ATG GCAGGCCTTGCGC - -3 'e invertire una omologia 5'------- 50 pb sul filamento complementare - ------ AAG TGCCCCGGAGGATGAGATTTTCT -3 ' - Generare un insieme di accompagnamento oligos per PCR nelle fasi successive così come per il sequenziamento del fosmid. Questi sono standard oligo PCR che deve legare ~ 100 bp a monte ea valle del sito di inserzione.
II. Trasferimento Fosmid per SW016 batteri
Il fosmids dalla C. elegans fosmid biblioteca sono previste nel ceppo batterico EPI300 (F-MCRA Δ (MRR-hsdRMS-mcrBC) φ80dlacZΔM15 ΔlacX74 recA1 endA1 araD139 Δ (ara, leu) 7697 Galu galK λ-RPSL nupG trfA tona) (Biotecnologie Epicentre, Madison, WI ) che permette l'espressione fosmid essere aumentata di sopra di una sola copia per cella per migliorare la resa durante la purificazione del DNA (Tabella 2). Per recombineering, la fosmid dovranno essere trasferiti al ceppo batterico SW106 (MCRA Δ (MRR-hsdRMS-mcrBC) ΔlacX74 Deor endA1 araD139 Δ (ara, leu) 7697 RPSL recA1 nupG φ80dlacZΔM15 [λc1857 (cro-bioA) <> Tet ] (cro-bioA) <> AraC-PBAD Cre ΔgalK) (NCI-Frederick) ceppo che porta il λred ricombinazione omologa geni sotto il controllo di un λ sensibile alla temperatura e un repressore inducibile arabinosio cre ricombinasi (Tabella 2) 15.
- Ordina il clone fosmid di Gene di interesse (GOI) da GeneService (Cambridge, UK) utilizzando Wormbase come guida. Quando si selezionano i cloni, abbiamo scelto quelle che hanno il governo indiano nel centro della sequenza. Quelle che escludono i geni vicini potrebbe essere preferibile, ma potrebbe essere difficile da trovare.
- Cultura il clone fosmid del GOI in LB contenente 12,5 mg / ml cloramfenicolo a 37 ° C.
- Crescere una cultura di 1,5 ml durante la notte del fosmid, e mini-preparazione del DNA fosmid Epicentre fosmid utilizzando il kit di preparazione (Biotecnologie Epicentre, Madison, WI). Seguiamo il protocollo alternativo descritto nelle istruzioni che prevede di aggiungere il mix Riboshredder in un passaggio precedente.
- Determinare la concentrazione fosmid DNA con uno spettrofotometro.
- Preparare il electrocompetent SW106 cellule in crescita di 5 ml di cultura durante la notte in un SW106 14 mL snap-tappo della provetta con brodo LB con 12,5 mcg / mL cloramfenicolo a 32 ° C.
- Seminare 1 ml in 100 ml di LB con cloramfenicolo in un pallone 2 L. SW106 crescere i batteri in un OD 600 0,6-0,8. NON heat-shock.
- Pellet di centrifugazione a 5000xg per 5 minuti, risospendere il pellet vortexando gentilmente, e aggiungere 50 ml ghiacciata glicerolo 10%. Ripetere questa fase di lavaggio una volta.
- Agglomerare le SW106 per centrifugazione ed aspirare tutti, ma ~ 500 microlitri di ciascun supernatante
- Risospendere il pellet vortexando gentilmente. Congelamento aliquote di 100 microlitri in azoto liquido o ghiaccio secco, e conservare a -80 ° C per un uso futuro.
- Trasformare il DNA in fosmid electrocompetent SW106 cellule electroporating i batteri con ~ 50 ng di DNA fosmid utilizzando un Eppendorf 2510 elettroporatore a 1350 volt in cuvette centimetri 0,1 gap.
- Recuperare i batteri in 1 LB mL per 1 ora a 32 ° C.
- Aliquote piastra su piastre LB con cloramfenicolo (12,5 mg / ml) e incubare a 32 ° C durante la notte.
- Verificare la presenza del governo indiano di colonia PCR. Crescere una cultura 5 ml pernottamento in LB con 12,5 mcg / mL cloramfenicolo a 32 ° C. Aggiungere 0,5 l di cultura per una reazione standard di PCR con l'oligo di accompagnamento, e aumentare l'iniziale 95 ° C di incubazione a 5 minuti per lisare i batteri prima di PCR.
- Preparare uno stock di glicerolo per la conservazione a lungo termine.
III. L'inserimento del gene galK da recombineering
Nella prima fase di modifica fosmid, galK il gene viene inserito nel fosmid per ricombinazione omologa, e la fosmids correttamente modificati vengono selezionati dalla crescita sul terreno minimo contenente galattosio come unica fonte di carbonio (Figura 2A). Il SW106 batteri crescono lentamente sul terreno minimo e 3-5 giorni per vedere colonie.
- Preparare piatti MOPS minimo supporti contenenti 0,2% galattosio. Mezzi di MOPS minimo è disponibile da Teknova Inc. (Hollister, CA) (catalogo # M2106), ma non utilizzare il glucosio incluso.
Mezzi di MOPS minimo con lo 0,2% galattosio (1 L)
Autoclave 15 grammi di agar in 870 ml di acqua
Raffreddare a 55 ° C e aggiungere:100 ml MOPS 10X terreno minimo 5 ml 0,2 mg / ml d-biotina (sterile filtrata) 4,5 ml 10 mg / ml L-leucina (1%, riscaldata, poi raffreddata e filtrata sterile) 10 mL 20% di galattosio (autoclave) 1 ml 12,5 mg / ml di cloramfenicolo in EtOH 2,55 ml 20% di NH 4 Cl 10 mL 0,132 M di potassio fosfato bibasico - PCR amplifica la pMOD4 galK-G o pMOD4 galK GT-cassette con i primer disegnato sopra. Abbiamo utilizzato Phusion (New England Biolabs, Ipswich, MA) o GoTaq (Promega, Madison, WI).
- Gel purificare la banda risultante. Quantificare il rendimento di gel o spettrofotometro NanoDrop. Questo prodotto PCR è pronto per il passo 3.14.
- Inoculare una cultura della notte SW106 cellule contenenti il DNA fosmid in 5 ml di LB con cloramfenicolo (12,5 mcg / mL). Crescere a 32 ° C.
- Impostare scuotendo bagnomaria a 42 ° C a warm-up con una sterile matraccio da 250 ml nel supporto. L'uso di un bagno d'acqua agitazione è fondamentale per ottenere un elevato rendimento.
- Aggiungere 1 ml di cultura durante la notte a 100 ml di LB e cloramfenicolo in un pallone 2 L. Crescere ad un OD 0,6-0,8. Questo richiede di solito 3-4 ore.
- Trasferire 50 ml di cellule SW106 nel matraccio da 250 ml e shock termico a 42 ° C per esattamente 20 minuti. a bagnomaria agitazione a 100 rpm Lascia i batteri rimasti a 32 ° C come il controllo uninduced.
- Raffreddare i batteri indotti e uninduced in ghiaccio per 10 minuti.
- Trasferire i campioni a due provette da centrifuga sterili e pellet a ~ 5000xg per 5 minuti.
- Eliminare tutti i supernatante e risospendere il sedimento in 1 ml ghiacciata glicerolo 10% vortexando gentilmente (ossia regolazione 3-4).
- Quando risospeso, aggiungere un altro 49 ml ghiacciata glicerolo 10%, e il pellet campioni a ~ 5000xg per 5 minuti.
- Ripetere il punto 3.9, 3.10, e 3.11 di nuovo.
- Rimuovere tutti i sopranatante invertendo i tubi, e risospendere il pellet nel liquido rimanente (circa 500 microlitri ciascuno). Aliquota in 100 microlitri campioni, congelare il ghiaccio secco, e conservare a -80 ° C. Questi sono buoni per settimane o mesi. (Di solito fermarsi qui ed eseguire l'elettroporazione del giorno successivo).
- Electroporate l'indotto e uninduced SW106 cellule con 150 ng di prodotto PCR utilizzando cuvette gap 0,1 cm in un insieme elettroporatore Eppendorf 2510 a 1350 volt.
- Recuperare i batteri in 1 LB mL in un tubo 14 ml Falcon. Incubare a 32 ° C per 4,5 ore.
- Pellet i batteri in una microcentrifuga a 13.200 rpm per 15 secondi. I batteri vengono risospesi in M9 e quindi lavato due volte rimuovere qualsiasi mezzo ricco (vedi sotto per ricetta).
- M9 media (1 L)
- 6g Na 2 HPO 4
- 3g KH 2 PO 4
- 1 g di NH 4 Cl
- 0,5 g di NaCl
- AUTOCLAVE
- Dopo il secondo lavaggio, il sopranatante viene rimosso e il pellet viene risospeso in 1 ml M9 prima della placcatura diluizioni seriali in M9 (100 l, 100 ml di una diluizione 1:10, e 100 l 1:100) su supporti MOPS minimo.
- Incubare 3-5 giorni a 32 ° C in un incubatore. Nota: Sii paziente come veri positivi crescono lentamente.
- Streak alcune colonie su piastre di agar indicatore MacConkey (BD # 281810) integrata con 1% di galattosio e cloramfenicolo 12,5 mcg / ml. Tutte le colonie che appare dopo l'ultimo passaggio dovrebbe essere + galK, ma al fine di sbarazzarsi di qualsiasi galK - contaminanti, è importante ottenere singolo, colonie rosa prima di procedere alla seconda fase. Il galK - colonie sarà bianco / incolore e batteri + galK sarà di colore rosso / rosa a causa di un cambiamento di pH risultante dalla fermentazione galattosio dopo una notte di incubazione a 32 ° C.
- Scegli una singola colonia e inoculare in 5 ml di LB + cloramfenicolo cultura durante la notte per la crescita a 32 ° C.
- Confermare l'inserimento del gene galK nella posizione corretta tramite PCR utilizzando i oligo accompagnamento. Aggiungere 0,5 microlitri della cultura ad una reazione standard di PCR e aumentare l'iniziale 95 ° C di incubazione a 5 minuti per lisare i batteri. Il prodotto PCR dovrebbe essere upshifted di dimensioni a causa della presenza del gene galK.
- Preparare uno stock di glicerolo per la conservazione.
IV. Sostituzione di galK con sequenze da tag recombineering
In questa fase il gene galK è sostituito dal sequenze di tag desiderata e la fosmids correttamente modificati vengono selezionati dalla selezione contro il gene galK dal tossici galattosio analogico deoxygalactose (DOG) (Figura 2B).
- Preparare piatti MOPS minimo supporti contenenti 0,2% deoxygalactose (DOG) e 0,2% glicerolo. Mezzi di MOPS minimo è disponibile da Teknova Inc. (Hollister, CA) (catalogo # M2106), ma non utilizzare il glucosio incluso.
Mezzi di MOPS minimo con lo 0,2% DOG e glicerolo (1 L)
Autoclave 15 grammi di agar in 860 ml di acqua
Raffreddare a 55 ° C e aggiungere:100 ml MOPS 10X terreno minimo 5 ml 0,2 mg / ml d-biotina (sterile filtrata) 4,5 ml 10 mg / ml L-leucina (1%, riscaldata, poi raffreddata e filtrata sterile) 10 mL 20% deoxygalactose (sterile filtrata) 10 mL 20% di glicerolo (autoclave) 1 ml 12,5 mg / ml di cloramfenicolo in EtOH 2,55 ml 20% di NH 4 Cl 10 mL 0,132 M di potassio fosfato bibasico - PCR amplifica i frammenti tag da pMOD4 GFP, pBS1761 (N-termine TAP), o pBS1479 (C-termine TAP) utilizzando la oligo stesso utilizzato nel primo turno o utilizzando più breve GFP o TAP-specifici oligonucleotidi (le sequenze interne al passo 1,2 ). Se si stanno facendo i costrutti più, è particolarmente utile per utilizzare il oligo più breve come lo stesso prodotto della PCR può essere utilizzato per tutti i costrutti.
- Gel purificare il prodotto di PCR e misurare la concentrazione tramite gel o spettrofotometria.
- Generare indotto e uninduced SW106 competente che il fosmid con il gene inserito galK seguenti passaggi 3,4-3,13 sopra.
- Electroporate l'indotto e uninduced SW106 cellule con ~ 100 ng di prodotto PCR utilizzando cuvette gap 0,1 cm in un insieme elettroporatore Eppendorf 2510 a 1350 volt.
- Recuperare in 1 LB ml in 14 ml snap-tappo della provetta ed incubare a 32 ° C shaker per 4,5 ore.
- Lavare e diluire i batteri, come nei passi 3.16 e 3.17. Batteri su piastra MOPS piastre minimo supporti contenenti 0,2% 2-deossi-galattosio (DOG) e 0,2% glicerolo.
- Incubare a 32 ° C per tre giorni.
- Quattro colonie sono usati per fare 5 mlculture pernottamento in LB con 12,5 mcg / mL cloramfenicolo. Questi sono usati per colonia PCR come sopra per confermare che la cassetta è stata inserita. Noi usiamo sia il più breve GFP / TAP oligo specifici e la oligos di accompagnamento per dimostrare l'inserto giusto e il sito giusto. GFP è ~ 800 bp e TAP è ~ 550 bp, mentre galK è di 1,4 kb.
- Preparare uno stock di glicerolo.
Aggiunta V. unc-119 da Gene crea-loxP ricombinazione
Un modo comune di generare animali transgenici con il fosmids modificato è attraverso l'uso di bombardamento biolistic. Questa tecnica utilizza rivestite di DNA particelle d'oro per introdurre DNA fosmid in C. elegans. Animali transgenici sono in genere identificate tramite salvataggio del unc-119 mutante con un unc-119 transgene. In questa fase, il gene unc-119 si aggiunge alla spina dorsale fosmid in cis da crea-loxP ricombinazione con l'pLoxP unc-119 plasmide (Figura 2C).
- Preparare competente SW106 batteri portando la fosmid modificato dal punto 4.9 Uso passi 2,5-2,9. NON indurre a 42 ° C.
- Electroporate con 50 ng. pLoxP unc-119 da un mini-prep utilizzando cuvette gap 0,1 cm in elettroporatore Eppendorf 2510 impostato a 1350 volt.
- Recupera batteri in LB contenente 0,1% arabinosio per 1 ora a 32 ° C.
- Aliquote piastra su piastre LB contenente ampicillina 50μg/mL e 12,5 mcg / mL cloramfenicolo. Incubare a 32 ° C durante la notte. che seleziona per l'integrazione di pLoxP unc-119 nel fosmid.
- Crescere una cultura pernottamento in LB contenente ampicillina 50μg/mL e 12,5 mg / ml cloramfenicolo. Utilizzate da 0,5 microlitri per PCR verificare la presenza del gene unc-119 con l'unc-119 F (5'-CAAATCCGTGACCTCGACAC-3 ') e unc-119 R (5'-CACAGTTGTTTCTCGAATTTGG-3') oligos (Tabella 1).
- Fai uno stock di glicerolo del fosmid finale.
VI. Grande scala Preparazione Fosmid
Per facilitare l'ottenimento le maggiori quantità di DNA fosmid necessari per il bombardamento, in questa fase il fosmid è trasferito al EPI300 batteri. Questa varietà ha la capacità di aumentare il numero fosmid copia per aumentare la resa durante la preparazione del DNA.
- Crescere una cultura 5 ml durante la notte dei batteri dal punto 5.5 in LB contenente ampicillina e cloramfenicolo a 32 ° C. Usa Epicentre fosmid kit di preparazione per isolare il fosmid da 1,5 ml di coltura.
- Electroporate ~ 50 ng nel EPI300 batteri utilizzando cuvette gap 0,1 cm in un insieme elettroporatore Eppendorf 2510 a 1350 volt. Il EPI300 batteri possono essere acquistati presso Biotecnologie Epicentre (Madison, WI).
- Recupera batteri in LB per 1 ora a 37 ° C. Aliquote piastra su agar LB contenente 50 mg / ml e 12,5 mcg / mL cloramfenicolo.
- Crescere e di indurre la EPI300 batteri che contiene il fosmid modificati utilizzando le istruzioni incluse. Una cultura da 50 ml indotto darà> 10 mg di DNA purificato fosmid. Purificare il fosmid con il fosmid Epicentre preparazione kit.
VII. Bombardamento
- Uso 10 mcg. del DNA fosmid per bombardare la DP38 ceppo worm come descritto (D. Hochbaum, A. Ferguson, e A. Fisher, Giove, in stampa).
VIII. Rappresentante Risultati
La modifica del fosmids via recombineering robusta e il tasso di successo di> 90% nella fase di selezione negativa sono regolarmente osservati 2. Questo protocollo prende anche ~ 2 settimane per completare il che rende la preparazione dei transgeni abbastanza rapido. Il protocollo è stato provato da altri laboratori con successo 20.
| Oligo | Sequenza |
| C-TAP termine F | ATGGAAAAGAGAAGATGGAAAAAG |
| C-TAP termine R | GGTTGACTTCCCCGC |
| BANDIERA GFP-F | ATGGATTACAAGGACGATGACGATAAGATGAG |
| FLAG-GFP R | CAAAGCTTGTGGGCTTTTGTATAG |
| N-TAP termine F | ATGGCAGGCCTTGCGC |
| N-TAP termine R | AAGTGCCCCGGAGGATGAGATTTTCT |
| galK F | CCTGTTGACAATTAATCATCGGCA |
| galK R | TCAGCACTGTCCTGCTCCT |
| unc-119 F | CAAATCCGTGACCTCGACAC |
| unc-119 R | CACAGTTGTTTCTCGAATTTGG |
Tabella 1. Oligonucleotidi utilizzati per la PCR.
| Plasmidi | Fonte | Disponibile all'indirizzo |
| Fosmid clone | Geneservice Ltd. | Geneservice |
| pGalK | 15 | NCI-Frederick |
| pMOD4-RT-G | 2 | Addgene |
| pMOD4-galK-G | ||
| pMOD4-galK-GT | ||
| pLoxP-unc-119 | ||
| pMOD4-GFP | ||
| Batteri | ||
| SW106 | 15 | NCI-Frederick |
| EPI300 | Epicentre Biotecnologie | Epicentre |
| EC100D pir-116 | ||
Tabella 2. Strain e disponibilità vettore.
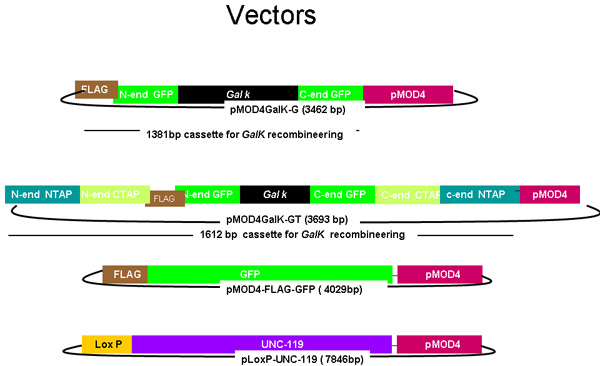
Figura 1.
Schema di pMOD4-galK-G, e pMOD4-galK-GT, pMOD4 GFP, pLoxP-unc-119
Il pMOD4-galk-G plasmide consiste nella cassetta galK (nero), affiancato da 50 regioni nucleotide identico al 5 'e 3' estremità della FLAG (Brown)-GFP (verde), mentre PMOD 4-galK-GT è costituito dai galK cassetta affiancato da entrambi i FLAG-GFP regioni omologia nucleotidica e 50 regioni identico al 5 'e 3' estremità N-terminale e C-terminale TAP (blu e arancio, rispettivamente). pMOD4-FLAG-GFP consiste nella cassetta GFP completa con un 5 'FLAG tag e pLoxP unc-119 consiste nella unc-119 sequenza genomica (viola) in un plasmide contenente un sito loxP. Tutti i plasmidi utilizzare il R6K basato pMOD4 (rosso), spina dorsale, che è in grado di replicare in SW106.
Figura 2.
Panoramica del processo recombineering galK
Figura 2A-2C cifre distinte che mostrano le fasi e tempi necessari per recombineering si utilizza il cassetto galK. Queste sono le stesse figure che si fondono nella figura 2d, ma separatamente per chiarezza e facilità di lettura. Un fosmid di interesse viene prima modificato in due fasi che coinvolgono l'inserimento della cassetta galK affiancato da 50 regioni bp di omologia di FLAG-GFP o TAP (Figura 2A), seguita dalla sostituzione di questa cassetta di FLAG-GFP o TAP ( Figura 2B). Più tardi la unc-119 marker per l'impiego nella generazione di animali transgenici è inserito nel sito loxP sulla dorsale fosmid (Figura 2C).
Figura 2D mostra una figura fusa di procedura recombineering galK.
Schema di procedura recombineering galK come descritto nel tempo sopra anche richiesto per ogni passo dalla fusione Figura 2A-2C.
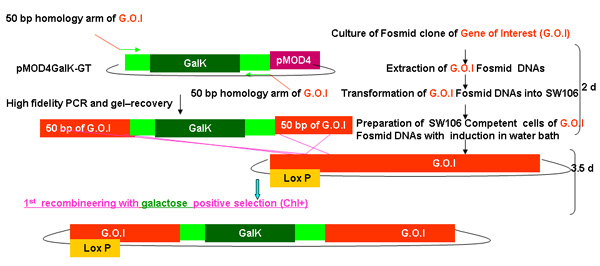
Figura 2a. L'inserimento galk in recombineering galk.
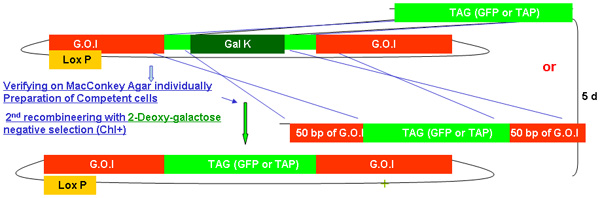
Figura 2b. L'(GFP / TAP) inserimento nel TAG recombineering galk.
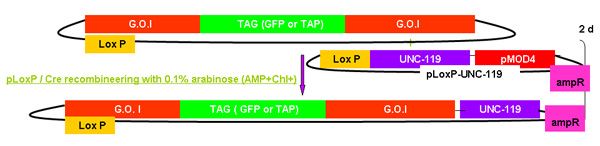
Figura 2c. L'aggiunta di unc-119.
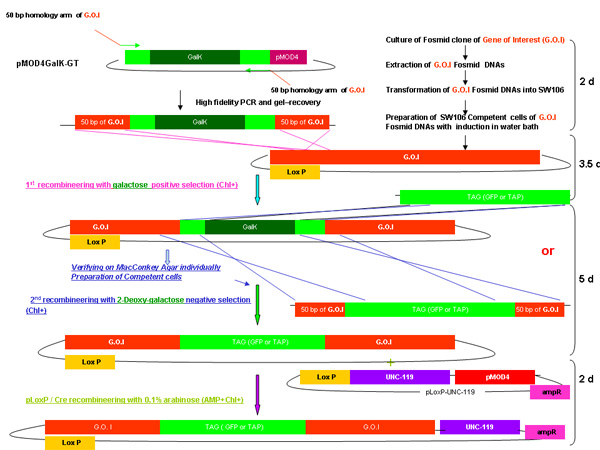
Figura 2d. La panoramica fusa di recombineering galk.
Discussione
La generazione di transgeni da fosmids offre il vantaggio di mantenere tutti gli elementi promotore nativo, varianti di splicing, e 3 'UTR elementi di regolamentazione. Questo può portare alla costruzione di un transgene che è più riflettente del pattern di espressione nativa, o la costruzione di un transgene funzionale quando altri approcci non 5. I transgeni risultante può trasportare una varietà di etichette tra cui epitopo GFP o un tag TAP.
La costruzione di transgeni...
Divulgazioni
Riconoscimenti
Gli autori desiderano ringraziare Lindsey Nash aiuto per sviluppare la tecnica. Questo lavoro è stato finanziato dal NIH concedere AG028977 di ALF, una borsa di progetto pilota presso l'Università di Pittsburgh OAIC (AG024827), e fondi presso l'Università di Pittsburgh.
Materiali
| Name | Company | Catalog Number | Comments |
| FosmidMAX kit | Epicentre Biotechnologies | FMAX046 | |
| GoTaq | Promega Corp. | M7122 | |
| MOPS Media | TEKnova, Inc. | M2120 | |
| 0.132 M Potassium phosphate solution | TEKnova, Inc. | M2102 | |
| D-galactose | Sigma-Aldrich | G0750 | |
| 2-deoxygalactose | Sigma-Aldrich | D4407 | |
| Biotin | Sigma-Aldrich | B4639 | |
| Leucine | Sigma-Aldrich | L8000 | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Phusion DNA polymerase | New England Biolabs | F-530S | |
| MacConkey agar base | BD Biosciences | 281810 | |
| Arabinose | Sigma-Aldrich | A3131 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S5136 | |
| Potassium phosphate monobasic | Sigma-Aldrich | P5655 | |
| Sodium chloride | Sigma-Aldrich | S5886 | |
| Glycerol | Sigma-Aldrich | G2025 | |
| Bacto Agar | BD Biosciences | 214010 |
Riferimenti
- Mello, C., Fire, A. DNA transformation. Methods Cell Biol. 48, 451-482 (1995).
- Zhang, Y., Nash, L., Fisher, A. L. A simplified, robust, and streamlined procedure for the production of C. elegans transgenes via recombineering. BMC Dev Biol. 8, 119-119 (2008).
- Antebi, A., Yeh, W. H., Tait, D., Hedgecock, E. M., Riddle, D. L. daf-12 encodes a nuclear receptor that regulates the dauer diapause and developmental age in C. elegans. Genes and Development. 14, 1512-1527 (2000).
- Snow, M. I., Larsen, P. L. Structure and expression of daf-12: a nuclear hormone receptor with three isoforms that are involved in development and aging in Caenorhabditis elegans. Biochim. Biophys. Acta. 1494, 104-116 (2000).
- Fisher, A. L., Page, K. E., Lithgow, G. J., Nash, L. The Caenorhabditis elegans K10C2.4 Gene Encodes a Member of the Fumarylacetoacetate Hydrolase Family. A CAENORHABDITIS ELEGANS MODEL OF TYPE I TYROSINEMIA. J Biol.Chem. 283, 9127-9135 (2008).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75, 855-862 (1993).
- Lehrbach, N. J. LIN-28 and the poly(U) polymerase PUP-2 regulate let-7 microRNA processing in Caenorhabditis elegans. Nat Struct Mol Biol. 16, 1016-1020 (2009).
- Tursun, B., Cochella, L., Carrera, I., Hobert, O. A toolkit and robust pipeline for the generation of fosmid-based reporter genes in C. elegans. PLoS One. 4, e4625-e4625 (2009).
- Bamps, S., Hope, I. A. Large-scale gene expression pattern analysis, in situ, in Caenorhabditis elegans. Brief. Funct. Genomic. Proteomic. , (2008).
- Dolphin, C. T., Hope, I. A. Caenorhabditis elegans reporter fusion genes generated by seamless modification of large genomic DNA clones. Nucleic Acids Res. 34, e72-e72 (2006).
- Sarov, M. A recombineering pipeline for functional genomics applied to Caenorhabditis elegans. Nat. Methods. 3, 839-844 (2006).
- Yang, X. W., Model, P., Heintz, N. Homologous recombination based modification in Escherichia coli and germline transmission in transgenic mice of a bacterial artificial chromosome. Nat Biotechnol. 15, 859-865 (1997).
- Court, D. L., Sawitzke, J. A., Thomason, L. C. Genetic engineering using homologous recombination. Annu.Rev.Genet. 36, 361-388 (2002).
- Westenberg, M., Bamps, S., Soedling, H., Hope, I. A., Dolphin, C. T. Escherichia coli MW005: lambda Red-mediated recombineering and copy-number induction of oriV-equipped constructs in a single host. BMC Biotechnol. 10, 27-27 (2010).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36-e36 (2005).
- Penfold, R. J., Pemberton, J. M. An improved suicide vector for construction of chromosomal insertion mutations in bacteria. Gene. 118, 145-146 (1992).
- Praitis, V., Casey, E., Collar, D., Austin, J. Creation of low-copy integrated transgenic lines in Caenorhabditis elegans. Genetics. 157, 1217-1226 (2001).
- Puig, O. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Rigaut, G. A generic protein purification method for protein complex characterization and proteome exploration. Nat.Biotechnol. 17, 1030-1032 (1999).
- Achilleos, A., Wehman, A. M., Nance, J. PAR-3 mediates the initial clustering and apical localization of junction and polarity proteins during C. elegans intestinal epithelial cell polarization. Development. 137, 1833-1842 (2010).
- Maduro, M., Pilgrim, D. Identification and cloning of unc-119, a gene expressed in the Caenorhabditis elegans nervous system. Genetics. 141, 977-988 (1995).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon